表1 用于质粒构建的引物
Table 1 Primers used for plasmid construction

引物名称序列(5′-3′)purC-FCATGCCATGGGGGTGGAAAAAGAAACGCTGCTTTpurC-RCCAAGCTTTTATTTTGTCTTTTCCAATAGATTTAAAAC
乳酸菌可发酵糖类并产生以乳酸为主的代谢产物,其属于厌氧或兼性厌氧的革兰氏阳性菌[1],大多数乳酸菌作为重要的工业微生物,广泛应用于医药、食品、精细化工等领域中[2-4]。乳酸菌在上述应用中会面临多种环境胁迫,比如酸、冷冻、渗透压、氧等[5],酸胁迫是其中影响乳酸菌存活能力最严重的挑战之一。乳酸的过量积累使乳酸菌遭受严重的酸胁迫,影响菌株的正常生长和代谢,从而进一步抑制乳酸菌益生功能的发挥,因此,提高乳酸菌抵抗酸胁迫的能力尤为迫切[6-7]。
目前,提高乳酸菌酸胁迫抗性的策略主要有预适应及交叉保护[8-9]、适应性进化[10-11]、外源添加保护剂[12]和代谢工程改造抗酸元器件[13-14]等。其中抗酸元器件包括一些具有抗酸功能的关键基因、应激蛋白和代谢途径等。酸胁迫条件下,乳酸乳球菌可以通过柠檬酸代谢生成乙偶姻、丁二醇和乙二酰等,这些产物能够帮助细胞抵御酸胁迫[15]。氨基酸是维持细胞正常生长代谢的重要原料物质之一,其在乳酸菌的生长代谢和抵抗酸胁迫中,发挥着重要的作用。外源添加天冬氨酸可有效提高乳酸乳球菌和干酪乳杆菌对酸胁迫的耐受性[16-17],张梦汝等[18]通过添加亮氨酸提高了乳酸乳球菌在pH 4.0酸胁迫环境中的存活率,研究表明,酸胁迫条件下乳酸菌可以通过调节氨基酸如天冬氨酸和亮氨酸的代谢方式提高细胞酸耐受性能。目前,关于乳酸菌中酸胁迫耐受性提升的研究主要集中于氨基酸代谢途径,而关于其他类型代谢途径在帮助乳酸菌细胞提升酸胁迫耐受性方面的研究相对较少。嘌呤核苷酸对能量代谢和各种合成代谢过程如DNA、RNA和蛋白质合成有着至关重要的作用[19],调控嘌呤代谢可在一定程度缓解非生物胁迫的侵扰[20]。目前,有关嘌呤代谢途径在改善乳酸乳球菌酸胁迫抗性方面的研究还非常有限,仅有的研究发现,嘌呤代谢中的PurA和GuaB催化转化腺苷和鸟苷一磷酸(AMP和GMP)有利于乳酸乳球菌抵御氧胁迫和酸胁迫[21-22]。
编码磷酸核糖基氨基咪唑-琥珀酰胺合酶的purC是IMP生物合成途径的关键基因,本身也是嘌呤代谢的一部分,该基因能够催化转化CAIR(羧酰氨基咪唑核糖核苷酸)生成SAICAR(琥珀酰氨基咪唑甲酰胺核苷酸)。研究发现,purC基因沉默会引起嘌呤代谢水平改变,产生嘌呤代谢产物的缺陷可导致细菌生长延缓[23]。本文通过在L.lactis NZ9000中过表达嘌呤代谢途径中的purC基因,考察了重组菌株生长能力和存活能力,通过检测胞内ATP和氨基酸含量,研究重组菌株耐受酸胁迫的具体生理响应机制。
1.1.1 菌株及培养条件
大肠杆菌E.coli MC1061,实验室保藏;乳酸乳球菌L.lactis NZ9000 (保藏编号:LLN,CP002094;荷兰“NIZO Food Research”奶制品研究所);质粒pNZ8148,德国MoBiTec公司;E.coli MC1061在LB培养基(10 g/L蛋白胨、5 g/L酵母粉、10 g/L NaCl)中37 ℃,220 r/min振荡培养;L.lactis NZ9000在GM17培养基(M17培养基中添加5 g/L的葡萄糖)于30 ℃恒温培养箱静置培养。
1.1.2 试剂
蛋白胨、酵母粉和M17肉汤培养基,Oxoid公司;NcoI、HindⅢ快速内切酶和GeneJET PCR纯化试剂盒,Thermo Scientific公司;Solution I、PrimerSTAR HS (Premix),大连宝生物工程有限公司;SanPrep柱式质粒小量抽提试剂盒,生工生物工程(上海)有限公司;nisin,Sigma公司;BCA蛋白定量试剂盒,天根生化科技有限公司;ATP测定试剂盒,碧云天生物技术研究所;其他化学试剂均为国产分析纯。
恒温培养箱,上海医疗器械研究所;组织和细胞匀浆仪,MP Biomedicals公司;M-100生物传感器分析仪,深圳市西尔曼科技有限公司;Agilent 1260高效液相色谱仪,安捷伦科技(中国)有限公司;多功能酶标仪,Thermo Scientific公司;凝胶成像仪、电转化仪,Bio-Rad公司。
1.3.1 目的片段的扩增及纯化
根据NCBI数据库公布的磷酸核糖基氨基咪唑-琥珀酰胺合成酶PurC基因序列并利用Primer Primier 5.0设计引物purC-F和purC-R(表1),上、下游引物的5′端分别加入NcoI和HindⅢ两个酶切位点(引物中划线部分)。
表1 用于质粒构建的引物
Table 1 Primers used for plasmid construction

引物名称序列(5′-3′)purC-FCATGCCATGGGGGTGGAAAAAGAAACGCTGCTTTpurC-RCCAAGCTTTTATTTTGTCTTTTCCAATAGATTTAAAAC
以L.lactis NZ9000的基因组为模板,通过PCR扩增获得purC基因。PCR反应体系:2×Prime STAR HS (Premix) 25 μL,purC-F和purC-R (10 μmol/L)各1 μL,L.lactis NZ9000的基因组模板1 μL,无菌水22 μL。反应条件:95 ℃、3 min;95 ℃、10 s;55 ℃、15 s; 72 ℃、1 min,30个循环,使用10 g/L的琼脂糖凝胶核酸电泳初步确定,并进行胶回收获得目的基因片段。
1.3.2 表达载体的构建
将PCR产物以及质粒pNZ8148采用限制性内切酶NcoI和HindⅢ 同时进行双酶切,并纯化回收。将酶切的PCR产物以及pNZ8148使用Solution I连接酶16 ℃ 过夜连接。连接产物通过热激法转入E.coli MC1061感受态中,转化后的重组子涂布于含有100 μg/mL 氯霉素的LB固体培养基中,37 ℃培养12 h。挑取氯霉素平板上的转化子进行菌落PCR验证。将获得的菌体进行培养,提取pNZ8148-purC重组质粒,进行双酶切验证,测序验证。通过电转化的方式将测序成功的重组质粒和空质粒转入L.lactis NZ9000,并通过菌落PCR验证和10 μg/mL的氯霉素抗性筛选获得重组菌株L.lactis NZ9000 (pNZ8148-purC)和对照菌株L.lactis NZ9000 (pNZ8148)。
1.3.3 重组菌株生长曲线的测定和乳酸发酵实验
将菌株L.lactis NZ9000 (pNZ8148-purC)和L.lactis NZ9000 (pNZ8148)接种于加了10 μg/mL氯霉素的GM17液体培养基中进行活化,放在30 ℃培养箱中静置培养过夜。再以2%的接种量将种子液转接至含有氯霉素(10 μg/mL)的GM17液体培养基中,30 ℃静置培养。每隔一定时间取样,培养至OD600=0.4时加入10 ng/mL的nisin诱导表达PurC蛋白,测定OD600 nm值,使用生物传感器分析仪测定各个时间点的葡萄糖质量浓度和乳酸产量。以时间为横坐标,OD600值为纵坐标,绘制生长曲线。以时间为横坐标,葡萄糖质量浓度和乳酸产量为纵坐标,绘制重组菌株和对照菌株的乳酸发酵曲线。
1.3.4 胁迫条件下重组菌株酸胁迫抗性的分析
在含有10 μg/mL氯霉素的GM17培养基中培养重组菌株和对照菌株至OD600约为0.4,加入10 ng/mL的nisin诱导蛋白表达,待菌株生长至对数中后期,离心(8 000 r/min)3 min,使用8.5 g/L生理盐水等体积洗涤2次、离心、收集菌体,重悬于等体积的GM17培养基中(乳酸调节,pH 4.0)。随后于0、1、2、3、4 h各取900 μL菌液,离心,等体积洗涤并进行梯度稀释,取10 μL合适梯度的重悬液点种于GM17固体平板上,30 ℃下培养24 h,活菌计数并计算存活率[24]。
1.3.5 胁迫条件下重组菌株胞内ATP和氨基酸含量的测定
nisin诱导蛋白表达后,重组菌株和对照菌株生长至对数中后期,离心(8 000 r/min)4 min,使用8.5 g/L 生理盐水等体积洗涤2次、离心、收集菌体,重悬于等体积的GM17培养基中(pH 4.0),胁迫一定时间取样,收集菌体,破碎细胞,取细胞破碎液测定胞内ATP和氨基酸。选用ATP测定试剂盒测定胞内ATP,高效液相色谱仪测定胞内氨基酸[25]。
1.3.6 蛋白浓度的测定
蛋白浓度测定采用BCA蛋白浓度测定试剂盒,具体按说明书操作。
以L.lactis NZ9000基因组为模板,利用引物purC-F和purC-R进行目的片段的扩增,得到目的PCR产物,核酸电泳结果显示,目的基因条带单一,与理论711 bp大小符合(图1)。

图1 目的基因purC的PCR扩增
Fig.1 PCR amplification of genepurC
按照1.3.2的方式构建重组质粒pNZ8148-purC,结果如图2所示。将上述获得的重组质粒导入到E.coli MC1061的感受态细胞中,挑取经菌落PCR验证的阳性克隆子培养并提取质粒,采用NcoI和HindⅢ进行双酶切验证,核酸电泳结果显示,目标条带在500~750 bp,与已知目的片段大小711 bp相符合,其双酶切验证结果如图3所示。将验证成功的重组质粒进行测序验证,并电转入感受态L.lactis NZ9000,获得重组菌株L.lactis NZ9000 (pNZ8148-purC)。
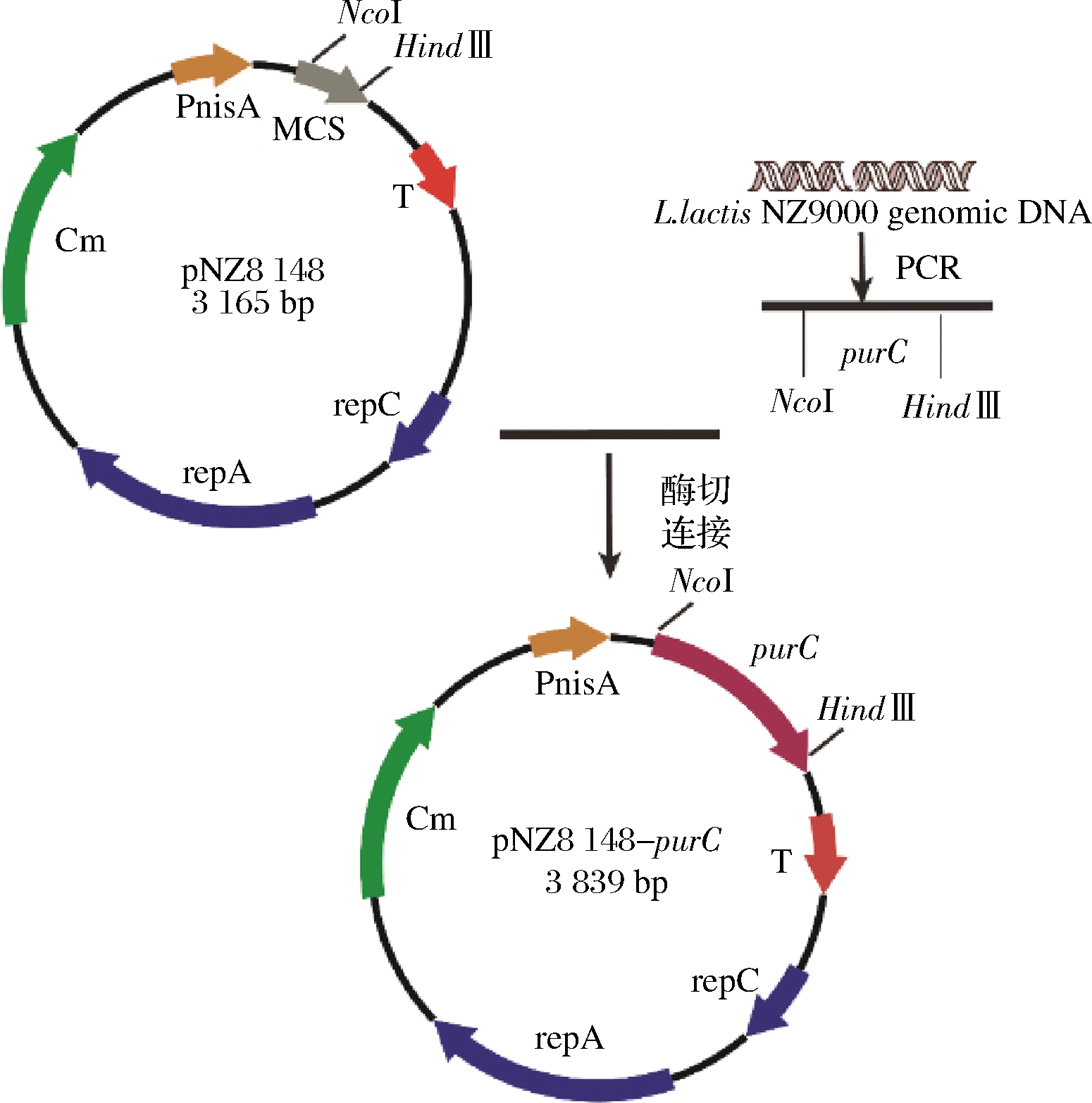
图2 重组质粒pNZ8148-purC的构建
Fig.2 Construction of recombinant plasmid of pNZ8148-purC
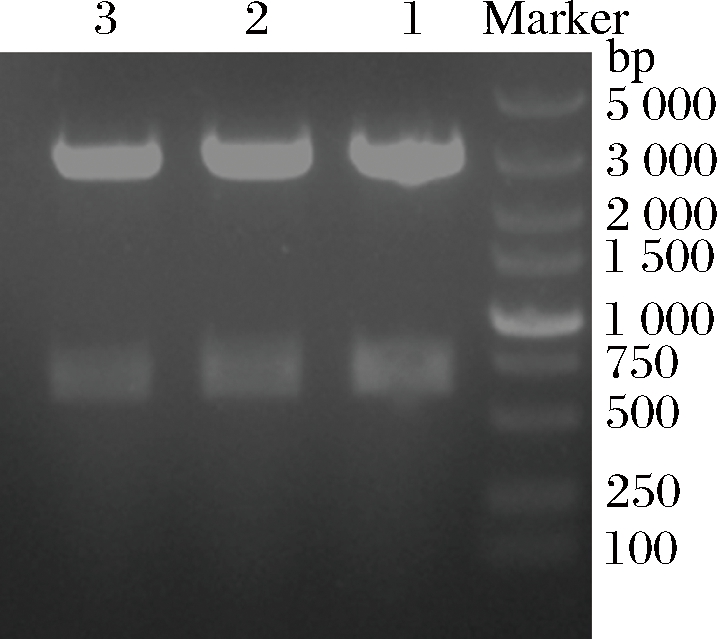
图3 重组质粒pNZ8148-purC双酶切验证
Fig.3 Identification of recombinant plasmid pNZ8148-purC by digestion with restriction endonuclease
为了研究purC基因的过表达对L.lactis NZ9000生长性能的影响,实验测定了对照菌株和重组菌株在初始pH 7.0条件下的生长曲线。由图4可知,重组菌株与对照菌株生长状态基本一致,说明purC的过表达并未影响L.lactis NZ9000的正常生长。

图4L.lactis NZ9000 (pNZ8148-purC)和
L.lactis NZ9000 (pNZ8148)的生长性能
Fig.4 Growth curves ofL.lactis NZ9000 (pNZ8148-purC) andL.lactis NZ9000 (pNZ8148)
乳酸菌主要依靠乳酸等有机酸代谢产物来发挥益生功能,提高乳酸菌酸胁迫耐受性的前提应是确保其正常的乳酸产量,为了进一步考察在初始pH 7.0条件下,重组菌株及对照菌株的发酵情况,作者对菌株的乳酸产量和葡萄糖消耗进行测定。由图5可知,随着菌株生长时间的延长,葡萄糖残留量逐渐降低,乳酸产量逐渐提高,并且菌株生长至8 h后葡萄糖与乳酸含量均趋于平缓。重组菌株的乳酸产量和葡萄糖消耗与对照菌株相比没有明显的区别。说明表达purC基因并未影响重组菌株的正常发酵生产。

图5L.lactis NZ9000 (pNZ8148-purC)和L.lactis NZ9000 (pNZ8148)的葡萄糖和乳酸发酵曲线
Fig.5 The concents of glucose and lactic acid ofL.lactis
NZ9000 (pNZ8148-purC) andL.lactis NZ9000 (pNZ8148)
上述实验结果表明,过量表达purC基因并未影响重组菌株的正常生长和发酵生产。为了进一步考察过表达purC基因对菌株酸胁迫抗性的影响,对L.lactis NZ9000 (pNZ8148-purC)和L.lactis NZ9000 (pNZ8148)进行乳酸胁迫培养实验,测定菌株的存活率。L.lactis生长的最适pH接近中性,随着其主要代谢产物乳酸产量的积累,胞外pH逐渐下降,最低可降至pH 4.5~5.0[26],而在pH 4.0的胁迫环境下,L.lactis的存活率下降最为明显,因此本实验选择pH 4.0作为酸胁迫条件来研究L.lactis的存活率及相关生理指标[27]。如图6所示,为培养在GM17(pH 7.0)培养基中的L.lactis NZ9000经pH 4.0胁迫后存活率的变化情况。
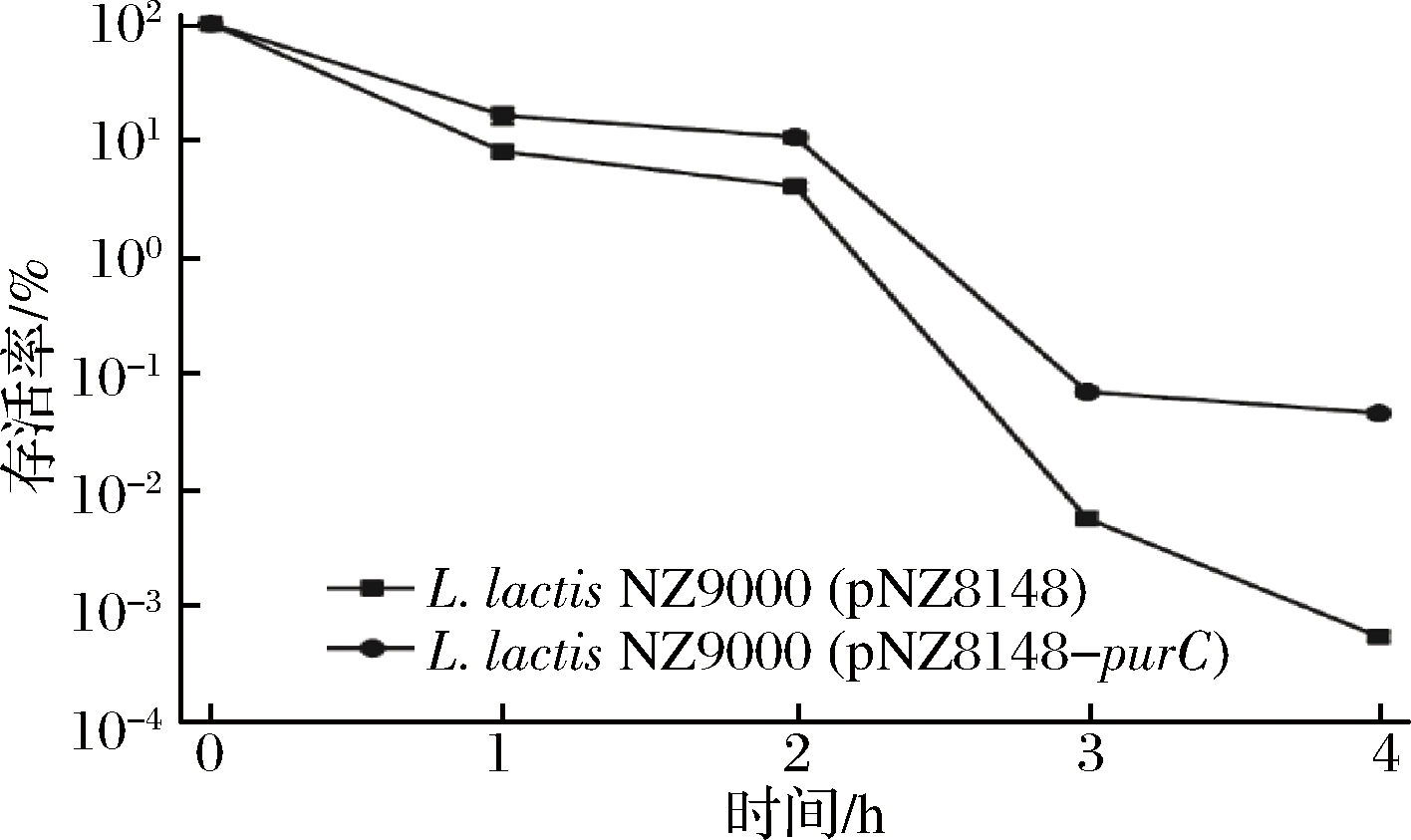
图6 胁迫条件下L.lactis NZ9000 (pNZ8148-purC)与
L.lactis NZ9000 (pNZ8148)的存活率
Fig.6 The survival rates ofL.lactis NZ9000 (pNZ8148-purC) andL.lactis NZ9000 (pNZ8148) under acid stress
由图6可知,过量表达了purC基因的L.lactis NZ9000经pH 4.0胁迫处理后的存活率明显高于未表达purC基因的L.lactis NZ9000,且随着时间的延长,两者之间存活率的差距变大。经pH 4.0胁迫培养3 h后,重组菌株L.lactis NZ9000 (pNZ8148-purC)的存活率是对照菌株的12.3倍,而在pH 4.0胁迫培养4 h后,重组菌株的存活率是对照菌株的83.2倍,结果表明,通过基因工程手段过表达purC基因能明显提高L.lactis NZ9000的酸胁迫抗性(图6)。
当乳酸菌在酸胁迫环境的情况下,维持胞内pH的平衡最主要的应对方式之一是将胞内的H+排出到胞外,此过程是1个耗能的过程,需要消耗大量ATP来产生能量。因此实验考察了酸胁迫条件下菌株胞内ATP浓度的变化(图7)。在pH 4.0胁迫0、1、3 h,胞内ATP含量分别为对照菌株的4.8、6.3和4.3倍,重组菌株L.lactis NZ9000 (pNZ8148-purC)与对照菌株L.lactis NZ9000 (pNZ8148)相比保持了相对较高的ATP浓度,因此,重组菌株可以释放更多的ATP,满足胁迫条件下细胞对能量的需求,从而提高细胞的酸胁迫抗性。

图7 胁迫条件下L.lactis NZ9000 (pNZ8148-purC)和
L.lactis NZ9000 (pNZ8148)胞内ATP含量
Fig.7 The contents of intracellular ATP pool ofL.lactis NZ9000 (pNZ8148-purC) andL.lactis NZ9000 (pNZ8148) under acid stress
氨基酸作为细胞生长代谢的关键营养成分,是多种大分子如蛋白质、脂肪酸、核酸等合成的重要原料物质之一,其还可以在酶的催化作用下生成与细胞能量代谢相关的中间产物[28]。一些氨基酸如天冬氨酸和苏氨酸可以参与嘌呤合成[29],在乳酸乳球菌中,嘌呤代谢与氨基酸代谢之间的联系如图8所示,从图中可以看出,嘌呤代谢途径可以促进ATP的合成,同时与一些氨基酸的合成如天冬氨酸(aspartate)、谷氨酸(glutamate)、苏氨酸(threonine)和γ-氨基丁酸(γ-aminobutyric acid)有着密切的联系。
为了进一步研究过量表达嘌呤代谢途径中的purC基因作用效果,测定了胁迫培养后L.lactis NZ9000 (pNZ8148-purC)的胞内氨基酸含量。结果如图9所示,酸胁迫前后重组菌株胞内天冬氨酸(Asp)、苏氨酸(Thr)、谷氨酸(Glu)和γ-氨基丁酸(GABA)均高于对照菌株。
pH 4.0条件下胁迫2 h后重组菌株胞内Asp、Thr、Glu和GABA含量分别为对照菌株的2.1、1.7、1.8和1.4倍。
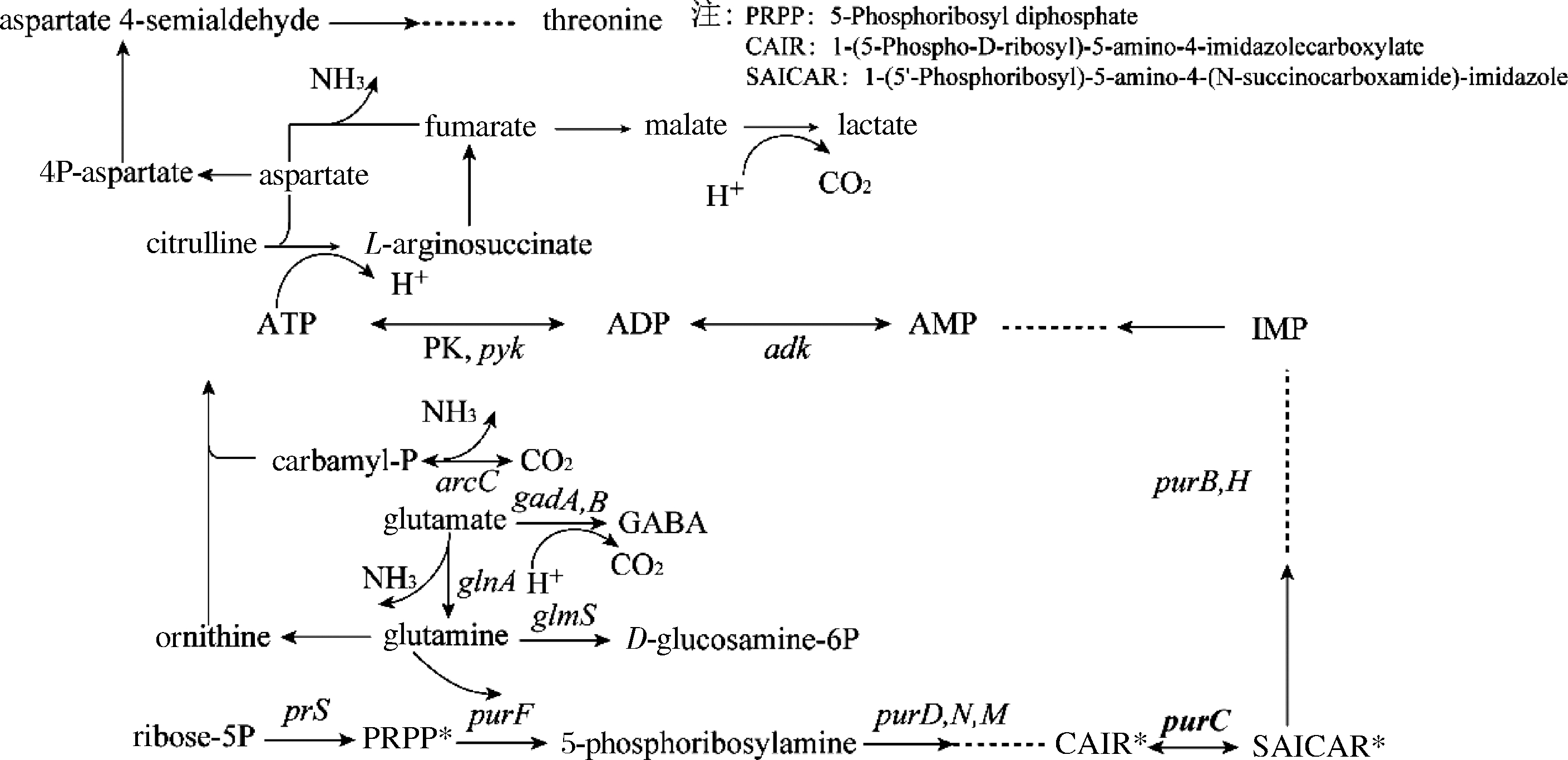
图8L.lactis NZ9000中嘌呤和氨基酸代谢机制
Fig.8 Schematic representation of purine and amino acid pathway inL.lactis NZ9000
Asp在保护乳酸菌抵御酸胁迫过程中具有重要作用[30],Asp生成富马酸(fumarate)的过程中伴随着NH3的产生,同时富马酸能够进一步转化为苹果酸,后者可以通过苹果酸乳酸发酵途径消耗胞内的H+,减少酸胁迫环境对乳酸菌细胞造成的损伤[31],进而增强L.lactis NZ9000的酸胁迫抗性[32]。Thr在苏氨酸脱氨酶的作用下可释放NH3中和H+[33]。氨基酸脱羧是乳酸菌抵御酸胁迫的一种重要途径,乳酸菌可以通过脱羧产生CO2,与此同时消耗H+,从而维持胞内pH值的动态平衡。如图8所示,Glu在谷氨酸脱羧酶催化下脱掉羧基消耗H+,并产生CO2,H+的消耗可提高胞内的pH值。同时,脱羧反应生成的GABA作为一种碱性物质可被反向转运到胞外,进一步减轻细胞的酸胁迫压力[34]。综上,作者证实了在酸胁迫条件下,过量表达嘌呤代谢途径中的purC基因,为细胞提供更多ATP的同时,重组菌株能够产生较多的胞内天冬氨酸、苏氨酸、谷氨酸和γ-氨基丁酸,通过生成碱性物质和消耗胞内质子的方式,维持胞内pH稳态,进而增强L.lactis NZ9000的酸胁迫抗性。

A-天冬氨酸含量;B-苏氨酸含量;C-谷氨酸含量;D-γ-氨基丁酸含量
图9 胁迫条件下L.lactis NZ9000 (pNZ8148-purC)和L.lactis NZ9000 (pNZ8148)胞内氨基酸含量
Fig.9 The contents of intracellular amino acids ofL.lactis NZ9000 (pNZ8148-purC) andL.lactis NZ9000 (pNZ8148) under acid stress
本研究利用NICE系统诱导表达PurC蛋白提高乳酸菌的酸胁迫抗性,相关研究发现,在酸胁迫条件下进行诱导表达的主要是热休克蛋白以及与DNA修复相关的蛋白[35-36]。在乳酸乳球菌(L.lactis)中异源表达来源于大肠杆菌的热休克蛋白DanK,工程菌对酸(体积分数0.5%乳酸,pH 5.47)的耐受性显著提高。WU等[13]通过在L.lactis NZ9000中表达DNA修复蛋白RecO,重组菌株的存活率是对照菌株的2.9倍(pH 4.0胁迫5 h)。除此之外,部分外源功能因子在微生物细胞抵抗酸胁迫中发挥的作用也有相关报道,将L.lactis中海藻糖编码基因trePP、pgmB及otsB在L.lactis NZ9000中进行表达,重组菌对酸胁迫(pH 3.0)的耐受性提高了5~10倍[37]。嘌呤核苷酸对能量代谢和各种合成代谢过程有着至关重要的作用[19],嘌呤代谢途径中的purC基因沉默会引起嘌呤代谢水平改变,影响细胞的正常生长[23]。本研究通过在L.lactis NZ9000过量表达嘌呤代谢途径中的purC基因,在pH 4.0胁迫4 h后,重组菌株的存活率是对照菌株的83.2倍,结果表明,通过基因工程手段过表达purC基因能明显提高L.lactis NZ9000的酸胁迫抗性。进一步研究发现,重组菌株可以在酸胁迫条件下维持更高的胞内ATP浓度和氨基酸含量,分别通过满足细胞对能量的需求以及生成碱性物质和消耗胞内质子的方式缓解细胞受到的酸胁迫压力,从而帮助细胞抵御酸胁迫。本文首次报道了在乳酸乳球菌中过量表达purC基因提高酸胁迫耐受性的作用效果,为进一步通过对嘌呤代谢途径改造提高细胞酸胁迫耐受性提供了新的思路。同时,上述研究结果也对关于purC基因在其他工业微生物中的应用提供了重要的参考。
[1] BENBOUZIANE B, RIBELLES P, AUBRY C, et al. Development of a stress-inducible controlled expression (SICE) system inLactococcus lactis for the production and delivery of therapeutic molecules at mucosal surfaces [J]. Journal of Biotechnology, 2013, 168(2): 120-129.
[2] BURGES C, O'CONNELL-MOTHERWAY M, SYBESMA W, et al. Riboflavin production inLactococcus lactis: Potential for in situ production of vitamin-enriched foods [J]. Applied and Environmental Microbiology, 2004, 70(10): 5 769-5 777.
[3] ZHU Yan, ZHANG Yangping, LI Yin. Understanding the industrial application potential of lactic acid bacteria through genomics [J]. Applied Microbiology and Biotechnology, 2009, 83(4): 597-610.
[4] CARALHEIRO F, MONIZ P, DUARTE L C, et al. Mannitol production by lactic acid bacteria grown in supplemented carob syrup [J]. Journal of Industrial Microbiology & Biotechnology, 2011, 38(1): 221-227.
[5] ZHANG Mingyang, ZHANG Juan, LIU Long, et al. Influence of key acid-resistant genes in arginine metabolism on stress tolerance inLactococcus lactis NZ9000 [J]. Microbiology China, 2017, 44(2): 314-324.
[6] AZIZAN K A, RESSOM H W, MENDOZA E R, et al.13C based proteinogenic amino acid (PAA) and metabolic flux ratio analysis ofLactococcus lactis reveals changes in pentose phosphate (PP) pathway in response to agitation and temperature related stresses [J]. Peerj, 2017, 5(7): 24.
[7] WEIDMANN S, MAITRE M, LAURENT J, et al. Production of the small heat shock protein Lo18 fromOenococcus oeni inLactococcus lactis improves its stress tolerance [J]. International Journal of Food Microbiology, 2017, 247: 18-23.
[8] JIN Junhua, QIN Qian, GUO Huiyuan, et al. Effect of pre-stressing on the acid-stress response inBifidobacterium revealed using proteomic and physiological approaches [J]. Plos One, 2015, 10(2): 10-14.
[9] DE A M, DI C R, HUET C, et al. Heat shock response inlactobacillus plantarum [J]. Applied and Environmental Microbiology, 2004, 70(3): 1 336-1 346.
[10] ZHANG Juan, WU Chongde, DU Guocheng, et al. Enhanced acid tolerance inLactobacillus casei by adaptive evolution and compared stress response during acid stress [J]. Biotechnology and Bioprocess Engineering, 2012, 17(2): 283-289.
[11] BAS T, ANNE W, LEO J, et al. Understanding the adaptive growth strategy ofLactobacillus plantarum by in silico optimisation [J]. Plos Computational Biology, 2009, 5(6): e1000410.
[12] ZHANG Juan, FU Ruiyan, HUGENHOLTZ J, et al. Glutathione protectsLactococcus lactis against acid stress [J]. Applied and Environmental Microbiology, 2007, 73(16): 5 268-5 275.
[13] WU Chongde, ZHANG Juan, DU Guocheng, et al. Heterologous expression ofLactobacillus casei RecO improved the multiple-stress tolerance and lactic acid production inLactococcus lactis NZ9000 during salt stress [J]. Bioresource Technology, 2013, 143(6): 238-241.
[14] ALMARZA O, NUNEZ D, TOLEDO H. The DNA-Binding protein HU has a regulatory role in the acid stress response mechanism inHelicobacter pylori [J]. Helicobacter, 2015, 20(1): 29-40.
[15] BOVE C G, ANGELIS M D, GATTI M, et al. Metabolic and proteomic adaptation ofLactobacillus rhamnosus strains during growth under cheese-like environmental conditions compared to de Man, Rogosa, and Sharpe medium [J]. Proteomics, 2012, 12(21): 3 206-3 218.
[16] WU Chongde, ZHANG Juan, CHEN Wei, et al. A combined physiological and proteomic approach to reveal lactic-acid-induced alterations inLactobacillus casei Zhang and its mutant with enhanced lactic acid tolerance [J]. Applied Microbiology and Biotechnology, 2012, 93(2): 707-722.
[17] 张彦位, 张娟,堵国成,等. 天冬氨酸提高乳酸乳球菌Lactococcus lactis NZ9000酸胁迫抗性的作用机制[J]. 微生物学通报,2018,45(12): 2 563-2 515.
[18] 张梦汝, 张娟,堵国成,等. 外源添加亮氨酸提高乳酸乳球菌酸胁迫抗性[J]. 食品与生物技术学报, 2015, 34(2): 134-139.
[19] CHEN J, VESTERGAARD M, JENSEN T G, et al. Finding the needle in the gaystack-the use of microfluidic droplet technology to identify vitamin-secreting lactic acid bacteria [J]. Mbio, 2017, 8(3): 12.
[20] MA Xiaofeng, WANG Wenming, BITTNER F, et al. Dual and opposing roles of xanthine dehydrogenase in defense-associated reactive oxygen species metabolism in arabidopsis [J]. Plant Cell, 2016, 28(5): 1 108-1 126.
[21] XIE Y, CHOU L S, CUTLER A, et al. DNA macroarray profiling ofLactococcus lactis subsp lactis IL1403 gene expression during environmental stresses [J]. Applied and Environmental Microbiology, 2004, 70(11): 6 738-6 747.
[22] RYSSEL M, HVIID A M M, DAWISH M S, et al. Multi-stress resistance inLactococcus lactis is actually escape from purine-induced stress sensitivity [J]. Microbiology, 2014, 160(Pt11): 2 551-2 559.
[23] YUAN Zhihui, WANG Li, SUN Shutao, et al. Genetic and proteomic analyses of aXanthomonas campestris pv. campestrispurC mutant deficient in purine biosynthesis and virulence [J]. Journal of Genetics and Genomics, 2013, 40(9): 473-487.
[24] 张明阳.argG、argH和argR基因对Lactococcus lactis NZ9000胁迫抗性的影响[D].无锡:江南大学, 2016.
[25] 苏建坤, 王雪,卢建秋,等. OPA-FMOC在线衍生化法测定氨基酸的含量[J]. 中国实验方剂学杂志, 2012, 18(15): 135-138.
[26] OSULLIVAN E, CONDON S. Intracellular pH is a major factor in the induction of tolerance to acid and other stresses inLactococcus lactis [J]. Applied and Environmental Microbiology, 1997, 63(11): 4 210-4 215.
[27] RALLU F, GRUSS A, MAGUIN E.Lactococcus lactis and stress [J]. Antonie Van Leeuwenhoek International Journal of General and Molecular Microbiology, 1996, 70(2-4): 243-251.
[28] FERNANDEZ M, ZUNIGA M. Amino acid catabolic pathways of lactic acid bacteria [J]. Critical Reviews in Microbiology, 2006, 32(3): 155-183.
[29] WU Guoyao. Amino acids: metabolism, functions, and nutrition [J]. Amino Acids, 2009, 37(1): 1-17.
[30] 吴重德, 黄钧,周荣清. 调控乳酸菌酸胁迫抗性研究进展[J]. 微生物学报, 2014, 54(7): 721-727.
[31] BROADBENT J R, LARSEN R L, DEIBEL V, et al. Physiological and transcriptional response ofLactobacillus casei ATCC 334 to acid stress [J]. Journal of Bacteriology, 2010, 192(9): 2 445-2 458.
[32] 张彦位. 调控天冬氨酸代谢途径提高Lactococcus lactis NZ9000的酸胁迫抗性[D].无锡:江南大学, 2018.
[33] GUCHTE M V D, SERROR P, CHERVAUX C, et al. Stress responses in lactic acid bacteria [J]. Antonie Van Leeuwenhoek, 2002, 82(1-4): 187-216.
[34] DAMIANO M A, BASTIANELLI D, DAHOUK S A, et al. Glutamate decarboxylase-dependent acid resistance inBrucella spp.: Distribution and contribution to fitness under extremely acidic conditions [J]. Applied and Environmental Microbiology, 2015, 81(2): 578-586.
[35] WU Chongde, ZHANG Juan, CHEN Wei, et al. A combined physiological and proteomic approach to reveal lactic-acid-induced alterations inLactobacillus casei Zhang and its mutant with enhanced lactic acid tolerance [J]. Applied Microbiology and Biotechnology, 2012, 93(2): 707-722.
[36] WU Rina, ZHANG Wenyi, SUN Tiansong, et al. Proteomic analysis of responses of a new probiotic bacteriumLactobacillus casei Zhang to low acid stress [J]. International Journal of Food Microbiology, 2011, 147(3): 181-187.
[37] CARVALHO A L, CARDOSO F S, BOHN A, et al. Engineering trehalose synthesis inLactococcus lactis for improved stress tolerance [J]. Applied and Environmental Microbiology, 2011, 77(12): 4 189-4 199.