干酪乳杆菌是干酪制品生产中常用乳酸菌发酵剂的重要组成部分。在干酪成熟过程中,风味形成不仅需要分泌到胞外的蛋白酶,同时也需要胞内的脂肪酶和肽酶,而正是乳酸菌自溶过程中释放的肽酶和脂肪酶将牛乳中的脂肪、蛋白和肽水解为风味物质,才赋予其独特的风味和优良的品质[1-3]。发酵剂的自溶会导致蛋白水解物的增加,尤其是游离氨基酸的增加(在干酪成熟过程中可以增加2~3倍),苦味降低,从而使干酪产生更好的风味[4-6]。除了赋予干酪制品独特的风味外,发酵剂自溶的适当提高可以加速干酪的成熟,缩短干酪成熟的生产周期,当自溶菌株与非自溶菌株联合使用时,可以有效地缩短切达干酪的成熟时间[7-8]。
低能量离子注入作为一种较新的人工诱变技术,与其他传统诱变方式(如γ射线、紫外照射等)相比具有损伤率低、突变率高和突变域广的优点,被广泛应用于植物[9-10]、微生物[11-12]育种领域。常用的离子束有He+,Ar+,N+,所用能量一般在数十至数百keV,原理主要为经过质量选择和加速的低能量离子束在真空条件下被注入生物体细胞时,离子束本身的能量可以直接造成胞内重要的生物大分子如蛋白质被分解,双链DNA的解链,单链DNA和碱基的损伤[13]。目前用于诱变的低能量离子束的能量要比传统的离子能量束能量至少低3个数量级,最初主要应用于干的植物种子的诱变,在微生物体系中的应用则因高水分含量限制了低能量离子束的发射范围,使得离子能量不能有效到达生物样品的内部并产生预期的生物效果[14]。此后,随着研究人员对低能量离子注入与生物体间的相互作用关系,从能量发射与吸收、质量堆积效应和电荷积累和转移等方面进行了深入的研究,最终确定了目前可用于微生物诱变的低能量离子诱变技术,并使其在工业应用微生物的育种筛选方面得到广泛应用[15]。
本研究中,通过不同能量的低能N+注入的方式诱变干酪乳杆菌116-a,以自溶度为考察指标,筛选高自溶突变菌株,并通过扫描电镜观察突变前后自溶菌体的形态变化,然后利用荧光定量PCR来检测高自溶突变菌株与野生菌株间N-乙酰胞壁质酶(AcmA)和N-乙酰氨基葡糖胺糖苷酶(GlcNAcase)的表达差异,从而确定在干酪乳杆菌116-a自溶中起关键调控作用的肽聚糖水解酶。
1 材料和方法
1.1 材料与主要仪器
干酪乳杆菌 116-a于-20 ℃保存;MRS肉汤培养基和MRS(北京陆桥技术有限责任公司);细菌gDNA提取试剂盒、100 mg/mL RNase A溶液、20 mg/mL溶菌酶、RNAPrep Pure细菌总RNA提取试剂盒(北京天根生化科技有限公司);2×Bench TopTM Taq Master Mix与琼脂糖凝胶/PCR产物纯化试剂盒(美国Biomiga公司);Biowest常规琼脂糖G-10(香港Gene company Limited);甘氨酸(超纯级);Goldview核酸染料(北京拜尔迪生物技术有限公司);Triton X-100(分析纯,国药化学试剂有限公司);反转录试剂盒(美国 Promega公司);SYBR Green Ⅰ试剂、Prism 光学反应管(美国 ABI公司);DEPC(美国Amresco公司)。
DU800核酸蛋白分析仪,美国Backman;SIGMA 3K-15台式高速冷冻离心机,Sigma公司;LDZX-50KB立式压力蒸汽灭菌锅,上海申安医疗器械厂;UV2300分光光度计,上海天美科学仪器有限公司;DL-CJ-IN高性能无菌试验台,哈尔滨市东联电子技术开发有限公司;Alpha Ease FC凝胶成像分析系统,美国Alpha Innotech;双稳定时电泳仪DYY-6C,北京六一仪器厂;TP600梯度PCR仪,日本TAKARA;DHP-9082电热恒温培养箱,上海一恒科学仪器有限公司;AUW220电子分析天平,日本Shimadzu公司;WH-3微型旋涡振荡器,海门市其林贝尔仪器制造有限公司;ABI 7500荧光定量PCR仪,美国Applied Biosystems;400 keV离子注入机,北京师范大学低能核物理研究所;Delta 320 pH计,法国梅特勒。
1.2 试验方法
1.2.1 菌株保藏及培养基制备
将贮藏于-20 ℃冰箱的冻干菌粉,在37 ℃条件下采用MRS肉汤培养基活化3次以上,短期内可储存于4 ℃冰箱备用。MRS肉汤培养基依照产品说明书标示比例进行配制,灭菌条件:115 ℃灭菌20 min。
1.2.2 样品制备和N+注入
将活化后的干酪乳杆菌116-a培养至对数生长期后,以4%(体积分数)接种量接入MRS肉汤培养基于37 ℃恒温培养箱中培养至OD600nm=1.5左右,使用无菌MRS肉汤培养基稀释菌悬液细胞量为2×108 CFU/mL左右。
在超净工作台中添加100 μL上述菌悬液于直径9.0 cm的洁净无菌培养平皿中,涂布棒涂布均匀后,无菌风吹干形成菌膜,光学显微镜镜检无细胞重叠后,进行离子注入。取原种子菌悬液经稀释后,计算活菌落数。
采用注入能量30 keV的N+源,离子注入剂量分别为0.5×1015、1.0×1015、1.5×1015、2.0×1015、2.5×1015、3.0 ×1015 ions/cm2。设置未进行离子注入操作的真空对照和空气对照。离子注入后,立即采用无菌MRS培养基进行冲洗,将上述冲洗液经适当稀释后取100 μL均匀涂布于MRS琼脂培养基上,37 ℃培养48 h,记录稀释度与其对应的平板菌落数,按公式(1)计算存活率。
存活率![]()
(1)
1.2.3 突变菌株的筛选
分别挑取N+注入后的单菌落于MRS肉汤培养中培养至OD600nm值为0.8~1.0,然后离心(12 000 r/min,10 min,4 ℃)收集菌体检测自溶度。
突变率:以菌体自溶度变化作为考察指标,以原始菌株为空白参照,当突变菌株自溶度的变化幅度不超过±10%时定义为无意义突变,当突变菌株自溶度变化低于-10%为负突变株,高于10%为正突变株。正突变率,负突变率和总突变率按公式(2)、(3)、(4)进行计算:
总突变率![]()
(2)
正突变率![]()
(3)
负突变率![]()
(4)
1.2.4 自溶度的检测
经活化的实验菌株培养至对数生长期后,以4%的接种量接种于MRS肉汤培养基,37 ℃下培养至目标菌体浓度后,离心(5 000 r/min,4 ℃,10 min)收集菌体。无菌生理盐水悬浮洗涤菌体,重复洗涤3次,无菌备用Tri-HCl缓冲液(50 mmol/L Tris,50 mmol/L EDTA)洗涤菌体1次,悬浮收集菌体于pH 6.5的Tri-HCl缓冲液,至菌悬液(OD600 nm值为0.4~0.6),测定初始OD600 nm,读数记为A0。其余上述菌悬液置于37 ℃培养箱温育24 h后,测定OD600 nm,读数记为A24h。则菌体自溶度按公式(5)计算求得:
自溶度![]()
(5)
1.2.5 实时荧光定量PCR引物设计及合成
分别以干酪乳杆菌BL-23基因组为模板,以 Primer premier 5.0软件设计N-乙酰氨基葡糖胺糖苷酶,N-乙酰胞壁质酶和内参基因16S核糖体DNA的引物,并采用NCBI-Primer blast对引物特异性进行检验。采用mfold 在线软件(http://mfold.rna.albany.edu/?q=DINAMelt/Quickfold)对引物扩增子的二级结构进行预测。所设计引物序列(表1)送至北京华大基因进行合成,合成引物于-20 ℃保存备用。
表1 实时荧光定量PCR所用引物序列
Table 1 The primers sequence of GlcNAcase,AcmA and 16S rRNA for RT-qPCR

名称引物序列(5’端-3’端)退火温度/℃产物大小/bpN-乙酰氨基葡糖苷酶(GlcNAcase)上游引物:GCATTGCGATTGCTACT下游引物:CATCCATGTCGGTTTGC58148N-乙酰胞壁质酶(AcmA)上游引物:CACCACGCTGGGTTGTAAG下游引物:TACGGCAGCACGGTAATCT5811216S核糖体RNA(16SrRNA)上游引物:AACCTTACCAGGTCTTGACA下游引物:GGGACTTAACCCAACATCTC58122
1.2.6 细菌总RNA的提取及cDNA的合成
总RNA的提取参考试剂盒中操作说明。
cDNA合成:参照反转试剂盒用户手册。具体反转体系为:25 mmol/L MaCl2 4 μL;Reverse Transcription 10 ×buffer 2 μL;10 mmol/L dNTP Mix 2 μL;rRNasin RNase inhibitor 0.5 μL;AMV Reverse transcriptase 15 U;Random primers 0.5 μg,总RNA template 1 μg,添加无核酸酶水(nuclease-free water)至总反应体积20 μL。反应条件为:室温下放置 10 min,42 ℃反应60 min,95 ℃,5min灭活反转录酶活性,0~5 ℃放置5 min,于-20 ℃保存备用。
1.2.7 实时荧光定量PCR
PCR 反应体系如下:总反应体积为20 μL,10 μL 2×PCR mix (含SYBR-Green I),4 μL cDNA模板,10 μmol/L 上下游引物各 2 μL和2 μL无核酸酶双蒸水。PCR扩增条件为:94 ℃预变性5 min;94 ℃,30 s,58 ℃,30s,72 ℃,1 min (40个循环);72 ℃ 延伸10 min。在延伸阶段检测荧光强度,收集信号。分别对引物进行融解曲线的分析:加热样品从60 ℃ 到95 ℃,每隔0.5 ℃ 停留1 s 检测1 次荧光强度变化。设置无模板空白对照(双蒸水为模板)和RT-阴性对照(以RNA为模板)。
1.2.8 扫描电镜观察
分别低速离心收获处于对数生长期的高自溶突变菌株菌体细胞和野生型菌株细胞,生理盐水洗涤3次,TE缓冲液洗涤1次后,重悬于TE缓冲液,置于37 ℃培养箱孵育24 h,离心收集菌体,重悬于电镜样品固定液,备用。将高自溶突变菌株自溶细胞和野生型菌株自溶细胞经临界点干燥后,样品放置于SEM盘,表面喷金后放置于扫描电子显微镜下观察。
1.2.9 数据处理与分析
应用Statistics Analysis System(SAS)9.2进行单因素方差分析,P<0.05视为差异显著。
2 结果与分析
2.1 N+注入干酪乳杆菌116-a的存活率
离子注入的能量和剂量决定着突变菌株的存活率和突变率。如图1所示,N+注入会显著降低菌体的存活率,相比对照组,经过N+注入之后,菌株存活率均在32%以下。但菌体存活率并不随着注入剂量的增大而降低,而是在不同的N+注入剂量下表现出典型的“马鞍型”特征,使其显著区别于紫外、X射线和γ射线。
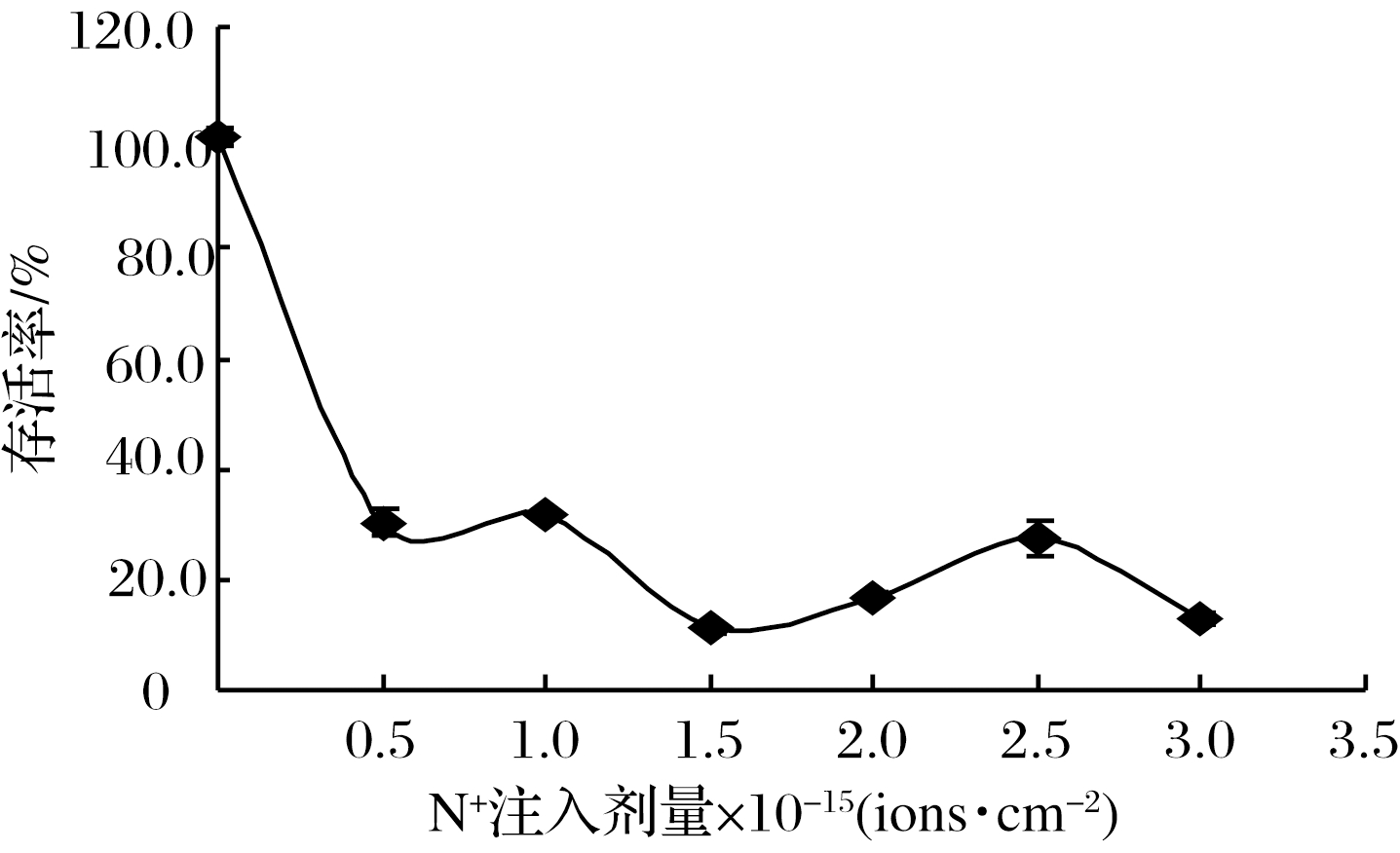
图1 30 keV离子能量N+注入不同注入剂量下干酪乳杆菌116-a的存活率
Fig.1 Survival rate of L.casei 116-a at different doses of 30 keV N+ implantation
N+注入之所以能够造成菌体的大幅度死亡是因为当离子注入细胞时会由于离子刻蚀效果在细胞表面产生类囊泡结构和孔洞,甚至破坏细胞壁的完整性,此外离子注入所引入的能量和电荷的积累则会损伤菌体细胞内的核酸结构和大分子活性物质[16]。而“马鞍型”剂量效应曲线的可能产生机制是低剂量离子注入时以离子的动量转移效应和能量沉积效应为主,两者的共同作用则会极大地损伤细胞内的核酸和生物膜,最终导致菌体的死亡;当注入剂量逐渐提高至某一阈值时,细胞自身的修复机制会被激活从而产生系列修复行为以维持菌体的存活,此时在细胞表面堆积的电荷产生的库伦斥力也在一定程度上阻碍了后续离子对细胞造成的损伤;当注入剂量继续增大时,细胞自身的修复速度将远远低于细胞损伤的速度而电荷积累效应到达临界点时也会产生库伦爆炸,此时保护屏障被破坏而自我修复能力的不足,则导致细胞存活率再度降低[17]。
2.2 干酪乳杆菌116-a突变株的筛选
2.2.1 N+注入干酪乳杆菌116-a的突变率
如图2所示,在0.5×1015~2.5×1015 ions/cm2之间,随着注入剂量的增加菌株突变率也随之增加。注入剂量在0.5×1015~3.0×1015 ions/cm2之间时,最大正突变率和负突变率分别在注入剂量2.5×1015 ions/cm2和2.0×1015 ions/cm2处。而最大注入剂量时则导致最低的菌株突变率,可能是由于存活率的降低导致了突变率的降低。而在注入剂量1.5×1015 ions/cm2和2.0×1015 ions/cm2时,分别只有负突变菌株和正突变菌株。
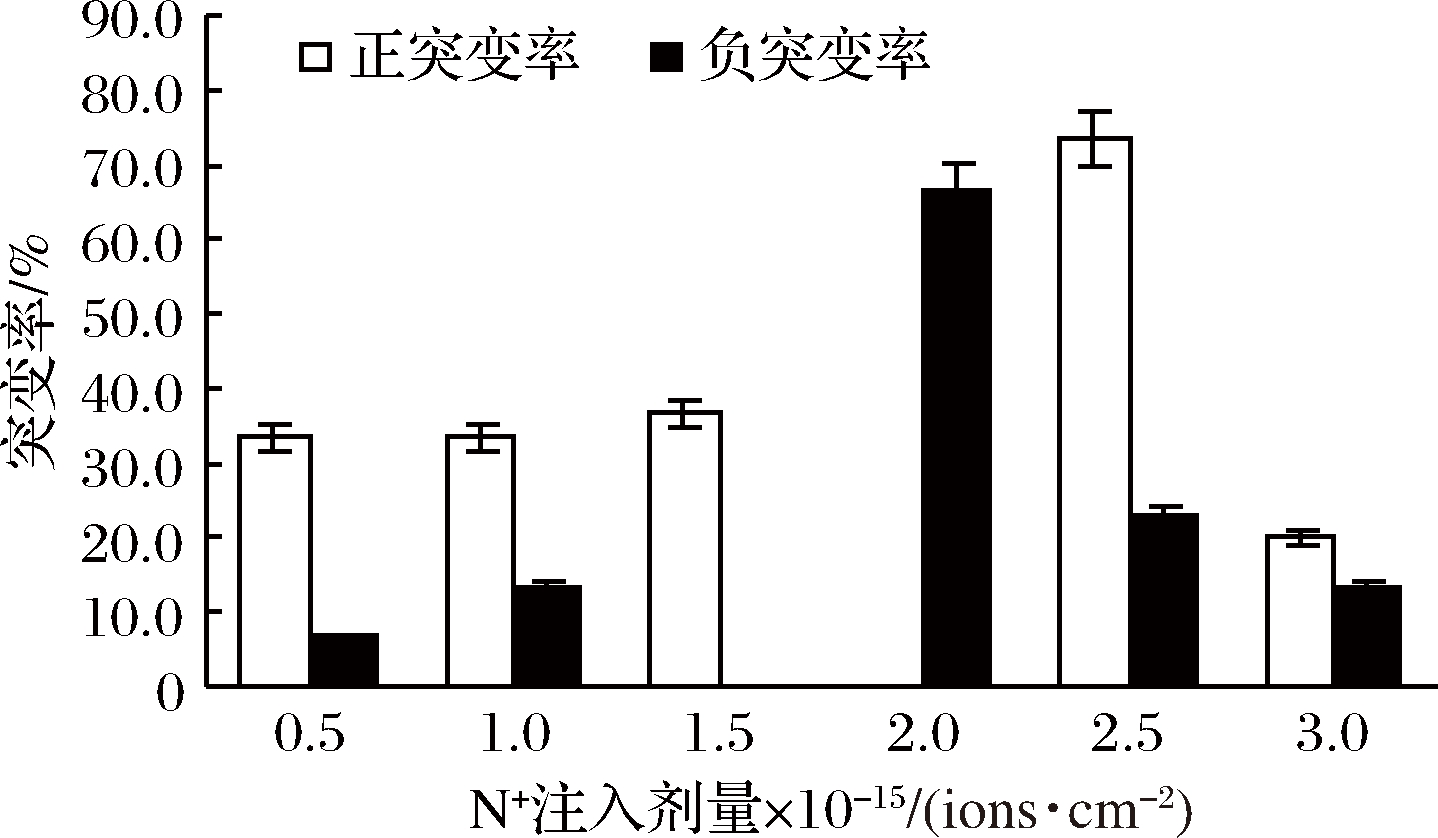
图2 离子注入剂量对干酪乳杆菌116-a突变率的影响
Fig.2 Effect of dose of ion beam implantation on mutation rate of L.casei 116-a
2.2.2 干酪乳杆菌突变体的自溶度
由图3可知,不同剂量N+注入共筛选到正突变变菌株23株,按正突变菌株数量排序可得其中自溶度增大超过15%菌株共有10株,占所有正突变菌株的43.5%。其中自溶度增大最多的正突变菌株出现在注入剂量0.5×1015 ions/cm2处,最大2株正突变菌株的自溶度分别增加了25.7%和20.1%。
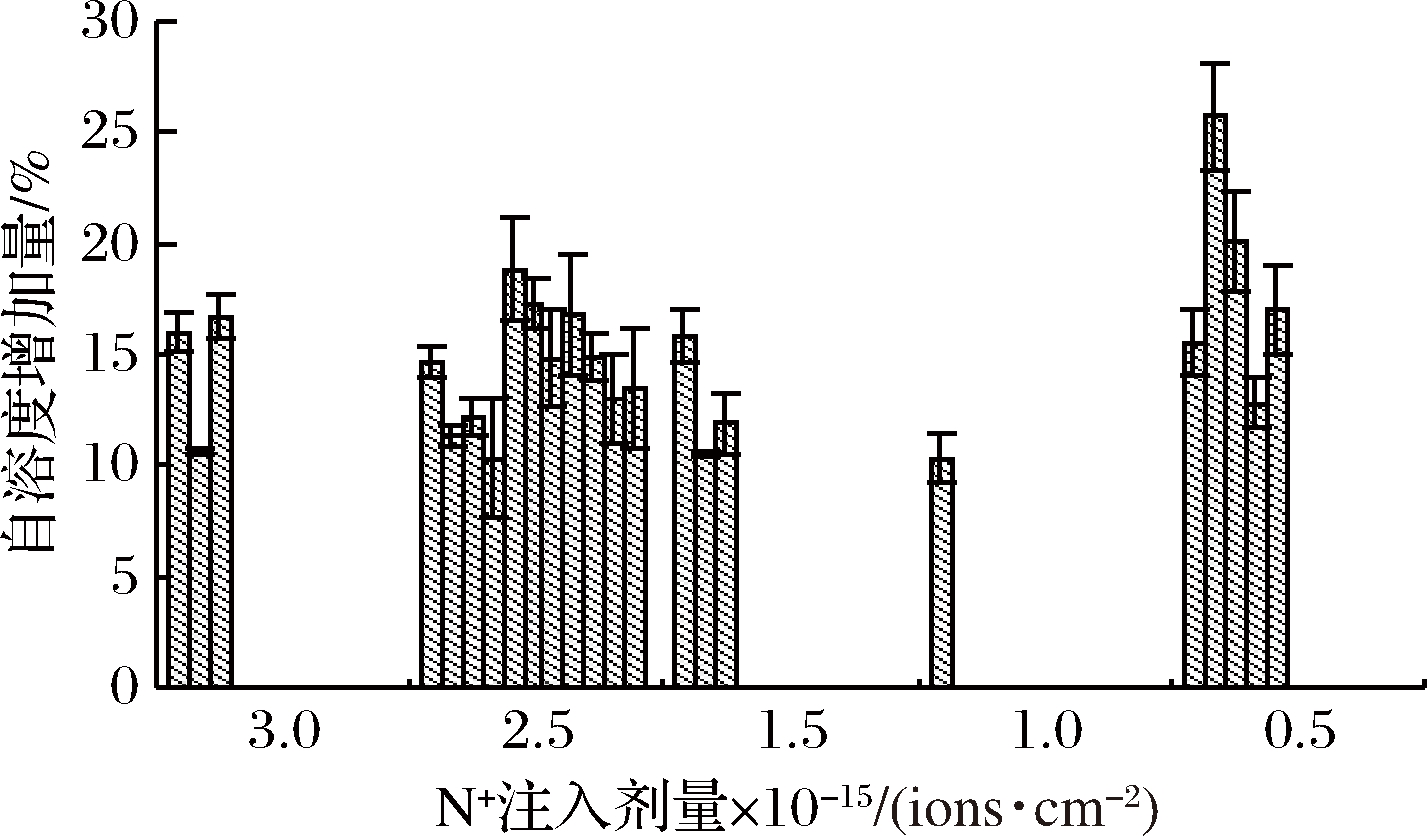
图3 正突变菌株的自溶度
Fig.3 Autolysis rate of positive mutants
N+注入之所以能够造成基因的突变是因为在离子注入过程中离子束或因离子注入作用而产生的大量自由基在生物体细胞内的积累,可以造成DNA、蛋白质等生物大分子的损伤[18]。而一旦核酸损伤发生,则细胞内的修复机制就会对损伤进行修复,主要的修复作用为同源重组和非同源末端连接,一般前一修复机制比较准确基本不会发生错误,而后一修复机制则具有较高的出错率,从而经常导致碱基的改变、缺失或错位插入,从而使生物体产生突变体[19]。
2.3 最大正突变菌株的遗传稳定性
由于生物体细胞内存在着严格的修复和纠错机制,所以筛选得到的氮离子诱变所导致的突变菌株在多次的传代培养中,有可能在生物体本身的同源重组修复机制的作用下,将产生的错误进行修复[18,20]。因此,需要采用连续传代培养的方式,确保所得突变的遗传稳定性。本研究中选取上述研究结果中的最大正突变的菌株,进行连续的传代培养并分别检测每一代的自溶度,连续传代培养10代的各代菌株自溶度结果如图4所示,其中最大自溶度为56.73%,最小自溶度为52.67%,则最大的自溶度差异为4.08%。因此,经过最大正突变菌株经过10代传代培养后,10代间自溶度波动范围都在5%以内且通过统计分析代与代之间自溶度不存在统计学上的显著性差异(P>0.05),表明经过N+注入诱变后,最大正突变菌株获得了具有良好遗传稳定性的自溶度突变菌株。

图4 最大正突变菌株的遗传稳定性
Fig.4 The genetics stability of irradiated positive mutant of L.casei 116-a mutant with N+ implantation
2.4 最大正突变菌株与野生型菌株自溶过程中形态变化
如图5中所示,在相同的自溶条件和自溶时间下,野生型干酪乳杆菌116-a属于低自溶菌株,所以经过诱导自溶后仅有少量细胞发生胞内内容物的逸出物,而菌体仍整体保持较饱满的状态;而在最大正突变菌株中由于自溶度的增大,菌体细胞的饱满度开始降低,菌体细胞出现较明显的凹陷和萎缩,部分细胞可以看到小孔洞的出现,同时更多菌体细胞内容物出现逸出。

a-最大正突变菌株;b-野生菌株自溶细胞
图5 最大正突变菌株和野生菌株自溶细胞的扫描电镜图片
Fig.5 Electron micrographs showing the autolysis cells of mutant and wild type strain
2.5 AcmA和GlcNAcase实时荧光定量PCR的引物特异性和扩增效率
所有用于RT-qPCR的引物,在使用前都需要保证其特异性,不能存在非特异性产物和引物二聚体[21]。本试验中用到的各引物的扩增产物结果如图6中所示,各引物扩增产物均只存在单一条带,各条带所在位置与设计引物产物大小相符,在100 bp附近也不存在引物二聚体。此外,在RT-qPCR过程中检测到的熔解曲线,也都只出现单一的信号峰,其特异性熔解温度分别为16S,82.4 ℃;AcmA,83.6 ℃;GlcNAcase,72.7 ℃进一步表明试验中所用引物具有优良的特异性,保证了试验结果的可靠性。

M-100 bp DNA ladder;1-16s rRNA;2-AcmA;3-GlcNAcase
图6 荧光定量引物的PCR产物
Fig.6 Agarose gel electrophoresis analysis of RT-qPCR product of primers
为了确保引物的扩增效率,本试验中以梯度稀释的cDNA为模板,分别进行RT-qPCR扩增,绘制各基因的标准曲线,得到引物的相关参数如图7所示。从图中可以看到内参基因16S rRNA和2个目的基因的线性均良好,线性相关系数均在0.99~1.00范围内,其中16S rRNA:y=-3.191 7x+21.661,R2=0.991 3;AcmA:y=-3.249 8x+30.221,R2=0.997 4;GlcNAcase:y=-3.401 7x+33.747,R2=0.994 7。依据公式E=(10-1/slope-1)×100[22-23]计算各引物对扩增效率,分别为16S rRNA:105.7%,AcmA:103.1%,GlcNAcase:96.8%。实验中所用的扩增引物得扩增效率均处于90%~110%之间,符合RT-qPCR对引物扩增效率,保证了实验数据的准确和可靠。
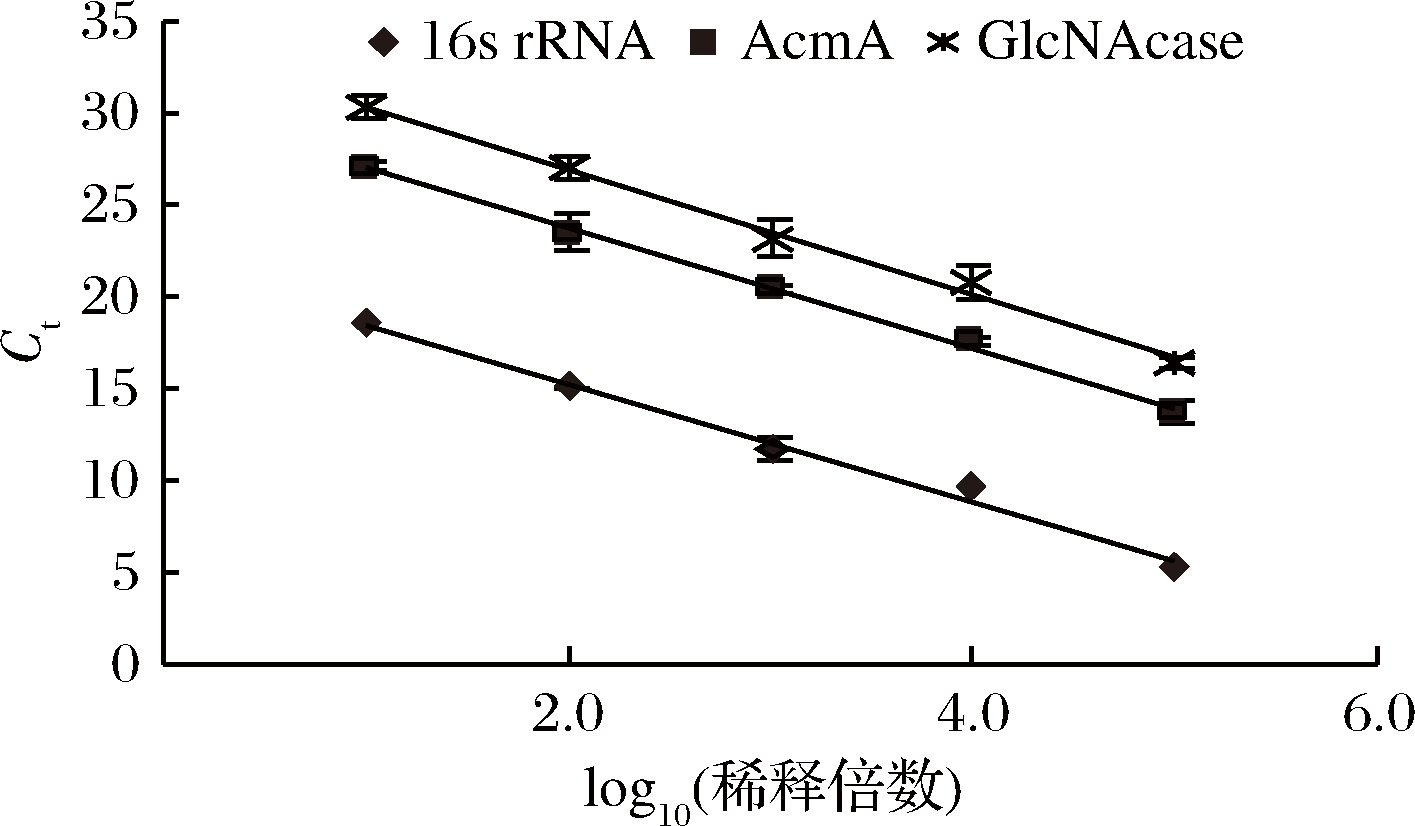
图7 RT-qPCR引物扩增效率
Fig.7 Amplification efficiency of RT-qPCR primers
2.6 AcmA和GlcNAcase在野生菌和最大正突变菌株中的表达差异
通过RT-qPCR研究AcmA和GlcNAcase在野生菌和最大正突变菌株中的表达差异发现,AcmA和GlcNAcase在最大正突变菌株中的表达均比野生菌株中的表达发生了上调,其中最大正突变菌株中GlcNAcase的基因表达比野生菌中的表达上调了55倍,而AcmA的基因表达仅上调了2.8倍(结果如图8所示)。这表明在最大正突变菌株中GlcNAcase表达的增加可能是导致菌体自溶度增加的重要原因。

图8 最大正突变菌与野生菌中GlcNAcase和AcmA的相对表达量
Fig.8 The mean values of the relative expression ratio of GlcNAcase gene and AcmA between positive mutant and wild type strain
AcmA和GlcNAcase是肽聚糖水解酶中的重要组成,两者均可用于水解连接N-乙酰胞壁质酸和N-乙酰葡萄糖胺之间的β-1,4糖苷键,其中前者通过解离N-乙酰胞壁质酸的还原基团实现糖苷键的破坏,而后者则通过解离N-乙酰葡萄糖胺的还原基团来实现糖苷键的破坏[24-25]。在正常的生长环境中,肽聚糖水解酶被精确的调控用于母代子代细胞间的细胞壁分离,而当正常调控系统受到外界的阻挠或环境胁迫时,对肽聚糖水解酶的调控就失去控制,此时肽聚糖水解酶将导致菌体的自溶[26-27]。因此,菌体的自溶与肽聚糖水解酶的表达量和活性大小都密切相关。这也是GlcNAcase的表达量上调导致菌体自溶增加的原因。此外,干酪乳杆菌中存在的肽聚糖水解酶约有十几种,并不是所有的肽聚糖水解酶均在菌体的自溶中发挥重要作用[28]。从上述结果来看GlcNAcase是干酪乳杆菌自溶中关键肽聚糖水解酶可以有效调节自溶的发生,而AcmA在干酪乳杆菌自溶中所起的作用则有限。
3 结论
(1)低剂量的N+注入诱变作为一种较新型的微生物诱变育种技术,可以高效地用于微生物菌种的诱变和选育,本研究中野生型低自溶干酪乳杆菌116-a经过注入剂量0.5×1015 ions/cm2的N+注入诱变筛选得到自溶度增加25.6%的自溶突变菌株。
(2)实时荧光定量PCR分析比较野生菌株和突变菌株的AcmA和GlcNAcase的基因表达发现与野生菌株中的肽聚糖水解酶GlcNAcase的表达量相比,最大正突变菌株中的肽聚糖水解酶GlcNAcase的表达量上调了55倍,表明肽聚糖水解酶GlcNAcase是影响干酪乳杆菌116-a自溶的重要肽聚糖水解酶,在调节干酪乳酸菌自溶中起着作用,而肽聚糖水解表达量的增加可以有效的提高菌体的自溶度。
[1] LAZZI C,POVOLO M,LOCCI F,et al.Can the development and autolysis of lactic acid bacteria influence the cheese volatile fraction?The case of Grana Padano[J].International Journal of Food Microbiology,2016,233:20-28.
[2] THIERRY A,VALENCE F,DEUTSCH S M,et al.Strain-to-strain differences within lactic and propionic acid bacteria species strongly impact the properties of cheese-A review[J].Dairy Science &Technology,2015,95(6):895-918.
[3] ZHAO C J,SCHIEBER A,G NZLE M G.Formation of taste-active amino acids,amino acid derivatives and peptides in food fermentations-A review[J].Food Research International,2016,89:39-47.
NZLE M G.Formation of taste-active amino acids,amino acid derivatives and peptides in food fermentations-A review[J].Food Research International,2016,89:39-47.
[4] CAMARA S P,DAPKEVICIUS A,RIQUELME C,et al.Potential of lactic acid bacteria from Pico cheese for starter culture development[J].Food Science and Technology International,2019,25(4):303-317.
[5] BLAYA J,BARZIDEH Z,LAPOINTE G.Symposium review:Interaction of starter cultures and nonstarter lactic acid bacteria in the cheese environment[J].Journal of Dairy Science,2018,101(4):3 611-3 629.
[6] ÖZER E,KESENKA H.The effect of using different starter culture combinations on ripening parameters,microbiological and sensory properties of Mihaliç cheese[J].Journal of Food Science and Technology,2019,56(3):1 202-1 211.
H.The effect of using different starter culture combinations on ripening parameters,microbiological and sensory properties of Mihaliç cheese[J].Journal of Food Science and Technology,2019,56(3):1 202-1 211.
[7] SAIKI R,HAGI T,NARITA T,et al.Effects of the addition of non-starter lactic acid bacteria on free amino acid production during cheese ripening[J].Food Science and Technology Research,2018,24(2):299-309.
[8] HANNON J A,WILKINSON M G,DELAHUNTY C M,et al.Use of autolytic starter systems to accelerate the ripening of Cheddar cheese[J].International Dairy Journal,2003,13 (4):313-323.
[9] LI Y,WANG W,WANG T,et al.Regulation through microRNAs in response to low-energy N+ ion irradiation in Oryza sativa[J].Radiation Research,2018,191(2):189-200.
[10] ENKHBILEG E,FENYVESI A,B R
R B,et al.Mutation induction in sweet basil (Ocimum basilicum L.) by fast neutron irradiation[J].International Journal of Horticultural Science,2019,25(1-2):30-38.
B,et al.Mutation induction in sweet basil (Ocimum basilicum L.) by fast neutron irradiation[J].International Journal of Horticultural Science,2019,25(1-2):30-38.
[11] GUO X,ZHANG M,GAO Y,et al.A genome-wide view of mutations in respiration-deficient mutants of Saccharomyces cerevisiae selected following carbon ion beam irradiation[J].Applied Microbiology and Biotechnology,2019,103(4):1 851-1 864.
[12] ZHANG N,JIANG J,YANG J,et al.Screening of thermotolerant yeast by low-energy ion implantation for cellulosic ethanol fermentation[J].Energy Sources,Part A:Recovery,Utilization,and Environmental Effects,2018,40(9):1 084-1 090.
[13] GU S B,LI S C,FENG H Y,et al.A novel approach to microbial breeding-low-energy ion implantation[J].Applied Microbiology and Biotechnology,2008,78(2):201-209.
[14] LIU Y,WANG L,ZHENG Z,et al.Improvement of Vitamin K2 production by Escherichia sp.with nitrogen ion beam implantation induction[J].Plasma Science and Technology,2015,17(2):159-166.
[15] 虞龙 姚驰亚,吴晓菲,等.低能氮离子注入和紫外线复合诱变选育高产酿酒酵母[J].辐射研究与辐射工艺学报,2017(2):6.
[16] WANG P,LI J,WANG L,et al.L(+)-lactic acid production by co-fermentation of glucose and xylose with Rhizopus oryzae obtained by low-energy ion beam irradiation[J].Journal of Industrial Microbiology &Biotechnology,2009,36(11):1 363-1 368.
[17] GUO X,ZHANG M,GAO Y,et al.“Saddle-shaped” dose-survival effect,is it a general and valuable phenomenon in microbes in response to heavy ion beam irradiation?[J].Annals of Microbiology,2019,69(3):221-232.
[18] VILAITHONG T,YU L D,APAVATJRUT P,et al.Heavy ion induced DNA transfer in biological cells[J].Radiation Physics and Chemistry,2004,71(3-4):927-935.
[19] THOPAN P,YU L D,TIPPAWAN U.Critical low-energy Ar-ion beam conditions to induce direct DNA double strand break[J].Surface and Coatings Technology,2016,306:313-318.
[20] SPRINGALL L,HUGHES C,SIMONS M,et al.A novel DNA repair mechanism for the processing of low-level UV-induced damage in bacteria[J].Biophysical Journal,2018,114(3):81a-82a.
[21] ONGOL M P,TANAKA M,SONE T,et al.A real-time PCR method targeting a gene sequence encoding 16s rRNA processing protein,rimM,for detection and enumeration of Streptococcus thermophilus in dairy products[J].Food Research International,2009,42(8):893-898.
[22] FOROOTAN A,SJÖBACK R,BJÖRKMAN J,et al.Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR)[J].Biomolecular Detection and Quantification,2017,12(C):1-6.
[23] DUARY R K,BATISH V K,GROVER S.Expression of the atpD gene in probiotic Lactobacillus plantarum strains under in vitro acidic conditions using RT-qPCR[J].Research in Microbiology,2010,161(5):399-405.
[24] INAGAKI N,IGUCHI A,YOKOYAMA T,et al.Molecular properties of the glucosaminidase AcmA from Lactococcus lactis MG1363:mutational and biochemical analyses[J].Gene,2009,447(2):61-71.
[25] PANG X,ZHANG S,LU J,et al.Identification and functional validation of autolysis—associated genes in Lactobacillus bulgaricus ATCC BAA-365[J].Frontiers in Microbiology,2017,8:1 367.
[26] CAVENEY N A,LI F KK,STRYNADKA N CJ.Enzyme structures of the bacterial peptidoglycan and wall teichoic acid biogenesis pathways[J].Current Opinion in Structural Biology,2018,53:45-58.
[27] XU Y,WANG T,KONG J,et al.Identification and functional characterization of AclB,a novel cell-separating enzyme from Lactobacillus casei[J].International Journal of Food Microbiology,2015,203:93-100.
[28] REGULSKI K,COURTIN P,MEYRAND M,et al.Analysis of the peptidoglycan hydrolase complement of Lactobacillus casei and characterization of the major gamma-D-glutamyl-L-lysyl-endopeptidase[J].PloS One,2012,7(2):e32301.