表1 菌株及PCR扩增结果
Table 1 The bacteria strains used in the PCR specificity test
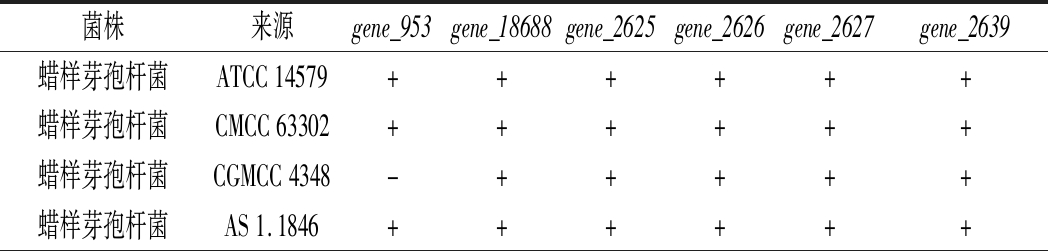
菌株来源gene_953gene_18688gene_2625gene_2626gene_2627gene_2639蜡样芽孢杆菌ATCC14579++++++蜡样芽孢杆菌CMCC63302++++++蜡样芽孢杆菌CGMCC4348-+++++蜡样芽孢杆菌AS1.1846++++++
蜡样芽孢杆菌(Bacillus cereus)是食品中常见的致病菌。其引起的食物中毒主要症状为呕吐、腹泻,严重可导致局部或全身感染[1-4]。蜡样芽孢杆菌可污染谷物类、调味品、乳制品和肉制品等多种食品[5-6],在低温和低pH条件下可以存活[7],因此低温保存的食品也会受到蜡样芽孢杆菌的污染,如蜡样芽孢杆菌是影响低温乳制品货架期的一个主要因素[8-9]。据统计,2002—2015年发生在校园的生物污染食源性疾病事件中蜡样芽孢杆菌、金黄色葡萄球菌和沙门氏菌位居前三位[10]。
我国对食品中蜡样芽孢杆菌的检测主要采用GB4789.14—2014《食品微生物学检验 蜡样芽孢杆菌检验》进行[11-12]。该法周期长,当蜡样芽孢杆菌数较少时结果不准确。目前基于特异性靶点的PCR检测技术已广泛应用于食品病原微生物检测。如基于rpoB[13]和ompA[14]对克罗诺杆菌属进行PCR检测,基于gene_3105[15]对甲型副伤寒沙门氏菌进行PCR检测以及基于LMOf2365_2721,AX25_00730[16]对单增李斯特菌进行PCR检测等。
目前基于特异性靶点基因对蜡样芽孢杆菌的PCR检测方法已有较多报道,包括基于gyrB、16S rRNA[17]、pcplc[18]和HBLA[19]等特异性靶点。但是一些靶点的特异性不强,如HBLA在苏云金芽孢杆菌中也存在。因此为了丰富蜡样芽孢杆菌的PCR快速检测方法,本研究应用生物信息学和比较基因组学方法,从蜡样芽孢杆菌全基因数据中筛选特异性基因,以筛选到的特异性基因为靶点设计特异性引物,建立蜡样芽孢杆菌的PCR检测体系,并将其应用于牛肉和猪肉实际样品中,为食品中蜡样芽孢杆菌的快速检测提供了技术支持。
1.1.1 试验材料
牛肉和猪肉样品,购自河南省新乡市某农贸市场。菌株信息如表1所示。
表1 菌株及PCR扩增结果
Table 1 The bacteria strains used in the PCR specificity test

菌株来源gene_953gene_18688gene_2625gene_2626gene_2627gene_2639蜡样芽孢杆菌ATCC14579++++++蜡样芽孢杆菌CMCC63302++++++蜡样芽孢杆菌CGMCC4348-+++++蜡样芽孢杆菌AS1.1846++++++
续表1
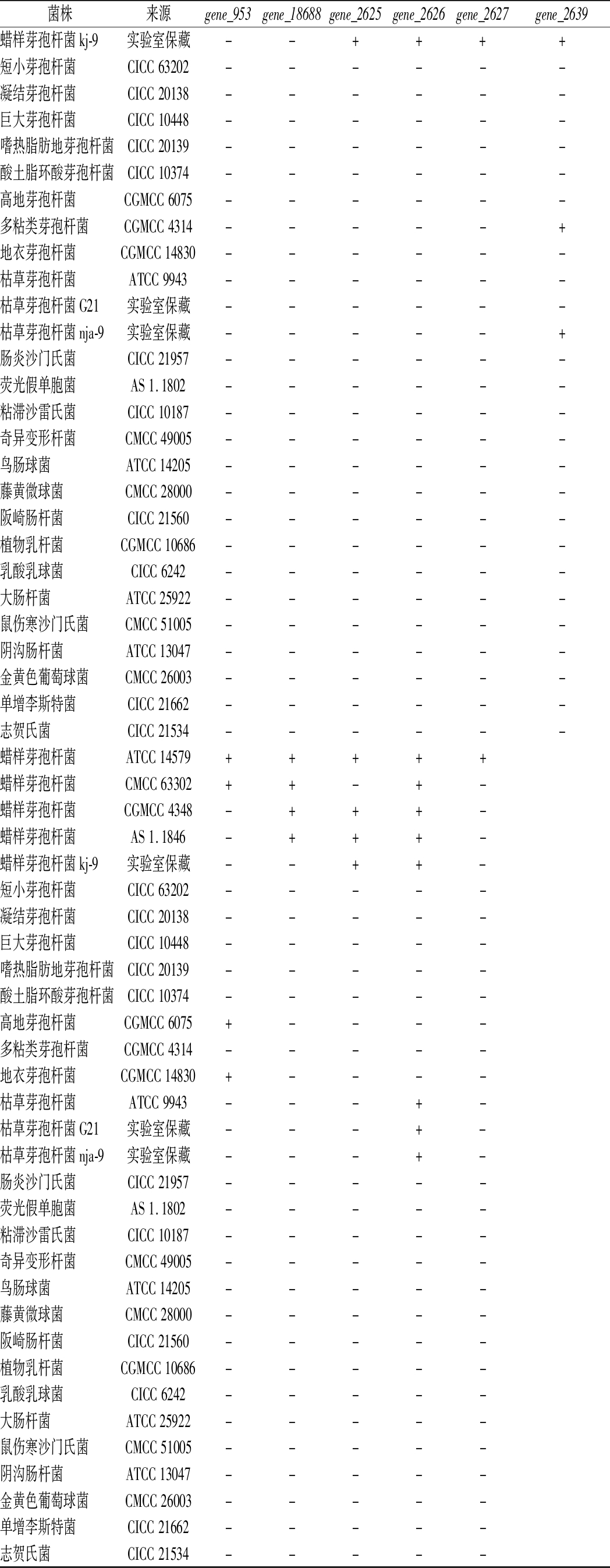
菌株来源gene_953gene_18688gene_2625gene_2626gene_2627gene_2639蜡样芽孢杆菌kj-9实验室保藏--++++短小芽孢杆菌CICC63202------凝结芽孢杆菌CICC20138------巨大芽孢杆菌CICC10448------嗜热脂肪地芽孢杆菌CICC20139------酸土脂环酸芽孢杆菌CICC10374------高地芽孢杆菌CGMCC6075------多粘类芽孢杆菌CGMCC4314-----+地衣芽孢杆菌CGMCC14830------枯草芽孢杆菌ATCC9943------枯草芽孢杆菌G21实验室保藏------枯草芽孢杆菌nja-9实验室保藏-----+肠炎沙门氏菌CICC21957------荧光假单胞菌AS1.1802------粘滞沙雷氏菌CICC10187------奇异变形杆菌CMCC49005------鸟肠球菌ATCC14205------藤黄微球菌CMCC28000------阪崎肠杆菌CICC21560------植物乳杆菌CGMCC10686------乳酸乳球菌CICC6242------大肠杆菌ATCC25922------鼠伤寒沙门氏菌CMCC51005------阴沟肠杆菌ATCC13047------金黄色葡萄球菌CMCC26003------单增李斯特菌CICC21662------志贺氏菌CICC21534------蜡样芽孢杆菌ATCC14579+++++蜡样芽孢杆菌CMCC63302++-+-蜡样芽孢杆菌CGMCC4348-+++-蜡样芽孢杆菌AS1.1846-+++-蜡样芽孢杆菌kj-9实验室保藏--++-短小芽孢杆菌CICC63202-----凝结芽孢杆菌CICC20138-----巨大芽孢杆菌CICC10448-----嗜热脂肪地芽孢杆菌CICC20139-----酸土脂环酸芽孢杆菌CICC10374-----高地芽孢杆菌CGMCC6075+----多粘类芽孢杆菌CGMCC4314-----地衣芽孢杆菌CGMCC14830+----枯草芽孢杆菌ATCC9943---+-枯草芽孢杆菌G21实验室保藏---+-枯草芽孢杆菌nja-9实验室保藏---+-肠炎沙门氏菌CICC21957-----荧光假单胞菌AS1.1802-----粘滞沙雷氏菌CICC10187-----奇异变形杆菌CMCC49005-----鸟肠球菌ATCC14205-----藤黄微球菌CMCC28000-----阪崎肠杆菌CICC21560-----植物乳杆菌CGMCC10686-----乳酸乳球菌CICC6242-----大肠杆菌ATCC25922-----鼠伤寒沙门氏菌CMCC51005-----阴沟肠杆菌ATCC13047-----金黄色葡萄球菌CMCC26003-----单增李斯特菌CICC21662-----志贺氏菌CICC21534-----
注:+表示PCR阳性结果;-表示PCR阴性结果(下同)
1.1.2 试验试剂
营养琼脂(NA培养基),英国Oxoid公司;Ezup柱式细菌总DNA提取试剂盒,北京索莱宝科技有限公司;引物合成,武汉金开瑞生物工程有限公司;GoldView核酸染料,北京索莱宝科技有限公司;2×Taq Mix、dNTP、6×loading buffer和DNA标准分子质量DL2000 Marker,南京诺维赞生物科技有限公司。
5418高速离心机,德国Eppendorf公司;JS-380C全自动数码凝胶成像分析仪,上海培清科技有限公司;PowPacTM HC164-5052高电流电泳仪,美国Bio-Rad公司;PTC-100TMPCR仪,美国MJ Research公司;NV3000C微量紫外分光光度计仪,北京凯奥科技发展有限公司。
1.3.1 特异性基因的筛选
从NCBI(ftp://ftp.ncbi.nih.gov/genbank/genomes/Bacteria/)基因组公共数据库中,获取蜡样芽孢杆菌ATCC 14579的基因组DNA序列。将全基因组DNA序列中的每个基因在NCBI的BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome)程序中进行比对。选择Query cover值为100%的同源性最好,同时与其他芽孢菌属的菌株不具有同源性的基因作为该蜡样芽孢杆菌的准特异性基因。
1.3.2 引物设计
使用Premier 5.0软件(Premier Biosoft International Inc.,USA)选择序列长度在400 bp以上的准特异性基因设计引物。获得的引物再通过Oligo 6.0软件和BLAST网站进行分析,选择与非模板DNA同源性较低的引物作为该基因的特异性引物(表2)。
表2 引物序列及扩增片段大小
Table 2 Primer sequences and the length of amplification fragments

特异性基因特异性引物引物序列退火温度/℃扩增大小/bpgene_2625gFA1-fTCCAAGTTGTACCAGAGATTGATGCT59269gFA1-rGCGAATAGTGCGGAGTATGGATTCTATA59.6gene_2626gFA2-fGGCTAGGCAGTACCGAGGAAGAA59.9750gFA2-rTAGCGTATCCTGGTTGAACTGGTAATTG60.2gene_2627gFA3-fCCACGCTACTTCCTGGAATCTACAA59591gFA3-rGGAATGCCTGTTCTCTTGCTACTGA59.2
1.3.3 实验菌株基因组DNA提取
使用Ezup柱式细菌基因组提取试剂盒提取基因组DNA。
1.3.4 引物特异性评价
以基因组DNA为模板,采用引物gFA1、gFA2、gFA3分别进行PCR扩增。反应体系为2×PCR Taq Master 12.5 μL、上下游引物(0.4 μmol/L)各1 μL、DNA模板1 μL和ddH2O 9.5 μL;总反应体25 μL。反应条件为95 ℃预变性5 min; 95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸45 s以上过程进行35个循环;最后72 ℃延伸10 min。PCR扩增产物在1%的琼脂糖凝胶中进行电泳,并在凝胶成像仪中观察电泳结果。
1.3.5 PCR检测体系的建立
以蜡样芽孢杆菌ATCC 14579的基因组DNA为模板,对PCR检测体系进行优化。确定最佳反应体系为10×PCR buffer(Mg2+ free)3 μL、dNTP 0.5 μmol/L、MgCl2 4.5 mmol/L、Taq聚合酶0.6 U、引物gFA2 0.8 μmol/L、DNA模板2 μL、ddH2O 4 μL;总反应体25 μL。优化的PCR反应条件为95 ℃预变性5 min;再95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸60 s以上过程进行35个循环;最后72 ℃延伸10 min。
1.3.6 蜡样芽孢杆菌PCR检测引物灵敏度评价
通过核酸浓度测定仪测定初始基因组DNA浓度,再利用无菌蒸馏水10倍梯度稀释。以稀释后的DNA溶液为模板,利用上述PCR体系及程序进行反应,确定灵敏度较高的引物对。
1.3.7 PCR检测体系的抗干扰能力评价
将牛肉和猪肉样品采用国标方法检测,确定无蜡样芽孢杆菌污染后,分别取牛肉和猪肉各25 g,分别在LB培养基中增菌12 h获得各自的背景增菌液,采用平板菌落计数法测定菌液浓度。同时培养蜡样芽孢杆菌ATCC 14579,采用平板菌落计数法测定菌液初始浓度,使用0.9%(质量分数)无菌生理盐水进行10倍梯度稀释。取5 mL 108 CFU/g至101 CFU/g的蜡样芽孢杆菌菌液分别与牛肉背景菌群浓度为5.28×107 CFU/mL和猪肉背景菌群浓度为7.75×106 CFU/mL混合均匀,按照1.3.3提取菌液的总基因组DNA。以混合菌液总DNA为模板进行PCR扩增,同时以无菌水作空白对照。
1.3.8 人工污染实验
人工污染菌为蜡样芽孢杆菌ATCC 14579,接种于LB培养基增菌12 h后,用0.9%无菌生理盐水进行10倍梯度稀释。取不同稀释度的菌液接种于经巴氏杀菌(85 ℃处理15 s)的牛肉和猪肉样品中,每个处理3个重复。每个样品取25 g,分别用1 mL不同稀释度的蜡样芽孢杆菌菌液污染后,无菌均质2 min,置入225 mL的LB液体培养基中,在37 ℃、180 r/min增菌培养12 h。在培养过程中从4 h开始每隔2 h取1 mL的增菌液提取DNA,按照1.3.5进行PCR扩增,同时以无菌水作为对照。
将蜡样芽孢杆菌ATCC 14579的基因通过NCBI数据库的BLAST程序(http://blast.ncbi.nlm.nih.gov/)进行序列比对。共获得11个准特异性基因分别为gene_953(genebank登录号BC0954)、gene_1868(genebank登录号BC1872)、gene_2625(genebank登录号BC2629)、gene_2626(genebank登录号BC2630)、gene_2627(genebank登录号BC2631)、gene_2639(genebank登录号BC2643)、gene_2643(genebank登录号BC2647)、gene_4437(genebank登录号BC4448)、gene_4438(genebank登录号BC4449)、gene_4439(genebank登录号BC4450)和gene_4440(genebank登录号BC4451)。选取以上6个基因为模板,使用Primer 5.0设计引物。以5株蜡样芽孢杆菌和26株非蜡样芽孢杆菌的基因组DNA为模板,进行PCR验证。PCR扩增结果见表1。结果表明基因gene_2625、gene_2626、gene_2627表现出更好的特异性,被确定为蜡样芽孢杆菌的特异性基因。
通过PCR检测结果发现引物gFA2扩增时条带最明显且无非特异性条带产生。因此选取引物gFA2优化蜡样芽孢杆菌的PCR检测体系。通过优化引物的浓度、Mg2+浓度及Taq聚合酶浓度最终确定反应体系和反应条件见1.3.5。优化的反应体系检测结果如图1所示。
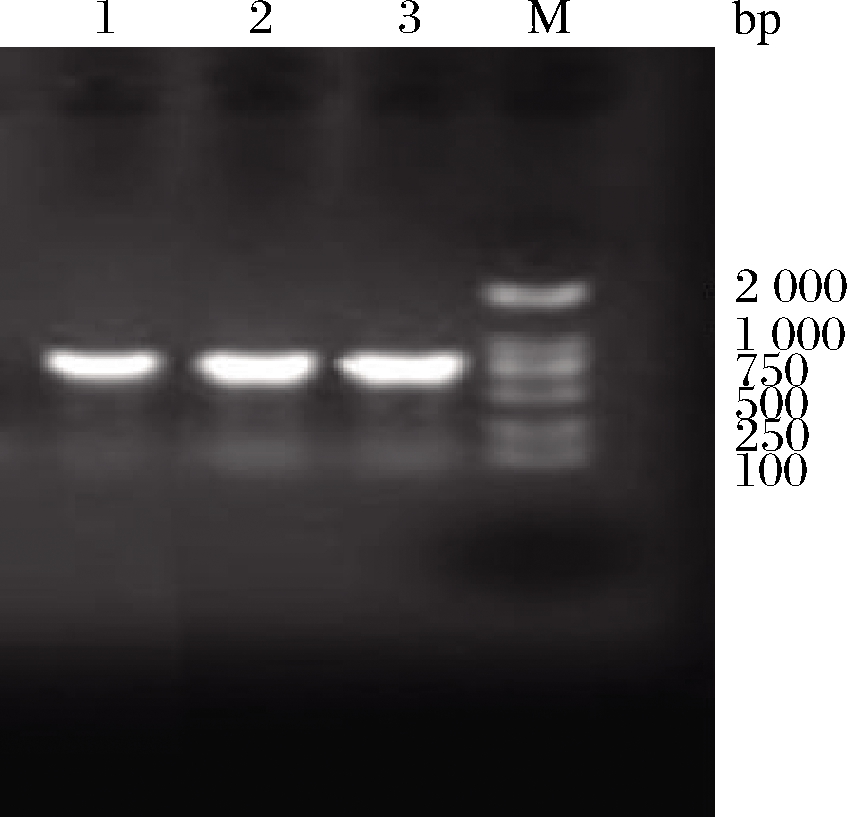
泳道1、2、3-Bacillus cereus;M-DL2000 marker
图1 检测蜡样芽孢杆菌的PCR反应阳性结果
Fig.1 Positive results obtained in the PCR detection of Bacillus cereus
以表2中所列菌株的基因组DNA为模板,对优化的PCR体系进行特异性验证,结果如图2所示。结果表明,引物gFA1、gFA2、gFA3分别扩增出269、750、591 bp的特异性条带,而非蜡样芽孢杆菌基因组DNA没有条带。

M-DL2000 marker;1-蜡样芽孢杆菌ATCC 14579;2-蜡样芽孢杆菌CMCC 63202;3-蜡样芽孢杆菌CGMCC 4348;4-蜡样芽孢杆菌AS 1.1846;5-蜡样芽孢杆菌;6-短小芽孢杆菌;7-凝结芽孢杆菌;8-巨大芽孢杆菌;9-嗜热脂肪地芽孢杆菌;10-酸土脂环酸芽孢杆菌;11-高地芽孢杆菌;12-多黏类芽孢杆菌;13-地衣芽孢杆菌;14-枯草芽孢杆菌;15-枯草芽孢杆菌G21;16-枯草芽孢杆菌nja-9;17-肠炎沙门氏菌;18-荧光假单胞菌;19-黏滞沙雷氏菌;20-奇异变形杆菌;21-鸟肠球菌;22-藤黄微球菌;23-阪崎肠杆菌;24-植物乳杆菌;25-乳酸乳球菌;26-大肠杆菌;27-鼠伤寒沙门氏菌;28-阴沟肠杆菌;29-金黄色葡萄球菌;30-单增李斯特菌;31-志贺氏菌
a-gFA1;b-gFA2;c-gFA3
图2 三种引物的特异性检测结果
Fig.2 PCR detection specificity of used three primers
蜡样芽孢杆菌ATCC 14579基因组DNA质量浓度为35.95 ng/μL,用无菌水进行10倍梯度稀释,将系列稀释的DNA作为模板,应用引物gFA1、gFA2、gFA3进行PCR扩增,以确定检测体系的灵敏度,结果如图3所示。结果表明,gFA2灵敏度较高可达359.5 fg/μL,gene_2625、gene_2627灵敏度可达3.595 pg/μL。CAWTHOM等[20]的研究报道用于检测阪崎肠杆菌的特异性引物Eask的最低检测灵敏度为10 pg/μL,TAO等[21]报道用于检测单增李斯特菌的Lm8引物的最低灵敏度为430 fg/μL,因此本研究中所筛选的引物具有较高的检测灵敏度。
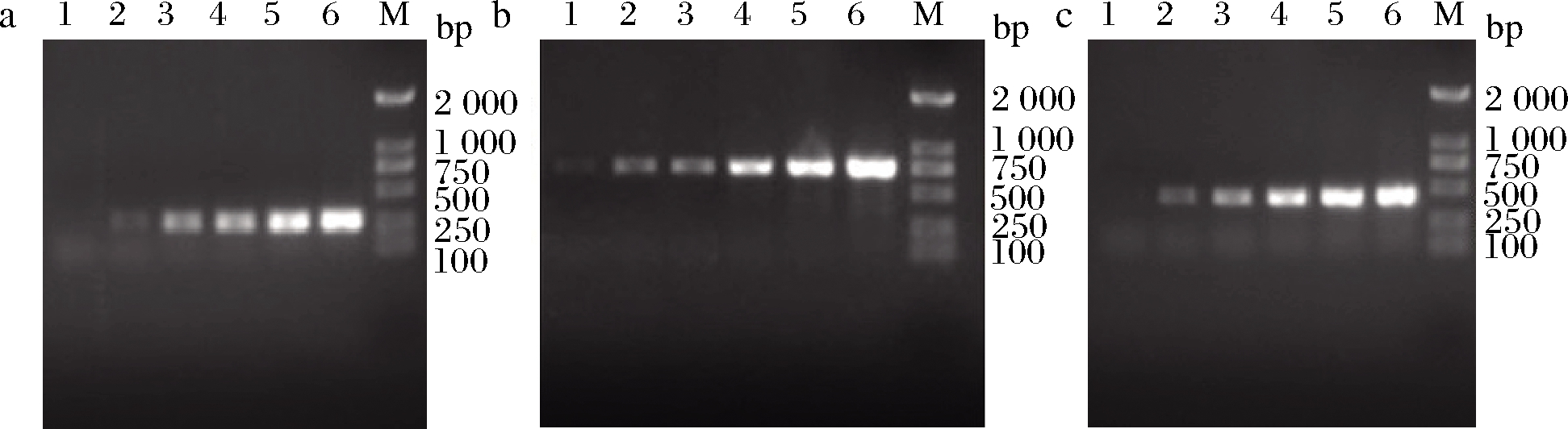
泳道1~6-359.5 fg/μL、3.595 pg/μL、35.95 pg/μL、359.5 pg/μL、3.595 ng/μL、35.95 ng/μL的DNA模板;M-DL2000 marker
a-gFA1;b-gFA2;c-gFA3
图3 三种引物的灵敏度检测结果
Fig.3 PCR detection sensitivity of used three primers
食品样品中通常存在多种微生物,而非蜡样芽孢杆菌菌属的基因组DNA可能会影响PCR反应的准确度。因此,本研究对特异性引物的抗干扰能力进行了评价。将购自农贸市场的牛肉和猪肉样品经国标方法检测无蜡样芽孢杆菌污染后,进行增菌培养,牛肉的增菌浓度为5.28×107 CFU/mL,猪肉的增菌浓度为7.75×106 CFU/mL。将不同稀释度的蜡样芽孢杆菌培养液分别与牛肉和猪肉样品增菌液混合后提取总基因组DNA进行PCR检测,结果如表3和表4所示。由表3可知,当牛肉背景菌液浓度为5.28×107 CFU/mL时,蜡样芽孢杆菌浓度高于3.74×104 CFU/mL的样品可以扩增得到gene_2625、gene_2626和gene_2627的条带。而当蜡样芽孢杆菌浓度为3.74×103 CFU/mL时,仅可以扩增出gene_2626的条带,其他2个基因无条带。说明此时样品中蜡样芽孢杆菌浓度较低,牛肉样品背景菌液浓度较高,对检测结果产生了干扰。由表4可知,当猪肉背景菌液浓度为7.75×106 CFU/mL时,蜡样芽孢杆菌浓度高于3.74×103 CFU/mL的样品可以扩增得到gene_2625、gene_2626和gene_2627的条带。而当蜡样芽孢杆菌浓度低于3.74×103 CFU/mL时,3个目的基因均无扩增条带产生。说明猪肉样品背景菌液浓度较高,对检测结果产生了干扰。本研究中在牛肉背景菌浓度5.28×107 CFU/mL和猪肉背景菌浓度7.75×106 CFU/mL的干扰下,当蜡样芽孢杆菌的浓度高于3.74×103 CFU/mL,均可检测到目的条带。说明本研究中蜡样芽孢杆菌特异性引物具有较强的抗干扰能力。
表3 牛肉微生物菌群对蜡样芽孢杆菌检测的干扰
Table 3 Effect of pork background flora to detection of Bacillus cereus

蜡样芽孢杆浓度/(CFU·mL-1)gene_2625gene_2626gene_26273.74×107+++3.74×106+++3.74×105+++3.74×104+++3.74×103-+-3.74×102---3.74×101---0---
表4 猪肉微生物菌群对蜡样芽孢杆菌检测的干扰
Table 4 Effect of pork background flora to detection of Bacillus cereus

蜡样芽孢杆浓度/(CFU·mL-1)gene_2625gene_2626gene_26273.74×107+++3.74×106+++3.74×105+++3.74×104+++3.74×103+++3.74×102---3.74×101---0---
食品样品中组分较为复杂,一些成分会影响PCR反应,从而影响PCR检测结果,因此需要通过检测人工污染实验来评价检测结果的可靠性[22]。本研究选用牛肉和猪肉作为人工污染对象,牛肉样品中蜡样芽孢杆菌的污染浓度分别为4.62×103、4.62×102、4.62×101、4.62 CFU/g。当增菌培养10 h后,3对引物检测各污染浓度结果均呈阳性。增菌4 h时,只有污染浓度为4.62×103 CFU/g的样品呈阳性,结果如表5所示。猪肉样品中蜡样芽孢杆菌的污染浓度分别为8.38×103、8.38×102、8.38×101、8.38 CFU/g。当增菌培养10 h后,3对引物检测各污染浓度结果均呈阳性。增菌4 h时,只有污染浓度为8.38×102和8.38×103 CFU/g的样品呈阳性,结果如表6所示。以上试验结果表明,牛肉和猪肉样品经人工污染蜡样芽孢杆菌增菌培养10 h后,本研究所用的3对引物对蜡样芽孢杆菌的检出限能达到N CFU/g(0<N<10),证明该检测方法具有较强的可靠性。
表5 人工污染牛肉样品PCR扩增结果
Table 5 PCR amplification results of Bacillus cereus in artificially contaminated beef samples
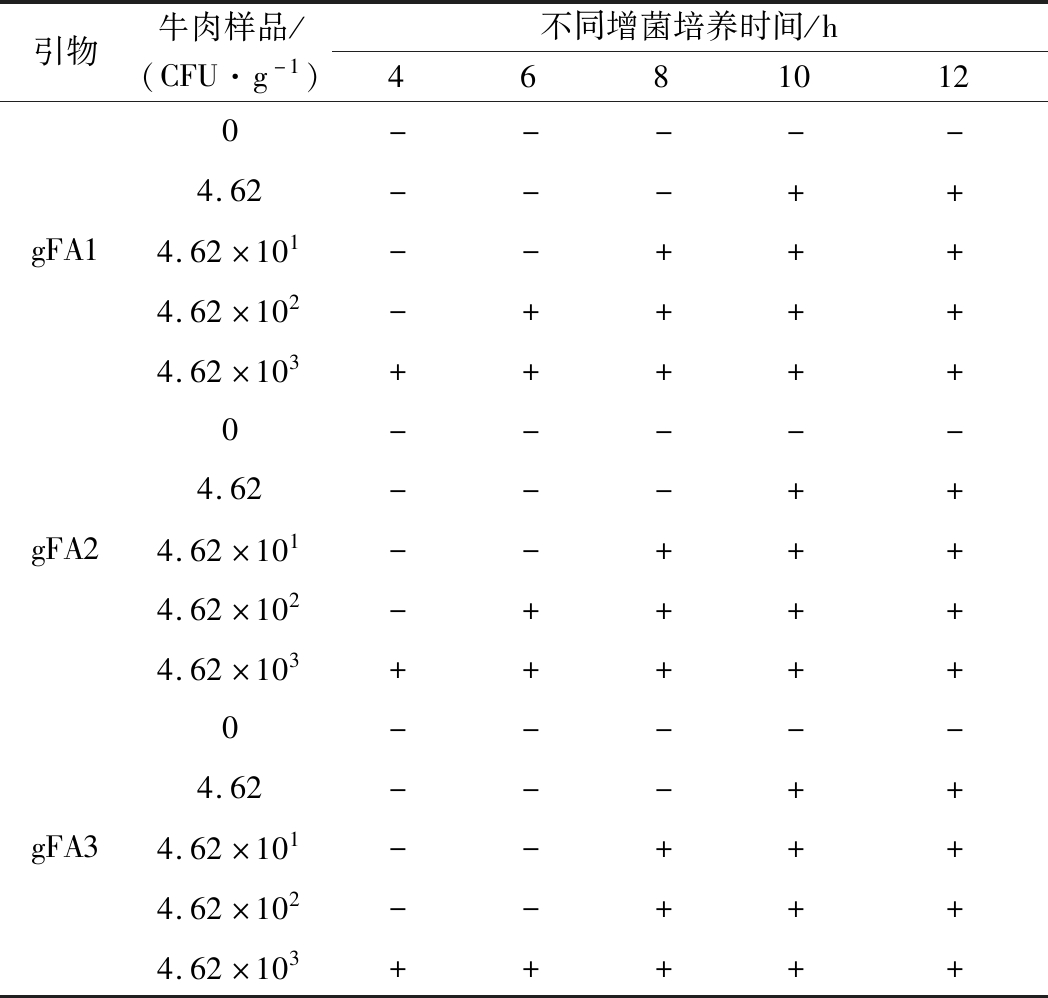
引物牛肉样品/(CFU·g-1)不同增菌培养时间/h46810120-----4.62---++gFA14.62×101--+++4.62×102-++++4.62×103+++++0-----4.62---++gFA24.62×101--+++4.62×102-++++4.62×103+++++0-----4.62---++gFA34.62×101--+++4.62×102--+++4.62×103+++++
表6 人工污染猪肉样品PCR扩增结果
Table 6 PCR amplification results of Bacillus cereus in artificially contaminated pork samples
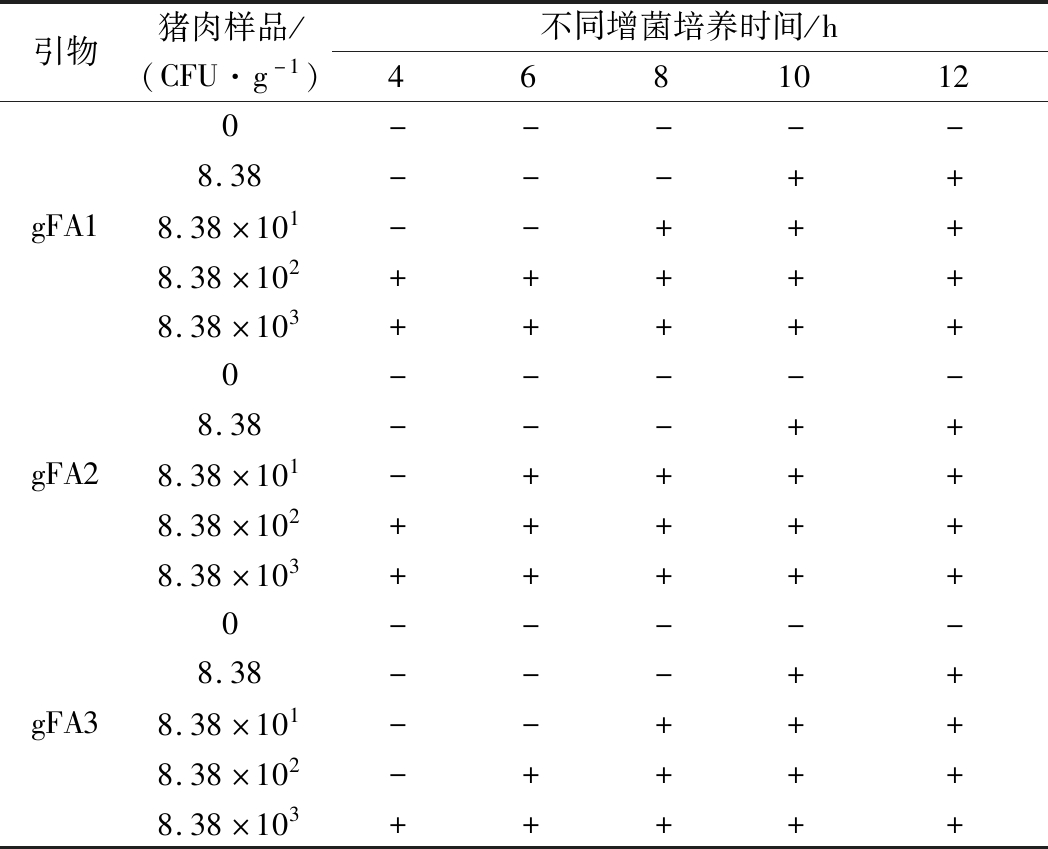
引物猪肉样品/(CFU·g-1)不同增菌培养时间/h46810120-----8.38---++gFA18.38×101--+++8.38×102+++++8.38×103+++++0-----8.38---++gFA28.38×101-++++8.38×102+++++8.38×103+++++0-----8.38---++gFA38.38×101--+++8.38×102-++++8.38×103+++++
蜡样芽孢杆菌是食品中常见的条件致病菌,在我国常发生由蜡样芽孢杆菌引起的食源性疾病[23-24]。目前分子生物学检测方法已成为食品中蜡样芽孢杆菌检测的研究热点,该检测方法的关键点是筛选获得特异性检测靶点[25]。已报道的特异性蜡样芽孢杆菌基因有16S rDNA、gyrB、motB、tuf和rpoB等[11-12,26-27],CAAMA O等[28]确定tuf基因是检测蜡样芽孢杆菌的特异性基因,还有研究采用蜡样芽孢杆菌的肠毒素基因如entFM、呕吐毒素基因如ces、非溶血性毒素nhe等进行分子生物学检测[29-30]。使用毒力基因作为特异性靶点进行检测存在一些问题,有些菌株可能不含有该毒力基因,造成出现假阴性检测结果;一些毒力基因如nhe和hbl[31]在苏云金芽孢杆菌中也存在,因此也会出现假阳性结果。本研究试图寻找更有效的新型特异性检测靶点基因。利用比较基因组学的方法,筛选获得蜡样芽孢杆菌ATCC 14579中的特异性基因,设计的引物经PCR验证,具有良好的特异性和较强的抗干扰能力,人工污染实验结果表明,牛肉和猪肉样品经人工污染蜡样芽孢杆菌增菌培养10 h后,均可以有效检测出蜡样芽孢杆菌。同时本研究中建立的检测方法可在12 h内完成对蜡样芽孢杆菌的检测。因此本研究建立的检测方法具有检测时间短、灵敏度高等优点,更适合大量食品样品的快速检测。
O等[28]确定tuf基因是检测蜡样芽孢杆菌的特异性基因,还有研究采用蜡样芽孢杆菌的肠毒素基因如entFM、呕吐毒素基因如ces、非溶血性毒素nhe等进行分子生物学检测[29-30]。使用毒力基因作为特异性靶点进行检测存在一些问题,有些菌株可能不含有该毒力基因,造成出现假阴性检测结果;一些毒力基因如nhe和hbl[31]在苏云金芽孢杆菌中也存在,因此也会出现假阳性结果。本研究试图寻找更有效的新型特异性检测靶点基因。利用比较基因组学的方法,筛选获得蜡样芽孢杆菌ATCC 14579中的特异性基因,设计的引物经PCR验证,具有良好的特异性和较强的抗干扰能力,人工污染实验结果表明,牛肉和猪肉样品经人工污染蜡样芽孢杆菌增菌培养10 h后,均可以有效检测出蜡样芽孢杆菌。同时本研究中建立的检测方法可在12 h内完成对蜡样芽孢杆菌的检测。因此本研究建立的检测方法具有检测时间短、灵敏度高等优点,更适合大量食品样品的快速检测。
本研究利用比较基因组学方法,以蜡样芽孢杆菌ATCC 14579基因组序列为模板,筛选得到11个特异性基因,设计引物进行PCR验证后最终确定基因gene_2625、gene_2626、gene_2627对蜡样芽孢杆菌的特异性较好,确定其相应引物的检测灵敏度分别为3.595 pg/μL、359.5 fg/μL和3.595 pg/μL。在牛肉和猪肉背景菌群的干扰下,蜡样芽孢杆菌的检出限为3.74×103 CFU/mL。经增菌培养10 h后,本研究所用的3对引物对人工污染的牛肉和猪肉样品中蜡样芽孢杆菌的检出限分别达到4.62和8.38 CFU/g。
[1] PIRHONEN T I, ANDERSSON M A, JAASKELAINE E L, et al. Biochemical and toxic diversity of Bacillus cereus in a pasta and meat dish associated with a food-poisoning case[J]. Food Microbiology, 2005, 22(1): 87-91.
[2] VALERO M, HEMANDEZ-HERRERO L A, FEMANDEZ P S, et al. Characterization of Bacillus cereus isolates from fresh vegetables and refrigerated minimally processed foods by biochemical and physiological tests[J]. Food Microbiology, 2002, 19(5): 491-499.
[3] HAN J Z, ZHAO S M, MA Z, et al. The antibacterial activity and modes of LI-F type antimicrobial peptides against Bacillus cereus in vitro[J]. Journal of Applied Microbiology, 2017, 123(3): 602-614.
[4] MOLS M, KRANENBURG R V, PELAARS M H, et al. Comparative analysis of transcriptional and physiological responses of Bacillus cereus to organic and inorganic acid shocks[J]. International Journal of Food Microbiology, 2010, 137(1): 13-21.
[5] TEWARI A, ABDULLAH S. Bacillus cereus food poisoning: International and Indian perspective[J]. Journal of Food Science and Technology, 2014, 52(5): 2 500-2 511.
[6] CETIN K H, NEWMAN M C. Antimicrobial efficacy of phytochemicals against Bacillus cereus in reconstituted infant rice cereal[J]. Food Microbiology, 2018, 2(69):189-195.
[7] BROUSSOLLE V, PANDIANI F, HADDAD N, et al. Insertional mutagenesis reveals genes involved in Bacillus cereus ATCC 14579 growth at low temperature[J]. FEMS Microbiology Letters, 2010, 306(2): 177-183.
[8] WALKLINGRIBEIRO M, RODR GUZGONZ
GUZGONZ LEZ O, JAYARAM S, et al. Microbial inactivation and shelf life comparison of ′cold′ hurdle processing with pulsed electric fields and microfiltration, and conventional thermal pasteurisation in skim milk[J]. International Journal of Food Microbiology, 2011, 144(3): 379-386.
LEZ O, JAYARAM S, et al. Microbial inactivation and shelf life comparison of ′cold′ hurdle processing with pulsed electric fields and microfiltration, and conventional thermal pasteurisation in skim milk[J]. International Journal of Food Microbiology, 2011, 144(3): 379-386.
[9] 曹飞扬, 王娉, 江连洲, 等. 蜡样芽孢杆菌分型方法研究进展[J]. 食品科学, 2017, 38(17): 286-290.
[10] 潘娜, 李薇薇, 杨淑香, 等. 中国2002-2015年学校食源性疾病暴发事件归因分析[J]. 中国学校卫生, 2018, 39(4): 570-572.
[11] 黄晶菁, 罗静, 何宏轩. 蜡样芽孢杆菌检测方法研究进展[J]. 中国畜牧兽医, 2018, 45(3): 635-642.
[12] 张志鸿, 许恒毅, 魏华. 基于PCR方法检测蜡样芽孢杆菌的研究进展[J]. 食品工业科技, 2013, 34(22): 335-338.
[13] LI Y, CHEN Q, JIANG H, et al. Novel development of a qPCR assay based on the rpoB gene for rapid detection of Cronobacter spp[J]. Current Microbiology, 2016, 72(4): 436-443.
[14] NAIR M, VENKITANARAYANAN K. Cloning and sequencing of the ompA gene of Enterobacter sakazakii and development of an ompA-targeted PCR for rapid detection of Enterobacter sakazakii in infant formula[J]. Applied and Environmental Microbiology, 2006, 72(4): 2 539-2 546.
[15] 翟立公, 王俊颖, 张小雨, 等. 甲型副伤寒沙门菌特异性基因筛选及PCR检测体系建立[J]. 食品科学, 2018, 39(2): 151-157.
[16] TAO T T, CHEN Q M, BIE X M, et al. Investigation on prevalence of Listeria spp. and Listeria monocytogenes in animal-derived foods by multiplex PCR assay targeting novel genes[J]. Food Control, 2017,73(PartB):704-711.
[17] DZIECIOL M, FRICKER M, WAGNER M, et al. A novel diagnostic real-time PCR assay for quantification and differentiation of emetic and non-emetic Bacillus cereus[J]. Food Control, 2013, 32(1): 176-185.
[18] MART NE-BLANCH J F, S
NE-BLANCH J F, S NCHEZ G, GARAY E, et al. Detection and quantification of viable Bacillus cereus in food by RT-qPCR[J]. European Food Research and Technology, 2011, 232: 951-955.
NCHEZ G, GARAY E, et al. Detection and quantification of viable Bacillus cereus in food by RT-qPCR[J]. European Food Research and Technology, 2011, 232: 951-955.
[19] 贾雅菁, 付博宇, 王羽, 等. 实时荧光环介导等温扩增技术检测牛乳中的蜡样芽孢杆菌[J]. 食品科学, 2016, 37(6): 184-189.
[20] CAWTHOM D M, BOTHA S, WITTHUHN R C. Evaluation of different methods for the detection and identification of Enterobacter sakazakii isolated from South African infant formula milks and the processing environment[J]. International Journal of Food Microbiology, 2008, 127(1-2): 129-138.
[21] TAO T T, CHEN Q M, BIE X M, et al. Mining of novel species-specific primers for PCR detection of Listeria monocytogenes based on genomic approach[J]. World Journal of Microbiology and Biotechnology, 2015, 31(12): 1 955-1 966.
[22] 陈启明. 克罗诺杆菌免疫学和分子生物学检测方法研究[D].南京:南京农业大学, 2016:102-103.
[23] PEI X Y, YANG S R, ZhAN L, et al. Prevalence of Bacillus cereus in powdered infant and powdered follow-up formula in China[J]. Food Control, 2018, 93: 101-105.
[24] RENTZEL H, THANH M D, KRAUSE G, et al. Quantification and differentiation of Bacillus cereus group species in spices and herbs by real-time PCR[J]. Food control, 2018, 83: 99-108.
[25] LIU B, ZHOU X J, ZHANG L D, et al. Development of a novel multiplex PCR assay for the identification of Salmonella enterica Typhimurium and Enteritidis[J]. Food Control, 2012, 27(1): 87-93.
[26] ARUNRUT N, KIATPATHOMCHAI W, ANANCHAIPATTANA C. Multiplex PCR assay and lyophilization for detection of Salmonella spp., Staphylococcus aureus and Bacillus cereus in pork products[J]. Food Science and Biotechnology, 2018, 27: 867-875.
[27] OLIWA-STASIAK K, KOLAJ-ROBIN O, AADLEY C C. Development of real-time PCR assays for detection and quantification of Bacillus cereus group species: Differentiation of B. weihenstephanensis and rhizoid B. pseudomycoides isolates from milk[J]. Applied and Environmental Microbiology, 2011, 77(1): 80-88.
[28] CAAMA O-ANTELO S, FERN
O-ANTELO S, FERN NDEZ NOIE, BÖHME K, et al. Genetic discrimination of foodborne pathogenic and spoilage Bacillus spp. based on three housekeeping genes[J]. Food Microbiology, 2015, 46: 288-298.
NDEZ NOIE, BÖHME K, et al. Genetic discrimination of foodborne pathogenic and spoilage Bacillus spp. based on three housekeeping genes[J]. Food Microbiology, 2015, 46: 288-298.
[29] WALKERYORKMOORE L, MOORE S C, FOX E M. Characterization of enterotoxigenic Bacillus cereus sensu lato and Staphylococcus aureus isolates and associated enterotoxin production dynamics in milk or meat-based broth[J]. Toxins, 2017, 9(7): 225.
[30] KIM M J, HAN J K, PARK J S, et al. Various enterotoxin and other virulence factor genes widespread among Bacillus cereus and Bacillus thuringiensis strains[J]. Journal of Microbiology and Biotechnology, 2015, 25(6): 872-879.
[31] KAMINSKA P S, YERNAZAROVA A, MURAWSKS E, et al. Comparative analysis of quantitative reverse transcription real-time PCR and commercial enzyme imunoassays for detection of enterotoxigenic Bacillus thuringiensis isolates[J]. FEMS Microbiology Letters, 2014, 357(1): 34-39.