S-腺苷甲硫氨酸(S-adenosylmethionine,SAM),是生物体内含硫氨基酸代谢的一个关键组成部分,由甲硫氨酸和三磷酸腺苷通过蛋氨酸腺苷转移酶合成[1]。SAM在细胞中主要参与转甲基化[2]、转硫化[3]和脂肪多胺合成[4],被视为生物体中的关键代谢物,常用于预防和改善肝病、关节炎、抑郁症、阿尔茨海默氏症等多种疾病[5-7]。欧美国家已将其批准用作膳食补充剂[8]。因此,开发SAM相关产品具有重要的意义。
微生物发酵法是生产SAM的主要方法。早期的研究普遍通过添加大量甲硫氨酸前体物来实现SAM的高效合成,产量可达10 g/L以上[9],但甲硫氨酸添加量大、成本高,且转化率低,导致SAM价格昂贵[10]。进而有研究者以廉价糖类碳源从头合成SAM,以期降低发酵成本[11]。然而,目前从头合成SAM的产量仍较低,尚需开展深入研究。课题组前期通过解淀粉芽胞杆菌从头合成SAM,通过途径耦合代谢工程策略强化了SAM的合成[12],该法在SAM的从头合成中具有重要的应用潜力,但仍需开展大量研究工作。
解淀粉芽胞杆菌可大量合成两亲性脂肽类表面活性剂副产物[13],不仅消耗大量的能量,而且这些表面活性剂可导致发酵过程起泡严重,影响发酵过程[14]。芽胞杆菌产生的脂肽类表面活性剂主要包括iturin和surfactin,其中iturin主要由ituDABC操纵子基因编码[15-16],surfactin主要由srfA-ABCD操纵子基因编码[17-18]。通过基因敲除技术阻断iturin和surfactin的合成有望促进SAM的合成,然而目前还未见相关报道。因此,本研究通过基因敲除技术敲除解淀粉芽孢杆菌中的ituD和SrfAC基因,阻断iturin和surfactin的合成,以期降低发酵过程脂肽类副产物和发酵泡沫,以期提高SAM产量。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒
本研究所用到的菌株和质粒如表1和表2所示。其中大肠杆菌DH5α用于构建载体。
表1 实验用菌株
Table 1 Strains used in this study

菌株基因型或表型来源大肠杆菌(Escherichia coli)DH5αF-Φ80d/lacZΔM15,Δ(lacZYA-argF) U169, recA1, endA1, hsdR17(rK-, mK+),phoA, supE44, λ-, thi-1, gyrA96, relA1本实验室保存解淀粉芽胞杆菌(Bacillusamyloliquefaciens) HZ-12CCTCC M2015234, 野生菌本实验室保存HSAM4HZ12整合酿酒酵母SAM2基因、 E.coli metA和metB基因,敲除mccA、sucC和thrB基因本实验室保存HSAM4ΔituDHSAM4菌株中缺失ituD基因本研究HSAM4ΔsrfACHSAM4菌株中缺失srfAC基因本研究HSAM4ΔituDΔsrfACHSAM4菌株中缺失ituD和srfAC基因本研究
表2 实验用质粒
Table 2 Plasmids used in this study

质粒基因型或表现型来源T2(2)-oriE. coli-B. amyloliquefaciens穿梭质粒,温度敏感型质粒,orits,Kanr本实验室保存T2(2)-oriΔsrfACT2(2)-ori含有敲除srfAC基因所需的同源臂序列,orits,Kanr本实验室保存T2(2)-oriΔituDT2(2)-ori含有敲除ituD基因所需的同源臂序列,orits,Kanr本研究
注:Tetr:四环素抗性;Kanr:卡那霉素抗性;orits:温敏型复制子
1.1.2 PCR引物
本研究所用的主要引物见表3。
表3 本研究所使用的PCR引物
Table 3 Primers used for PCR in this study

引物名称引物序列(5′→3′)T2-FATGTGATAACTCGGCGTAT2-RGCAAGCAGCAGATTACGCΔituD-AFGGACTAGTTCTGAACGGCTGTTTTTTTATΔituD-ARCCTGGGCGCTTTATTTTAATATTGAACAGAAATACAATATCCTΔituD-BFAGGATATTGTATTTCTGTTCAATATTAAAATAAAGCGCCCAGGΔituD-BRGCTCTAGATCAGGTTCATAAAACGAAGCΔituD-YFTGTCAGTGAAGGAAATCGCTΔituD-YRCTTTCGAATGAACGGCTAGΔsrfAC-YFATACAATGCCCTGCGAGAAΔsrfAC-YRCGGCGGGAACGCAAACAGT
注:粗体为限制性内切酶酶切位点,划线为重叠延伸PCR (SOE-PCR)的重叠区域
1.1.3 主要培养基
SAM发酵培养基(g/L):蔗糖80,蛋白胨10,玉米浆5,天冬氨酸3,尿素2,NaCl 2.5,(NH4)2SO4 6.3,KH2PO4 3,MgSO4·7H2O 4.2,pH 6.5。
LB培养基(g/L):蛋白胨10.0,酵母浸出粉5.0,NaCl 10.0,pH 7.2,固体培养基加琼脂1.5%。
1.1.4 工具酶和试剂
DNA抽提试剂盒,武汉楚诚正茂科技工程有限公司;TransStartR FastPfu DNA聚合酶和TransStartR easyTaq DNA聚合酶,北京全式金公司;DNA限制性内切酶、T4 DNA连接酶,DNA回收试剂盒和质粒抽提试剂盒,美国OmegaBio-Tek公司;DL5000 Marker,TaKaRa公司;琼脂糖,Spanish公司;Kan、Tet、溶菌酶、山梨醇、甘露醇等试剂等均为进口分装,武汉楚诚正茂科技工程有限公司,biosharp;甲醇为色谱级,其他主要生化试剂均为国产分析级纯或进口分装。
1.1.5 仪器与设备
Agilent Technologies 1260高效液相色谱,美国Agilent公司;Thermal Cycler (My Cycler) PCR仪,美国 Bio-Rad 公司;DYY-8C型电泳仪,北京六一仪器厂;CR21G高速冷冻离心机,日本Hitachi公司;HQL300B恒温大幅振荡摇床,武汉中科科仪技术发展有限责任公司;SKP-02.420电热恒温培养箱,黄石市恒丰医疗器械有限公司;Legend Micro17R高速冷冻离心机,美国Thermo Fisher Scientific 公司。
1.2 实验方法
1.2.1 敲除载体的构建
以ituD基因为例,选取B. amyloliquefaciens HZ-12的基因组DNA作为模板,利用上游同源臂引物(ΔituD-AF, ΔituD-AR)和下游同源臂引物(ΔituD-BF,ΔituD-BR)分别扩增出同源臂序列A和B,产物纯化回收后通过SOE-PCR用引物ΔituD-AF、ΔituD-BR将A和B连接起来,并与T2(2)-ori载体同时用限制性内切酶SpeI和XbaI进行双酶切,回收后用T4 DNA连接酶连接,采用CaCl2转化法转化到E.coli DH5α感受态细胞。钙转化成功后提取质粒进行双酶切验证并送往测序公司测序,构建成功的敲除质粒命名为T2(2)-oriΔituD。
1.2.2 基因敲除菌株的构建[12]
以ituD基因为例,将构建成功的质粒电转化入HSAM4感受态细胞中,涂布至含Kan的抗性平板,37 ℃静置培养16~18 h。挑选转化子单菌落于Kan平板上划线,培养8~12 h后进行PCR菌落验证(引物T2-F,T2-R)。将电转正确的菌株在45 ℃下进行单交换,ΔituD-YF/YR引物和T2引物验证;获得的单交换菌株在37 ℃下进行双交换,挑选在LB板上长而在Kan抗性板上不长的单菌落,用ΔituD-YF和ΔituD-YR验证,筛选到敲除菌株,命名为HSAM4ΔituD。
1.2.3 发酵培养条件
从超低温冰箱中取出保藏的菌种划线LB平板培养基中,37 ℃培养基12 h。挑取适量菌体于装有50 mL LB液体培养基的250 mL三角瓶中,37 ℃,180 r/min培养9 h。吸取体积分数1.5%的菌液接种于装有25 mL发酵培养基的250 mL三角瓶中,37 ℃、180 r/min发酵培养,每个菌设置3个摇瓶重复。
1.2.4 SAM液相检测[12]
取发酵液0.5 mL,加1.5 mL 0.4 mol/L高氯酸,提取1 h(每15 min振荡1次),10 000 r/min离心5 min。取800 μL上清液,加100 μL,2 mol/L NaOH调pH。利用HPLC检测,色谱柱为XDB-C18(规格:5 μm,4.6 mm×150 mm),检测器为紫外检测器,流动相为V(甲醇)∶V(40 mmol/L NH4H2PO4 2 mmol/L庚烷磺酸钠)=18∶82,流速为0.8 mL/min,进样量为20 μL,柱温30 ℃,检测波长为254 nm。
1.2.5 数据统计分析
每组实验设计3个重复,采用SPSS 20.0进行数据统计分析,评估数据差异的显著性,计算出平均值和标准偏差,采用Origin 8.5进行图表绘制。
2 结果与分析
2.1 ituD基因缺失对解淀粉芽孢杆菌合成SAM的影响
2.1.1 ituD基因缺失菌株的构建
利用1.2.1小节中所述的方法构建重组载体T2(2)-oriΔituD,利用表3中相应的引物ΔituD-AF/AR,ΔituD-BF/BR扩增用于构建T2(2)-oriΔituD的上下游同源臂。敲除ituD基因的上下游同源臂大小分别为513和504 bp,上下游同源臂经SOE-PCR融合后的产物大小为1 017 bp,上下游同源臂SOE-PCR融合片段与T2(2)-ori质粒经酶切和酶连后转化至E.coli DH5α,选取验证正确的阳性转化子,提取质粒并且测序结果正确无误,表明敲除质粒T2(2)-oriΔituD构建成功。
将已构建完成的重组质粒T2(2)-oriΔituD电转化至HASM4中,筛选出阳性转化子,并按照1.2.2的方法进行单交换和双交换筛选突变株。经菌落PCR验证正确的双交换菌株,提取基因组DNA,并利用表3中相应的引物ΔituD-YF/YR验证原始菌株HSAM4和阳性交换突变株,片段大小分别为2 508、1 290 bp,两片段相差的长度正好是ituD基因的长度,PCR验证电泳图如图1所示。且纯化回收阳性交换突变株PCR产物,经测序确认碱基正确无误,说明缺失ituD基因的菌株HSAM4ΔituD构建成功。
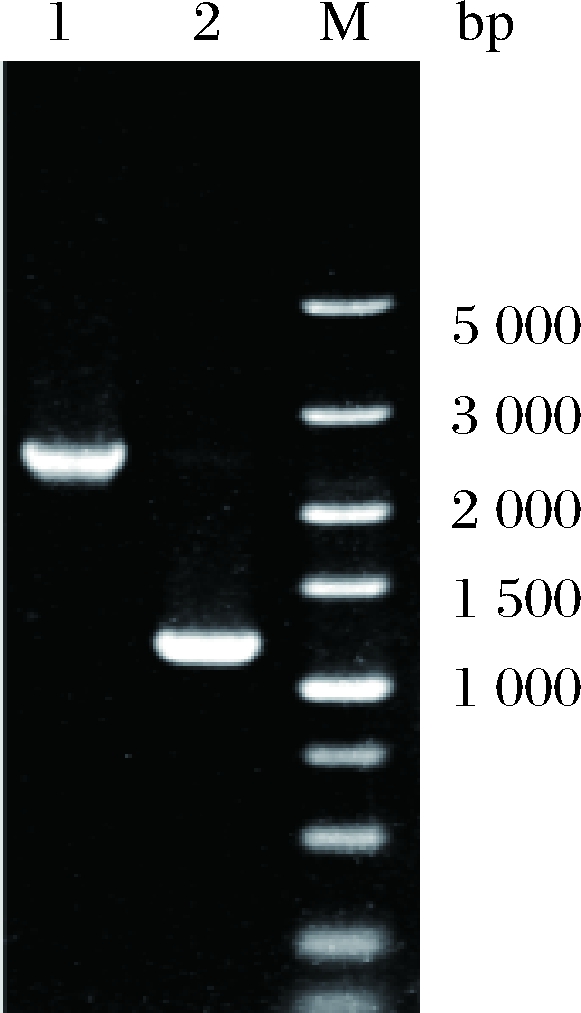
1-ΔituD-YF 和ΔituD-YR PCR验证HSAM4(2 508 bp);
2-ΔituD-YF 和ΔituD-YR PCR验证HSAM4ΔituD(1 290 bp)
图1 HSAM4ΔituD菌株的PCR验证电泳图
Fig.1 Electrophoretogram of PCR validation for
HSAM4ΔituD
2.1.2 ituD基因缺失对SAM合成的影响
为了考察ituD基因的缺失对B. amyloliquefaciens HSAM4合成SAM的影响,按照1.1.3和1.2.3小节的发酵培养基和培养条件对工程菌株HSAM4ΔituD进行发酵验证,发酵周期为60 h。发酵结束后取样检测SAM产量和菌体生物量(OD600),结果如图2所示。

图2 缺失ituD基因对SAM合成的影响
Fig.2 Effect of ituD knockout on SAM fermentation
由图2可以看出,原始菌 HSAM4与HSAM4ΔituD的生物量无显著差异,说明ituD基因缺失对菌体生长无影响。此外,缺失菌株HSAM4ΔituD的SAM产量与原始菌 HSAM4相比,没有显著性提高。上述结果说明敲除ituD基因对菌体生长和产物的合成未产生显著性影响。
2.2 srfAC基因缺失对解淀粉芽孢杆菌合成SAM的影响
2.2.1 srfAC基因缺失菌株的构建
敲除质粒T2(2)-oriΔsrfAC来自本实验室保存。将T2(2)-oriΔsrfAC电转化至HASM4中,筛选出阳性转化子,并按照1.2.2的方法进行单交换和双交换筛选突变株。经菌落PCR验证正确的双交换菌株,提取基因组DNA,并利用表3中相应的引物ΔsrfAC-YF/YR验证原始菌株HSAM4和阳性交换突变株,片段大小分别为 2 704和1 657 bp,两片段相差的长度正好是srfAC基因的长度,PCR验证电泳图如图3所示。纯化回收阳性交换突变株PCR产物,经测序确认碱基正确无误,说明缺失srfAC基因的菌株HSAM4ΔsrfAC构建成功。
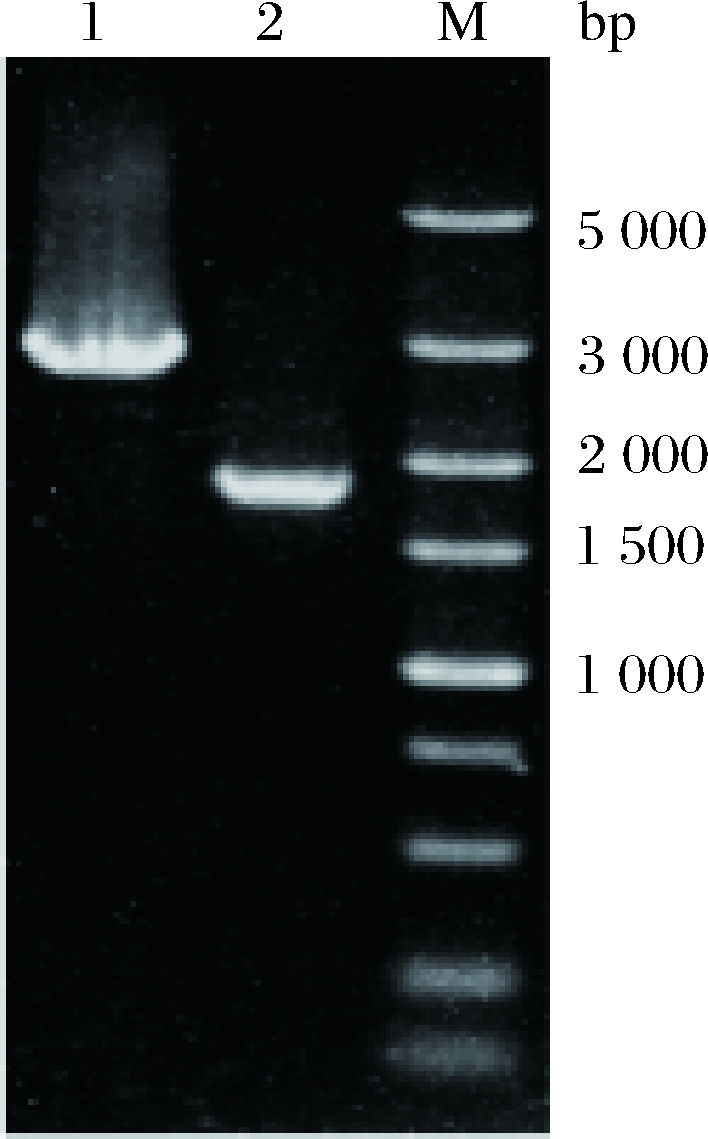
1-ΔsrfAC-YF 和ΔsrfAC-YR PCR验证HSAM4(2 704 bp);
2-ΔsrfAC-YF 和ΔsrfAC-YR PCR验证HSAM4ΔsrfAC(1 657 bp)
图3 HSAM4ΔsrfAC菌株的PCR验证电泳图
Fig.3 Electrophoretogram of PCR validation for
HSAM4ΔsrfAC
2.2.2 srfAC 基因缺失对SAM合成的影响
为了考察 srfAC 基因的缺失对B. amyloliquefaciens HSAM4合成SAM的影响,按照1.1.3和1.2.3小节的发酵培养基和培养条件对工程菌株HSAM4ΔsrfAC进行发酵验证,发酵周期为60 h。发酵结束后取样检测SAM产量和菌体生物量(OD600),结果如图4所示。
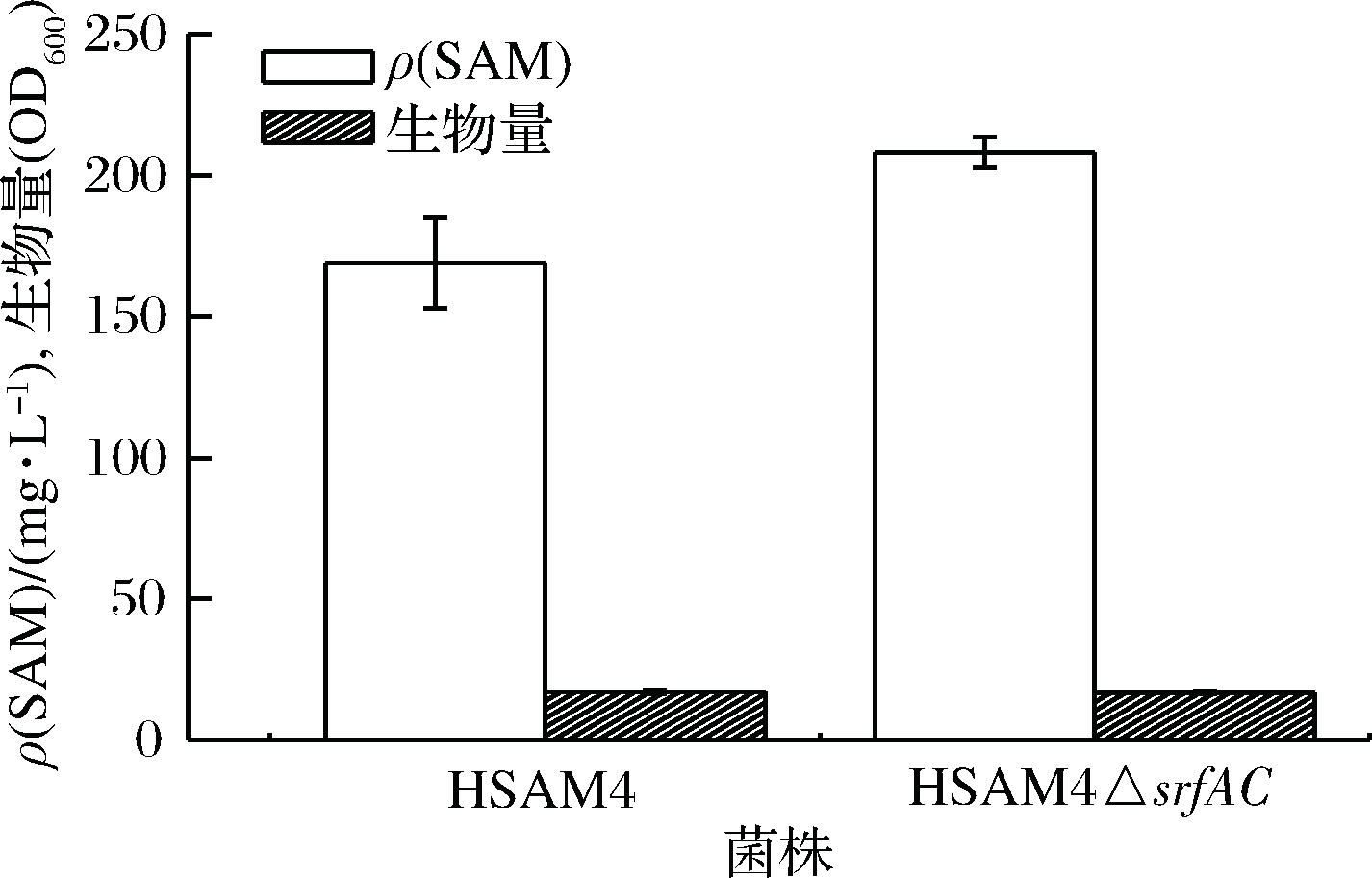
图4 缺失srfAC基因对SAM合成的影响
Fig.4 Effect of srfAC knockout on SAM fermentation
由图4可以看出,工程菌 HSAM4ΔsrfAC的生物量相比于原始菌没有发生显著性变化。缺失菌株 HSAM4ΔsrfAC的SAM 产量为 208.24 mg/L,比原始菌HSAM4提高了23%,差异显著。推测其原因,srfAC基因编码surfactin合成酶[17],影响脂肽的环化与释放,减少了表面活性剂surfactin的合成,可降低副产物和发酵产泡现象,从而促进SAM的合成。
2.3 ituD基因和srfAC基因双缺失对解淀粉芽孢杆菌合成SAM的影响
2.3.1 ituD基因和srfAC基因双缺失菌株的构建
将T2(2)-oriΔsrfAC电转化至HASM4ΔituD中,筛选出阳性转化子,并按照1.2.2的方法进行单交换和双交换筛选突变株。经菌落PCR验证正确的双交换菌株,提取基因组DNA,并利用表3中相应的引物ΔituD-YF/YR和ΔsrfAC-YF/YR验证原始菌株HSAM4和阳性交换突变株,与原始菌相比分别相差ituD基因和srfAC基因的长度,PCR验证电泳图如图5所示。纯化回收阳性交换突变株PCR产物,经测序确认碱基正确无误,说明缺失ituD和srfAC基因的菌株HSAM4ΔituDΔsrfAC构建成功。
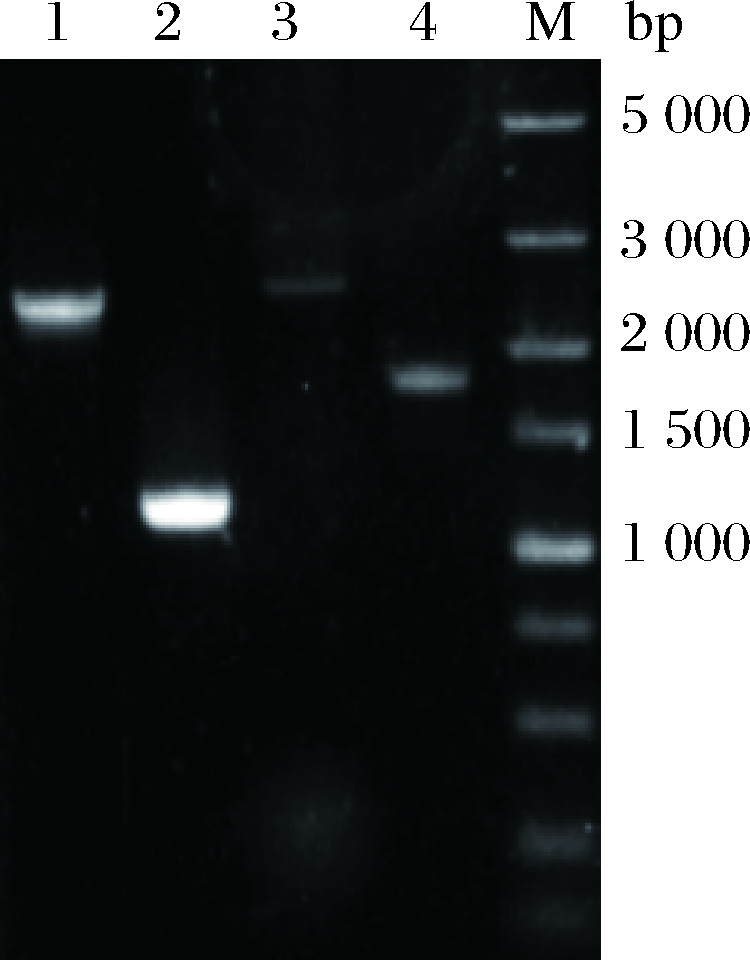
1-ΔituD-YF 和ΔituD-YR PCR验证HSAM4(2 508 bp);
2-ΔituD-YF 和ΔituD-YR PCR验证HSAM4ΔituDΔsrfAC(1 290 bp);
3-ΔsrfAC-YF 和ΔsrfAC-YR PCR验证HSAM4(2 704 bp);
4-ΔsrfAC-YF 和ΔsrfAC-YR PCR
验证HSAM4ΔituDΔsrfAC(1 657 bp)
图5 HSAM4ΔituDΔsrfAC菌株的PCR验证电泳图
Fig.5 Electrophoretogram of PCR validation for
HSAM4ΔituDΔsrfAC
2.3.2 ituD基因和srfAC基因双缺失对SAM合成的影响
按照1.1.3和1.2.3小节的发酵培养基和培养条件对工程菌株HSAM4ΔituDΔsrfAC进行发酵验证,发酵周期为60 h。发酵结束后取样检测 SAM 产量和菌体生物量(OD600),结果如图6所示。

图6 缺失ituD,srfAC基因对SAM合成的影响
Fig.6 Effect of ituD,srfAC knockout on SAM
fermentation
由图6可以看出,工程菌HSAM4ΔituDΔsrfAC的生物量相比于原始菌 HSAM4无明显变化,说明 ituD和srfAC基因双缺失对菌体的生长无影响。缺失菌株HSAM4ΔituDΔsrfAC的SAM的产量达222.95 mg/L,相比于原始菌 HSAM4提高了32%。这可能是敲除ituD基因和srfAC基因,同时阻断了iturin和surfactin的合成[15,17],降低发酵副产物和发酵产泡现象,进一步提高了SAM产量。
3 结论
S-腺苷甲硫氨酸是存在于生物体中的必需功能因子[19]。在不添加甲硫氨酸的前提下,通过廉价碳源实现SAM的高效从头合成具有重要的意义。解淀粉芽胞杆菌在从头合成SAM的过程中显示了初步的效果[12],为有效提高SAM的从头合成产量,本文基于基因敲除技术,成功构建了ituD 基因缺失菌株HSAM4ΔituD,srfAC 基因缺失菌株HSAM4ΔsrfAC,ituD、srfAC基因双缺失菌株 HSAM4ΔituDΔsrfAC。SAM发酵结果显示,缺失ituD、srfAC基因对菌株生长几乎无影响,双缺失菌株HSAM4ΔituDΔsrfAC的SAM产量可提高至原始菌株HSAM4的1.32倍。
ZHANG等[20]研究发现,利用Bacillus subtilis ATCC 6051a发酵生产环糊精糖基转移酶过程中,敲除该菌株的srfAC基因可有效控制发酵过程中的泡沫。Iturin和surfactin作为两亲性脂肽类表面活性物质,是液体深层发酵产泡的重要原因。由此推测,本研究ituD和srfAC基因的缺失,阻断了iturin和surfactin的产生,不仅可减少发酵过程中副产物的生成及不必要的能量消耗,而且可通过降低表面活性剂改善发酵产泡现象,这可能是SAM产量提高的主要原因。本研究为SAM从头合成的代谢工程育种提供了新的思路和方向。
[1] PANZA F, FRISARDI V, CAPURSO C, et al. Polyunsaturated fatty acid and S-adenosylmethionine supplementation in predementia syndromes and Alzheimer's disease: a review[J]. The Scientific World Journal, 2009, 9: 373-389.
[2] KRUGLOVA M P, GRACHEV S V, BULGAKOVA P O, et al. Low S-adenosylmethionine/S-adenosylhomocysteine ratio in urine is associated with chronic kidney disease[J]. Laboratory Medicine, 2020, 51(1): 80-85.
[3] SINNER E K, LICHSTRAHL M S, LI R, et al. Methylations in complex carbapenem biosynthesis are catalyzed by a single cobalamin-dependent radical S-adenosylmethionine enzyme[J]. Chemical Communications, 2019, 55(99): 14 934-14 937.
[4] CAMPONESCHI F, MUZZIOLI R, CIOFI-BAFFONI S, et al. Paramagnetic 1H NMR spectroscopy to investigate the catalytic mechanism of radical S-adenosylmethionine enzymes[J]. Journal of Molecular Biology, 2019, 431(22): 4 514-4 522.
[5] RAIKHELSON K L, KONDRASHINA E A. Аdеmethionine in the treatment of fatigue in liver diseases: a systematic review[J]. Terapevticheskii Arkhiv, 2019, 91(2): 134-142.
[6] LANDGRAF B J, MCCARTHY E L, BOOKER S J. Radical S-adenosylmethionine enzymes in human health and disease[J]. Annual Review of Biochemistry, 2016, 85: 485-514.
[7] MART NEZ-U
NEZ-U A M, VARELA-REY M, MESTRE D, et al. S-Adenosylmethionine increases circulating very-low density lipoprotein clearance in non-alcoholic fatty liver disease[J]. Journal of Hepatology, 2015, 62(3): 673-681.
A M, VARELA-REY M, MESTRE D, et al. S-Adenosylmethionine increases circulating very-low density lipoprotein clearance in non-alcoholic fatty liver disease[J]. Journal of Hepatology, 2015, 62(3): 673-681.
[8] MATO J M, MART NEZ-CHANTAR M L, LU S C. S-adenosylmethionine metabolism and liver disease[J]. Annals of Hepatology, 2015, 12(2): 183-189.
NEZ-CHANTAR M L, LU S C. S-adenosylmethionine metabolism and liver disease[J]. Annals of Hepatology, 2015, 12(2): 183-189.
[9] KANAI M, MIZUNUMA M, FUJII T, et al. A genetic method to enhance the accumulation of S-adenosylmethionine in yeast[J]. Applied Microbiology and Biotechnology, 2017, 101(4): 1 351-1 357.
[10] CHU J, QIAN J, ZHUANG Y, et al. Progress in the research of S-adenosyl-L-methionine production[J]. Applied Microbiology and Biotechnology, 2013, 97(1): 41-49.
[11] CHEN Y, XU D, FAN L, et al. Manipulating multi-system of NADPH regulation in Escherichia coli for enhanced S-adenosylmethionine production[J]. RSC Advances, 2015, 5(51): 41 103-41 111.
[12] RUAN L, LI L, ZOU D, et al. Metabolic engineering of Bacillus amyloliquefaciens for enhanced production of S-adenosylmethionine by coupling of an engineered S-adenosylmethionine pathway and the tricarboxylic acid cycle[J]. Biotechnology for Biofuels, 2019, 12(1): 211.
[13] 卢彩鸽, 张殿朋, 刘霆, 等. 解淀粉芽胞杆菌 MH71 的生防活性及脂肽类抗生素基因检测[J].植物保护, 2015, 41(3): 12-18.
[14] HOEKS F W, VAN WEES-TANGERMAN C, LUYBEN K C A M, et al. Stirring as foam disruption (SAFD) technique in fermentation processes[J]. The Canadian Journal of Chemical Engineering, 1997, 75(6): 1 018-1 029.
[15] 郭丽清. 苏云金芽孢杆菌中脂肽类抗生素 Iturins 的研究[D].福州:福建农林大学, 2010.
[16] LE ES F L, VELHO R V, CALDAS D G G, et al. Influence of pH and temperature on the expression of sboA and ituD genes in Bacillus sp. P11[J]. Antonie Van Leeuwenhoek, 2013, 104(1): 149-154.
ES F L, VELHO R V, CALDAS D G G, et al. Influence of pH and temperature on the expression of sboA and ituD genes in Bacillus sp. P11[J]. Antonie Van Leeuwenhoek, 2013, 104(1): 149-154.
[17] 刘丽霞. Surfactin合成酶相关基因研究[D].南京:南京农业大学, 2012.
[18] WANG X, CHEN Z, FENG H, et al. Genetic variants of the oppA gene are involved in metabolic regulation of surfactin in Bacillus subtilis[J]. Microbial Cell Factories, 2019, 18(1): 141.
[19] YAN L, LIANG X, HUANG H, et al. S-adenosylmethionine affects cell cycle pathways and suppresses proliferation in liver cells[J]. Journal of Cancer, 2019, 10(18): 4 368-4 379.
[20] ZHANG K, DUAN X, WU J. Multigene disruption in undomesticated Bacillus subtilis ATCC 6051a using the CRISPR/Cas9 system[J]. Scientific Reports, 2016, 6(1): 1-11.