乳酸菌因发酵糖类产生大量乳酸而得名,是一类不产孢子,过氧化氢酶促反应为阴性,培养条件为厌氧或兼性厌氧的原核微生物[1-3]。部分乳酸菌通过产生乳酸、醋酸、苯乳酸、细菌素等代谢产物而显示出抗菌活性,可抑制致病菌和腐败菌的繁殖,防止食品发生腐败变质,通常被用作食品的天然生物防腐剂[4-5],在乳制品、泡菜等发酵食品中有着悠久的应用历史。
乳酸乳球菌是乳酸菌属中重要成员之一,由于基因组相对较小,在基因工程及代谢方面已成为理想的模式菌株[6]、微生物细胞工厂[7]、口服疫苗传递载体[8]、异源蛋白质生产的宿主[9]。近年来,利用分子生物学手段可以在乳酸乳球菌中过表达外源基因,赋予宿主新的生物特性。在乳酸菌的表达系统中,外源蛋白的表达离不开启动子的调控。启动子是基因表达重要的调控元件,决定了基因表达的强度和时机,启动子的强弱,决定了蛋白质表达量的多少[10]。启动子根据作用方式分为不同的类型:一种是不受调节因子或生长条件限制,可持续表达的组成型启动子;一种是驱动细胞适应外界环境进行基因表达,需要添加诱导物的诱导型启动子[7]。随着现代生物技术的发展,人工合成启动子的出现实现了对代谢工程更精细的调控且有助于基因低水平的表达[11]。
本文以增强型绿色荧光蛋白(enhance green fluorescent protein,eGFP)基因为报告基因,将人工合成启动子P11、组成型启动子P23、P32、P44分别克隆至报告质粒中,利用电转化得到携带目标质粒的阳性转化子,接着利用酶标仪对各启动子调控的eGFP进行表达强度的定量分析,以此表征各启动子的转录活性,从中筛选出乳酸乳球菌中具有较强转录活性的启动子。
1 材料与方法
1.1 菌株、质粒及培养条件
本文所用菌株、质粒见表1。LB(Luria-Bertani)培养基用于大肠杆菌(Escherichia coli)的培养,配方(g/L)如下:胰蛋白胨10、酵母浸出粉5、NaCl 10、琼脂粉15。乳酸乳球菌的培养基为GM17培养基,配方( g/L):胰蛋白胨 5、大豆蛋白胨5、牛肉膏5、酵母浸出粉2.5、β-磷酸甘油二钠19、MgSO4·7H2O 0.25、葡萄糖5、琼脂粉(Agar)15。上述培养基若为液体培养基,则不加琼脂粉。筛选标记为红霉素(erythromycin),大肠杆菌工作质量浓度为300 μg/mL;乳酸乳球菌工作质量浓度为10 μg/mL。复苏培养基:含2 mmol/L CaCl2和20 mmol/L MgCl2的GM17液体培养基。20 mmol/L Tris-HCl (pH 7.4):2.432 g Tris溶于1 L灭菌去离子水中,用HCl调节pH至7.4。
表1 菌株和质粒
Table 1 Strains and plasmids

菌株或质粒特性来源菌株 E.coli Top 10克隆宿主细胞本实验室 L. lactis NZ9000乳酸乳球菌,转化宿主本实验室 L. lactis NZ9000-pIB184-eGFPpIB184-eGFP电转至L. lactis NZ9000本研究 L. lactis NZ9000-pLL06pLL06电转至L. lactis NZ9000本研究 L. lactis NZ9000-pLL07pLL07电转至L. lactis NZ9000本研究 L. lactis NZ9000-pLL37pLL37电转至L. lactis NZ9000本研究 L. lactis NZ9000-pLL15pLL15电转至L. lactis NZ9000本研究质粒 pIB184Emr, P23, 大肠杆菌-乳酸菌穿梭质粒江南大学食品学院 pMG36eEmr, P32, 大肠杆菌-乳酸菌穿梭质粒江南大学食品学院 pNZ44Cmr, P44, 大肠杆菌-乳酸菌穿梭质粒江南大学食品学院 pLpCas9-0537RepA101(Ts), Kanr, P11, RepD, RepE and Emr from pIB184本实验室 pIB184-eGFPEmr, pIB184含有eGFP编码序列本实验室 pLL06Emr, pIB184-eGFP, P32替换P23启动子本研究 pLL07Emr, pIB184-eGFP, P44替换P23启动子本研究 pLL37Emr, pIB184-eGFP, P11替换P23启动子本研究 pLL15pIB184-eGFP无P23启动子本研究
1.2 主要试剂及仪器
PCR高保真酶PrimeSTAR HS(Premix)、Premix TaqTM、限制性内切酶Pst I、BamH I、Apa I、EcoR I、Bgl II及T4 DNA Polymerase、T4 DNA Ligase、DNA Marker,Takara宝生物工程(大连)有限公司;ClonExpress II One Step Cloning Kit,南京诺唯赞生物科技有限公司;KOD FX Neo,日本Toyobo公司;割胶回收试剂盒、质粒DNA提取试剂盒,Axygen公司;红霉素、琼脂糖、Tris,生工生物工程(上海)股份有限公司;其他试剂,国药集团,分析纯。利用Vector NTI设计引物,由生工生物工程(上海)股份有限公司合成,引物序列见表2。
表2 引物序列
Table 2 Primer sequence

引物名称引物序列(5’-3’)pIB184-P11-FCTCTTGGTCGTCAGACTGATGGGCCCAGCGCTATAGTTGTTGACAGAA(Apa I)P11-eGFP-RTCCTCGCCCTTGCTCACCATGGATCCATATATCATAGTATGTCCATTCTGTCeGFP-P11-FATGGACATACTATGATATATGGATCCATGGTGAGCAAGGGCGAGGAeGFP-RTCGATAGATCTCGAGCTCTAGAATTCTTACTTGTACAGCTCGTCCAT(EcoR I)P32-FAATCGATAAGCTTGGCTGCAGGAATTCGGTCCTCGGGATATG(Pst I)P32-RGCCCTTGCTCACCATGGATCCGTCGACTCTAGAGGATCGATCCC(BamH I)P44-FAATCGATAAGCTTGGCTGCAGAGATCTAACAATTGTAACCCATCCAG(Pst I)P44-RGCCCTTGCTCACCATGGATCCTTTGGCCATAAAAGCGCCT(BamH I)pIB184-eGFP-FTGCGTTATCCCCTGATTCTGTGpIB184-eGFP-RTTACTTGTACAGCTCGTCCATGCC
注:下划线代表酶切位点
S1000TM Thermal Cycler PCR仪、Powerpac basic凝胶电泳仪、凝胶成像仪、MicroPulser电击转化仪、2 mm电转杯,美国Bio-Rad公司;超微量核酸蛋白定量仪Nanodrop 2000/2000c,Thermo Fisher Scientific公司;高速冷冻离心机,德国Sigma公司;Spectra Maxi3x酶标仪,美谷分子仪器(上海)有限公司。
1.3 实验方法
1.3.1 E. coli Top10感受态细胞的制备
(1)取冻藏于-80 ℃冰箱的甘油管E. coli Top10划线于LB固体平板,37 ℃培养箱静置培养12~16 h,单菌落状态直径为1~2 mm。
(2)挑取单菌落,接种至装有4 mL LB液体培养基的试管中,于37 ℃下200 r/min摇床振荡培养12~16 h。
(3)以1%的接种量接种至装有50 mL LB液体培养基的250 mL锥形瓶中,于37 ℃下200 r/min摇床振荡培养至OD600值为0.3~0.5。转移至50 mL离心管中,冰上静置10 min。于4 ℃下4 500 r/min离心10 min,收集菌体,弃上清液。
(4)用预冷的0.1 mol/L CaCl2溶液重悬洗涤细胞2次,4 ℃下4 500 r/min离心2~5 min,收集菌体,弃上清液。重悬于预冷的0.1 mol/L CaCl2-10%甘油溶液中。每100 μL感受态细胞分装于预冷的1.5 mL EP管中,冻藏于-80 ℃冰箱中,备用。
1.3.2 表达质粒的构建
以pMG36e、pNZ44质粒为模板,P32-F/R,P44-F/R为引物,分别进行PCR扩增,获得启动子片段P32、P44。以pLpCas9-0537、pIB184-eGFP为模板,pIB184-P11-F、P11-eGFP-R,eGFP-P11-F、eGFP-R为引物,分别扩增获得片段P11、eGFP。再以片段P11、eGFP为模板,pIB184-P11-F、eGFP-R为引物进行overlap PCR,获得片段eGFP-P11。PCR反应程序如下:95 ℃预变性3 min,98 ℃变性10 s,55 ℃退火15 s(PCR扩增用高保真酶PrimeSTAR HS;若PCR验证用Premix TaqTM,退火时间为30 s),72 ℃延伸1 min/kb;变性至延伸循环30次,72 ℃总延伸10 min。
用Pst I/BamH I、Apa I/EcoR I分别双酶切pIB184-eGFP骨架,将酶切后的DNA片段进行1%琼脂糖凝胶电泳,割胶回收6 565、5 880 bp片段,获得线性化载体;启动子片段P32、P44,eGFP-P11分别与Pst I/BamH I、Apa I/EcoR I双酶切后的线性化载体骨架经Clon Express II One Step Cloning Kit无缝克隆连接,以构建带有不同启动子的质粒。
经Pst I/BamH I双酶切线性化载体pIB184-eGFP,70 ℃金属浴5 min,使限制性内切酶失活,冷却至室温后加入dNTPs、T4 DNA Polymerase,37 ℃保温40~60 min,割胶回收,T4 DNA Ligase连接,16 ℃金属浴4~5 h,以构建不含启动子的空载对照。
将以上连接产物及对照分别转化至100 μL E. coli Top10感受态细胞,冰上静置30 min,42 ℃水浴45~90 s,冰浴2~5 min后加入37 ℃预热的900 μL LB液体培养基,37 ℃下200 r/min摇床复苏1 h,涂布于含有红霉素的LB固体培养基,37 ℃静置培养 12~24 h。挑取转化子进行菌落PCR验证,分别用引物P32-F/R,P44-F/R,pIB184-P11-F、eGFP-R,pIB184-eGFP-F/R验证pLL06、pLL07、pLL37、pLL15。验证正确的转化子接种于液体LB+Em培养基扩大培养,37 ℃下200 r/min摇床培养12~16 h,按照Axygen公司质粒DNA提取试剂盒中说明书抽提质粒,进一步酶切验证。挑选酶切片段正确的质粒,送至上海生工生物工程有限公司测序。以上质粒分别命名为pLL06(携带P32启动子)、pLL07(携带P44启动子)、pLL37(携带P11启动子)、pLL15(无启动子)。
1.3.3 乳酸乳球菌的转化
将pLL06、pLL07、pLL37、pLL15及pIB184-eGFP分别电转至乳酸乳球菌NZ9000感受态细胞中,每100 μL细胞中转化200 ng左右的质粒,感受态细胞制备及转化方法参照文献[12]进行,复苏后涂布于含有红霉素的GM17固体平板,30 ℃静置培养24~36 h后挑取转化子进行菌落PCR验证,分别用引物P32-F、pIB184-eGFP-R,P44-F、pIB184-eGFP-R,pIB184-P11-F、eGFP-R,pIB184-eGFP-F/R验证pLL06、pLL07、pLL37、pLL15。阳性转化子命名为L. lactis NZ9000-pLL06、L. lactis NZ9000-pLL07、L. lactis NZ9000-pLL37、L. lactis NZ9000-pLL15、L. lactis NZ9000-pIB184-eGFP。PCR反应程序如下:94 ℃预变性2 min,98 ℃变性10 s,55 ℃退火30 s(KOD FX Neo),68 ℃延伸30 s/kb;变性至延伸循环30次,68 ℃总延伸10 min。
1.3.4 荧光强度的测定
将L. lactis NZ9000-pLL06、L. lactis NZ9000-pLL07、L. lactis NZ9000-pLL37、L. lactis NZ9000-pLL15、L. lactis NZ9000-pIB184-eGFP分别划线活化,挑取单菌落转接至GM17+Em液体培养基中。30 ℃静置培养至对数期,1.5 mL菌液于12 000×g下离心1 min,弃上清液;20 mmol/L Tris-HCl (pH 7.4)洗涤2次后重悬菌体,混匀后每个样品取100 μL加入96孔板。在激发波长480 nm、发射波长525 nm下,用酶标仪测eGFP的表达强度及OD600值。每个样品3个平行测定[13]。
2 结果与分析
2.1 目的基因片段的扩增
分别以pMG36e、pNZ44为模板,引物P32-F/R、P44-F/R扩增启动子P32、P44,经1%琼脂糖凝胶电泳检测,如图1所示。
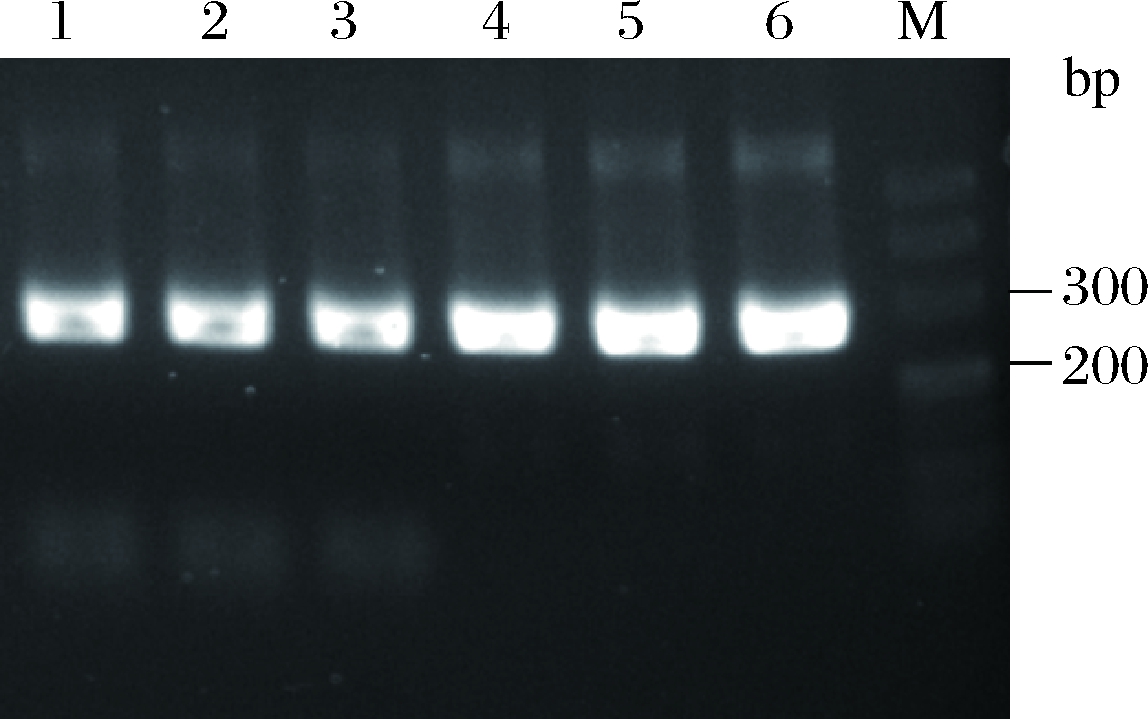
M-DL 500 DNA Marker; 1~3-启动子P32; 4~6-启动子P44
图1 PCR扩增启动子P32、P44
Fig.1 PCR amplified the promter of P32, P44
泳道1~6在近300 bp处有特异性清晰条带,与启动子P32、P44片段大小287、288 bp相符。以pLpCas9-0537、pIB184-eGFP为模板,引物pIB184-P11-F、P11-eGFP-R,eGFP-P11-F、eGFP-R分别扩增片段P11、eGFP。如图2所示,泳道1~3是P11的PCR条带93 bp(图2-a)。在750 bp处有明亮的条带,与eGFP片段772 bp的大小相符(图2-b)。片段P11及eGFP经overlap PCR后在琼脂糖凝胶1 000和750 bp间有单一条带,与819 bp的eGFP-P11片段大小相符(图2-c)。

a-M-DL 500 DNA Marker, 1~3-PCR扩增P11;b-M-DL 2000 DNA
Marker, PCR扩增eGFP;c-M-DL 2000 DNA Marker,1~3-overlap
扩增eGFP-P11
图2 PCR扩增P11、eGFP、eGFP-P11
Fig.2 PCR amplified the fragment P11, eGFP and eGFP-P11
2.2 酶切载体的获得
用Pst I/BamH I、Apa I/EcoR I分别双酶切质粒pIB184-eGFP,将线性化的DNA片段6 565、5 880 bp进行1%琼脂糖凝胶电泳,结果见图3。如图3-a所示,1~2为质粒pIB184-eGFP经Pst I/BamH I双酶切出6 565、239 bp(图中未显示)的条带。图3-b中,1为质粒pIB184-eGFP经Apa I/EcoR I双酶切出5 880、924 bp的条带。
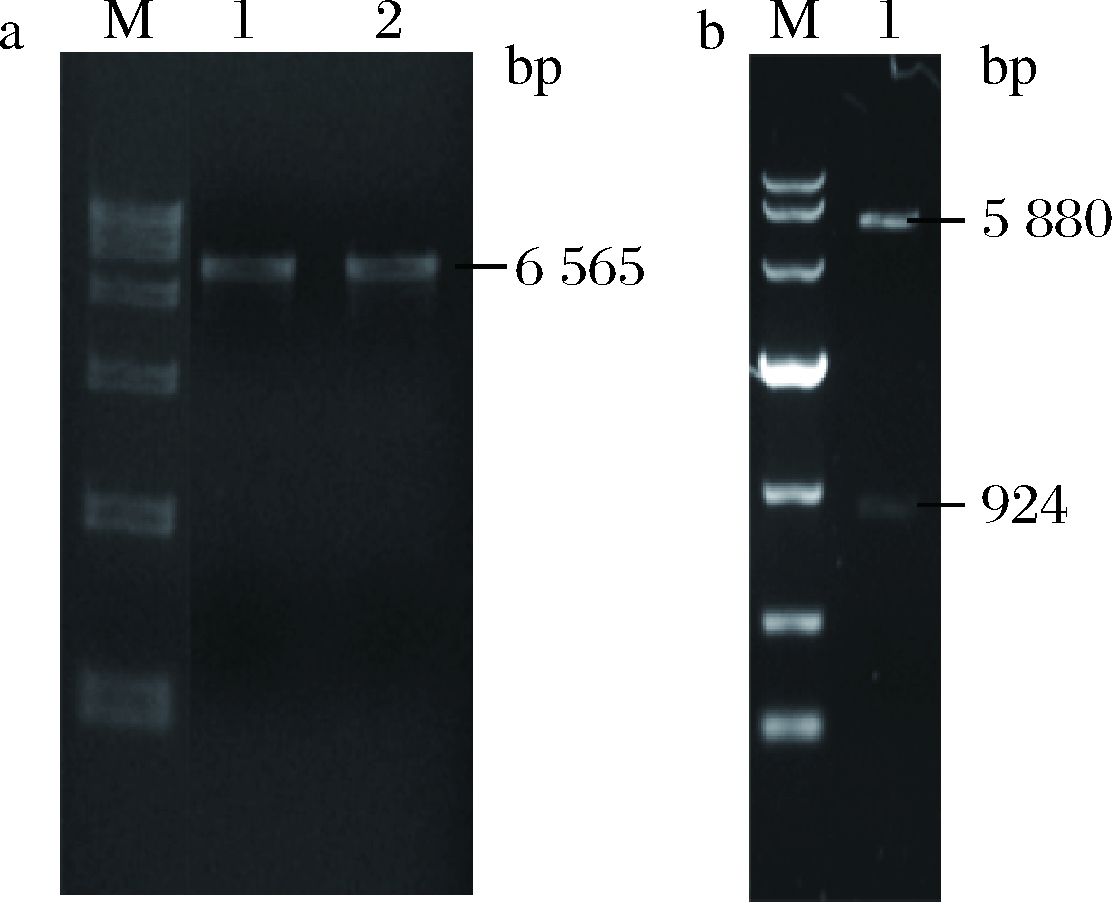
a-M-DL 15000 DNA Marker, Pst I/BamH I酶切pIB184-eGFP;
b-M-DL 10000 DNA Marker, Apa I/EcoR I酶切pIB184-eGFP
图3 双酶切载体pIB184-eGFP
Fig.3 Double enzyme digested the vector pIB184-eGFP
2.3 重组质粒的构建及鉴定
分别挑取pLL06、pLL07、pLL37、pLL15转化子进行菌落PCR验证。如图4-a所示,泳道1~2为pLL07的插入片段P44的验证结果,泳道3~4为pLL06的插入片段P32的验证结果。图4-b是pLL37的插入片段eGFP-P11验证结果,均与预期片段大小相符。挑菌落PCR验证正确的单菌落,转接至液体培养基,过夜培养后收集菌体提质粒,进一步进行酶切验证。如图5-a所示,泳道1为pLL06经Pst I/Bgl II双酶切,获得5 816、1000 bp的条带;泳道2为pLL07经Pst I/EcoR I双酶切,获得5 827、990 bp的条带;泳道3为pLL37经Apa I/EcoR I双酶切,获得5 880、773 bp的条带。图5-b中,泳道1为EcoR I单酶切pLL15,片段大小为6 551 bp;泳道2为对照质粒pIB184-eGFP经EcoR I单酶切,片段大小为6 804 bp。酶切出的片段大小均与预期相符,说明pLL06、pLL07、pLL15、pLL37系列质粒构建成功。

a-M-DL 500 DNA Marker; 1~2-pLL07; 3~4-pLL06;
b-M-DL 1000 DNA Marker; 1~4-pLL37
图4 PCR验证pLL06、pLL07、pLL37
Fig.4 PCR verified pLL06, pLL07 and pLL37
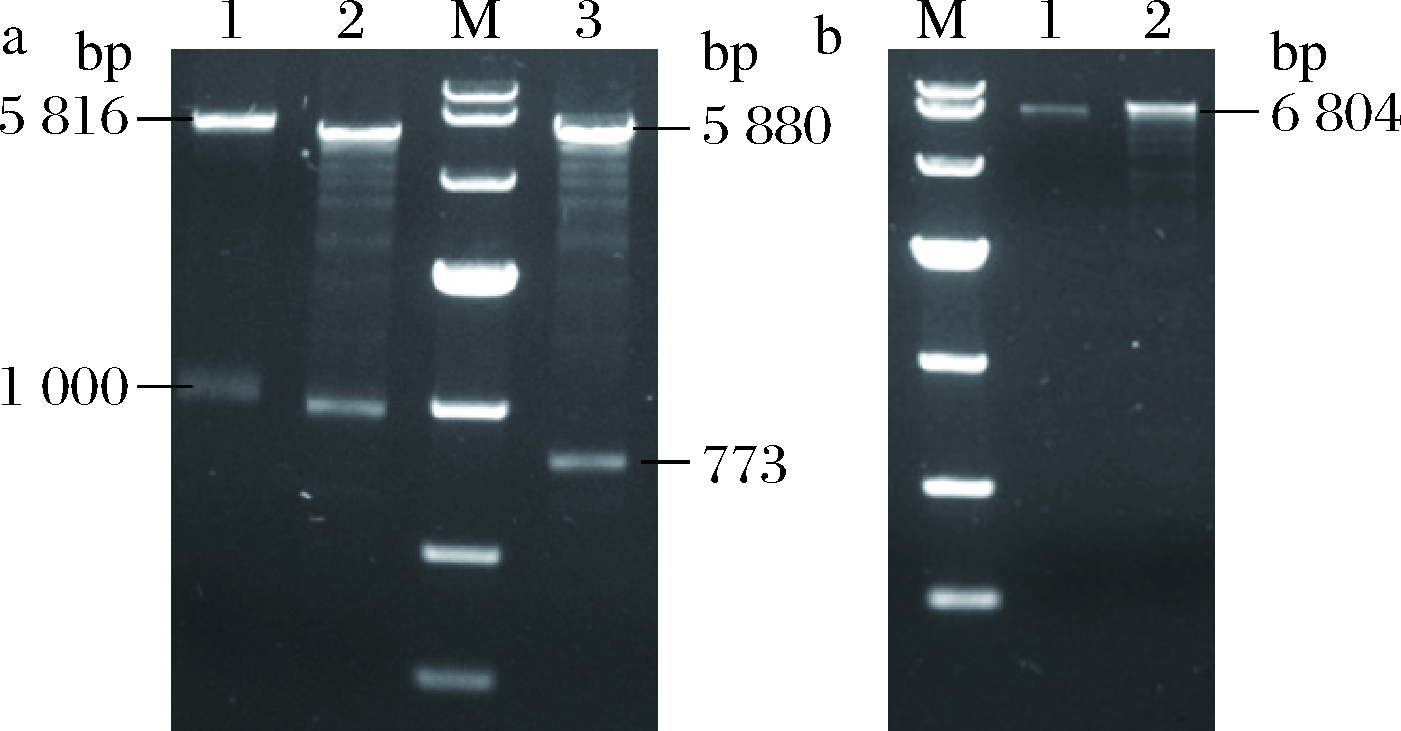
M-DL 10000 DNA Marker; a-1-pLL06; 2-pLL07; 3-pLL37;
b-1-pLL15; 2-对照pIB184-eGFP
图5 酶切验证不同启动子的质粒
Fig.5 Identification of plasmids of different promoters
by enzyme digestion
2.4 荧光强度的测定
将重组质粒pLL06、pLL07、pLL37、pIB184-eGFP及pLL15电转至乳酸乳球菌NZ9000感受态细胞中,挑取单菌落进行PCR验证。如图6-a所示,泳道1~3为pLL15用引物pIB184-eGFP-F/R进行验证,片段大小为863 bp;泳道4~6为pLL07用引物P44-F、pIB184-eGFP-R进行验证,片段大小为993 bp;泳道7~8为pLL06用引物P32-F、pIB184-eGFP-R进行验证,片段大小为992 bp。如图6-b所示,pLL37用引物pIB184-P11-F、eGFP-R进行验证,片段大小为819 bp。结果显示所有重组质粒均成功转化至乳酸乳球菌NZ9000中。挑选阳性转化子,测量对数期不同启动子调控eGFP的表达强度,如图7所示。结果表明,启动子P11的活力十分微弱,启动子P23、P32、P44分别比P11高约23.9、7.8、4.1倍。
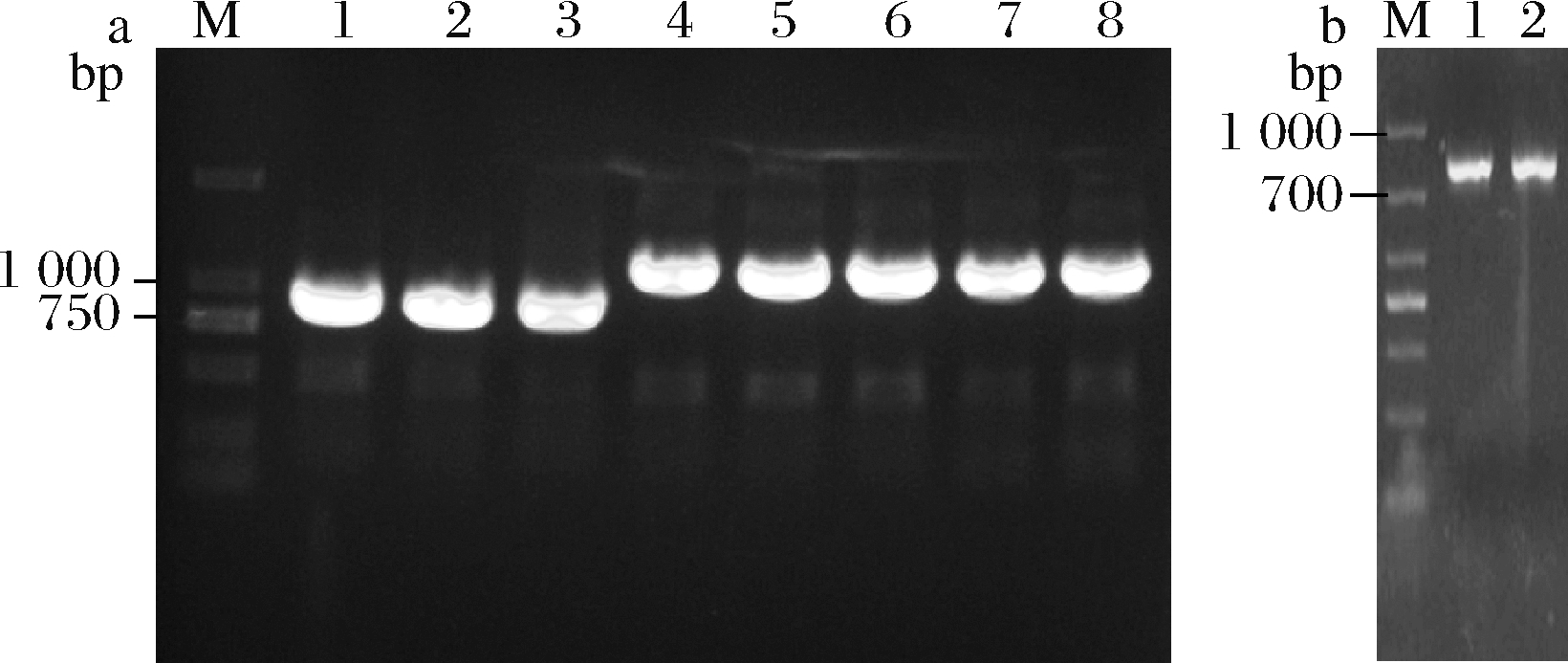
a-M-DL 2000 DNA Marker; 1~3-pLL15; 4~6-pLL07;
7~8-pLL06;b-M-DL 1000 DNA Marker; 1~2-pLL37
图6 PCR验证NZ9000-pLL06、pLL07、pLL15、pLL37
Fig.6 PCR verified NZ9000-pLL06, pLL07, pLL15
and pLL37
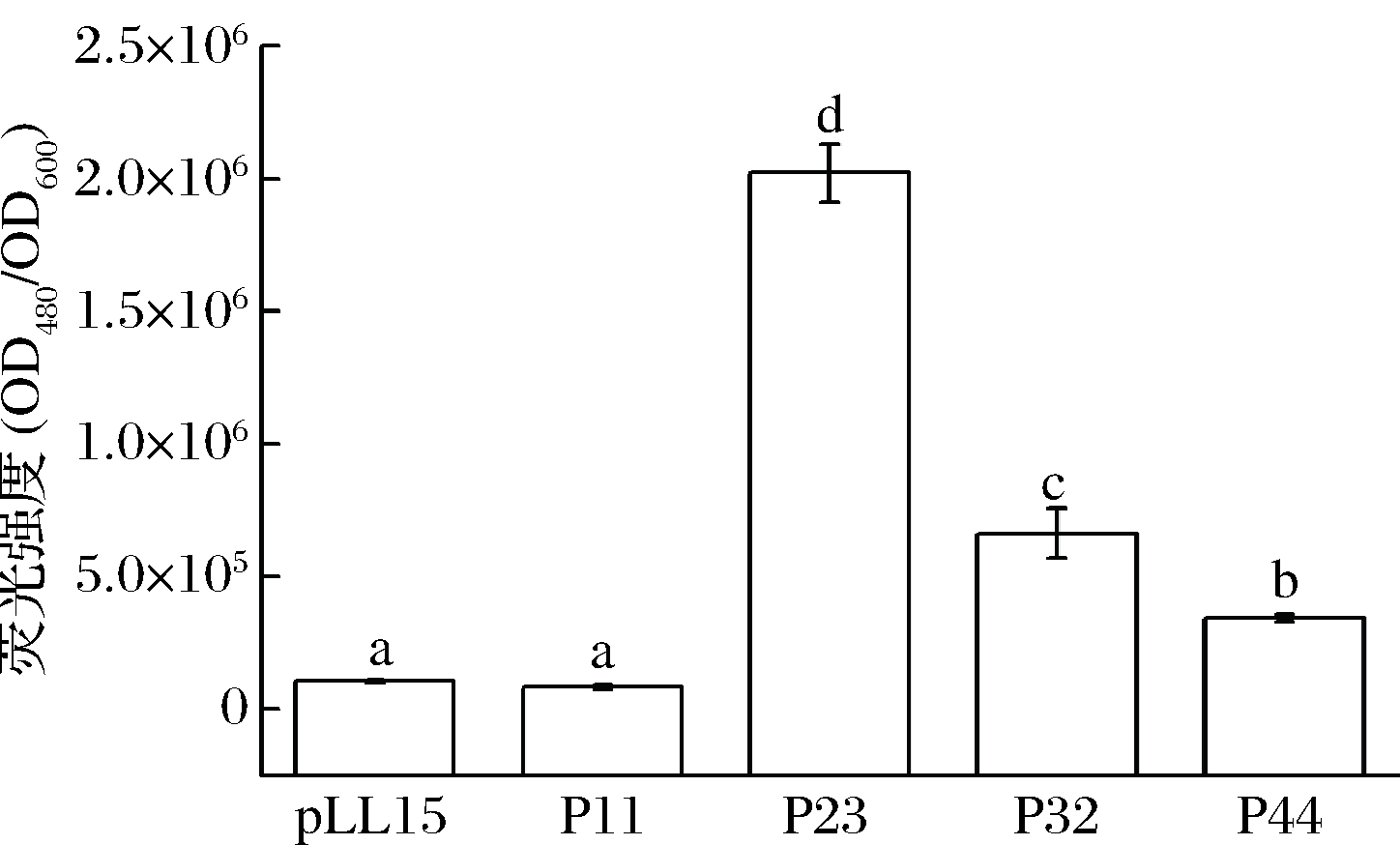
图7 利用基于eGFP的报告系统对不同启动子
强度的测定
Fig.7 Evaluation of the transcriptional activities of different
promoters via an eGFP reporting system
注:图中不同字母表示存在极显著性差异(P<0.01)
3 结果与讨论
在乳酸乳球菌中,蛋白质的表达可通过组成型启动子进行调控使基因持续性表达,也可通过诱导型系统添加诱导肽诱导相应启动子发挥活性以调控相应蛋白的表达,如Nisin-controlled expression system[14],Sugar-inducible expression system[15]。诱导型系统可以降低毒性蛋白产生的毒性,避免细胞死亡,但系统的表达需要添加诱导肽,不易调控,操作繁琐。pIB184是广宿主大肠杆菌-乳酸菌穿梭质粒,常用做乳酸菌过表达质粒载体[16-17]。为筛选出易于调控、适用于乳酸乳球菌、启动强度高的启动子,本研究以eGFP为报告基因、pIB184质粒为骨架,建立了乳酸乳球菌NZ9000报告系统。通过无缝克隆的方式,获得一系列携带不同启动子的重组质粒,并将其分别转化至乳酸乳球菌中。利用酶标仪测定eGFP的表达强度,比较启动子P11、P23、P32、P44在乳酸乳球菌NZ9000中调控基因表达能力的强弱。结果表明启动子P11转录活性较弱,这可能是由于乳酸菌对启动子具有严格的宿主选择性[18-19]。启动子P23转录活性最强,这与VOSSEN等[20]在乳脂链球菌中的研究结果一致。宋馨[13]在干酪乳杆菌中对P23与Pcas、PJ23119、Pldh进行了比较,结果表明P23调控eGFP表达的能力最强。因此,推断P23是目前应用在乳酸菌中转录活性最强的启动子。本研究建立了乳酸乳球菌适用的报告系统,可被应用于筛选不同转录活性的启动子。同时利用该系统,鉴定了不同启动子在乳酸乳球菌中的转录活性,获得了不同强度的启动子,为外源基因在乳酸乳球菌中的表达奠定了基础。
[1] 李全阳, 夏文水. 乳酸菌胞外多糖的研究[J]. 食品与发酵工业, 2003, 29(5): 86-90.
[2] 张金宝, 乌云塔娜. 乳酸菌食品级高效表达载体系统的研究进展[J]. 畜牧兽医杂志, 2008, 27(6): 42-44.
[3] 张虎成. 乳酸菌表达系统的初步构建及应用[D]. 北京: 中国人民解放军军事医学科学院, 2007.
[4] 贡汉生, 孟祥晨. 乳酸菌细菌素分类与作用机制[J].食品与发酵工业, 2008, 34(1): 105-109.
[5] 程璐, 缪铭, 张涛, 等. 食品生物防腐剂——抗真菌乳酸菌研究进展[J]. 食品与发酵工业, 2010, 36(9): 129-133.
[6] ZHU D, LIU F, XU H, et al. Isolation of strong constitutive promoters from Lactococcus lactis subsp. lactis N8[J]. FEMS Microbiology Letters, 2015, 362(16): 1-6.
[7] MORELLO E, L G BENMUDEZ-HUMARAN, LLULL D, et al. Lactococcus lactis, an efficient cell factory for recombinant protein production and secretion[J]. Journal of Molecular Microbiology & Biotechnology, 2008, 14:48-58.
[8] GU Q, SONG D, ZHU M. Oral vaccination of mice against Helicobacter pylori with recombinant Lactococcus lactis expressing urease subunit B[J]. FEMS Immunology & Medical Microbiology, 2009, 56(3): 197-203.
[9] SONG A L, INL LA, LIM SHE, et al. A review on Lactococcus lactis: from food to factory[J]. Microbial Cell Factories, 2017, 16(1): 1-15.
[10] 孙海烨, 张梁, 李由然, 等. 利用增强型绿色荧光蛋白研究不同启动子在乳酸克鲁维酵母中的功能[J]. 生物技术通报, 2017, 33(6): 197-206.
[11] 肖红庆, 李晓薇, JAMEEL A, 等. 人工合成启动子的应用[J].农业与技术, 2018, 38(9): 20-23.
[12] 韦云莹, 王立峰, 熊智强, 等. 响应面法优化乳酸乳球菌电转化效率研究[J]. 上海理工大学学报, 2018, 40(6): 566-571.
[13] 宋馨. 干酪乳杆菌遗传操作系统建立及胞外多糖合成关键基因解析[D]. 上海: 上海理工大学, 2018.
[14] KLEEREBEZEM M, BEERTHUYZEN M M, VAUGHAN E E, et al. Controlled gene expression systems for lactic acid bacteria: transferable Nisin-inducible expression cassettes for Lactococcus, Leuconostoc, and Lactobacillus spp.[J]. Applied and Environmental Microbiology, 1997, 63(11):4 581-4 584.
[15] DE V W M. Gene expression system for lactic acid bacteria[J].Current Opinion in Microbiology, 1999, 2(3): 289-295.
[16] 王巧惠, 王光强, 宋馨, 等. 不同质粒电转三种乳酸菌的比较研究[J]. 工业微生物, 2016, 46(1): 36-41.
[17] SONG X, XIONG Z Q, KONG L H, et al. Relationship between putative eps genes and production of exopolysaccharide in Lactobacillus casei LC2W[J]. Frontiers in Microbiology, 2018, 9:1-9.
[18] 于泽. 基于乳酸菌自身分泌蛋白的组成型强启动子探测及功能分析[D]. 哈尔滨: 东北农业大学, 2013.
[19] 秦思. 基于短乳杆菌SlpA启动子的组成型表达载体构建及其活性分析[D]. 哈尔滨: 东北农业大学, 2013.
[20] VOSSEN J M V D, LELIE D V D, VENEMA G. Isolation and characterization of Streptococcus cremoris Wg2-specific promoters[J]. Applied and Environmental Microbiology, 1987, 53(10):2 452-2 457.