解淀粉芽胞杆菌(Bacillus amyloliquefaciens)是芽孢杆菌属的一种具有重要研究价值的菌种,在发酵工业中可用于生产淀粉酶[1]、蛋白酶[2-3]、纤维素酶[4]等多种工业用酶,此外还可以表达几丁质酶[5]、伊枯草菌素[6-7]等具有重要研究价值的生物活性物质。但由于芽胞杆菌属中各菌种间保守序列高度相似,无法单独依靠常用的16S rDNA/RNA测序来准确定位到种, 通常需要结合生理生化鉴定[8-9],或者其他序列,如5′端16S-23S ITS核苷酸序列[10]、gyrB基因[11]、gyrA[12]甚至全基因序列[13-14]进行分析。CHEN等[15]、XU等[16]在筛选解淀粉芽胞杆菌时,使用16S与gyrA基因分别建树的方法进行鉴定;LIM等[17]采用16S序列比对结合API系统鉴定1株产广谱细菌素的解淀粉芽胞杆菌;NEL等[18]采用16S rDNA扩增片段限制性内切酶分析与rpoB基因建树结合的方法,区分并鉴定南非制糖厂中筛选得到的解淀粉芽胞杆菌与枯草芽胞杆菌。鉴定方法较为繁琐,目前还没有通用、简便、快速的鉴定方法。
gyrB基因编码DNA促旋酶的B亚单位,在细菌中广泛存在,序列相对保守,但进化速度快于16S,目前在细菌鉴定中的使用逐渐增加[19],积累了较为丰富的比对信息,但单独使用时部分芽胞杆菌也难以准确定位,而16S与gyrB序列分别比对建树的方法容易出现进化树结构冲突,干扰定位的情况。利用16S与gyrB基因联合建树,以较少的序列数和进化树数实现对筛选菌株快速鉴定的方法,可提高数据的利用率,减少数据冲突,为芽胞杆菌种群的分子生物学鉴定方法开发提供参考。
1 材料与方法
1.1 实验材料
1.1.1 土壤样本
土壤样本,采集自厦门同安国家农业科技园区竹坝农场。
1.1.2 培养基与试剂
营养琼脂培养基,广东环凯生物科技有限公司;苯丙氨酸脱氨酶鉴定管、MR-VP鉴定管、SIM培养基(用于硫化氢和吲哚试验)等,青岛海博生物科技有限公司。
细菌基因组DNA提取试剂盒、胶回收试剂盒、聚合链式反应(polymerase chain reaction,PCR)试剂,生工生物工程(上海)股份有限公司生产;16S通用引物27F/1492R、gyrB扩增引物UP-1/UP-2r、gyrB测序引物UP-1S/UP-2Sr[20],生工生物工程(上海)股份有限公司合成。
1.1.3 仪器与设备
Aeris-bg096PCR仪,新加坡ESCO公司;HE99核酸电泳仪,美国GE公司;211B摇床,上海苏坤公司;LRH-250生化培养箱,上海一恒公司。
1.2 实验方法
1.2.1 样本采集
于农场蔬菜种植区取土壤25 g加入225 mL蛋白胨生理盐水稀释均质后稀释涂布营养琼脂平板,30 ℃培养24 h,取单菌落进行纯化。
1.2.2 16S与gyrB序列提取
用细菌基因组DNA抽提试剂盒,提取菌株的总DNA;分别用16S与gyrB引物扩增,所得PCR产物电泳纯化后,送由生工生物工程(上海)股份有限公司测序。
16S扩增条件:94 ℃预变性4 min;94 ℃变性30 s,65 ℃退火40 s,72 ℃延伸90 s,共35个循环;72 ℃延伸10 min。
gyrB扩增条件[20]:94 ℃预变性4 min;94 ℃变性1 min,60 ℃退火1 min,72 ℃延伸2 min,共35个循环;72 ℃延伸10 min。
1.2.3 16S与gyrB序列比对与进化分析
将测序结果在Genbank进行比对。为保障数据的准确性,取与受试菌gyrB序列相似度较高的前1 000个序列中,来源于各菌种保藏中心菌株的序列进行比对,包括ATCC、BCRC、CGMCC、CIP、CECT等。下载相似菌株的16S与gyrB序列、分别用BioEdit[21]和Clustal X[22]进行比对和编辑,取有效区间,分别试验单独使用16S序列、单独使用gyrB序列和使用16S与gyrB线形拼接序列建树[23],采用最大简约算法,用Paup* 4.0构建进化树[24-25],选择大肠杆菌ATCC 25922[26]作为外群。
1.2.4 生理生化验证
参考《伯杰细菌鉴定手册》[27]进行验证。
1.2.5 联合建树方法适用性分析
为分析16S与gyrB基因联合建树的方法是否适用于其他解淀粉芽胞杆菌的鉴定,于Genbank下载16S与gyrB基因均可得的解淀粉芽胞杆菌序列,按照上述方法进行序列拼接。选择《伯杰细菌鉴定手册》[27]中芽胞杆菌属模式菌株,于Genbak和EZBiocloud下载16S和gyrB序列,选择大肠杆菌ATCC 25922作为外群,构建进化树。
2 结果与分析
2.1 菌株分离纯化
从土壤中筛选得到1株革兰氏阳性杆菌,产芽胞,菌落白色、干燥、表面粗糙,编号为HX2016004。
2.2 16S与gyrB基因测序
PCR扩增所得16S序列有效区共1 436 bp,gyrB序列有效区共1 207 bp。
2.3 序列比对与进化树构建
16S序列比对结果中,高相似度菌株过多,且多数菌株无法获得更多的遗传标记序列;而gyrB序列比对结果中,多数菌株同时可获取16S序列,因此,以gyrB相似菌株作为建树群,下载16S与gyrB基因序列,如表1所示,比较分别使用16S、gyrB和16S与gyrB线形拼接序列建树结果。
16S比对后去除头尾不整齐片段,保留中心区域1 467 bp(含gap);gyrB比对后去除头尾不整齐片段,保留中心区域1 175 bp(含gap)。构建进化树如图1所示,A树基于16S序列构建(图1-a),b树基于gyrB序列构建(图1-b),c树基于16S与gyrB拼接序列构建(图1-c),计算次数为1 000,参数如表2所示。
表1 Genbank中拥有相似16S与gyrB序列的菌株
Table 1 Bacterial strains in Genbank with similar 16S and gyrB sequences
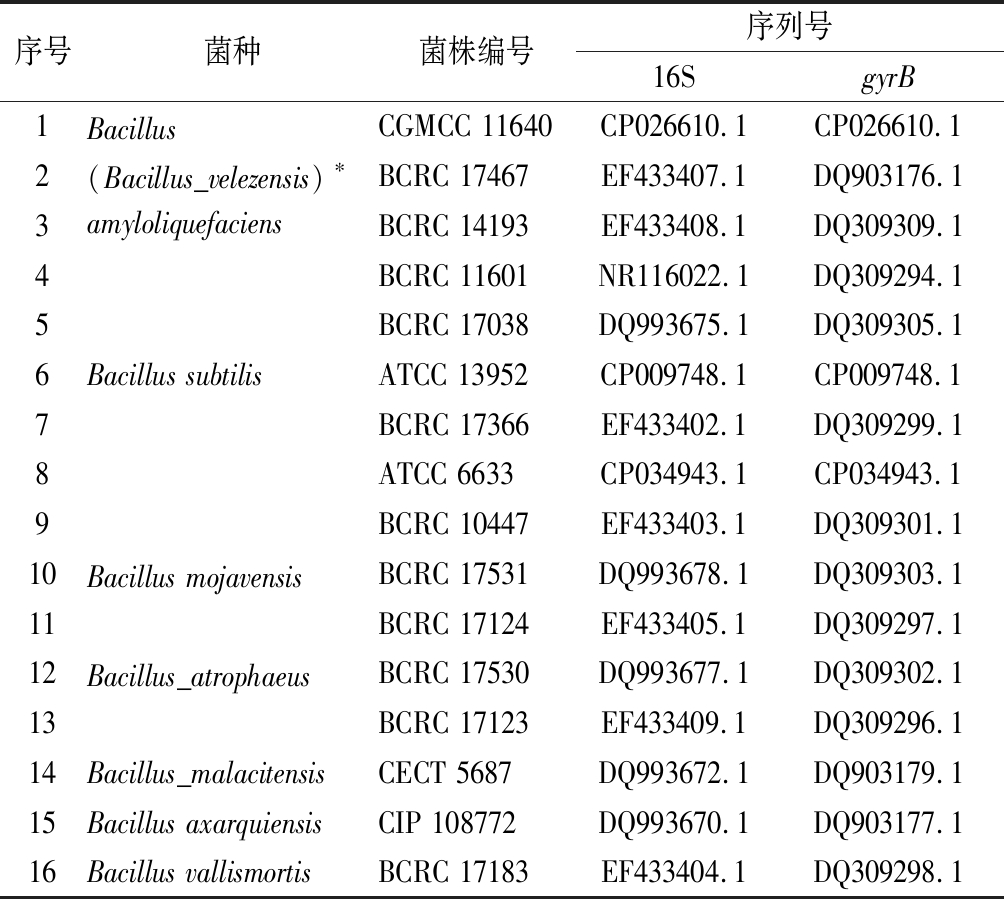
序号菌种菌株编号序列号16SgyrB12345Bacillus(Bacillus_velezensis)∗amyloliquefaciensCGMCC 11640CP026610.1CP026610.1BCRC 17467EF433407.1DQ903176.1BCRC 14193EF433408.1DQ309309.1BCRC 11601NR116022.1DQ309294.1BCRC 17038DQ993675.1DQ309305.16789Bacillus subtilisATCC 13952CP009748.1CP009748.1BCRC 17366EF433402.1DQ309299.1ATCC 6633CP034943.1CP034943.1BCRC 10447EF433403.1DQ309301.11011Bacillus mojavensis BCRC 17531DQ993678.1DQ309303.1BCRC 17124EF433405.1DQ309297.112Bacillus_atrophaeusBCRC 17530DQ993677.1DQ309302.113BCRC 17123EF433409.1DQ309296.114Bacillus_malacitensisCECT 5687DQ993672.1DQ903179.115Bacillus axarquiensisCIP 108772DQ993670.1DQ903177.116Bacillus vallismortisBCRC 17183EF433404.1DQ309298.1
注:*表示 Bacillus_velezensis为Bacillus amyloliquefaciens的异名[28-29]
表2 基于16S与gyrB序列的进化树参数表
Table 2 Parameters of phylogenetic trees based on the combined sequences of 16S and gyrB

进化树树长一致性指数保留指数同型指数a6660.9830.8280.017b1 0110.7860.8760.214c1 6820.8620.8710.138
由图1可知,仅使用16S序列构建的进化树a分枝自展值低,无法准确定位HX2016004菌株的族群;仅使用gyrB序列构建的进化树b较a整体分枝自展值高,但一致性指数(consistency index,CI)相对较低,且与a树存在较大的结构冲突;使用16S与gyrB拼接序列构建的进化树c结构与b一致,表明建树群中16S的差异较小,不足以影响分类结构,HX2016004菌株与解淀粉杆菌聚合在一枝,自展值为94,表明该菌株为解淀粉芽胞杆菌可能性高。c树中Bacillus subtilis ATCC 13952的进化关系存在疑问,有待进一步分析,但不影响HX2016004菌株的鉴定。

a-基于16S的进化树;b-gyrB基因的进化树; c-基于16S与gyrB基因的进化树
图1 HX2016004菌株基于16S与gyrB序列的进化树
Fig.1 Phylogenetic trees about HX2016004 based on sequences of 16S and gyrB
2.4 生理生化验证
菌株HX2016004生理生化特征如表3所示,与《伯杰细菌鉴定手册》中的解淀粉芽胞杆菌特征吻合,与进化分析结果一致,命名为Bacillus amyloliquefaciens HX2016004。
表3 菌株HX2016004生理生化特征
Table 3 Physiological and biochemical characteristics of HX2016004

生理生化特征HX2016004解淀粉芽胞杆菌芽胞位置中生中生葡萄糖产酸++淀粉水解++厌氧生长--3 ℃生长--明胶液化++V.P反应++接触酶++柠檬酸盐利用++硝酸盐还原--苯丙氨酸脱氨酶--吲哚试验--甲基红试验--硫化氢试验--
注:+代表革兰氏染色呈阳性;-代表革兰氏染色呈阴性
2.5 联合建树方法适用性
Genbank中16S与gyrB序列同时可得,且鉴定结果较为可靠的解淀粉芽胞杆菌菌株共有37株,另选择亲缘关系较近的芽胞杆菌属其他菌种模式菌株16株,拼接16S与gyrB基因序列后构建进化树如图2所示。其中所有的解淀粉芽胞杆菌很好的聚合在一簇,与亲缘关系相近菌种Bacillus subtilis、Bacillus licheniformis、Bacillus pumilus具有较高的种间自展值,分类结构可靠。也就是说,如这37株菌中的任意1株菌种还未鉴定的情况下,利用该方法建树时均与其他36株解淀粉芽胞杆菌聚合在1枝,可准确鉴定为解淀粉芽胞杆菌。表明16S与gyrB基因联合建树的方法适用于已知的37株解淀粉芽胞杆菌鉴定,可以预测该方法在解淀粉芽胞杆菌鉴定中具有普适性。

图2 Genbank已知菌株基于16S与gyrB序列的进化树
Fig.2 Phylogenetic tree based on sequences of 16S and gyrB in Genbank
3 结论
利用16S与gyrB基因联合建树的方法,课题组快速将1株从土壤中筛选得到的芽胞杆菌HX2016004准确鉴定为解淀粉芽胞杆菌。该方法中使用的引物均为通用引物,只需要1次扩增即可得到16S与gyrB基因序列,测序量小,时间短,数据利用率高;与多基因分别建树的方法相比,可避免进化树结构之间的冲突和数据紊乱,提高了进化树准确性,适用于16S序列高度相似的菌群鉴定。经验证,该方法在解淀粉芽胞杆菌的鉴定中具有较普遍的适用性,课题组下一步将继续研究该方法在其他菌群鉴别中的有效性。
[1] GANGADHARAN D, SIVARAMAKRISHNAN S, NAMPOOTHIRI K M, et al. Solid culturing of Bacillus amyloliquefaciens for α-amylase production[J]. Food Technology and Biotechnology, 2006, 44(2): 269-274.
[2] CORTEZI M, CONTIERO J, LIMA C J B, et al. Characterization of a feather degrading by Bacillus amyloliquefaciens protease: A new strain[J]. World Journal of Agricultural Sciences, 2008, 4(5): 648-656.
[3] 钟晨滑, 贠婷婷, 綦文涛, 等. 基于解淀粉芽胞杆菌的发酵谷朊粉制备工艺[J]. 食品与发酵工业, 2016, 42(4): 12-16.
[4] KALAWONG R, WAKAYAMA M, ANUNTALABHOCHAI S, et al. Comparison and characterization of purified cellulase and xylanase from Bacillus amyloliquefaciens CX1 and Bacillus subtilis B4[J]. Chiang Mai Journal of Science, 2018, 45(1): 92-105.
[5] WANG S L, SHIH I L, LIANG T W, et al. Purification and characterization of two antifungal chitinases extracellularly produced by Bacillus amyloliquefaciens V656 in a shrimp and crab shell powder medium[J]. Journal of Agricultural and Food Chemistry, 2002, 50(8): 2 241-2 248.
[6] HIRADATE S, YOSHIDA S, SUGIE H, et al. Mulberry anthracnose antagonists (iturins) produced by Bacillus amyloliquefaciens RC-2[J]. Phytochemistry, 2002, 61(6): 693-698.
[7] YU G Y, SINCLAIR J B, HARTMAN G L, et al. Production of iturin A by Bacillus amyloliquefaciens suppressing Rhizoctonia solani[J]. Soil Biology and Biochemistry, 2002, 34(7): 955-963.
[8] LOGAN N A, BERKELEY R C W. Identification of Bacillus strains using the API system[J]. Microbiology, 1984, 130(7): 1 871-1 882.
[9] O′DONNELL A G, NORRIS J R, BERKELEY R C W, et al. Characterization of Bacillus subtilis, Bacillus pumilus, Bacillus licheniformis, and Bacillus amyloliquefaciens by pyrolysis gas-liquid chromatography, deoxyribonucleic acid-deoxyribonucleic acid hybridization, biochemical tests, and API systems[J]. International Journal of Systematic and Evolutionary Microbiology, 1980, 30(2): 448-459.
[10] XU D, COTE J C. Phylogenetic relationships between Bacillus species and related genera inferred from comparison of 3′ end 16S rDNA and 5′ end 16S-23S ITS nucleotide sequences[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(3): 695-704.
[11] WANG L T, LEE F L, TAI C J, et al. Comparison of gyrB gene sequences, 16S rRNA gene sequences and DNA-DNA hybridization in the Bacillus subtilis group[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(8): 1 846-1 850.
[12] 喻国辉, 牛春艳, 陈远凤, 等. 利用16S rDNA结合gyrA和gyrB基因对生防芽孢杆菌R31的快速鉴定[J]. 中国生物防治学报, 2010, 26(2): 160-166.
[13] LAPIDUS A, GALLERON N, ANDERSEN J T, et al. Co-linear scaffold of the Bacillus licheniformis and Bacillus subtilis genomes and its use to compare their competence genes[J]. FEMS Microbiology Letters, 2002, 209(1): 23-30.
[14] REY M W, RAMAIYA P, NELSON B A, et al. Complete genome sequence of the industrial bacterium Bacillus licheniformis and comparisons with closely related Bacillus species[J]. Genome biology, 2004, 5(10): 77.
[15] CHEN X, ZHANG Y, FU X, et al. Isolation and characterization of Bacillus amyloliquefaciens PG12 for the biological control of apple ring rot[J]. Postharvest Biology and Technology, 2016, 115: 113-121.
[16] XU J, WANG H, ZHU Z, et al. Isolation and characterization of Bacillus amyloliquefaciens ZDS-1: Exploring the degradation of Zearalenone by Bacillus spp[J]. Food Control, 2016, 68: 244-250.
[17] LIM K B, BALOLONG M P, KIM S H, et al. Isolation and characterization of a broad spectrum bacteriocin from Bacillus amyloliquefaciens RX7[J/OL]. BioMed Research International, 2016,https.//doi.org/10.1155/2016/8521476.
[18] NEL S, DAVIS S B, ENDO A, et al. Differentiation between Bacillus amyloliquefaciens and Bacillus subtilis isolated from a South African sugarcane processing factory using ARDRA and rpoB gene sequencing[J]. Archives of microbiology, 2019, 201(10): 1 453-1 457.
[19] WATANABE K, NELSON J, HARAYAMA S, et al. ICB database: The gyrB database for identification and classification of bacteria[J]. Nucleic Acids Research, 2001, 29(1): 344-345.
[20] YAMAMOTO S, HARAYAMA S. PCR amplification and direct sequencing of gyrB genes with universal primers and their application to the detection and taxonomic analysis of Pseudomonas putida strains[J]. Applied and Environmental Microbiology, 1995, 61(3): 1 104-1 109.
[21] HALL T A. Bioedit: A user—friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41:95-98.
[22] THOMSON J D, GIBSON T J, PLEWNIAK F, et al. The Clustal_X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research,1997, 25:4 876-4 882.
[23] ZHAO C L, CUI B K, DAI Y C. New species and phylogeny of Perenniporia based on morphological and molecular characters[J]. Fungal Diversity,2013,58(1): 47-60.
[24] JIANG X Z, YU H Y, XIANG M C, et al. Echinochlamydosporium variabile, a new genus and species of Zygomycota from soil nematodes[J]. Fungal Diversity,2011,46(1):43-51.
[25] 肖玉娟, 傅奇, 沈金海, 等. 一株引起厦门地区不同作物感染的Poitrasia circinans分离与鉴定[J]. 科学技术与工程, 2018, 18(14): 20-23.
[26] MINOGUE T D, DALIGAULT H A, DAVENPORT K W, et al. Complete genome assembly of Escherichia coli ATCC 25922, a serotype O6 reference strain[J]. Genome Announcements, 2014, 2(5): e00 969-14.
[27] CLAUS D. The Genus Bacillus[M]. New York: Springer, 1986: 751-752.
[28] WANG L T, LEE F L, TAI C J, et al. Bacillus velezensis is a later heterotypic synonym of Bacillus amyloliquefaciens[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(3): 671-675.
[29] 张彩文, 程坤, 张欣, 等. 贝莱斯芽胞杆菌 (Bacillus velezensis) 分类学及功能研究进展[J]. 食品与发酵工业, 2019, 45(17): 258-265.