酸汤是贵州黔东南苗族、侗族人民嗜食的传统发酵食品,包括白酸汤和红酸汤两种,成品红酸汤由辣椒红酸汤、番茄红酸汤分别发酵、混合而成。白酸汤的主要原材料有大米、糯米、玉米等,做法是将清米汤直接放置在密闭容器内,接种老酸汤,自然发酵。白酸汤具有开胃健脾、增进食欲、生津止渴、清热解暑、助消化等保健功效。白酸汤中添加蔬菜,煮制菜汤,是当地人的日常饮食习惯,当地有“三天不吃酸,走路打串串”之说。此外,白酸汤可加工成火锅底料、调味品、饮料等[1]。
红酸汤的主要原材料为番茄、辣椒。制作工艺可分为两个发酵阶段:一是番茄和辣椒分别发酵为半成品;二是将两者按一定的比例混合进行第2次发酵。第1个阶段决定了红酸汤的酸度,是红酸汤风味形成的关键阶段[2]。红酸汤、白酸汤可以作为底料配以蔬菜,牛肉、鲜鱼、排骨等制成酸汤火锅。酸汤中富含多种益生菌群和有机酸、矿物质等,有调节人体肠道微生态平衡、帮助消化、防止便秘、防止细胞老化、降低胆固醇以及调剂人体生理机能等保健作用[3]。
苗家酸汤含有一定量的酒石酸、苹果酸、乳酸、乙酸、柠檬酸和少量的丁二酸。富含丰富的矿物质,如钙、磷、铁等对保持神经、肌肉的兴奋性以及维持肌体的酸碱平衡具有重要的作用[4]。酸汤发酵中主要有乳酸杆菌、醋酸杆菌、双歧杆菌和酵母菌等多种微生物,发酵阶段的微生物是酸汤品质风味形成的重要因素。自然发酵的酸汤品质极不稳定[5]。
目前,对于酸汤的报道大多为单个白酸汤、辣椒红酸汤、番茄红酸汤的研究。如红酸汤的发酵工艺;主要营养成分和功能成分的分析研究[6];苗族发酵型白酸汤的生产工艺研究;凯里白酸汤的工艺优化;苗族白酸汤饮料的开发;贵州红酸汤火锅调料的研究进展及工业化进程等。
运用高通量测序(Illumina MiSeq)技术分析贵州凯里酸汤独特风味的优势菌群尚无相关报道。在酸汤中接种老酸汤发酵时,可能存在老酸汤质量问题和接种时操作规范问题。由于发酵过程为自然发酵,最终形成的品质风味受外界环境影响巨大。因此,揭示酸汤发酵过程中的微生物多样性优势菌群种类对酸汤生产具有重大意义。因此对贵州黔东南酸汤的研究解释有助于白酸汤和红酸汤发酵过程的精准调控,指导大规模生产。
1 材料与方法
1.1 样品采集
白酸汤、辣椒红酸汤和番茄红酸汤,采自贵州千里苗疆农业开发有限公司的9个不同车间,采集样品后将其装入密封器皿中,低温运输至实验室,放于4 ℃短期存放,提取样品总DNA。
1.2 总DNA抽提
1.2.1 试剂盒提取样品DNA
量取100 mL白酸汤,100 mL辣椒红酸汤和100 mL番茄红酸汤,4 000 r/min离心5 min后弃上清取沉淀,采用E.Z.N.A.®Soil DNA Kit 试剂盒,按照试剂盒操作说明书进行微生物总DNA 提取。
1.2.2 预洗处理的聚乙烯吡咯烷酮(polyvinyl pyrrolidone,PVP)法
称取2.0 g已处理样品于10 mL离心管内,按照PVP法提取样品总DNA[7]。
将2种方法提取的基因组DNA按比例混合,送至上海美吉生物医药科技有限公司测序,并根据测序得到的数据使用分析软件进行各项指标分析。
1.3 引物及PCR扩增、产物检测与纯化
16S rRNA基因V3~V4区扩展引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGTWTCTAAT-3′)[8-9];ITS区引物为ITS1F(5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS2R (5′-GCTGCGTTCTTCATCGATGC-3′)[10]。PCR正式试验采用TransStart FastPfu DNA Polymerase,20 μL反应体系为:5×FastPfu Buffer 4 μL,dNTPs (2.5 mmol/L) 2 μL,正、反向引物(5 μmol/L)各0.8 μL,FastPfu Polymerase (2.5 U/μL) 0.4 μL,BSA (0.8 μg/μL) 0.2 μL,DNA 10 ng,ddH2O补至20 μL。PCR反应程序:95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸45 s,共32个循环;72 ℃延伸10 min。PCR产物使用2%琼脂糖凝胶电泳检测,根据PCR产物浓度进行等量混样,充分混匀后,再用2%琼脂糖凝胶电泳检测PCR产物,对目的条带使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收。
1.4 PCR产物检测与纯化
PCR产物使用2%琼脂糖凝胶电泳检测,根据PCR产物浓度进行等量混样,充分混匀后,再用2%琼脂糖凝胶电泳检测PCR产物,对目的条带使用AxyPrepDNA凝胶回收试剂盒切胶回收。
1.5 荧光定量、文库构建和上机测序
参照电泳结果,将PCR产物用QuantiFLuorTM-ST蓝色荧光定量系统(PRomega公司)进行检测定量。参照沈馨等[11]方法,将同一样本的PCR产物混合后使用2%琼脂糖凝胶回收PCR产物,利用AxyPrep DNA Gel Extraction Kit (Axygen Biosciences, Union City, CA, USA) 进行回收产物纯化,2%琼脂糖凝胶电泳检测,并用QuantusTM Fluorometer (Promega, USA) 对回收产物进行检测定量。使用NEXTFLEX Rapid DNA-Seq Kit进行建库,文库合格后,进行上机测序。
1.6 数据分析
测序得到的原始数据(raw data)存在一定比例的干扰,为了得到更加准确、可靠的信息分析结果,对原始数据进行拼接、过滤处理得到有效数据(clean data)。基于有效数据采用 Usearch 软件进行OTUs (operational taxonomic units) 聚类和物种分析。根据OTUs聚类分析及各个分类水平下的物种比对结果,利用QIIME软件对每个OTU的代表序列进行多种多样性指数分析,基于分类学信息,在各个分类水平上进行群落结构的统计分析[12]。采用 Sliva 细菌数据库比对细菌得到的OTU代表序列, 采用Unite真菌数据库比对真菌得到的OTU代表序列。进行物种注释分析,获得对应的物种信息和基于物种的丰度分布情况。
同时,对OTUs进行Alpha多样性指数统计、Beta多样性、群落结构、群落热图等分析。从而得到样品内物种丰富度和均匀度等信息、不同样品或分组间的共有和特有OTUs信息以及Beta多样性指数组间差异分析等。
2 结果与分析
2.1 测序数据统计及Alpha多样性统计
用Ace和Chao指数来评估菌群丰富度,Ace指数显示的细菌物种丰度为番茄红酸汤>辣椒红酸汤>白酸汤,真菌物种丰富度为辣椒红酸汤>白酸汤>番茄红酸汤;Chao指数显示的细菌物种丰富度为番茄红酸汤>辣椒红酸汤>白酸汤,真菌物种丰富度为辣椒红酸汤>白酸汤>番茄红酸汤(表1)。Shannon和Simpson指数评估群落多样性,Shannon指数指示的细菌群落多样性为番茄红酸汤>白酸汤>辣椒红酸汤,真菌群落多样性为辣椒红酸汤>番茄红酸汤>白酸汤;Simpson指数指示的细菌群落多样性表现为番茄红酸汤>白酸汤>辣椒红酸汤,真菌群落多样性表现为辣椒红酸汤>番茄红酸汤>白酸汤(表1)。上述8组数据显示,红酸汤的细菌群落多样性>白酸汤,红酸汤的真菌群落多样性>白酸汤。
表1 OTU聚类与多样性分析
Table 1 cluster and diversity analysis of OTU
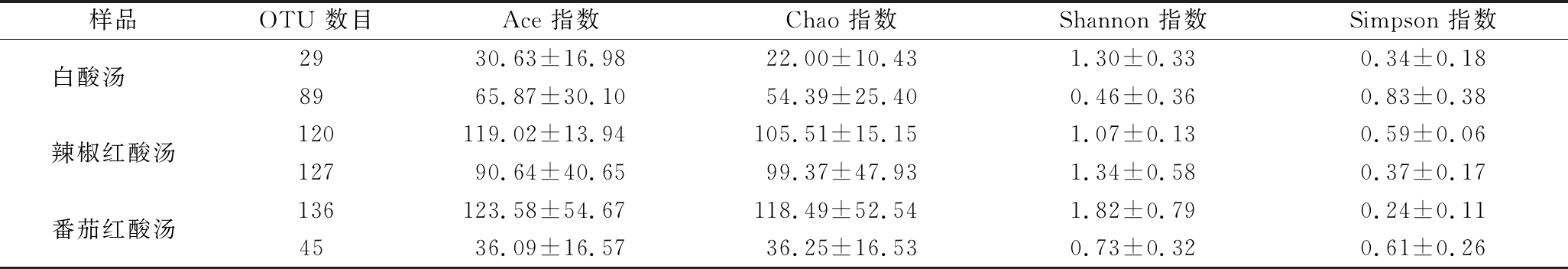
样品OTU 数目Ace 指数Chao 指数Shannon 指数Simpson 指数白酸汤 29 30.63±16.9822.00±10.431.30±0.330.34±0.1889 65.87±30.1054.39±25.400.46±0.36 0.83±0.38辣椒红酸汤120119.02±13.94105.51±15.151.07±0.130.59±0.0612790.64±40.6599.37±47.931.34±0.580.37±0.17番茄红酸汤136123.58±54.67118.49±52.541.82±0.790.24±0.1145 36.09±16.5736.25±16.530.73±0.320.61±0.26
2.2 样品间Beta多样性指数分析
为了评价微生物β多样性,对传统厌氧发酵的白酸汤、辣椒红酸汤、番茄红酸汤进行种水平上的样本层级聚类分析,主要反映了发酵过程中细菌和真菌群落结构的总体差异。两样品越接近,它们之间的种就越相似,根据距离远近进行分析, 白酸汤、辣椒红酸汤、番茄红酸汤细菌菌群明显的分为3个簇,呈鲜明的差异性(图1-a),白酸汤、辣椒红酸汤和番茄红酸汤真菌菌群具有明显的差异性,但是辣椒红酸汤和番茄红酸汤没有明显的差异性(图1-b)。

a-细菌层级聚类;b-真菌层级聚类
图1 样本种水平上的层级聚类
Fig.1 Hierarchical clustering analysis at the species level of sample
注:BST为白酸汤;HSTC为辣椒红酸汤;HSTL为番茄红酸汤(下同)
2.3 OTU聚类、注释及多样性分析
白酸汤、辣椒红酸汤和番茄红酸汤通过测序共获得173个细菌OTUs(图2-a)。其中白酸汤特有细菌菌种1个;辣椒红酸汤特有菌种32个;番茄红酸汤特有菌种49个。白酸汤,辣椒红酸汤和番茄红酸汤共有菌种21个;白酸汤和辣椒红酸汤共有菌种3个;白酸汤和番茄红酸汤共有菌种2个;辣椒红酸汤和番茄红酸汤共有菌种50个。
白酸汤、辣椒红酸汤和番茄红酸汤通过测序共获得99个真菌OTUs(图2-b)。白酸汤特有菌种13个;辣椒红酸汤特有菌种41个;番茄红酸汤特有菌种有2个。白酸汤,辣椒红酸汤和番茄红酸汤共有菌种13个;白酸汤和辣椒红酸汤共有菌种21个; 白酸汤和番茄红酸汤共有菌种2个;辣椒红酸汤和番茄红酸汤共有菌种7个。
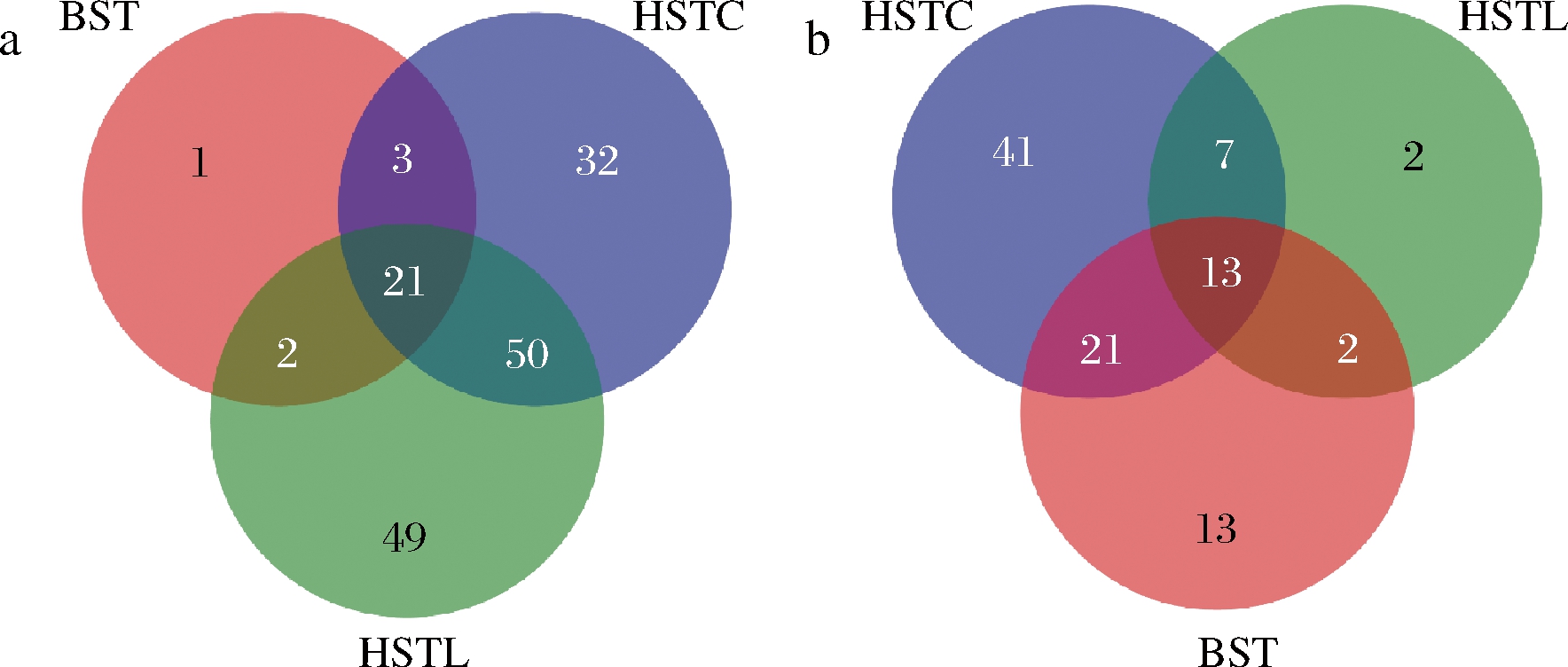
a-细菌Venn图;b-真菌Venn图
图2 样品种水平Venn图
Fig.2 VENN analyies at the species level of sample
2.4 群落结构组分分析
2.4.1 门和属水平上群落结构组分分析
经统计分析表明,白酸汤细菌检测到4个门(Phylum),分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria)、放线菌门(Actinobacteria)。分别为微小杆菌属(Exiguobacterium)、双歧杆菌属(Bifidobacterium)、乳杆菌属(Lactobacillus)、假单胞菌(Pseudomonas)、醋酸杆菌属(Acetobacter)、葡萄醋酸杆菌属(Gluconacetobacter)等19个属(Genus),30个OTUs;真菌有4个门(Phylum)、分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)等。39个属(Genus),分别为青霉属(Penicillium)、篮状菌属(Talaromyces)、Nigrospora、Meyerozyma、链格孢属(Alternaria)等89个OTUs。
辣椒红酸汤细菌检测到9个细菌门(Phylum),分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria)、放线菌门(Actinobacteria)、螺旋体门(Spirochaetes)等。71个属,分别为普雷沃氏菌属(Prevotella)、布氏杆菌属(Bryobacter)、葡萄糖醋酸杆菌(Gluconacetobacter)、慢生根瘤菌(Bradyrhizobium)、酸丙酸杆菌(Acidipropionibacterium)等,120个OTUs;真菌有4个门(Phylum),分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)等。60个属,分别为篮状菌属(Talaromyces)、酵母属(Saccharomyces)、Cyberlindnera、Meyerozyma 、Cystofilobasidium、链格孢属(Alternaria)等,127个OTUs。
番茄红酸汤检测到8个细菌门(Phylum),分别为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、蓝细菌门(Cyanobacteria)等。88个属,分别为Sinomicrobium、拟杆菌属(Bacteroides)、嗜热菌属(Anoxybacillus)、Rosenbergiella、海细菌属(Marinobacterium)、Dysgonomonas、海杆菌属(Marinobacter)等,136个OTUs;真菌有4个门(Phylum)分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、丝孢菌门(Mortierellomycota)等。26个属,分别为Hanseniaspora、Saitozyma、毕赤酵母属(Pichia)、哈萨克斯坦酵母属(Kazachstania)、双足囊菌属(Dipodascus),Plectosphaerella,假丝酵母(Candida)等,45个OTUs。
2.4.2 种水平上群落结构组分分析
白酸汤的细菌优势菌种为43.46%Lactobacillus harbinensis、31.25%布氏乳杆菌(Lactobacillus buchneri)、10.05%干酪乳杆菌(Lactobacillus casei)、9.90%unclassified_g_Acetobacter、1.79%植物乳杆菌(L. plantarum subsp. plantarum)、1.39%液化葡萄糖醋酸杆菌(Gluconacetobacter liquefaciens)、1.38%发酵乳杆菌(Lactobacillus fermentum);辣椒红酸汤的细菌优势菌种为76.25%耐酸乳酸菌(Lactobacillus acetotolerans)、7.35%耐乙醇片球菌(Pediococcus ethanolidurans)、6.03%布氏乳杆菌(L. buchneri)、2.95%面包乳杆菌(Lactobacillus panis)、1.81%uncultured_bacterium_g_Lactobacillus、1.23%未分类的乳酸杆菌(unclassified_g_Lactobacillus)、1.19%unclassified_g_norank_o_Chloroplast、0.74%uncultured_Lactobacillus_p_g_Lactobacillus、0.55%植物乳杆菌(L. plantarum subsp. plantarum);番茄红酸汤的细菌优势菌种为33.38%Lactobacillus zymae、31.18%布氏乳杆菌(L. buchneri)、14.82%耐酸乳酸菌(L.acetotolerans)、9.99%unclassified_g_norank_o_Chloroplast、2.09%耐乙醇片球菌(P. ethanolidurans)、1.73%面包乳杆菌(L. panis)、1.33%类短乳酸菌(Lactobacillus parabrevis)、1.23%未分类的乳酸杆菌(unclassified_g_Lactobacillus)、1.18%uncultured_Lactobacillus_sp._g_Lactobacillus、1.08%植物乳杆菌(L. plantarum subsp. plantarum)、0.87%uncultured_bacterium_g_Lactobacillus(图3-a)。
白酸汤在种水平的优势真菌分别为88.31%膜璞毕赤酵母(Pichia membranifaciens)、6.39%博伊丁假丝酵母(Candida boidinii)、2.86%未分类的子囊菌门(unclassified_p_Ascomycota)、0.95%地丝双足囊菌(Dipodascus geotrichum);辣椒红酸汤在种水平上的优势真菌为74.36%Kazachstania humilis、19.02%酿酒酵母(Saccharomyces cerevisiae)、2.13%地丝双足囊菌(D. geotrichum)、0.96%unclassified_p_Ascomycota、0.84%膜璞毕赤酵母(P. membranifaciens);番茄红酸汤在种水平上的优势真菌为99.17%K. humilis、0.32%膜璞毕赤酵母(P. membranifaciens)(图3-b)。

a-细菌的组成情况;b-真菌的组成情况
图3 样品种水平上的组成情况
Fig.3 Composition at the species level of sample
2.5 Heatmap热图分析
通过对白酸汤、辣椒红酸汤和番茄红酸汤丰度排名前35的物种进行热图分析,反映白酸汤、辣椒红酸汤和番茄红酸汤在属水平上群落组成的相似性和差异性。白酸汤、辣椒红酸汤和番茄红酸汤细菌在属水平丰度不同,但主要分布在变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、蓝细菌门(Cyanobacteria)、拟杆菌门(Bacteroidetes)5个门中。白酸汤优势菌属为葡萄糖醋酸杆菌属(Gluconacetobacter)、双歧杆菌属(Bifidobacterium)、假单胞菌属(Pseudomonas)等;辣椒红酸汤优势菌属为片球菌属(Pediococcus)、乳杆菌属(Lactobacillus)、魏斯氏属(Weissella)、克雷伯氏菌属(Klebsiella)、奈瑟菌属(Neisseria)等;番茄红酸汤优势菌属为假单胞菌属(Pseudomonas)、青枯菌属(Ralstonia)、不动杆菌属(Acinetobacter)、明串珠菌属(Leuconostoc)等(图4-a)。
白酸汤、辣椒红酸汤和番茄红酸汤真菌在属水平丰度不同,但主要分布在子囊菌门(Ascomycota)、毛霉亚门(Mucoromycota)、被孢霉门(Mortierellomycota)、担子菌门(Basidiomycota)4个门中。白酸汤的优势菌属为毕赤酵母属(Pichia)、假丝酵母属(Candida)、双足囊菌属(Dipodascus)、青霉菌属(Penicillium);辣椒红酸汤的优势菌属为分子孢子菌属(Cladosporium)、Kodamaea、针壳孢属(Septoria)等;番茄红酸汤的优势菌属为Kazachstania、毕赤酵母属(Pichia)、双足囊菌属(Dipodascus)、小囊菌属(Microascus)(图4-b)。

a-细菌相对丰度热图;b-真菌相对丰度热图
图4 样本属水平上的相对丰度热图
Fig.4 Relative abundance heatmap at the genus level of sample
2.6 样本与物种的关系
白酸汤、辣椒红酸汤和番茄红酸汤的第一优势菌属均为乳酸菌属(Lactobacillus),第二优势菌属为醋酸杆菌属(Acetobacter)、葡萄糖醋酸杆菌属(Gluconacetobacter)、双球菌属(Pediococcus)、norank_o_Chloroplast,且在辣椒红酸汤和番茄红酸汤中的平均丰度比白酸汤中高(图5-a)。
白酸汤的优势真菌菌属为毕赤酵母属(Pichia)和假丝酵母属(Candida)。辣椒红酸汤和番茄红酸汤的优势菌属为哈萨克斯坦酵母属(Kazachstania)、酵母属(Saccharomyces)。白酸汤、辣椒红酸汤和番茄红酸汤的共有菌属为未分类的子囊菌门(unclassified_p_Ascomycota)、双足囊菌属(Dipodascus),且unclassified_p_Ascomycota在白酸汤、辣椒红酸汤和番茄红酸汤中的平均丰度依次为74%、25%、1.0%;Dipodascus在白酸汤、辣椒红酸汤和番茄红酸汤中的平均丰度依次为29%、65%、6.3%(图5-b)。

a-细菌Circos图;b-真菌Circos图
图5 样本在属水平上的Circos图
Fig.5 Circos at the genus level of sample
3 讨论
酸汤独特风味的形成与微生物发酵紧密相关。酸汤的微生物发酵作用以乳酸为主并伴有少量的酒精发酵和微量的醋酸发酵。白酸汤和辣椒红酸汤、番茄红酸汤的微生物优势菌属不同。白酸汤是将大米、糯米、鲜玉米煮成米汤,接种老酸汤密封发酵而成;红酸汤是将辣椒、番茄处理干净,剁成酱,分别加入适量的糯米汤、食盐、白酒、木姜子进行密封发酵而成。推测造成白酸汤和红酸汤菌落结构差异的主要原因是老酸汤中微生物以及在接种老酸汤时带入的环境微生物,辣椒和番茄表面附着微生物及其内生菌。有机酸是决定发酵食品风味的因素之一。蔬菜本身带有有机酸,发酵过程中也会产生有机酸,不同的蔬菜,有机酸种类和含量也不同[13]。不同的有机酸形成的口感风味不同。
白酸汤,辣椒红酸汤和番茄红酸汤的共同菌种有植物乳杆菌(L. plantarum subsp. plantarum)。植物乳杆菌(L. plantarum subsp. plantarum)在酸汤发酵时使其品质稳定、口味上乘、开胃爽口并且减短发酵周期。植物乳杆菌在干酪成熟过程中起到重要作用,能提高干酪中的游离氨基酸(free amino acid, FAA)含量,其是风味物质的重要前体[14],从而增强干酪的风味[15]。AGUILAR-TOAL 等发现从植物乳杆菌发酵的发酵乳中提取的肽碎片具有抗感染、抗溶血、抗氧化的功能,其中<3 kDa的肽碎片具有更高活性[16]。ZAGO等研究发现植物乳杆菌(L.plantarum subsp. plantarum)能产生叶酸、核黄素,且在发酵乳中具有较好的抗氧化能力[17]。白酸汤中的优势真菌膜璞毕赤酵母(P.membranifaciens)是一种生防酵母,膜璞毕赤酵母(P.membranifaciens)的大量繁殖会使发酵食品表面生花,长白膜,产生不愉快的酸臭味,导致产品变质,氧化酵母能利用乳酸和较低的盐水,而致使其他腐败菌生长,但这只在有氧条件下才发生[18]。推测造成白酸汤中有膜璞毕赤酵母(P.membranifaciens)的原因是白酸汤发酵时老酸汤接种不当或者厌氧发酵环境不好。因此,在后期的生产中应该严格把控老酸汤质量和厌氧发酵环境。白酸汤中的膜璞毕赤酵母(P.membranifaciens)、博伊丁假丝酵母(C.boidinii)是形成其风味的主要微生物。辣椒红酸汤和番茄红酸汤的优势菌属大致相似,但其相对丰度不同,因此推测形成差异的主要原因在于原材料的内生菌差异。辣椒红酸汤和番茄红酸汤中的耐酸乳酸菌(L.acetotoleran)和K.humilis是形成红酸风味特征的主要菌群。耐酸乳酸菌(L.acetotoleran)在酸汤发酵时可以抑制杂菌的生成但是发现在生产过程中,耐酸乳酸菌(L.acetotoleran)能够将葡萄糖等底物转化为醋酸和乳酸,丰富醋的风味。一旦其出现在成品醋中就会引起醋的变质和腐败[19]。K.humilis会增加食品的体外抗氧化活性,而且能提高油炸面团游离氨基酸总量[20]。
等发现从植物乳杆菌发酵的发酵乳中提取的肽碎片具有抗感染、抗溶血、抗氧化的功能,其中<3 kDa的肽碎片具有更高活性[16]。ZAGO等研究发现植物乳杆菌(L.plantarum subsp. plantarum)能产生叶酸、核黄素,且在发酵乳中具有较好的抗氧化能力[17]。白酸汤中的优势真菌膜璞毕赤酵母(P.membranifaciens)是一种生防酵母,膜璞毕赤酵母(P.membranifaciens)的大量繁殖会使发酵食品表面生花,长白膜,产生不愉快的酸臭味,导致产品变质,氧化酵母能利用乳酸和较低的盐水,而致使其他腐败菌生长,但这只在有氧条件下才发生[18]。推测造成白酸汤中有膜璞毕赤酵母(P.membranifaciens)的原因是白酸汤发酵时老酸汤接种不当或者厌氧发酵环境不好。因此,在后期的生产中应该严格把控老酸汤质量和厌氧发酵环境。白酸汤中的膜璞毕赤酵母(P.membranifaciens)、博伊丁假丝酵母(C.boidinii)是形成其风味的主要微生物。辣椒红酸汤和番茄红酸汤的优势菌属大致相似,但其相对丰度不同,因此推测形成差异的主要原因在于原材料的内生菌差异。辣椒红酸汤和番茄红酸汤中的耐酸乳酸菌(L.acetotoleran)和K.humilis是形成红酸风味特征的主要菌群。耐酸乳酸菌(L.acetotoleran)在酸汤发酵时可以抑制杂菌的生成但是发现在生产过程中,耐酸乳酸菌(L.acetotoleran)能够将葡萄糖等底物转化为醋酸和乳酸,丰富醋的风味。一旦其出现在成品醋中就会引起醋的变质和腐败[19]。K.humilis会增加食品的体外抗氧化活性,而且能提高油炸面团游离氨基酸总量[20]。
白酸汤中特有微生物有液化葡糖醋酸杆菌(G.liquefaciens)、哈尔滨乳杆菌(L.harbinensis)、干酪乳杆菌(L.casei)、发酵乳杆菌(L.fermentum)、博伊丁假丝酵母(C.boidinii)。液化葡糖醋酸杆菌(G.liquefaciens)产生醋酸,使得白酸汤酸爽可口,可以和发酵环境中的乙醇发生酯化反应,产生酯类化合物乙酸乙酯,具有芳香气味,赋予酸汤独特的香气。但醋酸含量过高也会影响酸汤成品的品质。因此,在发酵过程中要始终保持好的厌氧环境,缺氧状态可以抑制醋酸菌的生长代谢活动,以保障酸汤的风味品质。发酵乳杆菌(L.fermentum)作为异型发酵乳酸菌,常被用于传统谷物类食品的发酵生产[21]。如在发酵豆乳应用,可消除豆乳中胀气因子α-半乳糖苷类低聚糖的含量[22]。在对不同发酵乳杆菌(L.fermentum)菌株的生理特征研究时发现,很多菌株都具备益生菌的优良特性[23-26]。由于发酵乳杆菌(L.fermentum)具有耐受肠道环境的能力,因此还能在动物肠道中起到原位杀菌或抑菌的效果。干酪乳杆菌(L.casei)具有良好的耐酸及胆汁抗性,可降低血浆胆固醇,增强宿主对微生物病原体的非特异性抵抗力,加快清除肠道内病原体,治疗肠道菌群紊乱和增强肠道透性,从而防止食物过敏和急性腹泻。此外,干酪乳杆菌(L.casei)还可使抗低密度氧化脂抗体和淋巴细胞增加,使粒细胞的噬菌作用明显增强,对宿主进行免疫调节,防止肿瘤的产生[27]。博伊丁假丝酵母(C.boidinii)属于产酯酵母,使得白酸汤有独特的香味。辣椒红酸汤中特有的微生物为酿酒酵母(S.cerevisiae),酵母菌通过酒精发酵产生酒精,既可以增加醇香,也可以抑制有害菌生长,还可以进一步与有机酸反应产生酯类等芳香成分,使发酵食品的风味更加丰富[28]。此外,酵母菌的代谢产物有利于乳酸菌的生长[29-32]。酿酒酵母(S.cerevisiae)能够阻止其pH进一步下降,降低总酸生成量,可有效解决辣椒红酸汤的过度酸化。番茄红酸汤中特有的微生物为L.zymae、Lactobacillus parabrevis。L.zymae在食品中发酵时会产生γ-氨基丁酸(γ-aminobutyric acid,GABA)。GABA是一种非蛋白氨基酸,广泛分布于微生物、植物和动物中。在微生物中,GABA增加耐酸能力和ATP的产生[33]。它是一种神经递质抑制剂,在动物中具有多种重要的生理功能,包括降压、抗焦虑、通便、镇痛和利尿等[34]。因此含有L.zymae的药物或食品具有很好的开发价值。
[1] 曾荣妹,韩琳,黄平. 贵州红酸汤火锅调料的研究进展及工业化进程[J]. 食品与发酵科技, 2014, 50(5): 72-75.
[2] 鲁杨,王楠兰,李贤,等. 凯里红酸汤主要营养和功能成分的分析研究[J]. 食品研究与开发, 2019, 40(7): 163-166.
[3] 田永峰. 贵州苗族发酵型酸汤中功能性乳酸菌的筛选[D].贵阳:贵州大学, 2008.
[4] 郑莎莎,宋煜婷,罗意,等. 凯里白酸汤的优化工艺[J]. 山地农业生物学报, 2016, 35(2): 80-85.
[5] 周文美. 苗家珍味——酸汤[J]. 江苏调味副食品, 2004, 21(4): 27-28.
[6] 张璇. 贵州 “红酸汤” 半成品中微生物区系的研究[D].重庆:西南大学,2011.
[7] 刘昕昕,孟令波,潘明阳,等. 东北设施黑土土壤微生物总DNA提取方法探讨[J]. 生物技术, 2013, 23(2): 45-48.
[8] WALTER J,TANNOCK G W,TILSALA-TIMISJARVI A, et al. Detection and identification of gastrointestinal Lactobacillus species by using denaturing gradient gel electrophoresis and species-specific PCR primers[J]. Applied and Environmental Microbiology, 2000, 66(1): 297-303.
[9] YU Y, LEE C, KIM J, et al. Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction[J]. Biotechnology and Bioengineering, 2005, 89(6): 670-679.
[10] GARDES M,BRUNS T D. Its primers with enhanced specificity for basidiomycetes-application to the identification of mycorrhizae and rusts[J]. Molecular Ecology, 1993, 2(2): 113-118.
[11] 沈馨,尚雪娇,董蕴,等. 基于MiSeq高通量测序技术对3个孝感凤窝酒曲细菌多样性的评价[J]. 中国微生态学杂志, 2018, 30(5): 525-530;544.
[12] EDGAR R C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998.
[13] 郝明玉. 直投式发酵泡菜与自然发酵泡菜的比较研究[D]. 南昌: 南昌大学, 2013.
[14] FOX P F,WALLACE J M. Formation of flavor compounds in cheese[J]. Advances in Applied Microbiology, 1997, 45: 17-85.
[15] SETTANNI L,MOSCHETTI G. Non-starter lactic acid bacteria used to improve cheese quality and provide health benefits[J]. Food Microbiology, 2010, 27(6): 691-697.
[16] AGUILAR-TOAL E J,SANTIAGO-L
E J,SANTIAGO-L PEZ L,PERES C M, et al. Assessment of multifunctional activity of bioactive peptides derived from fermented milk by specific Lactobacillus plantarum strains[J]. Journal of Dairy Science, 2017, 100(1): 65-75.
PEZ L,PERES C M, et al. Assessment of multifunctional activity of bioactive peptides derived from fermented milk by specific Lactobacillus plantarum strains[J]. Journal of Dairy Science, 2017, 100(1): 65-75.
[17] ZAGO M,SCALTRITI E,BONVINI B, et al. Genomic diversity and immunomodulatory activity of Lactobacillus plantarum isolated from dairy products[J]. Beneficial Microbes, 2017, 8(4): 597-604.
[18] 曾骏. 传统四川泡菜发酵过程中酵母菌的动态变化规律及发酵性能研究[D].雅安:四川农业大学, 2013.
[19] 翟磊,程宵宵,苏姣姣,等. 一株食醋污染菌CICC10774的鉴定及其生长代谢特性[J]. 微生物学通报, 2016, 43(7): 1 524-1 531.
[20] MICHELA P,MONICA A,ANTONELLA C, et al. Exploitation of autochthonous tuscan sourdough yeasts as potential starters[J]. International Journal of Food Microbiology, 2019, 302: 59-68.
[21] FENG X M, ERIKSSON A R B, SCHNÜRER J. Growth of lactic acid bacteria and Rhizopus oligosporus during barley tempeh fermentation[J]. International Journal of Food Microbiology, 2005, 104(3): 249-256.
[22] GARRO M S, DE VALDEZ G F, DE GIORI G S. Temperature effect on the biological activity of Bifidobacterium longum CRL 849 and Lactobacillus fermentum CRL 251 in pure and mixed cultures grown in soymilk[J]. Food Microbiology, 2004, 21(5): 511-518.
[23] ZOUMPOPOULOU G, FOLIGNE B, CHRISTODOULOU K, et al. Lactobacillus fermentum ACA-DC 179 displays probiotic potential in vitro and protects against trinitrobenzene sulfonic acid (TNBS)-induced colitis and Salmonella infection in murine models[J]. International Journal of Food Microbiology, 2008, 121(1): 18-26.
[24] FAZELI M R, SHAHVERDI A R, SEDAGHAT B, et al. Sourdough-isolated Lactobacillus fermentum as a potent anti-mould preservative of a traditional Iranian bread[J].European Food Research and Technology, 2004, 218(6): 554-556.
[25] ZENG X Q, PAN D D, ZHOU P D. Functional characteristics of Lactobacillus fermentum F1[J]. Current microbiology, 2011, 62(1): 27-31.
[26] J RVENP
RVENP
 S, TAHVONEN R L, OUWEHAND A C, et al. A probiotic, Lactobacillus fermentum ME-3, has antioxidative capacity in soft cheese spreads with different fats[J]. Journal of dairy science, 2007, 90(7): 3 171-3 177.
S, TAHVONEN R L, OUWEHAND A C, et al. A probiotic, Lactobacillus fermentum ME-3, has antioxidative capacity in soft cheese spreads with different fats[J]. Journal of dairy science, 2007, 90(7): 3 171-3 177.
[27] 张书光,张云娟,代卫东,等. Viili乳制品中干酪乳杆菌的分离鉴定[J]. 微生物学杂志, 2012, 32(3): 47-52.
[28] TOFALO R,PERPETUINI G,SCHIRONE M, et al. Yeast biota associated to naturally fermented table olives from different Italian cultivars[J].International Journal of Food Microbiology, 2013, 161(3): 203-208.
[29] ALVES M,GONÇALVES T,QUINTAS C. Microbial quality and yeast population dynamics in cracked green table olives′ fermentations[J]. Food Control, 2012, 23(2): 363-368.
[30] HERN NDEZ A,MART
NDEZ A,MART N A,ARANDA E, et al. Identification and characterization of yeast isolated from the elaboration of seasoned green table olives[J]. Food Microbiology, 2006, 24(4): 346-351.
N A,ARANDA E, et al. Identification and characterization of yeast isolated from the elaboration of seasoned green table olives[J]. Food Microbiology, 2006, 24(4): 346-351.
[31] ARROYO-L PEZ F,QUEROL A,BAUTISTA-GALLEGO J, et al. Role of yeasts in table olive production[J]. International Journal of Food Microbiology, 2008, 128(2): 189-196.
PEZ F,QUEROL A,BAUTISTA-GALLEGO J, et al. Role of yeasts in table olive production[J]. International Journal of Food Microbiology, 2008, 128(2): 189-196.
[32] 赵欠,王巧碧,周才琼. 酿酒酵母相关营养功能成分的研究及应用进展[J]. 中国酿造, 2015, 34(6): 15-18.
[33] JOHNSON B S,SINGH N K,CHERRY J H, et al. Purification and characterization of glutamate decarboxylase from cowpea[J]. Phytochemistry, 1997, 46(1): 39-44.
[34] PARK J Y,JEONG S,KIM J H. Characterization of a glutamate decarboxylase (GAD) gene from Lactobacillus zymae[J]. Biotechnology Letters, 2014, 36(9): 1 791-1 799.