谷氨酰胺转氨酶(EC 2.3.2.13,transglutaminase,TGase),能催化蛋白质肽链中谷氨酰胺残基的γ-羧酰胺基(酰基供体)转移至赖氨酸残基的ε-氨基(酰基受体),形成ε-(γ-谷氨酰基)赖氨酸共价键,导致蛋白质分子发生交联[1]。目前,TGase已被广泛应用于肉制品、乳制品和豆制品等蛋白类食品的质构改良[2]。随着对其催化本质认识的加深,TGase的应用开始向生物制药[3]、制革、纺织[4]及生物技术[5]等多个非食品领域拓展。1989年,日本天野公司首次从土壤中分离了1株能够分泌TGase的茂源链霉菌(Streptomyces mobaraensis)[6]。与其他来源的TGase相比,微生物发酵生产的TGase具有非Ca2+依赖性的特点,且生产周期短,成本优势明显。随着应用领域的拓展,链霉菌TGase的产量及酶学性质(底物特异性及热稳定性)的研究逐渐限制了对它的应用需求。构建高产TGase的基因工程菌,并进行酶学性质改造成为相关领域的研究热点。
链霉菌TGase是一种胞外酶,在野生菌中以无活性的酶原(pro-TGase)形式分泌,pro-TGase被内源的蛋白酶切除N端酶原区后,转化成活性TGase。由于N端酶原区对TGase的正确折叠及分泌有重要影响[7-9],通常其在异源宿主中需以pro-TGase形式表达。值得注意的是,细菌和真菌异源表达宿主通常缺少能专一切割pro-TGase酶原区的蛋白酶。因此,如何将pro-TGase高效转化为活性TGase成为其异源表达的关键。目前,主要有3种策略获得活性重组TGase:(1)共表达pro-TGase与特异性蛋白酶(SAM-P45[10]或人鼻病毒3C蛋白酶[11]);(2)共表达酶原区与TGase [8,12-13];(3)仅表达pro-TGase,体外添加活化蛋白酶[14]。由于共表达蛋白酶可以对TGase的持续降解[10]或缺少共价连接的酶原区促进其分泌[12],该策略获得的TGase表达量(16.30 U/mL)[15]仍远低于仅表达pro-TGase获得的(51.1 U/mL)[14]。因此,仅表达pro-TGase再体外添加蛋白酶活化的策略,对于制备活性TGase仍具有产量和成本优势。
目前,用于切割N端酶原区使pro-TGase活化的蛋白酶可以从产TGase的野生菌中分离(如S. mobaraensis金属蛋白酶TAMEP)[16-17]或其他链霉菌中提取(如Streptomyces albogriseolus 分泌的SAM-P20、SAM-P26及SAM-P45等)[18]。此外,一系列商品化的蛋白酶同样能活化pro-TGase,如dispase、中性蛋白酶和胰蛋白酶等[19]。与其他的蛋白酶相比,S. mobaraensis来源的TAMEP能够精准地切割pro-TGase的N端酶原区,活化效率更高且不会对活化后的TGase进行持续降解[20]。研究发现,S. mobaraensis来源TAMEP属M4蛋白酶,在野生菌中仅有少量分泌至胞外,且活性受到链霉菌、枯草杆菌蛋白酶抑制剂的抑制[16,20-21]。最近,S. mobaraensis TAMEP被成功表达于大肠杆菌,但仅存在于细胞周质空间,并不利于其大规模发酵制备[20]。基于此,实现TAMEP高效分泌表达将有助于降低重组pro-TGase的活化成本,提高TGase生产效率。
本研究将来源于S. mobaraensis的TAMEP表达于大肠杆菌,通过信号肽类型优化、信号肽N端密码子同义替换及诱导条件优化,实现了TAMEP的高效分泌,并分析了其酶学性质及对pro-TGase的活化效率。研究结果将有助于TAMEP的规模化制备。
1 材料与方法
1.1 材料与试剂
菌株与质粒:表达质粒pET-22b(+)、大肠杆菌Escherichia coli JM109和E. coli BL21(DE3)由本实验室保存。产S. mobaraensis pro-TGase的生产菌Yarrowia lipolytica/pINA1297/TGase由本实验前期构建[22]。编码S. mobaraensis TAMEP及天然信号肽TSP基因[20]的质粒pUC18/TSP-TAMEP由上海生工合成。
LB培养基(g/L):酵母提取物 5,胰蛋白胨10,NaCl 10。TB培养基(g/L):胰蛋白胨 12,酵母提取物 24,K2HPO4·3H2O 16.37,KH2PO4 2.31,甘油 5。Y. lipolytica 发酵培养基(g/L):甘油 15,酵母膏 20,NH4Cl 2.64,KH2PO4 0.32,MgSO4 0.25,维生素B1 3.34×10-4,调pH至8.0。YPD培养基(g/L):酵母膏 10,蛋白胨 20,葡萄糖 20。LB培养基、TB培养基在接种前需加入终质量浓度为50 μg/mL的氨苄青霉素。
酶、试剂、引物和DNA测序:限制性内切酶、DNA聚合酶购自Takara公司。dispase、GSH、质粒提取试剂盒、DNA回收试剂盒和抗生素购自上海生工公司。Z-Gln-Gly-OH、L-谷氨酸-γ-单羟肟酸、组氨酸标签蛋白抗体和显色剂购自赛默飞公司。BCA蛋白浓度检测试剂盒和SDS-PAGE试剂盒购于碧云天公司。引物构建、DNA测序由上海生工公司完成。
1.2 实验方法
1.2.1 TAMEP表达质粒的构建与转化
以pUC18/TAMEP为模板,分别用引物对TSPTAMEP1/TAMEP2和TAMEP1/TAMEP2进行PCR扩增,得到片段TSP-TAMEP(天然信号肽TSP基因+TAMEP基因)和TAMEP。PCR反应条件为: 98 ℃预变性90 s;随后进行30个循环扩增(98 ℃ 15 s,55 ℃ 15 s,72 ℃ 30 s);最后72 ℃保温5 min。将TSP-TAMEP和TAMEP分别克隆至pET-22b(+)的Nde I-Xho I和Nco I-Xho I酶切位点,得到表达载体pET22b-TSP-TAMEP和pET22b-pelB-TAMEP。将所得表达载体转化E. coli BL21(DE3),得到重组菌E. coli BL21(DE3)(pET22b-TSP-TAMEP)和E. coli BL21(DE3)(pET22b-pelB-TAMEP)。质粒构建相关引物见表1。
表1 本研究所用引物
Table 1 Primers used in this study

引物名称引物序列(5’-3’)TAMEP1CATGCCATGGATGGGCAGGACAAGGCTAMEP2CCGCTCGAGGAAGGTCAGCCGCCATSPTAMEP1GGAATTCCATATGAGACCCACCCCCCAGAGAMut-A1TTAACTTTAAGAAGGAGATATACATAT-GCGNCCNACHCCNCARCGNCGNGCNGTNGCNACCGGCGCCCTCGTCGCCMut-A2TTAACTTTAAGAAGGAGATATACATAT-GCGNCCNACHCCNCARCGNAGRGCNGTNGCNACCGGCGCCCTCGTCGCCMut-A3TTAACTTTAAGAAGGAGATATACATAT-GCGNCCNACHCCNCARAGRCGNGCNGTNGCNACCGGCGCCCTCGTCGCCMut-A4TTAACTTTAAGAAGGAGATATACATAT-GCGNCCNACHCCNCARAGRAGRGCNGTNGCNACCGGCGCCCTCGTCGCCMut-A5TTAACTTTAAGAAGGAGATATACATAT-GAGRCCNACHCCNCARCGNCGNGCNGTNGCNACCGGCGCCCTCGTCGCCMut-A6TTAACTTTAAGAAGGAGATATACATAT-GAGRCCNACHCCNCARCGNAGRGCNGTNGCNACCGGCGCCCTCGTCGCCMut-A7TTAACTTTAAGAAGGAGATATACATAT-GAGRCCNACHCCNCARAGRCGNGCNGTNGCNACCGGCGCCCTCGTCGCCMut-A8TTAACTTTAAGAAGGAGATATACATAT-GAGRCCNACHCCNCARAGRAGRGCNGTNGCNACCGGCGCCCTCGTCGCCMut-BCATATGTATATCTCCTTCTTAAAGTTAAACAAAATTATT
1.2.2 优化TAMEP信号肽N端密码子同义随机替换
以Mut-A1、Mut-A2、Mut-A3、Mut-A4、Mut-A5、Mut-A6、Mut-A7、Mut-A8为上游引物,以Mut-B为下游引物,对pET22b-TSP-TAMEP进行PCR扩增,在TAMEP信号肽TSP的 N端前10个氨基编码基因中随机引入同义密码子。PCR反应条件为: 98 ℃预变性90 s;随后进行30个循环扩增(98 ℃ 15 s,55 ℃ 15 s,72 ℃ 3 min);最后72 ℃保温5 min。PCR产物混合,并经Dpn I处理后转化,转化后涂布氨苄青霉素抗性的LB培养基平板。挑取转化子至含LB培养基的96深孔板中培养,37 ℃培养8 h;转接装有TB培养基的96深孔板,37 ℃培养2 h,加入终浓度10 μmol/L IPTG,20 ℃继续培养40 h。将高产转化子转接摇瓶培养复筛。
1.2.3 重组菌发酵优化
重组大肠杆菌发酵:取TAMEP生产菌液100 μL接入LB培养基,37 ℃培养10 h转接至TB培养基于37 ℃培养;当菌体OD600达到一定值(0.5~3.0)时,加入一定量的IPTG(0~200 μmol/L),同时降温培养(16~30 ℃)培养40 h;为提高TAMEP胞外表达水平,分别添加2~10 g/L的甘氨酸、葡萄糖或体积分数为0.2%~1.0%乙醇。各培养基使用前添加终质量浓度50 μg/mL的氨苄青霉素。
重组Y. lipolytica发酵:将pro-TGase生产菌液接种于YPD液体培养基中,于28 ℃培养24 h;将种子液以10%的接种量转接于发酵培养基中,于28 ℃摇瓶培养120 h。
1.2.4 蛋白质分离纯化
采用镍柱对TAMEP及pro-TGase进行纯化。重组菌发酵上清液经0.22 μmol/L膜过滤制成上样样品。将样品注入经结合缓冲液平衡的镍柱,并用结合缓冲液洗去未结合蛋白;随后以0~500 mmol/L咪唑溶液进行梯度洗脱,收集含有TAMEP或TGase催化活性的部分(pro-TGase样品经dispase活化)。所得纯化样品经脱盐柱去除盐离子。
1.2.5 TAMEP酶学性质检测
最适反应温度:在反应温度为20~80 ℃测定TAMEP活性,以所测得的最高酶活力为100%,计算其他反应温度下的相对酶活力。
最适反应pH:测定pH为5~10条件下TAMEP活性(pH 5:50 mmol/L柠檬酸-柠檬酸钠缓冲液;pH 6~8:50 mmol/L K2HPO4-KH2PO4缓冲液;pH 9~10:50 mmol/L甘氨酸-NaOH缓冲液),以所测得的最高活性为100%,计算其他反应pH下的相对酶活力。
热稳定性:将TAMEP分别于30~60 ℃下孵育0~60 min后,冰水浴5 min并测定其催化活性,未经热处理的酶活力设为100%,计算其他条件下的相对酶活力。
pH稳定性:将TAMEP用pH为5~10缓冲液稀释相同倍数,于30 ℃水浴中孵育60 min测定酶活力,以测得最高酶活力为100%,计算其他pH下孵育的相对酶活力。
1.2.6 pro-TGase活化分析
将纯化的pro-TGase与不同浓度的TAMEP溶液混合均匀,于37 ℃恒温30 min。在该反应体系中,pro-TGase浓度为6.91 μmol/L,TAMEP浓度分别为0.133、0.066、0.033和0.017 μmol/L。反应开始后,每间隔10 min取样50 μL,进行TGase活力测定;同时取样10 μL与SDS-PAGE电泳上样缓冲液混合于90 ℃加热10 min后进电泳分析。
1.2.7 酶活力检测
按照GB/T 23527—2009《蛋白酶制剂》中福林法测蛋白酶活性方法测定TAMEP酶活力。1 U TAMEP酶活力定义为40 ℃下每1 min水解酪蛋白产生1 μg酪氨酸的酶量。以用α-N-CBZ-Gln-Gly和盐酸羟胺为作用底物测定TGase酶活力[7]。1 U TGase酶活力定义为37 ℃每1 min催化生成1 μmol L-谷氨酸-γ-单羟肟酸的酶量。
1.2.8 蛋白质分析
使用BCA蛋白浓度检测和采用碧云天试剂盒制备SDS-PAGE电泳凝胶,具体操作参考产品说明书。使用浓缩胶质量浓度为50 g/L,分离胶质量浓度为120 g/L,以质量浓度1 g/L考马斯亮蓝R-250进行染色。使用His-tag标签抗体进行Western blot,具体方法参考前期工作[23]。
2 结果与分析
2.1 信号肽来源优化提高TAMEP在E. coli中的分泌表达
在E. coli中,融合于N端的信号肽可介导蛋白质分泌[24]。因此,分别将TAMEP天然信号肽[20]和大肠杆菌pelB信号肽融合至TAMEP的N端,构建得到表达质粒pET22b-TSP-TAMEP和pET22b-pelB-TAMEP(图1)。转化至E. coli BL21(DE3)分别得到重组菌E. coli BL21(DE3)(pET22b-TSP-TAMEP)和E. coli BL21(DE3)(pET22b-pelB-TAMEP)。
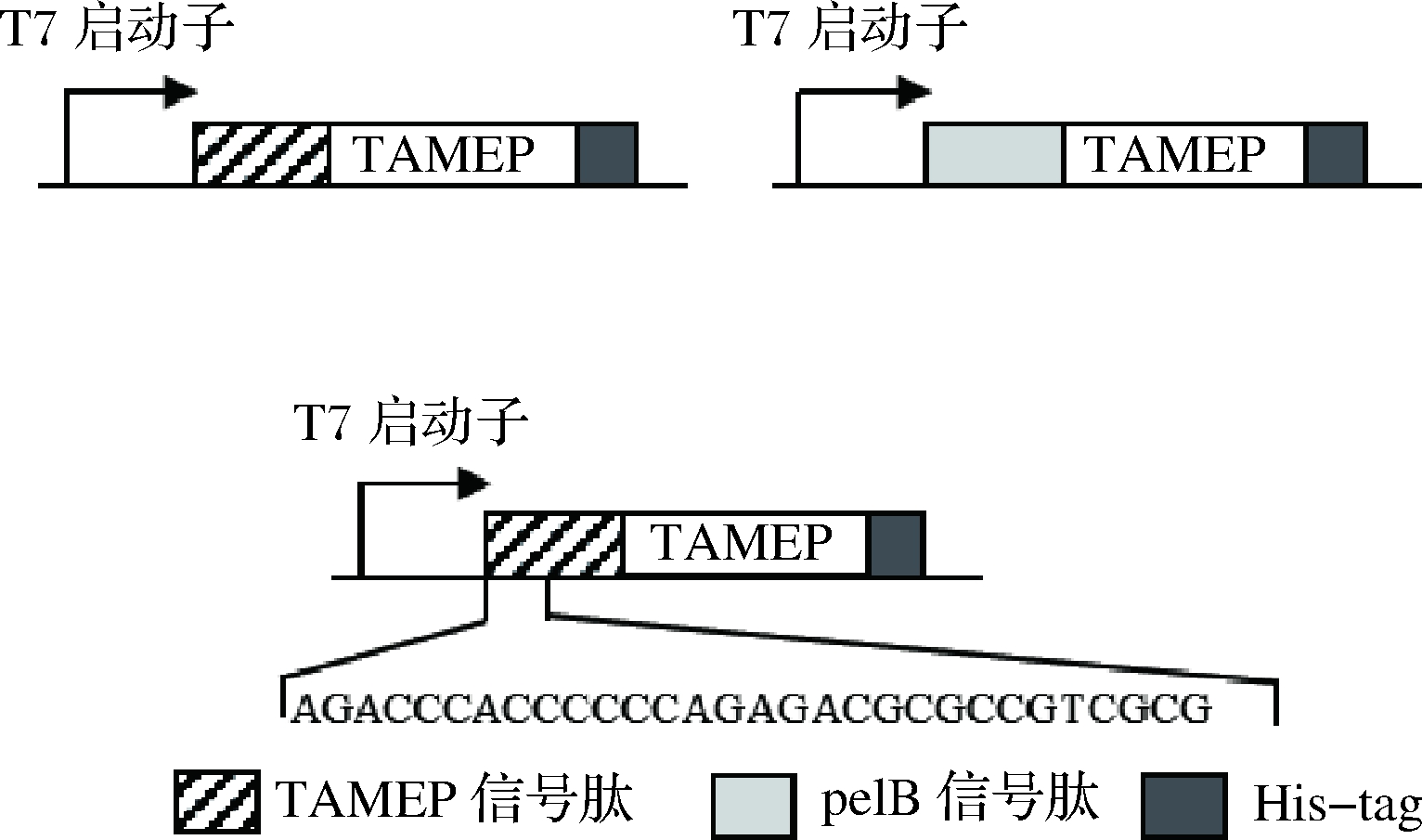
图1 表达质粒示意图
Fig.1 The scheme of expression plasmids
将E.coli BL21(DE3)(pET22b-TSP-TAMEP)、E. coli BL21(DE3)(pET22b-pelB-TAMEP)及对照菌E. coli BL21(DE3)(pET-22b(+))于TB培养基中进行诱导表达。诱导温度、诱导起始菌体浓度(OD600)、IPTG浓度和诱导时间,分别为20 ℃、0.8、100 μmol/L和40 h。如图2-a所示,使用TAMEP天然信号肽的重组菌胞外蛋白酶活力达到20.8 U/mL,较pelB信号肽条件下酶活力提高92.6%;由于存在一定量的内源蛋白酶,对照菌仍能检测到一定的蛋白酶活力。由于胞外蛋白酶条带较多,TAMEP的条带在SDS-PAGE电泳图中并不清晰(图2-b)。Western Blot分析结果显示,在E. coli BL21(DE3)(pET22b-TSP-TAMEP)、E. coli BL21(DE3)(pET22b-pelB-TAMEP)发酵上清液中均能检测到条带,且与TAMEP理论分子质量56.4 kDa接近(图2-c)。对照E. coli BL21(DE3)(pET-22b(+))样品,未能检测到相同条带(图2-c)。上述结果表明,TAMEP天然信号肽能有效介导TAMEP在E. coli BL21(DE3)中的分泌,且性能优于E. coli内源信号肽pelB。

a-重组菌胞外TAMEP酶活力;b-重组菌胞外SDS-PAGE电泳;
c-重组菌胞外Western Blot
M-蛋白质标准分子质量;1-E. coli BL21(DE3)(pET22b-TSP-
TAMEP);2-E. coli BL21(DE3)(pET22b-pelB-TAMEP);
3-E. coli BL21(DE3)(pET-22b(+))
图2 分泌信号肽类型对TAMEP表达的影响
Fig.2 Type of signal peptide on the expression of TAMEP
注:TAMEP位置用箭头标出
2.2 信号肽N端密码子同义突变提高TAMEP在E. coli中的表达
研究表明,蛋白质N端编码区基因序列对其表达效率有重要影响,但确切的机制尚不清楚[25-26]。为进一步提高TAMEP在E. coli中的表达,基于pET22b-TSP-TAMEP对TAMEP天然信号肽N端前10个氨基酸的密码子进行随机同义突变(图1),并转化E. coli BL21(DE3)。通过微孔板对1 656个转化子进行初筛,选取了20株蛋白酶活力较高的菌株进行摇瓶复筛。如图3-a所示,20株菌中17株菌的酶活力较E. coli BL21(DE3)(pET22b-TSP-TAMEP)高;其中,菌株17较出发菌提高了30%,达到了27.0 U/mL。基因序列分析显示,上述17株菌中TAMEP信号肽N端的前10个氨基酸编码基因均发生了不同程度的同义突变。
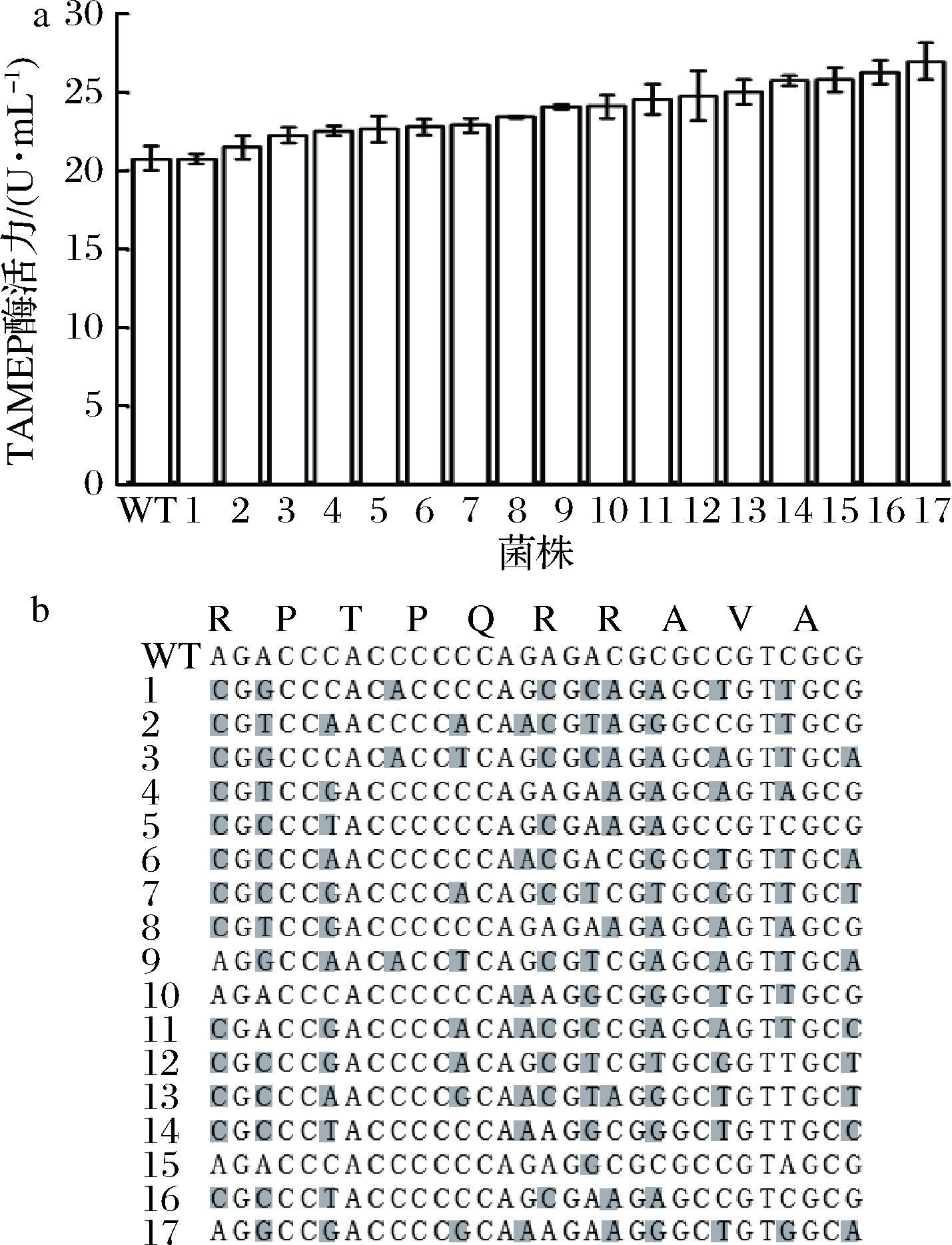
a-突变株摇瓶发酵上清液TAMEP酶活力;b-突变株TAMEP信号肽
N端基因序列
WT-E. coli BL21(DE3)(pET22b-TSP-TAMEP);
1-17-TAMEP信号肽N端密码子同义突变体
图3 信号肽N端前10个氨基酸密码子同义突变对
TAMEP在E. coli中表达的影响
Fig.3 Effect of synonymous mutation within first 10 residues
of the TAMEP signal peptide on its expression in E.coli
2.3 提高TAMEP在E.coli中表达的发酵条件优化
为提高TAMEP在E. coli BL21(+)中的表达量,通过单因素实验依次对上述17株菌的诱导温度、诱导剂IPTG浓度、诱导起始菌体浓度及添加剂进行优化。如图4所示,对TAMEP分泌表达影响最大的是添加剂,其次为诱导温度和诱导剂浓度;诱导起始菌体浓度对酶表达的影响最小。在所选的添加剂中,乙醇能够刺激细胞热休克蛋白的表达,防止所表达蛋白的错误折叠;葡萄糖可提高宿主细胞中的质粒稳定性;甘氨酸、曲通及吐温等可以改善细胞膜通透性,提高重组蛋白向胞外转运的效率[27]。葡萄糖对TAMEP分泌表达的促进作用最大,表明质粒稳定性可能是其高效表达的限制性因素。最终确定TAMEP在E. coli中高效分泌表达的条件为:诱导温度20 ℃、IPTG浓度为10 μmol/L、诱导起始OD600值为1.5及添加10 g/L葡萄糖。在此条件下,胞外TAMEP酶活力达到186.3 U/mL,较优化前提高了5.9倍。
2.4 TAMEP的纯化及酶学性质研究
本研究中表达的重组TAMEP和前期研究中表达的重组pro-TGase均在C端添加了由6个组氨酸构成的His-tag,故采用镍柱对这2个蛋白进行亲和纯化。经洗脱条件优化,确定TAMEP和pro-TGase洗脱的咪唑浓度分别为50和250 mol/L。如图5所示,纯化的蛋白无明显杂带。经脱盐柱去除咪唑,测定TAMEP比酶活力为437 U/mg。

a-诱导温度TAMEP表达的影响;b-IPTG浓度TAMEP表达的影响;c-诱导起始菌株浓度TAMEP表达的影响;d-添加剂TAMEP表达的影响
图4 诱导表达条件对TAMEP表达的影响
Fig.4 Effects of induction conditions on the TAMEP expression

M-蛋白质标准分子质量;1-纯化TAMEP 50 mol/L洗脱液;
2-纯化pro-TGase 250 mol/L洗脱液
图5 SDS-PAGE电泳分析TAMEP
Fig.5 SDS-PAGE analysis of TAMEP
注:TAMEP和pro-TGase位置分别用单箭头和双箭头标出(下同)
对纯化后的TAMEP进行了酶学性质分析。如图6-a和图6-b所示,TAMEP的最适反应温度和最适反应pH分别为55 ℃和7.0。稳定性分析结果显示,TAMEP在30~50 ℃孵育60 min酶活力保持在50%以上,60 ℃孵育相同时间酶活力几乎全部损失(图6-c);TAMEP在pH 6.2~8.9最稳定,并且在碱性环境中比在酸性环境中更稳定。上述性质与S. mobaraensis TAMEP相似,将有助于高效活化pro-TGase的同时,确保活化后TGase的稳定性。
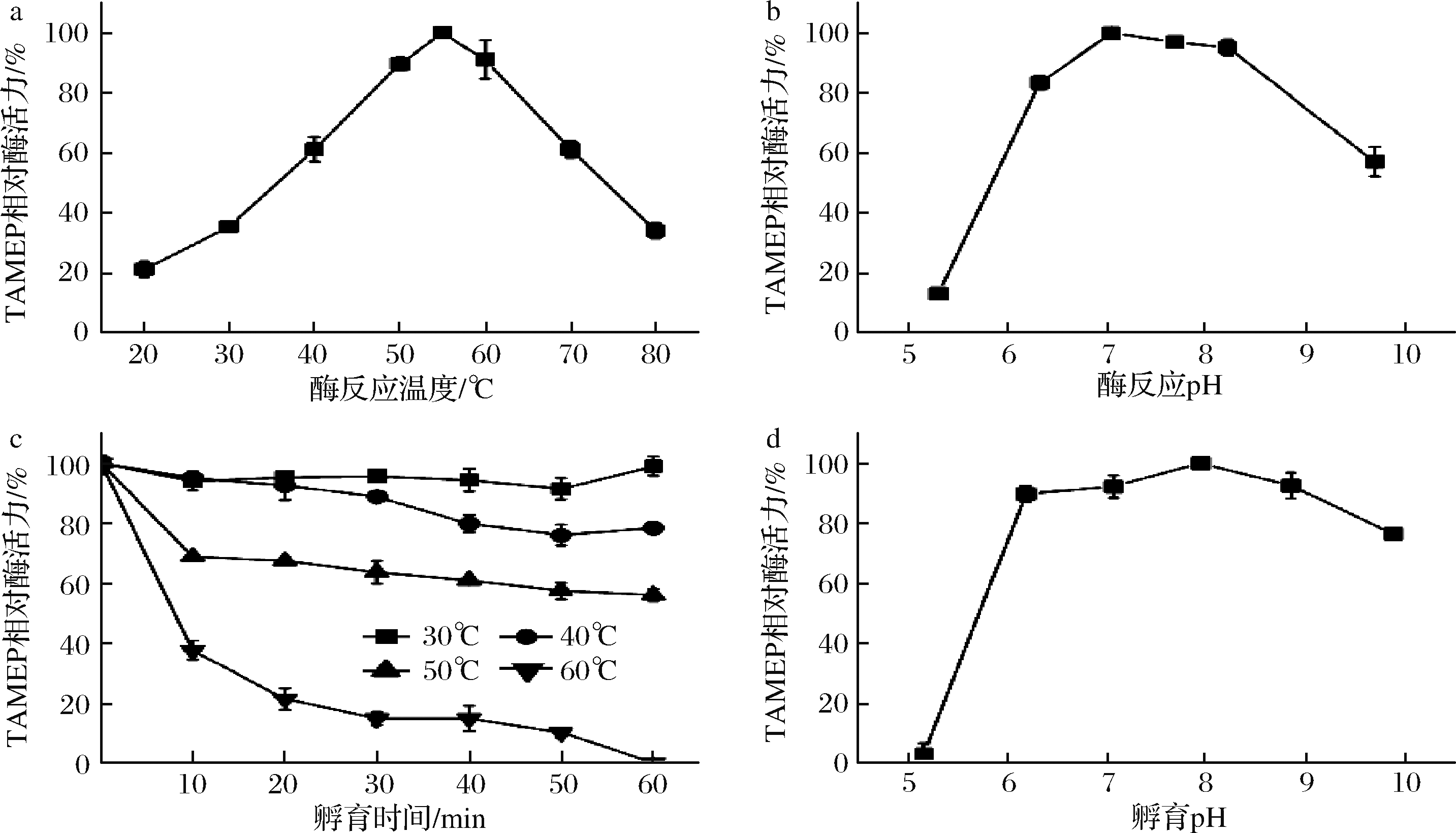
a-温度对TAMEP催化活性影响;b-pH对TAMEP催化活性影响;c-温度对TAMEP稳定性影响;d-pH对TAMEP稳定性影响
图6 TAMEP酶学性质分析
Fig.6 Catalytic property analysis of TAMEP
2.5 TAMEP活化pro-TGase
为研究重组TAMEP对pro-TGase的活化,用0.017~0.133 μmol/L TAMEP活化6.91 μmol/L pro-TGase 30 min,每隔10 min取样进行TGase酶活力检测和电泳分析。TGase酶活力分析显示,TGase活性随着活化反应进行而逐渐升高;相同的活化时间下,TGase活性随着加入TAMEP的浓度升高而升高(图7-a)。
如图7-b所示,随着活化反应的进行,pro-TGase(43.4 kDa)迅速转化为TGase(39.2 kDa),较高浓度TAMEP可加快这个转化过程,并且未产生其他小的蛋白条带。上述结果表明,重组TAMEP能高效催化pro-TGase活化,并不会降解活化后的TGase。
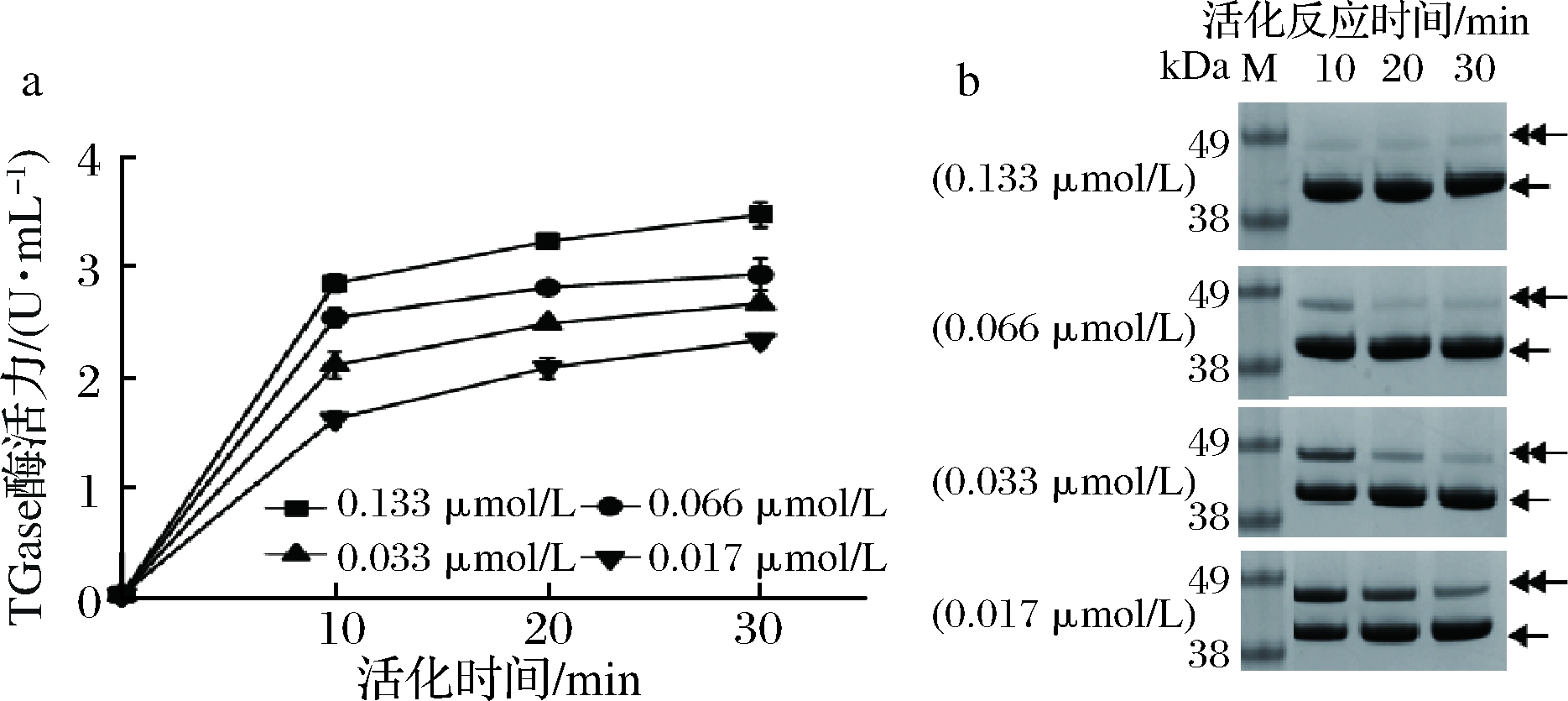
a-TGase酶活;b-SDS-PAGE电泳
图7 重组TAMEP的浓度对pro-TGase活化的影响
Fig.7 Effect of TAMEP concentration on the
activation of pro-TGase
注:SDS-PAGE左侧括号中的数值为TAMEP浓度
3 结论与讨论
重组链霉菌pro-TGase异源表达后,需要高效且专一性强的活化蛋白将其转化为活性TGase。S. mobaraensis TAMEP作为pro-TGase天然活化蛋白,较其他蛋白酶更具优势。本研究,我们将S. mobaraensis来源的TAMEP表达于E. coli,并通过多重优化策略(信号肽类型、N端同义密码子优化、优化诱导条件),实现了TAMEP的高效分泌表达,胞外TAMEP酶活力达到了186.3 U/mL。与胞内表达相比[20],高效分泌表达将有助于TAMEP的分离提取,降低产酶成本。酶学性质分析显示,重组TAMEP 与TGase具有近似稳定pH和温度范围,将有助于前者对后者的活化。研究结果为TAMEP规模化生产提供了生产菌株和基础数据。值得注意的是,本研究采用价格较高的IPTG作为诱导剂,同时活化过程中TAMEP不能重复利用。因此,进一步考查自诱导表达的条件及研究TAMEP的固化策略将有助于其工业化应用。
[1] YOKOYAMA K,NIO N,KIKUCHI Y.Properties and applications of microbial transglutaminase[J].Applied Microbiology and Biotechnology,2004,64(4):447-454.
[2] TAGHI G S M,SHIMA Y,CHRONAKIS I S.Microbial transglutaminase in noodle and pasta processing[J].Critical Reviews in Food Science and Nutrition,2019,59(2):313-327.
[3] SOO K C,THEAM S L.Bioengineering of microbial transglutaminase for biomedical applications[J].Applied Microbiology and Biotechnology,2019,103(7):2 973-2 984.
[4] DUARTE L,MATTE C R,BIZARRO C V,et al.Review transglutaminases: part II-industrial applications in food,biotechnology,textiles and leather products[J].World Journal of Microbiology & Biotechnology,2019,36(1):11.
[5] STROP P.Versatility of microbial transglutaminase[J].Bioconjugate Chemistry,2014,25(5):855-862.
[6] ANDO H,ADACHI M,UMEDA K,et al.Purification and characteristics of a novel transglutaminase derived from microorganisms[J].Agricultural and Biological Chemistry,1989,53(10):2 613-2 617.
[7] LIU S,ZHANG D,WANG M,et al.The pro-region of Streptomyces hygroscopicus transglutaminase affects its secretion by Escherichia coli[J].FEMS Microbiology Letters,2011,324(2):98-105.
[8] YURIMOTO H,YAMANE M,KIKUCHI Y,et al.The pro-peptide of Streptomyces mobaraensis transglutaminase functions in cis and in trans to mediate efficient secretion of active enzyme from methylotropic yeasts[J].Bioscience Biotechnology and Biochemistry,2004,68(10):2 058-2 069.
[9] LIU X,YANG X,XIE F,et al.On-column refolding and purification of transglutaminase from Streptomyces fradiae expressed as inclusion bodies in Escherichia coli[J].Protein Expression and Purification,2007,51(2):179-186.
[10] KIKUCHI Y,DATE M,YOKOYAMA K,et al.Secretion of active-form Streptoverticillium mobaraense transglutaminase by Corynebacterium glutamicum: processing of the pro-transglutaminase by a cosecreted subtilisin-Like protease from Streptomyces albogriseolus[J].Applied and Environmental Microbiology 2003,69(1):358-366.
[11] ZHAO X,SHAW A C,WANG J,et al.A novel high-throughput screening method for microbial transglutaminases with high specificity toward Gln141 of human growth hormone[J].Journal of Biomolecular Screening,2010,15(2):206-212.
[12] LIU S,ZHANG D,WANG M,et al.The order of expression is a key factor in the production of active transglutaminase in Escherichia coli by co-expression with its pro-peptide[J].Microbial Cell Factories,2011,10(1):112.
[13] 李鹏飞,孙红兵,游丽金,等.利用毕赤酵母系统直接分泌表达具有活性的谷氨酰胺转胺酶[J].生物工程学报,2013,29(2):180-188.
[14] ITAYA H,KIKUCHI Y.Secretion of Streptomyces mobaraensis pro-transglutaminase by coryneform bacteria[J].Applied Microbiology and Biotechnology,2008,78(4):621-625.
[15] DATE M,YOKOYAMA K I,UMEZAWA Y,et al.Highlevel expression of Streptomyces mobaraensis transglutaminase in Corynebacterium glutamicum using a chimeric pro-region from Streptomyces cinnamoneus transglutaminase[J].Journal of Biotechnology,2004,110(3):219-226.
[16] ZOTZEL J,KELLER P,Fuchsbauer H L.Transglutaminase from Streptomyces mobaraensis is activated by an endogenous metalloprotease[J].European Journal of Biochemistry,2003,270(15):3 214-3 222.
[17] ZOTZEL J,PASTERNACK R,PELZER C,et al.Activated transglutaminase from Streptomyces mobaraensis is processed by a tripeptidyl aminopeptidase in the final step[J].European Journal of Biochemistry,2003,270(20):4 149-4 155.
[18] TAGUCHI S,ARAKAWA K,YOKOYAMA K,et al.Overexpression and purification of microbial pro-transglutaminase from Streptomyces cinnamoneum and in vitro processing by Streptomyces albogriseolus proteases[J].Journal of Bioscience and Bioengineering,2002,94(5):478-481.
[19] MARX C K,HERTEL T C,PIETZSCH M.Purification and activation of a recombinant histidine-tagged pro-transglutaminase after soluble expression in Escherichia coli and partial characterization of the active enzyme[J].Enzyme and Microbial Technology,2008,42(7):568-575.
[20] JUETTNER N E,CLASSEN M,COLIN F,et al.Features of the transglutaminase-activating metalloprotease from Streptomyces mobaraensis DSM 40847 produced in Escherichia coli[J].Journal of Biotechnology,2018,281:115-122.
[21] ZHANG L,HAN X,ZHANG L.Determination of key protease for TGase yield from Streptomyces mobaraensis[J].Food and Fermentation Industries,2014,40(2):6-9.
[22] 任蕊蕊,刘松,李江华,等.分子改造提高谷氨酰胺转氨酶的催化活性[J].食品与发酵工业,2018,44(9):9-14.
[23] ZHAO W,LIU L,DU G,et al.A multifunctional tag with the ability to benefit the expression,purification,thermostability and activity of recombinant proteins[J].Journal of Biotechnology,2018,283:1-10.
[24] CHEN W,NIE Y,XU Y.Signal peptide-independent secretory expression and characterization of pullulanase from a newly isolated Klebsiella variicola SHN-1 in Escherichia coli[J].Applied Biochemistry and Biotechnology,2013,169(1):41-54.
[25] YANG H,LIU L,SHIN H,et al.Comparative analysis of heterologous expression,biochemical characterization optimal production of an alkalineα-amylase from alkaliphilic Alkalimonas amylolytica in Escherichia coli and Pichia pastoris[J].Biotechnology Progress,2013,29(1):39-47.
[26] CHEONG D E,KO K C,HAN Y,et al.Enhancing functional expression of heterologous proteins through random substitution of genetic codes in the 5′ coding region[J].Biotechnology and Bioengineering,2015,112(4):822-826.
[27] BOSTROM M,MARKLAND K,SANDEN A M,et al.Effect of substrate feed rate on recombinant protein secretion,degradation and inclusion body formation in Escherichia coli[J].Applied Microbiology and Biotechnology,2005,68(1):82-90.