随着社会经济的发展和人们生活水平的改善,食品安全问题越来越得到人们的重视,但食源性疾病依然在各个国家广泛爆发并导致严重后果[1]。造成人类食源性疾病的主要致病菌有大肠杆菌O157:H7、金黄色葡萄球菌、沙门氏菌、单增李斯特菌、空肠弯曲菌和弧菌等[2-3],其中检出率最高的是沙门氏菌,其次是志贺氏菌,之后分别为金黄色葡萄球菌、致泻性大肠埃希菌和蜡样芽孢杆菌[4]等。食品中富含各种营养物质,是致病菌生长繁殖的良好基质,在加工、包装、配送和储存等过程中极易受到致病菌污染。消费者食用了含有致病菌的食物,会出现呕吐、腹泻等症状甚至导致死亡[5]。因此,食源性致病菌的检测对于保障消费者安全至关重要。
食源性致病菌的鉴定、检测方法有很多。其中传统的方法是平板菌落计数法,然而这种方法劳动强度大且耗时长,往往需要7~10 d才能得到最终结果,无法满足快速、原位检测致病菌的需求。此外还有基于免疫学的方法(抗原抗体相互作用)、循环介导等温扩增和三磷酸腺苷生物发光法(adenosine triphosphate bioluminescence, ATP)生物发光技术等,尽管这些方法灵敏并且可以同时检测一种或多种微生物,但普遍存在耗时费力等缺点。因此,为了确保食品安全,防止食源性疾病传播,迫切需要快速、准确的食源性致病菌检测方法。
随着纳米材料的快速发展,电子、光学和电化学等科学的不断进步,目前人们已探究并建立了各种新颖的检测分析方法,如微流体法[6]、流式细胞术、基于核酸的分析方法[7]、生物传感器、电子鼻和电子舌[8]、光谱成像技术[9]等,都适合食源性致病菌的快速检测。通过对上述各种技术的对比分析,本文综述了适合快速检测最有潜力的3种方法:振动光谱学技术、聚合酶链式反应(polymerase chain reaction,PCR)和生物传感器。本文在简介了相关背景和技术原理后,详细分析讨论了其在食源性致病菌快速检测中的应用,为食源性致病菌检测方法的研究和食品产业的应用选择提供参考。
1 振动光谱学技术
在致病菌的检测方法中,振动光谱技术具有快速、无损、可以实时在线检测并且样品制备简单的优点,在食品产业中的应用最具前景和吸引力。近年来,拉曼光谱和近红外光谱技术在致病菌的定量和定性检测中显示出巨大潜力。细菌的不同菌种和菌株的振动光谱具有极大的相似性,但由于不同菌种或菌株间的生化成分和含量往往存在差异,因此需要结合化学计量法,例如层次聚类分析(hierarchical Cclustering analysis,HCA)、主成分分析(principal component analysis,PCA)、人工神经网络和支持向量机(support vector machine, SVM)等,可以对同种或多种致病菌进行分类和鉴别。
1.1 拉曼光谱技术
拉曼光谱属于振动光谱技术。单色光和振动分子间相互作用时可以产生弹性碰撞和非弹性碰撞。光子与振动分子的弹性碰撞产生的散射称为“瑞利散射”;在光子与振动分子的非弹性碰撞中,能量要么从光子转移到分子,产生“斯托克斯拉曼散射”;要么从分子转移到光子产生“反斯托克斯拉曼散射”。由于“斯托克斯拉曼散射”比“反斯托克斯拉曼散射”更为强烈,因此“斯托克斯拉曼散射”被用于传统的拉曼光谱。拉曼光谱可以对食品中的致病菌进行快速、无损的实时原位检测,作为传统致病菌检测方法和分子生物学方法的补充。拉曼光谱结合适当的光谱预处理和化学计量法,能够提供完整细胞和组织内部的物质浓度、结构以及相互作用的信息,因此,拉曼光谱可以获取单个细菌细胞的成分信息,对污染食源性致病菌的食品基质进行检测,还可以对不同菌属以及同一菌属不同血清型的致病菌进行区分鉴定[10]。
众多研究表明,拉曼光谱可以在单细胞水平上用最少的样品来检测致病菌。ASSAF等[11]将拉曼光谱扫描引入到检测沙门氏菌的ISO(6 579:2 002)标准中的3种不同途径,检测时选择在指数生长期的沙门氏菌,以提高对于近缘菌种的鉴别区分能力。结合PCA结果显示,拉曼光谱鉴定沙门氏菌的相关系数(R2)为0.985,检测时间从100 h减少到50 h,表明ISO(6579:2002)结合拉曼光谱技术大大缩短了检测时间,提高了准确性。MEISEL等[12]建立了基于拉曼光谱的三级分类SVM模型,从人工污染的碎牛肉和鸡胸肉中分离出单增李斯特菌、沙门氏菌、金黄色葡萄球菌和小肠结肠耶尔森菌,对革兰氏水平、属水平和种水平的分类模型实现了90.6%~99.5%的准确性,实现了对低浓度致病菌污染的快速检测,避免了耗时的预浓缩步骤。
为了增强拉曼光谱的信号、减少干扰,科研人员开发了表面增强拉曼散射(surface-enhanced raman scattering,SERS)技术。SERS是一种特殊的拉曼光谱技术,通常使用硬币金属,特别是金或者银作为SERS的底物,通过小金属颗粒或粗糙金属表面与目标分子的相互作用使光谱信号强度比普通拉曼光谱提高了7个数量级[13-14]。SERS的应用提高了拉曼光谱在食源性致病菌检测中的分析能力。LIU等[15]使用种子介导法合成金纳米粒子作为SERS的增强底物,扫描鸡胸肉中分离出的4种假单胞菌,得到的SERS光谱结合PCA和HCA对4种假单胞菌进行鉴别,结果显示,它们的遗传关系与16S rRNA的结果完全一致,分类准确率高达100%,实现了快速、准确检测。LUO等[16]使用胶体银作为SERS的底物对2种单增李斯特菌、伊诺特氏李斯特菌、大肠杆菌O157:H7和鼠伤寒氏沙门氏菌进行拉曼光谱分析,结果表明,胶体银可有效增强拉曼光谱信号,并对4种食源性致病菌进行准确鉴定。此外,有人使用SERS结合免疫分析法准确检测到了低浓度病毒,SERS的使用大大缩短了检测时间。KOGLER等[17]以纳米金为SERS的底物,成功鉴别了大肠杆菌和单增李斯特菌。
1.2 红外光谱技术
红外光谱(infrared spectroscopy,IR)区域覆盖电磁波中12 500~33 cm-1的频率,可分为近红外(12 500~4 000 cm-1)、中红外(4 000~400 cm-1)和远红外(400~33 cm-1)区域,其中,近红外和中红外区域常用于化学分析,并用于致病菌检测。微生物的细胞膜和细胞壁具有独特的化学成分,如核酸、蛋白质、碳水化合物和脂肪酸等,可以提供独特的红外吸收光谱。虽然大多数微生物的化学成分略有不同,但仍具有相似的红外光谱。因此,IR技术也需要与光谱预处理和化学计量法相结合,才能用于致病菌的定量和不同菌种之间的分类[18]。
基于细胞成分的红外吸收技术,可在菌种、亚种、菌株、血清型和单倍体水平上对各种致病菌进行鉴别和分类。DAVIS等[19]研究表明,红外光谱结合HCA和典型变量分析可在单倍体、血清型和菌株水平上鉴别单核增生李斯特菌,其鉴别准确率分别为91.7%、96.6%和100%,所有的分类都可在18 h内完成。ERNEST等[20]使用可见近红外高光谱成像技术结合判别分析和支持向量机模型将大肠杆菌O157:H7、单增李斯特菌、金黄色葡萄球菌和鼠伤寒沙门氏菌分类鉴别,判别分析和支持向量机的预测准确率均较高,分别为90.7%和82.2%,结果证明该方法可以准确、快速识别和鉴定食源性致病菌。TITO等[21]使用近红外光谱结合PCA和偏最小二乘回归(partial least squares regression,PLSR)预测大西洋鲑鱼中的腐败菌落数量,相关系数(R2)为0.95,标准误差(root mean squared error,RMSE)为0.12 lg CFU/g,该方法建立的预测模型可较好地运用到食源性致病菌的近红外光谱检测中。然而,上述研究多使用昂贵、大体积的台式仪器进行,在实际应用中的适应性较差。DUAN等[22]利用便携式近红外光谱仪检测牙鲆鱼片中的菌落总数,通过将遗传算法(genetic algorithm,GA)和反向传播人工神经网络(back propagation artificial neural network,BP-ANN)算法与600~1 100 nm的红外光谱结合,建立了预测模型,结果具有良好的预测准确度(R2=0.985, RMSE=0.095)。应用便携式设备有利于方便应用,值得进一步研究并建立检测方法。
以振动光谱学为基础检测食源性致病菌的例子如表1所示。
表1 用于食源性致病菌检测的振动光谱学技术
Table 1 Vibration spectroscopy techniques for detection of foodborne pathogens
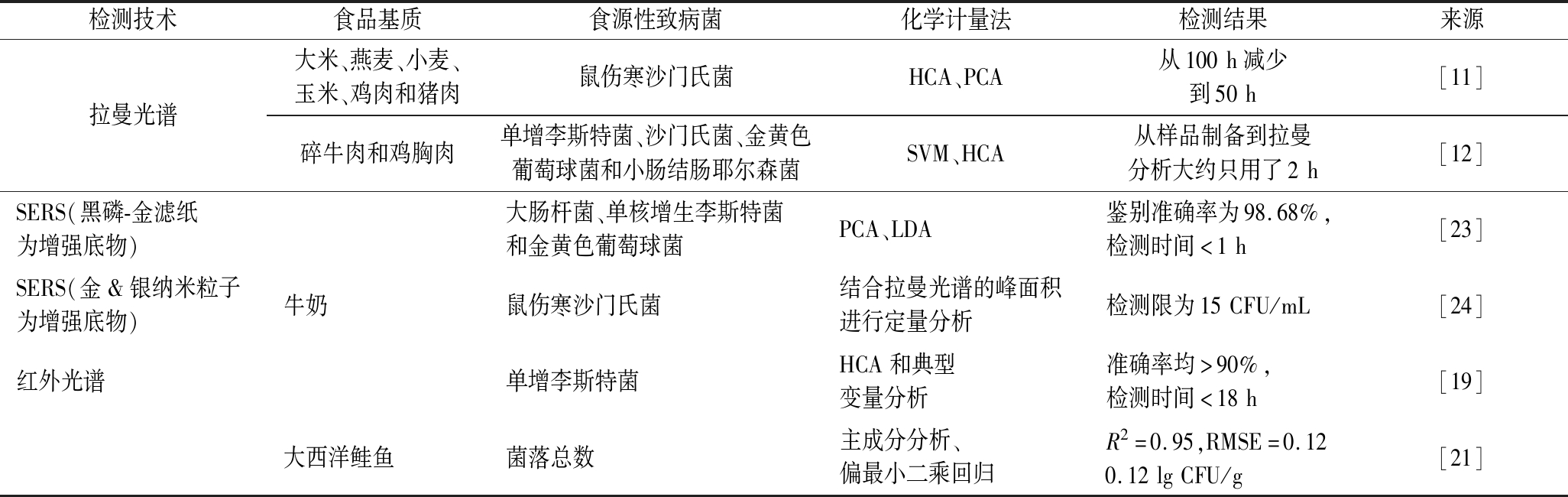
检测技术食品基质食源性致病菌化学计量法检测结果来源拉曼光谱大米、燕麦、小麦、玉米、鸡肉和猪肉鼠伤寒沙门氏菌HCA、PCA从100h减少到50h[11]碎牛肉和鸡胸肉单增李斯特菌、沙门氏菌、金黄色葡萄球菌和小肠结肠耶尔森菌SVM、HCA从样品制备到拉曼分析大约只用了2h[12]SERS(黑磷-金滤纸为增强底物)大肠杆菌、单核增生李斯特菌和金黄色葡萄球菌PCA、LDA鉴别准确率为98.68%,检测时间<1h[23]SERS(金&银纳米粒子为增强底物)牛奶鼠伤寒沙门氏菌结合拉曼光谱的峰面积进行定量分析检测限为15CFU/mL[24]红外光谱单增李斯特菌HCA和典型变量分析准确率均>90%,检测时间<18h[19]大西洋鲑鱼菌落总数主成分分析、偏最小二乘回归R2=0.95,RMSE=0.120.12lgCFU/g[21]
2 PCR
PCR是1986年发展起来的一种体外扩增DNA的方法[25],具有分析时间短、特异性高和自动化的优点,是食品中常见的致病菌检测方法之一[26]。许多使用PCR检测食源性致病菌基因和基因组区域的方法已被开发出来,并应用于食源性致病菌检测。由于食物成分通常会干扰酶反应,所以食物样本在PCR前需要预处理,通过离心或磁分离技术分离或浓缩病原体,然后从浓缩的细胞中提取DNA作为PCR的模板,以提高PCR检测的灵敏度和准确性。GORDILLO等[27]使用免疫磁分离技术筛选肉制品中的大肠杆菌O157:H7,富集后使用PCR检测大肠杆菌的基因型,检测限(limit of detection,LOD)为103 CFU/mL。TAYLOR等[28]使用双强度氧化钛氢氧化物对碎牛肉中的大肠杆菌O157:H7进行富集,之后提取DNA并进行PCR检测,得到LOD为103 CFU/mL,做到了对低浓度大肠杆菌O157:H7的检测,省去了耗时的富集等预处理步骤。
2.1 多重PCR
多重PCR(multiplex PCR,mPCR)是一种应用广泛的分子诊断技术,该技术在单次PCR实验中使用2组或2组以上引物对不同的目标基因序列进行扩增,每组引物都能够检测1个基因、1个基因变体或1个特定生物体的基因组标记[29]。自1988年实施以来,mPCR被广泛用于食源性致病菌的检测。WEI等[2]以rfbE、nuc和invA基因为靶点设计引物,鉴定大肠杆菌O157:H7、金黄色葡萄球菌和沙门氏菌,检测灵敏度为103 CFU/mL,与单重PCR的检测结果一致,可见mPCR是一种简便易行而又快速的检测方法。GORDILLO等[27]在免疫磁分离和选择性富集之后,使用mPCR检测大肠杆菌O157:H7基因型,结果显示,不同引物对相对应的基因型有极高的特异性,实现了高效快速检测。还有学者用mPCR在未富集的情况下,对牛奶和鸡肉中的金黄色葡萄球菌、阪崎克罗诺杆菌、大肠杆菌O157:H7、蜡样芽孢杆菌、副溶血性弧菌和沙门氏菌6种致病菌进行扩增检测,所有6种病原体均被检出,LOD为103~104 CFU/mL,在进行富集之后,将2种多重PCR结合,LOD则低至100 CFU/mL,且仅用了10 h就得到结果[30]。
尽管mPCR技术是一种检测食源性致病菌的可靠方法,但由于mPCR在一次反应中需要扩增多个基因序列,因此所有的引物要在相同的温度下退火才有效。另外,引物之间可能会相互作用,导致引物二聚体或者不需要的目标序列扩增,对实验结果产生干扰,因此,分析前要确定每个引物的合理浓度。
2.2 实时荧光定量PCR
实时荧光定量PCR(quantitative real-time PCR,qPCR)检测是向反应体系中加入荧光基团,通过连续监测PCR指数扩增期间荧光信号的强弱变化,实时监测特异性产物的量,最后通过标准曲线对目的基因进行定量分析的PCR技术[31]。
qPCR常用的检测方法之一是使用荧光染料,这是一种插入染料,可以与双链DNA结合,DNA的增加与荧光强度成正比,因此可以通过插入染料的荧光强度来量化扩增的DNA。在食源性致病菌的检测中通常使用SYBR Green作为qPCR的荧光染料,SYBR Green荧光染料是一种具有绿色激发波长的不对称菁类荧光素,最大吸收波长和发射波长分别约为497和520 nm。D’SOUZA等[32]使用SYBR Green结合qPCR对人工接种创伤弧菌的50多个海鲜样本进行检测,以创伤弧菌的gyrB基因为检测目标基因设计引物,LOD为102 CFU/mL,明显高于常规PCR,且整个检测过程仅需9~12 h。
qPCR的另一种检测方法是使用探针,包括Taqman荧光探针和分子信标。在对食源性致病菌的检测中,使用Taqman探针较为普遍。一项使用TaqMan qPCR检测大肠杆菌O157:H7的研究表明,该技术在牛肉中的LOD可达到100 CFU/mL[33]。
PCR检测食源性致病菌的例子及比较如表2所示。
表2 用于食源性致病菌检测的PCR技术
Table 2 PCR techniques for detection of foodborne pathogens

检测技术食品基质食源性致病菌检测限检测时间/h来源多重PCR肉沙门氏菌、志贺氏菌和金黄色葡萄球菌101CFU/g<30[34]猪肉沙门氏菌、单增李斯特菌和大肠杆菌O157:H7每25g接种的猪肉样本中有1个细胞30[35]大肠杆菌O157:H7、金黄色葡萄球菌、沙门氏菌每25g样品中可检测出10个细胞[2]实时定量PCR卷心菜、生肉糜单增李斯特菌和金黄色葡萄球菌7和2CFU/mL24[36]牛肉大肠杆菌O157:H7100CFU/mL<3[33]海鲜食品样本创伤弧菌102CFU/mL9~12[32]
3 生物传感器
生物传感器(biosensesor)是对生物物质敏感并能将其浓度转换为电信号进行检测的设备。生物传感器的优点是可以对食品样品中的微生物活性进行快速、经济、高效的分析。生物传感器由识别元件和转导元件组成,其中,识别元件是生物衍生分子,转导元件则是将对识别元件产生的物理化学反应转化为可测量的信号[37]。生物传感器通常是根据转导元件的类型进行分类,可分为光学生物传感器、电化学生物传感器、热学生物传感器、压电生物传感器和半导体生物传感器等。其中,光学生物传感器、电化学生物传感器在食源性致病菌检测中具有较高的特异性和灵敏度,并可对食源性致病菌进行原位快速检测,因此应用较广泛。
3.1 光学生物传感器
光学生物传感器是食源性致病菌检测中应用最广泛的生物传感器,目前已经开发出商品化产品。光学生物传感器基于多种成熟可靠的技术,包括光吸收、比色、化学发光、荧光、光偏转、表面等离子共振体(surface plasmon resonance,SPR)和总内部反射率。其中,SPR生物传感器具有突出的灵敏度和复用能力,这些独特的优势使得光学生物传感器在食源性致病菌的检测中发展迅速;比色生物传感器易于使用,对食源性致病菌的检测效率极高,而且便携、成本低,具有较好的应用前景。
SPR生物传感器具有无标签检测、实时监测生物分子相互作用、传感芯片可重复使用以及将光纤和波导结构用于光传输的小型化等优点。SPR现象是指当偏振光以特定的角度入射到2种不同折射率的介质(通常是玻璃或水和附着在玻璃表面的金属或金属氧化物)界面时,就产生了表面等离子体激元,从而使反射光的强度在一个特定的角度上减小,这个角度叫做共振角,通过测量反射率、角度或波长随时间的变化可以得到传感器信号。在所有的构象中,SPR现象可以使传感器表面的折射率发生直接的、无标签的、实时的变化,这些变化与生物分子的浓度成比例[38],因此,SPR生物传感器使得实时测量生物分子检测食源性致病菌成为可能,并且具有很高的灵敏度。KUSHWAHA等[39]使用以ZnO为基质的石墨烯SPR生物传感器对假单胞菌进行检测时发现,氧化锌大大提高了SPR信号,使得石墨烯上的分子识别位点与假单胞菌细胞紧密结合,该SPR生物传感器较传统的生物传感器具有更高的灵敏度,为187.43 deg/RIU。
比色生物传感器可以通过反应液颜色的变化,无需任何分析仪器实时地用肉眼观察到食物样品中的致病菌。SRISA-ART等[40]使用比色生物传感器检测牛奶样品中的鼠伤寒沙门氏菌,LOD为103 CFU/mL,表明可以做到快速定量检测。比色生物传感器检测法在食品致病菌中展现出的优势,将推动其在肉制品加工中的研究和应用。
3.2 电化学生物传感器
电化学生物传感器是基于电极和样品之间的界面发生化学反应而引起的电流或电位差的变化检测微生物的生物传感器,具有良好的灵敏度、简便性和快速性[41]。在电化学生物传感器中,广泛使用分子、抗体、适配体和核酸等作为传感层,传感层的选择对于检测的准确度和灵敏度起着关键作用[42]。用于设计电极的材料包括金、铂、石墨、导电聚合物和改性碳作为介体分子,电化学生物传感器可以通过使用介体分子增强检测的灵敏度,因为介体分子具有放大载点基础产生响应电流的能力[37]。VASQUEZ等[43]开发了一种电化学生物传感器,通过使用固定在丝网刷碳电极表面的生物素抗体对罗非鱼的无乳链球菌进行检测,可以在101~107 CFU/mL的较低浓度检测到无乳链球菌。CHEN等[44]使用碳纳米管(carbon nanotube,CNT)作为氧化还原探针,该纳米管与信号探针相连,用于放大电化学信号以检测结合分歧杆菌。
生物传感器检测食源性致病菌的例子及比较如表3所示。
表3 用于食源性致病菌检测的生物传感器技术
Table 3 Biosensesor techniques for detection of foodborne pathogens

检测技术食源性致病菌检测限/(CFU·mL-1)检测时间/h来源光学生物传感器牛奶中的鼠伤寒沙门氏菌10330[45]大肠杆菌、鼠伤寒沙门氏菌和金黄色葡萄球菌101[46]金黄色葡萄球菌、铜绿假单胞菌、粪肠球菌、大肠杆菌10[47]电化学生物传感器大肠杆菌O157:H7102[48]蜡样芽胞杆菌35~880.1[49]
4 结语
振动光谱学、PCR和生物传感器检测技术结合适当的化学计量等分析方法可以做到对食源性致病菌的快速检测,在食品产业中具有较好的应用前景,但各技术又具有自己的优势和问题。
振动光谱学技术结合化学计量法具有良好的特异性、准确性,并且可以做到原位、无损的快速检测。然而,食品组分复杂多变,食品基质自身的荧光会对光谱产生影响,这给使用光谱学技术检测食源性致病菌带来挑战。因此,需要大量的光谱数据并结合科学的光谱预处理技术、适当的化学计量学,才能建立可靠的预测模型。PCR和生物传感器检测技术,通常成本较高,同时生物传感器检测方法需要专业的研究人员,且设备复杂,但是由于检测时间短、特异性强,通常比传统的培养方法更具有时间效益。
食源性致病菌的快速检测是食品行业亟待解决的关键点,不仅需要持续开发新技术,还需要对现有快速检测技术进行对比分析以明确最有效的检测方法并推广应用,提高食品安全性。
[1] DANIELLE M T,ELLVN P M,PARTICIA M G,et al.Preliminary incidence and trends of infections with pathogens transmitted commonly through food—Foodborne diseases active surveillance network,10 US.sites,2015—2018[J].American Journal of Transplantation,2019,19(6):1 859-1 863.
[2] WEI Caijiao,ZHONG Junliang,HU Ting,et al.Simultaneous detection of Escherichia coli O157:H7,Staphylococcus aureus,and Salmonella,by multiplex PCR in milk[J].Biotech,2018,8(1):76.
[3] MINAROVICOVA J,WEGHOVA A,KACLIKOVA E.Evaluation of DNA extraction methods for culture-independent Real-Time PCR-Based detection of Listeria monocytogenes in Cheese[J].Food Analytical Methods,2020,13(3):667-677.
[4] 韩进兰,食源性疾病监测中病原微生物检验结果分析[J].临床检验杂志电子版,2020,9(1):122.
[5] JONES T F,YACKLEY J,Foodborne disease outbreaks in the United States:A historical overview[J].Foodborne Pathogens & Disease,2018,15(1):11-15.
[6] ZHAO Xihong,LI Mei,LIU Yao.Microfluidic-based approaches for foodborne pathogen detection[J].Microorganisms,2019,7(10):381.
[7] FARKAS K,MANNION F,HILLARY S,et al.Emerging technologies for the rapid detection of enteric viruses in the aquatic environment[J].Current Opinion in Environmental Science & Health,2020,16:1-6.
[8] KODOGIANNIS,VASSIILIS S.Application of an electronic nose coupled with fuzzy-wavelet network for the detection of meat spoilage[J].Food and Bioprocess Technology,2017,10(4):730-749.
[9] MUNGROO N A,OLIVEIRA G,NEETHIRAJAN S.SERS baced point-of-care detection of food-borne pathogens[J].Microchimica Acta,2015,183(2):697-707.
[10] LORENZ B,WICHMANN C,STOCKEL S,et al.Cultivation-free Raman spectroscopic investigations of bacteria[J].Trends in Microbiology,2017,25(5):413-424.
[11] ASSAF A,COEDELLA C B Y,THOUAND G.Raman spectroscopy applied to the horizontal methods ISO 6579:2002 to identify Salmonella spp. in the food industry[J].Analytical and Bioanalytical Chemistry,2014,406(20):4 899-4 910.
[12] MEISEL S,STOCKEL S,ROESCH P,et al.Identification of meat-associated pathogens via Raman microspectroscopy[J].Food Microbiology,2014,38:36-43.
[13] CIALLA D,MORZ A,RENE B,et al.Surface-enhanced Raman spectroscopy(SERS):progress and trends[J].Analytical and Bioanalytical Chemistry,2012,403(1):27-54.
[14] PAHLOW S,MARZ A,SEISE B,et al.Bioanalytical application of surface and tip enhanced Raman spectroscopy[J].Engineering in Life Science,2012,12(2):131-143.
[15] LIU Shuangshuang,LI Huanhuan,HASSAN M M,et al.Amplification of Raman spectra by gold nanorods combines with chemometrics for rapid classification of four Pseudomonas[J].International Journal of Food Microbiology,2019,304:58-67.
[16] LUO B S,LIN M I N.A portable Raman system for the identification of foodborne pathogenic bacteria[J].Journal of Rapid Method & Automation in Microbiology,2008,16(3):238-255.
[17] KOGLER M,RYABCHIKOV Y U,UUSITALO S,et al.Bare laser-synthesized Au-based nanoparticles as nondisturbing surface-enhanced Raman scattering probes for bacteria identification[J].Journal of Biophotonics,2018,11(7).DOI:10.1002/jbi0.201700225.
[18] SHAPAVAL V,WALCZAK B,GOGNIES S,et al.FTIR spectroscopic characterization of differently cultivated food related yeasts[J].The Analyst,2013,138(14):4 129-4 138.
[19] DAVIS R,MAUER L J.Subtyping of Listeria monocytogenes at the halptype level by Fourier transform infrared(FT-IR) spectroscopy and multivariate statistical analysis[J].International Journal of Food Microbiology,2011,150(2-3):140-149.
[20] ERNEST B,HUANG Xingyi,YI Ren,et al.Vis-NIR hyperspectral imaging for the classification of bacterial foodborne pathogens based on pixel-wise analysis and a novel CARS-PSO-SVM model[J].Infrared Physics and Technology,2020,105.DOI:10.1016/j.infrared.2020.103220.
[21] TITO N B,RODEMANN T,POWELL S M.Use of near infrared spectroscopy to predict microbial numbers on Atlantic salmon[J].Food Microbiology,2012,32(2):431-436.
[22] DUAN Cui,CHEN Chunguang,KHAN M N,et al.Non-destructive determination of the total bacteria in flounder filet by portable near infrared spectrometer[J].Food Control,2014,48:18-22.
[23] HUANG Deqiu,ZHUANG Zhengfei,WANG Zhen,et al.Black phosphorus-Au filter paper-based three-dimensional SERS substrate for rapid detection of foodborne bacteria[J].Applied Surface Science,2019,497.DOI:10.1016/j.apsusc.2019.143825.
[24] DUAN Nuo,CHANG Boya,ZHANG Hui,et al.Salmonella typhimurium detection using a surface-enhanced Raman scattering-based aptasensor[J]International Journal of Food Microbiology, 2016,218:38-43.
[25] MULLIS K B,FALOONA F A,SCHARF S J,et al.Specific enzymatic amplification of DNA in vitro:the polymerase chain reaction[J].Food Control,2014,48:18-22.
[26] FORGHANI F,WEI Shuai,OH D H.A rapid multiplex real-time PCR high-resolution melt curve assay for the simultaneous detection of Bacillus cereus,Listeriia monocytogenes and Staphylococcus aureus in food[J].Journal of Food Protection,2016,79(5):810-815.
[27] GORDILLO R,JUAN J C,MARIA J A,et al.Development of PCR assays for detection of Escherichia coli O157:H7 in meat products[J].Meat Scinece,2011,88(4):763-773.
[28] TAYLOR T M,ELHANAFI D,DRAKE M,et al.Effect of food matrix and cell growth on PCR-based detection of Escherichia coli O157:H7 in ground beef[J].Journal of Food Protection,2005,68(2):225-232.
[29] WANG Yun,SALAZAR J K.Culture-independent rapid detection methods for bacterial pathogens and toxins in food matrices[J].Comprehensive Reviews in Food Science and Food Safety,2016,15(1):183-205.
[30] CHIANG Yucheng, TSEN Hauyang,CHEN Hsinyen,et al.Multiplex PCR and a chromogenic DNA macroarray for the detection of Listeria monocytogens,Staphylococcus aureus,Streptococcus agalactiae,Enterobacter sakazakii,Escherichia coli O157:H7,Vibrio parahaemolyticus,Salmonella spp.and Pseudomonas fluoresc[J].Journal of Microbiol Methods,2012,88(1):110-116.
[31] 曲勤凤.重要食品掺假检测技术研究鱼糜制品中主料含量的测定 (荧光 PCR 法)[D].上海:复旦大学, 2011
[32] D’SOUZA C,KUMAR B K,RAI P,et al.Application of gyrB targeted SYBR green based qPCR assay for the specific and rapid detection of Vibrio vulnificus in seafood[J].Journal of Microbiological Methods, 2019.DOI:10.1016/j.mimet.2019.105747.
[33] HSU C F,TSAI T Y,PAN T M.Use of the duplex TaqMan PCR system for detection of Shiga-like toxin-producing Escherichia coli O157[J].Journal of Clinical Microbiology, 2005,43(6):2 668-2 673.
[34] WANG Lijun,YE Chenlian,XU Hengyi,et al.Development of an SD-PMA-mPCR assay with internal amplification control for rapid and sensitive detection of viable Salmonella spp.Shigella spp.and Staphylococcus aureus in food products[J].Food Control,2015,57:314-320.
[35] KIM H J,LEE H J,LEE K H,et al.Simultaneous detection of pathogenic Vibrio species using multiplex realtime PCR[J].Food Control,2012,23:491-498.
[36] MARTINON A,WILKINSON M G.Selection of optimal primer sets for use in a duplex sybr green-based,real-time polymerase chain reaction protocol for the detection of Listeria monocytogenes and Staphyloccocus aureus in foods[J].Food Saf,2011,31:297-312.
[37] JAYAN H,PU Hongbin,SUN Dawen.Recent development in rapid detection techniques for microorganism activities in food matrices using bio-recognition:A review[J].Trends in Food Science & Technology, 2020:233-246.DOI:10.1016/j.tifs.2019.10.007.
[38] KHANSILI N,RATTU G,KRISHNA P M.Label-free optical biosensors for food and biological sensor applications[J].Sensors and Actuators B:Chemical,2018,265:35-49.
[39] KUSHWAHA A S,ANIL K,RAJEEV K,et al.Zinc oxide,gold and graphene-based surface plasmon resonance (SPR) biosensor for detection of Pseudomonas like bacteria:A comparative study[J].Optik,2018:697-707.DOI:10.1016/j.ijleo.2018.07.066.
[40] SRISA-ART M,BOEHLE K E,GEISS B J,et al.Highly sensitive detection of Salmonella typhimurium using a colorimetric paper-based analytical device coupled with immunomagnetic separation[J].Analytical Chemistry,2018,90(1):1 035-1 043.
[41] SHARMA H,MUTHARASAN R.Review of biosensors for foodborne pathogens and toxins[J].Sensors and Actuators B:Chemical,2013,183:535-549.
[42] MAJDINASAB M,HAYAT A,MARTY J L.Aptamer-based assays and aptasensors for detection of pathogenic bacteria in food samples[J].TrAC Trends in Analytical Chemistry,2018,107:60-77.
[43] VASQUEZ G,REY A,RIVERA C,et al.Amperometric biosensor based on a single antibody of dual function for rapid detection of Streptococcus agalactiae[J].Biosensors and Bioelectronics,2017,87:453-458.
[44] CHEN Yuhan,GUO Shuliang,ZHAO Min,et al.Amperometric DNA biosensor for Mycobacterium tuberculosis detection using flower-like carbon nanotubes-polyaniline nanohybrid and enzyme-assisted signal amplification strategy[J].Biosensors and Bioelectronics,2018,119:215-220.
[45] LI Yong,MUSTAPHA A.Simultaneous detection of Escherichia coli O157:H7,Salmonella,and Shigella in apple cider and produce by a multiplex PCR[J].Food Prot,2004,67:27-33.
[46] KEARNS H,GOODACRE R,JAMIESON L,et al.SERS detection of multiple anti-microbial resistant pathogens using nanosensors[J].Analytical Chemistry,2017,89(23):12 666-12 673.
[47] CAREY J R,SUSLICK K S,HULKOWER K I,et al.Rapid identification of bacteria with a disposable colorimetric sensing array[J].Journal of the American Chemical Society,2011,133(19):7 571-7 576.
[48] LIN Yuehhui,CHEN Szhau,CHUANG Yaochen,et al.Disposable amperometric immunosensing strips fabricated by Au nanoparticles-modified screenprinted carbon electrodes for the detection of foodborne pathogen Escherichia coli O157:H7[J].Biosens Bioelectron,2008,23:1 832-1 837.
[49] PAL S,YING W,ALOCILIA E C,et al.Sensitivity and specificity performance of a direct-charge transfer biosensor for detecting Bacillus cereus in selected food matrices[J].Biosystems Engineering,2008,99:46.