脂肪氧合酶(lipoxygenase,LOX,EC. 1.13.11.12)属于脂质过氧化酶的异构家族,主要催化具有一个或多个cis,cis-1,4-戊二烯结构的多不饱和脂肪酸的特异性双加氧反应[1],生成氢过氧化物,进一步转化为一系列具有生物活性的氧化脂类[2]。在食品加工领域,LOX可以介导小麦面筋中二硫键的形成,起到强筋、改善面团流变性的作用[3]。LOX也可与过氧化氢酶、异构酶和脱氢酶协同作用,增强水果和蔬菜的自然香气[4]。基于LOX开发的漂白剂和洗涤剂具有天然、温和、多效等特点[5]。此外,在制药行业中,LOX则可用于氯丙嗪的去除[6]。
现阶段,商业化的LOX主要来源于植物提取,但由于原料批次间成分不稳定及提取方法低效等问题,LOX导致成品质量不稳定,成本较高。随着生物技术的发展,微生物发酵生产LOX因其生产成本低,绿色高效而得到广泛的关注。常用的模式菌株Escherichia coli具有遗传背景清晰、生长周期短、培养成本低、产量高等优点,常被用作重组蛋白的表达宿主[7]。细菌来源的LOX在E.coli表达系统中具有高效表达的潜力。其中,Pseudomonas aeruginosa来源的LOX被研究的较多[8],Burkholderia thailandensis来源的LOX因其能有效转化亚油酸为羟基不饱和脂肪酸等,用于化妆品防腐剂也被关注[5, 9]。目前,重组LOX存在表达量低、稳定性差等问题,限制了其工业化生产与应用。随着现代酶工程技术的发展,通过对不同来源LOX酶学性质的分析比较,利用分子改造与表达优化等策略将有利于实现LOX的高效异源表达。本研究通过构建重组E.coli分别表达P. aeruginosa与B.thailandensis来源的LOX并对其酶学性质比较分析,选择酶学性能优良的PaLOX进一步表达优化,提高其可溶性表达水平,为构建高效的LOX表达系统奠定了基础。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
本研究所用质粒与菌株见表1。
1.1.2 试剂
DNA聚合酶、DNA连接酶、E. coli感受态,TaKaRa公司;ClonExpress®Ⅱ One step Cloning Kit试剂盒,南京诺唯赞公司;质粒提取试剂盒、异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)等,生工生物工程(上海)股份有限公司;亚油酸,麦克林(上海)公司。所有化学试剂均为国产分析纯。
表1 研究所用质粒和菌株
Table 1 Plasmids and strains used in this work
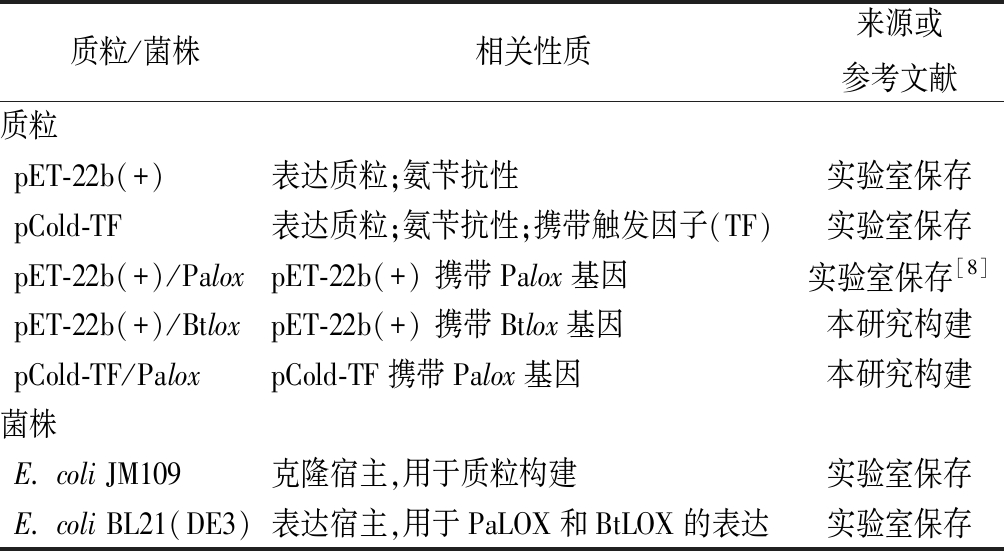
质粒/菌株相关性质来源或参考文献质粒pET-22b(+)表达质粒;氨苄抗性实验室保存pCold-TF表达质粒;氨苄抗性;携带触发因子(TF)实验室保存pET-22b(+)/PaloxpET-22b(+) 携带Palox基因实验室保存[8]pET-22b(+)/BtloxpET-22b(+) 携带Btlox基因本研究构建pCold-TF/PaloxpCold-TF携带Palox基因本研究构建菌株E. coli JM109克隆宿主,用于质粒构建实验室保存E. coli BL21(DE3)表达宿主,用于PaLOX和BtLOX的表达实验室保存
1.1.3 培养基和缓冲液
LB(Luria-Bertani)培养基(g/L):酵母粉5、胰蛋白胨10、NaCl 10、自然pH。
TB(Terrific-broth)培养基(g/L):酵母粉24、胰蛋白胨12、甘油5、KH2PO4 2.31、K2HPO4·3H2O 16.43、pH 7.0。
固体培养基在此基础上添加质量分数为1.5%~2%的琼脂粉。
缓冲液A:20 mmol/L KH2PO4-K2HPO4,pH 7.5。
缓冲液B:20 mmol/L KH2PO4-K2HPO4,500 mmol/L咪唑,pH 7.5。
1.2 实验方法
1.2.1 质粒构建
基于E.coli密码子偏好性优化合成Btlox基因(序列号:YP_442874.1)。为便于纯化,设计引物F1、R1(见表2)在N端引入组氨酸标签,PCR扩增得到Btlox片段。质粒pET-22b(+)经BamH I和Hind III双酶切后得到载体pET-22b(+)的线性化片段。经重组、转化即得到表达载体pET-22b(+)/Btlox。
表2 质粒构建过程中所用引物
Table 2 Primers used in the construction of plasmids

引物名称引物序列F1GTGCGGCCGCAAGCTTTCAGATGTTAGTAGAAGCAGGGATACGF2CGAAGGTAGGCATATGATGCATCATCATCATCATCATGCAGAAGCR1GAGATATACATATGATGCATCATCATCATCATCATGTTAACCATAAAACTGR2GCAGGTCGACAAGCTTTCAGATATTGGTGCTCGCCGGGATAC
质粒pCold-TF经BamH I和Hind III双酶切后得到载体pCold-TF的线性化片段。利用引物F2、R2(见表2)扩增得到目的基因Palox片段,经重组、转化得到表达载体pCold-TF/Palox。引物、基因合成与DNA测序均由生工生物工程(上海)股份有限公司完成。
1.2.2 重组LOX的表达
将各株重组甘油菌划线于LB固体培养基中,37 ℃条件下培养12 h,单菌落接种于装有10% LB培养基的250 mL 摇瓶中,37 ℃,220 r/min条件下培养8~10 h。按4%的接种量接入装有10% TB培养基的250 mL摇瓶中,于37 ℃,220 r/min条件下培养至菌体密度(OD600)达到0.5~0.6,加入终浓度为1 mmol/L的IPTG,20 ℃诱导培养24 h。以上培养过程均需添加100 μg/mL氨苄青霉素以维持质粒稳定及防止杂菌污染。
1.2.3 重组LOX的纯化
利用镍柱亲和层析的方法对LOX蛋白进行分离纯化。具体过程如下:不同重组菌的发酵液及细胞提取物均在4 ℃,14 000 r/min下离心15 min取上清液,并用0.22 μm的混合纤维素膜过滤除杂。将样品上样于已用缓冲液A预平衡的镍亲和层析柱中,待再次平衡后,用8%缓冲液B洗脱杂蛋白。用20%缓冲液B洗脱目标LOX,随后于预冷的缓冲液A中在4 ℃环境下透析获得纯蛋白。
1.2.4 菌体浓度OD600值的测定
发酵液用缓冲液A稀释相应倍数后,检测600 nm下的吸光值,以OD600值表示。OD600值与菌体干重之间的换算关系为当OD600值为1时,菌体质量浓度为0.578 g/L,可以计算出相应OD600值所对应的菌体干重。
1.2.5 LOX酶活力的测定
LOX的酶活力检测方法根据STEFAN[10]的方法进行了改进。酶活力测定条件:亚油酸在缓冲液A中稀释至终浓度0.32 mmol/L,将200 μL亚油酸底物缓冲液与20 μL稀释的LOX酶液混匀,25 ℃孵育4 min。每隔10 s向混合物中添加10 μL 10 mg/mL可溶性淀粉和40 μL饱和碘化钾溶液。最后,添加500 μL 体积分数15.0%的乙酸溶液显色10 min,测定470 nm下的吸光值。在所有的实验中,以失活LOX作为空白对照。单位LOX酶活力定义:1 min内470 nm下的吸光值增加0.001即为1个酶活力单位(U)。
1.2.6 SDS-PAGE电泳及蛋白浓度检测
10%的NuPAGE® SDS-PAGE蛋白胶用于分析蛋白的表达水平,以2-(N-吗啡啉)乙磺酸缓冲液为电泳缓冲液,上样量为15 μL,电泳电压为120 V。采用Brandford方法[11]测定蛋白浓度。
1.2.7 LOX最适反应温度和热稳定性的测定
最适反应温度:将溶有底物的缓冲液A分别在20~60 ℃的条件下孵育10 min,在不同温度下测定LOX酶活力,以酶活力最高者为100%。
热稳定性:重组LOX的热稳定性用30 ℃和50 ℃下酶活力的半衰期(t1/2, min)来表示。将纯酶用缓冲液A稀释到100 μg/mL,在不同温度下保温,间隔5 min取样测定残余酶活力,将残余酶活力按照文献所述方式拟合并计算半衰期(t1/2, min)[12]。
1.2.8 LOX最适反应pH和pH稳定性的测定
最适反应pH:分别用浓度为20 mmol/L的CH3COOH-CH3COONa (pH 3~6)、K2HPO4-KH2PO4 (pH 6~8)、C2H5 NO2-NaOH (pH 8~11)的缓冲液配制底物,测定纯酶在不同pH下的酶活力,将最高酶活力值定义为100%,采用非线性回归拟合曲线。
pH稳定性:将纯酶液用上述不同pH缓冲液稀释至100 μg/mL,在25 ℃下放置1 h,测定残余酶活力,以未保温酶液的酶活力为100%,采用非线性回归拟合曲线。
1.2.9 金属离子对酶活力的影响
将纯酶在pH 7.5,20 mmol/L Tris-HCl缓冲液中透析以置换缓冲液A。在底物中分别添加终浓度为0.1 mol/L和1 mol/L的一价(Li+、Na+)、二价金属离子(Ca2+、Cu2+、Zn2+、Mg2+、Mn2+、Fe2+),测定添加金属离子后纯化酶的酶活力,以无任何添加物下的酶活力为100%。
1.2.10 底物促溶剂对酶活力的影响
在缓冲液A中分别添加体积分数为2%和6%的有机溶剂(甲醇、乙醇、丙酮、异丙醇、二甲基亚砜)促进底物在缓冲液内分散,测定添加底物促溶剂后纯酶的酶活力,以无任何添加条件下的酶活力为100%。
1.2.11 LOX的动力学常数测定
用缓冲液A配制不同浓度(0.02~0.2 mmol/L)的亚油酸底物溶液,在25 ℃下分别测定LOX催化不同浓度亚油酸生成氢过氧化物的速率,采用非线性回归拟合计算Km和Kcat。
2 结果与分析
2.1 LOX基因进化树构建与性质分析
通过MEGA 6.06软件对12种来源的LOX基因序列进行分析,用Neighbor-Joining法构建系统发育树,通过Kimura 2-Parameter Distance模型计算进化距离。如图1所示,来源于P. aeruginosa PA01和B. thailandensis E264的LOX进化距离接近。结合前期研究报道(见表3),来源于P. aeruginosa、B. thailandensis的LOX的底物亲和力相对其他原核细菌来源的高,具有较大的开发潜力。因此,我们选取来源于P. aeruginosa和B. thailandensis的LOX基因进行异源表达,并比较分析其酶学性质。
2.2 LOX在E. coli BL21(DE3)中的表达
为研究LOX的表达情况,分别在E.coli BL21(DE3)中构建表达PaLOX、BtLOX的重组质粒pET-22b(+)/Palox、pET-22b(+)/Btlox(图2-a),并在20 ℃条件下诱导表达。

图1 LOX的系统发育树
Fig.1 Phylogenetic tree on the basis of LOXs
表3 LOX酶学性质分析
Table 3 Comparison of LOXs properties from different sources
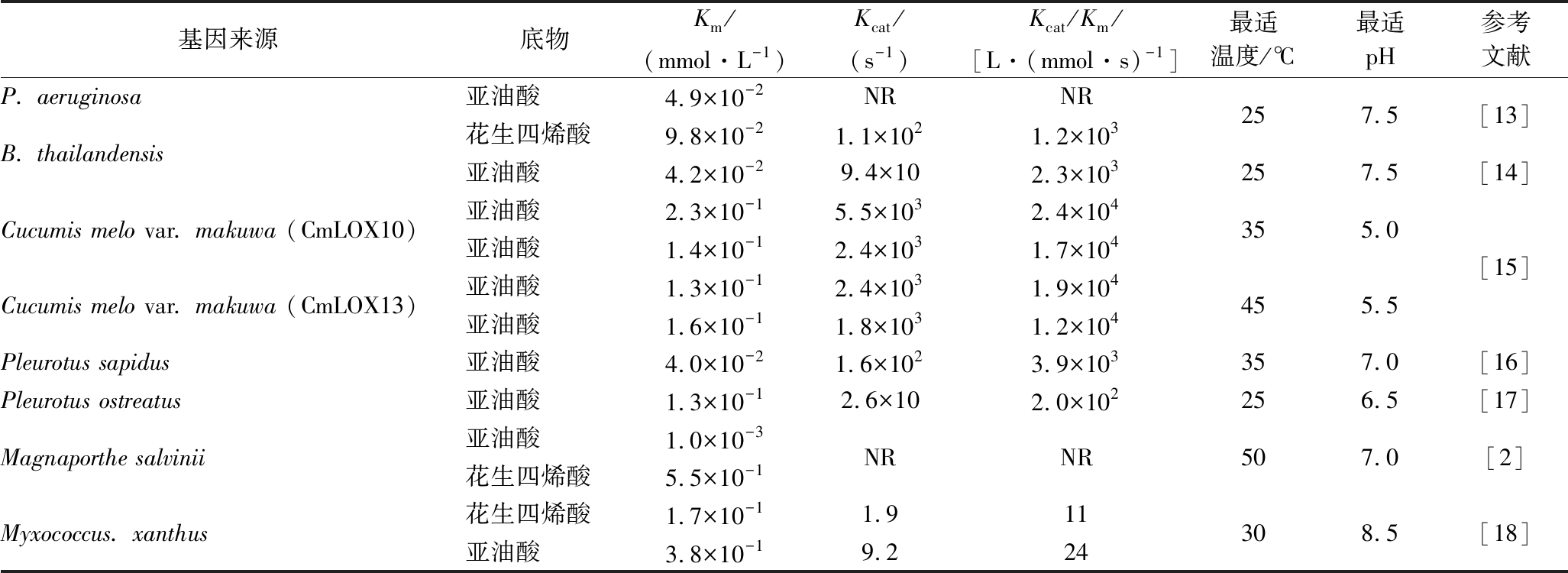
基因来源底物Km/(mmol·L-1)Kcat/(s-1)Kcat/Km/[L·(mmol·s) -1]最适温度/℃最适pH 参考文献P. aeruginosa亚油酸4.9×10-2NRNR257.5[13]B. thailandensis花生四烯酸9.8×10-21.1×1021.2×103亚油酸4.2×10-29.4×102.3×103257.5[14]Cucumis melo var. makuwa (CmLOX10)亚油酸2.3×10-15.5×1032.4×104355.0亚油酸1.4×10-12.4×1031.7×104[15]Cucumis melo var. makuwa (CmLOX13)亚油酸1.3×10-12.4×1031.9×104455.5亚油酸1.6×10-11.8×1031.2×104Pleurotus sapidus亚油酸4.0×10-21.6×1023.9×103357.0[16]Pleurotus ostreatus亚油酸1.3×10-12.6×102.0×102256.5[17]Magnaporthe salvinii亚油酸1.0×10-3NRNR507.0[2]花生四烯酸5.5×10-1Myxococcus. xanthus花生四烯酸1.7×10-11.911308.5[18]亚油酸3.8×10-19.224
注:NR文献未报道
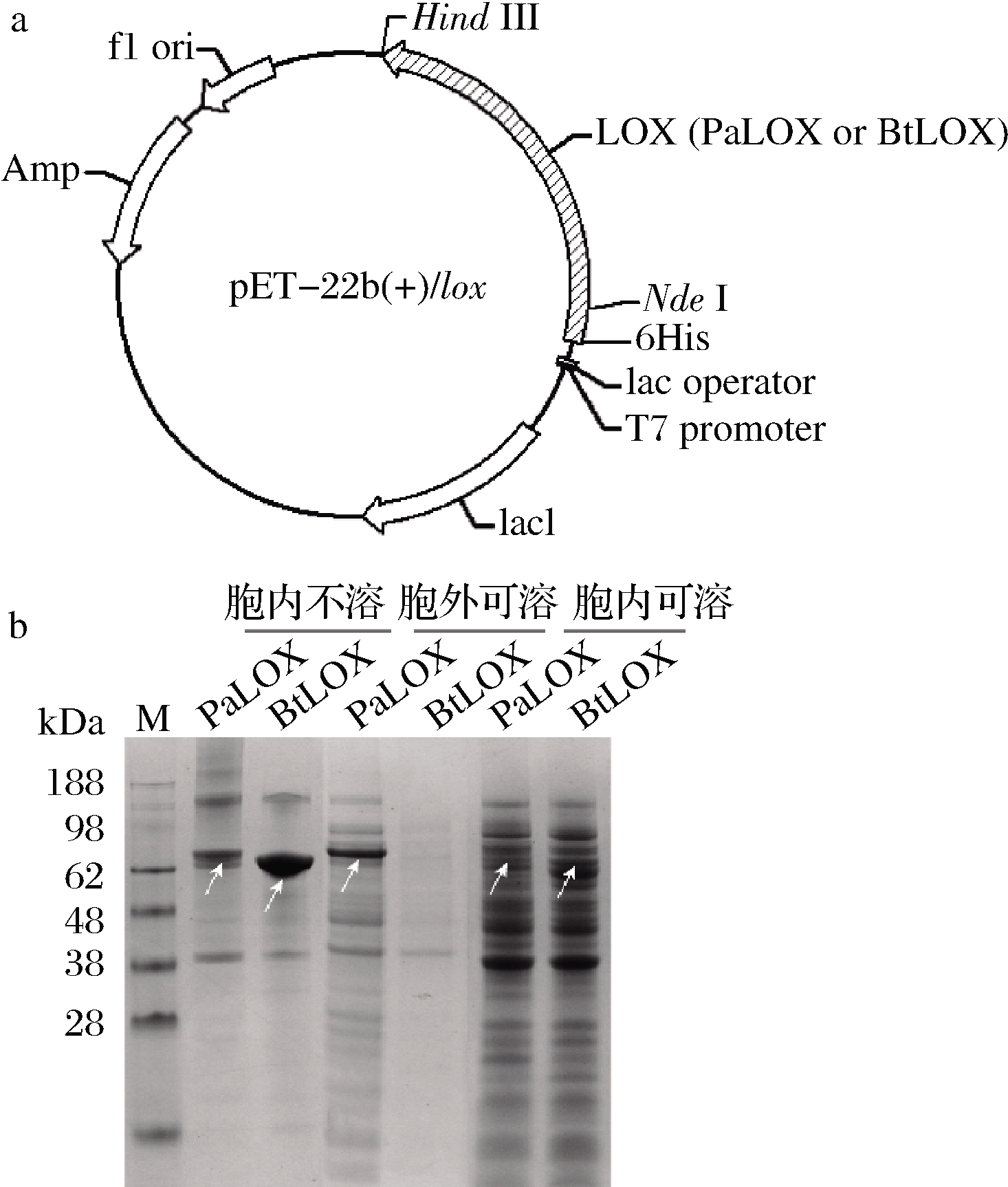
a-LOX表达式质粒;b-SDS-PAGE电泳分析图 M-蛋白质标准分子量;箭头表示PaLOX和BtLOX蛋白条带
图2 构建的LOX表达质粒和Palox、Btlox 表达蛋白SDS-PAGE电泳分析图
Fig.2 Construction of LOX expression plasmid and SDS-PAGE analysis of the expressed Palox and Btlox
通过蛋白表达分析发现,PaLOX、BtLOX在胞内可溶和不溶部分均有明显条带,其大小约为70 kDa。而BtLOX胞外未见重组蛋白条带(图2-b)。LOX活性结果显示,PaLOX发酵上清液LOX酶活力为351 U/DCW(Units per Dry Cell Weight),胞内为726 U/DCW;BtLOX胞内酶活力为597 U/DCW。上述结果表明,Palox和Btlox基因在E. coli中均实现了可溶表达,与以往研究报道[13]不一致的是,BtLOX表达过程中产生了大量的包涵体,可能是由于诱导温度高于16 ℃所致。
2.3 最适反应温度和热稳定性
为评估LOX的应用潜力,对LOX的最适反应温度和热稳定性进行了比较分析。结果表明,重组PaLOX和BtLOX的最适反应温度分别为25和35 ℃(图3-a)。随着反应温度的不断升高,当反应温度超过50 ℃时,2种来源的LOX活力均显著降低,但BtLOX活性下降较PaLOX明显。进一步研究LOXs的热稳定性发现,在30 ℃的条件下孵育,PaLOX和BtLOX的酶活力下降趋势缓慢,t1/2分别为59 min和157 min。在50 ℃的条件下孵育,随着时间的延长,二者活力下降趋势均较明显,PaLOX和BtLOX的t1/2分别为7 min和5 min(图3-b)。上述结果表明,30 ℃条件下重组BtLOX的稳定性较PaLOX好,而在50 ℃条件下则较PaLOX差。
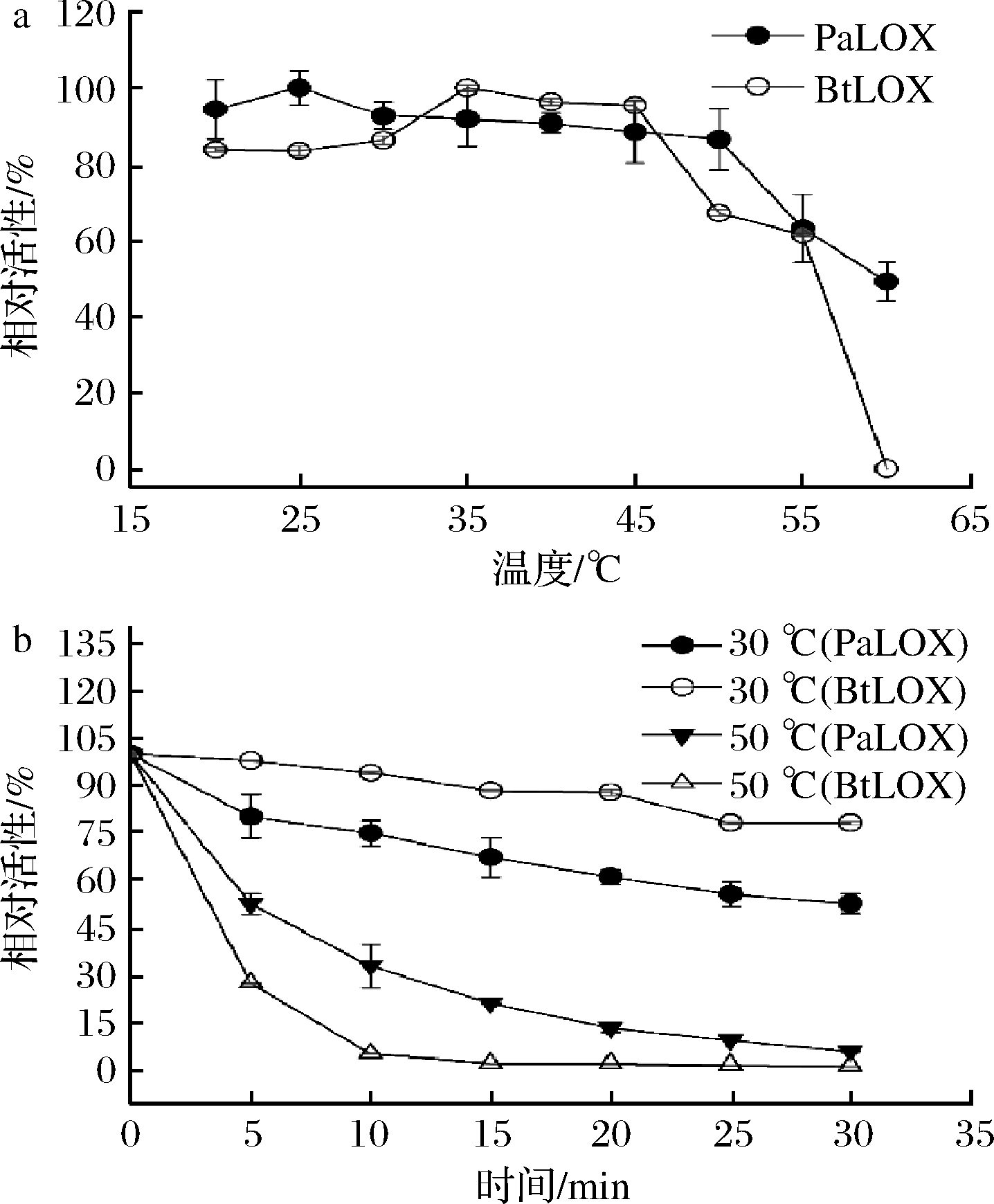
a-最适反应温度;b-不同温度下的热稳定性
图3 温度对LOX催化活性及稳定性的影响
Fig.3 Effects of temperature on the catalytic efficiency and stability of LOX
2.4 最适反应pH和pH稳定性
为了评估pH对LOX的影响,测定了pH在3.0~11.0之间LOX的酶活力变化。结果显示,重组PaLOX和BtLOX的最适反应pH分别为7.0和7.5,pH值>8.0或<6.5均会使LOX的催化活性快速下降;当pH<4.5或>10.0时,LOX的催化活性基本完全丧失(图4-a)。对LOX的pH稳定性进行分析,结果表明重组PaLOX在pH 6.0~10.0之间均较BtLOX稳定,而在pH<5.0时,BtLOX的稳定性有所提高(图4-b)。
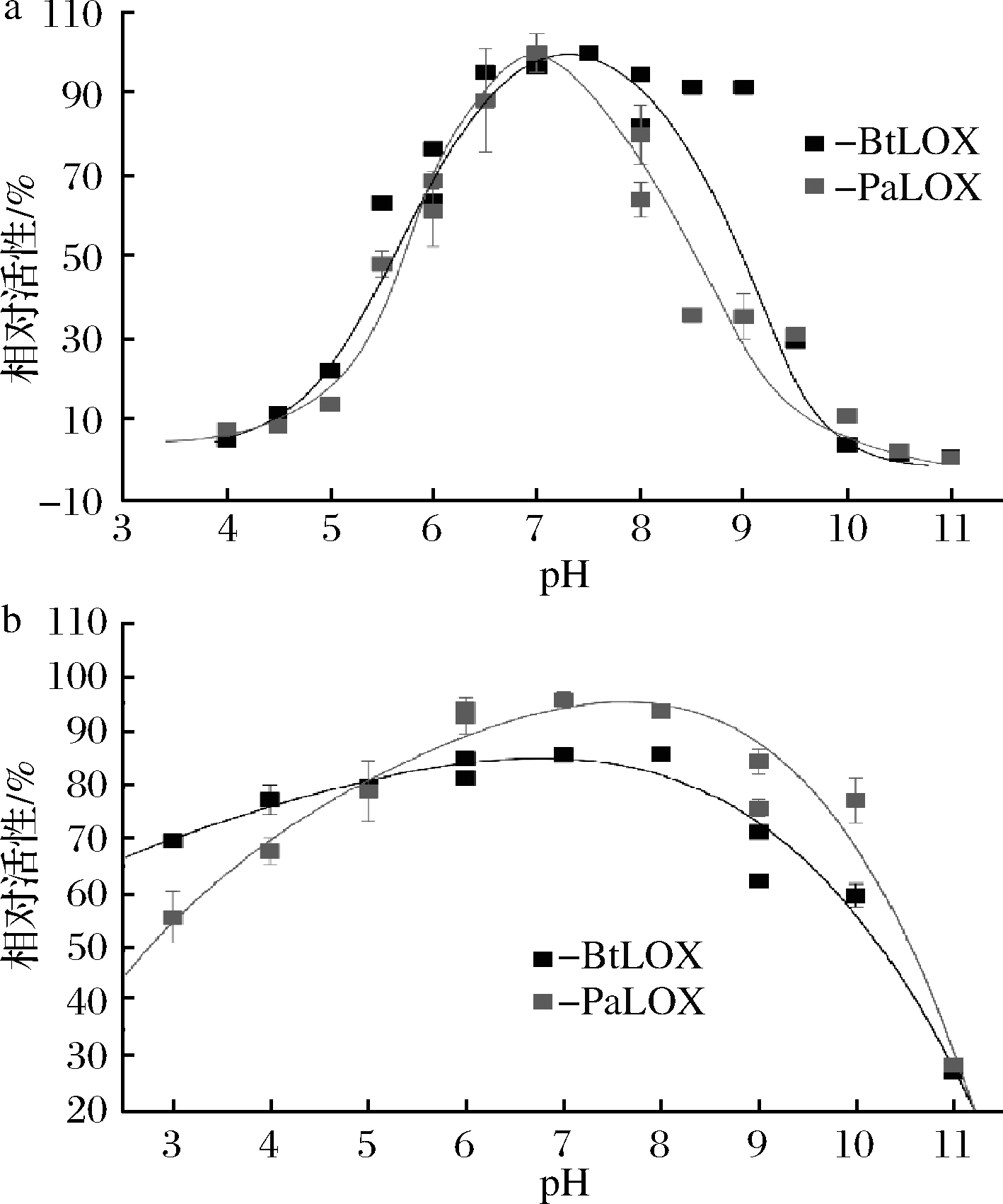
a-不同pH条件下LOX的催化活性; b-不同pH条件下LOX的稳定性
图4 pH对LOX酶活力及稳定性的影响
Fig.4 Effects of pH on the catalytic efficiency and stability of LOX
2.5 金属离子对LOX酶活力的影响
对酶有激活作用的激活剂,能在酶与底物之间形成一种酶-金属离子-底物的三元复合物,有利于底物与酶的活性中心结合;而对酶活力有抑制作用的,则可作为酶的抑制剂。因此,实验研究了金属离子对2种LOX酶活力的影响(图5)。其中Mg2+和Na+对PaLOX和BtLOX有激活作用,Cu2+、Zn2+对二者有明显的抑制作用。值得注意的是,低浓度的Fe2+对BtLOX有激活作用,而对PaLOX则有抑制作用。

a-金属离子对BtLOX活性的影响; b-金属离子对PaLOX活性的影响
图5 金属离子对LOX催化活性的影响
Fig.5 Effect of metal ions on catalytic activity of LOX
2.6 底物促溶剂对LOX酶活力的影响
酶和底物之间的结合能力是酶催化反应的主要推动力,其中底物的溶解度是直接影响酶和底物结合的因素。通过比较不同底物促溶剂对LOX酶活力的影响,发现体积分数为2%的甲醇、乙醇、乙二醇、异丙醇和丙酮对PaLOX的酶活力影响不大。在体积分数为6%下,PaLOX酶活力受到了抑制,可能是高浓度的有机溶剂使部分酶失活所致。体积分数为2%和6%的甲醇、乙醇、乙二醇对BtLOX酶活力均有不同程度的提高,特别是甲醇和乙二醇能显著提高40%的酶活力,可能是由于甲醇和乙二醇改善了底物亚油酸的溶解度,进而提高了BtLOX的酶活力(图6)。AN等[14]曾报道体积分数为6%的甲醇可以作为底物促溶剂有效改善LOX的催化效率。

a-促溶剂对PaLOX酶活力的影响;b-促溶剂对BtLOX酶活力的影响 1-甲醇;2-乙醇;3-乙二醇;4-丙酮;5-异丙酮;6-二甲基亚砜
图6 促溶剂对LOX催化活性的影响
Fig.6 Effect of dissolvant on catalytic activity of LOX
2.7 LOX酶动力学分析
酶的动力学参数是评价酶与底物的亲和力及催化活性的重要指标。据报道,野生型PaLOX以亚油酸为底物的Km值为660 μmol/L[19],而进行重组表达后,Km值则为48.9 μmol/L[8],表明重组表达有利于提高PaLOX对底物的亲和力。因此,分析比较了重组PaLOX和BtLOX在不同亚油酸浓度下的酶促动力学参数(表4)。结果表明,以亚油酸作为底物时,重组表达的PaLOX的底物亲和力较BtLOX高14.9倍; Kcat/Km较BtLOX高16.7倍。因此,PaLOX在底物亲和力、催化效率以及表达效果上优势明显,具有更好的开发应用潜力。
表4 LOX酶动力学参数
Table 4 LOX kinetic parameters

酶种类Km/(mmol·L-1)Vmax/(U·min-1)Kcat/(s-1)Kcat/Km/[L·(mmol·s)-1]PaLOX6.4×10-2196.5×1031.0×105BtLOX4.3×10-1122.6×1036.0×103
2.8 诱导剂优化对PaLOX表达水平的影响
如何提升外源蛋白高效、可溶表达是工业酶生产与应用的瓶颈。前期研究也发现,虽然2种细菌来源的LOX均能异源表达,但表达过程中存在着大量的包涵体,可溶表达水平较低。为进一步实现LOX的高效可溶表达,选择可溶性表达与应用潜力较好的PaLOX,通过设计不同浓度的IPTG和乳糖的组合添加,进行表达条件优化。结果表明,随IPTG浓度的降低,乳糖浓度的增加,PaLOX的酶活力在不断增加。当单独使用乳糖作为诱导剂时,PaLOX酶活力较IPTG作为诱导剂时提高了1.53倍,可溶表达水平提高了2.41倍,不可溶部分也提高了1.29倍(图7)。虽然更换诱导剂可以大幅提高PaLOX的表达水平与酶活力,但未能完全解决包涵体问题。
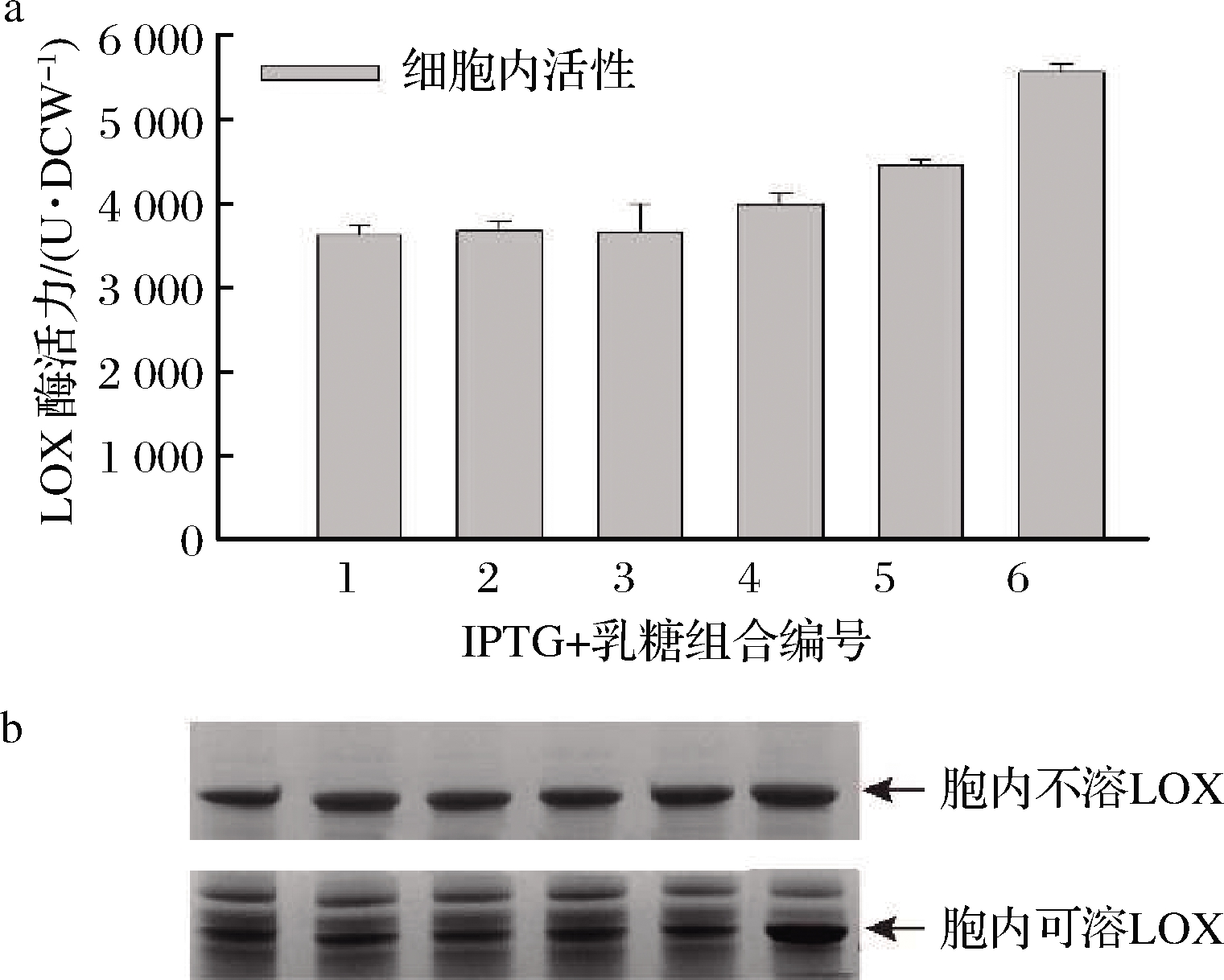
1-1 mmol/L IPTG;2-0.8 mmol/L IPTG+0.56 mmol/L乳糖; 3-0.6 mmol/L IPTG+1.11 mmol/L乳糖;4-0.4 mmol/L IPTG+ 1.67 mmol/L乳糖;5-0.2 mmol/L IPTG+2.22 mmol/L 乳糖;6-2.78 mmol/L乳糖
图7 不同浓度IPTG和乳糖浓度组合对LOX活性 和表达水平的影响
Fig.7 Effect of combination of IPTG and lactose concentration on LOX activity and expression level
2.9 分子伴侣促进PaLOX的可溶表达
在重组蛋白的表达过程中,合适的载体和分子伴侣对重组蛋白的正确折叠,防止包涵体的形成具有积极作用[20]。触发因子(triggering factor,TF)是一种来源于E.coli的核糖体结合伴侣蛋白,是第1个与新生多肽链相互作用并协助蛋白质共翻译折叠的伴侣[21],可有效促进蛋白的可溶表达。因此,构建了分子伴侣TF融合表达质粒pCold-TF/Palox(图8-a),分析TF对PaLOX可溶表达的影响。结果表明,TF-PaLOX融合酶活力较PaLOX提高了1.85倍,达到4520 U/DCW(图8-b)。SDS-PAGE分析显示,胞内不溶部分基本消失,可溶表达水平提高了3.19倍(图8-c)。与其他来源的标签相比,TF表达效率高,可溶性效果好,能有效促进PaLOX的可溶表达,具有重要的应用价值。
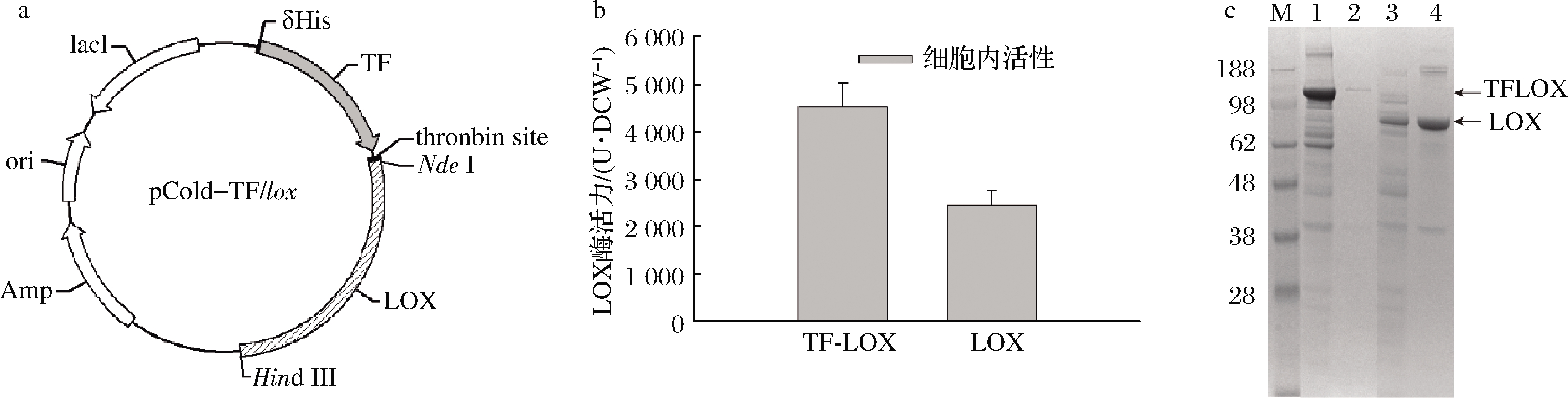
a-pCold-TF/lox表达质粒示意图;b-胞内LOX活性分析;c-SDS-PAGE电泳分析 M-蛋白质标准相对分子质量;1,2-TF-PaLOX的SDS-PAGE分析;3,4-PaLOX的SDS-PAGE分析;其中1,3-胞内可溶部分;2,4-胞内不溶部分
图8 TF对LOX表达的影响
Fig.8 Effect of TF on LOX expression
3 结论与讨论
由于LOX在食品、医药、造纸等领域有着广泛的应用,研究者对其进行了大量研究,包括从天然酶的分离提取[22-23]到LOX异源表达与分子改造等[17, 24]。到目前为止,已经有大量来自植物[25]、动物[26-27]和真菌[16, 24]等的LOX基因被克隆并成功表达。本研究以原核细菌来源的P. aeruginosa、B.thailandensis LOX基因为研究对象,通过将其在E.coli中异源表达,比较酶学性质,并利用优化诱导剂及分子伴侣融合表达策略,最终实现了LOX在E.coli表达系统中的高效可溶性表达。实验结果表明,TF作为促折叠的分子伴侣,通过与未折叠的蛋白结合,有效促进LOX的可溶表达。另外,TF催化肽基脯氨酰键的顺反异构化,可能也有利于改善蛋白质折叠过程中的限速步骤[21]。
目前研究表明,细菌来源的LOX的最适反应温度和热稳定性较其他来源的LOX差,限制了其工业应用。已有对LOX热稳定性研究的报道,例如通过在LOX的N-端和C-端的loop结构域(L6)插入1~3个L6序列[28],或融合双亲短肽[8],以及高柔性loop区域改造[29]等策略,可有效提高其热稳定性。因此,在后期研究中,进一步利用理性改造、定向进化等策略进行LOX分子改良,并结合高效的蛋白表达系统,将有利于实现LOX的高效生物合成,有效拓展LOX的工业应用范围。
[1] ELLEN H, SUSAN K, ALENA L, et al. Identification of an amino acid determinant of pH regiospecificity in a seed lipoxygenase from Momordica charantia [J]. Phytochemistry, 2008, 69(16): 2 774-2 780.
[2] ANON. Evolution and expansion of the prokaryote-like lipoxygenase family in the brown alga Saccharina japonica [J]. Frontiers in Plant Science, 2017, 8: 2 018.
[3] RODCASEY, HUGHES R K J F B. Recombinant lipoxygenases and oxylipin metabolism in relation to food quality [J]. Food Biotechnology, 2004, 18(2): 135-170.
[4] CASEY R, WEST S I, HARDY D, et al. New frontiers in food enzymology: Recombinant lipoxygenases [J]. Trends in Food Science & Technology, 1999, 10(9): 297-302.
[5] ZHANG C, TAO T, YING Q, et al. Extracellular production of lipoxygenase from Anabaena sp. PCC 7120 in Bacillus subtilis and its effect on wheat protein [J]. Applied Microbiology and Biotechnology, 2012, 94(4): 949-958.
[6] SANTANO E, PINTO M D C, PEDRO M. Chlorpromazine oxidation by hydroperoxidase activity of covalent immobilized lipoxygenase [J]. Biotechnology and Applied Biochemistry, 2002, 36(2): 95-100.
[7] MAHALIK S, SHARMA A K, MUKHERJEE K J. Genome engineering for improved recombinant protein expression in Escherichia coli [J]. Microbial Cell Factories, 2014, 13(1): 177.
[8] LU X, LIU S, ZHANG D, et al. Enhanced thermal stability and specific activity of Pseudomonas aeruginosa lipoxygenase by fusing with self-assembling amphipathic peptides [J]. Applied Microbiology and Biotechnology, 2013, 97(21): 9 419-9 427.
[9] KATERYNA G, SABINE S, DAGMAR H, et al. Functional characterization of a novel arachidonic acid 12S-lipoxygenase in the halotolerant bacterium Myxococcus fulvus exhibiting complex social living patterns [J]. Microbiology Open, 2019, 8(7): e775.
[10] STEFAN H, TERTIUS C, PIETER S. Lipoxygenases: From isolation to application [J]. Comprehensive Reviews in Food Science and Food Safety, 2017, 16(1): 199-211.
[11] RUSSELL R J, GERIKE U, DANSON M J, et al. Structural adaptations of the cold-active citrate synthase from an Antarctic bacterium [J]. Structure, 1998, 6(6): 351-361.
[12] CIAR N
N  ’ F
’ F G
G IN. Enzyme stabilization—recent experimental progress [J]. Enzyme and Microbial Technology, 2003, 33(2): 137-149.
IN. Enzyme stabilization—recent experimental progress [J]. Enzyme and Microbial Technology, 2003, 33(2): 137-149.
[13] LU X, ZHANG J, LIU S, et al. Overproduction, purification, and characterization of extracellular lipoxygenase of Pseudomonas aeruginosa in Escherichia coli [J]. Applied Microbiology and Biotechnology, 2013, 97(13): 5 793-5 800.
[14] AN J U, KIM B J, HONG S H, et al. Characterization of an omega-6 linoleate lipoxygenase from Burkholderia thailandensis and its application in the production of 13-hydroxyoctadecadienoic acid [J]. Applied Microbiology and Biotechnology, 2015, 99(13): 5 487-5 497.
[15] CAO S, CHEN H, ZHANG C, et al. Heterologous expression and biochemical characterization of two lipoxygenases in oriental melon, Cucumis melo var. makuwa Makino [J]. Plos One, 2016, 11(4): e0153801.
[16] KELLE S, ZELENA K, KRINGS U, et al. Expression of soluble recombinant lipoxygenase from Pleurotus sapidus in Pichia pastoris [J]. Protein Expression and Puripication, 2014, 95: 233-239.
[17] TASAKI Y, TOYAMA S, KURIBAYASHI T, et al. Molecular characterization of a lipoxygenase from the basidiomycete mushroom Pleurotus ostreatus [J]. Bioscience Biotechnology and Biochemistry, 2013, 77(1): 38-45.
[18] HUI Q, BINGJIE X, YUJUN H, et al. Expression, purification, and characterization of a novel acidic Lipoxygenase from Myxococcus xanthus [J]. Protein Expres. Purif., 2017, 138: 13-17.
[19] VIDAL-MAS J, BUSQUETS M, MANRESA A. Cloning and expression of a lipoxygenase from Pseudomonas aeruginosa 42A2 [J]. Antonie Van Leeuwenhoek, 2005, 87(3): 245-251.
[20] BALCHIN D, HAYER-HARTL M, HARTL F U. In vivo aspects of protein folding and quality control [J]. Science, 2016, 353(6 294): aac 4354.
[21] KRAMER G, PATZELT H, RAUCH T, et al. Trigger factor peptidyl-prolyl cis/trans isomerase activity is not essential for the folding of cytosolic proteins in Escherichia coli [J]. Journal of Biological Chemistry, 2004, 279(14): 14 165-14 170.
[22] SHARMA B, CHUGH L K. Two isoforms of lipoxygenase from mature grains of pearl millet [Pennisetum glaucum (L.) R. Br.]: Purification and physico-chemico-kinetic characterization [J]. Journal of Food Science & Technology, 2017, 54(6): 1 577-1 584.
[23] 王卫东, 杨万根, 付湘晋. 白鲢鱼肌肉脂肪氧合酶的分离纯化与鉴定 [J]. 食品科学, 2010, 31(23): 164-166.
[24] AKIKO S, HENRIK S L, KENJI M, et al. Characterization of two fungal lipoxygenases expressed in Aspergillus oryzae [J]. Journal of Bioscience and Bioengineering, 2018, 126(4): 436-444.
[25] EGMOND M R, BRUNORI M, FASELLA P M. The steady-state kinetics of the oxygenation of linoleic acid catalysed by soybean lipoxygenase [J]. European Journal of Biochemistry, 2010, 61(1): 93-100.
[26] JIN G, ZHANG J, YU X, et al. Crude lipoxygenase from pig muscle: Partial characterization and interactions of temperature, NaCl and pH on its activity [J]. Meat Science, 2011, 87(3): 257-263.
[27] KUHN H, WALTHER M, KUBAN R J. Mammalian arachidonate 15-lipoxygenases-Structure, function, and biological implications [J]. Prostaglamdins & Other Lipid Mediators, 2002, 68-69: 263-290.
[28] 王广圣,徐智,刘松,陈坚,堵国成.重组脂肪氧合酶分子改造[J].食品科技,2014,39(9):2-7.
[29] LU X, LIU S, FENG Y, et al. Enhanced thermal stability of Pseudomonas aeruginosa lipoxygenase through modification of two highly flexible regions [J]. Applied Microbiology & Biotechnology, 2014, 98(4): 1 663-1 669.