木糖异构酶是一种极具价值的工业酶,在细胞体内可将木糖异构化为木酮糖;在体外可转化葡萄糖形成果糖, 并已经用于高果糖浆的工业生产[1]。由于木糖异构酶可以很好地弥补氧化还原途径时产生大量中间产物这个缺陷,木糖异构酶被用于基因工程等研究,提高发酵效率[2]。木糖异构酶的作用与二价阳离子相关,对Mn2+有极强的亲和力。豆科类木糖异构酶的晶体结构显示了与其他类木糖异构酶不同的活性位点。其D-木糖结合位点包含2个色氨酸、1个催化组氨酸以及2个由保守的天冬氨酸和谷氨酸羧基形成的金属结合位点。金属配位残基的结合位置和构象因金属种类的不同而略有不同,这可能导致木糖异构酶的活性依赖金属[3-4]。木糖异构酶催化D-葡萄糖转化为D-果糖的异构反应速率快。近年来,木糖异构酶在乙醇生产中的应用也成为研究热点[5-6]。
豆科,属于双子叶植物纲,多为灌木、亚灌木,乔木,根部有根瘤菌,是自然界为数不多的固氮的生物。种类繁多与分布广泛的豆科植物为人类的生活提供了很大的帮助,如重要的经济价值、营养价值、药用价值、生态价值以及观赏价值等[7]。我国豆类品种繁多,开发豆类更多的价值具有很大的应用潜力[7-8]。
随着分子生物学的发展,大麦木糖异构酶基因[9]、枸杞木糖异构酶基因[10]等越来越多的木糖异构酶基因被克隆。但有关木糖醇异构酶基因家族的研究尚鲜见报道。本研究通过生物信息学方法研究豆科植物木糖异构酶基因家族的分类、基因结构、系统进化关系等,为深入研究豆科植物中木糖异构酶基因的功能奠定理论基础。
1 材料与方法
1.1 材料获取
利用美国国家生物信息中心(National Center for Biotechnology Information,NCBI)网站(https://www.ncbi.nlm.nih.gov/)的BLAST功能,将大豆木糖异构酶基因XP_003549495进行比对。经过BLAST比对、鉴定筛选出34条木糖异构酶基因序列,并从NCBI网站下载。
1.2 木糖异构酶基因亚细胞定位、基序(Motif)分析、保守区分析
木糖异构酶基因亚细胞定位的预测是利用WoLF PSORT网站(http://www.genscript.com/wolf-psort.html?src=leftbar)获得。木糖异构酶基因的Motif分析利用MEME网站(http://meme-suit.org/tools/meme)。利用NCBI中的Domain position(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsd.cgi) 分析34条木糖异构酶基因保守区[11-15]。木糖异构酶基因的基因结构由TBtools软件程序绘制[16]。
1.3 木糖异构酶基因一级结构和二级结构预测
利用ExPASy网站(https://web.expasy.org/protparam/)对一级结构进行分析[17],将结果绘制成表格。蛋白质二级结构利用PredictProtein[18] (http://www.predictprotein.org/)分析完成,将结果分别绘制成表格。
1.4 进化树分析
利用Clustal x1.83[19]软件对34条木糖异构酶基因进行比对分析,利用MEGA7[20]软件中的邻接 (neighbor-joining,NJ)法(Bootstrap,重复数=1 000)来构建系统发生树。
2 结果与分析
2.1 基因序列分析和亚细胞定位
34条木糖异构酶基因序列主要集中在落花生属、大豆属、牧豆树属、羽扇豆属及豇豆属的植物当中。这些基因序列的长度不一,大部分的长度都在1 413~1 443 bp,在落花生属当中的序列都比较长,分别是:Arachis ipaensis 2 421 bp、Arachis hypogaea 1 866 bp、Arachis duranensis 1 815 bp(表1)。
通过WoLF PSORT网站得到34条序列的亚细胞定位的预测结果表明(表1),不同属的木糖异构酶亚细胞定位的预测主要位置在线粒体、细胞质、内质网。大豆属中的木糖异构酶的预测位置主要在内质网中;豇豆属在线粒体中;牧豆属在细胞骨架或者细胞质中;羽扇豆属主要是在细胞质与内质网当中;而落花生属的豆科植物的亚细胞预测位置出现了不同,有的出现在细胞质当中,有的出现在内质网,还有一部分出现在线粒体。
2.2 进化树、Motif及保守区域分析
34条木糖异构酶蛋白质序列利用MEGA软件构建进化树(图1)。34个蛋白序列在进化树上有5个可信度高的分支。分支1有13条序列,全部来自落花生属(Arachis);第2分支亲缘关系较高,都是羽扇豆属(Lupinus);分支3中包括落花生属(Arachis)和牧豆树属(Prosopis);分支4都是大豆属的序列;第5分支4条序列都是豇豆属的。
表1 豆科中木糖异构酶亚细胞定位和基因信息
Table 1 Subcellular location and gene information of xylose isomerase in Leguminosae
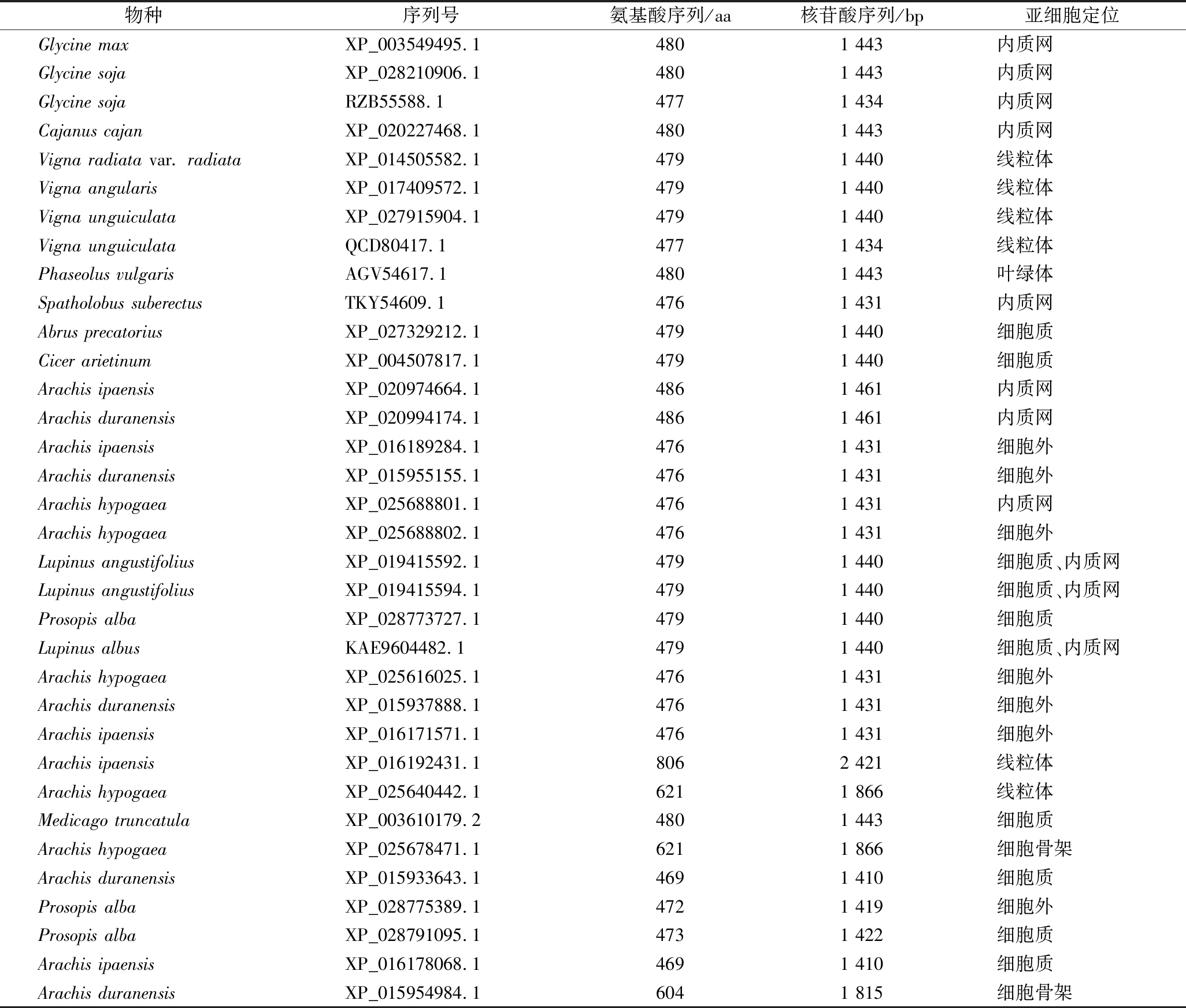
物种序列号氨基酸序列/aa核苷酸序列/bp亚细胞定位Glycine maxXP_003549495.14801 443内质网Glycine sojaXP_028210906.14801 443内质网Glycine sojaRZB55588.14771 434内质网Cajanus cajanXP_020227468.14801 443内质网Vigna radiata var. radiataXP_014505582.14791 440线粒体Vigna angularisXP_017409572.14791 440线粒体Vigna unguiculataXP_027915904.14791 440线粒体Vigna unguiculataQCD80417.14771 434线粒体Phaseolus vulgarisAGV54617.14801 443叶绿体Spatholobus suberectusTKY54609.14761 431内质网Abrus precatoriusXP_027329212.14791 440细胞质Cicer arietinumXP_004507817.14791 440细胞质Arachis ipaensisXP_020974664.14861 461内质网Arachis duranensisXP_020994174.14861 461内质网Arachis ipaensisXP_016189284.14761 431细胞外Arachis duranensisXP_015955155.14761 431细胞外Arachis hypogaeaXP_025688801.14761 431内质网Arachis hypogaeaXP_025688802.14761 431细胞外Lupinus angustifoliusXP_019415592.14791 440细胞质、内质网Lupinus angustifoliusXP_019415594.14791 440细胞质、内质网Prosopis albaXP_028773727.14791 440细胞质Lupinus albusKAE9604482.14791 440细胞质、内质网Arachis hypogaeaXP_025616025.14761 431细胞外Arachis duranensisXP_015937888.14761 431细胞外Arachis ipaensisXP_016171571.14761 431细胞外Arachis ipaensisXP_016192431.18062 421线粒体Arachis hypogaeaXP_025640442.16211 866线粒体Medicago truncatulaXP_003610179.24801 443细胞质Arachis hypogaeaXP_025678471.16211 866细胞骨架Arachis duranensisXP_015933643.14691 410细胞质Prosopis albaXP_028775389.14721 419细胞外Prosopis albaXP_028791095.14731 422细胞质Arachis ipaensisXP_016178068.14691 410细胞质Arachis duranensisXP_015954984.16041 815细胞骨架
利用MEME网站分析34条不同物种当中的木糖异构酶序列获得了15个不同的Motif(图1)。其中有17条序列包含所有的Motif;缺少Motif 15的序列较多,其中大部分都是落花生属的,也有少数几个是牧豆树属的;缺少Motif 13的有4条序列,这4条序列都是落花生属的;缺少Motif 11的有3条序列,分别是XP_015933643.1、XP_016178068.1、XP_015954984.1;而XP_028775389.1和XP_028791095.1缺少Motif 7;唯一一个缺少Motif 12的序列是TKY54609.1。在NCBI网站中下载了这34条序列的保守区间(图1),结果表明所有序列都有木糖异构酶特有的保守区(PLN02923),大部分的序列保守区域都是在1~480 bp。进一步分析可以发现Motif 11、Moti 6、Motif 2、Motif 7、Motif 4、Motif 12、Motif 3、Motif 9、Motif 1、Motif 5、Motif 10、Motif 15都在保守区当中。
2.3 一级结构和二级结构
从ExPASy网站中可以获得豆科中木糖异构酶基因一级结构如表2所示。从表格中发现这34条序列的分子量大部分都在52 952.24~54 793.33 Da,最大的是落花生属的Arachis ipaensis (XP_016192431.1 )90 485.51 Da,最小的是牧豆树属的Prosopis alba (XP_028775389.1)52 957.1 Da。等电点的范围为pH 5.52~7.95,影响等电点的因素有很多,但主要还是看蛋白质的一级结构当中的疏水键以及R基团。将这些排序之后,发现分子量与等电点之间没有任何线形关系。
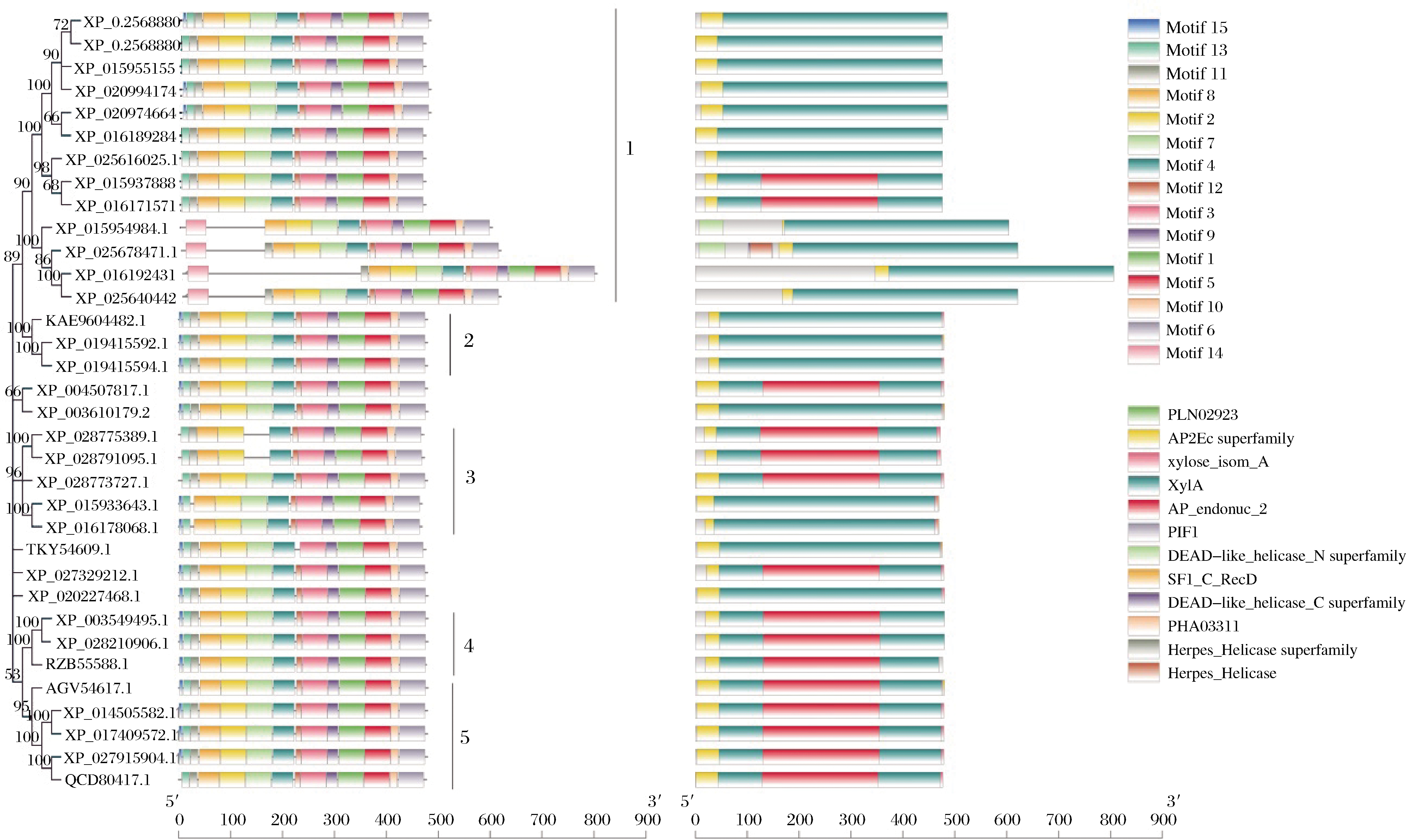
图1 豆科中木糖异构酶基因系统进化树
Fig.1 Phylogenetic tree of xylose isomerase genes in Leguminosae
利用Predict Protein分析豆科中木糖异构酶基因二级结构数据如表2所示。无规则卷曲的占比最高,是蛋白质结构重要的组成部分,α螺旋的占比次之,最少的部分是β折叠。其中,无规则卷曲的占比最高可达69.35%,为序列XP_016192431.1中,α螺旋的占比相差不大,较多的序列中α螺旋的占比在42.23%~46.46%,而β折叠的比例超过10%的只有8条序列。
表2 蛋白质的一级和二级结构信息
Table 2 Molecular weight and isoelectric point of protein

序列号分子质量/Da等电点(pI)α螺旋/%β折叠/%无规则卷曲/%XP_003549495.154 209.886.0145.428.5446.04XP_028210906.154 193.836.0145.218.7546.04RZB55588.153 910.496.3545.919.6444.44XP_020227468.154 087.545.7146.468.3345.21XP_014505582.154 109.395.8444.2610.4445.30XP_017409572.154 123.385.8444.899.8145.30XP_027915904.153 944.245.8345.519.6044.89QCD80417.153 684.875.7444.389.7945.83AGV54617.153 903.445.6544.389.7945.83TKY54609.153 576.255.9643.7010.0846.22XP_027329212.153 871.355.7645.309.1945.51XP_020974664.154 793.335.5245.889.4744.65XP_020994174.154 751.295.5245.689.8844.44
续表2
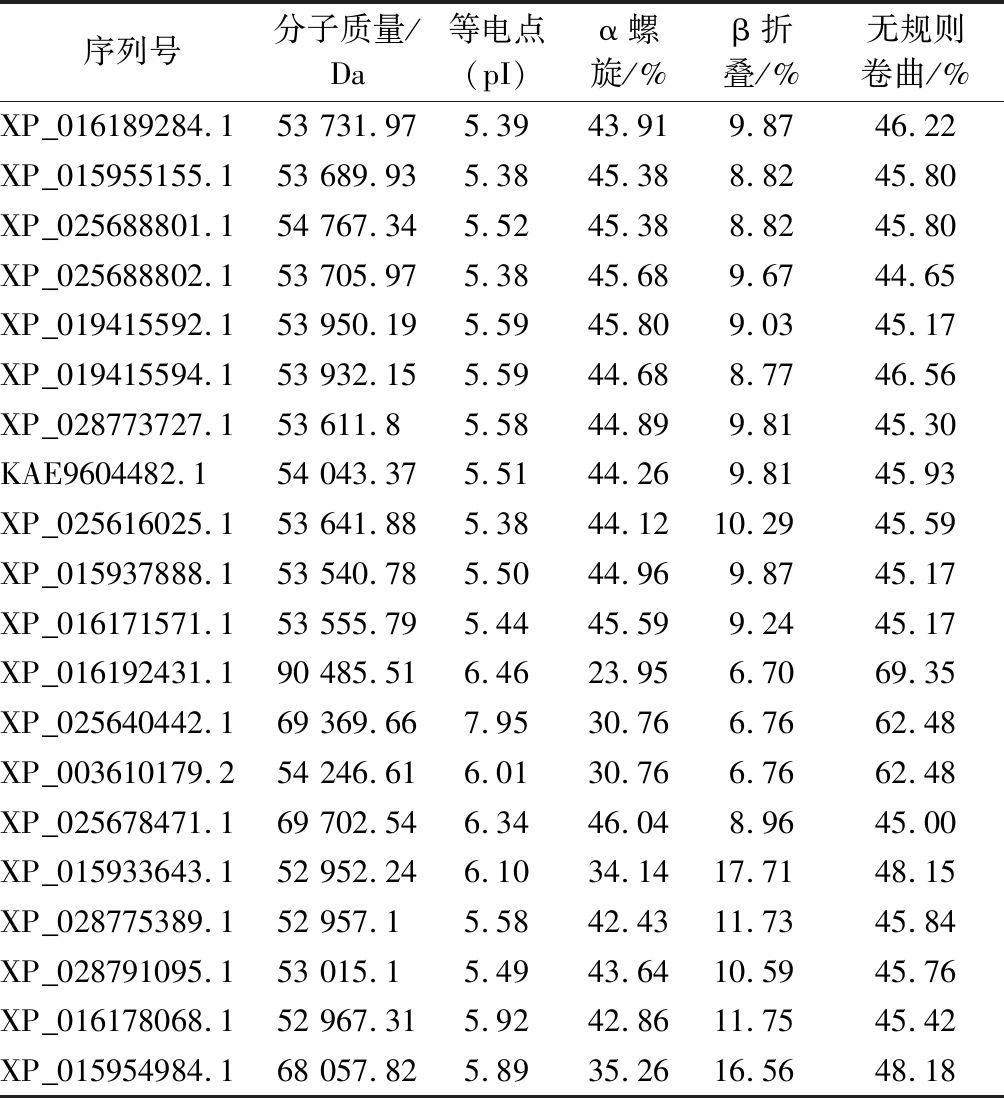
序列号分子质量/Da等电点(pI)α螺旋/%β折叠/%无规则卷曲/%XP_016189284.153 731.975.3943.919.8746.22XP_015955155.153 689.935.3845.388.8245.80XP_025688801.154 767.345.5245.388.8245.80XP_025688802.153 705.975.3845.689.6744.65XP_019415592.153 950.195.5945.809.0345.17XP_019415594.153 932.155.5944.688.7746.56XP_028773727.153 611.85.5844.899.8145.30KAE9604482.154 043.375.5144.269.8145.93XP_025616025.153 641.885.3844.1210.2945.59XP_015937888.153 540.785.5044.969.8745.17XP_016171571.153 555.795.4445.599.2445.17XP_016192431.190 485.516.4623.956.7069.35XP_025640442.169 369.667.9530.766.7662.48XP_003610179.254 246.616.0130.766.7662.48XP_025678471.169 702.546.3446.048.9645.00XP_015933643.152 952.246.1034.1417.7148.15XP_028775389.152 957.15.5842.4311.7345.84XP_028791095.153 015.15.4943.6410.5945.76XP_016178068.152 967.315.9242.8611.7545.42XP_015954984.168 057.825.8935.2616.5648.18
3 讨论与结论
本文分析了豆科家族中34条木糖异构酶基因,研究表明在氨基酸序列当中,最长的1个是落花生属的A.ipaensis (XP_016192431.1 )氨基酸序列,长度为806 aa,最短的1个是牧豆树属的P.alba (XP_028775389.1)基因,长度为472 aa,大部分的序列长度都集中在476~ 480 aa。亚细胞定位分析表明,34条序列大体可分为5个属,5个属的亚细胞定位都不大相同,但出现概率最大的还是线粒体、内质网以及细胞质这3个位点。Motif和保守域的分析表明,MEME网站获得的信息中,序列中的Motif总共有15个,其中Motif1、2、3、4、5、6、8、9、10、14在每段序列中都有,而Motif 15在多段序列中丢失。每段序列的保守区域都很接近,XP_016192431.1、XP_025640442.1、XP_025678471.1以及XP_015954984.1都有其他序列没有的Motif 14,并且在Motif 14与后一段Motif中间有较长的一段间隔,出现这段间隔的原因可能是在进化过程中有其他序列插入[21]。
从豆科家族中34条木糖异构酶基因的一级结构进行分析可知,大部分序列分子质量在52 952.24~54 793.33Da,等电点在pH 5.52~7.95。利用PredictProtein分析二级结构表明,无规则卷曲在所有序列中的占比最大,β-折叠的占比最小。这与CHANG等[22]的研究结果很相似。CHANG等通过用x射线结晶学方法测定了T.thermophilus木糖异构酶(TthXI)和T.caldophilus木糖异构酶(TcaXI)的晶体结构。在TcaXI和TthXI中,以222对称性为特征的亚基的四聚体排列和每个亚基的3次折叠与其他木糖异构酶基本相同。TcaXI的每个单体包含10个-链、15个-螺旋和6个310-螺旋,而TthXI的每个单体包含10个-链、16个-螺旋和5个310-螺旋[22]。
[1] PALAZZI E, CONVERTI A. Generalized linearization of kinetics of glucose isomerization to fructose by immobilized glucose isomerase[J]. Biotechnology & Bioengineering, 1999, 63(3): 273-284.
[2] 李云成,孟凡冰,苟敏,等.基于木糖异构酶途径的木糖发酵酿酒酵母菌株构建研究进展[J]. 生物技术通报, 2017, 33(10): 88-96.
[3] LEE M, ROZEBOOM H J, DE W P P, et al. Metal dependence of the xylose isomerase from Piromyces sp. e2 explored by activity profiling and protein crystallography[J]. Biochemistry, 2017, 56(45): 5 991-6 005.
[4] ZHU X, TENG M, NIU L, et al. Structure of xylose isomerase from Streptomyces diastaticus No. 7 strain M1033 at 1.85 a resolution[J]. Acta Crystallogra D Biol Crystallogr, 2000, 56(Pt2):129-136.
[5] ZHANG B, LI X L, FU J, et al. Production of acetoin through simultaneous utilization of glucose, xylose, and arabinose by engineered Bacillus subtilis[J]. PLoS One, 2016, 11(7): e0159298.
[6] TANINO T, HOTTA A, ITO T, et al. Construction of a xylose-metabolizing yeast by genome integration of xylose isomerase gene and investigation of the effect of xylitol on fermentation[J]. Applied Microbiology and Biotechnology, 2010, 88(5): 1 215-1 221.
[7] 刘虹,易丽莎,蒲乙琴,等.中国野生豆科植物资源及豆类蛋白研究概况[J]. 生物资源, 2019, 41(3): 185-194.
[8] 薛洁, 苑璐, 张丽,等. 青岛崂山豆科药用植物资源多样性调查[J]. 山东农业科学, 2015,47(6): 56-60;82.
[9] KRISTO P, SAARELAINEN R, FAGERSTRÖM R, et al. Protein purification, and cloning and characterization of the cDNA and gene for xylose isomerase of barley[J]. European Journal of Biochemistry, 1996, 237(1): 240-246.
[10] 赵建华,李浩霞,尹跃,等.枸杞木糖异构酶基因LbxylA的克隆、原核表达及多克隆抗体的制备[J]. 食品科学, 2019, 40(10): 77-83.
[11] LU Shennan, WANG Jiyao, CHITSAZ F, et al. CDD/SPARCLE: the conserved domain database in 2020[J]. Nucleic Acids Research, 2020, 48(D1): 265-268.
[12] MARCHLER B A, BO Yu, HAN Lianyi, et al. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures[J]. Nucleic Acids Research, 2017, 45(D): 200-203.
[13] MARCHLER-BAUER A, DERBYSHIRE M K, GONZALES N R, et al. CDD: NCBI's conserved domain database[J]. Nucleic Acids Research, 2015, 43(D): 222-226.
[14] MARCHLER-BAUER B A, LU Shennan, ANDERSON J B, et al. CDD: A conserved domain database for the functional annotation of proteins[J]. Nucleic Acids Research, 2011, 39(D), 225-229.
[15] MARCHLER-BAUER A, BRYANT S H. CD-Search: protein domain annotations on the fly[J]. Nucleic Acids Research, 2004, 32(W): 327-331.
[16] CHEN Chengjie, XIA Rui, CHEN Hao, et al. TBtools, a Toolkit for Biologists integrating various HTS-data handling tools with a user-friendly interface[J]. BiorXiv, 2018, 289660.
[17] ELISABETH G, CHRISTINE H, ALEXANDRE G, et al. Protein Identification and Analysis Tools on the ExPASy Server[M]. Humana Press: Proteomics Protocols Handbook, 2005: 571-607.
[18] ROST B, YACHDAV G, LIU Jinfeng. The predictprotein server[J]. Nucleic Acids Research, 2004, 32(W): 321-326.
[19] THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The clustal x windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25(24): 4 876-4 882.
[20] KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33: 1 870-1 874.
[21] 齐燕妮,李闻娟,王利民,等.胡麻SUC基因家族的鉴定与生物信息学分析[J].甘肃农业科技, 2019, 35(8): 35-40.
[22] CHANG C, PARK B C, LEE D S, et al. Crystal structures of thermostable xylose isomerases from Thermus caldophilus and Thermus thermophilus: Possible structural determinants of thermostability[J]. Journal of Molecular Biology, 1999, 288(4): 623-634.