中国白酒是世界六大蒸馏酒之一,已有1 000多年的历史,是我国最具民族文化特征的产品之一,对我国国民经济和产业发展起到了重要的推动作用。迄今为止,中国白酒根据其风味特征已形成12种类型,包括浓香型、酱香型、清香型、凤香型、药香型、米香型、芝麻香型、豉香型、兼香型、老白干香型、馥郁香型、特香型。白酒酿造过程是在开放的环境下进行的,以谷物为发酵基质,大曲为糖化发酵剂,酿造过程复杂,受多种因素的影响。酿酒原料在微生物和酶的作用下产生的微量成分,构成了白酒中微量成分的主要来源,对白酒的产品质量有着重要的影响。白酒中的微量成分含量为2%~3%,是决定白酒香气口感和风格的关键。根据化学性质,可以将酒中的微量成分分为醇类、醛类、酸类、酯类、酮类、内酯类化合物、硫化物、缩醛类化合物、吡嗪类化合物、呋喃类化合物、芳香族化合物[1]。
浓香型白酒是中国白酒的典型代表之一,在中国占有70 %以上的市场份额[2]。浓香型白酒是以粮谷为原料,经传统固态法发酵、蒸馏、陈酿、勾兑而成的,未添加食用酒精及非白酒发酵产生的呈香物质,以己酸乙酯为主体复合香的白酒[3]。众所周知,微生物在白酒酿造过程中发挥着重要的作用,处于核心地位,浓香型白酒也是不同种类微生物的多微共酵作用形成的,其中大曲、窖泥及酒醅是浓香型白酒酿造的关键,依赖于其中微生物的多微共酵,形成复杂的白酒成分[4],最终形成了浓香型白酒的独特风格。随着酿酒工艺的改善和发展,浓香型白酒生产工艺日趋成熟,但其酿造机理和呈香味机制相关研究还处于起步阶段[5],微生物在白酒酿造过程中的作用机理尚未明确,微生物代谢与白酒风味物质形成的内在联系还未完全清晰。
传统的平板培养、变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)、16S rRNA基因克隆文库等技术对于分析环境微生物生态具有很大的局限性。传统的平板培养方法只能获得环境中极少数微生物的信息,不能充分反映环境微生物生态功能。ZHANG等通过PCR变性梯度凝胶电泳(DGGE)检测了窖泥中的一些不可培养的芽胞杆菌和梭菌[6]。然而,由于PCR-DGGE在很大程度上依赖于所设计的PCR引物的特异性,因此该技术不能准确反映真实的微生物群落组成。16S rRNA基因的克隆文库分析依赖于PCR扩增产物的连接转化效率,因此不能排除在表征细菌多样性时产生信息遗漏的可能性[7]。而高通量测序技术具有高通量、高分辨率的优势,已被证明是分析复杂微生物菌群的强有力手段[8],能更可靠地定量估计微生物群落结构[9]。随着高通量测序价格的持续走低,基于宏基因组测序的微生物生态学研究逐渐增多。宏基因组测序技术可以绕开“大部分环境微生物不可培养”的壁垒,不仅可以获得微生物群落的多样性和功能组成,还可以此结果为依据,发掘环境中新的功能酶或基因,可谓是将微生物分类-功能组成-应用开发结合了起来,因此在目前环境微生物学研究中占据主导地位。李斌等采用高通量测序技术分析了浓香型和芝麻香型白酒酒曲细菌群落结构,结果表明浓香型白酒中高温酒曲的优势类群主要包括乳杆菌属(Lactobacillus sp.)、魏斯氏菌属(Weissella sp.)和片球菌属(Pediococcus sp.)等[10];邓杰等[11]采用高通量测序技术发现浓香型白酒30年窖龄窖泥中古菌类群主要集中在热原体科(Thermoplasmataceae),甲烷杆菌科(Methanobacteriaceae),甲烷微菌科(Methanomicrobiaceae)、甲烷八叠球菌科(Methanosarcinaceae)和甲烷粒菌科(Methanocorpusculaceae)。
本研究拟利用高通量测序技术,全面解析浓香型白酒特殊酿造生境中微生物的群落结构,在分子生物学水平上探究特征性微生物资源,为定向筛选和深度挖掘浓香型白酒特殊酿造生境中潜在新种和特征性微生物资源奠定基础,为全面解析白酒酿造机理,提升白酒品质提供理论基础和材料保障。
1 材料与方法
1.1 样品采集
样品采集于宜宾产区浓香型白酒酿造生境,包括窖泥(N)、酒醅(J)、大曲(D)和黄水(H)。采用五点采样法,对宜宾产区的5个白酒生产企业的7个车间的浓香型白酒酿造生境进行采样。除大曲样品外,其他三类样品均是从当天出池的窖池中采样获得,采集后的样品密封,-80℃保存。
1.2 样品宏基因组DNA的提取与纯化[12]
准确称取5 g样品,加入13.5 mL DNA提取液(100 mmol/L Tris-HCl,100 mmol/L EDTA,100 mmol/L磷酸钠缓冲液,1.5 mol/L NaCl, 1% CTAB,pH 8.0)和100 μL蛋白酶K(10 mg/mL),37 ℃,220 r/min振荡处理30 min。加入1.5 mL 20% SDS, 65 ℃温育2 h。6 000 r/min离心10 min,收集上清。在沉淀中加入4.5 mL DNA提取液和0.5 mL 20% SDS,漩涡振荡混匀,65 ℃温育10 min,6 000 r/min离心10 min,合并上清。加入等体积酚-氯仿-异丙醇(25∶24∶1)溶液,漩涡振荡混匀,静置10 min后,12 000 r/min离心20 min。收集上层水相,加入0.7倍体积异丙醇,室温沉淀1 h,12 000 r/min离心20 min,弃上清。向沉淀中加入1 mL 70 %乙醇,洗涤2次后,加入100 μL无菌蒸馏水过夜溶解,即得到样品的粗DNA。为了满足后续操作要求,使用Cycle-pure Kit (D6492,Omega)对提取的粗DNA进行纯化,待用。
1.3 PCR扩增及测序
以纯化的宏基因组DNA为模板,使用16S rRNA基因V4区通用引物338F (5′-ACTCCTACGGGAGGCAGCA-3′)和806R (5′-GGACTACHVGGGTWTCTAA T-3′)进行PCR扩增。PCR扩增体系:5×FastPfu缓冲液8 μL,2.5 mmol/L dNTPs 4 μL,10 μmol/L 上、下游引物各2 μL,FastPfu酶1 μL,DNA 30 ng,ddH2O补足至40 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 45 s,72 ℃ 45 s,共27个循环;72 ℃ 10 min[13]。对PCR 产物进行切胶纯化回收,定量后进行文库构建,具体步骤如下:使用T4 DNA Polymerase、Klenow DNA Polymerase和T4 PNK将打断形成的黏性末端修复成平末端,再通过3′端加碱基“A”,使得DNA片段能与3′端带有“T”碱基的特殊接头连接,使用illumina平台对质控合格的文库进行测序。
1.4 测序数据分析
利用QIIME 2[14]的DADA2方法[15]对下机数据进行质控,去除低质量序列、嵌合体并得到特征表(feature table)和代表序列(feature data),利用QIIME2中q2-phylogeny方法和iTOL[16]构建系统发育树,利用QIIME2中q2-diversity方法进行Alpha和Beta多样性分析,利用shannon指数,评估样品微生物多样性;通过Bray-Curtis距离矩阵进行主坐标轴分析(PCoA, principle coordinates analysis),利用QIIME2 q2-feature-classifier方法对物种进行分类,统计作图利用ggplot2[17]和STAMP软件[18]完成。
2 结果与分析
2.1 样品的采集
通过文献查阅和实地调研,发现宜宾市区和南溪区是宜宾浓香型白酒的两个主要产区。因此,本研究分别从宜宾市区的叙府酒业和川雄酒业,以及南溪区金南福酒业、今良造酒业和六尺巷酒业的7个车间进行采样,并详细记录采样信息,包括样品名称、采样地区、采样地点、样品类型和窖池窖龄等信息,其中样品以采样酒厂、采样车间和样品类型来命名。采用五点法,采集了来自上述5家酒企的窖泥(N)、大曲(D)、出池阶段的酒醅(J)和黄水(H)样品,共计26份,其中叙府酒业的3个不同车间窖池使用的是同一批次的大曲,因此,只采集了五车间大曲样品。
宜宾市区的白酒生产企业的窖池窖龄普遍较高,其中川雄酒业窖池使用了40年,叙府酒业的3个车间窖池的窖龄分别为15、20和30年;南溪区白酒生产企业的窖池窖龄范围较大,既包括金南福酒业使用30年的窖池,也包括六尺巷酒业仅使用3年的新窖池,而今良造酒业窖池比较特殊,窖泥是从宜宾市区整体转移到南溪区,继续进行白酒生产的,在南溪区使用了5年,整体窖龄为20年。因此,本研究采集的样品既包括了宜宾地区浓香型白酒的两个主要产区,又包括不同窖龄的窖池中窖泥、大曲、酒醅和黄水,具有良好的代表性,具体采样信息见表1。
表1 浓香型白酒酿造生境采样信息表
Table 1 Sampling information of strong flavour Chinese Baijiu fermented habitat

样品名称采样地区采样地点样品类型窖池窖龄XF_2叙府酒业2车间2排7号窖泥、酒醅、黄水30年XF_3宜宾市区叙府酒业3车间2排21号窖泥、酒醅、黄水20年XF_5叙府酒业5车间1排4号窖泥、大曲、酒醅、黄水15年CX川雄酒业1排6号窖泥、大曲、酒醅、黄水40年JNF金南福酒业5排2号窖泥、大曲、酒醅、黄水30年JLZ南溪区今良造酒业1排31号窖泥、大曲、酒醅、黄水5年(20年)*LCX六尺巷酒业3017号窖泥、大曲、酒醅、黄水3年
注:*,今良造酒业的窖泥是从宜宾市区整体转移到南溪区,在南溪区使用了5年,整体窖龄为20年。
2.2 样品宏基因组的提取、纯化与测序分析
本研究采集的窖泥、大曲、酒醅、黄水均为复杂基质的样品,提取的宏基因组DNA含有大量的腐殖酸,会影响后续的PCR扩增与测序分析。因此,本研究使用Cycle Pure Kit(D6492,Omega)对提取的粗DNA进行纯化回收,纯化后的宏基因组DNA的浓度和纯度均满足PCR扩增与测序的要求。利用Illumina PE150测序平台对宜宾产区浓香型白酒特殊酿造生境中细菌进行高通量测序,解析其微生物群落结构。测序结果表明,26个样品共获得674.21 Mbp数据,1 345 405条有效序列,序列平均长度为498 bp,平均每个样品的有效序列超过51 000条,数据量超过25 Mbp,优质序列覆盖率均高于97%,且稀释曲线均趋于平稳,表明本研究的测序数据质量满足微生物群落结构的分析的要求,具体测序信息见表2。
表2 样品高通量测序信息表
Table 2 High-throughput sequencing information of samples
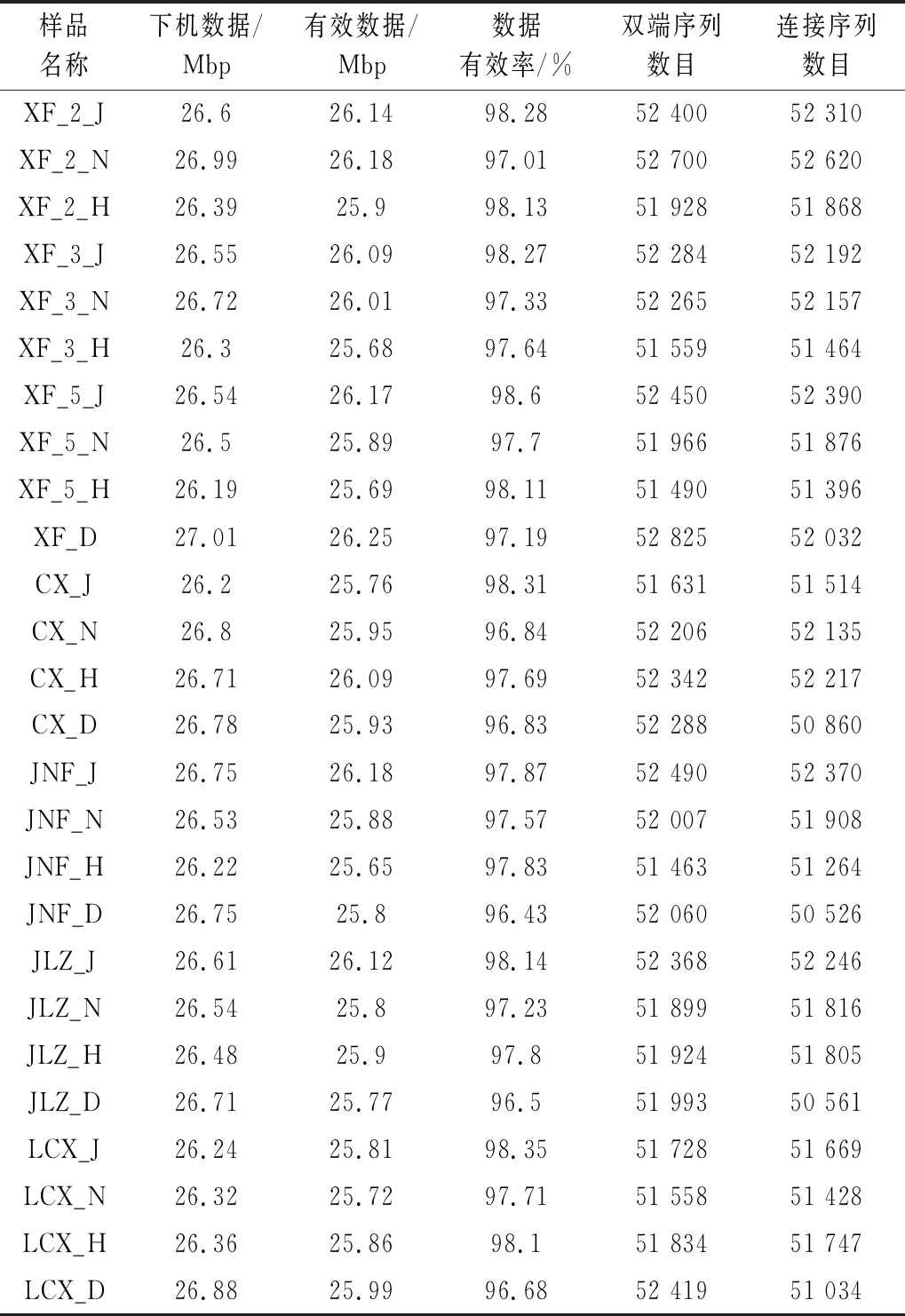
样品名称下机数据/Mbp有效数据/Mbp数据有效率/%双端序列数目连接序列数目XF_2_J26.626.1498.2852 40052 310XF_2_N26.9926.1897.0152 70052 620XF_2_H26.3925.998.1351 92851 868XF_3_J26.5526.0998.2752 28452 192XF_3_N26.7226.0197.3352 26552 157XF_3_H26.325.6897.6451 55951 464XF_5_J26.5426.1798.652 45052 390XF_5_N26.525.8997.751 96651 876XF_5_H26.1925.6998.1151 49051 396XF_D27.0126.2597.1952 82552 032CX_J26.225.7698.3151 63151 514CX_N26.825.9596.8452 20652 135CX_H26.7126.0997.6952 34252 217CX_D26.7825.9396.8352 28850 860JNF_J26.7526.1897.8752 49052 370JNF_N26.5325.8897.5752 00751 908JNF_H26.2225.6597.8351 46351 264JNF_D26.7525.896.4352 06050 526JLZ_J26.6126.1298.1452 36852 246JLZ_N26.5425.897.2351 89951 816JLZ_H26.4825.997.851 92451 805JLZ_D26.7125.7796.551 99350 561LCX_J26.2425.8198.3551 72851 669LCX_N26.3225.7297.7151 55851 428LCX_H26.3625.8698.151 83451 747LCX_D26.8825.9996.6852 41951 034
2.3 浓香型白酒酿造生境细菌系统发育学分析
基于16S rRNA基因V4区对细菌种类的区分度,本研究选择在属水平上解析宜宾产区浓香型白酒酿造生境细菌群落结构。利用QIIME2中q2-phylogeny方法,对26个样品中的细菌种类和丰度进行分析,共注释到来自于201个属的1 493个OTUs,主要分布于拟杆菌门(Bacteroidete)、变形菌门(Proteobacteria)和厚壁菌门(Firmicutes),其中5个属来自于古菌域的广古菌门(Euryarchaeota),利用iTOL构建这些物种的系统发育树(图1),这表明浓香型白酒酿造生境中的细菌多样性非常丰富,浓香型白酒的酿造是多种微生物共同作用的结果。
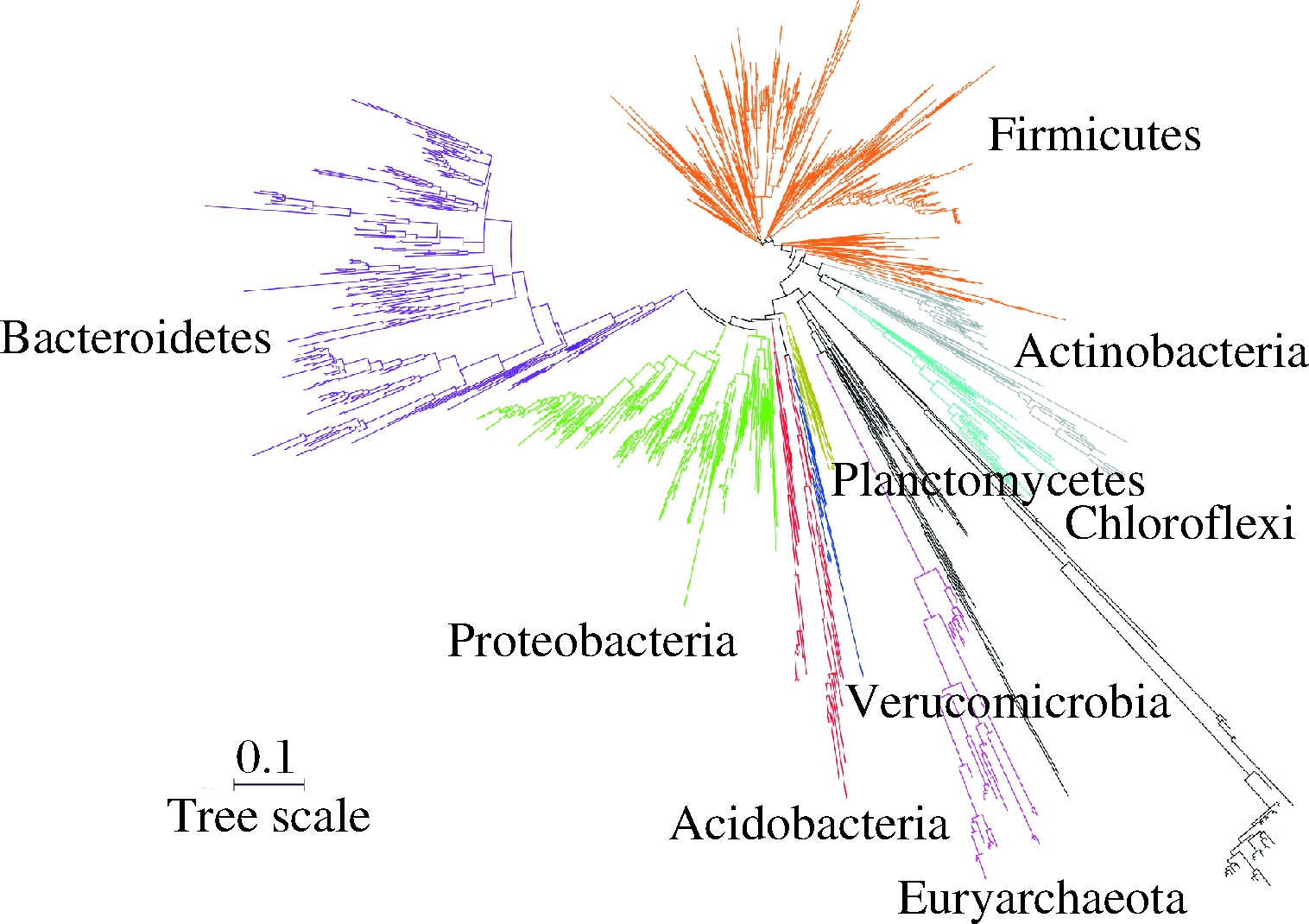
图1 宜宾产区浓香型白酒酿造生境细菌系统发育树
Fig.1 Bacterial phylogenetic tree in the fermented habitats of strong flavour Chinese Baijiu from Yibin region
2.4 浓香型白酒酿造生境细菌多样性分析
利用QIIME2中q2-diversity方法进行多样性分析,利用shannon指数评估样品中微生物多样性,以评价样品中细菌的物种丰富度(图2)。从整体上看,浓香型白酒酿造生境中的大曲和窖泥样品的物种丰富度显著高于黄水和酒醅样品,其中窖泥样品shannon指数的平均值最高,达到5.022,其中XF_3_N样品的shannon指数达到7.809;大曲样品shannon指数的平均值也达到4.805,XF_5_D样品的shannon指数达到5.513;黄水样品shannon指数波动较大,在1.994~5.156,平均值为2.987;酒醅样品的shannon指数最低,平均值仅为1.533,其中XF_2_J的shannon指数仅为0.930。从每家浓香型白酒生产企业来看,细菌多样性变化趋势与整体变化趋势也是一致的,即大曲和窖泥样品的物种多样性显著高于黄水和酒醅样品,这表明浓香型白酒酿造是从多种微生物共同作用开始,通过环境和工艺的选择,最终逐渐形成功能微生物为主的群落结构。
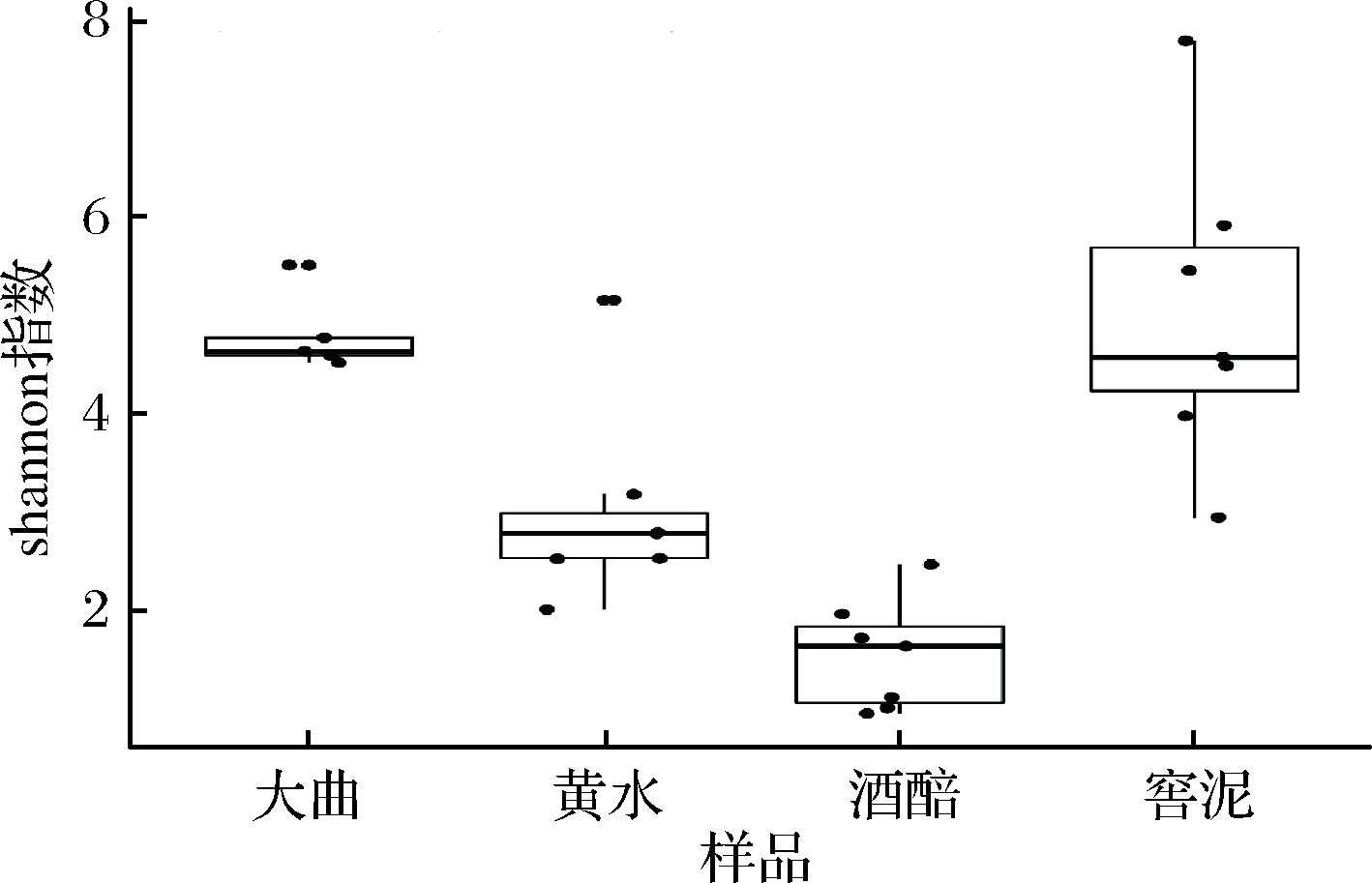
图2 宜宾产区浓香型白酒酿造生境细菌多样性分析
Fig.2 Bacterial diversity analysis in the fermented habitats of strong flavour Chinese Baijiu from Yibin region
2.5 浓香型白酒酿造生境细菌种类和丰度分析
利用QIIME 2 q2-feature-classifier方法对物种进行分类,通过ggplot2绘制堆积图来展示每个样品的微生物类别和丰度(图3)。生产浓香型白酒,窖泥是基础,大曲是动力,工艺是关键。大曲是我国固态白酒酿造特有的糖化发酵剂,含有大量微生物和酶类,在传统白酒酿造过程中发挥重要的作用。在本研究的大曲样品中,未鉴定到属水平的细菌为主要类群,占比在60%~89%。除此之外,芽胞杆菌属(Bacillus sp.)、片球菌属(Pedicoccus sp.)、乳杆菌属(Lactobacillus sp.)、魏斯氏菌属(Weissella sp.)、明串珠菌属(Leuconostoc sp.)、高温放线菌属(Thermoactinomyces sp.)和醋杆菌属(Acetobacter sp.)为大曲中的主要微生物类群。其中芽胞杆菌能够分泌多种水解酶,通过水解作用提高大曲原料的利用率,提升白酒的风味成分;丰富的乳酸菌资源为后续厌氧发酵提供了重要的功能菌种资源;高温放线菌属菌株能够产生发达的菌丝体,疏松大曲内部结构,在维持大曲骨架方面发挥重要作用[19-20]。
窖泥的好坏直接决定着浓香型白酒酒质的优劣。细菌群落结构分析结果表明,甲烷杆菌属(Methanobacterium sp.)、甲烷袋状菌(Methanoculleus sp.)、甲烷粒菌属(Methanocorpusculum sp.)和甲烷短杆菌属(Methanobrevibacter sp.)、瘤胃球菌属(Ruminococcus sp.)、梭菌属(Clostridium sp.)、乳杆菌属(Lactobacillus sp.)、片球菌属(Pediococcus sp.)、紫单胞菌属(Petrimonas sp.)和互养单胞菌属(Syntrophomonas sp.)是窖泥中的主要细菌菌群。其中,以甲烷菌为主的古菌在窖泥中发挥重要的作用,催化H2和CO2产生甲烷,从而解除了累积的H2对酸代谢的反馈抑制作用,提高了浓香型白酒主要香味成分己酸乙酯的含量[21-22]。紫单胞菌属(Petrimonas sp.)和互养单胞菌属(Syntrophomonas sp.)菌株主要与产甲烷菌互营共生,为甲烷菌生长提供代谢底物[21]。
黄水和酒醅中的生物物种显著降低,整个浓香型白酒酿造过程细菌群落呈现从多种微生物共同作用到功能优势微生物发挥主要作用的转变过程。黄水和酒醅中主要以片球菌属、乳杆菌属和和瘤胃球菌属为主,其中片球菌属为优势菌种,丰度在65.5%~99.7%,其中XF_2_J样品的片球菌属的丰度达到了99.7%。瘤胃球菌属主要参与丁酸等有机酸的合成,增加浓香型白酒中的呈香物质。而以片球菌和乳杆菌为主的乳酸菌是白酒发酵中后期的优势细菌,随着发酵的进行,发酵体系内酸度和乙醇浓度的升高,氧气含量逐渐减少,大部分的微生物不能够耐受高酸度、高乙醇浓度、厌氧等不利条件而逐渐衰亡,来自于大曲和窖泥的乳酸菌逐渐成为优势菌种。乳酸菌的主要产物是乳酸,能够减少白酒的刺激感,增加酒体的醇厚感,对白酒产品风味的形成有重要影响。同时,乳酸能够与其他微生物代谢产生的乙醇发生反应,生成乳酸乙酯,乳酸乙酯能够增加酒体的醇甜感、醇厚度,是重要的呈香物质[23]。
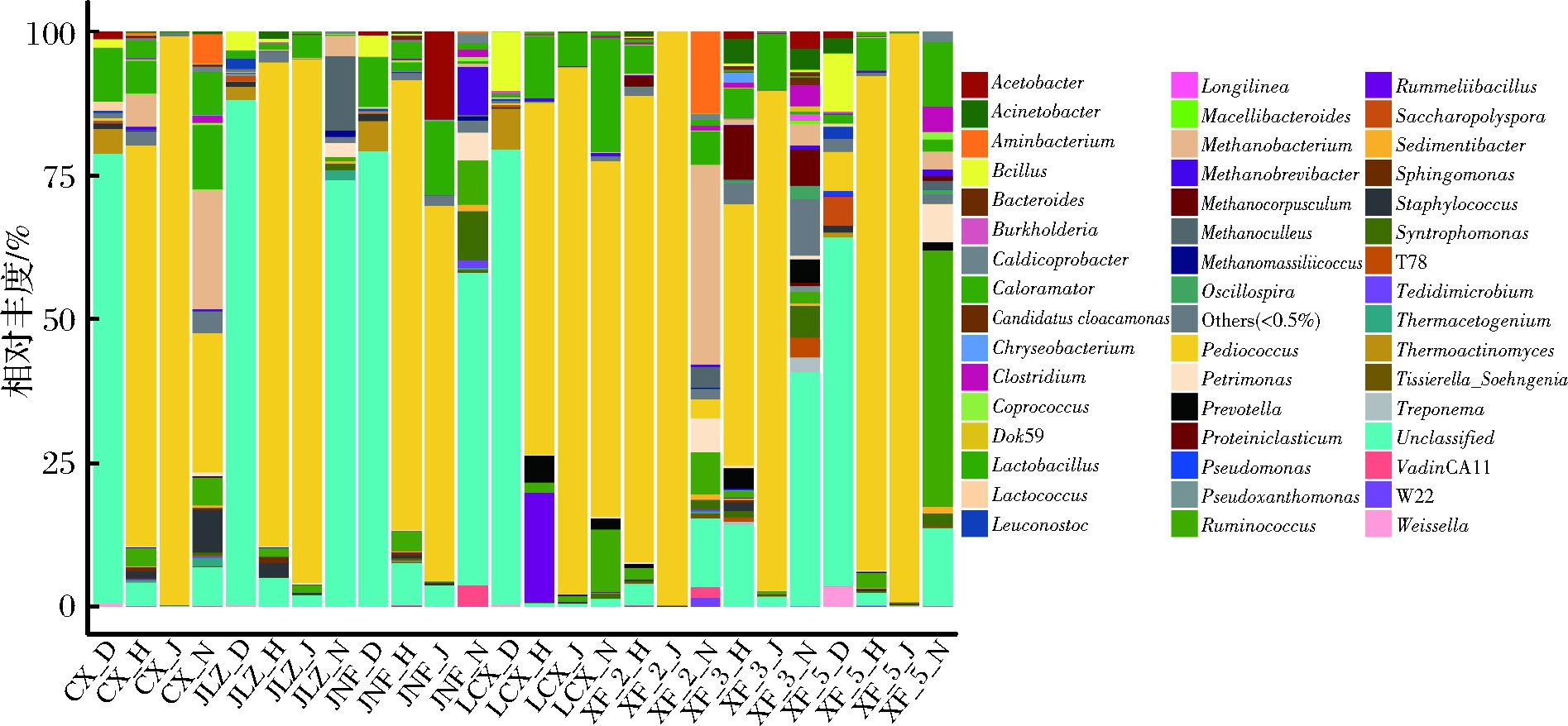
图3 细菌种类和丰度分析
Fig.3 Bacterial species and abundance analysis
2.6 浓香型白酒酿造生境不同种类样品的聚类分析
通过Bray-Curtis距离矩阵进行主坐标轴(PCoA)分析不同样品间的相关性(图4)。研究结果发现,尽管样品来源不同,但是5个大曲样品能够聚类在一起,且与其他样品有明显的差异。7个窖泥,除了窖龄最短的LCX_N(3年)外,其他6个窖龄超过15年的窖泥样品也能够聚类在一起,这说明窖泥微生物群落结构与窖龄的关系密切,推测六尺巷酒业的窖泥正在成熟过程,其中的细菌群落结构正在活跃变化之中。另外,由于黄水来源于酒醅,因此这两种样品不能很好的区分,而聚为一个大的类群,且与其他样品有明显的差异。
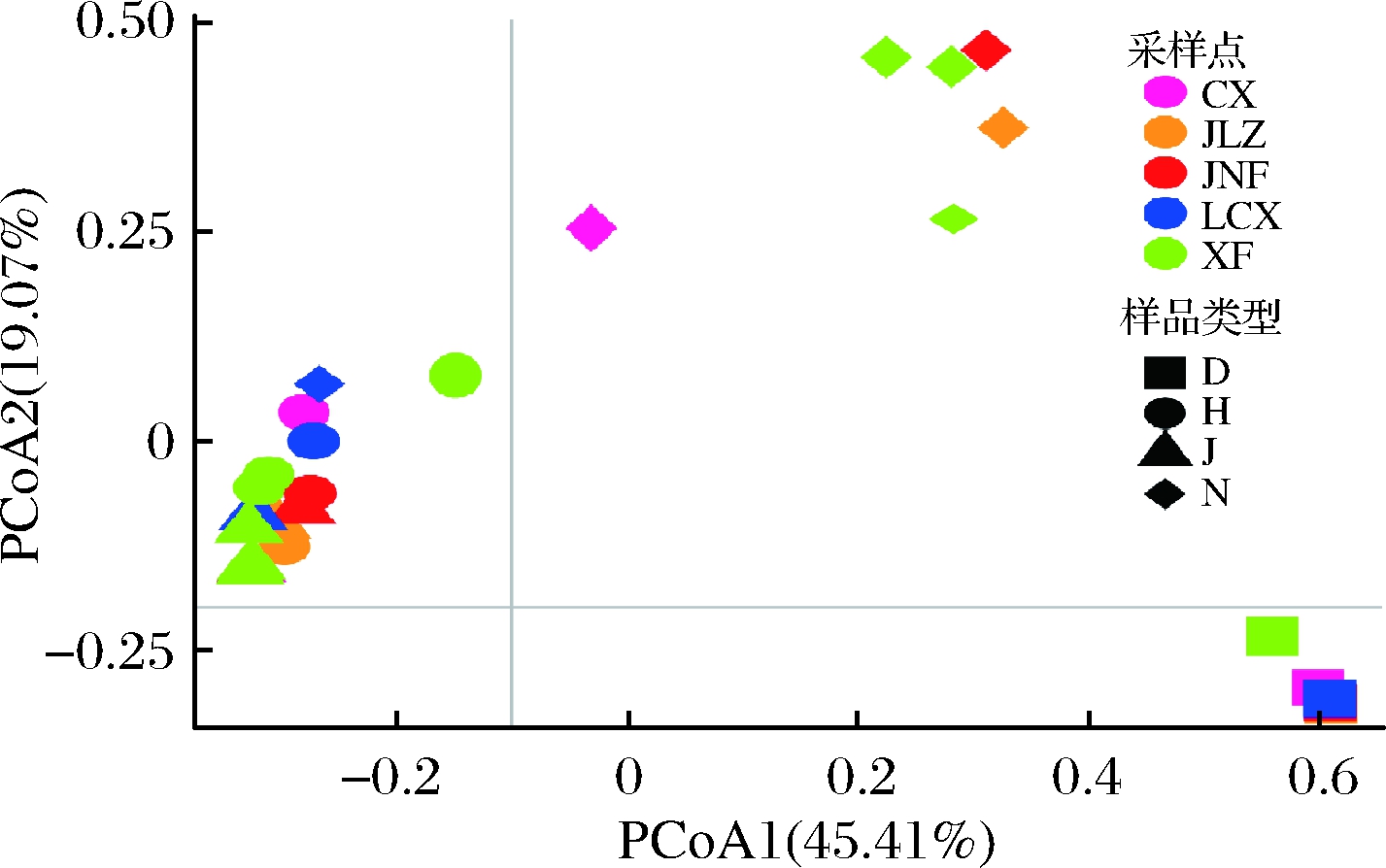
图4 主坐标轴(PCoA)分析
Fig.4 Principle coordinates analysis
通过主成分分析比较不同地区的大曲和窖泥样品的差异。分析结果发现,宜宾市区和南溪区的大曲分别聚类在一起(图5-a),这表明大曲样品微生物群落受区域环境影响较大,表现出明显的地域性。而不同区域的窖泥样品微生物群落结构并没有显示出明显的地域性,而与窖泥窖龄的关系更为相关,推测这与窖泥在慢慢熟化的过程中微生物类群不断变化有关。如图5-b所示,窖龄最短的六尺巷样品(3年)与其他样品距离较远,窖龄最长(40年)的川雄样品次之,而窖龄在15~30年的样品较好地聚类在一起,这表明窖龄对于窖泥微生物群落结构的影响高于区域环境。
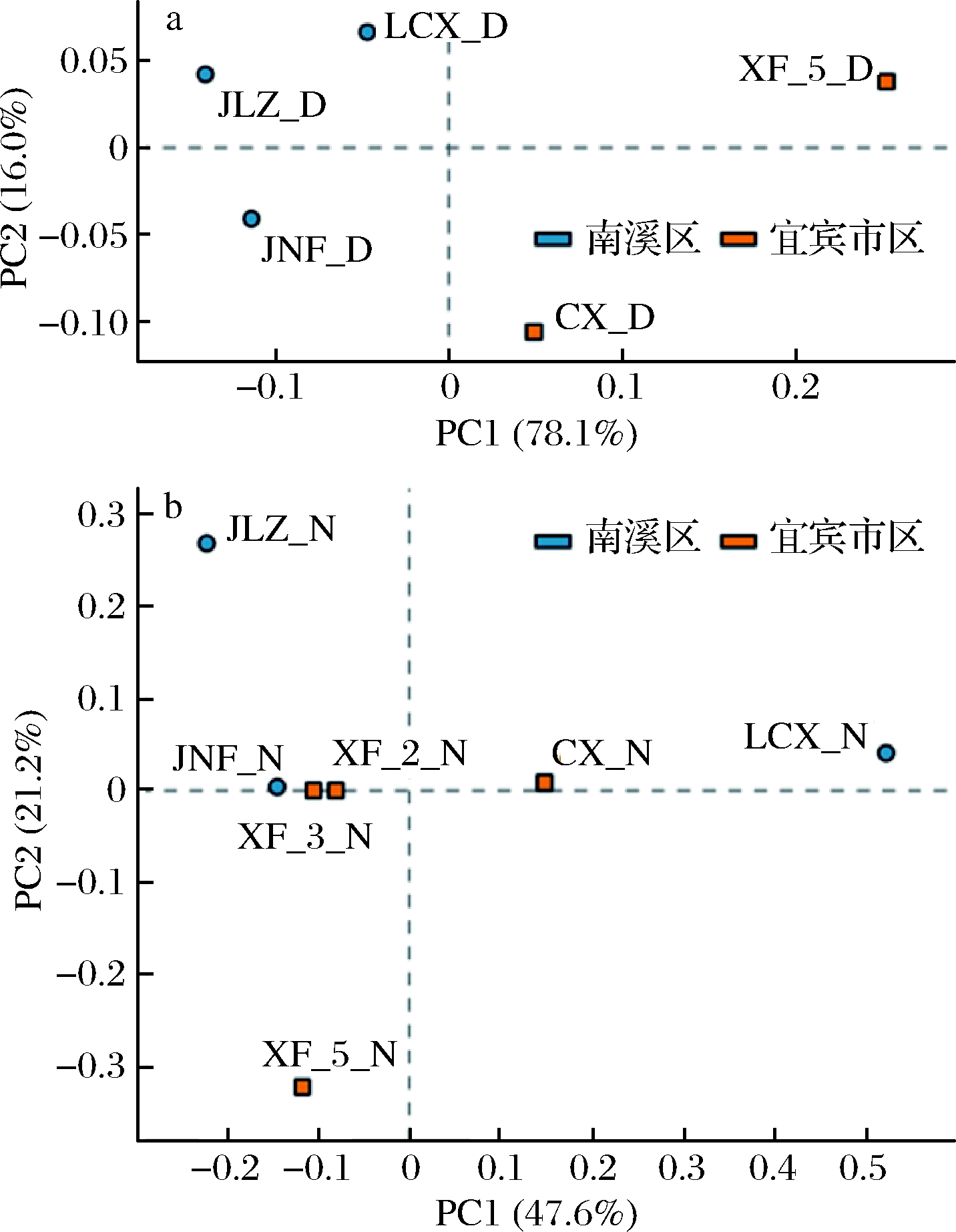
a-大曲样品;b-窖泥样品
图5 大曲和窖泥样品主成分分析
Fig.5 Principal component analysis of Daqu and pit mud samples
2.7 窖泥微生物群落结构与其窖龄的相关性研究
窖泥在浓香型白酒生产过程中发挥着重要的作用,而且窖泥的质量和窖龄息息相关,直接影响着浓香型白酒的质量。多样性指数是反映群落结构特征的重要指标,随着窖龄的增加,微生物繁衍交替,窖泥微生物多样性呈增加趋势。本研究发现,7个窖泥样品中的shannon指数在2.933~7.809,其中XF_3_N样品最高,表明该样品中微生物群落结构最为丰富;而3年窖龄的LCX_N样品shannon指数最低,推测该样品中的微生物群落正在逐步形成过程中,物种的丰富程度有待加强。
古菌在窖泥中发挥着重要的作用,能够利用己酸菌代谢产生H2产生甲烷,从而解除了累积的H2对己酸代谢的反馈抑制作用,提高了浓香型白酒主要香味成分含量。不同窖龄的样品比较发现,在窖龄最短的六尺巷窖泥样品(3年)中并未发现古菌的存在,而其他窖泥样品中的古菌主要以甲烷杆菌属、甲烷袋状菌属、甲烷粒菌属和甲烷短杆菌属为主,这些甲烷菌均为氢营养型产甲烷菌,能够利用H2和CO2生成甲烷,降低窖泥中H2含量。其中窖龄较长的CX_N(40年)和XF_2_N(30年)窖泥样品中以甲烷杆菌属菌株为主(图6),推测该属菌株可以为窖泥窖龄判断的指示菌。
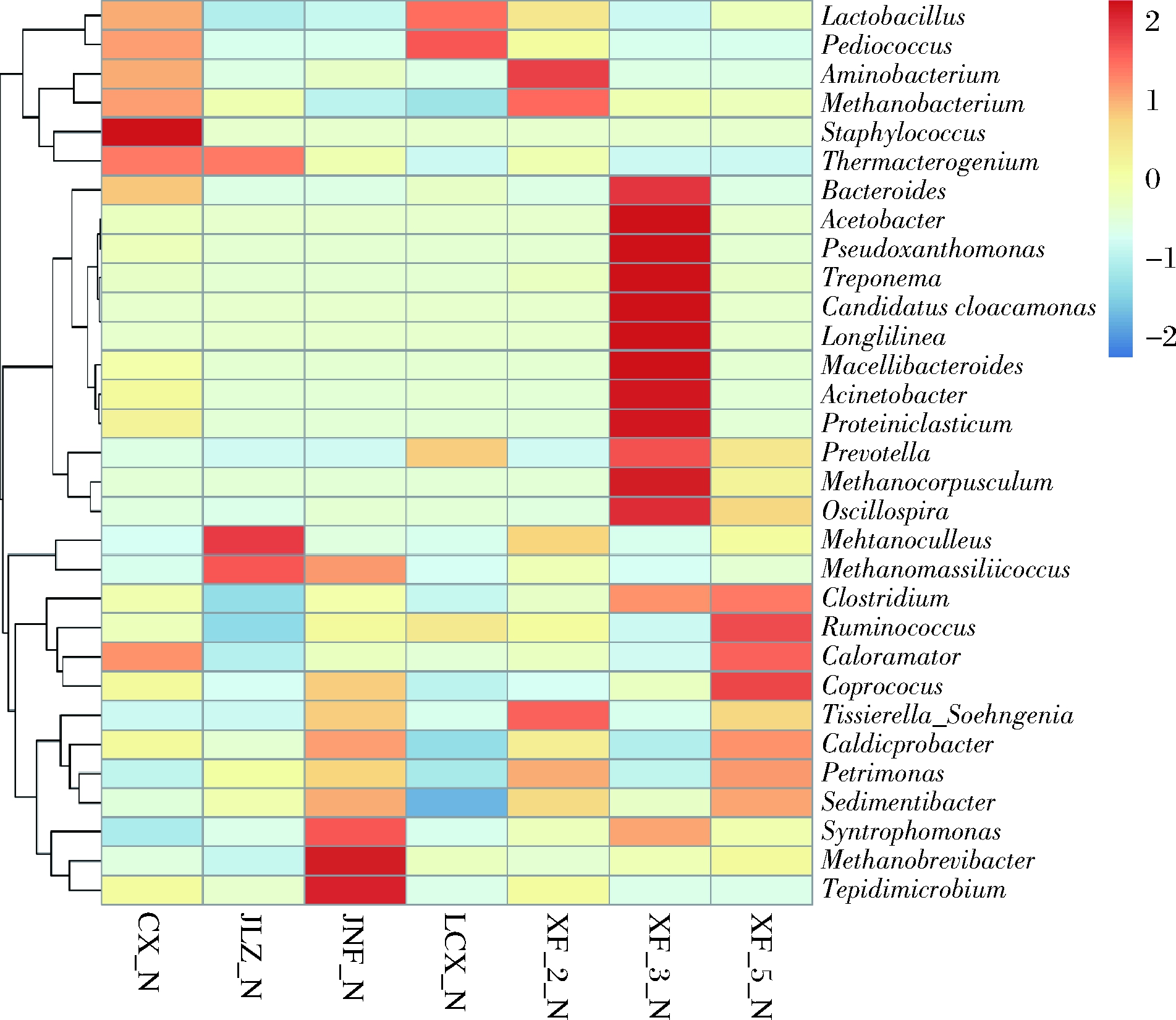
图6 窖泥样品中微生物丰度热图
Fig.6 Heatmap of microbial abundance in pit mud samples
2.8 叙府酒业酿造生境中细菌群落结构分析
分别从叙府酒业3个不同车间(窖龄不同)的窖池中取样,比较其微生物群落结构的异同。分析结果表明,酒醅中主要以片球菌属为主,在二车间和五车间样品中分别达到99.74%和98.89%,而在三车间中片球菌占比87.01%,乳杆菌占比9.42%。而黄水样品中,二车间和五车间主要以乳杆菌属、片球菌属和瘤胃球菌属为主要类群,而三车间样品的微生物多样性较为丰富,丰度高于1%的细菌有10个类群之多。3个车间的窖泥样品的微生物群落差异较大,其中窖龄最长的二车间主要以甲烷杆菌属菌株为主,占比达到35.23%。随着窖泥的发酵,代谢产生的H2含量不断积累,而甲烷菌的存在能够利用H2和CO2生成甲烷,降低窖泥中H2含量而提升窖泥质量,这表明二车间的窖泥已经处于成熟阶段。窖龄最短的五车间主要以产丁酸的瘤胃球菌属为主,占比达到43.44%,这表明五车间的窖泥处于酸代谢活跃时期。窖龄居中的三车间窖泥样品的微生物多样性明显高于其他2个车间,而且占比最高的甲烷粒菌属菌株仅为6.66%,推测该车间窖泥中的微生物类群正处于活跃的演替阶段,物质和能量代谢正在从活跃阶段向稳定阶段转变(表3)。
表3 叙府酒业酿造生境中细菌群落结构比较
Table 3 Comparisons of bacterial community structures in fermented habitats from Xufu

样品*XF-2aXF-3bXF-5c酒醅(J)Pediococcus (99.74%)Pediococcus (87.01%)Lactobacillus (9.42%)Pediococcus (98.89%)窖泥(N)Methanobacterium (35.23%)Aminobacterium 1(3.97%)Methanoculleus (3.82%)Ruminococcus (7.37%)Lactobacillus (6.28%)Petrimonas (5.20%)Pediococcus (3.62%)Syntrophomonas (1.67%)Caldicoprobacter (1.12%)Caloramator (1.01%)Methanocorpusculum (6.66%)Acinetobacter (3.36%)Syntrophomonas (5.06%)Methanobacterium (4.14%)Prevotella (3.84%)Treponema (2.90%)Clostridium (3.60%)Acetobacter (2.84%)Ruminococcus (2.26%)Oscillospira (2.06%)Candidatus (1.18%)Ruminococcus (43.44%)Caloramator (10.64%)Petrimonas (6.74%)Clostridium (4.63%)Methanobacterium (3.65%)Lactobacillus (2.39%)Syntrophomonas (2.11%)Caldicoprobacter (1.98%)Methanobrevibacter (1.49%)Coprococcus (1.46%)Sedimentibacter (1.04%)Prevotella (1.34%)Methanoculleus (1.34%)黄水(H)Pediococcus (81.95%)Lactobacillus (4.78%)Ruminococcus (1.82%)Methanocorpusculum (1.62%)Pediococcus (45.01%)Methanocorpusculum (9.20%)Lactobacillus (4.72%)Acinetobacter (4.15%)Prevotella (3.56%)Chryseobacterium (1.72%)Staphylococcus (1.36%)Acetobacter (1.26%)Syntrophomonas (1.27%)Ruminococcus (1.29%)Pediococcus (86.52%)Lactobacillus (5.52%)Ruminococcus (2.11%)
注:*,属水平上进行细菌群落结构分析,且只显示丰度大于1%的物种。a,窖龄30年;b,窖龄20年;c,窖龄15年。
3 结论
本研究利用高通量测序技术全面解析宜宾产区浓香型白酒酿造生境中的微生物菌群结构,采集的样品均呈现出从多种微生物共同作用到功能微生物主要发挥作用的趋势,片球菌和乳杆菌为酒醅发酵结束期的优势微生物。本研究为后续探究浓香型白酒酿造过程中优势微生物的功能,解析白酒酿造机理,提升白酒品质提供了重要依据。
[1] 康文怀, 徐岩. 中国白酒风味分析及其影响机制的研究[J]. 北京工商大学学报:自然科学版, 2012, 30(3): 53-58.
[2] XU Y, SUN B, FAN G, et al. The brewing process and microbial diversity of strong flavour Chinese spirits: A review[J]. Journal of the Institute of Brewing, 2017, 123(1): 5-12.
[3] GB/T 10781.1—2006 浓香型白酒[S].北京:中国标准出版社, 2007.
[4] 王媚, 傅小红, 冯学愚, 等. 浓香型白酒发酵微生物多样性研究进展与技术创新[J]. 酿酒科技, 2014 (8): 85-88.
[5] 张宿义, 沈才洪, 许德富. 浓香型白酒的技术发展回顾[J]. 酿酒, 2009, 36 (1): 8-10.
[6] ZHANG L, ZHOU R, NIU M, et al. Difference of microbial community stressed in artificial pit muds for Luzhou-flavour liquor brewing revealed by multiphase culture-independent technology[J]. Journal of Applied Microbiology, 2015, 119(5): 1 345-1 356.
[7] SCHIRMER M, IJAZ U Z, D' AMORE R, et al. Insight into biases and sequencing errors for amplicon sequencing with the Illumina MiSeq platform[J]. Nucleic Acids Research, 2015, 43(6): e37.
[8] ALBANESE D, DONATI C. Strain profiling and epidemiology of bacterial species from metagenomic sequencing[J]. Nature Communications, 2017, 8(1): 2 260.
[9] GUO M, HOU C, BIAN M, et al. Characterization of microbial community profiles associated with quality of Chinese strong-flavour liquor through metagenomics[J]. Journal of Applied Microbiology, 2019, 127(3): 750-762.
[10] 李斌, 闫志鹏, 李慧星, 等. 基于高通量测序技术的浓香型和芝麻香型白酒酒曲细菌群落结构分析[J]. 中国酿造, 2018,37(8): 148-152.
[11] 邓杰, 卫春会, 罗惠波, 等. 基于高通量测序的浓香型白酒窖池古菌群落结构分析[J]. 现代食品科技, 2015,31(8): 205-210.
[12] ZHOU J, BRUNS M A, TIEDJE J M. DNA recovery from soils of diverse composition[J]. Appl Environ Microbiol, 1996, 62(2): 316-322.
[13] ZHANG X L, TIAN X Q, MA L Y, et al. Biodiversity of the symbiotic bacteria associated with toxic marine Dinoflagellate Alexandrium tamarense[J]. Journal of Biosciences and Medicines, 2015, 3(6): 23-28
[14] BOLYEN E, RIDEOUT J R, DILLON M R. et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2[J]. Nature Biotechnology, 2019, 37(8): 852-857.
[15] CALLAHAN B J, MCMURDIE P J, ROSEN M J, et al. DADA2: High-resolution sample inference from Illumina amplicon data[J]. Nature Methods, 2016,13(7):581.
[16] LETUNIC I, BORK P. Interactive tree of life (iTOL) v3: An online tool for the display and annotation of phylogenetic and other trees[J]. Nucleic Acids Research, 2016,44(W1):W242-W245.
[17] GINESTET C. ggplot2: Elegant graphics for data analysis[J]. Journal of the Royal Statistical Society, 2011, 174(1):245-246.
[18] CHI C M, CHUNG J W, KOZYRAKIS C, et al. STAMP: Stanford Transactional Applications for Multi-Processing[C]//2008 IEEE International Symposium on workload Characterization.IEEE,2008:35-36.
[19] 冯慧军, 翟磊, 程坤, 等. 高温放线菌属研究进展[J]. 食品与发酵工业, 2017,43(11):257-261.
[20] YAO S, LIU Y, ZHANG M, et al. Thermoactinomyces daqus sp. nov. a thermophilic bacterium isolated from high-temperature Daqu[J]. Int J Syst Evol Microbiol, 2014, 64(1):206-210.
[21] 王明跃, 张文学, 王海英, 等. 不同窖龄窖泥古菌的系统发育多样性分析[J]. 应用与环境生物学报, 2012, 18(6):1 043-1 048.
[22] 邓杰, 卫春会, 边名鸿, 等. 浓香型白酒不同窖龄窖池窖泥中古菌群落结构分析[J]. 食品科学, 2017,38(8):45-50.
[23] 邢敏钰, 杜海, 徐岩. 芝麻香型白酒发酵过程中乳酸菌多样性及其演替规律[J]. 微生物学通报, 2018, 45(1):19-28.