涪陵榨菜采用茎瘤芥(Brassica juncea,俗称青菜头)腌制加工,是世界三大名腌菜之一[1-2]。榨菜加工工艺包括3次腌制和3次压榨:第1次腌制采用菜块质量分数2%~4%的食盐腌制约7 d,第2次加入4%~8%食盐腌制20~30 d,第3次以10%左右食盐用量腌制4~6月,每次腌制结束时都将榨菜起池、压榨去水[3]。
榨菜腌制过程中偶尔出现菜块表面变红,或者表面有一层膜的现象,有时引起菜块变软甚至腐败。目前对引起菜块变红、产膜现象的原因不清楚。本研究采集变红、产膜的菜块样品,分离鉴定相关的微生物,并且用Illumina Miseq测序鉴定原料表面的微生物种类,分析是否来源于青菜头原料。
1 材料与方法
1.1 实验材料
1.1.1 实验原料
用无菌操作采集变红的榨菜样品4个,编号为H1~H4(图1-A),采集有膜的样品5个,编号为M1~M5(图1-B)。

A-变红;B-产膜
图1 榨菜腌制过程中的变红和产膜现象
Fig.1 The picture of the turning red or biofilm-forming phenomenon of Zhacai during the pickling process
1.1.2 实验试剂
酵母与霉菌培养基(yeast maltose,YM)、MRS、营养肉汤(nutrient broth, NB)、平板计数(plate count agar,PCA)培养基,青岛海博生物技术有限公司;细菌基因组DNA提取试剂盒(DP302)、酵母基因组DNA提取试剂盒(DP307)、普通琼脂糖凝胶DNA回收试剂盒(DP209),天根生化科技(北京)有限公司;DNeasy mericon Food Kit (69514),德国Qiagen公司;琼脂糖Regular agarose G-10,西班牙BIOWEST;1×TE缓冲液(B548106-0500)、50×TAE缓冲液(B548101)、PBS缓冲液(pH 7.3~7.5,B640435),生工生物工程(上海)股份有限公司;Premix Taq、RNase-free water、DL2 000 DNA ladder marker,宝生物工程(大连)有限公司。
引物:本研究所用引物如表1所示,委托生工生物工程(上海)股份有限公司合成。
表1 引物序列
Table 1 Sequences of primers

引物引物序列(5'-3')扩增物长度/bp27F1492RAGAGTTTGATCCTGGCTCAGGGYTTACCTTGTTACGACTT1 465 [4]338F806RACTCCTACGGGAGGCAGCAGGGACTACHVGGGTWTCTAAT468 [5]NL1NL4GCATATCAATAAGCGGAGGAAAAGGGTCCGTGTTTCAAGACGG600 [6-7]
1.1.3 仪器与设备
JJ1000 电子天平,常熟市双杰测试仪器厂;303-4B电热恒温培养箱,上海叶拓仪器仪表有限公司;HBM-400D拍击式均质仪,天津市恒奥科技发展有限公司;BX43F 生物显微镜,OLYMPUS;T100 PCR仪、Power pacbasic电泳仪,美国Bio-Rad公司;SW-CJ-2F洁净工作台,苏州安泰空气技术有限公司;G:BOXEF凝胶成像系统,美国gene公司;P100/P100+超微量分光光度计,美国Pultton公司;Mini-Q超级纯水仪,法国Synergy公司。
1.2 实验方法
1.2.1 榨菜变红或产膜相关菌株的分离纯化和形态学鉴定
变红的榨菜样品:取变红部位,加入无菌均质袋中,加入已灭菌50 mL PBS缓冲液,用拍击式均质仪均质1 min,溶液用0.22 μm滤膜过滤,用接种针挑取滤膜上的微生物分别在添加80 g/L NaCl的YM、MRS、NB、PCA培养基上划线分离,于(30±1)℃培养3 d,选出红色菌落的菌株,经过至少连续3次划线分离,得到纯化的菌株,将菌株接种在已灭菌的榨菜中,每株菌接种3份榨菜,另设1组作为空白对照,于(30±1)℃培养,观察是否出现变红现象,如果变红,再从此榨菜上分离纯化得到纯菌株,鉴定其性状是否与接种的菌株相同[8]。
产膜的榨菜样品:用接种针挑取菜块上的膜,分别在添加80 g/L NaCl的YM、MRS、NB、PCA培养基上划线分离,于(30±1)℃培养3 d,经过至少连续3次划线分离,得到纯化的菌株,将菌株逐一接种到适宜的液体培养基中培养48 h,如果在培养液表面形成菌膜,将菌株接种在已灭菌的榨菜中,每株菌接种3份榨菜,另设1组作为空白对照,于(30±1)℃培养,观察是否出现产膜现象,如果产膜,再从此榨菜上分离纯化得到纯菌株,鉴定其性状是否与接种的菌株相同。
将变红和产膜菌株用适当的培养基培养,观察菌落形态,做革兰氏染色,观察显微形态并且拍照。
1.2.2 菌株的分子生物学鉴定
利用天根公司细菌或者酵母基因组DNA提取试剂盒提取分离微生物的基因组DNA。通过PCR扩增细菌16S rDNA序列、酵母菌26S rDNA序列,25 μL PCR反应体系包括2×Premix Taq buffer (TAKARA) 12.5 μL、前后引物(10 μmol/L)各0.8 μL、DNA 2 μL、ddH2O 8.9 μL;扩增程序为:94 ℃预变性3 min,30个循环(94 ℃变性40 s,适宜的温度退火40 s,72 ℃延伸1 min),最后72 ℃延伸10 min。其中细菌的引物为27F和1492R,退火温度为57 ℃[4];真菌的引物为NL1和NL4,退火温度为54 ℃[6-7]。扩增产物用1.2%琼脂糖凝胶电泳,用凝胶成像系统观察条带并且拍照。将PCR产物用琼脂糖凝胶DNA回收试剂盒纯化后,送生工生物(上海)有限公司测序,测序结果通过NCBI BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)比对鉴定,并用MEGA7.0软件进行系统发育分析。
1.2.3 菌株产酶实验
将酪蛋白或者可溶性淀粉以10 g/L的量加入培养基中,接种菌株,(30±1)℃培养48 h,采用平板水解圈法初步检测菌株是否产生蛋白酶或者淀粉酶。
1.2.4 Illumina MiSeq法分析青菜头原料表面微生物的种类
取原料堆中不同部位的青菜头10颗,用灭菌的PBS溶液清洗青菜头表面,洗液用0.22 μm滤膜过滤,用20 mL 灭菌的PBS溶液充分洗下滤膜上的微生物,用DNeasy mericon Food Kit试剂盒按照说明书的步骤提取微生物的宏基因组DNA[9]。用338F和806R引物扩增16S rDNA的V3-V4可变区,PCR反应体系和程序与“1.2.2”相同,退火温度为55 ℃[5]。经过验证的宏基因组DNA送上海美吉生物医药科技有限公司,采用Illumina MiSeq PE300平台对16S rDNA的V3-V4可变区进行高通量测序,测序引物为338F和806R,长度为468 bp[5]。
将获得的序列采用QIIME软件包(quantitative insights into microbial ecology,定量分析微生物群落,version 1.7)[10]进行生物信息分析。采用UCLUST软件在97%的相似度水平下对序列进行聚类分析,获得分类操作单元(operational taxonomic units,OTUs)[11],将序列基于Silva(silva128/16s_bacteria,https://www.arb-silva.de/documentation/release-128/)[12-14]细菌数据库进行比对,分类置信度0.7,得到每个OTU对应的物种分类信息,生成不同分类水平上的物种丰度表。用R绘制样品在属水平下的物种构成图。部分数据分析在上海美吉生物医药科技有限公司的I-Sanger云平台中完成(https://www.i-sanger.com/)。
1.2.5 NCBI序列号
本研究中菌株的16S rDNA序列均上传至NCBI的GenBank,变红相关菌株的序列注册号为MN445590至MN445593,产膜相关菌株的序列注册号为MN445594至MN445606。青菜头原料样品表面微生物的16S rDNA V3-V4序列存入NCBI 的Sequence Read Archive (SRA) 数据库(https://www.ncbi.nlm.nih.gov/sra),BioProject ID为PRJNA565341。
2 结果与分析
2.1 榨菜变红或产膜现象相关菌株的菌落形态和显微形态鉴定
本研究从变红榨菜样品中分离、纯化、验证得到4株引起变红现象的菌株,其菌落形态和显微形态如表2和图2所示。从产膜榨菜样品中鉴定出引起产膜现象的菌株有13株,其菌落形态和显微形态如表2和图3所示,图4为部分菌株产膜验证实验照片。
表2 榨菜变红或产膜现象相关菌株的菌落形态和显微形态
Table 2 The colony morphology and microscopic morphology of strains related to the turning red or biofilm-forming phenomenon of Zhacai during the pickling process
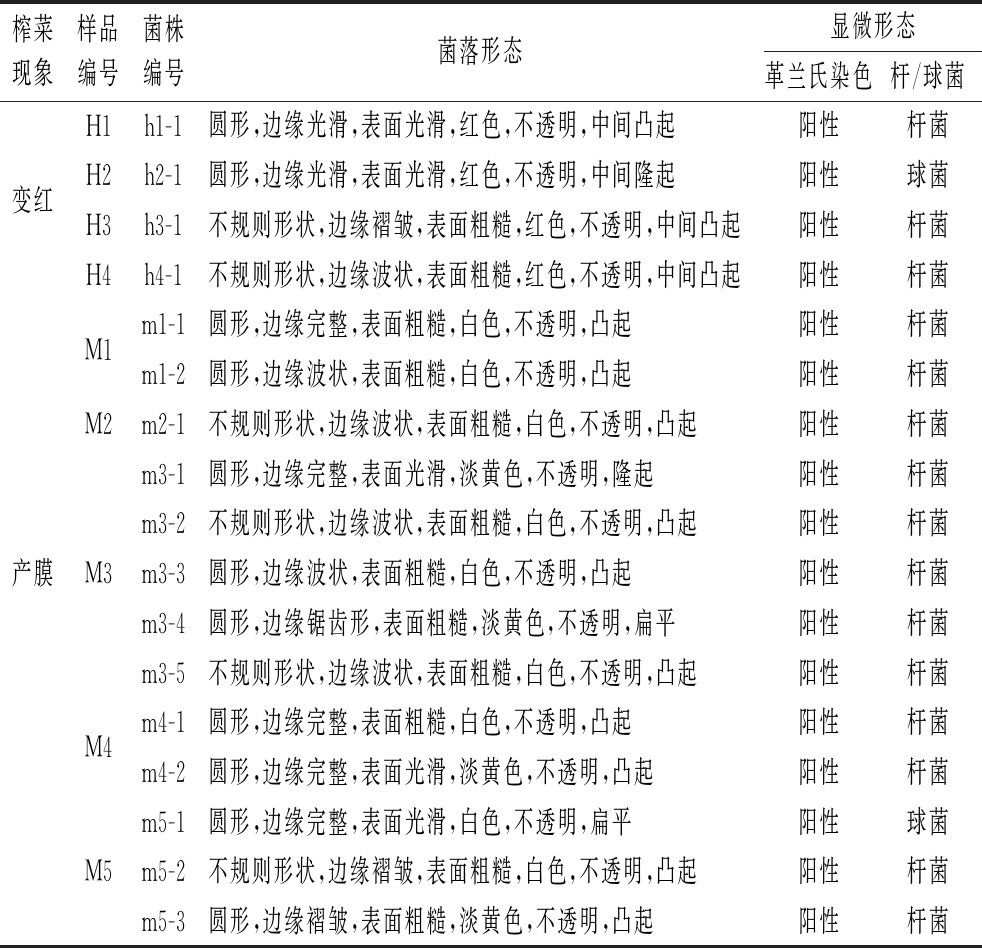
榨菜现象样品编号菌株编号菌落形态显微形态革兰氏染色杆/球菌H1h1-1圆形,边缘光滑,表面光滑,红色,不透明,中间凸起阳性杆菌变红H2h2-1圆形,边缘光滑,表面光滑,红色,不透明,中间隆起阳性球菌H3h3-1不规则形状,边缘褶皱,表面粗糙,红色,不透明,中间凸起阳性杆菌H4h4-1不规则形状,边缘波状,表面粗糙,红色,不透明,中间凸起阳性杆菌M1m1-1圆形,边缘完整,表面粗糙,白色,不透明,凸起阳性杆菌m1-2圆形,边缘波状,表面粗糙,白色,不透明,凸起阳性杆菌M2m2-1不规则形状,边缘波状,表面粗糙,白色,不透明,凸起阳性杆菌m3-1圆形,边缘完整,表面光滑,淡黄色,不透明,隆起阳性杆菌m3-2不规则形状,边缘波状,表面粗糙,白色,不透明,凸起阳性杆菌产膜M3m3-3圆形,边缘波状,表面粗糙,白色,不透明,凸起阳性杆菌m3-4圆形,边缘锯齿形,表面粗糙,淡黄色,不透明,扁平阳性杆菌m3-5不规则形状,边缘波状,表面粗糙,白色,不透明,凸起阳性杆菌M4m4-1圆形,边缘完整,表面粗糙,白色,不透明,凸起阳性杆菌m4-2圆形,边缘完整,表面光滑,淡黄色,不透明,凸起阳性杆菌m5-1圆形,边缘完整,表面光滑,白色,不透明,扁平阳性球菌M5m5-2不规则形状,边缘褶皱,表面粗糙,白色,不透明,凸起阳性杆菌m5-3圆形,边缘褶皱,表面粗糙,淡黄色,不透明,凸起阳性杆菌
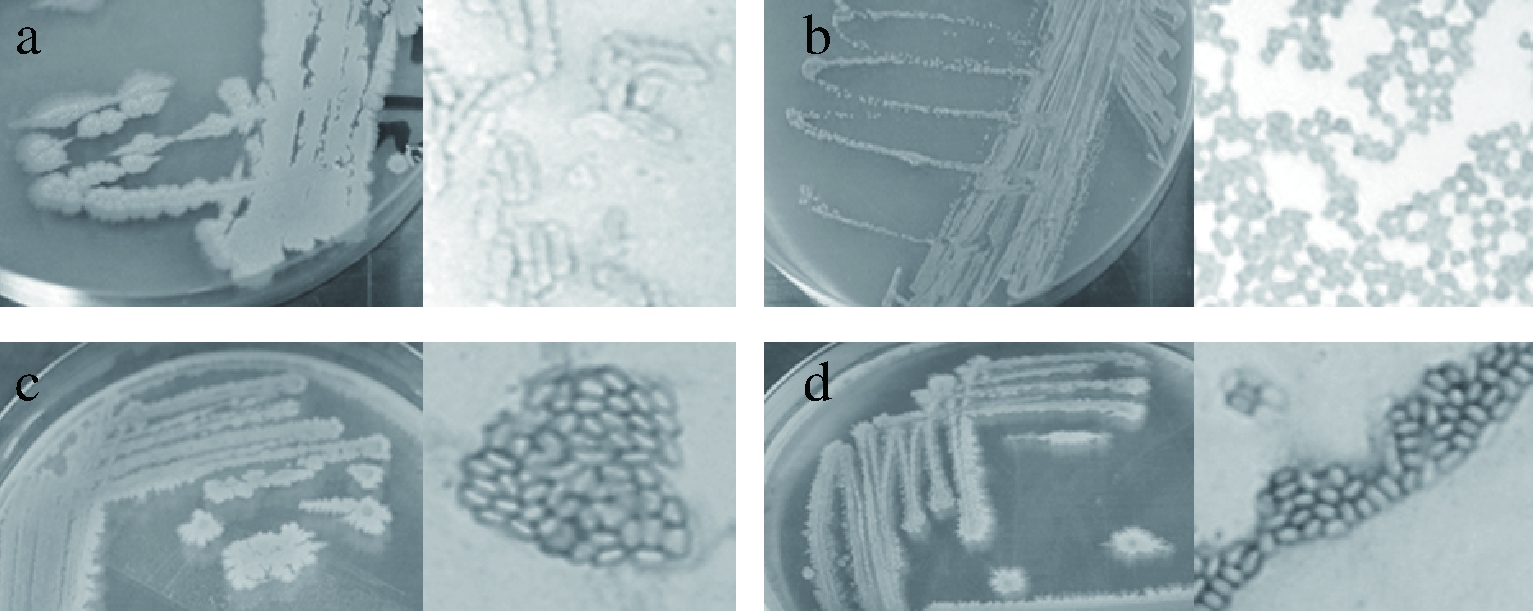
a-h1-1;b-h2-1;c-h3-1;d-h4-1
图2 榨菜变红现象相关菌株的菌落形态和显微形态
Fig.2 The colony morphology and microscopic morphology of strains related to the turning red phenomenon of Zhacai during the pickling process
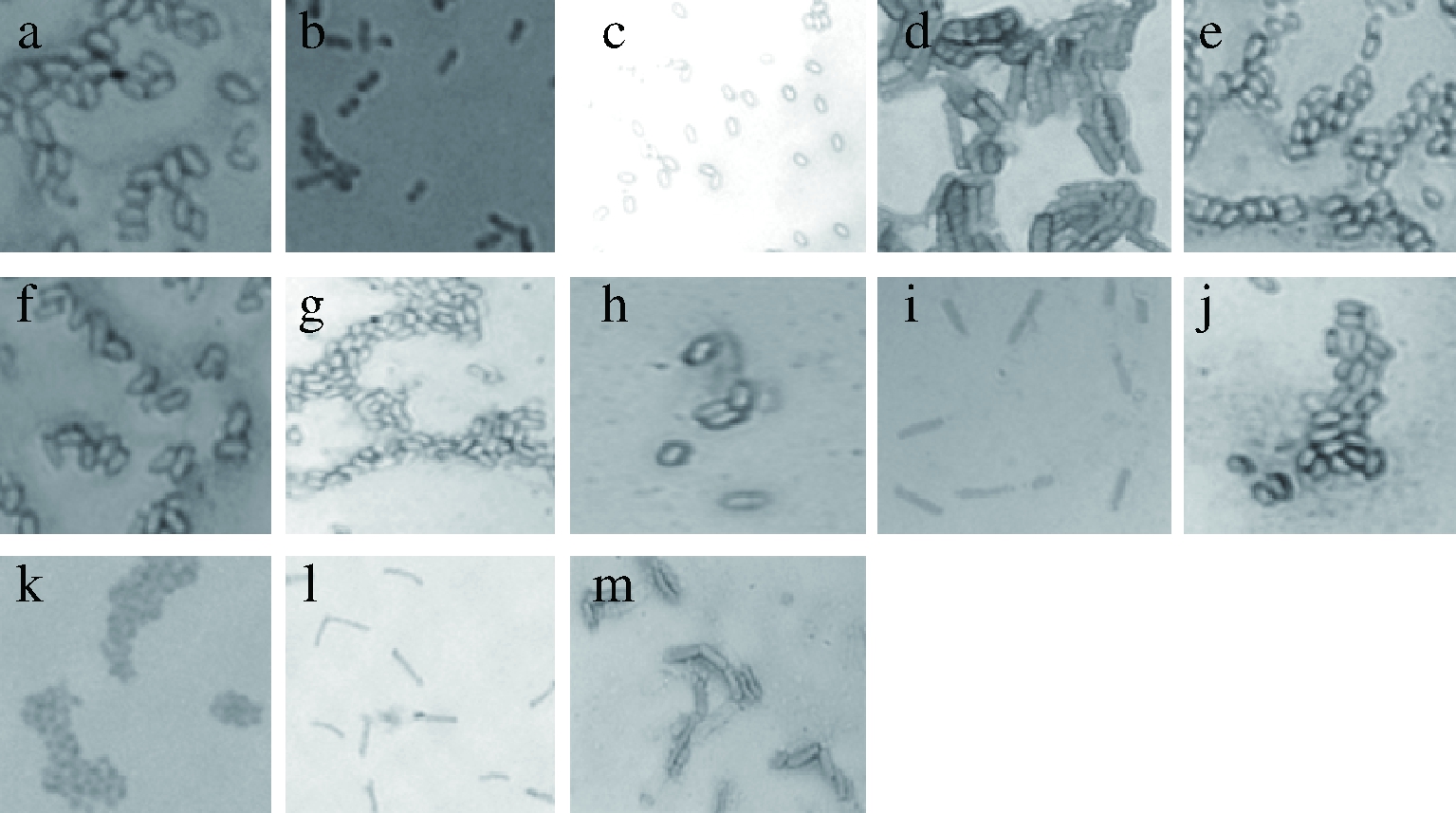
a-m1-1;b-m1-2;c-m2-1;d-m3-1;e-m3-2;f-m3-3;g-m3-4;h-m3-5;i-m4-1;j-m4-2;k-m5-1;l-m5-2,m-m5-3
图3 榨菜产膜现象相关菌株的显微形态
Fig.3 The microscopic morphology of strains related to the biofilm-forming phenomenon of Zhacai during the pickling process

a-m1-1;b-m2-1;c-m3-1;d-m4-1
图4 部分菌株产膜验证实验照片
Fig.4 The pictures of biofilm-forming verification experiment of partial strains
2.2 榨菜变红或产膜现象相关菌株的分子生物学鉴定
4株变红相关菌株和13株产膜菌株的基因组DNA均采用27F和1492R引物PCR扩增成功,用NL1和NL4引物扩增没有条带,所以菌株均为细菌。将菌株的16S rDNA序列输入NCBI blast网页中比对鉴定菌种,其结果如表3所示,菌株m4-1与参考序列的query coverage(覆盖度)为99%,percent identity(序列相似度)为99.48%,综合分析它与参考序列的相似度为98.49%,其余菌株与参考序列的相似度均大于99%。通常认为菌株间16S rDNA基因序列同源性大于97%很可能属于同一个种[15-16],根据NCBI比对结果列出了序列相似性最高的种。
图5为用MEGA7.0软件构建的榨菜变红现象相关菌株16S rDNA序列的进化树图,待测菌株的16S rDNA序列均与相应的参考序列正确聚类,验证了菌种鉴定的合理性。图6为产膜现象相关菌株的系统进化树图,除了Bacillus amyloliquefaciens与Bacillus velezensis的待测菌株和参考序列聚集在同一个分枝之外,其余菌种单独聚为一个分枝,并且待测菌株的序列均与相应的参考序列正确聚类。WANG等的文献报道Bacillus amyloliquefaciens与Bacillus velezensis的16S rDNA序列相似度达到99.7%,在构建的系统进化树中也聚集为一个分支[17],与本研究结果一致。
表3 菌株在NCBI blast网页中比对鉴定结果
Table 3 The identification results of relevant strains by sequence alignment method on the NCBI BLAST webpage
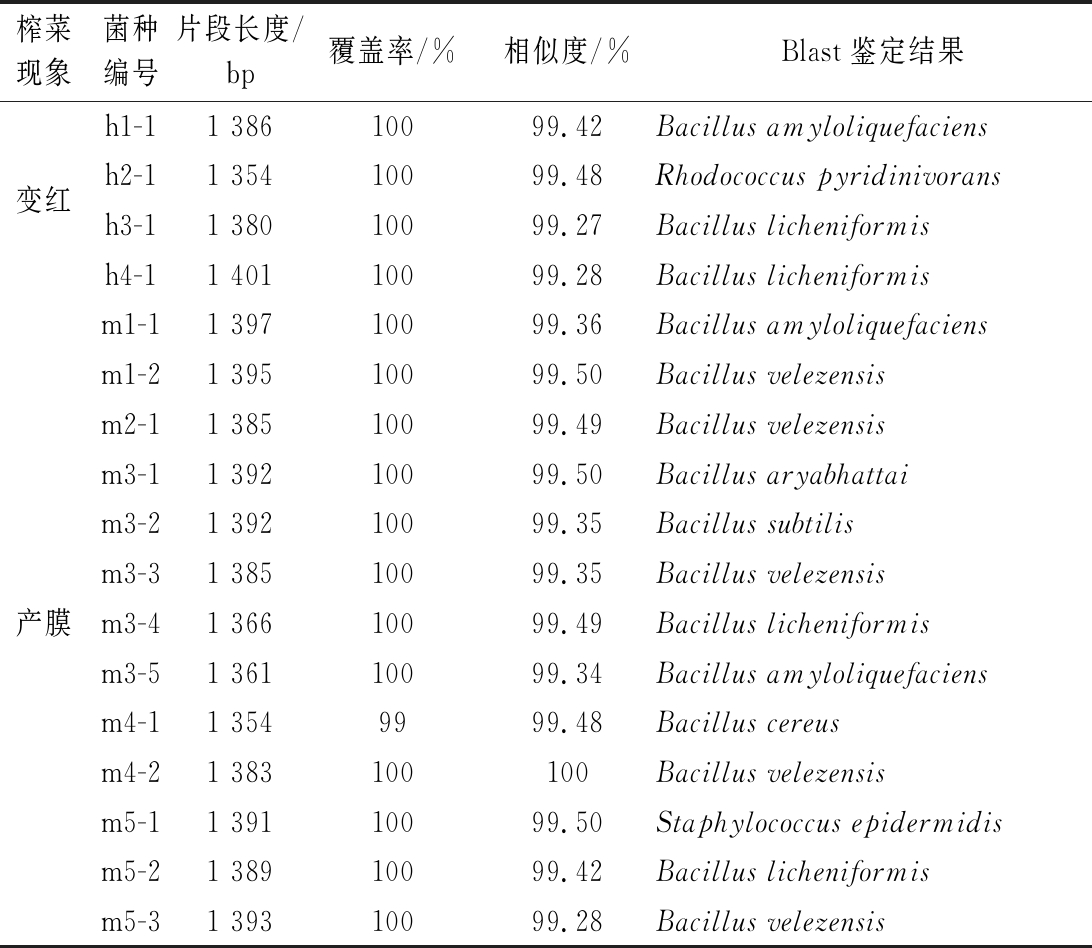
榨菜现象菌种编号片段长度/bp覆盖率/%相似度/%Blast鉴定结果变红h1-11 38610099.42Bacillus amyloliquefaciensh2-11 35410099.48Rhodococcus pyridinivoransh3-11 38010099.27Bacillus licheniformish4-11 40110099.28Bacillus licheniformis产膜m1-11 39710099.36Bacillus amyloliquefaciensm1-21 39510099.50Bacillus velezensism2-11 38510099.49Bacillus velezensism3-11 39210099.50Bacillus aryabhattaim3-21 39210099.35Bacillus subtilism3-31 38510099.35Bacillus velezensism3-41 36610099.49Bacillus licheniformism3-51 36110099.34Bacillus amyloliquefaciensm4-11 3549999.48Bacillus cereusm4-21 383100100Bacillus velezensism5-11 39110099.50Staphylococcus epidermidism5-21 38910099.42Bacillus licheniformism5-31 39310099.28Bacillus velezensis

图5 榨菜变红现象相关菌株的16S rDNA序列进化树图
Fig.5 The phylogenetic tree of 16S rDNA sequences of strains related to the turning red phenomenon of Zhacai
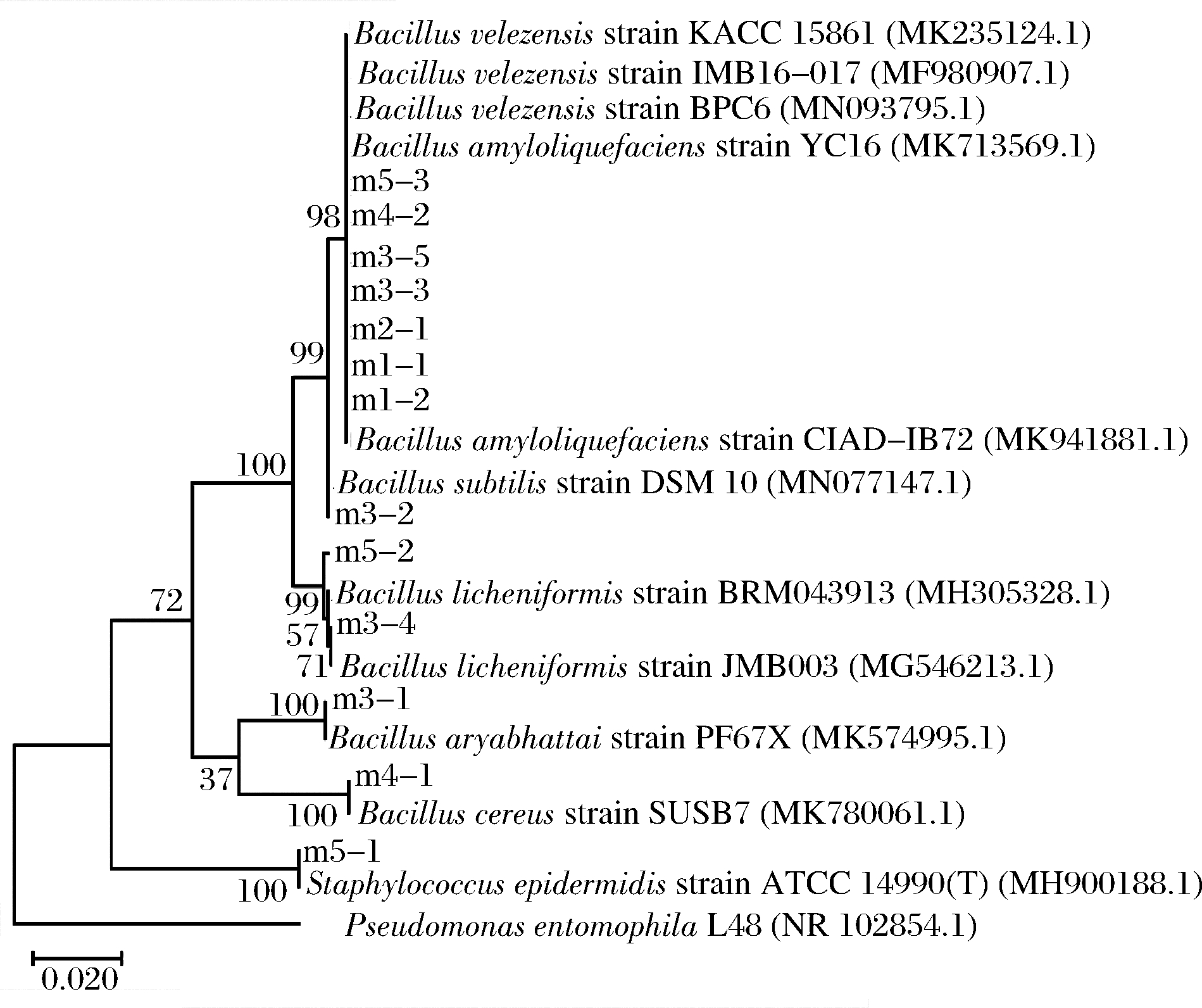
图6 榨菜产膜现象相关菌株的16S rDNA序列进化树图
Fig.6 The phylogenetic tree of 16S rDNA sequences of strains related to the biofilm-forming phenomenon of Zhacai
2.3 菌株产酶活性
能够产生蛋白酶(即水解酪蛋白)的菌株包括h1-1、h3-1、m1-1、m1-2、m2-1、m3-1、m3-3、m3-4、m4-1、m4-2、m5-2、m5-3。能够产生淀粉酶(即水解可溶性淀粉)的菌株包括h1-1、h3-1、h4-1、m1-1、m2-1、m3-1、m3-2、m3-4、m3-5、m4-2、m5-2、m5-3。部分菌株的产酶实验如图7所示。
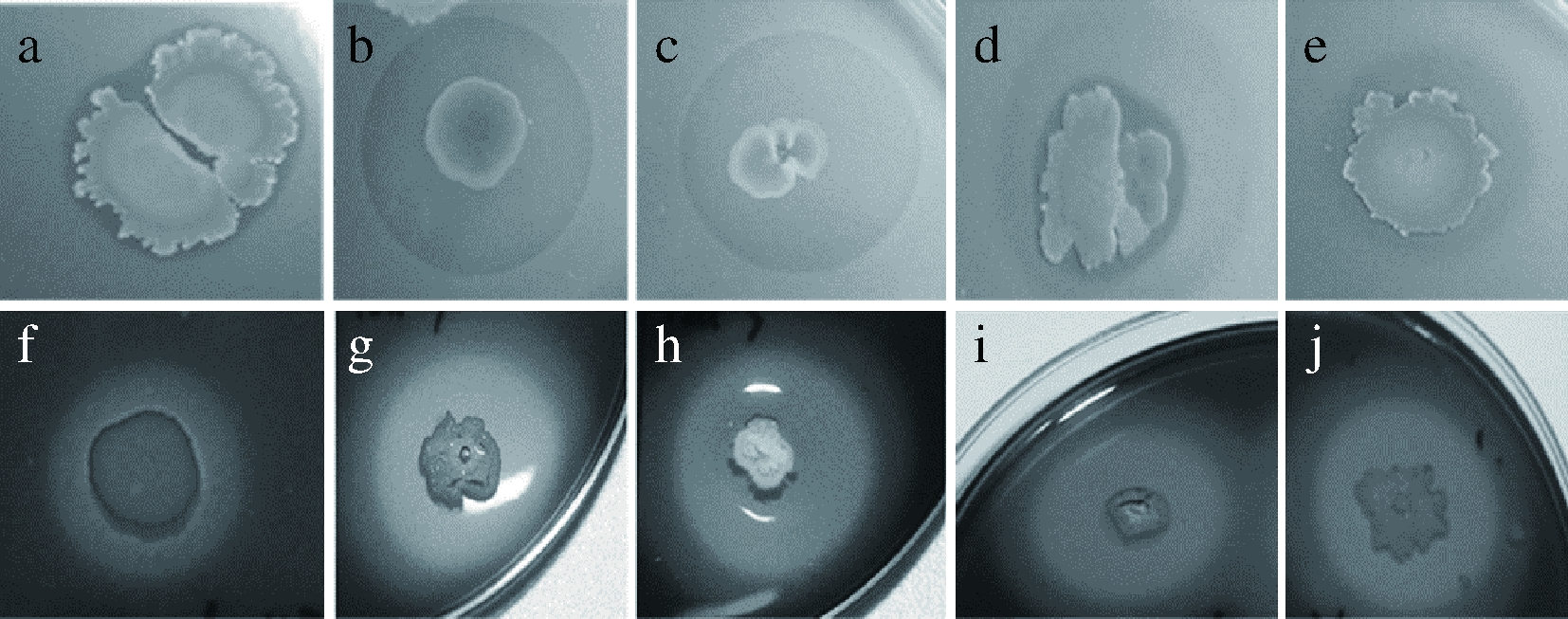
a~e-m3-1、m1-2、m3-1、m4-1、m5-2(产蛋白酶实验);f~j-h1-1、m2-1、m3-2、m4-2、m5-2(产淀粉酶实验)
图7 部分菌株产蛋白酶和淀粉酶的实验照片
Fig.7 The partial pictures of protease- and amylase-producing activity screening experiments of strains
2.4 青菜头原料表面微生物种类鉴定
采用Illumina Miseq对青菜头原料表面微生物DNA的16S rDNA基因V3-V4区段进行高通量测序,鉴定细菌的种类构成,总共产生了53 624条raw reads, 平均序列长度为447.58 bp,Coverage 1.000,测序深度满足生物信息学分析要求。鉴定出OTU总数为173,门、纲、目、科、属、种数量分别为:7、12、29、52、91、132。最优势的属为Pseudomonas (62.54%),其他优势属(相对丰度>1%的)还包括:unclassified_f_Enterobacteriaceae (6.34%), norank_c_Cyanobacteria (6.34%),Erwinia(3.80%),Acinetobacter (2.79%),Janthinobacterium(2.70%),Duganella (2.67%),Chryseobacterium (2.61%),Flavobacterium (2.34%)(见图8)。从青菜头原料中没有鉴定出芽孢杆菌属(Bacillus),然而细菌群落中存在很低丰度的Rhodococcus(0.184 8%)和Staphylococcus (0.026 4%),没有鉴定到种的水平,如表4所示。由此可初步推测,引起变红现象的嗜吡啶红球菌(Rhodococcus pyridinivorans)和引起产膜现象的表皮葡萄球菌(Staphylococcus epidermidis)有可能来源于青菜头原料。
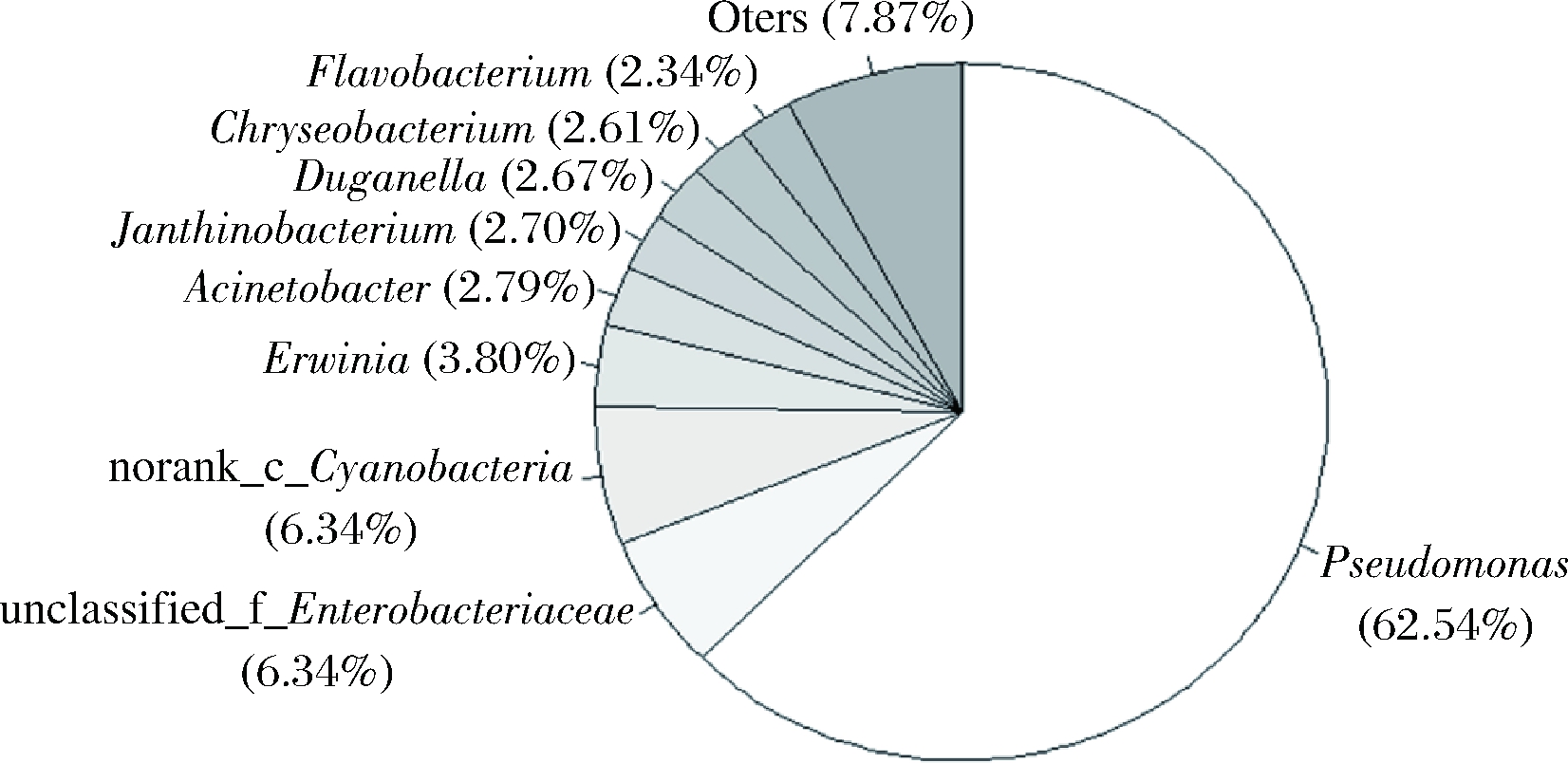
图8 青菜头原料表面细菌在属水平的种类构成图
Fig.8 The composition profile of surface bacteria of raw mustard tuber at the genus level
表4 从青菜头原料中鉴定出的Staphylococcus和Rhodococcus属信息统计
Table 4 The information statistics of the genus Staphylococcus and Rhodococcus identified from raw mustard tuber
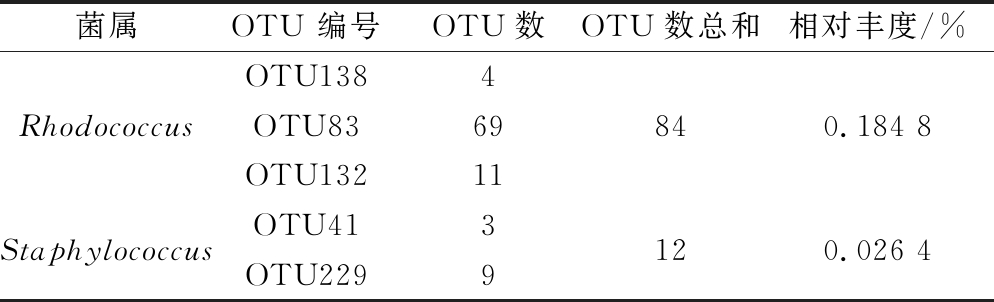
菌属OTU 编号OTU数OTU数总和相对丰度/%OTU1384RhodococcusOTU8369840.184 8OTU13211StaphylococcusOTU413120.026 4OTU2299
3 讨论
本研究鉴定出引起榨菜变红的菌种包括嗜吡啶红球菌(Rhodococcus pyridinivorans)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)、地衣芽孢杆菌(Bacillus licheniformis)3种。YOON等[18]将1株降解吡啶的菌株鉴定为嗜吡啶红球菌,革兰氏阳性球菌,菌落为橙色,与本研究中分离鉴定的嗜吡啶红球菌形态一致。郑晓冬等分析得出红球菌色素是类胡萝卜素,非单一色素[19]。TAKAICHI等的文献也指出红球菌属的色素为类胡萝卜素[20]。由此可推测,嗜吡啶红球菌可能因为产类胡萝卜素而使菌落呈橙(红)色。目前比较少文献报道Bacillus amyloliquefaciens、Bacillus licheniformis的菌落为红色。
本研究鉴定出产膜菌株种类包括:解淀粉芽孢杆菌 (Bacillus amyloliquefaciens)、贝莱斯芽孢杆菌 (Bacillus velezensis)、阿耶波多氏芽孢杆菌 (Bacillus aryabhattai)、枯草芽孢杆菌(Bacillus subtilis)、地衣芽胞杆菌(Bacillus licheniformis)、蜡状芽孢杆菌(Bacillus cereus)和表皮葡萄球菌(Staphylococcus epidermidis)。现有的文献EP映证了这7种菌都能形成生物膜[21-27]。芽孢杆菌属的菌体在温度、pH值、营养、或者菌体自身产生的胞外DNA、信号肽等条件的诱导下,吸附在固体表面,繁殖形成微菌落,菌体进一步生长并分泌大量的胞外多聚物(多糖、蛋白质或者核酸),将菌体细胞包被并聚集在一起,形成成熟的生物膜,菌体的运动功能相关基因表达下调,失去运动能力,以维持生物膜的稳定性。生物膜的形成有利于增强菌体对不良环境的耐受性[23-26]。表皮葡萄球菌形成生物膜主要经过2个阶段:菌体首先在蛋白质AtIE(具有黏附玻连蛋白活性)和Fbe(可能与宿主的纤维蛋白原结合)的帮助下黏附于固体表面,然后在PIA和AAP蛋白介导下相互聚集,形成成熟的生物膜[27]。在产膜相关菌种中,蜡状芽孢杆菌和表皮葡萄球菌是机会病原菌,蜡状芽孢杆菌的部分菌株产生毒素,引起腹泻或呕吐[28];表皮葡萄球菌是医院感染(伤口或者血流感染)的重要菌种之一[29]。
变红和产膜相关菌株大多产生淀粉酶或蛋白酶。涪陵青菜头原料的蛋白质含量高达5 800 mg/500 g,总糖含量为4 300 mg/500 g,菌株可能通过产生酶降解原料中的蛋白质或糖类成分引起榨菜腐败。
本研究采用高通量测序法分析了青菜头表面细菌的种类构成,结果显示葡萄球菌属(Staphylococcus)和红球菌属(Rhodococcus)以很低的丰度存在,没有发现芽孢杆菌属。由此推测,引起变红现象的嗜吡啶红球菌(Rhodococcus pyridinivorans)和引起产膜现象的表皮葡萄球菌(Staphylococcus epidermidis)有可能来源于青菜头原料,芽孢杆菌属菌株的来源有待进一步分析。
4 结论
本研究鉴定出与榨菜变红现象相关的菌株有4株,包括3个菌种:嗜吡啶红球菌(Rhodococcus pyridinivorans)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)、地衣芽孢杆菌(Bacillus licheniformis)。
本研究鉴定出引起榨菜产膜的菌株有13株,包括7个菌种:解淀粉芽孢杆菌 (Bacillus amyloliquefaciens)、贝莱斯芽孢杆菌 (Bacillus velezensis)、阿耶波多氏芽孢杆菌 (Bacillus aryabhattai)、枯草芽孢杆菌(Bacillus subtilis)、地衣芽胞杆菌(Bacillus licheniformis)、蜡状芽孢杆菌(Bacillus cereus)和表皮葡萄球菌(Staphylococcus epidermidis)。
嗜吡啶红球菌(Rhodococcus pyridinivorans)和表皮葡萄球菌(Staphylococcus epidermidis)有可能来源于青菜头原料。
[1] 庞杰,向珣.涪陵榨菜的加工及生产现状[J].长江蔬菜, 1999(2): 42-44.
[2] 苏扬,张聪,王朝辉.榨菜加工工业及其发展战略的研究[J].食品科技,2010,35(4):114-119.
[3] 贺云川,周斌全,刘德君.涪陵榨菜传统工艺概述[J].食品与发酵科技, 2013,49(4):57-60.
[4] 邓维琴,伍亚龙,陈功,等.多代发酵泡菜母水微生物多样性及其理化指标动态研究[J].食品与发酵工业, 2018,44(8):49-56.
[5] XU Nan,TAN Guangcai,WANG Hongyuan,et al.Effect of biochar additions to soil on nitrogen leaching, microbial biomass and bacterial community structure[J].European Journal of Soil Biology,2016,74:1-8.
[6] 何志刚,李维新,梁璋成,等.优良降酸酿酒酵母的分离和鉴定[J].中国食品学报,2013,13(5):187-193.
[7] 王珍珍,沙如意,蔡成岗,等.树莓酵素中耐高渗酵母菌的分离鉴定及生长特性研究[J].食品工业科技,2017, 38(8):178-182;188.
[8] 吴丹,陈健初,叶兴乾,等.榨菜腐败微生物的分离、鉴定及生物学特性研究[J].浙江大学学报(农业与生命科学版),2009,35(2):135-140.
[9] YANG Jixia,CAO Jialu,XU Haiyan,et al.Bacterial diversity and community structure in Chongqing radish paocai brines revealed using PacBio SMRT sequencing technology[J].Journal of the Science of Food and Agriculture,2018, 98(9):3 234-3 245.
[10] CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7(5):335-336.
[11] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26 (19):2 460-2 461.
[12] QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools[J].Nucleic Acids Research,2013,41(D1):D590-D596.
[13] YILMAZ P,PARFREY L W,YARZA P,et al. The SILVA and "all-species living tree project (LTP)" taxonomic frameworks[J].Nucleic Acids Research,2014,42(D1):D643-D648.
[14] LU Yingying,CHEN Jing,ZHENG Junyuan,et al.Mucosal adherent bacterial dysbiosis in patients with colorectal adenomas[J].Scientific reports,2016,6:26 337.
[15] 程池,刘光全,李金霞,等.55株芽孢杆菌16S rRNA基因序列测定与系统发育学分析[J].食品与发酵工业, 2006,32(10):20-24.
[16] STACKEBRANDT E,GOEBEL B M.Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J]. International Journal of Systematic Bacteriology,1994,44(4): 842-849.
[17] WANG L T,LEE F L,TAI C J,et al.Bacillus velezensis is a later heterotypic synonym of Bacillus amyloliquefaciens[J].International Journal of Systematic and Evolutionary Microbiology,2008,58(3):671-675.
[18] YOON J H,KANG S S,CHO Y G,et al.Rhodococcus pyridinivorans sp. nov. a pyridine-degrading bacterium[J]. International Journal of Systematic and Evolutionary Microbiology,2000,50(6):2 173-2 180.
[19] 郑晓冬,王友永.用红球菌生产类胡萝卜素的研究[J].浙江大学学报(农业与生命科学版),2000,26(5):516-520.
[20] TAKAICHI S,MAOKA T,AKIMOTO N,et al.Carotenoids in a corynebacterineae, Gordonia terrae AIST-1: Carotenoid glucosyl mycoloyl esters[J].Bioscience, Biotechnology and Biochemistry,2008,72(10):2 615-2 622.
[21] 刘振宇,曹阳,邓德法,等.解淀粉芽孢杆菌生物膜特征与生防活性[C].中国植物病理学会2012年学术年会论文集,2012:362.
[22] 蔡高磊,张凡,欧阳友香,等.贝莱斯芽孢杆菌(Bacillus velezensis)研究进展[J].北方园艺,2018(12):162-167.
[23] HONG B H,JOE M M,SELVAKUMAR G,et al.Influence of salinity variations on exocellular polysaccharide production, biofilm formation and flocculation in halotolerant bacteria[J].Journal of Environmental Biology,2017, 38(4):657-664.
[24] 刘伟杰,刘聪,蒋继宏.枯草芽孢杆菌形成生物被膜的研究进展[J].微生物学报,2014,54(9):977-983.
[25] RANDRIANJATOVO-GBALOU I,ROUQUETTE P,LEFEBVRE D,et al.In situ analysis of Bacillus licheniformis biofilms: Amyloid-like polymers and eDNA are involved in the adherence and aggregation of the extracellular matrix[J].Journal of Applied Microbiology,2017,122(5):1 262-1 274.
[26] MAJED R,FAILLE C,KALLASSY M,et al.Bacillus cereus biofilms-same, only different[J].Frontiers in Microbiology,2016,7:1 054.
[27] 李燕,李冬冬,陶传敏,等.表皮葡萄球菌生物膜形成及相关基因的检测及评价[J].中华医院感染学杂志, 2010,20(4):473-476.
[28] GRANUM P E,LUND T.Bacillus cereus and its food poisoning toxins[J].FEMS Microbiology Letters,1997, 157(2):223-228.
[29] LOWY F D,HAMMER S M.Staphylococcus epidermidis infections[J].Annals of Internal Medicine,1983,99(6): 834-839.