黄曲霉(Aspergillus flavus)是一种腐生型好氧真菌,它易污染玉米、花生、棉花、小麦等农作物,影响其产量及品质[1]。同烟曲霉一样,黄曲霉感染也能引起人鼻窦炎、脓毒性关节炎和系统性感染等一系列侵袭性疾病[2-3]。某些黄曲霉菌株还产生黄曲霉毒素(aflatoxin,AF),它主要包括AFB1、AFB2、AFG1和AFG2 4种类型,其中AFB1的毒性最强,具有致癌和致突变作用[1]。若奶牛摄入AFB1,1%~2%AFB1在奶牛体内会转化为AFM1,且AFM1主要通过牛奶排出[4]。牛奶和奶制品存在AFM1同样危害人类的健康,因此AFB1和AFM1被世界卫生组织国际癌症研究机构分别列为1类和2B类致癌物[5]。全面阐明黄曲霉致病及产毒分子机制,对控制黄曲霉感染和减轻黄曲霉毒素污染农产品均具有重要理论和实际意义。
黄曲霉毒素生物合成途径的分子调控机制已研究得较为清楚[6]。除了温度、培养基、水分活度、碳源、产毒和不产毒黄曲霉菌株双重培养等外部因素[7-10],黄曲霉毒素的生物合成还受到特异性转录因子和全局性转录因子的调控。AflR是调控黄曲霉毒素生物合成的特异性正调控因子,负责激活黄曲霉毒素生物合成基因簇的大部分基因表达[11-12]。近年来,越来越多的全局性转录因子已被证实参与调控黄曲霉毒素的生物合成,如LaeA[13]、LaeA同源蛋白Lael1[14]、LaeB[15]、Rum1[16]、mtfA[17]、Homeobox转录因子htf2[18]和HexA[19]。由此可见,黄曲霉毒素生物合成是一个多层次、多维度复杂的调控系统,可能涉及更多未知转录因子的调控。
2017年,OAKLEY等[20]首次报道一个新的全局转录因子McrA,并证实其参与调控构巢曲霉杂色曲霉素的生物合成,而杂色曲霉素是合成黄曲霉毒素最重要的前体物质。黑曲霉McrA对某些次级代谢产物合成起到正调控作用,而对某些次级代谢产物合成表现为负调控作用[21]。黄曲霉基因组存在McrA的同源基因,McrA是否参与黄曲霉毒素生物合成的调控目前尚不清楚。因此,本研究通过反转录聚合酶链式反应(reverse transcription-polymerase chain reaction,RT-PCR)克隆黄曲霉AfMcrA,并对其编码蛋白的基本特征、保守结构域、亚细胞定位、二级结构和功能进行预测分析,旨在为将来深入探究黄曲霉AfMcrA的生物学功能奠定理论基础。
1 材料与方法
1.1 菌种材料
黄曲霉(Aspergillus flavus),廊坊师范学院生命科学学院史振霞副教授馈赠,该菌株从河北廊坊九州具有典型褐腐病的鸭梨果实中分离;感受态细胞Escherichia coli Trans10,北京全式金生物技术有限公司。
1.2 实验试剂
Omega真菌DNA提取试剂盒,上海索莱宝生物科技有限公司;真菌RNA提取试剂盒、PCR产物纯化试剂盒、DNA胶回收试剂盒、氨苄青霉素钠,生工生物工程(上海)股份有限公司;质粒小量提取试剂盒、DNA Marker,天根生化科技(北京)有限公司;PCR扩增试剂盒、pTOPO-Blunt平末端克隆试剂盒、cDNA第一链合成试剂盒、实时荧光定量PCR试剂盒,北京艾德莱生物科技有限公司。
1.3 实验方法
1.3.1 黄曲霉RNA的提取及cDNA的合成
采用真菌RNA提取试剂盒分离黄曲霉总RNA。1.0%琼脂糖凝胶电泳检测RNA完整性,超微量分光光度计测定RNA的浓度和纯度。将高质量的RNA反转录合成cDNA第1链,其反应体系为5×TRUE Reaction Mix 4 μL,Oligo(dT) 1 μL,总RNA 400 ng,补RNase-free H2O至20 μL。反应程序为42 ℃、20 min; 85 ℃、5 s。
1.3.2 AfMcrA的克隆
根据黄曲霉AfMcrA的cDNA序列(Accession No. XM_002380888.1),设计并合成一对特异性引物McrAF和McrAR,如表1所示,PCR扩增AfMcrA基因。其反应体系如下:2×A8 PCR Master Mix 12.5 μL,cDNA 1 μL,McrAF和McrAR(10 μmol/L)各0.5 μL,补加ddH2O至25 μL。PCR程序为94 ℃、4 min;94 ℃、30 s,60 ℃、20 s,72 ℃、45 s,30 个循环;72 ℃最后延伸10 min。纯化的PCR产物直接克隆到平末端克隆载体pTOPO-Blunt,转化Escherichia coli Trans10感受态细胞。将菌落PCR鉴定的5个阳性转化子送上海生工进行测序,测序引物为载体通用引物M13F和M13R。
表1 本研究所有引物及序列
Table 1 Primers used in the study

引物名称序列McrAF5'-ATGTCCAGCAACCCCCACGATTTC-3'McrAR5'-TTACTGATTCAAGCCCCGCCGGTT-3'18S rRNAF5'-GCTCTTTTGGGTCTCGTAATTGG-3'18S rRNAR5'-TTACTGATTCAAGCCCCGCCGGTT-3'McrARTF5'-GACGGAAGATTAGGTGCTCG-3'McrARTR5'-GTTTCTCAAGTGTGGGTAGGC-3'
1.3.3 AfMcrA的外显子和内含子分析
采用真菌DNA提取试剂盒分离黄曲霉基因组DNA。以DNA为模板,McrAF和McrAR为特异引物进行PCR扩增。PCR产物经克隆测序获得AfMcrA的基因组DNA序列(gDNA)。采用DNAMAN软件对AfMcrA的gDNA和cDNA序列进行比对,以确定AfMcrA的内含子和外显子构成。
1.3.4 AfMcrA的生物信息学分析
利用一系列在线工具对AfMcrA的理化性质、亲疏水性、信号肽、跨膜结构域、亚细胞定位、保守结构域、二级结构等进行预测(表2)。DNAMAN对不同来源McrA进行多重序列比对。Mega 5.1构建系统发育NJ树(Bootstrap method并设定Bootstrap值为1 000)。
表2 AfMcrA的生物信息学预测项目及在线工具
Table 2 Items predicted and online tools

预测项目在线工具及网址理化性质ProtParam http://web.expasy.org/protparam/信号肽SignaIP 4.1 Server http://www.cbs.dtu. dk/serv-ices/SignalP/跨膜结构域TMpred https://embnet.vital-it.ch/software/TM-PRED_form.html亲疏水性Protscale http://web.expasy.org/protscale/磷酸化位点NetPhos 3.1 http://www.cbs.dtu.dk/services/Net-Phos/蛋白无序特征FoldIndex http://bioportal.weizmann.acil/fldbin /fin-dex保守结构域SMART http://smart.embl-heidelberg.de/smart/set_mode.cgi?NORMAL=1二级结构SOPMA https://npsa-prabi.ibcp.fr/cgi-bin/npsa_au-tomat.pl?page=npsa_sopma.htm
1.3.5 AfMcrA的表达分析
采用直径为5 mm的打孔器在黄曲霉菌落边缘打取菌饼,并等量接种到PD培养基中,28 ℃分别振荡培养2、4、6、8 d。分别提取不同培养时间黄曲霉总RNA,反转录合成cDNA第1链,-20 ℃保存备用。
采用实时荧光定量聚合酶链式反应(quantitative reverse transcription-polymerase chain reaction, qRT-PCR)分析不同培养时间AfMcrA的相对表达量。反应体系为20 μL,其中2× SYBR Green qPCR Mix 10 μL,cDNA模板1 μL,正向和反向引物(5 μmol/L)各1 μL。PCR程序为94 ℃、 2 min; 94 ℃、 10 s, 60 ℃、 30 s, 40个循环。AfMcrA及内参基因18S rRNA[22]的引物序列见表1。每个样本设置3个重复,反应结束后绘制熔解曲线。采用2-ΔΔCt计算AfMcrA的相对表达量,用Excel和SPSS进行柱形图绘制和差异显著性分析。
2 结果与分析
2.1 AfMcrA的克隆
1%琼脂糖凝胶电泳检测黄曲霉RNA的完整性,28S条带亮度约为18S的2倍(图1-a),表明所提取RNA的质量较好。以RNA反转录获得的cDNA为模板,McrAF和McrAR为特异引物,PCR扩增获得1条清晰、明亮条带,其大小与AfMcrA的理论值基本一致(图1-b)。经克隆测序,获得了黄曲霉AfMcrA的cDNA序列,全长1 101 bp。
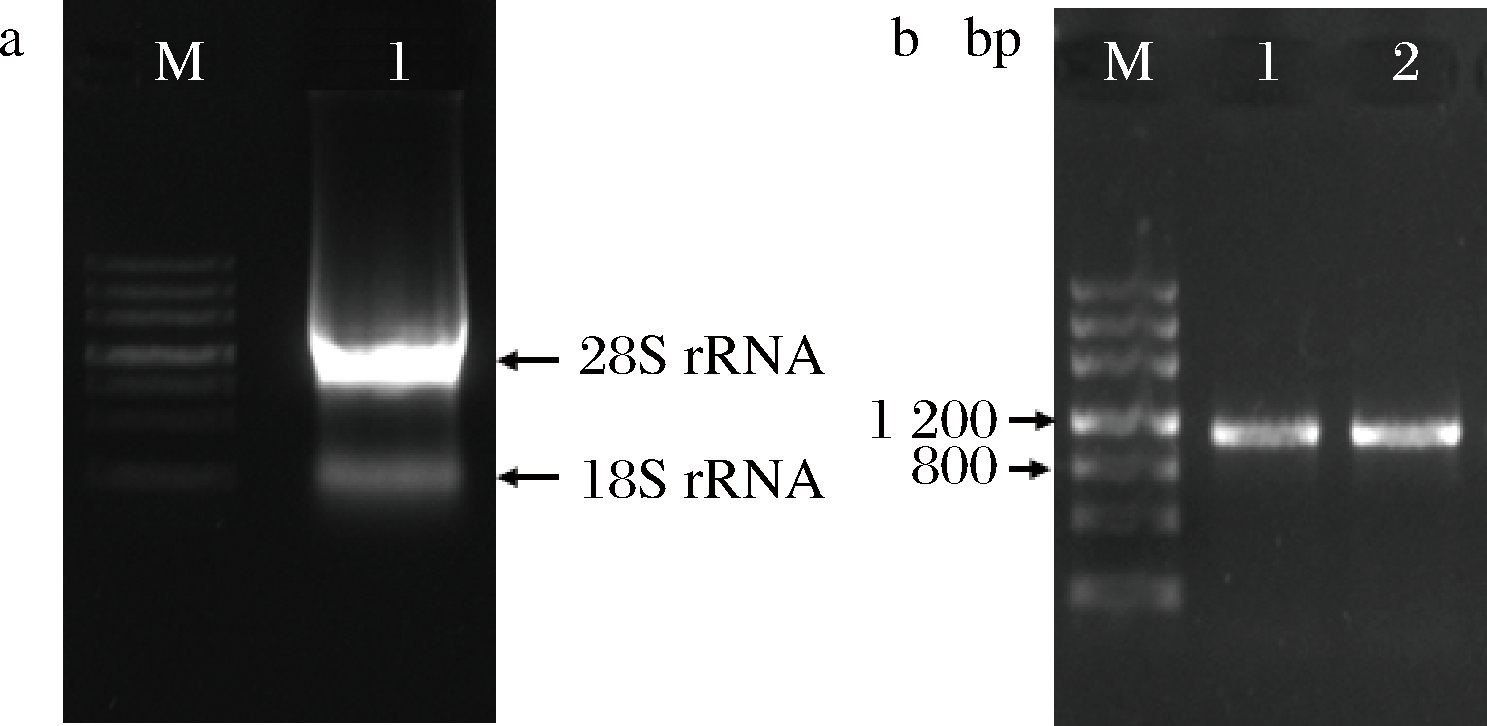
泳道M-DNA Marker Ⅲ;泳道a1-黄曲霉总RNA;
泳道b1~b2-AfMcrA的cDNA序列扩增产物
a-黄曲霉RNA;b-黄曲霉AfMcrA基因的PCR扩增
图1 黄曲霉RNA和AfMcrA的cDNA序列扩增
Fig.1 A. flavus RNA and amplification of AfMcrA cDNA fragment
2.2 AfMcrA基因的内含子和外显子分析
以黄曲霉gDNA为模板,McrAF和McrAR为特异引物,PCR扩增出1条1 200~2 000 bp的条带(图2)。克隆测序获得AfMcrA的gDNA序列为1 448 bp。采用DNAMAN软件对AfMcrA的cDNA和gDNA序列进行比对,发现AfMcrA由3个内含子和4个外显子组成,且内含子的剪切位点均符合真核生物的5′-GT和3′-AG原则(图3)。
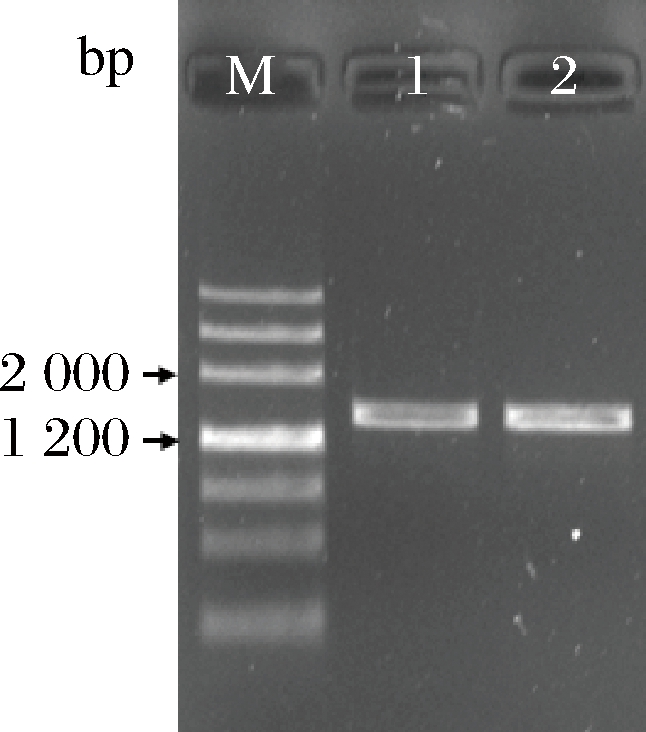
泳道M-DNA Marker Ⅲ;泳道1~2-AfMcrA的gDNA序列扩增产物
图2 AfMcrA的gDNA序列扩增
Fig.2 Amplification of gDNA fragment of AfMcrA
2.3 AfMcrA编码蛋白的生物信息学分析
2.3.1 AfMcrA的基本特征预测分析
AfMcrA共编码366个氨基酸(图3)。生物信息学预测,AfMcrA是1个分子质量为39.82 kDa,理论等电点为8.97,不稳定的亲水蛋白质,且不含信号肽和跨膜结构域。

黑三角形(▲)-与Zn2+结合的半胱氨酸残基:横线(-)-2个半胱氨酸之间的氨基酸残基的数目及种类
图3 AfMcrA的基因结构及编码的氨基酸序列
Fig.3 Gene structure and encoded amino acid sequence of AfMcrA
SMART预测,AfMcrA包含1个GAL4锌指蛋白结构域(氨基酸残基位点117~165),且氨基酸序列Cys-X2-Cys-X6-Cys-X10-Cys-X2-Cys-X6-Cys(图3)符合Zn(Ⅱ)2Cys6型转录因子典型的保守基因序列特征,其中6个半胱氨酸残基共结合2个Zn2+。
AfMcrA含有45个潜在的磷酸化位点,其中丝氨酸磷酸化位点29个,苏氨酸磷酸化位点7个,酪氨酸磷酸化位点9个。
2.3.2 AfMcrA亚细胞定位及结构预测
PredictNLS预测AfMcrA亚细胞定位于细胞核中,这与其作为转录因子相一致。AfMcrA二级结构预测显示,无规则卷曲最丰富(84.43%);其次是延伸链(6.83%)和α-螺旋(6.56%);β-转角最少,仅占2.19%。
2.3.3 AfMcrA无序化特征分析
无序化特征预测,发现AfMcrA包含4处无序区(氨基酸残基位点1~142,182~186,190~247,249~366),共323个无序氨基酸残基(图4)。进一步分析发现,脯氨酸、丝氨酸、谷氨酰胺、丙氨酸、酪氨酸、甘氨酸和精氨酸在AfMcrA无序区含量较为丰富,特别是脯氨酸和丝氨酸。
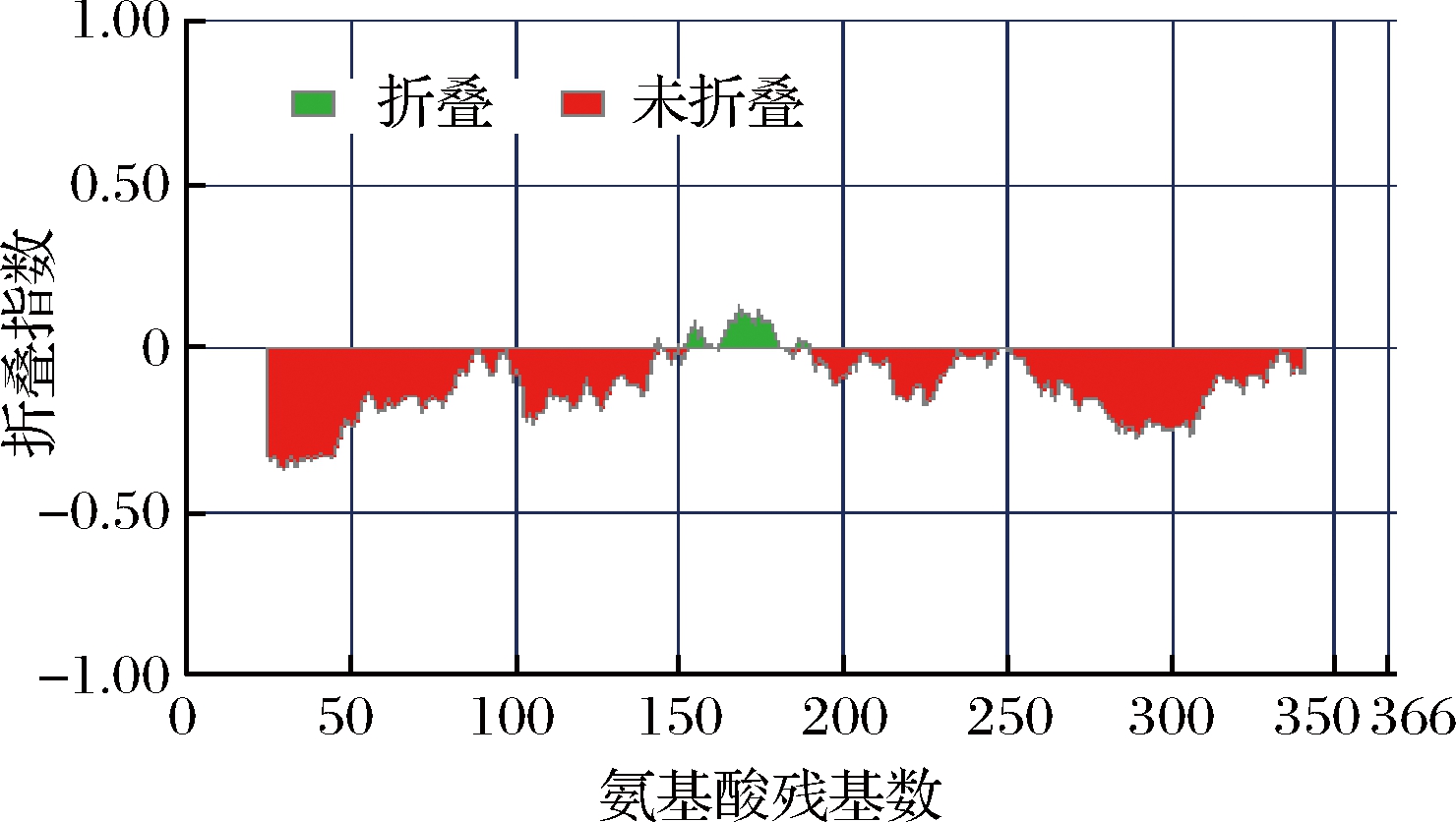
图4 AfMcrA的无序化特征预测
Fig.4 Disordered feature prediction of AfMcrA
2.4 AfMcrA的多序列比对分析
AfMcrA与不同曲霉McrA均具有较高同源性(83.42%~99.73%),表明曲霉属真菌McrA具有高度保守性。图5展示了曲霉McrA的多序列比对结果,发现其具有完全保守的Zn(Ⅱ)2Cys6 DNA结合结构域,但部分McrA存在18~22个氨基酸缺失。进一步分析发现,缺失的22个氨基酸恰好由1个内含子编码。
2.5 AfMcrA的系统进化分析
采用Mega 5.1构建曲霉转录因子McrA的系统发育树,青霉McrA为外类群。由图6可知,黄曲霉AfMcrA与米曲霉McrA聚在同一进化枝上,亲缘关系较近,这与米曲霉和黄曲霉同属曲霉属黄绿组的分类地位相一致。
2.6 AfMcrA的表达分析
qRT-PCR结果显示,随着培养时间的延长,AfMcrA的相对表达量呈现先升高后下降的趋势。以培养2 d的AfMcrA表达量为校正样本,AfMcrA在培养6 d时表达量最高,为2 d的15.79倍;培养4 d时AfMcrA的表达量为2 d的3.24倍(图7)。

图5 不同曲霉McrA蛋白的多序列比对
Fig.5 Multiple sequence alignment of different McrA
注:黑色区域表示比对相似度为100%的序列;灰色区域表示比对相似度≥75%的序列;黑色圆点线显示不同曲霉
McrA的Zn(Ⅱ)2Cys6双核簇DNA结合结构域;黑方框显示不同曲霉McrA的氨基酸缺失位点

图6 AfMcrA的系统进化树
Fig.6 Phylogenetic tree of AfMcrA

图7 不同培养时间AfMcrA的相对表达分析
Fig.7 Relative expression of AfMcrA in different culture periods
注:不同字母表示在P<0.05水平上有显著差异
3 结论与讨论
锌指蛋白作为真核生物中数量最多的一类转录因子,在基因表达调控、细胞分化、信号转导等方面发挥着重要作用[23]。根据保守基序的差异,锌指转录因子主要分为Cys2His2(C2H2)、Cys4(C4)和Cys6(C6)3种类型[24]。C6转录因子只发现于酵母和真菌中,包含一个Zn(Ⅱ)2Cys6双核簇结构域,参与碳和氮化合物的利用、次级代谢产物的合成、无性和有性发育等生物学过程[25-26]。2017年,OAKLEY等[20]首次报道1个新的C6型全局转录因子McrA,并在构巢曲霉、土曲霉和变灰青霉中证实了McrA参与众多次级代谢产物合成的调控。
以构巢曲霉McrA为查询序列进行Blastp分析,发现黄曲霉NRRL3557中的C6转录因子(XP_002380929.1)与构巢曲霉McrA具有较高的同源性(59.57%),但黄曲霉McrA的相关研究目前尚未见报道。因此,本研究通过RT-PCR克隆了黄曲霉AfMcrA,cDNA全长1 101 bp,编码366个氨基酸。AfMcrA具有典型保守基序Cys-X2-Cys-X6-Cys-X10-Cys-X2-Cys-X6-Cys,属于Zn(Ⅱ)2Cys6锌簇蛋白家族[24]。已有研究揭示黄曲霉基因组编码349个Zn(Ⅱ)2Cys6蛋白,其中94个蛋白仅有一个C6域,159个蛋白仅有fungi-specific TF域,96个蛋白具有C6域和TF域[24]。AfMcrA属于黄曲霉中只含有C6域的锌指蛋白,这与构巢曲霉McrA[20]和黑曲霉McrA[21]都只具有保守的C6结构域相一致。PredictNLS预测AfMcrA定位于细胞核内,推测AfMcrA可能在细胞质中合成,然后在核定位信号(YCRRRKIRCSGFE,氨基酸残基位点125~137)的介导下被转运到细胞核中发挥转录因子的调控作用。系统进化分析表明,黄曲霉AfMcrA与米曲霉McrA聚在同一进化枝上,这与Aspergillus oryzae属于A. flavus的同一居群的分类地位相一致。相对于其他曲霉,AfMcrA与Aspergillus arachidicola McrA的亲缘关系更近,这与MOORE等[27]研究结果相一致,A. arachidicola与A. flavus是由共同的原始祖先进化而来,它们之间具有较高的形态、遗传和化学相似性。
AfMcrA具有高比例的无规则卷曲(84.43%)、无序氨基酸残基(88.25%)和磷酸化位点(12.30%),暗示AfMcrA的生物学功能可能具有多样性。AfMcrA与参考序列Aspergillus flavus NRRL3357的McrA相比,缺失22个氨基酸,且这22个氨基酸恰好由1个内含子编码,表明黄曲霉AfMcrA可能存在内含子保留型可变剪切[28]。已有研究表明,内含子在基因表达中起着调控作用,通过选择性剪切使单基因表达多种蛋白,从而丰富蛋白多样性[29]。但黄曲霉AfMcrA是否通过可变剪切形成不同蛋白,执行不同生物学功能有待研究。
qRT-PCR发现,AfMcrA的相对表达量在培养6 d时达到峰值,分别为2 d和8 d的15.79倍和14.76倍,说明黄曲霉在生长前期AfMcrA表达量逐渐升高,而后由于养料的消耗和有害代谢产物的积累,菌丝自溶或生长减缓,导致生长后期AfMcrA表达量急剧下降。丝状真菌很多次生代谢产物是在衰退期合成,推测AfMcrA可能参与黄曲霉某些次级代谢产物合成的调控。构巢曲霉和黑曲霉McrA作为全局转录因子参与次级代谢产物合成的调控[20-21],可以为这一观点提供有力的支撑。值得注意的是,构巢曲霉McrA在杂色曲霉素的生物合成中发挥调控作用[20],而杂色曲霉素是合成黄曲霉毒素的重要前体物质。AfMcrA是否直接或间接参与黄曲霉毒素生物合成的调控是后续着重要解决的科学问题。目前我们正采用农杆菌介导的真菌遗传转化法(agrobacterium tumefaciens-mediated transformation,ATMT)对黄曲霉AfMcrA进行敲除和过表达,揭示其与黄曲霉毒素产生的关系,进一步完善黄曲霉毒素合成错综复杂的调控网络,旨在为制定黄曲霉毒素防控的策略提供理论依据。
[1] AMARE M G, KELLER N P. Molecular mechanisms of Aspergillus flavus secondary metabolism and development [J]. Fungal Genetics and Biology, 2014, 66: 11-18.
[2] HEDAYATI M, PASQUALOTTO A, WARN P, et al. Aspergillus flavus: Human pathogen, allergen and mycotoxin producer [J]. Microbiology, 2007, 153(6): 1 677-1 692.
[3] KUMAR M, THILAK J, ZAHOOR A, et al. Septic arthritis due to tubercular and Aspergillus co-infection [J]. Indian Journal of Orthopaedics, 2016, 50(3): 327-330.
[4] OMEIZA G K, KABIR J, KWAGA J K P, et al. A risk assessment study of the occurrence and distribution of aflatoxigenic Aspergillus flavus and aflatoxin B1 in dairy cattle feeds in a central northern state, Nigeria [J]. Toxicology Reports, 2018, 5: 846-856.
[5] MARCHESE S, POLO A, ARIANO A, et al. Aflatoxin B1 and M1: Biological properties and their involvement in cancer development [J]. Toxins, 2018, 10(6): 214-233.
[6] YU J. Current understanding on aflatoxin biosynthesis and future perspective in reducing aflatoxin contamination [J]. Toxins, 2012, 4(11): 1 024-1 057.
[7] KLICH M A. Aspergillus flavus:The major producer of aflatoxin [J]. Molecular Plant Pathology, 2010, 8(6):713-722.
[8] WANG P, CHANG P K, KONG Q, et al. Comparison of aflatoxin production of Aspergillus flavus at different temperatures and media: Proteome analysis based on TMT [J]. International Journal of Food Microbiology, 2019, 310: 108 313.
[9] LASRAM S, HAMDI Z, CHENENAOUI S, et al. Comparative study of toxigenic potential of Aspergillus flavus and Aspergillus niger isolated from Barley as affected by temperature, water activity and carbon source [J]. Journal of Stored Products Research, 2016, 69: 58-64.
[10] HUA S S T, PARFITT D E, SARREAL S B L. et al. Dual culture of atoxigenic and toxigenic strains of Aspergillus flavus to gain insight into repression of aflatoxin biosynthesis and fungal interaction [J]. Mycotoxin Research, 2019, 35(4): 381-389.
[11] YU J H, BUTCHKO R A E, FERNANDES M, et al. Conservation of structure and function of the aflatoxin regulatory gene aflR from Aspergillus nidulans and A. flavus [J]. Current Genetics, 1996, 29(6): 549-555.
[12] YABE K, NAKAJIMA H. Enzyme reactions and genes in aflatoxin biosynthesis [J]. Applied Microbiology and Biotechnology, 2004, 64(6):745-755.
[13] KALE S P, MILDE L T, TRAPP M K, et al. Requirement of LaeA for secondary metabolism and sclerotial production in Aspergillus flavus [J]. Fungal Genetics and Biology, 2008, 45(10): 1 422-1 429.
[14] YAO G, YUE Y, FU Y, et al. Exploration of the regulatory mechanism of secondary metabolism by comparative transcriptomics in Aspergillus flavus [J]. Frontiers in Microbiology, 2018, 9: 1 568.
[15] 魏鹏霖. 黄曲霉调控因子LaeB及孢子色素合成基因pks1功能的鉴定[D]. 福州: 福建农林大学, 2019.
[16] HU Y, YANG G, ZHANG D P, et al. The PHD transcription factor Rum1 regulates morphogenesis and aflatoxin biosynthesis in Aspergillus flavus [J]. Toxins, 2018, 10(7): 301-317.
[17] ZHUANG Z, LOHMAR J M,SATTERLEE T, et al. The master transcription factor mtfA governs aflatoxin production, morphological development and pathogenicity in the fungus Aspergillus flavus [J]. Toxins, 2016, 8(1): 29.
[18] 宋凤琴, 高晓庆, 梁林林, 等. Homeobox转录因子对黄曲霉生长和毒素合成的影响 [J]. 菌物学报, 2020, 39(3): 566-580.
[19] YUAN J, LI D, QIN L, et al. HexA is required for growth, aflatoxin biosynthesis and virulence in Aspergillus flavus [J]. BMC Molecular Biology, 2019, 20(1): 1-13.
[20] OAKLEY C E, AHUJA M, SUN W W, et al. Discovery of McrA, a master regulator of Aspergillus secondary metabolism [J]. Molecular Microbiology, 2017, 103(2): 347-365.
[21] 李执. 全局调控因子McrA调控下的Aspergillus niger CBS 513.88的表型及次级代谢产物研究 [D]. 福州: 福建农林大学, 2019.
[22] 陈茹, 刘钟滨. 黄曲霉菌aflR基因启动子序列变异与黄曲霉毒素产生相关联 [J]. 细胞生物学杂志, 2006, 28(6): 912-916.
[23] 杨帆, 王娟. 锌指蛋白在丝状真菌纤维素酶基因表达调控中的研究进展 [J]. 纤维素科学与技术, 2014, 22(4): 75-81; 64.
[24] MACPHERSON S, LAROCHELLE M, TURCOTTE B. A fungal family of transcriptional regulators: The zinc cluster proteins [J]. Microbiology and Molecular Biology Reviews, 2006, 70(3): 583-604.
[25] CHANG P K, EHRLICH K C. Genome-wide analysis of the Zn(Ⅱ)2Cys6 zinc cluster-encoding gene family in Aspergillus flavus [J]. Applied Microbiology and Biotechnology, 2013, 97(10): 4 289-4 300.
[26] GUO Z, HAO T, WANG Y, et al. VerZ, a Zn(Ⅱ)2Cys6 DNA-binding protein, regulates the biosynthesis of verticillin in Clonostachys rogersoniana [J]. Microbiology, 2017, 163(11): 1 654-1 663.
[27] MOORE G G, MACK B M, BELTZ S B, et al. Genome sequence of an aflatoxigenic pathogen of Argentinian peanut, Aspergillus arachidicola [J]. BMC Genomics, 2018, 19(1):189-201.
[28] KEREN H, LEV-MAOR G, AST G. Alternative splicing and evolution: Diversification, exon definition and function [J]. Nature Reviews Genetics, 2010, 11(5): 345-355.
[29] LANE C E, HEUVEL K V D, KOZERA C, et al. Nucleomorph genome of Hemiselmis andersenii reveals complete intron loss and compaction as a driver of protein structure and function [J]. Proceedings of the National Academy of Sciences, 2007, 104(50): 19 908-19 913.