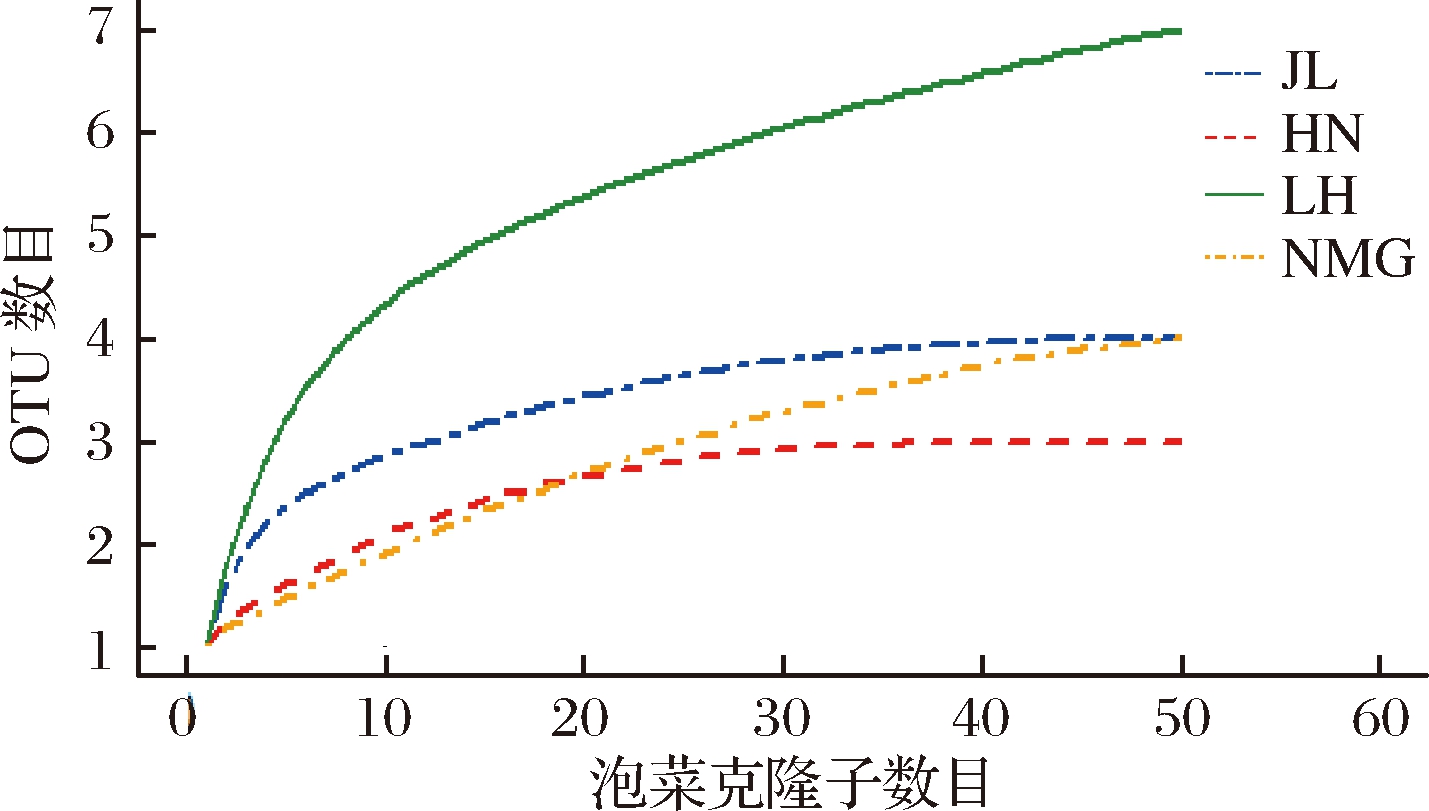
图1 细菌16S rDNA克隆文库稀释曲线
Fig.1 Rarefaction analysis of bacteria 16S rDNA clone library
泡菜是以新鲜蔬菜为原料,经过一定浓度盐水腌渍,由优势菌群乳酸菌以厌氧主导发酵而成的蔬菜食品[1]。中国泡菜有着悠久的历史,在3千多年前的商周时期家家户户就开始制作和食用,数千年来流传广泛,延续至今,是珍贵的中国优良传统发酵食品。泡菜不仅质地鲜嫩,香脆爽口,而且含有大量的膳食纤维、维生素、抗酸化活性因子以及益生菌等,能够帮助机体排毒,促进身体新陈代谢,延缓肌肤衰老,利于胃肠道消化。
研究传统发酵泡菜中微生物多样性的主要方法是可培养和免培养。可培养方法即采用传统分离纯化鉴定的方法,例如党乔等[2]通过MRS培养基分离和16S rDNA测序,从不同厂家生产的泡菜中分离筛选出34株乳酸菌,并分析其种类和数量。刘境等[3]采用分离纯化方法在自然发酵锦州小菜中筛选得到24株疑似乳酸菌,通过16S rDNA序列分析对24株菌进行鉴定。由于传统纯培养技术只能获得环境中微生物总量的极少部分,因此对于展现环境中微生物的多样性存在很大的片面性[4]。随着分子生物学技术的迅速发展,通过直接提取环境微生物基因组DNA的方法,可以从分子水平上全面解释系统中微生物的多样性和群落结构。如陈希等[5]采用建立16S rDNA克隆文库的方法,对不同腌制阶段雪菜的细菌多样性以及相关指标的变化情况进行了研究。曹碧璇等[6]利用构建16S rRNA基因文库的方法,研究了东北自然发酵酸菜中的细菌多样性和群落的组成结构。CHANG等[7]运用变性梯度凝胶电泳(polymerase chain reaction denaturing gradient gel electrop-hores,PCR-DGGE)技术,对不同类型泡菜发酵过程中的细菌、古菌和酵母的动态变化进行了研究。应用这些方法可以更加清晰地了解泡菜中微生物的多样性和菌群结构,避免了传统纯培养方法获取微生物信息的局限性。因此,本研究基于发酵泡菜地区差异的因素,构建16S rDNA克隆文库对吉林、河南、辽宁、内蒙古4个地区发酵泡菜的细菌多样性和群落的组成结构进行研究,探讨不同地区发酵泡菜中微生物多样性的异同之处,以便确切了解不同地区泡菜中的微生物资源分布和泡菜发酵机理。
样品分别取自吉林(JL)、河南(HN)、辽宁(LN)和内蒙古(NMG)4个地区当地传统的自然发酵农家泡菜液,密封保存于-20 ℃备用。
2×Tap 聚合酶链式反应(polymerase chain reaction,PCR) Master Mix、TE缓冲液(pH=8.0),索莱宝科技有限公司;SuperRed/GelRed核酸染色剂、引物Eu27F、1490R,上海生工生物工程有限公司;细菌基因组DNA提取试剂盒、PCR纯化试剂盒,江苏科晶生物科技有限公司;DH5α-感受态细胞、TIANGEN、pGM-T克隆试剂盒,上海康朗生物科技有限公司;其余试剂均为分析纯。
ST16R高速冷冻离心机,赛默飞世尔有限公司;T100PCR仪,深圳瑞华希科技有限公司;R40-IIB2超净工作台,上海苏净实业有限公司;Tanon3500凝胶成像系统,上海天能科技有限公司;GI54DW高压蒸汽灭菌锅,厦门致微仪器有限公司;DYCP-31DN水平电泳仪,南京庚辰科学仪器有限公司; GH6000电热恒温培养箱,天津泰斯特设备有限公司;NanoDrop2000微量核酸测定仪,上海学静生物科技有限公司。
1.4.1 细菌基因组DNA提取
用细菌组DNA提取试剂盒提取泡菜液中的细菌DNA,按照试剂盒说明书进行具体操作。
1.4.2 细菌16S rDNA的PCR扩增
扩增引物:Eu27F:5′-AGAGTTTGATCCTGGCTCAG-3′,1490R:5′-CTACGGCTACCTTGTTACGA-3′[8]。PCR扩增反应体系(25 μL):模板DNA 1 μL(100 ng/μL),上下游引物各2 μL(10 pmol/μL),2×Tap PCR Master Mix 12.5 μL,无菌双蒸水7.5 μL。PCR扩增反应程序:94 ℃预变性4 min,94 ℃变性30 s,54 ℃退火40 s,72 ℃延伸70 s,循环30次,72 ℃延伸20 min后反应结束,4 ℃保温备用[9]。
1.4.3 克隆文库的构建
经纯化后的PCR产物,通过pGM-T克隆试剂盒与pGM-T载体进行连接,连接产物转化入E.coli DH5α-感受态细胞中,并在Amp-X-gal-IPTG抗性筛选培养基上进行蓝白斑筛选,挑选具有氨苄青霉素抗性的白色菌落。再通过菌液PCR检测筛选正确的阳性克隆子,然后建立16S rDNA克隆文库。每个地区泡菜样品中随机挑选50个阳性克隆菌液送上海生工生物工程有限公司测序。
1.4.4 16S rDNA序列分析和系统发育分析
将200个阳性克隆子进行测序后,用软件UCHIME对所测得的16S rDNA序列进行嵌合体识别,检查有无异常序列后再利用GeneBank的BLAST程序进行同源性比对选择相似度最高的16S rDNA序列。使用CLASSIFIER(RDP II)对所测序列进行分类[10]。用软件BioEdit将所有序列以相似性≥97%划分为1个操作分类单元(operational taxonomic unit,OTU)[11]。使用MAGA 5.0软件通过Neighbor-Joining法构建系统发育树[12],并用Bootstrap(1 000次) 检验发育树的置信度。
1.4.5 细菌群落多样性分析
文库的覆盖率可以反映所建文库对系统中细菌多样性的表现程度。采用覆盖率Coverage C评估泡菜细菌克隆文库的库容情况,如公式(1)所示:
Coverage C/%=[1-(n1×N-1)]×100
(1)
式中:n1代表文库中只出现1次的克隆子数目,N代表文库中克隆子总数[13]。
微生物多样性和均匀度指数显示群落中种类多寡及菌株分布等情况,采用4个指标即shannon指数,simpson优势度,pielou均匀度,sorensen similarity相似度指数评价泡菜样品细菌群落的多样性,以上均采用R语言中vegan及biodiversity r包进行统计分析并绘制稀释曲线(rarefaction curve)。根据不同地区泡菜样品的共同OTU数目和单独OTU数目,绘制维恩图,并结合菌群组成柱状图和热图分析菌群的群落结构多样性。
本研究根据样品的OTU数据结果,做出吉林、河南、辽宁、内蒙古4个地区泡菜样品的稀释曲线,如图1所示。随着每个地区泡菜细菌的克隆子数目增加,OTU数目逐渐增加, rarefaction曲线逐渐上升趋于平坦,这说明所测序的克隆数能够反映克隆构建文库中的细菌多样性,可以囊括泡菜样品中大多数的菌落。并且4个地区的细菌克隆文库的覆盖率均较高,依次为92%、94%、86%、92%,这也表明克隆文库的库容已足够,能够较完整地反映细菌群落多样性。

图1 细菌16S rDNA克隆文库稀释曲线
Fig.1 Rarefaction analysis of bacteria 16S rDNA clone library
经分析将200条合格序列按照97%序列相似性共划分为9个OTU。采用R语言工具统计并绘制OTU分布维恩图,可以直观展现4个地区发酵泡菜菌群在OTU构成上的相似性、特异性和交叠情况。结果如图2所示,吉林、河南、辽宁、内蒙古4个地区泡菜的OTU数目存在差异,分别为4、3、7、4个OTU,其中辽宁地区的OTU数目最多,河南地区的OTU数目最少。同时这4个地区的细菌群落组成有重叠,说明它们之间均存在共同的群落,其中4个地区中共有的细菌OTU数目为2。两两地区间也存在共同的OTU,吉林和内蒙古共有的细菌OTU数量为4;辽宁和河南共有的细菌OTU数目为3。另外在辽宁地区中特异性细菌OTU数目为4,而其他3个地区没有特异性细菌OTU,这说明辽宁地区泡菜的细菌群落结构多样,细菌群落组成特异性在这4个地区中最大。

图2 不同地区发酵泡菜OTU维恩图谱
Fig.2 OTU Venn diagram of fermented pickles in different regions
不同地区泡菜的细菌多样性见表1,辽宁地区和吉林地区的多样性指数分别为1.555和1.015,说明这两个地区泡菜的细菌群落具有较高的多样性,并且辽宁地区的细菌群落具有更为丰富的多样性,而河南和内蒙古地区的多样性指数分别为0.43和0.5,泡菜的细菌群落多样性相对较低。同时,除河南地区的其他3个地区泡菜的均匀度指数相差不大,为0.549~0.69,但河南地区泡菜的均匀度指数很低为0.384,说明河南地区泡菜的细菌种群的分布很不均匀。另外,相似系数显示了辽宁地区和其他3个地区的泡菜相似性较平均,均在0.5以上,而内蒙古和河南地区的泡菜之间的细菌相似度最差,相似系数仅为0.12。
表1 不同地区泡菜细菌的多样性指数和相似性
Table 1 The diversity index and similarity index of pickles bacteria with different regions

地区克隆数shamnon指数sipson指数均匀度吉林内蒙古辽宁河南相似度JL 501.0150.5890.69010.480.540.44NMG500.5000.2500.54910.50.12LN 501.5550.7500.67610.62HN 500.4300.1860.3841
本研究中200条克隆子序列经CLASSIFIER进行分类后,显示这些克隆子均为乳酸菌,属于硬壁菌门,并且分布于乳杆菌属(Lactobacillus)(83%),肠球菌属(Enterococcus)(15.5%),明串珠菌属(Leuconostoc)(1%)以及片球菌属(Pediococcus)(0.5%)。将所有序列在NCBI上和GeneBank数据库中的已知序列进行比对,根据比对结果得到不同地区泡菜的细菌群落结构组成,如图3所示。
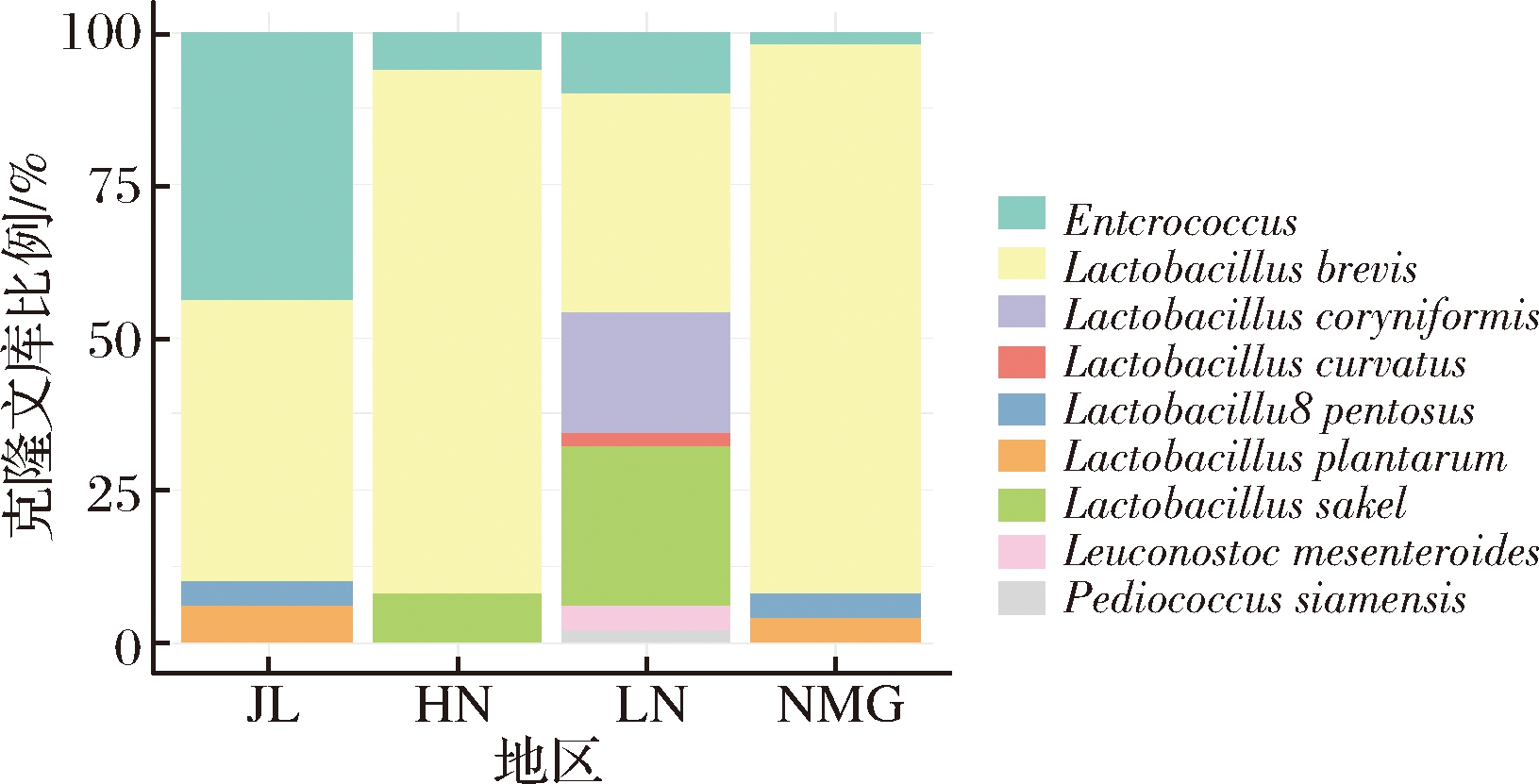
图3 不同地区泡菜的细菌16S rDNA克隆文库
克隆子分布图
Fig.3 Distribution map of the clones in the bacteria 16S rDNA clone libraries of pickle with different regions
STACKBRANDT等[14]认为16S rDNA基因序列同源性≥97%的菌株可以看作一个种。而对于基因序列高度相似的Enterococcus属只鉴定至属水平,因此在其余169个菌株中,129个(64.5%)经鉴定为短乳杆菌(Lactobacillus brevis),17个(8.5%)为沙克酸乳杆菌(Lactobacillus sakei),10个(5%)为棒状乳杆菌(Lactobacillus coryniformis),5个(2.5%)为植物乳杆菌(Lactobacillus plantarum),4个(2%)为戊糖乳杆菌(Lactobacillus pentosus),2个(1%)为肠系膜明串珠菌(Leuconostoc mesenteroides),1个(0.5%)为Pediococcus siamensis,1个(0.5%)为弯曲乳酸杆菌(Lactobacillus curvatus)。图3表明所有菌种在4个地区泡菜中呈现不均衡分布,在内蒙古和河南的泡菜中,L.brevis占了绝对的优势,是最优势菌群,分别占克隆文库比例为90%和86%;同样在吉林和辽宁,L.brevis所占的比例相对较大,为主要优势菌群,所占比例分别为46%、36%;而辽宁泡菜的主要优势菌群还有L.sakei和L.coryniformis,分别占克隆文库比例为26%和20%。从整体上来看Lactobacillus在4个地区泡菜微生物中均占据了绝大部分,根据以往的研究表明,泡菜中的乳酸菌大多数来自于Lactobacillus属,这与本研究结果相符。田伟等[15]通过构建16S rRNA基因文库,对四川发酵泡菜中的菌落组成进行分析,所分析的克隆子主要分布于Lactobacillus属和Pediococcus属,且Lactobacillus属占主导。武俊瑞等[16]运用PCR-DGGE 技术从东北发酵酸菜中分离和鉴定出Lactobacillus属的9个种。张先琴等[17]通过PCR和PCR-DGGE技术,分析四川家庭制作泡菜中的细菌群落结构,结果显示其中的Lactobacillus属是优势种群。而针对4个地区泡菜中均占据一定比例的Enterococcus属,在以前的研究中也有报道过[18-19]。
Heatmap图可以用颜色深浅度来反映样本间或物种丰度相似性聚类[20],将聚类结果展示在Heatmap图上采用R语言vegan包进行距离计算和聚类分析;得到4个地区泡菜细菌群落及其分类水平Heatmap图,如图4所示,图中每一行代表不同的种,不同的颜色代表不同的丰度,图中颜色越接近红色,表示在对应地区泡菜中的菌种丰度越大。反之,颜色越接近蓝色则物种丰度越小。由图4可知,不同细菌物种在各地区泡菜中所占丰度不同,其中L.pentosus在吉林和内蒙古地区泡菜中丰度较高;L.brevis在河南和内蒙古地区泡菜中丰度较高;Enterococcus属和L. plantarum在吉林地区的泡菜中丰度较高,但辽宁泡菜的细菌菌种比较多,也是各地区菌种差异的主要来源,主要集中于L.coryniformis,Leu.mesenteroides,L.curvatus,Ped.siamensis。而在吉林,河南和内蒙古地区的泡菜中细菌菌种较少,微生物种类多样性相对较低,这说明不同地区泡菜的细菌群落结构存在差异。

图4 不同地区泡菜的微生物多样性Heatmap图
Fig.4 Heatmap analysis of microbiological diversity in pickles with different regions
由于Lactobacillus属在4个地区泡菜细菌中占据绝大部分,因此这里只针对Lactobacillus属进行系统发育分析。选择本研究中Lactobacillus属的部分代表序列和NCBI数据库中的已知菌株序列建立系统发育树。结果如图5所示,在系统发育树中所有菌株被划分在4个分支,CD18112100961和CD18112100941与L.coryniformis在同一分支,相似性分别为98%,99%。CD18112100902,CD18112100911,CD18112100963与L.curvatus和L.sakei在一个分支,其中CD18112100963与L.sakei的相似性为99%,其余2株与L.curvatus相似度分别是96%、98%。CD18071300657、CD18101301379、CD18101301341与L.plantarum和L.pentosus聚为1个分支,CD18071300657和L.plantarum有97%的相似性,CD18101301379、CD18101301341与L.pentosus的相似性分别为98%,99%。CD18071300538、CD18112 100951、CD18071300677、CD18071300625、CD180713 00620、CD18071300646、CD18112100925和CD180713 00621都与L.brevis在同一分支,其中除了CD181121 00951和CD18071300538与L.brevis的相似性为96%,其他菌株的相似度均在97%~99%。另外,从4个地区泡菜中共有的菌种L.brevis来看出现了很多分支,说明来自不同地区的同一菌种在进化关系上也出现了差异,这可能是不同地域环境因素造成的。而来自同一地区属于同一物种的菌株之间在进化上也有差别,这可能和同一地区中不同泡菜样品的来源有关。

图5 根据16S rDNA序列构建的系统发育树
Fig.5 Phylogenetic tree based on 16S rDNA sequences
本实验以不同地区传统发酵泡菜为研究对象,采用构建克隆文库的方法,对克隆子的16S rDNA序列进行测序,在此基础上研究自然发酵泡菜液微生物群落组成及其多样性。尽管利用该技术不能反映样品中所有的细菌类群,但该方法可以反映样品中的优势细菌类群[21]。通过构建16S rDNA基因文库,所分析的200个克隆子分布于Lactobacillus属,Enterococcus属,Leuconostoc属以及Pediococcus属。其中Lactobacillus属在数量上占有绝对优势,是4个地区泡菜共有的主要优势菌属,这种现象与多数研究结果相似。一般情况下,在泡菜发酵初期,微厌氧、酸耐受性弱的乳酸菌如Leuconostoc属的Leu. mesenteroides较多,随着泡菜的不断发酵,乳酸逐渐积累,O2逐渐匮乏,此时酸耐受性强、严格厌氧的乳酸菌如Lactobacillus属的L. sakei、L. plantarum等逐渐占据优势[22]。王秋霞等[23]发现在泡菜发酵初期以乳球菌为主,其中Leu.mesenteroides等是主要的微生物类型,而在泡菜发酵中后期,以乳杆菌如L.plantarum、L.brevis和Lactobacillus casei等为主。XIONG等[24]证实在泡菜发酵启动阶段Leu.mesenteroides subsp.先占主导,然后逐步由Lactococcus lactis、Enterococcus faecalis替换,最后由L.casei和L.plantarum完成发酵。同时有研究表明成熟泡菜中优良菌株的筛选结果大多数来自于Lactobacillus属[25-27],优良的理化特性(如耐酸耐胆盐)不仅使Lactobacillus属在泡菜菌群中占据优势地位,而且在泡菜发酵过程中起着一定作用,是衡量泡菜发酵过程的指标。目前,根据我国不同传统发酵蔬菜的细菌多样性研究现状,虽然不同地域具有不同的代表性发酵蔬菜,但L.brevis、L. plantarum、Leu.mesenteroides等都是发酵蔬菜中常见的微生物[28-29]。本研究结果也表明,L.brevis是4个地区泡菜样品中的优势菌种,同时在辽宁地区泡菜中的主要优势菌群还有L. sakei和L.coryniformis,这些和其他的报道有一定差别。乌日娜等[30]通过PCR和PCR-DGGE技术,对发酵进程中酸菜的细菌群落动态变化进行分析,发现在酸菜发酵前期Leuconostoc sp.为优势菌群, 在发酵中后期L.plantarum为优势菌群。张蓓蓓等[31]在180多份泡菜中分离出447株乳酸菌,经鉴定分布于11个属34个种,并确定了Leu.mesenteroides、L.brevis、L.plantarum等为主要优势菌群。并且,本研究中除了Ped.siamensis,其余菌种在以前的研究中都有报道过[32-34]。
量化的多样性指数可以反映泡菜发酵过程中微生物群落结构的演替变化规律,本研究中多样性指数分析说明,辽宁和吉林地区泡菜的细菌多样性较丰富,但辽宁泡菜的细菌多样性相对高些,其主要原因是辽宁泡菜所特有的菌群L.coryniformis、Leu.mesenteroides、L.curvatus、Ped.siamensis也占据了一定的比例,使得辽宁泡菜的细菌多样性较高以及呈现出与其他3个地区泡菜的差异性。而内蒙古和河南地区泡菜的细菌群落多样性相对较低,原因在于细菌群落结构相对单一。4个地区泡菜的细菌群落分布不同,微生物多样性存在差异,究其原因,泡菜发酵是一个复杂的发酵体系,微生物种类繁多,不同的外部因素如气候、地域、原辅料、发酵方法等均能够影响发酵泡菜的微生物群落结构[35],从而也导致泡菜的口感、风味、营养的差异。
本研究通过构建16S rDNA克隆文库方法对吉林、河南、辽宁、内蒙古4个地区传统发酵泡菜的细菌群落多样性进行研究,从4个地区泡菜样品中随机挑取200个克隆子,经16S rDNA基因序列鉴定所得克隆子可分为4个属,其中Lactobacillus属6个种占据了绝对比例,Enterococcus属均占4个地区泡菜细菌的一定比例,另外在辽宁地区泡菜中还有Leu. mesenteroides和Ped.siamensis,因此辽宁地区泡菜的细菌种类数最多,而河南地区泡菜的细菌群落较为单一,但在4个地区泡菜中优势菌种均为L.brevis。多样性指数分析表明不同地区泡菜的菌株种类、优势度、均匀度存在差异,辽宁地区泡菜的种类较丰富,优势属突出,且分布均匀,而河南地区泡菜的多样性、优势度和均匀度较低,并且除辽宁地区泡菜外,其他3个地区之间的菌落相似度均较低。由于本研究只构建了4个地区泡菜的细菌16S rDNA克隆文库,没有研究泡菜液中其他微生物类群,因此需要进一步探讨。本研究通过比较分析不同地区泡菜的细菌多样性,可为后续深入研究泡菜发酵原理、风味物质的合成机制提供一定的理论依据,对优化泡菜发酵条件和开发利用泡菜微生物资源具有重要的研究意义。
[1] 张其圣,陈功,申文熹,等.中国泡菜乳酸菌群落结构动态变化研究进展[J].食品与发酵科技,2016,52(6):1-8.
[2] 党乔,孔令聪,刘洁,等.泡菜发酵乳酸菌的分离鉴定及耐药性分析[J].食品科学,2019,40(20):166-170.
[3] 刘境,孙慧君,李默,等.自然发酵锦州小菜中乳酸菌的分离筛选[J].食品科学,2019,40(2):73-78.
[4] WARD D M,WELLER R, BATESON M M.16S rRNA sequences reveal numerous uncultured microorganisms in a natural community[J]. Nature, 1990, 345(6 270):63-65.
[5] 陈希,沈锡权,翁佩芳,等.应用16S rDNA克隆文库法分析雪菜低盐腌制过程微生物群落的多样性[J].中国食品学报,2012,12(7):205-211.
[6] 曹碧璇,胡滨,刘爱平.利用16S rRNA基因克隆文库分析东北自然发酵酸菜中细菌多样性[J].食品与发酵工业,2015,41(11):76-80.
[7] CHANG H W, KIM K H, NAM Y D, et al. Analysis of yeast and archaeal population dynamics in kimchi using denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology, 2008, 126(1-2):159-166.
[8] 李小艳,刘书亮,蒲彪,等.泡菜中低温乳酸菌的筛选及其特性研究[J].中国食品学报,2014,14(10):108-116.
[9] 王营,孟祥晨.PCR-DGGE结合种特异性PCR技术检测市售酸奶中乳酸菌[J].食品科学,2013,37(8):253-258.
[10] TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology & Evolution, 2011, 28(10):2 731-2 739.
[11] COLE J R, WANG Q, CARDENAS E, et al. The ribosomal database project: Improved alignments and new tools for rRNA analysis[J]. Nucleic Acids Research,2009,34:141-145.
[12] PEI Z H, BINI E J, YANG L Y, et al. Bacterial biota in the human distal esophagus[J]. Proc NatI Acad Sci,2004, 101(12): 4 250-4 255.
[13] WU L, GE G, ZHU G F, et al. Diversity and composition of the bacterial community of Poyang Lake (China) as determined by 16S rRNA gene sequence analysis[J]. World Journal of Microbiology and Biotechnology, 2012, 28(1):233-244.
[14] STACKEBRANDT E, GOEBEL B M. Taxonomic note: A place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J]. International Journal of Systematic Bacteriology, 1994, 44(4):846-849.
[15] 田伟,张琦,邓珍珍,等.利用16S rRNA分析传统四川发酵泡菜中的细菌多样性[J].食品科学, 2013, 34(17):215-218.
[16] 武俊瑞, 岳喜庆, 石璞,等.PCR-DGGE分析东北自然发酵酸菜中乳酸菌多样性[J].食品与生物技术学报, 2014, 33(2):127-130.
[17] 张先琴, 张小平, 敖晓琳,等.PCR-DGGE分析四川地区家庭制作泡菜中微生物多样性[J].食品科学, 2013, 34(12):1 29-134.
[18] 盛海圆,郭艳萍,常艳,等.传统泡菜中乳酸菌多样性的分析[J].中国微生态学杂志,2010, 22(7):580-582;586.
[19] 王蓓,李淑英,张翠萍,等.PCR-DGGE分析玉溪地区水果泡菜中细菌多样性[J].云南农业大学学报(自然科学),2017,32(4):740-746.
[20] JAMI E, ISRAEL A, KOTSER A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. Isme Journal, 2013, 7(6):1 069-1 079.
[21] ZHENG Y,HOU L,LU M,et al.The bacterial diversity in surface sediments from Chongming Eastern tidal flat in summer and winter[J].China Environmental Science,2012,32 (2): 300-310.
[22] JI Y C, HAE C C. Improvements in the quality and shelf life of kimchi by fermentation with the induced bacteriocin-producing strain, Leuconostoc citreum GJ7 as a starter[J]. Journal of Food Science, 2010, 75(2):103-110.
[23] 王秋霞,路建东,金玲,等.乳酸菌菌种差异对泡菜发酵风味的影响[J].粮食与食品工业,2016,23(1):41-46.
[24] XIONG T, GUAN Q, SONG S, et al. Dynamic changes of lactic acid bacteria flora during Chinese sauerkraut fermentation[J]. Food Control, 2012, 26(1):178-181.
[25] 叶陵,李勇,王蓉蓉,等.剁辣椒中优良乳酸菌的分离鉴定及其生物学特性分析[J].食品科学,2018,39(10):112-117.
[26] 罗强,李幸洋,陈炼红,等.传统发酵泡菜中乳酸菌种群组成及优良菌株产酸耐酸特性分析[J/OL].食品科学,2020.DOI:107506/spkx1002-6630-20191020-207.
[27] 张晓娟,夏珊,陈洁,等.四川泡菜降解亚硝酸盐优良功能菌种的筛选、鉴定及其性能初探[J].中国调味品,2016,41(7):73-77.
[28] 叶陵,李勇,王蓉蓉,等.我国传统发酵蔬菜中乳酸菌多样性的研究进展[J].食品科学,2018,39(15):296-301.
[29] XIONG T, SONG S H,HUANG X H, et al. Screening and identification of functional lactobacillus specific for vegetable fermentation[J]. Journal of Food Science, 2013, 78(1):84-89.
[30] 乌日娜,于美玲,孟令帅,等.PCR-DGGE分析东北自然发酵酸菜中的微生物多样性[J].现代食品科技,2014,30(10):8-12;35.
[31] 张蓓蓓,王柱,王宪斌,等.四川地区泡菜微生物的多样性分析[J].食品与发酵科技,2016,52(1):1-5;10.
[32] YAN P M, XUE W T, TAN S S, et al. Effect of inoculating lactic acid bacteria starter cultures on the nitrite concentration of fermenting Chinese paocai[J]. Food Control, 2008, 19(1):50-55.
[33] 栗永乐,李秀丽,李传娟,等.内蒙古东部地区农家酸菜中乳酸菌的分离与初步鉴定[J].食品工业,2012,33(11):132-134.
[34] 翁佩芳,陈希,沈锡权,等.榨菜低盐腌制细菌群落多样性的分析[J].中国农业科学,2012,45(2):338-345.
[35] 邹伟,赵长青,赵兴秀,等.泡菜微生物群落结构及其动态机制研究概述[J].食品与发酵工业,2015,41(4):241.