豆豉作为国内传统发酵豆制品的代表,可分为曲霉型、毛霉型、细菌型等类型[1]。就曲霉型豆豉而言,其起源最早且分布最广,具有开胃消食、抗氧化[2]、溶血栓[3]等多重功效,是我国居民饮食生活中的调味品。虽然曲霉型豆豉的制作工艺不一,但均含制曲、发酵2个过程。其中,制曲周期较短且工艺差异较小,常由工业化的曲霉菌种完成;而发酵周期往往更长且工艺不一,许多发酵参数(如拌盐量、发酵温度等)常存在一定差别,造就了曲霉型豆豉营养风味的多样化[4]。由于微生物存在于制曲与发酵的全过程,其通过酶解及自身代谢可生成复杂多样化的产物,而不同工艺下曲霉型豆豉菌群结构差异一定程度上是造成营养风味差异的根本原因,这侧面表明了准确揭示豆豉菌群结构的必要性。
鉴于此,过去许多研究者利用诸类工具对豆豉菌群结构展开了探究。就可培养法而言,大量学者利用该技术对豆豉菌群结构进行了探究[5-7],而可培养法存在许多局限性,但也具有区分菌株活性并获得菌种资源的优势。也有学者利用分子生物学技术进行探究,如限制性片段长度多态性技术[8]、变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)技术[9],CHEN等[10]结合可培养法和DGGE技术解析了制曲阶段的菌群结构。伴随着测序技术的发展,以高准确率、高通量著称的二代测序技术应运而生。李晓然等[11]率先运用454测序平台阐明了云南细菌型豆豉的菌群结构,而测序成本逐渐降低,包括本课题组在内的一些学者[12-14]也对豆豉制曲及发酵中的菌群结构变化进行了研究。然而,高通量测序技术也存在一些弊端,如无法区分菌株存活和鉴定至种水平等[15],而可培养法恰与其形成优势互补。为此,已有一些学者利用可培养法结合高通量技术,对泡菜[16]、普洱茶[17]等发酵食品的菌群结构展开了探索,但关于豆豉的类似研究仍未见报道。
本研究以快速工艺与传统工艺下曲霉型豆豉为研究对象,基于两者发酵过程中重要理化因子的差异,一方面通过可培养法比较其优势细菌分布的整体差异,另一方面结合高通量测序技术深入剖析两者细菌群落的演替差异,并就发酵细菌基因进行了功能预测。本研究更科学地揭示2类豆豉发酵过程中细菌演替差异,同时,也旨在为后续曲霉型豆豉的风味改善试验提供有效理论支撑。
1 材料与方法
1.1 材料与试剂
脑心浸液肉汤(brain heart infusion,BHI)培养基、马铃薯葡萄糖琼脂(potato dextrose agar,PDA)培养基,海博生物公司;乳酸细菌(MRS)培养基,索莱宝生物公司;氯霉素、纳他霉素,阿拉丁生化公司;细菌基因组DNA提取试剂盒,天根生化科技公司;E.Z.N.A.Soil DNA提取试剂盒、凝胶回收试剂盒,OMEGA公司;DreamTaq Green PCR Mix,Thermo Fisher公司;常规生化试剂均为国产分析纯。
1.2 仪器与设备
TM902C数显温度计,安晟一电子科技公司;PB-10型pH计,Sartorius公司;DH4000II电热恒温培养箱,泰斯特仪器公司;ZQZY-GS8V电热恒温摇床,知楚仪器公司;HICO 21高速离心机,生工生物工程公司;T100 PCR仪、GelDoc XR凝胶成像仪,Biorad公司;DYY-6C凝胶电泳仪,六一仪器厂;Nanodrop-2000超微量分光光度计,Thermo Fisher公司;HiSeq 2500测序仪,Illumina公司。
1.3 实验方法
1.3.1 豆豉样品的采集
曲霉型豆豉样品,稻香园调味食品公司。样品含快速工艺和传统工艺2种,两者均先经7 d制曲阶段(米曲霉沪酿3.042,接种质量分数为0.001 5%),并经洗曲、堆积后,再运用不同工艺进行控温发酵(发酵罐7 d和发酵池约3周)。采样为同一发酵容器的3个区域分层(上、中、下)采样,同一区域的各层样品经充分混合后,装入同一无菌自封袋,即每个发酵阶段含3个样品。采样方式为初、中、末3个时期的跟踪采样,快速工艺在发酵第1、4、7天采样(编号:R1、R4、R7),传统工艺在发酵第1、7、21天采样(编号:T1,T7,T21),样品采集完毕即保存于冰盒,并尽快运回实验室备用。
1.3.2 重要理化因子的测定
豆豉采样时,使用数显金属温度计测定发酵温度。将2 g豆豉置于研钵充分碾碎,溶于50 mL纯水中,利用pH计进行pH测定。豆豉NaCl含量的测定采用银量法,具体参考GB 5009.44—2016《食品安全国家标准 食品中氯化物的测定》[18]。
1.3.3 豆豉微生物的分离培养与鉴定
豆豉微生物采用梯度稀释法(10-3~10-7梯度)涂布培养。细菌通过BHI培养基(另加0.1 g/L纳他霉素)于37 ℃培养24 h;真菌通过PDA培养基(另加0.025 g/L氯霉素)于28 ℃培养72 h;乳酸菌通过厌氧双层MRS培养基(另加0.1 g/L纳他霉素和1 g/L CaCO3)于37 ℃培养48 h。培养结束,取30~300菌落的平皿计数,并选取优势细菌(>5 log CFU/g)进一步分离。根据菌落外观结合光学显微镜鉴定,挑取具有一定形态差异的菌株,转接至对应液体培养基培养,培养完毕后短暂离心获得菌液沉淀。
参照试剂盒说明书,提取优势细菌基因组DNA,通过引物27F/1492R扩增16S rRNA 基因,引物序列及扩增参数参照文献[19]。扩增产物经电泳验证合格,送至生工生物工程公司测序。测序结果经拼接处理,通过NCBI网站的BLAST在线程序比对。通过MEGA软件采用最大简约法构建系统发育树,聚类树节点的置信度采用1 000次自举法[20]。
1.3.4 豆豉微生物宏基因的提取
等量称取各豆豉样品,使用无菌磷酸盐缓冲液(pH 7.0)洗涤3次,将豆豉样品洗脱下的物质用无菌双层纱布过滤。收集的滤液于4 ℃离心后弃上清液,参照试剂盒说明书完成宏基因提取。提取完毕,通过10 g/L的琼脂糖凝胶电泳及超微量分光光度计评估宏基因的完整性、浓度及污染程度。
1.3.5 PCR扩增与上机测序
以合格的豆豉宏基因为模板,采用含Barcode标签的引物338F/806R对细菌16S rRNA基因V3~V4区域进行PCR扩增,引物序列及扩增参数参照文献[12]。取少量扩增产物经电泳初步评估后,利用凝胶回收试剂盒完成回收。通过超微量分光光度计评估扩增产物的浓度及污染程度,所有样品均合格后,统一各样品的DNA总量后测序。上机测序使用的Hiseq 2500平台,采取双末端测序策略(150 bp×2),测序委托诺禾致源科技公司完成。
1.3.6 高通量测序数据的处理
获得原始序列后,通过FLASH、USEARCH和UCHIME软件获得不含嵌合体的tags序列。通过USEARCH软件在相似性97%的水平聚类OTU,以序列总数的0.005%为阈值过滤OTU[21]。序列经RDP Classifier软件比对Silva数据库(置信度阈值=0.8)[22]完成物种注释。采用mothur 软件计算细菌群落Alpha多样性指数;采用QIIME软件对细菌群落结构进行主坐标分析(principal coordinates analysis,PCoA)分析。通过Lefse软件分析各组间属水平上的标志生物(Biomaker),设置线性判别的得分筛选值为4[23]。参考KEGG数据库,通过PICRUSt软件进行豆豉样品的细菌基因功能预测。
1.4 统计分析与数据提交
试验结果以平均值±标准差表示,样品差异分析采用单因素方差法,差异显著(P<0.05)时通过Duncan氏法进行多重比较,分析由SPSS 24.0软件完成。
本文涉及18样品的原始序列已上传至NCBI网站SRA(Sequence Read Archive)数据库,项目检索号:SRP262092。
2 结果与分析
2.1 两种豆豉发酵过程中重要理化因子的比较
就重要的理化因子来看(图1),2种豆豉的pH值均持续下降,其中快速工艺下降较快,到发酵第7天即降至4.67;而传统工艺的pH下降偏慢,至发酵第7天仍为5.61,最终pH值为4.91。两者发酵中均采用人工控温策略,其中快速发酵工艺大都高达50~60 ℃,于第4天达到57.6 ℃的峰值,而传统工艺大都在40~50 ℃,最高温度为47.5 ℃。另外,由于发酵前未拌盐,快速工艺豆豉最高的NaCl含量仅为0.42%,而拌盐后的传统工艺豆豉NaCl含量最高为6.41%,因两者表面均覆盖盐层,发酵中盐层的逐步溶解,使得两者NaCl含量均小幅上升。
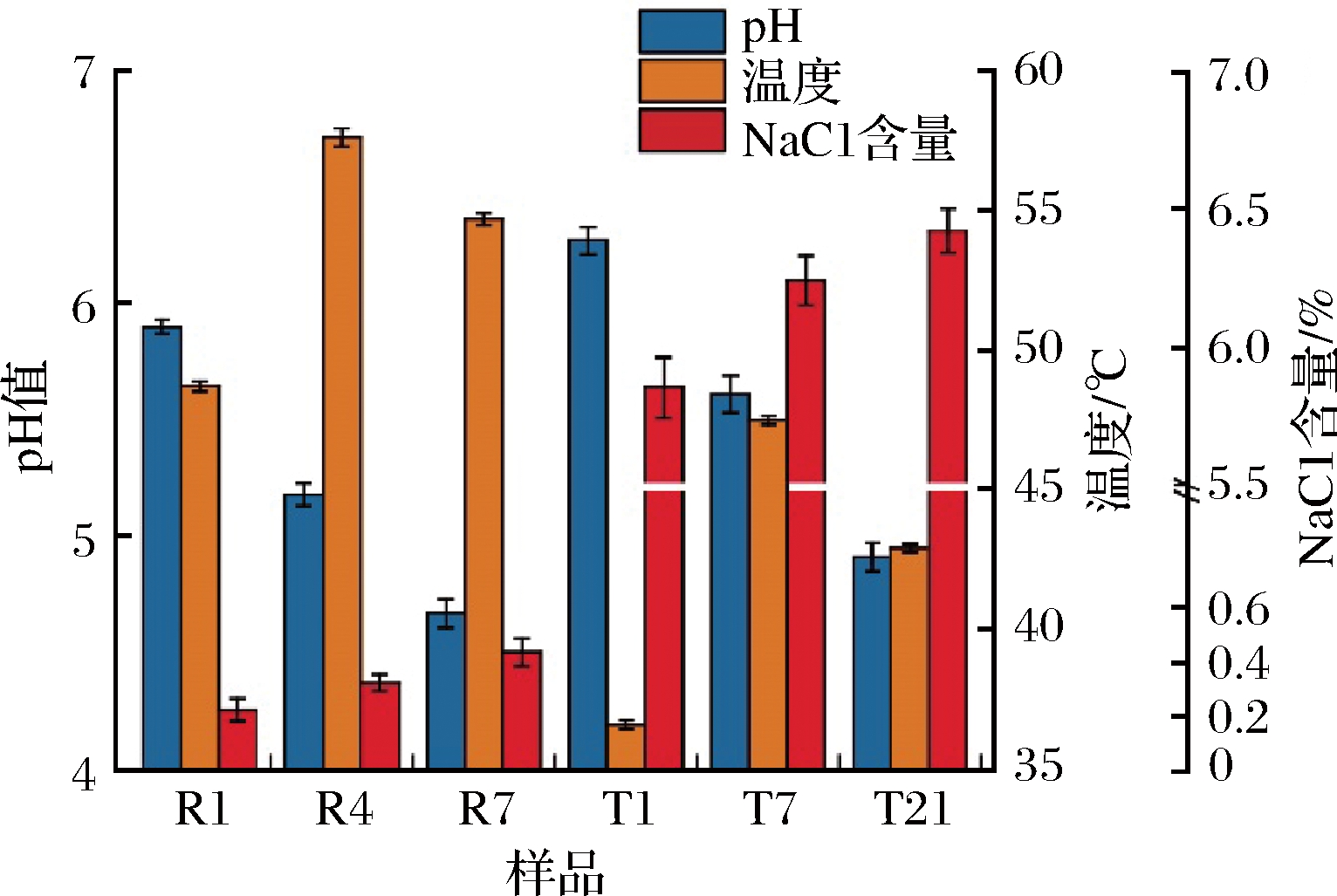
图1 两种豆豉发酵过程中重要理化因子的变化
Fig.1 Changes of vital physicochemical factors of two types of Douchi during fermentation
2.2 两种豆豉发酵过程中可培养微生物的比较
2.2.1 可培养微生物的丰度
经培养后计数发现(图2),2种工艺下豆豉可培养微生物的总量均呈下降趋势,而同时期传统工艺豆豉各类微生物的丰度更高(P<0.05)。具体来看,快速工艺下,各类微生物数目稳步下降,细菌由初期7.16 log CFU/g下降至末期的3.76 log CFU/g,而真菌、乳酸菌初期仅各为4.05和3.43 log CFU/g,末期甚至均未能分离。就传统工艺而言,细菌也占据着统治地位,各阶段数目分别为7.71、7.04和6.24 log CFU/g;真菌的数目最高仅为5.35 log CFU/g,相较于细菌,其在各阶段的数目均约低3个数量级;而从乳酸菌群体来看,其在初期达到6.85 log CFU/g,但在发酵中末期则均低于4 log CFU/g。
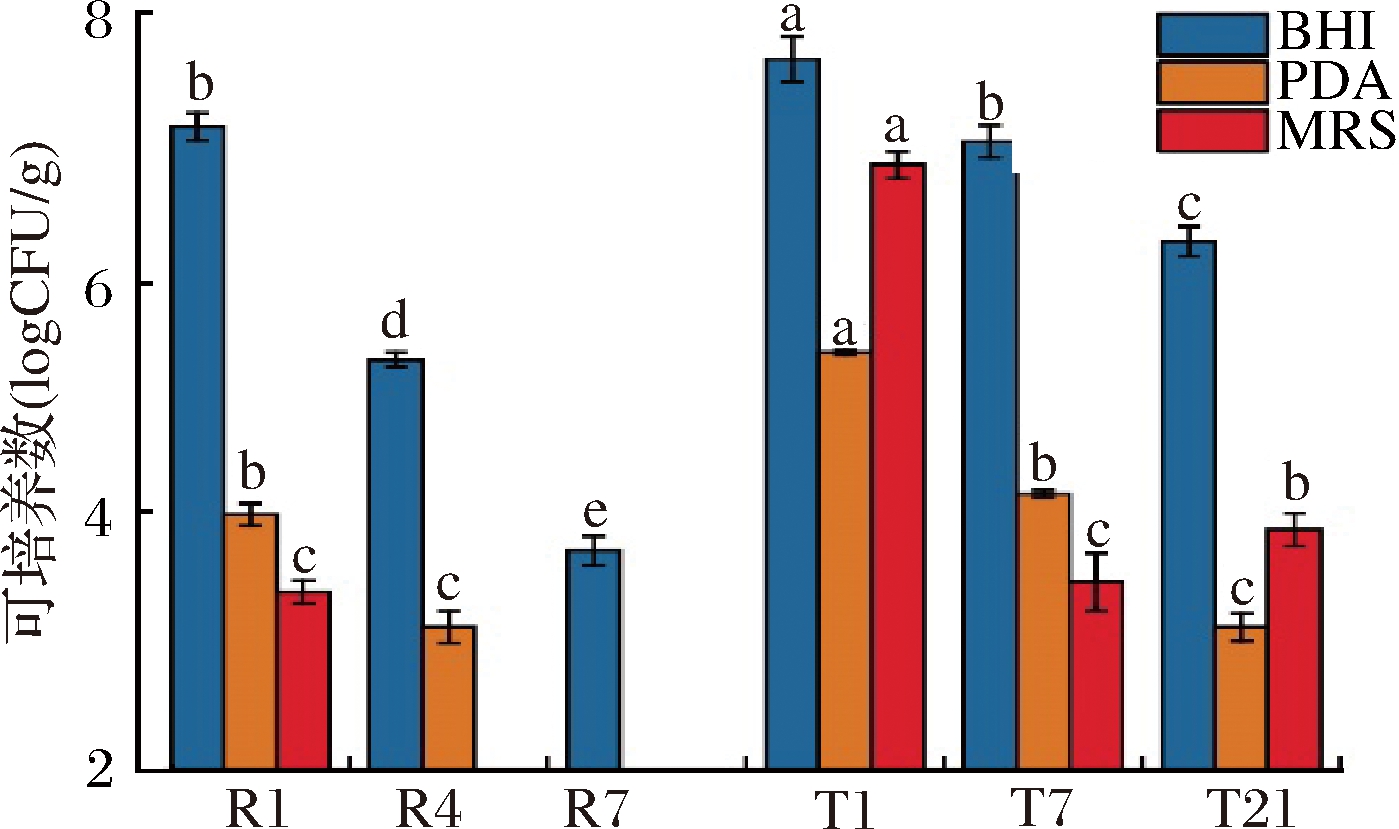
图2 两种豆豉发酵过程中可培养微生物的丰度变化
Fig.2 Changes in the abundance of cultured microbes of two types of Douchi during fermentation
注:不同的字母标注表示具显著性差异(P<0.05)(下同)
2.2.2 优势细菌的鉴定
由于2类工艺下豆豉中细菌的丰度均占绝对优势,为此后续重点选取优势细菌进行分离鉴定。分子鉴定的结果如图3所示,22株优势细菌可归为5个菌属下的7个种,其中2种为乳酸菌。从各菌种的分布来看,贝莱斯芽孢杆菌(Bacillus velezensis)分布最为广泛,且在传统工艺的各时期均为优势菌,而同属的地衣芽孢杆菌(Bacillus licheniformis)仅在2种工艺的发酵初期为优势菌。就特有优势菌而言,传统工艺下包括4种,分别为鸡葡萄球菌(Staphylococcus gallinarum)、松鼠葡萄球菌(Staphylococcus sciuri)、泰国魏斯氏菌(Weissella thailandensis)及发酵乳杆菌(Lactobacillus fermentum),分布于发酵初期及中期;与前者形成鲜明对照的是,快速工艺下特有优势菌仅为球形赖氨酸芽孢杆菌(Lysinibacillus sphaericus),且其仅在发酵初期出现。
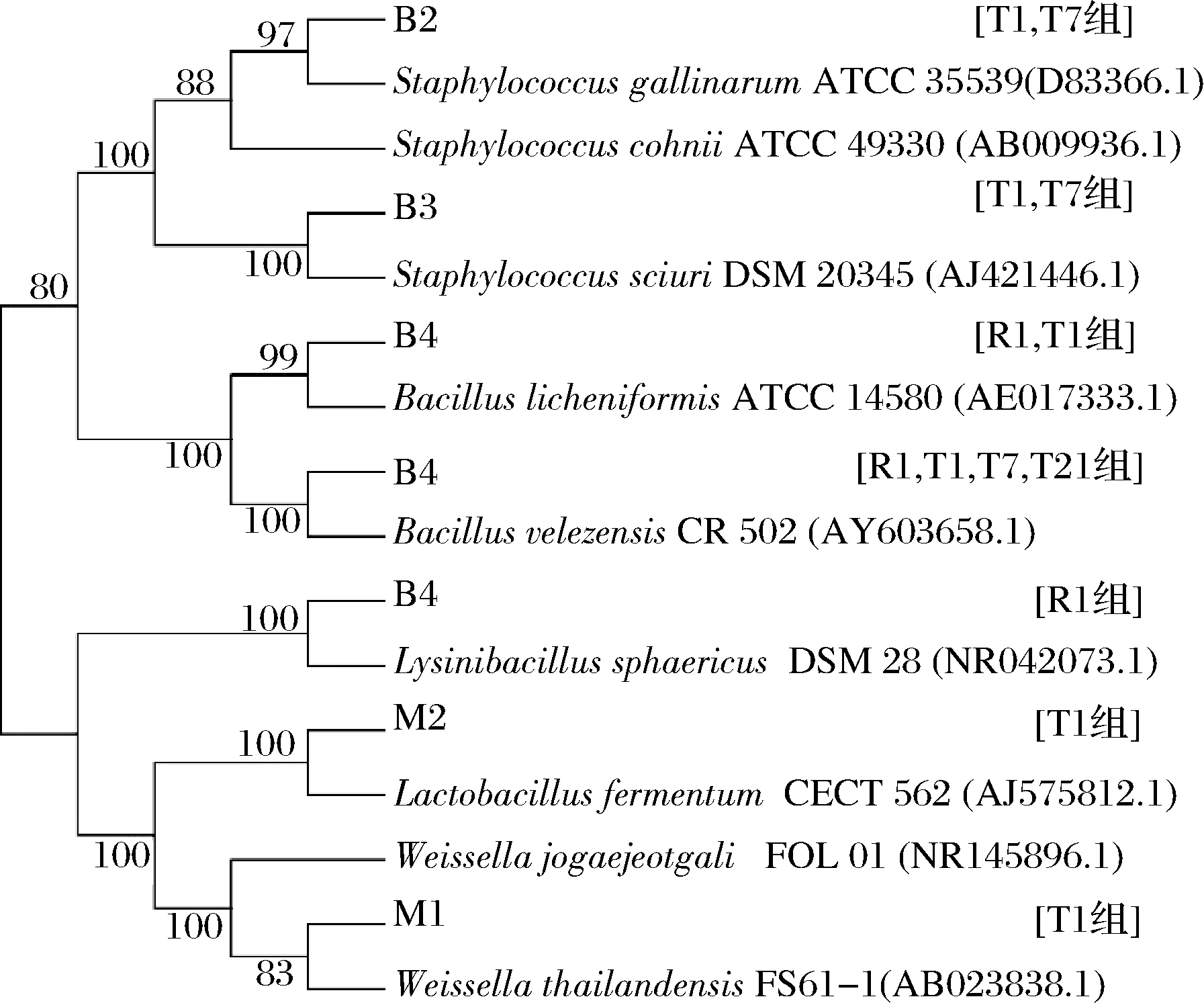
图3 豆豉样品中优势细菌的系统进化树
Fig.3 Phylogenetic tree of dominant bacteria in Douchi samples
2.3 两种豆豉发酵过程中细菌群落高通量测序的比较
2.3.1 豆豉细菌的Alpha多样性
对测序样品进行Alpha多样性分析后发现(表1),各豆豉样品的覆盖率(Coverage)>99.5%,说明测序中关键的物种信息已获得。就各项Alpha多样性指数来看,快速工艺下豆豉的Ace指数和Chao1指数整体低于传统工艺(P<0.05),说明传统工艺下细菌物种丰度即物种数量更多。而就Shannon指数与Simpson指数的分布来看,快速工艺与传统工艺则各有高低,但在传统工艺发酵第1天,两者的比值达到最大,表明此时物种多样性的程度最高。
表1 两种豆豉各样品细菌的Alpha多样性指数
Table 1 The Alpha diversity indexs of fermented bacteria in two types of Douchi samples
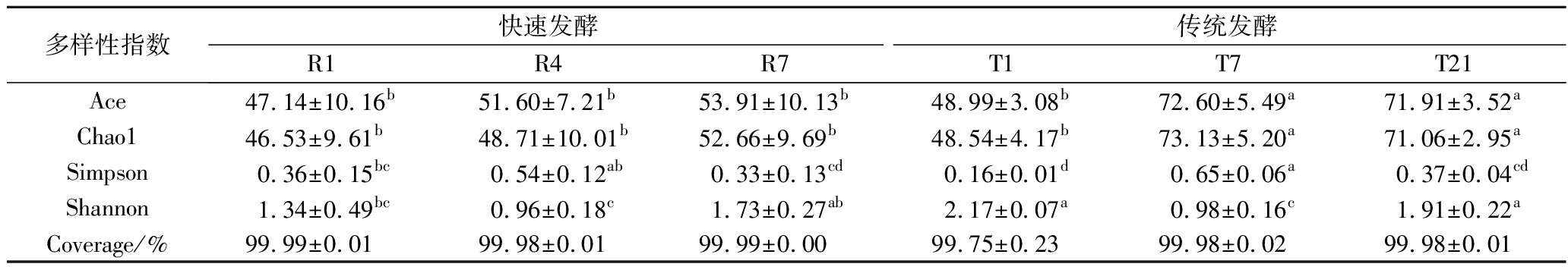
多样性指数快速发酵传统发酵R1R4R7T1T7T21Ace47.14±10.16b51.60±7.21b53.91±10.13b48.99±3.08b72.60±5.49a71.91±3.52aChao146.53±9.61b48.71±10.01b52.66±9.69b48.54±4.17b73.13±5.20a71.06±2.95aSimpson0.36±0.15bc0.54±0.12ab0.33±0.13cd0.16±0.01d0.65±0.06a0.37±0.04cdShannon1.34±0.49bc0.96±0.18c1.73±0.27ab2.17±0.07a0.98±0.16c1.91±0.22aCoverage/%99.99±0.0199.98±0.0199.99±0.0099.75±0.2399.98±0.0299.98±0.01
2.3.2 豆豉细菌的物种组成
测序数据经处理后,18个样品的测序信息共归为947个OTU,并获得由门至属水平的物种注释信息。从门水平的物种组成来看(图4),2种工艺下,厚壁菌门(Firmicutes)均是绝对优势门,虽然在传统工艺豆豉中,其占比相对更低,但最低相对丰度亦达到70.86%。其他主要门(平均相对丰度>1%)仅包括变形菌门(Proteobacteria)、放线菌门(Actinobacteria),其中快速工艺下,变形菌门的平均占比仅为1.38%,放线菌门更是均低于0.01%。相比之下,传统工艺下2门的比例明显更高,其中放线菌门在初期、中期相对丰度更高,而变形菌门更多地集中于末期,最高达到25.42%的占比。

图4 两种豆豉各样品门水平上细菌的相对丰度
Fig.4 Relative abundance of bacteria in two types of Douchi samples at phylum level
注:不同字母标注(a、b、c)表示同1个样品3组平行,图5同
具体到属水平上,2类豆豉在主要属(平均相对丰度>1%)组成及演替上表现出更大差异(图5-a)。在快速工艺豆豉中,芽孢杆菌属(Bacillus)占据绝对优势,其相对丰度为先升后降,在发酵第4天最高达96.91%;而次优势属包括赖氨酸芽孢杆菌属(Lysinibacillus)、类芽孢杆菌属(Paenibacillus)等,其中赖氨酸芽孢杆菌属在第1天最高达48.28%。就传统工艺而言,葡萄球菌属(Staphylococcus)全程占据优势,其相对丰度亦为先升后降,于发酵第7天最高达到88.82%的占比;其他次优势属主要包括乳杆菌属(Lactobacillus)、棒状杆菌属(Corynebacterium)、魏斯氏菌属(Weissella)、片球菌属(Pediococcus)等,值得注意的是,传统工艺下芽孢杆菌属的占比远不及快速工艺,其平均相对丰度仅为1.99%。另外,就2种工艺豆豉的菌属组成差异来看(图5-b),两者共有属达到38种,其中传统工艺下特有属的种类较快速发酵更多,而两者特有属的相对丰度和均未能超过5%。

a-相对丰度;b-韦恩图
图5 两种豆豉各样品属水平上细菌的分布特征
Fig.5 Distribution characteristics of bacteria in two types of Douchi samples at genus level
2.3.3 豆豉细菌结构的差异分析
未加权的PCoA表明(图6-a),PC1与PC2两主轴解释度和为75.08%,说明其已能够反映样品中大部分信息。就各样品分布来看,快速工艺与传统工艺样品相隔较远,占据着不同象限,表明两者菌群结构差异明显。而就同种豆豉来看,快速工艺样品分布整体较散,同一时期样品距离也较长,说明不同采样区域或发酵时期,其菌群结构均存在一定差别;而传统工艺样品虽整体也较为分散,但在同一时期分布较为密集,说明样品间差异更多来源于不同发酵时期之间。另外,结合Lefse分析后发现(图6-b),在属水平上,快速工艺豆豉的5种标识属多数为赖氨酸芽孢杆菌属、芽孢杆菌属等优势属,且发酵初期和末期的标志属较多;而传统工艺豆豉标志属的数目更多(9种),其中发酵初期的标志属有3种可归为乳酸菌,发酵末期更多是一些非优势属,如鞘脂杆菌属(Sphingobacterium)、柠檬酸杆菌属(Citrobacter)等。
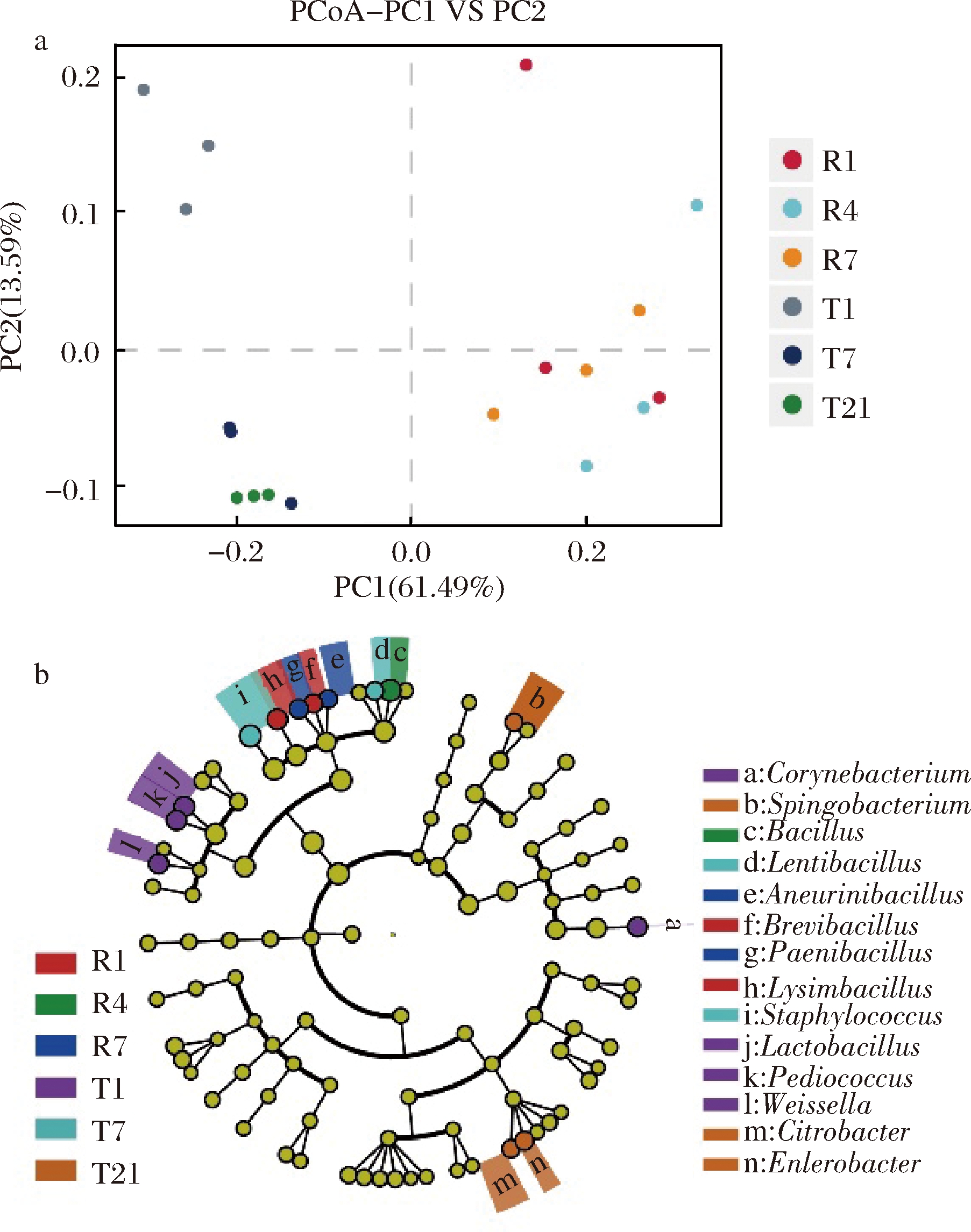
a-PcoA;b-Lefse
图6 两种豆豉各样品细菌结构的差异
Fig.6 Structural differences of bacteria in two types of Douchi samples
2.3.4 豆豉细菌基因的PICRUSt 功能预测
细菌基因功能预测的结果表明,在6类一级功能通路中(图7-a),注释到代谢(metabolism)、遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)更多,其中注释为代谢的基因占比均超过60%。与快速工艺相比,传统工艺下细菌基因注释到代谢、遗传信息处理的比例明显更高,注释到环境信息处理则明显更低(P<0.05)。在二级水平进一步注释后发现(图7-b), 47个二级功能通路中,主要的功能通路有14个(相对丰度>2%),较多注释到碳代谢(carbohydrate metabolism)、全局概览(global and overview maps)、氨基酸代谢(amino acid metabolism)等类别。就传统工艺对照来看,快速工艺下核酸代谢(nucleotide metabolism)、翻译(translation)及复制与修复(replication and repair)等通路的丰度明显更低,而关于信号转导(signal transduction)的通路丰度则明显更高(P<0.05)。
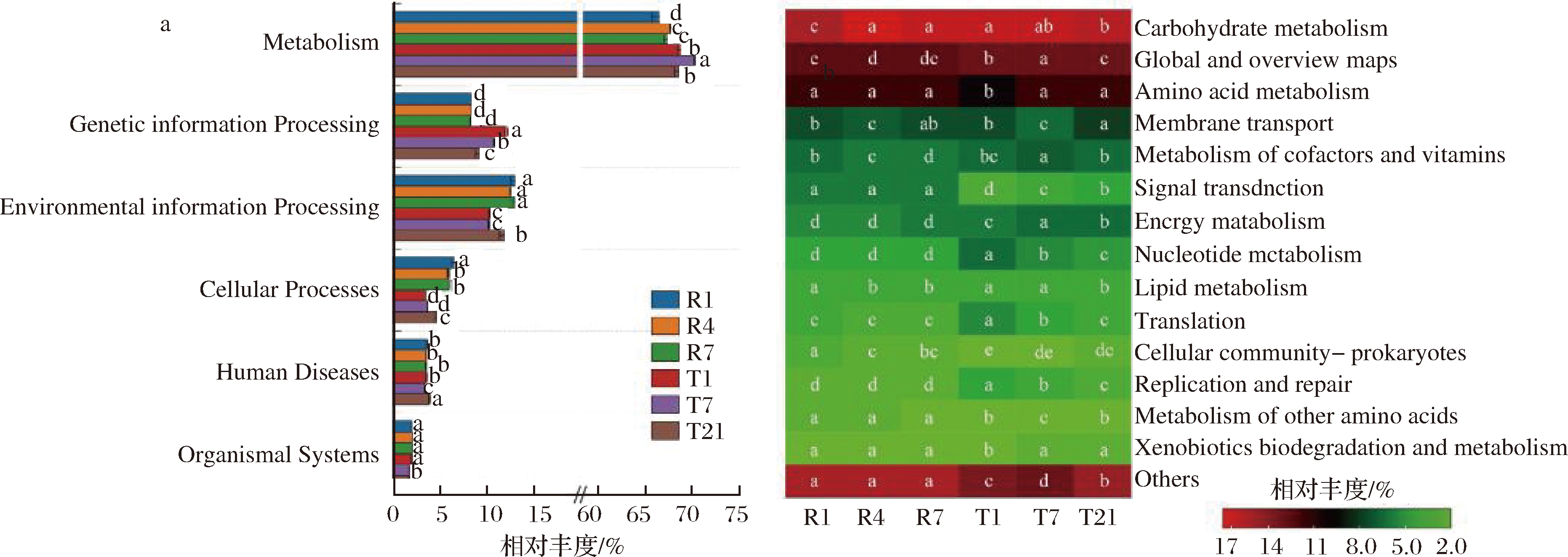
a-一级水平;b-二级水平
图7 两种豆豉各样品细菌基因功能预测的分布
Fig.7 Distribution of bacterial gene function prediction in two types of Douchi samples
3 讨论
豆豉的发酵环境可看作一种人为创设的微生物生态系统[24],即豆豉微生态。不同工艺下理化因子(温度、拌盐量、pH等)是豆豉微生态的重要组成,而豆豉微生物本质上是对发酵环境适应的体现。为此,本研究在比较2种工艺下菌群变化差异之前,针对性选取pH、含盐量、温度等理化因子进行了测定。研究发现,2种工艺下豆豉的pH在发酵中均稳步下降,其变化趋势与胡会萍等[25]报道一致。而传统工艺的含盐量约为6%,快速工艺更是不足1%,均不及阳江、永川等传统高盐豆豉[4],但2种工艺的发酵温度较上述传统高盐豆豉均明显更高并且成熟时间更短,虽然高温不利于菌群自身的生长代谢,但相对高温(50 ℃上下)可加快发酵中原料的酶促分解及化学反应速率[26-27],这或是快速工艺豆豉发酵期更短的重要原因。
可培养法的结果表明,2种工艺下菌群的总丰度均持续下降,而真菌丰度均远低于细菌丰度,其他学者[6, 28]结合可培养法分离得到更多发酵菌株,同时也发现豆豉发酵中的细菌占据绝对优势。这一方面或因制曲中的真菌经洗曲环节被大量去除,真菌较长的培养周期进一步限制其丰度的上升速率;更大可能则是在缺氧、高温的环境下,曲霉等主要真菌的增殖远不如一些细菌属活跃,由此奠定细菌在发酵中的绝对优势地位。而就分离的优势菌种来看,球形赖氨酸芽孢杆菌在以往豆豉的相关研究未见报道,但CHETTRI等[29]在当地发酵豆制品中分离到多株该菌。其他6种优势菌种在前人的分离试验中则常作为豆豉发酵优势菌存在[7, 30],且CHEN等[10]针对豆豉制曲阶段分离的细菌也有3种与本研究传统工艺一致,这进一步说明发酵中的部分优势菌或由制曲阶段带入。
就豆豉细菌的高通量测序来看,不同于部分报道对真菌、细菌均进行测序[12-13],本研究重点仅就豆豉细菌的演替展开比较。一方面是由于可培养试验已表明2种工艺下细菌丰度均占绝对优势,另一方面本课题组过去已探明豆豉发酵中真菌结构的波动较小,且优势属仅含曲霉、横梗霉2种[12]。为此,从细菌的整体组成看,高通量测序结果表明2类豆豉中共含53个细菌属,该数目远远超过可培养法中优势细菌的种类,而应用DGGE技术的相关研究仅可对豆豉优势菌株(约1%以上)进行分析[9-10]。从门水平来看,厚壁菌门在3个主要门中占据绝对优势,而厚壁菌门在以往曲霉型、细菌型豆豉的研究中也均归为强优势门[11-13],其占比大都在80%以上,且放线菌门、变形菌门亦占据较小比例,这与本文的结果大体一致。具体到属水平上,本次测序中发现的优势属在培养试验中也被归为优势细菌,说明2种探究手段的结果形成一定程度地互证。
而从2种工艺优势属的差异来看,快速工艺的特有优势属包括芽孢杆菌属、赖氨酸芽孢杆菌属、类芽孢杆菌属3种,其中芽孢杆菌属在以往豆豉的相关报道最多。如ZHANG等[31]发现甘肃两地豆豉的优势属均为芽孢杆菌属,其中陇南产地的豆豉中芽孢杆菌属的占比约为70%,这与本文快速工艺发酵末期该属的比例相近,而石聪等[13]亦发现芽孢杆菌在浏阳豆豉发酵中占据全程优势,且芽孢杆菌属在其他高温下发酵的食品也常作为优势属存在[32]。而就传统工艺而言,葡萄球菌属是唯一全程优势属,这与本课题组的研究[12]总体一致,其他学者在曲霉型豆豉[9]、细菌型豆豉[5,31]中虽也发现该属的存在,但大都为次优势菌的角色,此外,该属在其他盐分较高的发酵食品如豆酱[33]、香肠[34]中也占据优势地位;而包括魏斯氏菌属、乳杆菌属等在内的乳酸菌则广泛分布于豆豉[9,11-12]、豆酱[33]、泡菜[16]等诸多的常温发酵食品。结合主要的理化因子可以发现,除去共有的偏酸、低氧属性,快速工艺下豆豉细菌更多地面临高温胁迫,传统工艺下则是关于盐分、中高温的耐受考验。为此,2种工艺下,由于芽孢杆菌较强的高温耐受力、葡萄球菌较强的盐分耐受力,故两者分别维持着以芽孢杆菌、葡萄球菌为全程优势菌的局面,乳酸菌则仅集中于传统工艺温度较低的初期。值得注意的是,快速工艺下增殖相关的基因通路占比更少,其中发酵初期的细菌丰度及多样性更高,说明菌群在温度较低的初期更多地参与实质发酵进程。虽有研究表明部分芽孢杆菌在60 ℃以上仍具一定产酶能力[35],但高温往往可让多数菌群维持低活性或芽孢状态,表明发酵中后期或由残余酶的酶促分解及化学反应占主导,这可能不利于该工艺下豆豉风味的持续形成。因此,未来有必要结合代谢组学,对快速工艺豆豉的发酵温度、发酵周期等参数进一步改进。
[1] 赵德安.豆豉、纳豆和丹贝的简述[J].江苏调味副食品,2008,25(3):1-4.
ZHAO D A.Brief introduction of black bean sauce,natta and tempeh[J].Jiangsu Condiment and Subsidiary Food,2008,25(3):1-4.
[2] FRIEDMAN M,BRANDON D L.Nutritional and health benefits of soy proteins[J].Journal of Agricultural and Food Chemistry,2001,49(3):1 069-1 086.
[3] YUAN J,YANG J,ZHUANG Z,et al.Thrombolytic effects of Douchi fibrinolytic enzyme from Bacillus subtilis LD-8547 in vitro and in vivo[J].BMC Biotechnology,2012,12(1):36.
[4] 胡会萍.豆豉后酵中有益微生物及接菌发酵低盐豆豉品质、风味与功能性的研究[D].北京:中国农业大学,2012.
HU H P.Study on beneficial microorganisms during Douchi post-fermentation and the quality,flavor,and functionality of inoculated fermentation low-salt Douchi[D].Beijing:China Agricultural University,2012.
[5] LIU C,GONG F,LI X,et al.Natural populations of lactic acid bacteria in douchi from Yunnan Province,China[J].Journal of Zhejiang University-Science B,2012,13(4):298-306.
[6] 管泳宇,于海,葛庆丰,等.曲霉型豆豉后发酵过程的初步研究[J].食品与生物技术学报,2013,32(2):212-218.
GUAN Y Y,YU H,GE Q F,et al.Analysis of microbe on later fermentation process of Aspergillus-type-Douchi[J].Journal of Food Science and Biotechnology,2013,32(2):212-218.
[7] 胡会萍,刘丹赤,殷丽君,等.豆豉后发酵中优势菌株筛选及其生产性能[J].食品科学,2014,35(17):146-152.
HU H P,LIU D C,YIN L J,et al.Screening and production performance of predominant strains during Douchi post-fermentation[J].Food Science,2014,35(17):146-152.
[8] 李小永.细菌型豆豉后发酵期间菌相分析及产蛋白酶菌种的筛选[D].泰安:山东农业大学,2011.
LI X Y.The analysis of bacterial flora during Douchi post-fermentation and the screening of protease producing bacterial strains from Douchi[D].Taian:Shandong Agricultural University,2011.
[9] CHEN T,XIONG S,JIANG S,et al.Molecular identification of microbial community in Chinese Douchi during post-fermentation process[J].Food Science and Biotechnology,2011,20(6):1 633-1 638.
[10] CHEN T,WANG M,JIANG S,et al.Investigation of the microbial changes during koji-making process of Douchi by culture-dependent techniques and PCR-DGGE[J].International Journal of Food Science and Technology,2011,46(9):1 878-1 883.
[11] 李晓然,李洁,刘晓峰,等.利用高通量测序分析云南豆豉中细菌群落多样性[J].食品与生物技术学报,2014,33(2):137-142.
LI X R,LI J,LIU X F,et al.Analysis of bacterial community diversity in fermented soybean using pyrosequencing[J].Journal of Food Science and Biotechnology,2014,33(2):137-142.
[12] YANG L,YANG H,TU Z,et al.High-throughput sequencing of microbial community diversity and dynamics during Douchi fermentation[J].Plos One,2016,11(12):e168 166.
[13] 石聪,李世瑞,李跑,等.基于高通量测序浏阳豆豉不同发酵阶段微生物多样性分析[J].食品与发酵工业,2018,44(2):27-32.
SHI C,LI S R,LI P,et al.Analysis of microbial diversity in different fermented stages of Liuyang Douchi based on high throughput sequencing[J].Food and Fermentation Industries,2018,44(2):27-32.
[14] HE B,LI H,HU Z,et al.Difference in microbial community and taste compounds between Mucor-type and Aspergillus-type Douchi during koji-making[J].Food Research International,2019,121:136-143.
[15] VAN D E L,JASZCZYSZYN Y,NAQUIN D,et al.The third revolution in sequencing technology[J].Trends in Genetics,2018,34(9):666-681.
[16] LEE H,YOON S,KIM S,et al.Identification of microbial communities,with a focus on foodborne pathogens,during kimchi manufacturing process using culture-independent and-dependent analyses[J].LWT-Food Science and Technology,2017,81:153-159.
[17] 白飞荣,姚粟,田海霞,等.基于高通量测序和可培养方法的勐海发酵普洱茶真菌多样性分析[J].食品与发酵工业,2018,44(12):43-51.
BAI F R,YAO S,TIAN H X,et al.Fungal diversity in fermented Menhai Pu-erh tea using high throughput sequencing and culture isolations[J].Food and Fermentation Industries,2018,44(12):43-51.
[18] GB 5009.44—2016 食品安全国家标准 食品中氯化物的测定[S].北京:中国标准出版社,2016.
GB 5009.44—2016 National food safety standard Determination of chloride in food[S].Beijing:Standards Press of China,2016.
[19] 阮征,魏力,张延杰,等.高通量测序结合传统培养分析常温油沙冰皮月饼的菌群多样性[J].食品与发酵工业,2019,45(6):203-208.
RUAN ZH,WEI L,ZHANG Y J,et al.Analysis of microbial flora diversity of oil bean paste stuffing snow moon cake at room temperature by high-throughput sequencing combined with traditional culture isolation[J].Food and Fermentation Industries,2019,45(6):203-208.
[20] 伍玲,秦礼康.发酵豆制品中高产转氨酶菌株筛选及固态菌剂制备[J].食品科学,2012,33(3):105-109.
WU L,QIN L K.Screening and identification of aminotransferase-producing strain from fermented soybean products andpreparation of its solid starter culture[J].Food Science,2012,33(3):105-109.
[21] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-998.
[22] QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools[J].Nucleic Acids Research,2013,41(1):590-596.
[23] SEGATA N,IZARD J,WALDRON L,et al.Metagenomic biomarker discovery and explanation[J].Genome Biology,2011,12(6):1-18.
[24] BOKULICH N A,MILLS D A.Next-generation approaches to the microbial ecology of food fermentations[J].BMB Reports,2012,45(7):377-389.
[25] 胡会萍,程永强,袁娜,等.传统发酵豆豉含盐量及盐度对豆豉品质的影响[J].中国调味品,2011,36(8):37-41.
HU H P,CHENG Y Q,YUAN N,et al.Chinese traditional fermentation douchi salt content and impact of salt concentration on douchi qualities[J].China Condiment,2011,36(8):37-41.
[26] LEE N Y,KIM Y S,SHIN D H.Characterization of microbes for high temperature fermentation and the effect of high temperature fermentation of soy[J].Food Science and Biotechnology,2003,12(4):390-398.
[27] 朱薇玲,石豪,李金华,等.芽孢杆菌B15胞外蛋白酶和淀粉酶的酶学性质研究[J].中国酿造,2012,31(9):118-121.
ZHU W L,SHI H,LI J H,et al.Enzymatic properties of extracellular protease and amylase from Bacillus sp.B15[J].China Brewing,2012,31(9):118-121.
[28] CHEN T,WANG M,LI S,et al.Molecular identification of microbial community in surface and undersurface Douchi during postfermentation[J].Journal of Food Science,2014,79(4):M653-M658.
[29] CHETTRI R,TAMANG J P.Bacillus species isolated from tungrymbai and bekang,naturally fermented soybean foods of India[J].International Journal of Food Microbiology,2015,197:72-76.
[30] 董素琴,武晓丽,王报贵,等.豆豉中主要分离细菌对小鼠生化指标及肠道菌群的影响[J].食品工业科技,2014,35(7):343-347.
DONG S Q,WU X L,WANG B G,et al.The effect of the main bacteria isolated from Douchi on biochemical indexes and the intestinal flora in mice[J].Science and Technology of Food Industry,2014,35(7):343-347.
[31] ZHANG W,LUO Q,ZHU Y,et al.Microbial diversity in two traditional bacterial Douchi from Gansu province in northwest China using Illumina sequencing[J].Plos One,2018,13(3):e194 876.
[32] 王涛,赵东,田时平,等.宜宾浓香型白酒酿造过程中可培养细菌的系统发育多样性[J].微生物学报,2011,51(10):1 351-1 357.
WAN T,ZHAO D,TIAN SH P,et al.Phylogenetic diversity of cultivable bacteria during the brewing process of the Luzhou-flavor liquor in Yibin,Sichuan province,China[J].Acta Microbiologica Sinica,2011,51(10):1 351-1 357.
[33] WU J,TIAN T,LIU Y,et al.The dynamic changes of chemical components and microbiota during the natural fermentation process in Da-Jiang,a Chinese popular traditional fermented condiment[J].Food Research International,2018,112:457-467.
[34] WANG X,ZHANG Y,REN H,et al.Comparison of bacterial diversity profiles and microbial safety assessment of salami,Chinese dry-cured sausage and Chinese smoked-cured sausage by high-throughput sequencing[J].LWT-Food Science and Technology,2018,90:108-115.
[35] 周瑞平,王涛,陈云宗,等.偏高温大曲发酵过程中B.licheniformis和B.subtilis动态变化和生产特性[J].食品科学,2013,34(19):237-240.
ZHOU R P,WANG T,CHEN Y Z,et al.Dynamic change and production characteristics of B.licheniformis and B.subtilis during fermentation of high-temperature Daqu[J].Food Science,2013,34(19):237-240.