人体肠道内定植着1014个微生物,包括1 000多个种[1],肠道菌群组成受到基因、饮食、疾病等多种因素的影响。水苏糖天然存在于地黄或大豆中,是一种由半乳糖(α1→6)半乳糖(α1→6)葡萄糖(α1→2β)果糖组成的功能性低聚糖,具有预防龋齿、调节肠道菌群、缓解便秘、保护肝脏等功能[2-4]。水苏糖不能被人体消化吸收,会直接进入大肠,有些肠道细菌能利用水苏糖产气[5],因此摄入过量水苏糖可能会导致胀气等不良反应。
目前关于肠道菌利用水苏糖的相关研究较少。HAYAKAWA等在1990年研究大豆低聚糖对人类肠道菌群的影响时发现,实验所用的125株肠道细菌中,包括双歧杆菌属、乳杆菌属、多形杆状菌属、光岗菌属、巨单胞菌属、梭状芽孢杆菌属、克雷伯氏菌属在内的7个属的58株菌能够利用水苏糖[6]。WAGNER等[7]发现给大鼠饲喂水苏糖后会产H2,但未研究是哪些菌利用水苏糖产气;ZHONG等[8]发现水苏糖可以促进嗜酸乳杆菌CICC22162的生长;水苏糖与棉籽糖结构类似,从自然发酵的绿桌橄榄中分离到的乳酸菌几乎都能利用棉籽糖,其中81%的菌株也能发酵水苏糖[9]。
本实验从健康人粪便中筛选能够利用水苏糖的细菌,并结合培养液上清液中糖的组成、菌株产气情况、酶活以及基因组草图分析等,研究菌株对水苏糖的利用特性,对于水苏糖与人体健康关系的研究具有一定的指导意义。
1 材料与方法
1.1 实验试剂
水苏糖,上海源叶生物科技有限公司;棉籽糖,生工生物工程(上海)股份有限公司;酵母提取物、胰蛋白胨,英国Oxoid公司。
改良肠道微生物培养基(gut microbical culture medium,GMM):水苏糖8 g,胰蛋白胨4 g,KH2PO4 2 g,酵母提取物2 g,NaCl 0.08 g,CaCl2 0.008 g,MgSO4·7H2O 0.002 g,FeSO4·7H2O 0.73 mg,吐温-80 0.5 mL,L-半胱氨酸盐酸盐1 g,ATCC Trace Mineral Mix 10 mL,ATCC Vitamin Mix 10 mL。固体培养基在此基础上加入1.5%~2.0%(质量分数)的琼脂粉,并添加15 mL 0.5%(质量分数)的溴甲酚紫作为指示剂。
1.2 利用水苏糖的肠道菌的分离与鉴定
采集2名健康成年人的新鲜粪便样本,并将样本悬浮在0.9%(质量分数)的生理盐水中;梯度稀释,将样品涂布在以水苏糖作为唯一碳源、并添加溴甲酚紫指示剂的GMM固体培养基上[10],置于厌氧工作站37 ℃培养24~48 h;挑取能使培养基变黄的菌落,转接到GMM固体培养基上,划线分离、纯化菌株,并对菌株进行16S rRNA测序鉴定。
1.3 细菌培养上清液中水苏糖的高效液相色谱分析
菌株在以水苏糖作为唯一碳源的GMM培养基中37 ℃厌氧培养24 h,以8 000×g离心5 min收集上清液,采用高效液相色谱仪Water 1525测定上清液中水苏糖的含量,外标法定量。色谱条件:采用SugarPak1糖柱(300 mm,id 3.5 μm),柱温85 ℃,流动相为纯水,流量为0.4 mL/min。
1.4 细菌利用水苏糖生长的表征
将菌株接种于以水苏糖作为唯一碳源的GMM培养基中,37 ℃厌氧培养,每隔1 h取样,测定菌液的pH值和600 nm处的吸光度(OD600)。同时取1 mL待测菌液于1.5 mL离心管以8 000×g离心5 min,收集上清液备用。当菌株生长进入稳定期后,停止实验。
1.5 α-半乳糖苷酶活力测定
α-半乳糖苷酶活力测定主要参照SCALABRINI等[11]的方法:经8 000×g离心5 min得菌泥和上清液,其中菌泥经柠檬酸-柠檬酸钠缓冲液洗涤2次后重悬,与100 μL氯仿混合,振荡破碎,制成粗酶液。将250 μL粗酶液或上清液与对硝基苯α-D-氨基半乳糖苷混合,在37 ℃下反应30 min,加入0.2 mol/L 500 μL Na2CO3溶液终止反应,测定420 nm下的吸光值。菌泥测定结果对应胞内酶活,上清液测定结果对应胞外酶活。
酶活定义:在测定条件下,1 min释放1 μmol的对硝基苯酚所消耗的酶量(mL)为1个酶活力单位。
1.6 产气实验
将菌株以3%的接种量接种于装有倒置杜氏小管的水苏糖GMM培养基中,37 ℃厌氧培养24~48 h,观察产气情况并记录。
1.7 基因组草图的测定
将菌株以4%的接种量接种至5 mL MRS液体培养基中进行活化,置于厌氧工作站37 ℃恒温培养24 h。活化2次的菌株以4%的接种量接种至装有80 mL MRS液体培养基的蓝盖瓶中,置于厌氧工作站培养24~48 h。之后将菌液以8 000×g离心10 min得到菌泥,送至上海美吉生物医药科技有限公司进行基因组草图测序。
采用Illumina HiSeq 2000(Illumina Inc USA)[11]测序平台对送测菌株的基因组进行双末端测序,获得原始数据即大于基因组100倍的PE150测出数据量。然后对其碱基质量、碱基错误率、碱基分布进行质量控制,对低质量reads和adapter进行质量剪切,得到高质量的读长数据。上述步骤均由上海美吉生物医药科技有限公司完成。
使用短序列组装软件SOAPdenovo2进行从头组装[12],调试不同K-mer值,选取N50较长和Scaffold数量较小的K-mer值进行拼接,同时调节-u、-r、-R、-F等参数,获得初步组装结果。基于reads的paired-end和overlap关系,将reads对比到contigs上,获得最终组装结果。调用Gap Closer软件填补contigs间的内部空白[13],过滤500 bp读长以下的结果,获得完整序列。
采用GeneMark1.1[14]和Glimmer3.02[15]软件预测送测菌株草图序列中的蛋白质编码基因,已默认参数获取开放阅读框(open reading frame,ORF)的相关信息。在预测得到的编码基因基础上进行基因的功能注释,将草图基因的蛋白质序列与各大数据库进行对比,保留前5条序列相似性较高的结果,获得相应的功能注释信息,以此初步确定该蛋白序列与数据库中的蛋白质具备相似的功能。上述步骤均由上海美吉生物医药科技有限公司完成。本实验主要使用的是京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)的数据库,该数据库包括了完整或部分测序基因组的序列、细胞生化过程的图解以及酶分子、酶反应等信息[16]。
1.8 SignalP 5.0预测信号肽
在SignalP 5.0软件中输入菌株基因片段的氨基酸或核酸序列,选择物种(革兰氏阳性菌或革兰氏阴性菌)后,系统将对其进行信号肽预测。结果以图表形式呈现,根据出现Sec信号肽(Sec/SPI)、脂蛋白信号肽(Sec/SPII)、Tat信号肽(Tat/SPI)或无信号肽(other)的概率来确定测试基因片段是否存在信号肽。
1.9 数据分析
实验中pH值、OD600、糖含量与酶活力的测定均为3次平行,结果以平均值±标准差表示,采用Origin 9.0作图。
2 结果与分析
2.1 能利用水苏糖的肠道细菌的分离与鉴定
肠道细菌利用水苏糖会产酸,使指示剂变色。根据指示剂变色结果,并结合16S rRNA测序鉴定,从粪便样品中初步鉴定出23株能利用水苏糖的细菌,包括6个属、8个种(表1)。
表1 健康成年人粪便中分离到的能利用水苏糖的细菌
Table 1 The intestinal bacteria capable of utilizing stachyose isolated from the feces of healthy adults

属种初筛菌株克雷伯氏菌属(Klebsiella)肺炎克雷伯氏菌(Klebsiella pneumoniae)A3, A4, A9,A16肠球菌属(Enterococcus)粪肠球菌(Enterococcus faecalis)A12,A13,A36,B7,B30, B44,B54,B8H屎肠球菌(Enterococcus faecium)A6,A22,B8B鸟肠球菌(Enterococcus avium)A43,A48梭状芽孢杆菌属(Clostridium)产气荚膜梭状芽孢杆菌(Clostridium perfringens)B1,B15双歧杆菌属(Bifidobacterium)长双歧杆菌(Bifidobacterium longum)B39埃希氏菌属(Escherichia)大肠杆菌(Escherichia coli)B16,B34链球菌属(Streptococcus)唾液链球菌(Streptococcus salivarius)B9
注:A、B代表2份成人粪便样品(下同)
由于实验所使用的水苏糖纯度仅为85%,试剂中含有的其他成分也可能被菌株利用,因此进一步采用高效液相色谱法测定菌株上清液中水苏糖的含量,根据水苏糖的消耗量确定能够利用水苏糖的细菌。结果表明,实际能利用水苏糖的细菌有12株,包括3个属、4个种,分别为肺炎克雷伯氏菌、屎肠球菌、粪肠球菌和产气荚膜梭状芽孢杆菌(表2)。
表2 细菌培养上清液中水苏糖含量的测定 单位:g/L
Table 2 Determination of stachyose in the supernatant of bacterial culture
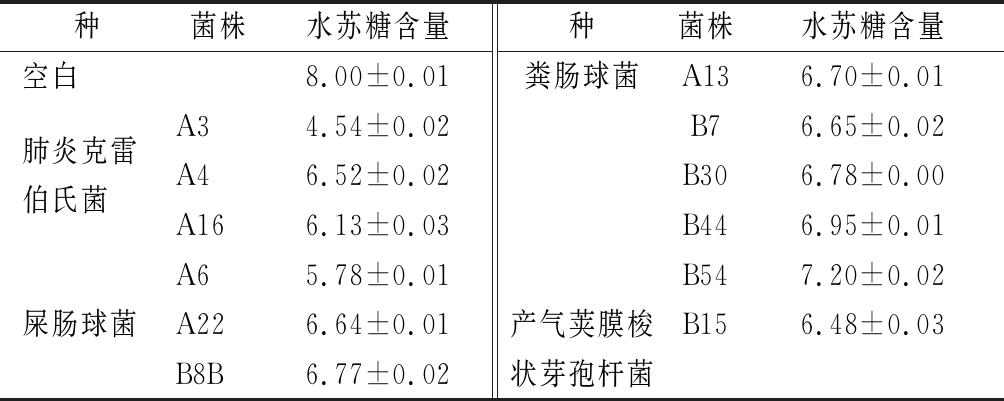
种菌株水苏糖含量种菌株水苏糖含量空白8.00±0.01粪肠球菌 A136.70±0.01肺炎克雷A34.54±0.02B76.65±0.02伯氏菌A46.52±0.02B306.78±0.00A166.13±0.03B446.95±0.01A65.78±0.01B547.20±0.02屎肠球菌A226.64±0.01产气荚膜梭B156.48±0.03B8B6.77±0.02状芽孢杆菌
已有文献报道短双歧杆菌、发酵乳杆菌、罗伊氏乳杆菌、肺炎克雷伯氏菌等多种菌株能够利用水苏糖[17-18],而屎肠球菌、粪肠球菌和产气荚膜梭状芽孢杆菌利用水苏糖的研究为首次报道。在BARBARA等[18]的实验中,屎肠球菌并不能利用水苏糖,而本实验成功筛选到多株能够利用水苏糖生长的屎肠球菌(A6、A22、B8B)。
2.2 肠道菌在GMM培养基中的生长曲线
为研究筛选出来的肠道菌对水苏糖的利用情况,从4种菌中各选1株测定其在以水苏糖作为唯一碳源的GMM培养基中的生长曲线(图1)。各菌株以相同的接种量、相同的培养条件进行生长,呈现出不同的生长情况。肺炎克雷伯氏菌A3生长最快,在3 h左右进入稳定期,培养至5 h结束实验;屎肠球菌A6、粪肠球菌A13、产气荚膜梭状芽孢杆菌B15在培养5 h后进入稳定期,培养至8 h结束实验。进入稳定期后,4株菌的上清液pH值为4.40~5.40,A6、A13和B15的OD600 达到1.30以上。

A-肺炎克雷伯氏菌A3;B-屎肠球菌A6;C-粪肠球菌A13;D-产气荚膜梭状芽孢杆菌B15
图1 菌株在以水苏糖作为唯一碳源的GMM培养基中的生长曲线
Fig.1 The growth curves of the strains in GMM medium with stachyose as the sole carbon source
2.3 肠道菌培养上清液中水苏糖及棉籽糖含量测定
由图1可知,不同菌株对水苏糖的利用情况不同。采用高效液相色谱跟踪测定菌株培养上清中的水苏糖和棉籽糖含量,发现所有菌株的上清液中水苏糖的含量随时间增长而减少(表3、表4),而棉籽糖含量的变化并不相同。其中,屎肠球菌A6和粪肠球菌A13的棉籽糖含量逐渐增加;而产气荚膜梭状芽孢杆菌B15上清液中的棉籽糖含量逐渐减少;肺炎克雷伯氏菌A3生长较快,棉籽糖含量呈现先增加后减少的趋势(表4)。
表3 不同时间菌株上清液中残留糖含量 单位:%
Table 3 Residual sugar content in strain supernatant at different times
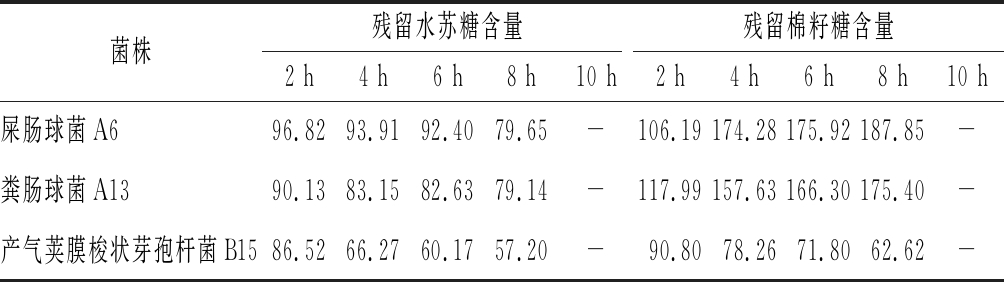
菌株残留水苏糖含量残留棉籽糖含量2 h4 h6 h8 h10 h2 h4 h6 h8 h10 h屎肠球菌A696.8293.9192.4079.65-106.19174.28175.92187.85-粪肠球菌A1390.1383.1582.6379.14-117.99157.63166.30175.40-产气荚膜梭状芽孢杆菌B1586.5266.2760.1757.20-90.8078.2671.8062.62-
注:残留水苏糖含量![]() 棉籽糖同理;“-”代表菌株生长已结束,未进行后续测定(下同)
棉籽糖同理;“-”代表菌株生长已结束,未进行后续测定(下同)
表4 不同时间肺炎克雷伯氏菌A3上清液中残留糖含量 单位:%
Table 4 Residual sugar content in Klebsiella pneumoniae A3 supernatant at different times

菌株残留水苏糖含量残留棉籽糖含量1 h2 h3 h4 h5 h1 h2 h3 h4 h5 h肺炎克雷伯氏菌A383.9083.8680.3779.0378.48101.62163.77152.4392.0290.15
2.4 肠道菌α-半乳糖苷酶活力测定
细菌水解水苏糖需要α-半乳糖苷酶,根据酶所处位置(胞外、胞内),可以进一步推断其利用机制。选择稳定期的菌株进行酶活测定,由表5可知,肺炎克雷伯氏菌A3具有较高的胞外α-半乳糖苷酶活力,是胞内酶活力的20倍;屎肠球菌A6、粪肠球菌A13的胞内酶活力较高,是胞外酶活力的30~40倍;产气荚膜梭状芽孢杆菌B15的胞内酶活力也较胞外酶活力高。由此可见,肺炎克雷伯氏菌A3的α-半乳糖苷酶以胞外为主,其余3株菌的α-半乳糖苷酶以胞内为主。α-半乳糖苷酶大多存在于细菌细胞内,除肺炎克雷伯氏菌A3外,实验结果基本与报道符合[19-20]。
表5 α-半乳糖苷酶酶活力测定 单位:U/L
Table 5 Determination of α-galactosidase enzyme activity

菌株胞内酶活力胞外酶活力肺炎克雷伯氏菌A30.004±0.0010.080±0.01屎肠球菌A60.080±0.010.001±0.003粪肠球菌A130.046±0.0050.002±0.001产气荚膜梭状芽孢杆菌B150.015±0.0050.002±0.001
2.5 菌株利用水苏糖产气情况
水苏糖进入人体后不被小肠消化吸收,可以达到大肠被肠道细菌发酵。摄入高剂量的水苏糖,可能会出现肠胃胀气、腹泻、消化不良等不适反应[21-22]。我们对筛选出的12株菌进行了产气实验,实验结果如表6所示,5株菌能够利用水苏糖产气,分别为肺炎克雷伯氏菌A3、A4、A16、屎肠球菌B8和产气荚膜梭状芽孢杆菌B15,而其余7株不能产气。其中,产气荚膜梭状芽孢杆菌B15产气最明显,杜氏小管被气泡充满。气体的产生能为产气荚膜梭状芽孢杆菌提供一个良好的厌氧环境,有助于它的生长[23]。
表6 不同菌株在以水苏糖作为唯一碳源的GMM培养基中的 产气情况
Table 6 Gas production of different strains in GMM medium with stachyose as the sole carbon source

菌株产气情况肺炎克雷伯氏菌A3, A4, A16+粪肠球菌A13, B7, B30, B44, B54-屎肠球菌A6, A22-屎肠球菌B8+产气荚膜梭状芽孢杆菌A15+
注:“+”表示产气;“-”表示不产气
肠道中的气体主要有N2、O2、CO2、H2、CH4以及各种痕量气体(如H2S、NO等)[24]。肠道菌发酵糖类产生的气体主要有两种:一是CO2,由戊糖磷酸途径代谢6-磷酸葡萄糖产生;二是H2,由丙酮酸脱羧过程产生。丙酮酸脱羧产氢也可分为梭状芽孢杆菌型和肠杆菌型[25]。在梭状芽孢杆菌型中,丙酮酸脱羧后形成焦磷酸硫胺素(thiamine pyrohosphate,TPP)-酶复合物,该复合物将电子转移给铁氧还蛋白(ferredoxin,Fd),还原型的Fd被氢化酶氧化并产生H2;在肠杆菌型中,丙酮酸转化为甲酸,通过甲酸裂解以及Fd氧化释放CO2和H2[26]。研究实验菌株产气所涉及的酶系,需要结合基因组数据进行分析。
2.6 基因组草图分析
对屎肠球菌A6、粪肠球菌A13、产气荚膜梭状芽孢杆菌B15、肺炎克雷伯氏菌A3进行基因组草图测序,4株菌的基因组草图数据已经上传至NCBI的SRA数据库,结果如表7所示。屎肠球菌和粪肠球菌的GC含量均在38%左右,与文献报道相符[27-28]。产气荚膜梭状芽孢杆菌B15的GC含量约为39%,基因数量最少;肺炎克雷伯氏菌A3在4株菌中GC含量最高。
表7 细菌的基因组草图信息
Table 7 Bacterial genome sketch information

菌株基因数量序列大小/MbpGC比例/%Scaffold/总数Scaffold N50/bp登录号屎肠球菌A65 3324.3738.3718289 484SRR12123670粪肠球菌A135 3854.5438.57145178 993SRR12123705产气荚膜梭状芽孢杆菌B152 6482.1639.1083128 544SRR12124348肺炎克雷伯氏菌A34 7314.0659.35132198 214SRR12123698
送测基因组草图的菌株中,肺炎克雷伯氏菌A3和产气荚膜梭状芽孢杆菌B15均能利用水苏糖产气。已有研究表明,肺炎克雷伯氏菌等兼性厌氧菌在产气过程中,通过丙酮酸甲酸裂解酶将丙酮酸转化为甲酸,再由甲酸裂解酶系统的多酶复合物分解甲酸产氢[29]。一般来说,甲酸裂解酶是由甲酸脱氢酶-H、氢化酶以及电子载体组成的复合物[30]。甲酸裂解酶-H的大亚基由fdh基因编码表现出催化性质,而其余部分是由hcyBCDEFG编码起还原氢分子的作用[31];hypABCDE编码合成氢化酶[32];fhlA作为决定性基因,是中央调节因子[33]。肺炎克雷伯氏菌A3的基因组草图数据中能够找到上述相关基因片段(图2)。
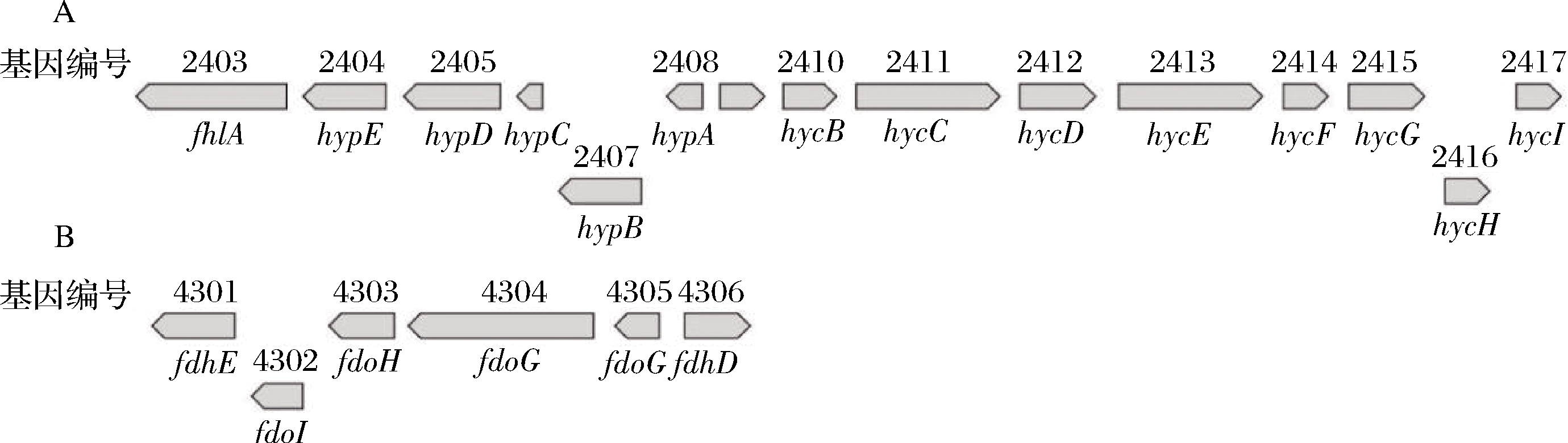
A-氢化酶以及电子载体部分基因片段;B-甲酸脱氢酶-H大亚基部分基因片段
图2 肺炎克雷伯氏菌A3中与产气相关的基因簇
Fig.2 Gene clusters related to gas production in Klebsiella pneumoniae A3
注:图谱上箭头的长度和方向分别代表了基因的长度和编码方向;如果出现基因上下错开的情况,说明该基因与上游或下游基因有重叠(下同)
产气荚膜梭状芽孢杆菌产氢途径是梭状芽孢杆菌型,产氢过程主要涉及丙酮酸脱氢酶复合物。在铜绿假单胞菌中该复合物由调节基因(pdhR),丙酮酸脱氢酶E1(aceE),二氢脂酰胺乙酰转移酶E2(aceF)和二氢脂酰胺脱氢酶E3(lpd)构成[34]。产气荚膜梭状杆菌B15存在类似的基因片段(图3),由丙酮酸脱氢酶E1的α-亚基(pdhA)、丙酮酸脱氢酶E1的β-亚基(pdhB),二氢脂酰胺乙酰转移酶E2(aceF)和二氢脂酰胺脱氢酶E3(lpd)构成。丙酮酸脱氢酶复合物在TPP作用下,将丙酮酸转化为乙酰辅酶A并释放CO2,并在由por基因编码的丙酮酸铁氧还蛋白还原酶作用下将Fe3+转化为Fe2+,Fe2+又被氢化酶氧化释放H2。
图3 产气荚膜梭状芽孢杆菌B15中丙酮酸脱氢酶 复合物基因片段
Fig.3 Gene fragment of pyruvate dehydrogenase complex in Clostridium perfringens B15
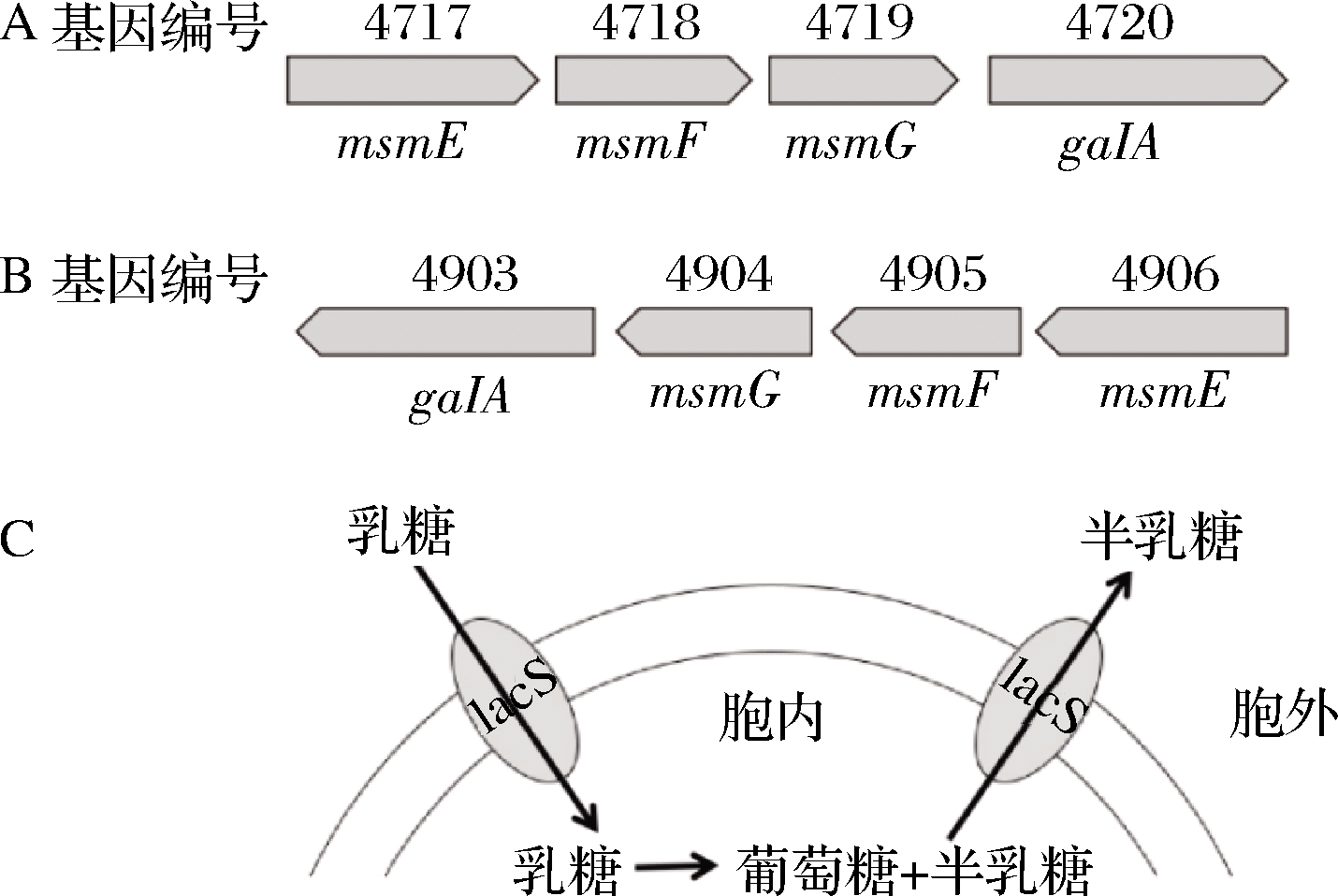
菌株彻底代谢水苏糖需要α-半乳糖苷酶、β-呋喃果糖苷酶以及相关转运蛋白[35]。ALEXANDER等[36]研究了变异链球菌中多糖代谢(msmEFGK)转运体,拥有msmEFGK转运体的变异链球菌能够转运代谢蜜二糖、棉籽糖、水苏糖、异麦芽糖、异麦芽糖醇(图4),缺失msmE基因时,菌株不能利用棉籽糖或水苏糖。aga、galA、melA等基因能够编码合成α-半乳糖苷酶参与水苏糖的水解,当水苏糖被α-半乳糖苷酶水解后2个半乳糖就生成了蔗糖[35,37]。据报道,代谢蔗糖的基因簇由5个基因组成,包括sacK(果糖激酶)、PTS(PTS转运系统)、sacA(β-呋喃果糖苷酶)、sacR(转录调节蛋白)以及agl(α-葡萄糖苷酶)[38]。
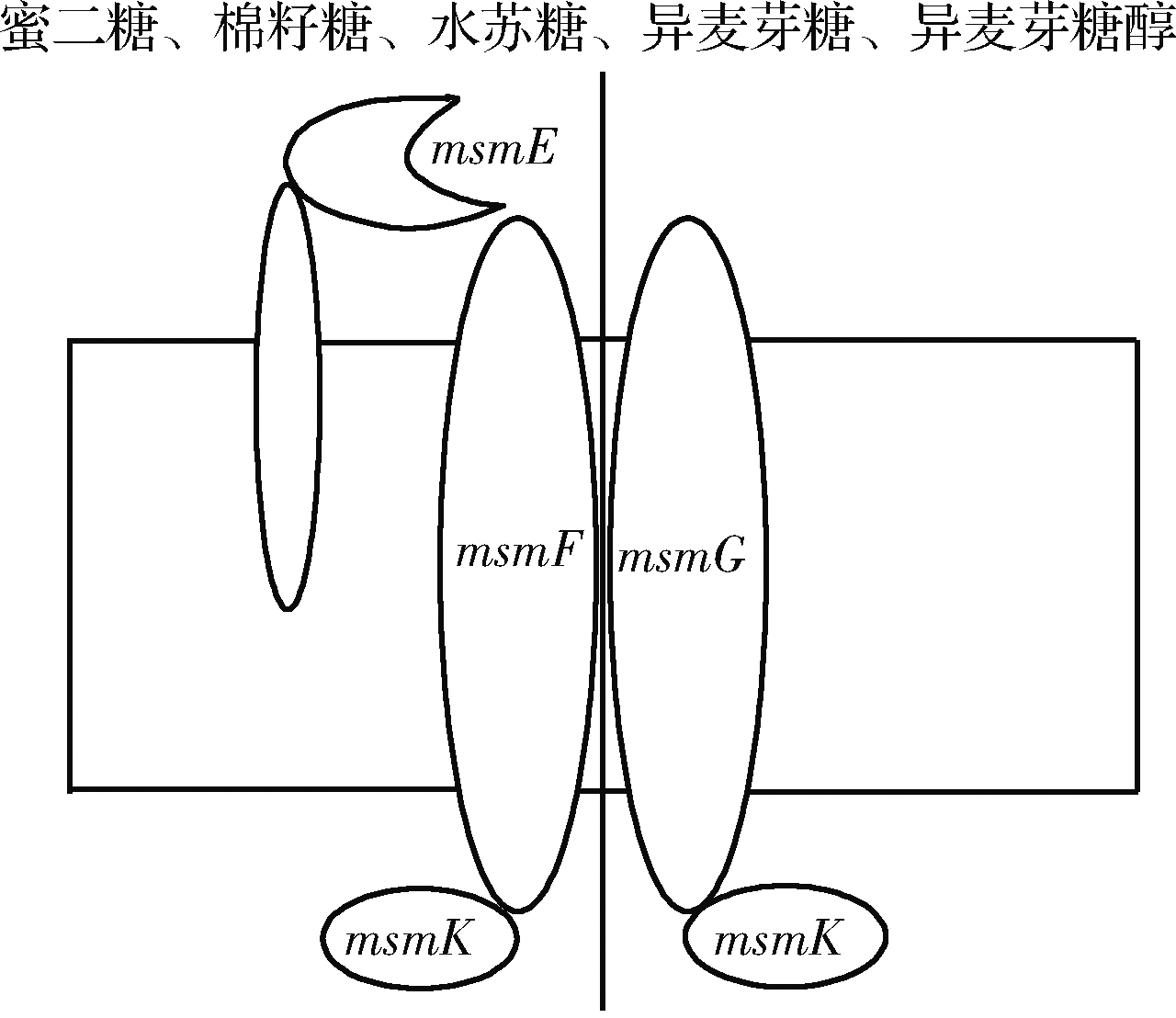
图4 变异链球菌msmEFGK转运体摄取碳水化合物
Fig.4 Carbohydrate uptake by msmEFGK transporters in Streptococcus mutans
通过分析菌株的基因组草图,发现产气荚膜梭状芽孢杆菌B15中与水苏糖利用相关的基因与变异链球菌相似[36](图4)。与变异链球菌相比,产气荚膜梭状芽孢杆菌B15缺少msmK基因,但转运水苏糖的功能并未受到影响,因此依靠msmEFG基因也能转运水苏糖。产气荚膜梭状芽孢杆菌B15利用msmEFG转运体转运水苏糖,由galA基因编码的α-半乳糖苷酶将其水解为2分子半乳糖和1分子蔗糖。由dexB(α-葡萄糖苷酶)、scrA(PTS系统)、sacA(β-呋喃果糖苷酶)、scrR(转录调节因子)、scrK(果糖激酶)构成的基因簇负责水解蔗糖并用于后续代谢(图5)。
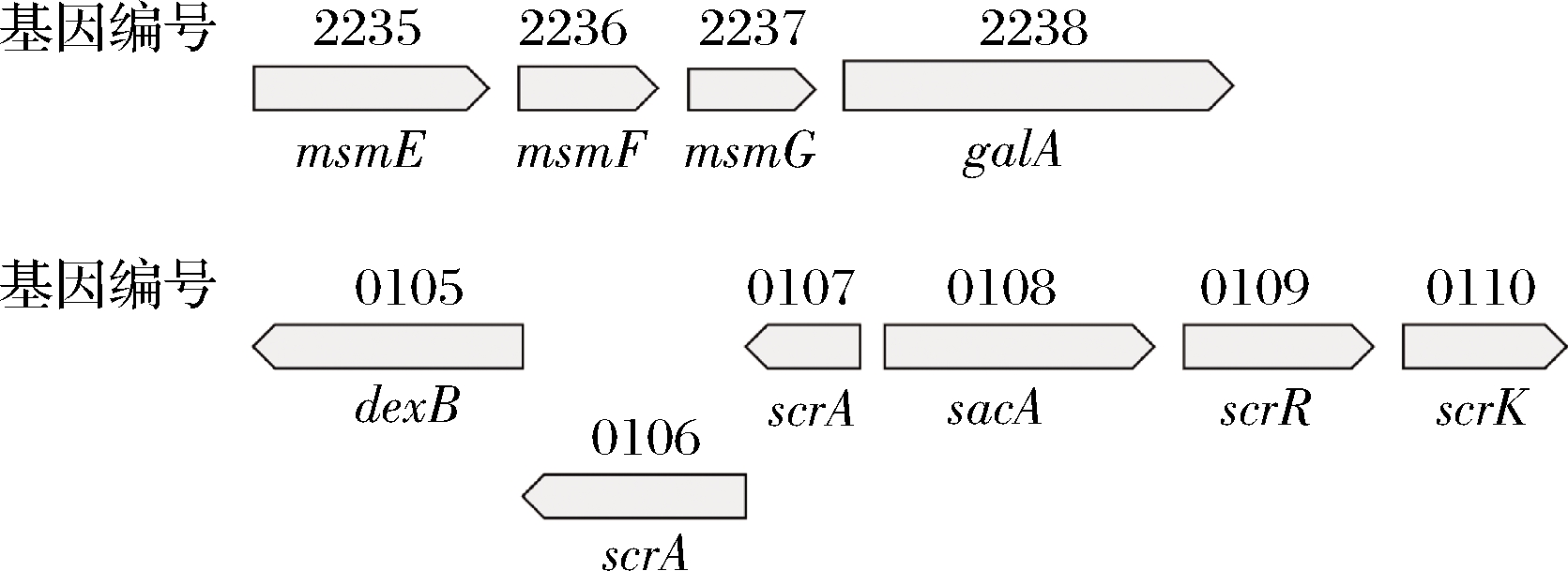
图5 产气荚膜梭状芽孢杆菌B15中与水苏糖利用 相关的基因簇
Fig.5 Gene clusters related to stachyose utilization in Clostridium perfringens B15
屎肠球菌A6和粪肠球菌A13在糖组成分析、α-半乳糖苷酶活力测定以及与水苏糖代谢相关的基因方面均呈现出一定的相似性(图6-A、图6-B),表明2株菌可能采用相同的方式利用水苏糖。通过查阅《伯杰氏系统细菌学手册》(《Bergey′s Manual of Systematic Bacteriology》),发现屎肠球菌和粪肠球菌不能利用棉籽糖[39]。高效液相色谱结果显示屎肠球菌A6和粪肠球菌A13在生长过程中上清液中的棉籽糖含量增加(表3),表明屎肠球菌A6和粪肠球菌A13均不能利用棉籽糖,这种现象与保加利亚乳杆菌利用乳糖的情况类似。保加利亚乳杆菌中存在乳糖/半乳糖逆向转运系统(图6-C),乳糖通过lacS基因编码的乳糖通透酶进入细胞内被水解成葡萄糖和半乳糖,葡萄糖被代谢利用,而游离的半乳糖则被释放到细胞外[40]。屎肠球菌A6、粪肠球菌A13都有一段连续的galA、msmE、msmF、msmG基因,可能通过msmEFG转运体将水苏糖转运胞内,由galA基因编码的α-半乳糖苷酶将水苏糖水解为半乳糖和棉籽糖,再将不能利用的棉籽糖释放到细胞外。而负责转运棉籽糖至胞外的转运蛋白,目前尚不清楚,需要进一步研究。
A-屎肠球菌A6;B-粪肠球菌A13;C-乳糖/半乳糖逆向转运系统
图6 屎肠球菌A6、粪肠球菌A13中水苏糖利用 相关基因及保加利亚乳杆菌中乳糖/半乳糖逆向转运系统
Fig.6 Gene clusters related to stachyose utilization in Enterococcus faecium A6 Enterococcus faecalis A13 and lactose/galactose reverse transport system in Lactobacillus bulgaricus
图谱上箭头的长度和方向分别代表了基因的长度和编码方向
根据α-半乳糖苷酶酶活力测定结果(表5),肺炎克雷伯氏菌A3可能在胞外水解水苏糖,但是分析基因组草图并未发现能将α-半乳糖苷酶释放至胞外的转运蛋白基因(图7)。利用SignalP 5.0对菌株基因片段中的信号肽进行预测,发现α-半乳糖苷酶的基因片段中并无信号肽,无法产生分泌蛋白(表8)。因此,肺炎克雷伯氏菌A3是否能在胞外水解水苏糖还有待验证。

图7 肺炎克雷伯氏菌A3中α-半乳糖苷酶及 β-呋喃果糖苷酶所在基因簇
Fig.7 Gene clusters of α-galactosidase and β-fructofuranosidase in Klebsiella pneumoniae A3
综上所述,初步判断菌株对水苏糖可能的利用方式为:
(1)产气荚膜梭状芽孢杆菌B15通过msmEFG转运体将水苏糖转运至胞内,然后由胞内α-半乳糖苷酶和β-呋喃果糖苷酶水解成单糖进行后续利用;
(2)屎肠球菌A6和粪肠球菌A13利用msmEFG转运体转运水苏糖,α-半乳糖苷酶将其水解为半乳糖和棉籽糖,通过类似乳糖/半乳糖逆向转运系统的方式,将不利用的棉籽糖释放到细胞外;
(3)肺炎克雷伯氏菌A3对水苏糖的利用方式目前尚不明确,仍需要进一步研究验证。
表8 肺炎克雷伯氏菌A3中α-半乳糖苷酶信号肽预测结果
Table 8 Prediction results of α-galactosidase signal peptide in Klebsiella pneumoniae A3

基因基因编号Sec信号肽(Sec/SPI)脂蛋白信号肽(Sec/SPII)Tat信号肽(Tat/SPI)无信号肽(other)melA32560.017 70.001 30.000 80.980 2galA43090.022 00.002 10.001 10.974 8
3 结论
本实验从粪便中成功筛选到12株能够利用水苏糖的肠道细菌,分别属于肺炎克雷伯氏菌、屎肠球菌、粪肠球菌和产气荚膜梭状芽孢杆菌。
不同菌株利用水苏糖的生长情况不同,利用水苏糖的方式也存在差异。大部分菌株以胞内α-半乳糖苷酶为主,肺炎克雷伯氏菌以胞外α-半乳糖苷酶为主,12株菌中仅肺炎克雷伯氏菌A3、A4、A16、屎肠球菌B8和产气荚膜梭状芽孢杆菌B15能够利用水苏糖产气。不同菌株的产气方式也不同,肺炎克雷伯氏菌A3通过丙酮酸甲酸裂解酶将丙酮酸转化为甲酸,再由甲酸裂解酶分解甲酸产气;产气荚膜梭状芽孢杆菌B15利用丙酮酸脱氢酶复合物产生CO2。
菌株对水苏糖的利用可能存在以下2种方式:(1)利用msmEFG转运体将水苏糖转运至胞内,由α-半乳糖苷酶和β-呋喃果糖苷酶将其水解利用;(2)利用msmEFG 转运体转运水苏糖,α-半乳糖苷酶将其水解为半乳糖和棉籽糖,将不利用的棉籽糖释放到细胞外。
本实验筛选得到了12株能利用水苏糖的肠道细菌,并采用高效液相色谱技术结合基因组学分析,研究了菌株对水苏糖的利用方式。今后可以更深入地研究肠道细菌对水苏糖的利用机制,揭示摄入过量水苏糖引起人体不适的机理,对于探究水苏糖与人体健康的关系具有一定的指导意义。
[1] LI T, LU X S, YANG X B.Evaluation of clinical safety and beneficial effects of stachyose-enriched α-galacto-oligosaccharides on gut microbiota and bowel function in humans [J].Food & Function, 2017, 8(1):262-269.
[2] CLARK J A, COOPERSMITH C M.Intestinal crosstalk-a new paradigm for understanding the gut as the “motor” of critical illness [J].Shock, 2007, 28(4):384-393.
[3] 雷金艳, 贾建伟.地黄中水苏糖的研究进展[J].医学综述, 2010, 16(6):935-937.
LEI J Y, JIA J W.Research progress in the stachyose in Rehmannia glufimom[J].Medical Recapitulate, 2010, 16(6):935-937.
[4] 高鹏, 刘薇薇, 杨建武.水苏糖的保健功能及其在饮料工业中的应用[J].饮料工业, 2012, 15(2):15-19.
GAO P, LIU W W, YANG J W.Health functions of stachyose and its use in beverage industry [J].The Beverage Industry, 2012, 15(2):15-19.
[5] ZHANG Y J, LI S, GAN R Y, et al.Impacts of gut bacteria on human health and diseases [J].International Journal of Molecular Sciences, 2015, 16(4):7 493-7 519.
[6] HAYAKAWA K, MIZUTANI J, WADA K, et al.Effects of soybean oligosaccharides on human faecal flora [J].Microbial Ecology in Health and Disease, 1990, 3(6):293-303.
[7] WANGNER J R, ROBERT B, GUMBMANN M R, et al.Hydrogen production in the rat following ingestion of raffinose, sachyose and oligosaccharide-free bean residue [J].Journal of Nutrition, 1976, 106(4):466-470.
[8] ZHONG X F, ZHANG Y B, HUANG G D, et al.Proteomic analysis of stachyose contributed to the growth of Lactobacillus acidophilus CICC22162[J].Food & Function, 2018, 9(5):2 979-2 988.
[9] ABRIOUEL H, BENOMAR N, COBO A, et al.Characterization of lactic acid bacteria from naturally-fermented Manzanilla Alore a green table olives[J].Food Microbiology, 2012, 32(2):308-316.
a green table olives[J].Food Microbiology, 2012, 32(2):308-316.
[10] 毛丙永. 功能性低聚糖对肠道细菌的影响及机制[D].无锡:江南大学, 2015.
MAO B Y.Effects of functional oligosaccharides on the gut bacteria and the mechanism [D].Wuxi:Jiangnan University, 2015.
[11] SCALABRINI P,ROSSI M,SPETTOLI P,et al.Characterization of Bifidobacterium stains for use in soymilk fermentation[J].International Jounal of Food Microbiology,1998,39(3):213-219.
[12] LUO R B, LIU B H, XIE Y L, et al.SOAPdenovo2:an empirically improved memory-efficient short-read de novo assembler [J].SOA Gigascience, 2012,1(1):18-19.
[13] STEFAN K, ADAM P, AETHUR L D, et al.Versatile and open software for comparing large genomes [J].Genome Biology, 2004, 5(12):1-9.
[14] BORODOVSKY M, MILLS R, BESEMER J.Prokaryotic gene prediction using genemark and genemark.hmm [J].Current Protocols in Bioinformatics, 2003, 1(1):4-5;16.
[15] ARTHUR L D, DOUGLAS H, SIMON K, et al.Improved microbial gene identification with GLIMMER [J].Nucleic Acids Research, 1999, 27(23):4 635-4 641.
[16] KANECHISA M, GOTO S.KEGG:Kyoto encyclopedia of genes and genomes[J].Nucleic Acids Research, 2000, 28(1):27-30.
[17] SCALABRINI P, ROSSI M, SPETTOLI P, et al.Characterization of Bifidobacterium strains for use in soymilk fermentation [J].International Journal of Food Microbiology, 1998, 39(3):213-219.
[18] BARBARA Z, KARINA S, RENATE L, et al.Fermentation of non-digestible raffinose family oligosaccharides and galactomannans by probiotics [J].Food & Function, 2018, 9(3):1 638-1 646.
[19] 肖敏, 刘树峰, 朱崇日, 等.短双歧杆菌α-D-半乳糖苷酶的纯化及性质[J].中华微生物学和免疫学杂志, 2001, 21(3):307-311.
XIAO M, LIU S F, ZHU C R, et al.Purification and characterization of α-D-galactosidase from Bifidobacterium breve[J].Chinese Journal of Microbiology and Immunology, 2001, 21(3):307-311.
[20] HOLT S, TERESI J, COTE G.Influence of alternansucrase-derived oligosaccharides and other carbohydrates on α-galactosidase and α-glucosidase activity in Bifidobacterium adolescentis [J].Letters in Applied Microbiology, 2008,46:73-79.
[21] ZARTL B, SILBERBAUER K, LOEPPERT R, et al.Fermentation of non-digestible raffinose family oligosaccharides and galactomannans by probiotics [J].Food & Function, 2018, 9(3):1 638-1 646.
[22] DIERKING E C, BILYEU K D.Raffinose and stachyose metabolism are not required for efficient soybean seed germination [J].Plant Physiology, 2009, 166(12):1 329-1 335.
[23] SHIMIZU T, OHTANI K, HIRAKAWA H, et al.Complete genome sequence of Clostridium perfringens, an anaerobic flesh-eater [J].PNAS, 2002, 99(2):996-1 001.
[24] SUAREZ F, FUME J, SPRINGFIELD J, et al.Insights into human colonic physiology obtained from the study of flatus composition [J].The American Journal of Physiology, 1997, 272(5):1 028-1 033.
[25] 张岩, 闫文辉, 程静, 等.厌氧发酵生物产氢技术的研究进展[J].能源与环境, 2008,2:66-67.
ZHANG Y, YAN W H, CHENG J, et al.Research progress of anaerobic fermentation biological hydrogen production technology[J].Energy and Environment, 2008,2:66-67.
[26] MONIQUE J, GARY S R.Ferredoxin has a pivotal role in the biosynthesis of the hydrogen-oxidizing hydrogenases in Escherichia coli [J].International Journal of Hydrogen Energy, 2014, 39(32):8 533-8 542.
[27] 王亚俊. 基于基因组的屎肠球菌进化和功能研究[D].上海:上海交通大学, 2012.
WANG Y J.Genome-based insights into the evolution and function of Enterococcus faecium[D].Shanghai:Shanghai Jiao Tong University, 2012.
[28] 杨唯枝. 体外诱导替加环素耐药粪肠球菌全基因测序及突变位点分析[D].广东:广东医学院, 2015.
YANG W Z.Complete genome sequencing of tigecycline-resistant Enterococcus faecalis induced in vitro and analyze the whole genome sequencing and mutation sites [D].Guangdong:Guangdong Medical University, 2015.
[29] SCHAUER N L, FERRY G J.Composition of the coenzyme F420-dependent formate dehydrogenase from Methanobacterium formicicum [J].Journal of Bacteriology, 1986, 165(2):405-411.
[30] PALLAVI S, SHANTONU R, DEBARATA D.Role of formate hydrogen lyase complex in hydrogen production in facultative anaerobes [J].International Journal of Hydrogen Energy, 2015, 40(29):8 806-8 815.
[31] BOHM R, SAUTER M, BOCK A.Nucleotide sequence andexpression of an operon in Escherichia coli coding for formate hydrogenlyase components [J].Molecular Microbiology, 1990, 4(2):231-243.
[32] SAUTE M, BOHM R, BOCK A.Mutational analysis of the operon (hyc) determining hydrogenase 3 formation in Escherichia coli [J].Molecular Microbiology, 1992, 6(11):1 523-1 532.
[33] SCHLENSOG V, BOCK A.Identification and sequence analysis of the gene encoding the transcriptional activator of the formate hydrogenlyase system of Escherichia coli [J].Molecular Microbiology, 1990, 4(8):1 319-1 327.
[34] RAE J L, CUTFIELD F J, LAMONT I L.Sequences and expression of pyruvate dehydrogenase genes from Pseudomonas aeruginosa[J].Journal of Bacteriology, 1997, 179(11):3 561-3 571.
[35] REHMS H, BARZ W.Degradation of stachyose, raffinose, melibiose and sucrose by different tempe-producing Rhizopus fungi [J].Applied Microbiology and Biotechnology,1995, 44(1-2):47-52.
[36] ALEXANDER J W, KAREN A H, ARTHUR H F.Two closely related ABC transporters in Streptococcus mutans are involved in disaccharide and/or oligosaccharide uptake[J].Journal of Bacteriology, 2008, 190(1):168-178.
[37] RUSSEL R, OPOKU A J, SUTCLIFFE I C, et al.A binding protein-dependent transport system in Streptococcus mutans responsible for multiple sugar metabolism [J].Journal of Biological Chemistry, 1992, 267(7):4 631-4 637.
[38] 陈臣,周方方,任婧,等.乳酸菌利用低聚果糖代谢机理的研究进展[J].食品与发酵工业, 2013, 39(10):156-160.
CHEN C, ZHOU F F, REN J, et al.The research progress on molecular mechanisms mediating fructooligosaccharides metabolism by lactic acid bacteria [J].Food and Fermentation Industries, 2013, 39(10):156-160.
[39] DEVOS P, GARRITY G M, JONES D, et al.Bergey’s Manual of Systematic Bacteriology (second edition)[M].Dordrecht,Heidelberg,London,New,York:Springer, 2009.
[40] 刘柯. 保加利亚乳杆菌与嗜热链球菌乳酸生成负反馈途径的构建[D].哈尔滨:哈尔滨工业大学, 2014.
LIU K.Construction of the negative feedback pathway of lactic acid in Lactobacillus bulgaricus and Streptococcus thermophiles [D].Harbin:Harbin Institute of Technology, 2014.