鸡蛋干是中国市面上常见的休闲蛋制品,因其营养丰富、风味独特而深受消费者喜爱。依据GB 2749—2015《食品安全国家标准 蛋与蛋制品》内容,在保质期内的鸡蛋干在感官方面应具有产品正常的色泽、滋味、气味,无异味,且具有产品正常的形状、形态、无酸败、霉变等;在微生物限量方面,其菌落总数应<104 CFU/g,大肠菌群应<10 CFU/g[1]。目前在实际生产过程中,鸡蛋干的保质期为9个月。鸡蛋干的生产原料以全蛋液或蛋清液为主,在加工制作过程中极易受到微生物污染而导致产品腐败变质[2]。相关调查研究表明,预包装鸡蛋干即便经过高压蒸汽灭菌后也可能出现胀包、汤汁浑浊、有异味的变质现象,尤其在夏秋高温季节,鸡蛋干发生腐败变质的概率更高[3]。最近数十年,鸡蛋干的市场在不断扩大,因此对鸡蛋干的防腐保鲜开展研究愈显重要。而明确鸡蛋干的优势致腐微生物,是防腐的首要关键。然而目前对于鸡蛋干的腐败微生物群落多样性的报道十分有限,贺燕等[3]从腐败鸡蛋干中分离鉴定得到2株芽孢杆菌,袁先铃等[4]在腐败鸡蛋干中检测到彭氏变形菌和奇异变形菌。因此,开展鸡蛋干中腐败微生物群落多样性的研究具有重要意义。
对于微生物群落多样性的研究,长期以来主要是基于《伯杰细菌鉴定手册》,对微生物菌落进行分离、纯化,进而通过形态学观察和一系列复杂的生理生化实验进行鉴定。这种传统方法不但耗费时间和精力,而且无法对微生物进行精确鉴定,不能检测到微生物群落多样性的真正概貌[5-6]。因为有些微生物的培养条件非常苛刻,甚至不可培养,常规方法难以检测到不可培养的微生物。近年来,在微生物群落多样性研究中已经越来越倾向于采用Illumina MiSeq高通量测序技术[7-10]。高通量测序技术也称“下一代测序技术”,可同时测序数百万个核酸分子,根据16S rDNA基因序列分析特定样品中微生物群落的构成情况[11-12]。目前高通量测序技术已成为监测食品贮藏过程中微生物群落演替、分析发酵及腐败食品微生物群落结构等食品微生物多样性研究的首选方法[11]。
本研究以保质期不同的腐败变质鸡蛋干为研究对象,基于Illumina Miseq测序平台对其16S rDNA V3~V4进行扩增子测序分析,研究其微生物群落结构组成和相对丰度,探究造成鸡蛋干腐败变质的核心优势微生物以及造成鸡蛋干保质期差异的微生物种类,为靶向抑制鸡蛋干的腐败变质提供理论依据。
1 材料与方法
1.1 材料与试剂
鸡蛋,北京德青源农业科技股份有限公司;细菌基因组DNA提取试剂盒,美国Omega科技公司;Axy Prep DNA凝胶回收试剂盒,美国Axygen公司;2×Taq PCR MasterMix、DNA Marker,天根生化科技(北京)有限公司。
1.2 仪器与设备
5418R高速离心机,德国Eppendorf公司;拍击式均质机,法国Interscience公司;RS-400型真空包装机,北京日上公司;T100 Thermal Cycler 聚合酶链式反应(polymerase chain reaction,PCR)仪、PowerPac Basic型电泳仪,美国Bio-Rad公司;ChampGel5000增强型全自动凝胶成像仪,北京赛智创业科技有限公司;Illumina MiSeq测序仪,美国Illumina公司。
1.3 试验方法
1.3.1 样品制备
鸡蛋干的制作工艺参照参考文献[13],具体为取预处理蛋液于模具中,水浴加热使其中心温度达到80 ℃,并维持20 min,待其冷却收缩后脱模。将鸡蛋干样品真空包装后,进行90 ℃、30 min灭菌,于25 ℃保存直至变质。预实验发现,对于使用不同批次原料、于不同的时间点制作的鸡蛋干样品,即使其加工工艺完全一致,样品的保质期也会有较大差异。为探究不同保质期的鸡蛋干样品中菌群结构的差异,采用上述加工工艺,分别使用不同批次原料,于不同的时间点制作了多批样品,从其中选取贮藏15 d、22 d和29 d腐败变质的3批鸡蛋干产品,取样保存于-80 ℃冰箱待测。将3批鸡蛋干样品分别编号为15 d、22 d和29 d,每组样品设置3个平行。
1.3.2 细菌总DNA的提取
参考高乾坤等[14]的方法,对样品进行处理后,采用美国Omega公司的细菌基因组DNA提取试剂盒,提取细菌总DNA,提取步骤参照试剂盒产品说明书,并进行1%(质量分数)的琼脂糖凝胶电泳,以检测提取效果。
1.3.3 PCR扩增及高通量测序
以检验合格的基因组DNA为模板,扩增细菌16S rDNA基因V3~V4区。正向引物为338F(5′-ACTCCTACGGGAGGCAGCA-3′),反向引物为806R(5′-GGACTACHVGGGTWTCTAAT-3′)。所得PCR产物经2%(质量分数)琼脂糖凝胶电泳检测扩增效果,然后将样品进行纯化并定量检测。将检测合格的样品送至上海美吉生物医药科技有限公司进行Illumina MiSeq高通量测序。
1.4 数据分析
对MiSeq测序得到的双端序列数据进行处理,首先将成对的reads拼接成一条序列,同时对reads的质量和拼接的效果进行质控过滤、得到有效序列,并校正序列方向[15]。
基于操作分类单元(operational taxonomic units, OTU)聚类分析结果,研究腐败变质鸡蛋干样品中微生物的多样性,通过Alpha多样性检测单样本测序深度,并分析微生物群落的丰度和多样性[16]。基于分类学信息,在各个分类水平上进行群落结构统计,综合讨论保质期不同的鸡蛋干样品中微生物群落结构组成和相对丰度。
2 结果与分析
2.1 测序数据统计与OTU分析
采用Illumina Miseq高通量测序获得了3种不同保质期鸡蛋干样品的原始序列数据后,经Fastp及FLASH软件优化数据后得到了9个样本共529 384条有效序列,序列平均长度为409.70~429.92 bp,主要集中在401~440 bp(图1-a)。稀释曲线可反映在高通量测序过程中的样品取样深度,因此是评价测序数据量是否全面覆盖微生物群落的常用指标[14]。为了探究鸡蛋干样品的测序数据量是否科学,从测序获得的所有原始数据中随机抽取数据,并统计其物种多样性指数,得到稀释曲线(图1-b和图1-c)。结果表明,随着数据量的增加,OTU曲线和Shannon-Wiener曲线的趋势已经逐渐平缓,即再增加数据量对于发现新物种也无显著影响。样本的OTU覆盖度已经基本饱和,更多的数据量对发现新OTU的边际贡献很小,这个结果说明测序数据量足够大,可以反映样本中绝大多数的微生物多样性信息[17]。

a-样品序列长度分布图;b-OTU曲线;c-Shannon-Wiener曲线
图1 样品序列长度分布图、OTU曲线和Shannon-Wiener曲线
Fig.1 Length distribution profile of trimed sequences, OTU curves and Shannon-Wiener curves
2.2 Alpha多样性分析
根据97%相似性水平下的OTU信息,采用Alpha多样性指标的Shannon、Simpson、ACE、Chao1及Coverage指数对鸡蛋干样品微生物物种的丰富度和多样性进行评估[14]。如表1所示,3组样品的Coverage指数都达到了0.999以上,即样品文库的覆盖率足够高,有未被检出序列的可能性较小,表明本次试验所建文库能够较准确地反映出待测鸡蛋干的微生物多样性情况。Chao1、ACE指数用于计算菌群丰度,二者的值越大,说明样品的群落丰富度越高;Shannon、Simpson指数用于计算菌群多样性指数,Shannon的值越大,说明样品群落多样性越高,而Simpson指数值越大,则说明其群落多样性越低[18]。22 d组的鸡蛋干样品ACE、Chao1及Shannon指数最高,Simpson值最低,这说明该组样品的细菌群落丰富度及多样性高于其他2组样品,可能由于该组样品的原料中及加工过程中污染的细菌种类最多;29 d组鸡蛋干样品的ACE、Chao1及Shannon指数均为3组中最小,这说明了该组样品中细菌群落丰度及多样性最低,可能由于该组样品的原料中及加工过程中受到的污染程度较低。综上,经Alpha多样性指数统计分析可知,鸡蛋干样品的物种多样性由高到低依次为22 d组、15 d组和29 d组。此结果显示,相比于污染的微生物物种的多样性,鸡蛋干所污染微生物的具体种类可能与鸡蛋干的保质期相关性更大,这与孙同辉等[11]的报道相一致。
2.3 OTU的Veen分析
基于样品总OTU数作Veen图,利用Venn图可以展示多组样品间共有和每组样品特有的OTU数目,并直观展示样品间OTU的重叠情况[19]。如图2所示,15 d组样品与22 d组样品共有的OTU数107个,15 d组与29 d组共有的OTU数131个,22 d组与29 d组共有的OTU数92个,3组样本共有的OTU数为81个,占各样本OTU数比例较小,而各样本特有的OTU数在113~259,表明各样本之间的菌群结构差异性较大。
表1 Alpha多样性指数
Table 1 Alpha diversity indexes
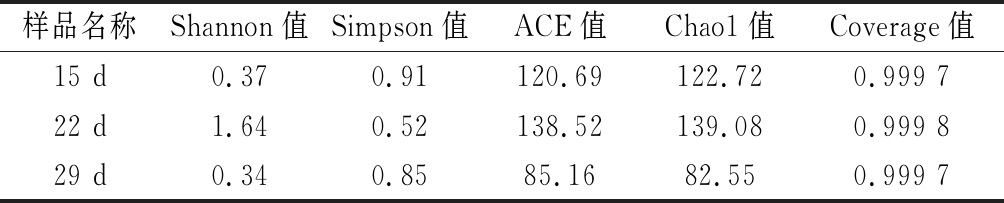
样品名称Shannon值Simpson值ACE值Chao1值Coverage值15 d0.370.91120.69122.720.999 722 d1.640.52138.52139.080.999 829 d0.340.8585.1682.550.999 7

图2 不同保质期样品中微生物物种间OUT分析图
Fig.2 OUT analysis diagram of microbial species in samples with different shelf life
2.4 不同保质期鸡蛋干样品细菌群落结构分析
2.4.1 基于门水平的细菌菌群结构分析
在门水平上,3组鸡蛋干样品的群落组成如图3所示。15 d组的鸡蛋干样品总细菌组成以厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为主,相对丰度分别为96.73%和1.96%。除此之外还包括少量的放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)微生物。而22 d组的鸡蛋干样品菌群结构以厚壁菌门、变形菌门和拟杆菌门为主,相对丰度分别为87.55%、9.41%和1.32%,同时还含有少量的放线菌门微生物。29 d组的鸡蛋干样品中微生物以厚壁菌门(70.94%)、放线菌门(27.81%)和变形菌门(1.09%)为主,还含有少量的拟杆菌门微生物。以上结果表明,在3组不同保质期的样品中,厚壁菌门的丰度均为最高,可见厚壁菌门是腐败变质鸡蛋干中的优势微生物。近年来许多研究表明,厚壁菌门微生物是高蛋白食品中的优势腐败菌,在乳制品、肉制品及水产品中都十分常见[18-21],这与本研究的结果一致。
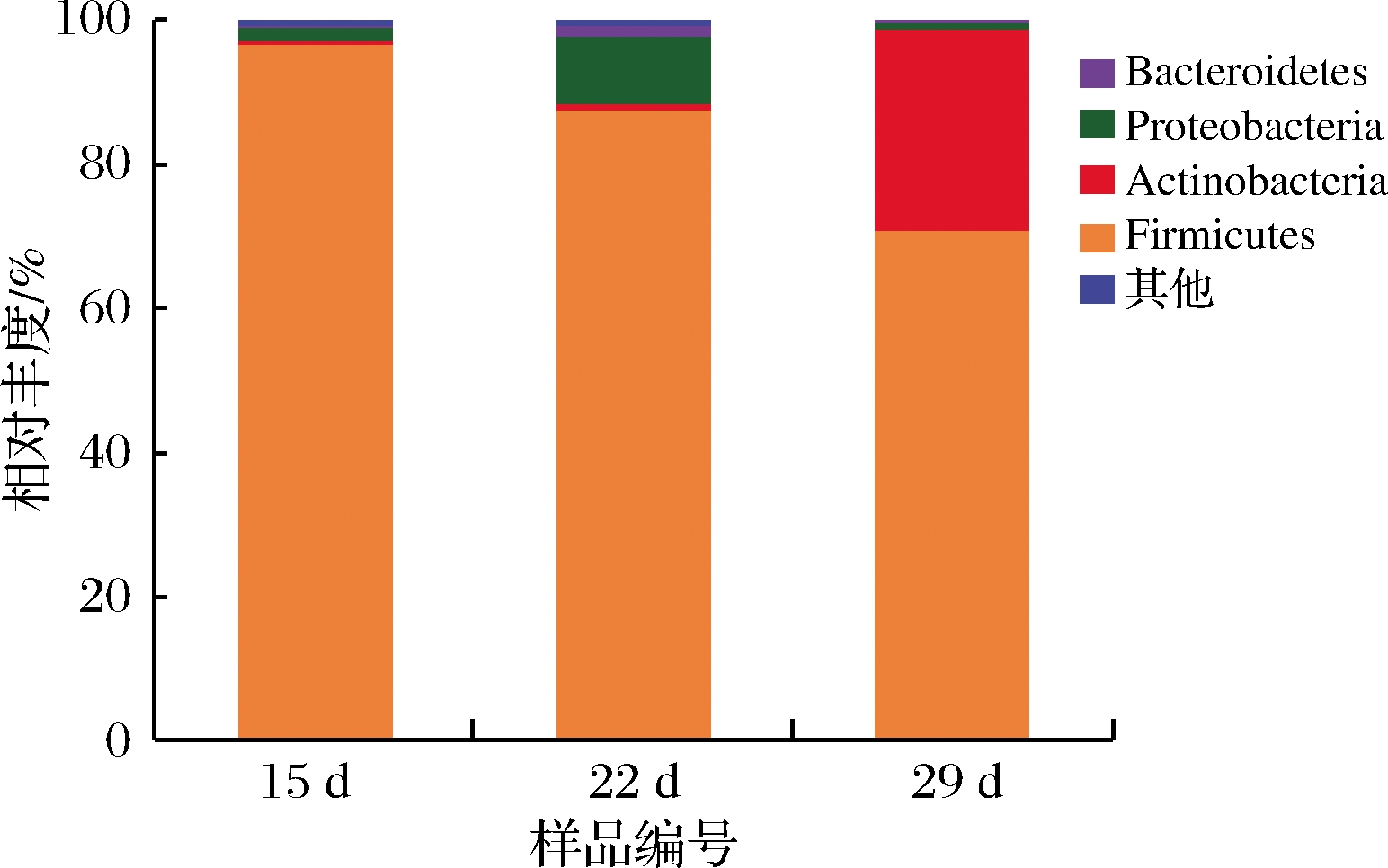
图3 基于门分类水平上的样品菌群分布图
Fig.3 Bacterial distribution pattern at the phylum level
2.4.2 基于科水平的细菌菌群结构分析
基于科分类水平的3组鸡蛋干样品的微生物群落结构如图4所示。测序结果表明,15 d组的鸡蛋干样品中类芽孢杆菌科(Paenibacillaceae)所占比例最大,为47.43%,其次为芽孢杆菌科(Bacillaceae)和梭菌科(Clostridiaceae_1),所占比例分别为33.14%和14.96%,还有乳杆菌科(Lactobacillaceae)、伯克氏菌科(Burkholderiaceae)、链球菌科(Streptococcaceae)、假单胞菌科(Pseudomonadaceae)等相对丰度较低的菌种,说明鸡蛋干在制作和包装过程中污染微生物的种类复杂,导致样品菌落结构呈现多样性。22 d组的鸡蛋干样品菌群结构以芽孢杆菌科、乳杆菌科、链球菌科、伯克氏菌科为主,相对丰度分别为47.39%、29.11%和3.76%、3.29%,除此之外还包括少量的红杆菌科(Rhodobacteraceae)、假单胞菌科、类芽孢杆菌科微生物。29 d组鸡蛋干样品中的菌群在科水平上主要有芽孢杆菌科(68.85%)和小单孢菌科(Micromonosporaceae,27.60%),还含有少量的假单胞菌科和伯克氏菌科微生物。以上结果表明,不同保质期鸡蛋干样品所含微生物种类和数量有明显差异,如相比于其他2组,29 d组样品中却未检出乳杆菌科微生物,可能由于该组样品的原料中及加工过程中污染的乳杆菌科较少。29 d组鸡蛋干样品的保质期最长,其微生物多样性低于其余2组,说明样品中污染的微生物种类和数量与鸡蛋干的保质期密切相关。22 d组样品微生物多样性最高,但此组保质期不是最短,可能由于样品中含有丰度较高的乳杆菌科微生物[22-23],对其他微生物产生拮抗作用、抑制其生长繁殖,相对延长了鸡蛋干保质期。此外,在15 d组样品中检测出梭菌科微生物,而其他2组中均未检出,梭菌科中有些菌种为厌氧菌,可在真空包装鸡蛋干中迅速生长繁殖,这可能是本组鸡蛋干保质期远小于其他2组的原因。3组不同保质期的样品中,均含有芽孢杆菌科微生物,且丰度较高,表明芽孢杆菌科是腐败变质鸡蛋干中的核心优势微生物。

图4 基于科分类水平上的样品菌群分布图
Fig.4 Bacterial distribution pattern at the family level
2.4.3 基于属水平的细菌菌群结构分析
选取在鸡蛋干样品丰度排名前15的属,根据其在每组鸡蛋干样品中的丰度信息聚类绘制成热图(图5-a)。热图可通过颜色变化直观地反映出各组鸡蛋干样品群落组成的相似性和差异性,更便于发现在各组样品中分布较多或较少的菌属[10]。图中颜色越偏砖红色,表明样品中所含该菌属比例越高,颜色越偏深蓝色,比例则越低。如图5-a所示,在属水平,22 d组和29 d组聚为一类,15 d组单独聚为一类,说明22 d组和29 d组菌群整体结构相似度较高,与15 d组相似度较低,表明鸡蛋干样品的保质期与其菌群结构相关性较大。
从图5-b各样品菌群柱状分布图中可以看出,在属水平上,15 d组优势菌群主要是芽孢杆菌属 (Bacillus,33.14%)、短芽孢杆菌属(Brevibacillus,30.96%)、类芽孢杆菌属(Paenibacillus,16.46%)和狭义梭菌属(Clostridium_sensu_stricto_13,14.86%)。在优势菌群中,芽孢杆菌属所占比例最大,它属于需氧或兼性厌氧菌,此属中大部分细菌为腐生菌,广泛分布于自然环境中。该菌属可形成芽孢,对不良环境条件有很强的抵抗力,不易杀灭。贺燕等[3]在腐败鸡蛋干中也有检测到芽孢杆菌属微生物。短芽孢杆菌属为需氧或兼性厌氧菌,近年来相关报道多集中于该属工程菌株的研究,鲜见有研究发现其在食品腐败中的作用[24-25]。类芽孢杆菌属为需氧或兼性厌氧菌,近年来许多研究表明,其为多种乳制品和即食食品的腐败微生物[26-27]。狭义梭菌属广泛分布于土壤、下水污泥、人和其他哺乳动物的肠道等环境中,是乳制品和肉制品中常见的腐败微生物[20, 28-29]。
22 d组优势菌群主要是芽孢杆菌属(47.38%)、乳杆菌属(Lactobacillus,28.26%)和链球菌属(Streptococcus,3.57%)。乳杆菌属为兼性厌氧菌,广泛分布于自然界中,污染乳制品、肉制品、水产品等高蛋白食品后可引起腐败变质。链球菌属为兼性厌氧菌,该属中有少数种为腐生菌,污染食品后可引起腐败变质[29]。

a-样品菌群相对丰度热图;b-样品菌群相对丰度柱形图
图5 基于属分类水平上的样品菌群相对丰度热图 和柱形图
Fig.5 Heat map and stacked-column of relative abundances of bacterial communities at the genus level
29 d组优势菌属主要是芽孢杆菌属(68.85%)和小单孢菌属(Micromonospora,27.60%)。小单孢菌属为好氧或微好氧的腐生菌,是土壤和水域的常见微生物群体[30],其在真空包装鸡蛋干中占比达到27.60%,可能由于鸡蛋干中初始污染小单孢菌属较多,且微好氧微生物能在较低的氧分压下生长,鸡蛋干虽然经过真空包装,但仍无法将样品中氧气完全排出,因此小单孢菌属中部分微好氧菌种可以生长。
以上结果表明,在3组不同保质期的样品中,芽孢杆菌属的相对丰度均为最高,可见芽孢杆菌属是鸡蛋干的核心致腐微生物。保质期最长的29 d组样品中优势菌属为芽孢杆菌属和小单孢菌属,其中小单孢菌属为好氧或微好氧菌,在真空包装鸡蛋干中生长缓慢,而芽孢杆菌属中部分菌种为兼性厌氧菌,可在真空包装条件下良好生长,因此芽孢杆菌属应为限制此组样品保质期的微生物。而相比于29 d组,22 d组样品中还含有较高丰度的乳杆菌属,这可能是造成本组鸡蛋干保质期<29 d的原因。此外,狭义梭菌属仅在保质期为15 d的鸡蛋干样品中检测到,它属于厌氧菌,因此在真空包装鸡蛋干中的生长速度大于非厌氧菌,可快速分解鸡蛋干中营养物质造成腐败变质,这可能是15 d组鸡蛋干保质期远低于其他2组的原因。
3 结论
本研究以保质期不同的3组腐败变质鸡蛋干为研究对象,采用Illumina Miseq高通量测序技术分析样品中菌群组成情况。结果表明,3组腐败变质鸡蛋干的微生物的多样性以及丰度存在较大差异,而总体来看,厚壁菌门为腐败变质鸡蛋干的优势菌门,芽孢杆菌科为优势菌科。在属水平,芽孢杆菌属在3组样品中的相对丰度均为最高,是鸡蛋干的核心致腐微生物。此外,造成鸡蛋干保质期差异的菌属包括乳杆菌属和狭义梭菌属,它们可加速鸡蛋干的腐败变质,是鸡蛋干保质期的限制因素,应在杀菌防腐环节重点关注。高通量测序技术检测不同保质期鸡蛋干样品的微生物组成,不仅可以分析鸡蛋干腐败过程中的核心优势菌群,还可以高效快速地分析样品中微生物种类的差异,为今后鸡蛋干质量安全监测及靶向研究鸡蛋干防腐保鲜技术提供理论依据。
[1] GB 2749—2015 食品安全国家标准 蛋与蛋制品[S].北京:中国标准出版社, 2017.
GB 2749—2015 National standards of food safety eggs and egg products[S].Beijing:China Standards Press, 2017.
[2] 薛艳蓉, 茂文, 赵瑞生, 等.鸡蛋在储存过程中微生物及蛋品质的变化研究[J].畜牧与饲料科学, 2017, 38(8):67-69;72.
XUE Y R, MAO W, ZHAO R S, et al.Dynamic changes of microbiological and qualitative features of chicken eggs during storage process[J].Animal Husbandry and Feed Science, 2017, 38(8):67-69;72.
[3] 贺燕, 谭纯良, 李娜, 等.复配防腐剂抑制鸡蛋干腐败菌效果研究[J].食品与机械, 2019, 35(11):159-164.
HE Y, TAN C L, LI N, et al.Study on the effect of compound preservatives on inhibiting spoilage of egg curd[J].Food and Machinery, 2019, 35(11):159-164.
[4] 袁先铃, 黄丹, 潘明.腐败鸡蛋干中主要微生物的分离纯化及鉴定[J].食品研究与开发, 2012, 33(7):57-59.
YUAN X L, HAUNG D, PAN M.Purification and identification of microbes in corruption dried eggcurd[J].Food Research and Development, 2012, 33(7):57-59.
[5] HANDELSMAN J.Metagenomics:Application of genomics to uncultured microorganisms[J].Microbiology and Molecular Biology Reviews, 2004, 68(4):669-685.
[6] 吴燕燕, 钱茜茜, 李来好, 等.基于Illumina MiSeq技术分析腌干鱼加工过程中微生物群落多样性[J].食品科学, 2017, 38(12):1-8.
WU Y Y, QIAN Q Q, LI L H, et al.Microbial community diversity in dried-salted fish during processing revealed by Illumina MiSeq sequencing[J].Food Science, 2017, 38(12):1-8.
[7] POO KA J, REBECCHI A, PISACANE V, et al.Bacterial diversity in typical Italian salami at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J].Food Microbiology, 2015, 46(4):342-356.
KA J, REBECCHI A, PISACANE V, et al.Bacterial diversity in typical Italian salami at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J].Food Microbiology, 2015, 46(4):342-356.
[8] ZHANG J B, LI Y, LIU X C, et al.Characterization of the microbial composition and quality of lightly salted grass carp (Ctenopharyngodon idellus) fillets with vacuum or modified atmosphere packaging[J].International Journal of Food Microbiology, 2019, 293(6):87-93.
[9] ZANG J H, XU Y S, XIA W S, et al.Dynamics and diversity of microbial community succession during fermentation of Suan yu, a Chinese traditional fermented fish, determined by high throughput sequencing[J].Food Research International, 2018, 111(9):565-573.
[10] 戴玲瑛, 齐曼婷, 王立梅, 等.虾酱中间产物细菌群落结构及多样性[J].食品科学, 2018, 39(22):173-178.
DAI L Y, QI M T, WANG L M, et al.Bacterial community structure and diversity in unsterilized shrimp sauce[J].Food Science, 2018, 39(22):173-178.
[11] 孙同辉, 陈洁, 李雪琴, 等.杀菌方式对年糕微生物多样性及品质的影响[J/OL].食品科学,2020.http://kns.cnki.net/kcms/detail/11.2206.TS.20200109.0934.012.html.
SUN T H, CHEN J, LI X Q, et al.Effect of sterilization method on microbial diversity and quality of rice cake[J/OL].Food Science,2020. http://kns.cnki.net/kcms/detail/11.2206.TS.202001 09.0934.012.html.
[12] SOON W W, HARIHARAN M, SNYDER M P.High-throughput sequencing for biology and medicine[J].Molecular Systems Biology, 2013, 9(1):640.
[13] 张伯男, 樊晓盼, 马俪珍, 等.风味鸡蛋干的加工工艺研究[J].食品研究与开发, 2018, 39(11):101-105.
ZHANG B N, FAN X P, MA L Z, et al.Study on processing technology of flavor egg curd[J].Food Research and Development, 2018, 39(11):101-105.
[14] 高乾坤, 焦琳舒, 杜贺超, 等.高通量测序分析不同产地带鱼冷藏时微生物群落多样性[J].食品科学, 2018, 39(18):127-132.
GAO Q K, JIAO L S, DU H C, et al.High-throughput sequencing different waters during chilled storage[J].Food Science, 2018, 39(18):127-132.
[15] ANDREEVSKAYA M, JOHANSSON P, LAINE P, et al.Genome sequence and transcriptome analysis of meat-spoilage-associated lactic acid bacterium Lactococcus piscium MKFS47[J].Applied and Environmental Microbiology, 2015, 81(11):3 800-3 811.
[16] LU X M, LU P Z.Characterization of bacterial communities in sediments receiving various wastewater effluents with high-throughput sequencing analysis[J].Microbial Ecology, 2014, 67(3):612-623.
[17] 江杨阳. 小龙虾低温贮藏品质变化规律及其腐败菌鉴定[D].杭州:浙江大学, 2019.
JIANG Y Y.Studies on the quality changes of red swamp crayfish and its specific spoilage organisms during low temperature storage[D].Hangzhou:Zhejiang University, 2019.
[18] 马岩石, 姜明, 李慧, 等.基于高通量测序技术分析东北豆酱的微生物多样性[J].食品工业科技,2020,41(12);100-105.
MA Y S, JIANG M, LI H, et al.Analysis of microbial diversity of northeast soy sauce based on high-throughput sequencing technology[J].Science and Technology of Food Industry,2020,41(12):100-105.
[19] 李春生, 王悦齐, 李来好, 等.传统鱼露发酵过程中细菌群落演替及对其挥发性风味形成的影响分析[J].食品科学, 2018, 39(24):85-92.
LI C S, WANG Y Q, LI L H, et al.Bacterial community succession during fermentation of Chinese fish sauce and its effect on formation of volatile flavor components[J].Food Science, 2018, 39(24):85-92.
[20] 毛海萍, 袁开, 金仁耀, 等.基于传统分离培养和高通量测序分析市售咸鳓鱼中微生物多样性[J].食品研究与开发, 2019, 40(21):193-201.
MAO H P, YUAN K, JIN R Y, et al.Study of microbial diversity in commercial salted Chinese herring using culture isolation and high-throughput sequencing[J].Food Research and Development, 2019, 40(21):193-201.
[21] 李伟程, 侯强川, 于洁, 等.传统发酵乳制品中微生物多样性研究[J].食品工业科技, 2018, 39(1):131-136.
LI W C, HOU Q C, YU J, et al.Study on the diversity of microbial in traditional fermented dairy products[J].Science and Technology of Food Industry, 2018, 39(1):131-136.
[22] 孙群, 易华西, 张冬, 等.潜在益生菌的筛选及抑菌功能特性评价[J].食品科技, 2019, 44(12):1-5.
SUN Q, YI H X, ZHANG D, et al.Screening of potential probiotics and evaluation of antibacterial function characteristics[J].Food Science and Technology, 2019, 44(12):1-5.
[23] 王玉文, 刘英华.乳酸菌的生物拮抗作用[J].中国乳品工业, 2008, 36(2):55-57.
WANG Y W, LIU Y H.State antagonism about lactic acid bacteria[J].China Dairy Industry, 2008, 36(2):55-57.
[24] 赵乃昕, 苑广盈.医学细菌名称及分类鉴定[M].济南:山东大学出版社, 2013.
ZHAO N X, YUAN G Y.Name and Classification of Medical Bacteria[M].Jinan:Shandong University Press, 2013.
[25] 邹亮, 吴敬, 陈晟.重组蔗糖异构酶在短短芽孢杆菌中的表达及发酵优化[J].食品与生物技术学报, 2019, 38(1):22-28.
ZOU L, WU J, CHEN S.Expression and fermentation optimization of recombinant sucrose isomerase in Brevibacillus choshinensis[J].Journal of Food Science and Biotechnology, 2019, 38(1):22-28.
[26] HELMOND M, NIEROP GROOT M N, HERMIEN B V D V.Characterization of four Paenibacillus species isolated from pasteurized, chilled ready-to-eat meals[J].International Journal of Food Microbiology, 2017, 252(13):35-41.
[27] TRMCIC A, MARTIN N H, BOOR K J, et al.A standard bacterial isolate set for research on contemporary dairy spoilage[J].Journal of Dairy Science, 2015, 98(8): 5 806-5 817.
[28] AKHTAR S, PAREDES-SABJA D, TORRES J A, et al.Strategy to inactivate Clostridium perfringens spores in meat products[J].Food Microbiology, 2009, 26(3):272-277.
[29] 何国庆, 贾英民, 丁立孝.食品微生物学[M].北京:中国农业大学出版社, 2009.
HE G Q, JIA Y M, DING L X.Food Microbiology[M].Beijing:China Agricultural University Press, 2009.
[30] 杨苏声. 细菌分类学[M].北京:中国农业大学出版社,1997.
YANG S S.Bacterial Taxonomy[M].Beijing:China Agricultural University Press, 1997.