半纤维素是自然界第二大丰富的多糖,在农业生产的废弃物中含有大量的半纤维素,合理高效的利用半纤维素除了能够解决农业生产废弃物的问题,还能在一定程度上缓解能源危机问题。木聚糖是植物半纤维素的主要组成成分,快速高效的降解木聚糖是利用植物半纤维素的一个关键点。近年来多采用生物转换法来降解木聚糖,如利用木聚糖酶。木聚糖酶是一类能降解植物半纤维素中的木聚糖的糖苷键骨架的水解酶,并通过其中各种组分的协同作用降解木聚糖[1]。50多年前,从曲霉当中部分纯化了木聚糖酶之后,陆续分离纯化了许多其他属的木聚糖酶[2-3]。木聚糖酶不仅仅用在植物能源方面,在食品和饮料、动物饲料、提取植物油和提高动物的营养价值等方面都有广泛的应用[4-5]。相关研究表明,在pH<4或5的酸性条件下,有较高酶活力的木聚糖酶都可以被称为酸性木聚糖酶[6-7]。大多数的木聚糖酶为碱性木聚糖酶,国外以及国内对于碱性木聚糖酶早有研究,并已经应用于造纸工业以及生物工程领域[8-9]。酸性木聚糖酶能够在酸性条件下保持相对较高的酶活性,这类木聚糖酶在较低的pH值下能够稳定的发挥作用,在饲料,酿酒以及果汁澄清等需要低pH值的行业具有很好的应用前景[10]。
木聚糖酶的产生受培养基、pH值、接种量和温度等因素的影响,不同的生产条件对木聚糖酶的产量和水解效率有很大的影响。降低酶的生产成本是提高工业生产经济性和可行性的重要途径[11]。随着木聚糖酶在各个领域和产业中的应用日益广泛,市场需求对木聚糖酶的生产和活性提出了更高的要求。本文旨在探索其酶学性质,获得最佳发酵条件,以更好地满足市场需求。
响应面分析法(response surface methodology,RSM)是一种统计方法,它使用合理的实验设计来寻找最佳工艺参数并解决多变量问题[12]。本文以解淀粉芽孢杆菌的基因为研究对象,并结合响应面分析对表达条件进行了优化,尝试找到木聚糖酶的最佳表达条件。本实验对其酶学性质进行了研究,为大规模生物合成生产高质量的木糖提供了理论依据。
1 材料与方法
1.1 材料
1.1.1 菌种与试剂
实验室从土壤样品中筛选出1株解淀粉芽孢杆菌。Taq聚合酶、质粒pMD18-T,Takara公司;凝胶提取试剂盒、细菌DNA提取试剂盒,Omega;质粒提取试剂盒,Biomiga公司;桦木木聚糖和燕麦木聚糖,Sigma公司;山毛榉木聚糖,Megazyme公司。
1.1.2 培养基
LB(Luria-Bertani)培养基(g/L):胰蛋白胨10,酵母浸膏5,NaCl 5。
SOC(super optimal broth with catabolic repression)培养基(g/L):蛋白胨20,酵母提取物5,NaCl 0.5,KCl 0.186,MgCl2 0.952,葡萄糖3.603。
2×YT(yeast peptone broth)培养基(g/L):蛋白胨16,酵母提取物5,NaCl 5。
M-TB(modified-terrific broth)培养基(g/L):甘油10,酵母提取物24,蛋白胨12,KH2PO4 2.314,KH2PO4·3H2O 16.432。
SB(super broth)培养基(g/L):蛋白胨30,酵母提取物20,葡萄糖20。
1.2 基因克隆与表达
利用细菌DNA提取试剂盒,从解淀粉芽孢杆菌中提取总基因序列,根据NCBI(登录码:KY849859.1)上记录的木聚糖酶基因序列设计特异性引物,获得目的基因。将聚合酶链式反应(polymerase chain reation,PCR)得到的目的片段与克隆载体pMD18-T连接,转染到大肠杆菌DH5α。基于载体上携带的抗性基因,每100 mL培养基中添加100 μL的100 mg/mL氨苄抗生素,在LB培养基中培养并测序验证,测序正确的基因在Nhe I和Xho I酶切位点与pET28a(+)连接,然后转移到大肠杆菌BL21(DE3)中,重组菌株编号为BA-TB-1。
1.3 木聚糖酶的纯化与分析
将含有重组质粒的细胞BA-TB-1转移到LB培养基中,在200 r/min、37 ℃条件下培养,然后用500 mmol/L异丙基β-D-1-硫代半乳吡喃糖苷(ispropyl-β-D-thinogalactoside,IPTG)溶液诱导表达。将诱导表达后的细胞溶液在4 ℃,5 000 r/min状态下离心5 min,去掉上清液后加入10 mL 50 mmol/L Tris-HCl缓冲液(pH 7.0)。粗酶液超声处理15 min,离心获得上清液,上清液在10 000 r/min下离心10 min,粗酶液经Ni-Sepharose-HP柱纯化[13]。采用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)测定蛋白质的分子质量,用聚氧基丙烯酸正丁酯(butyleyanoacrylate,BCA)蛋白质浓度试剂盒测定蛋白质浓度。
1.4 木聚糖酶活性测定
根据标准方法,木聚糖酶的1个单位被定义为1 min 释放1 μmol木聚糖酶的量。通过将0.9 mL的1 mg/mL 山毛榉木聚糖与0.1 mL适当稀释的木聚糖酶(50 mmol/L乙酸缓冲液,pH 5.5)在55 ℃、反应5 min 来测定[14],并用3,5-二硝基水杨酸法(3,5-dinitrosalicylic acid,DNS)计算释放的还原糖量[15]。
1.5 最佳pH和pH稳定性
将纯化后的酶放置在不同pH的缓冲溶液(甘氨酸-HCl缓冲液pH 2.0~3.5;柠檬酸缓冲液pH 3.0~6.5;NaH2PO4-Na2HPO4缓冲液pH 6.0~8.0;Tris-HCl缓冲液pH 7.5~8.5)中进行处理,得到最佳的pH(以最佳pH条件下的酶活力为100%)。纯化后的酶在37 ℃条件下,置于不同的pH(4.0~10.0)溶液中处理60 min,研究酶的pH稳定性(以未处理的酶活力为100%)。
1.6 最佳温度和热稳定性
将纯化后的酶在不同温度(10~90 ℃)下反应,确定最佳反应温度(以最佳温度条件下的酶活力为100%)。在适宜的pH和温度(20~90 ℃)下反应30 min后测定酶活性,研究酶的热稳定性(以未处理的酶活力为100%)。
1.7 酶的诱导表达优化
1.7.1 单因素设计
通过控制培养基、诱导温度、接种量、500 mmol/L IPTG用量、诱导时间和诱导点(加入IPTG前预培养时间)等变量,对木聚糖酶的表达条件进行了优化。将低温保存的菌株接种到LB培养基,在37 ℃恒温下培养14 h成为母液。
诱导温度对木聚糖酶诱导表达的影响:在LB培养基中,接种量1.0%,500 mmol/L IPTG 200 μL,诱导时间18 h,诱导时机3.5 h的条件下,研究诱导温度为15、18、21、24、27、30 ℃对木聚糖酶诱导表达的影响。
培养基对木聚糖酶诱导表达的影响:实验选择5种培养基进行优化。在诱导温度27 ℃,接种量1.0%,500 mmol/L IPTG 200 μL,诱导时间18 h的条件下,研究LB、2 × YT、SB、SOC、MTB培养基对木聚糖酶诱导表达的影响。
诱导点对木聚糖酶诱导表达的影响:在LB培养基中,诱导温度27 ℃,接种量1.0%,500 mmol/L IPTG 200 μL,诱导时间18 h的条件下,研究诱导点2.5、3、 3.5、 4、 4.5 h对木聚糖酶诱导表达的影响。
诱导时间对木聚糖酶诱导表达的影响:在LB培养基中,接种量1.0%,500 mmol/L IPTG 200 μL,诱导温度27 ℃,诱导时机3.5 h的条件下,研究诱导时间为12、15、18、21、24、27 h对木聚糖酶诱导表达的影响。
IPTG浓度对木聚糖酶诱导表达的影响:在LB培养基中,诱导时机3.5 h,诱导温度27 ℃,接种量1.0%,诱导时间18 h的条件下,研究500 mmol/L IPTG用量为100、150、200、250、300 μL对木聚糖酶诱导表达的影响。
接种量对木聚糖酶表达的影响:在500 mmol/L IPTG量200 μL,诱导点3.5 h,诱导时间18 h,诱导温度27 ℃,LB培养基的条件下,研究接种量为0.5%、1%、1.5%、2%、2.5%、3%、3.5%对木聚糖酶诱导表达的影响。
1.7.2 响应面设计
根据单因素实验结果,选择单因素中对木聚糖酶活力最大的3个因素,采用中心组合的设计原理,以诱导点,500 mmol/L IPTG用量,接种量为自变量,木聚糖酶的酶活力为响应值进行响应面设计。实验设计如表1所示。
表1 Central Composit试验因素与水平
Table 1 Central Composit test factors and levels

水平诱导点(A)/h500 mmol/L IPTG用量(B)接种量(C)/%-131502.503.52003142503.5
2 结果与分析
2.1 木聚糖酶的克隆与分析
以解淀粉芽孢杆菌总基因组为模板,NCBI上记录的木聚糖酶基因序列设计引物,进行PCR实验。将PCR 的扩增产物用1%(质量分数)琼脂糖凝胶电泳法进行检测。检测结果如图1所示,通过PCR扩增获得642 bp的条带,与木聚糖酶目的基因相吻合,说明具有很高的产物特异性。
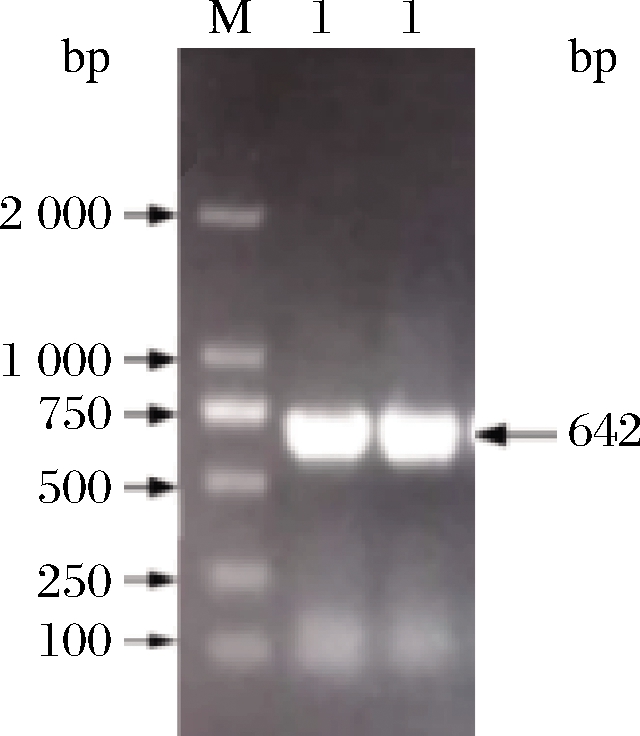
M-2 000 bp DNA Maker;条带1-目标DNA
图1 DNA琼脂糖凝胶电泳结果
Fig.1 Agarose gel electrophoresis of DNA
2.2 木聚糖酶的纯化
细胞置于超声波中进行破壁处理,将收集到的酶进行粉碎、离心和透析处理,粗酶经Ni-Sepharose-HP柱纯化。图2是酶的纯化结果,前2个条带分别是标记条带和空白对照。第3条带是破坏后的上清液。由图2可知,目标蛋白的表达量较高。第4条是纯化的酶,在23.28 kDa处有1条清晰的蛋白带。这与木聚糖酶的理论分子质量一致,表明得到了纯化的木聚糖酶。
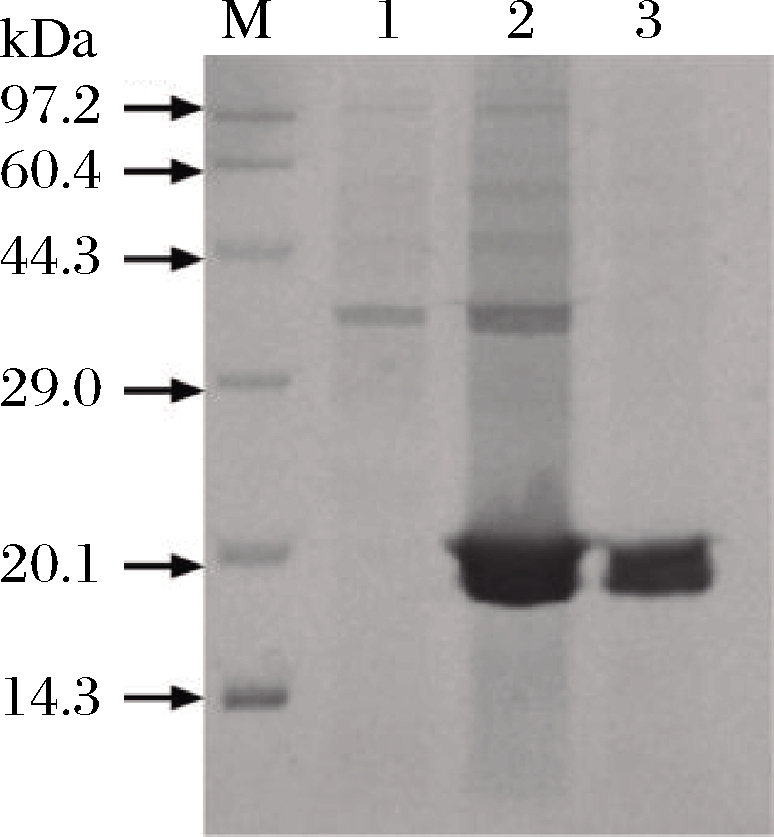
M-蛋白质Maker;1-空白对照;2-粗酶;3-纯化酶
图2 木聚糖酶的SDS-PAGE分析
Fig.2 Analysis of xylanase by SDS-PAGE
2.3 最佳pH和pH稳定性
以山毛榉木聚糖为底物,在缓冲溶液中测定木聚糖酶的最佳pH值。酶活性在4种不同的缓冲液中变化趋势相同,如图3-A所示。酶活性随着酸度的降低而增加,在pH值为5.0~6.0时有较高的酶活性。37 ℃条件下,将木聚糖酶在不同pH缓冲液中培养1 h,测定酶活力。在pH 5.0和pH 5.0~7.0条件下,残余酶活性分别保持在68%和50%以上(图3-B),表明木聚糖酶具有良好的耐酸性[16]。研究表明,来自于包括丙酮丁酸梭杆菌在内的不同生物体的木聚糖酶的最适pH值均在6.0[17]。
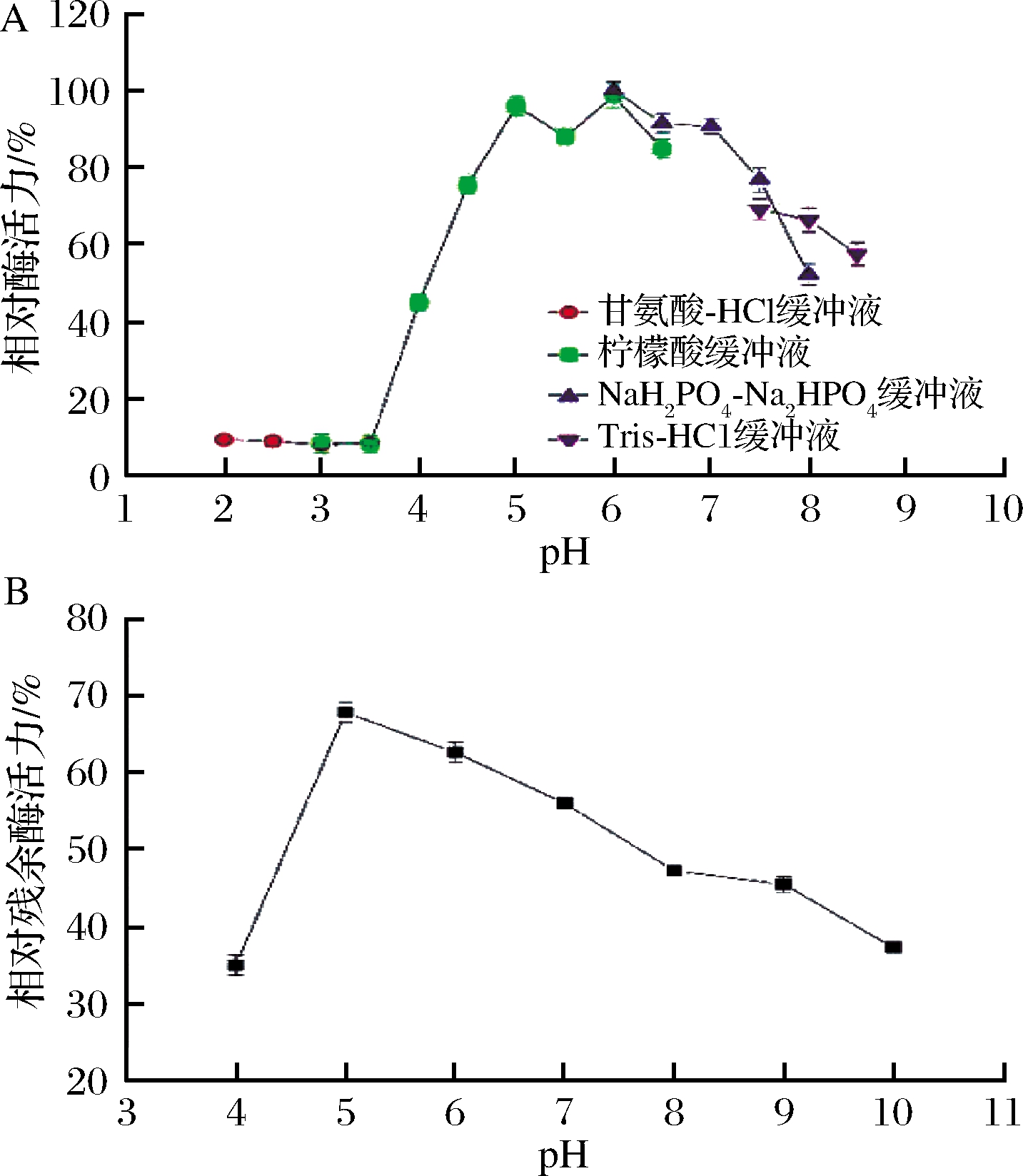
A-最佳pH;B-pH稳定性
图3 pH对木聚糖酶的影响
Fig.3 Effect of pH on xylanase
注:以未处理的木聚糖酶活性为100%
2.4 最佳温度和热稳定性
以山毛榉木聚糖为底物,纯化的木聚糖酶在不同温度下与底物进行反应。酶活性在30 ℃时较低,并且随着温度的升高而升高(图4-A)。该酶在60 ℃下表现出最大活性,高于其他菌株[18]。据相关研究报道,来源于Aspergillus sulphureus CGMCC0608菌株的木聚糖酶的最适温度仅为50 ℃[19],相比而言,菌株BA-TB-1的产生的木聚糖酶具有较好的热稳定性。在酶的生物催化过程中,温度对其催化效率有显著的影响,在较高温度下进行生物催化,酶可能失活[20-21]。如图4-A所示,实验中的木聚糖酶在温度超过90 ℃时,酶活性基本丧失。将酶置于不同温度下反应30 min,测定其热稳定性,图4-B显示残余酶活性随温度的升高而显著降低。60 ℃是食品工业应用木聚糖酶的理想温度,在此温度下,木聚糖酶可以增加面团的比体积和流变特性[18],在此温度下,酶的残余活性在30%左右。
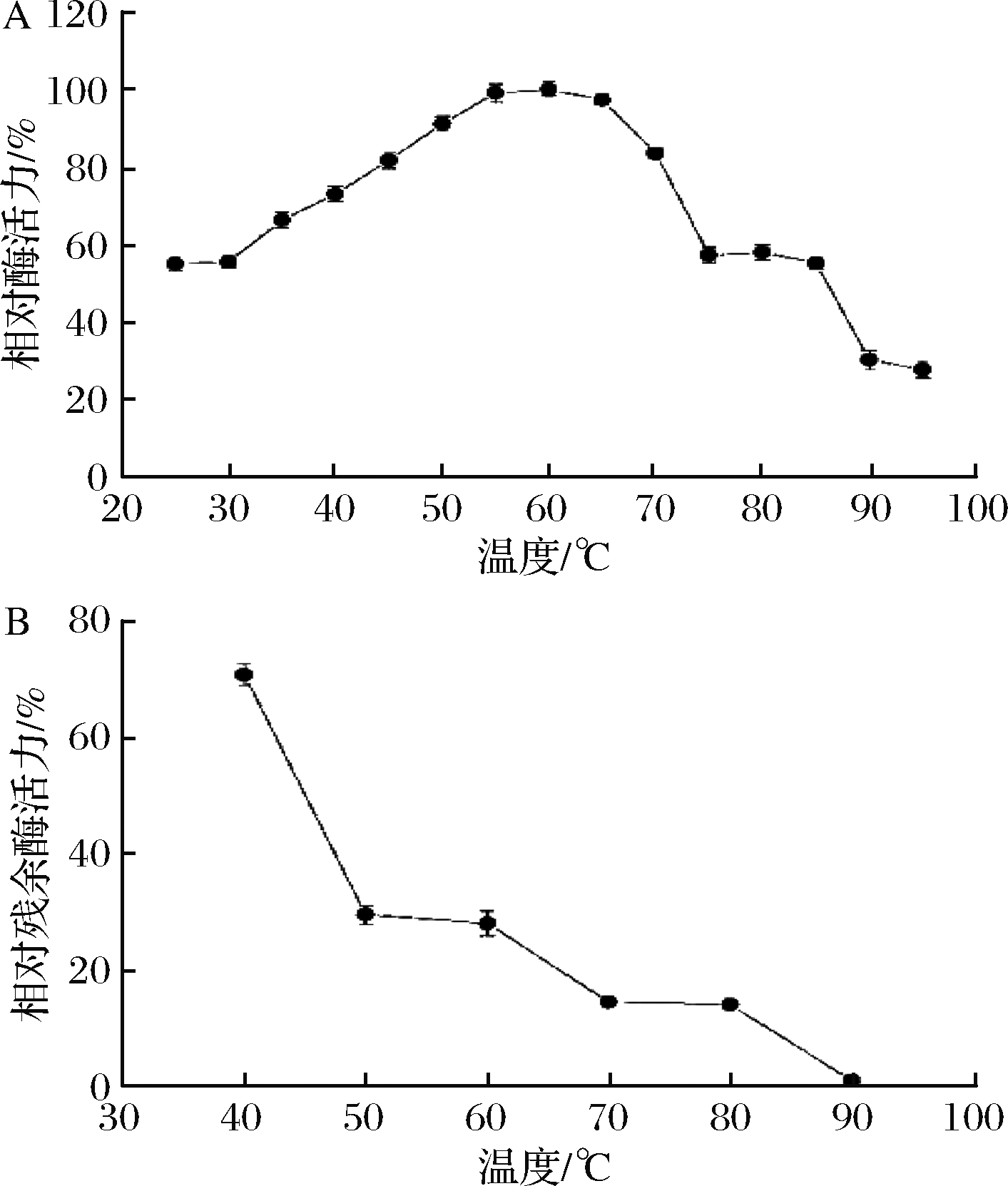
A-最佳温度;B-热稳定性
图4 温度对木聚糖酶的影响
Fig.4 Effect of temperature on xylanase
注:以未处理的酶活性为100%
2.5 木聚糖酶底物特异性和动力学参数
以山毛榉木聚糖为底物时的酶活力为100%。木聚糖酶对山毛榉木聚糖、桦木木聚糖、燕麦木聚糖的比活力分别为100%、65.53%、172.55%。山毛榉木聚糖的动力学常数Km和Vmax分别为(4.195±0.68) mg/mL和(1 757±6.31) mg/(min·μmol),低于大多数重组木聚糖酶的Km[22-23]。木聚糖酶对桦木木聚糖的比活力(65.53%)最低,对燕麦木聚糖的比活力(172.55%)最高,这与相关研究中的青霉菌木聚糖酶的结果相反[22]。当木聚糖酶和燕麦中的木聚糖相互作用时,活性位点的特征可能会改变[24]。Trichoderma inhamatum木聚糖酶对可溶性桦木木聚糖的催化专一性较高,这与本实验所研究的木聚糖酶相反[24]。
2.6 表达条件的优化
2.6.1 单因素实验
2.6.1.1 培养基优化
不同组成成分的培养基对促进细胞的生长,提升细胞生长数量有不同的效果,从而影响细胞的生长和调节[25]。添加不同碳源和氮源的培养基可促进重组纤维素酶的产量[26]。当选择2×YT培养基(含5 g/L酵母抽提物和16 g/L蛋白胨)时,其表达水平达到501.34 U/mL,略高于MTB培养基(含2.4 g/L酵母抽提物和12 g/L蛋白胨)(图5-A)。SUBRAMANIYAN等[27]用2.5 g/L蛋白胨和2.5 g/L酵母抽提物从芽孢杆菌SSP34中获得了最高的木聚糖酶产量。同时,一些报告表明,酵母提取物对芽孢杆菌产酶的影响最为重要[28]。这可能是因为以葡萄糖为碳源的培养基(SOC和SB)产生的细胞密度较低,而富集培养基(LB和MTB)提供的细胞密度较高,但不支持最佳的表达[25]。
2.6.1.2 诱导温度优化
诱导后培养温度的优化对于提高可溶性重组蛋白的产量具有重要意义[24]。当诱导温度过低时,酶活性没有实质性变化(图5-B)。当温度为24 ℃时,酶活性最高达到488.215 U/mL,与本实验的最佳诱导表达温度相同[25]。温度不仅对宿主细胞的生长有重要的影响,同时也影响异源性蛋白的合成。当诱导温度较高时,宿主生长加快,蛋白质合成速率过快,可能产生包涵体,导致细菌死亡[29]。
2.6.1.3 诱导时机优化
如图5-C所示,当诱导点为4 h时,木聚糖酶的酶活性达到最大值450.022 U/mL。重组酶在培养过程中主要依赖于细胞比生产力和细胞密度[30]。当培养时间过短时,细菌的代谢能力不强,细胞密度低,细菌的表达量低。然而,细菌相对较老时,培养基中的营养物质用于细胞的生长和繁殖,诱导点太晚时会导致酶的表达减少[31]。在低细胞密度的早期进行诱导会导致细胞代谢负担和生理负担,进而影响诱导后的蛋白质表达和细胞生长[32]。因此,有必要选择在适当的时间加入IPTG。
2.6.1.4 诱导时间优化
诱导时间影响大肠杆菌中表达蛋白的折叠、积累和产量[33]。当诱导时间太短时,菌群没有完全生长并繁殖到最佳状态,导致酶活性降低(图5-D),最适诱导时间为24 h,酶活性达到488.215 U/mL,诱导时间越长,会导致细胞衰老、自溶和活力下降,木聚糖酶活性越低。周质蛋白分泌随着诱导时间的延长而增加,这增加了其渗透性,削弱了外膜,这种延长培养时间的方法先前由DI Z-RINC
Z-RINC N[34]和DONOVAN[35]报道。
N[34]和DONOVAN[35]报道。
2.6.1.5 IPTG用量优化
IPTG是一种诱导分子,通常用于在lac操纵子下生产重组蛋白。目前研究表明,随着IPTG溶液用量的增加,重组蛋白的产量增加[25]。当500 mmol/L IPTG溶液用量为200 μL时,木聚糖酶显示出最大活性(图5-E)。但随着IPTG用量的增加,酶活性下降更为严重。选择最佳的诱导浓度时,可以确保膜的定位和高效运输,这也可以减少细胞的运转负荷[36]。然而,较高浓度的IPTG导致包涵体的形成[37],导致细菌死亡。
2.6.1.6 接种量优化
酶的生物合成受菌株年龄和接种量的影响[38]。图5-F表明,酶活性随着接种量的增加而呈现先升高后降低的情况,当接种量为3%时,酶活性最高,达到488 U/mL。过量接种时酶产量的下降可能是由于培养物生长速度加快和养分缺乏导致[39]。因此,最大的酶产量需要在有效养分和高产之间平衡[40]。
2.6.2 Central Composite试验设计与结果
依据单因素实验结果,运用Design-expert 8.0.6中Central Composite试验方法,采用响应面分析法对木聚糖酶的诱导表达条件中的影响因素500 mmol/L IPTG的用量、诱导时机、接种量进行优化,确定最佳的诱导表达条件。如表5所示,根据各个因素的F值,影响结果的次序为B>A>C,即影响因素IPTG用量>诱导点>接种量。模型的P值<0.000 1,差异显著,失拟项为0.125 8>0.05,表示结果差异不显著,说明模型是有效的。模型决定系数R2=0.997 5,表明木聚糖酶的实际酶活与预测值拟合度,校正决定系数 Adj R2=0.995 2。综上所述,该实验模型可进行真实值的分析,回归方程拟合度好,可用于木聚糖酶的表达优化。

A-培养基;B-诱导温度;C-诱导点;D-诱导时间;E-500 mmol/L IPTG用量;F-接种量
图5 木聚糖酶的表达优化条件
Fig.5 Optimization of expression conditions for xylanases
表2 Central Composite试验方案与结果
Table 2 Central Composite test plan and results
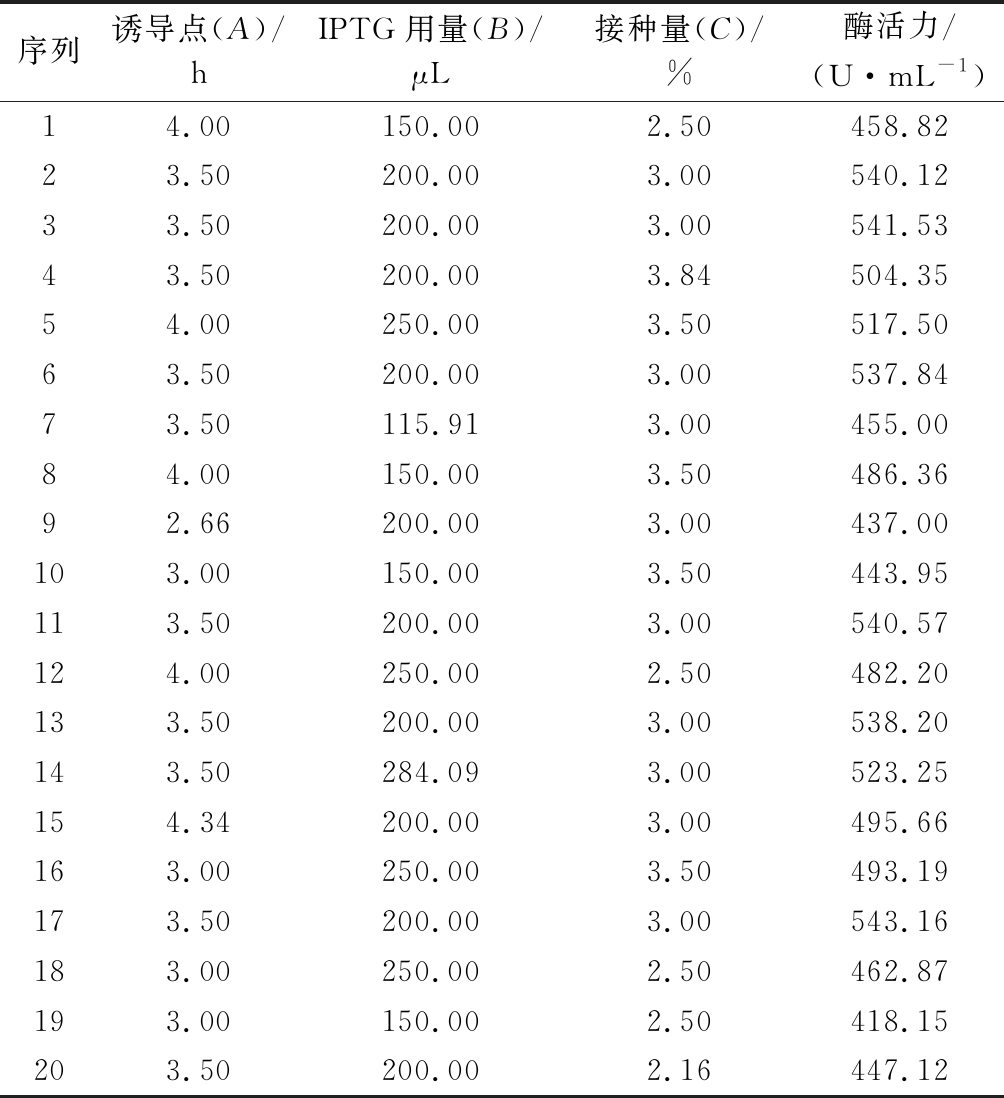
序列诱导点(A)/hIPTG用量(B)/μL接种量(C)/%酶活力/(U·mL-1)14.00150.002.50458.8223.50200.003.00540.1233.50200.003.00541.5343.50200.003.84504.3554.00250.003.50517.5063.50200.003.00537.8473.50115.913.00455.0084.00150.003.50486.3692.66200.003.00437.00103.00150.003.50443.95113.50200.003.00540.57124.00250.002.50482.20133.50200.003.00538.20143.50284.093.00523.25154.34200.003.00495.66163.00250.003.50493.19173.50200.003.00543.16183.00250.002.50462.87193.00150.002.50418.15203.50200.002.16447.12
2.6.3 响应面分析
根据Design-Expert 8.0.6软件分析得到的回归方程可知,预测的最佳表达优化条件为诱导点3.63 h、500 mmol/L IPTG 224.76 μL、接种量3.18%,此条件下的预测值为550.139 U/mL。为了实验的可操作性,取诱导点3.6 h、500 mmol/L IPTG 225 μL、接种量3.8%,在此实验条件下进行3次平行实验,平均木聚糖酶的活力为548.87 U/mL。实际值与预测值十分接近,说明响应面优化木聚糖酶的表达是可行的。
表3 回归模型方差分析
Table 3 Analysis of variance for regression models
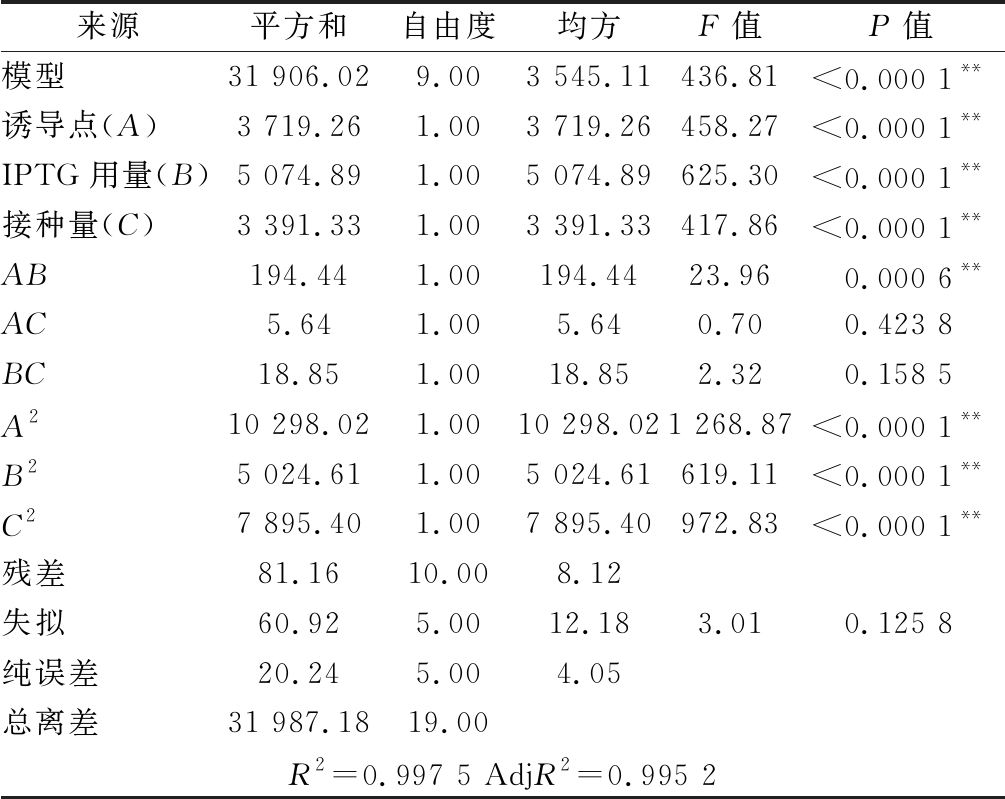
来源平方和自由度均方F值P值模型31 906.029.003 545.11436.81<0.000 1**诱导点(A)3 719.261.003 719.26458.27<0.000 1**IPTG用量(B)5 074.891.005 074.89625.30<0.000 1**接种量(C)3 391.331.003 391.33417.86<0.000 1**AB194.441.00194.4423.960.000 6**AC5.641.005.640.700.423 8BC18.851.0018.852.320.158 5A210 298.021.0010 298.021 268.87<0.000 1**B25 024.611.005 024.61619.11<0.000 1**C27 895.401.007 895.40972.83<0.000 1**残差81.1610.008.12失拟60.925.0012.183.010.125 8纯误差20.245.004.05总离差31 987.1819.00R2=0.997 5 AdjR2=0.995 2
注:**表示结果差异极显著(P<0.001);*表示结果差异显著(P<0.05)
3 结论
本文从解淀粉芽孢杆菌中克隆得到一个木聚糖酶基因,并在大肠杆菌DH5α中克隆保留,之后转入到BL21(DE3)中进行了异源表达。通过研究木聚糖酶的酶学性质,发现该木聚糖酶具有较好的耐酸性,这能为木聚糖酶在食品和动物饲料添加剂中的应用提供一定的理论依据。同时本实验采用响应面分析法来优化木聚糖酶的诱导表达条件,在最佳的诱导表达条件,获得更加稳定、高质量的木聚糖酶。木聚糖酶在中温条件下具有较高的耐热性,在较宽pH范围内的稳定性,可用于多种生物技术应用,且具有较高的耐热性和耐酸性,可以很好地应用于工业生产。

图6 诱导点、500 mmol/L IPTG的用量和接种量交互作用图
Fig.6 Interaction plot of inducing point, 500 mmol/L IPTG, and inoculation amount
[1] SANJIVKUMAR M, SILAMBARASAN T, BALAGURUNATHAN R, et al.Biosynthesis, molecular modeling and statistical optimization of xylanase from a mangrove associated actinobacterium Streptomyces variabilis (MAB3) using Box-Behnken design with its bioconversion efficacy[J]. International Journal of Biological Macromolecules,2018, 118:195-208.
[2] SØRENSEN H.Enzymatic hydrolysis of xylan[J].Nature,1953, 172(4 372):305-306.
[3] LI Q, SUN B G, XIONG K, et al.Improving special hydrolysis characterization into Talaromyces thermophilus F1208 xylanase by engineering of N-terminal extension and site-directed mutagenesis in C-terminal[J].International Journal of Biological Macromolecules,2017,96:451-458.
[4] MONTANÉ D, NABARLATZ D, MARTORELL A, et al.Removal of lignin and associated impurities from xylo-oligosaccharides by activated carbon adsorption[J].Industrial and Engineering Chemistry Research,2006, 45(7):2 294-2 302.
[5] KHURANA S, KAPOOR M, GUPTA S, et al.Statistical optimization of alkaline xylanase production from Streptomyces violaceoruber under submerged fermentation using response surface methodology[J].Indian Journal of Microbiology,2007,47(2):144-152.
[6] 刘明启,孙建义,郭爽.木聚糖酶嗜酸性、热稳定性研究进展[J].中国饲料,2004(13):21-22;25.
LIU Q M, SUN J Y, GUO S.Advances in the study of eosinophilic and thermal stability of wood polysaccharides[J].China Feed, 2004(13):21-22;25.
[7] 曹钰,陆健,李胤.酸性木聚糖酶的研究进展[J].工业微生物,2005(4):41-44;50.
CAO Y, LU J, LI Y, et al.Research advances in acid-stable xylanase[J].Industrial Microbiology,2005(4):41-44;50.
[8] VIIKARI L, KANTELLINEN A, SUNDQUIST J, et al.Xylanases in bleaching:From an idea to the industry[J].FEMS Microbiol Rev,1994,13(2-3):335-350.
[9] PRADE R A.Xylanases:From biology to biotechnology[J].Biotechnol Genet Eng Rev, 1996, 13:101-131.
[10] 王雅珍,李秀婷,孙宝国,等.微生物酸性木聚糖酶及其应用的研究进展[J].食品与发酵工业,2012,38(8):107-113.
WANG Y Z, LI X T, SUN B G, et al.Study on microbial acidic xylanase and its application[J].Food and Fermentation Industries, 2012, 38(8):107-113.
[11] GALBE M, SASSNER P, WINGREN A, et al.Process engineering economics of bioethanol production[J].Advances in Biochemical Engineering/Biotechnology,2007,108:303-327.
[12] SRIKANTH R, SIDDARTHA G, REDDY C H S S S, et al.Antioxidant and anti-inflammatory levan produced from Acetobacter xylinum NCIM2526 and its statistical optimization[J].Carbohydrate Polymers,2015,123:8-16.
[13] LI Q, SUN B G, LI X, et al.Improvement of the catalytic characteristics of a salt-tolerant GH10 xylanase from Streptomyce rochei L10904[J].International Journal of Biological Macromolecules, 2018,107:1 447-1 455.
[14] BAILEY M J, BIELY P, POUTANEN K.Interlaboratory testing of methods for assay of xylanase activity[J].Journal of Biotechnology,1992,23(3):257-270.
[15] MILLER G L.Use of dinitrosalicylic acid reagent for determination of reducing sugar analytical chemistry[J] Analytical Chemistry 1959,31(3): 426-428.
[16] LI Y H, ZHANG B, CHEN X, et al.Improvement of Aspergillus sulphureus endo-β-1,4-xylanase expression in Pichia pastoris by Codon optimization and analysis of the enzymic characterization[J].Applied Biochemistry and Biotechnology, 2010,160(5):1 321-1 331.
[17] ZAPPE H, JONES D T, WOODS D R, et al.Cloning and expression of a xylanase gene from Clostridium acetobutylicum P262 in Escherichia coli[J].Applied Microbiology and Biotechnology, 1987,27(1):57-63.
[18] LIU T Y, ZHANG J G.High-level expression and characterization of Aspergillus niger ATCC 1015 xylanase B in Komagataella phaffii[J].Applied Biological Chemistry,2018, 61(4):373- 381.
[19] WONG K K, TAN L U, SADDLER J N.Multiplicity of beta-1,4-xylanase in microorganisms:Functions and applications[J].Microbiological Reviews, 1988, 52(3):305-317.
[20] CHEN Y H, LI L L, LONG L K, et al.High cell-density cultivation of phenolic acid decarboxylase-expressing Escherichia coli and 4-vinylguaiacol bioproduction from ferulic acid by whole-cell catalysis[J].Journal of Chemical Technology and Biotechnology,2018, 93:2 415-2 421.
[21] HU H F, LI L L, DING S J.An organic solvent-tolerant phenolic acid decarboxylase from Bacillus licheniformis for the efficient bioconversion of hydroxycinnamic acids to vinyl phenol derivatives[J].Applied Microbiology and Biotechnology,2015, 99(12):5 071-5 081.
[22] LI Q, WU Q H, SUN B G, et al.Effect of disulfide bridge on hydrolytic characteristics of xylanase from Penicillium janthinellum[J].International Journal of Biological Macromolecules, 2018, 120: 405-413.
[23] LI Q, SUN B G, JIA H Y, et al.Engineering a xylanase from Streptomyce rochei L10904 by mutation to improve its catalytic characteristics[J].International Journal of Biological Macromolecules, 2017, 101:366-372.
[24] BAGEWADI Z K, MULLA S I, NINNEKAR H Z.Purification, characterization, gene cloning and expression of GH-10 xylanase (Penicillium citrinum isolate HZN13)[J].3 Biotech, 2016, 6(2):1-9.
[25] WEE M Y J, MURAD A M A, BAKAR F D A, et al.Expression of xylanase on Escherichia coli using a truncated ice nucleation protein of Erwinia ananas (InaA)[J].Process Biochemistry, 2019,78:25-32.
[26] KHALID A, TAYYAB M, HASHMI A S, et al.Optimization of conditions for maximal production of recombinant thermostable cellulase from Thermotoga naphthophila using E.coli BL21-codonPlus (DE3) as expression host[J].Pakistan journal of zoology, 2019, 51(4).
[27] SUBRAMANIYAN S, SANDHIA G S, PREMA P, et al.Control of xylanase production without protease activity in Bacillus sp. by selection of nitrogen source[J].Biotechnology Letters, 2001, 23(5):369-371.
[28] KR S.Cassava bagasse-low cost substrate for thermo-tolerant xylanase production using Bacillus subtilis[J].International Journal of Chem Tech Research, 2013, 5(1):394-400.
[29] GOLOTIN V A, BALABANOVA L A, NOSKOVA Y A, et al.Optimization of cold-adapted alpha-galactosidase expression in Escherichia coli.[J].Protein Expression and Purification,2016, 123:14-18.
[30] WANG H L, LI X M, MA Y H,et al.Process optimization of high-level extracellular production of alkaline pectate lyase in recombinant Escherichia coli BL21 (DE3)[J].Biochemical Engineering Journal,2015, 93:38-46.
[31] ZHANG H, SUN X, LI W J, et al.Expression and characterization of recombinant sucrose phosphorylase[J].Protein Journal,2018, 37(1): 93-100.
[32] CLAUSEN M, LAMB C J, MEGNET R, et al.PAD1 encodes phenylacrylic acid decarboxylase which confers resistance to cinnamic acid in Saccharomyces cerevisiae[J].Gene,1994, 142(1):107-112.
[33] MALIK A, ALSENAIDY A M, ELROBH M, et al.Optimization of expression and purfication of HSPA6 protein from Camelus dromedarius in E.coli.[J].Saudi Journal of Biological Sciences,2016,23(3):410-419.
[34] DI Z-RINC
Z-RINC N D J, DUQUE I, OSORIO E, et al.Production of recombinant Trichoderma reesei cellobiohydrolase II in a new expression system based on Wickerhamomyces anomalus[J].Enzyme Research, 2017, 2017:6 980 565.
N D J, DUQUE I, OSORIO E, et al.Production of recombinant Trichoderma reesei cellobiohydrolase II in a new expression system based on Wickerhamomyces anomalus[J].Enzyme Research, 2017, 2017:6 980 565.
[35] DONOVAN R S, ROBINSON C W, CLICK B R.Review:Optimizing inducer and culture conditions for expression of foreign proteins under the control of the lac promoter[J].Journal of Industrial Microbiology,1996, 16(3),145-154.
[36] LI Q Q, QI Y, CHEN J S, et al.Molecular characterization of an ice nucleation protein variant (InaQ) from Pseudomonas syringae and the analysis of its transmembrane transport activity in Escherichia coli[J].International Journal of Biological Sciences, 2012, 8(8):1 097-1 108.
[37] FAN L H, LIU N, YU M G, et al.Cell surface display of carbonic anhydrase on Escherichia coli using ice nucleation protein for CO2 sequestration[J].Biotechnology and Bioengineering,2011,108:2 853-2 864.
[38] LIU J G, XING J M, CHANG T S,et al.Optimization of nutritional conditions for nattokinase production by Bacillus natto NLSSE using statistical experimental methods[J].Process Biochemistry,2005, 40(8):2 757-2 762.
[39] ROUSHDY M M, DESOUKY S E, ESMAEL M E, et al.Optimization and characterization of tannin acyle hydrolase produced by Aspergillus flavus var.columnaris using solid state fermentation technique[J].In New York Science Journal,14,7(3):88-98.
[40] SANDHYA C, SUMANTHA A, SZAKACS G, et al.Comparative evaluation of neutral protease production by Aspergillus oryzae in submerged and solid-state fermentation[J].Process Biochemistry,2005, 40(8):2 689-2 694.