佛手(Citrus medica L. var. sarcodactylis Swingle) 又名佛手柑、五指橘,为芸香科柑橘属植物香橼的变种,因其形如手指故得名[1]。佛手药食同源,在我国种植和药用历史悠久,早在《本草纲目》[2]中就有记载,佛手柑“下气,除心头痰水;煮酒饮,治痰多咳嗽;煮汤,治心下气痛”。成熟的佛手中含有黄酮类、多酚、香豆素、活性多糖、蛋白质和矿物质等多种成分[3-4],具有和胃健脾、舒肝理气、止咳化痰、抗肿瘤等多种生理功效[5-6]。由佛手果经历九蒸九晒后制成的色黑如漆、绵软适口的老香黄是潮汕地区特有的药用凉果,受到潮汕人民的青睐[7]。其具有消积祛风、开胃理气、化痰生津等多种功效,而且久藏不坏,随着贮藏时间的延长其药效越佳[8]。
对样品中微生物的分析鉴定,传统的方法是先对样品中的微生物进行富集培养,然后分离培养获得纯培养物,再对其进行形态观察和生理生化反应鉴定,或提取纯培养物的DNA结合一代16S rRNA基因测序并与菌种库序列进行比对得出结论。但由于培养条件、微生物营养要求等方面的限制,能够培养的微生物往往只占其中所有微生物的很小一部分,因而传统纯培养的分析结果不能完全反映样本中微生物菌群情况。随着测序技术的发展,高通量测序在微生物菌群的研究表现出越来越突出的优势,目前已广泛应用于环境水土污染[9-10]、食品[11]、人体胃肠道[12]等多个方面。与传统分离鉴定方法相比,高通量测序技术无需经过微生物分离培养过程而是直接在分子层面进行测序,它既可以检出难以培养的优势微生物,还能确定样品中大多数微生物的相对丰度[13-14]。
老香黄作为佛手的腌制品,腌制年限越久药效越佳。但目前国内外尚无对老香黄的微生物多样性进行分析,为了初步了解加工前后微生物的变化以及老香黄药用功效与微生物之间的联系,本研究以加工前的佛手鲜果与其腌制品——老香黄为研究对象,采用高通量测序技术分析加工前后样品中的菌群组成及其变化,并结合纯培养方法进行分析比较,为老香黄的食用安全性与药用功效研究提供参考依据,为佛手的开发利用奠定基础。
1 材料与方法
1.1 材料
佛手鲜果和老香黄由广东某食品公司提供,以无菌采样袋采集后置于4 ℃恒温箱中于12 h内运至实验室,在超净工作台内进行无菌分割,于-80 ℃下保存。佛手鲜果编号为FF,老香黄编号为EP,编号1、2表示来自不同的鲜果和老香黄样品。
1.2 方法
1.2.1 微生物的分离培养和鉴定
1.2.1.1 分离培养
配制营养琼脂培养基(nutrient agar, NA)、GAM琼脂培养基、MRS培养基和改良MRS培养基,制作平板备用。分别称取佛手鲜果和老香黄各10 g,加入90 mL无菌生理盐水,在无菌条件下匀浆,制成10-1稀释液,并依次制备10-2、10-3和10-4等稀释液。取100 μL稀释液涂布于NA平板,将平板分别置于30 ℃和37 ℃恒温培养箱中培养36~48 h;在厌氧工作站中取100 μL稀释液涂布于GAM平板、MRS平板和改良MRS平板,于37 ℃无氧条件下培养36~48 h。各稀释度做3个平行。根据菌落的大小、形态、颜色等形态学特征挑取不同的单菌落,并划线纯化培养3次。
1.2.1.2 菌种鉴定
将分离纯化后的菌株进行革兰氏染色和镜检观察。同时采用SDS法提取菌株的DNA,以细菌通用引物27F(5’-AGAGTTTGATCCTGGCTCAG-3’)和1492R(5’-GGTTACCTTGTTACGACTT-3’)进行PCR扩增。PCR产物用2%的琼脂糖凝胶电泳进行检测,出现清晰的条带,将其送至上海生工进行测序,将所得序列在NCBI上进行序列比对。
1.2.2 高通量测序
1.2.2.1 基因组DNA的提取和PCR扩增
采用CTAB法[15]对样本的基因组DNA进行提取,以琼脂糖凝胶电泳检测DNA的纯度,取适量的样本DNA于离心管中,以无菌水稀释至1ng/μL备用。以稀释后的基因组DNA为模板,以16S V4区引物(515F和806R)进行PCR扩增。
515F:5’-GTTTCGGTGCCAGCMGCCGCGGTAA-3’
806R:5’-GCCAATGGACTACHVGGGTWTCTAAT-3’
1.2.2.2 PCR产物的混样和纯化
PCR产物以2%琼脂糖凝胶电泳进行检测,根据PCR产物浓度进行等量混样,充分混匀后以1×TAE的2%琼脂糖凝胶电泳纯化PCR产物,剪切回收目标条带,以Thermo Scientific公司GeneJET胶回收试剂盒回收产物。
1.2.2.3 文库构建和上机测序
采用Thermofisher公司的Ion Plus Fragment Library Kit 48 rxns建库试剂盒进行文库的构建,构建的文库经过Qubit定量和检测合格后,使用Thermofisher的Ion S5TMXL进行上机测序(由北京诺禾致源生物信息科技有限公司协助完成)。
1.2.2.4 测序数据处理与分析
采用Cutadapt先对reads进行低质量部分剪切等方法得到原始数据(raw reads),将reads序列通过BugBase[16]与物种注释数据库进行比对检测嵌合体序列并去除其中的嵌合体序列,得到最终的有效数据(clean reads)。利用Uparse软件[17]对所有样品的全部clean reads进行聚类,默认以97%的一致性将序列聚类成为OTUs(operational taxonomic units),最后用Mothur方法与SILVA132的SSUrRNA数据库对OTUs序列进行物种注释分析。
2 结果与分析
2.1 纯培养结果
在37 ℃下培养的NA平板上从佛手鲜果中分离到7种不同形态的菌株,将分离的菌株进行革兰氏染色、形态学观察与16S rRNA基因鉴定,结果表明分离的细菌以枯草芽孢杆菌(Bacillus subtilis)为主,还有产气克雷伯氏菌(Klebsiella aerogenes)、粘质沙雷氏菌(Serratia marcescens)、Rhodococcus hoagii、Enterobacter asburiae、Glutamicibacter creatinolyticus和假单胞菌(Pseudomonas sp.);老香黄中只分离出了枯草芽孢杆菌。菌落形态如图1所示。
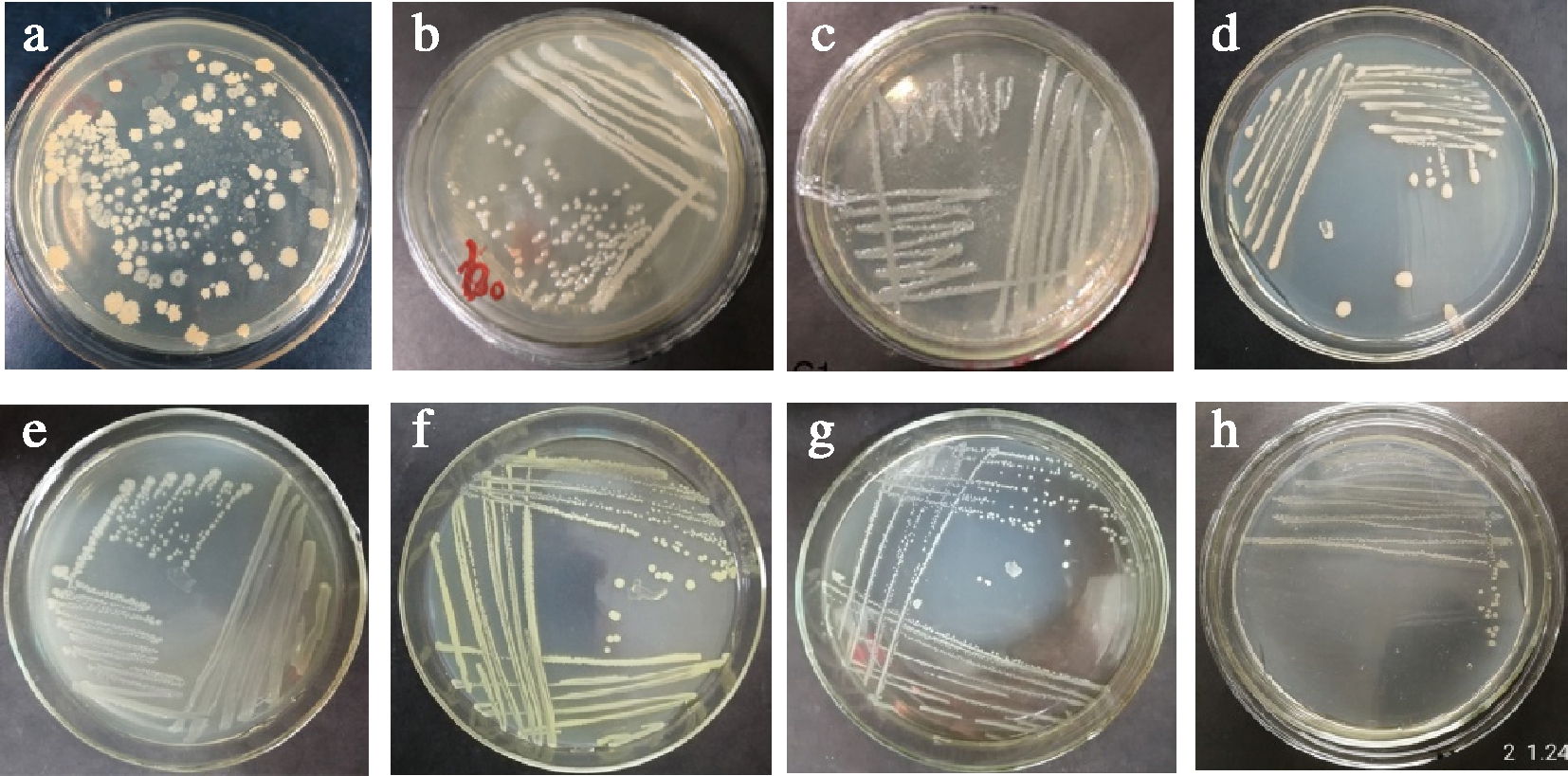
a、b、c、d、e、f、g为从鲜果中分离,h为从老香黄中分离的
细菌的菌落形态
a-B.subtilis;b-K.aerogenes;c-S.marcescens;
d-R.hoagii;e-E.asburiae;f-G.creatinolyticus;
g-Pseudomonas sp.;h-B.subtilis
图1 在NA培养基上37 ℃培养的菌株的菌落形态
Fig.1 Colony morphologies of the strains on NA at 37 ℃
30 ℃下,以NA平板从鲜果中分离出1株菌,经鉴定为分散泛菌(Pantoea dispersa),从老香黄中分离到1株菌,为枯草芽孢杆菌,菌落形态如图2所示。GAM平板、MRS平板和改良MRS平板上经过36~48 h的培养并未有微生物生长。分离菌株的16S rRNA基因测序及Blast比对结果见表1,比对相似度均达99%以上。
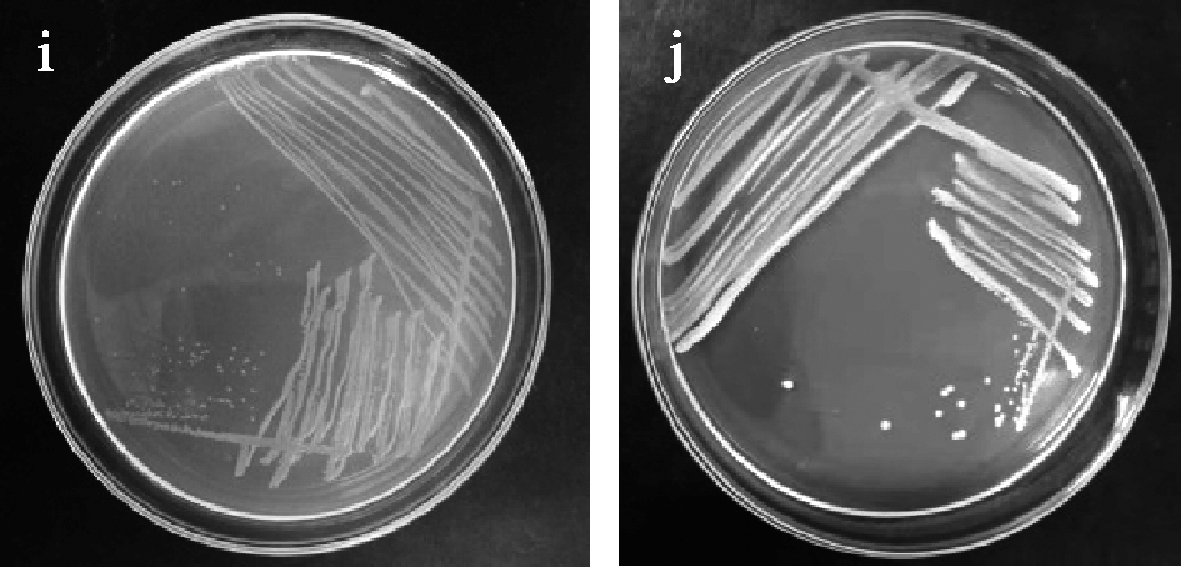
i-鲜果中分离菌株;j-老香黄中分离菌株
图2 在NA培养基上30 ℃下培养的菌株的菌落形态
Fig.2 Colony morphologies of the strains on NA at 30 ℃
表1 菌株16S rRNA基因比对结果
Table 1 16S rRNA gene alignment results of the strains
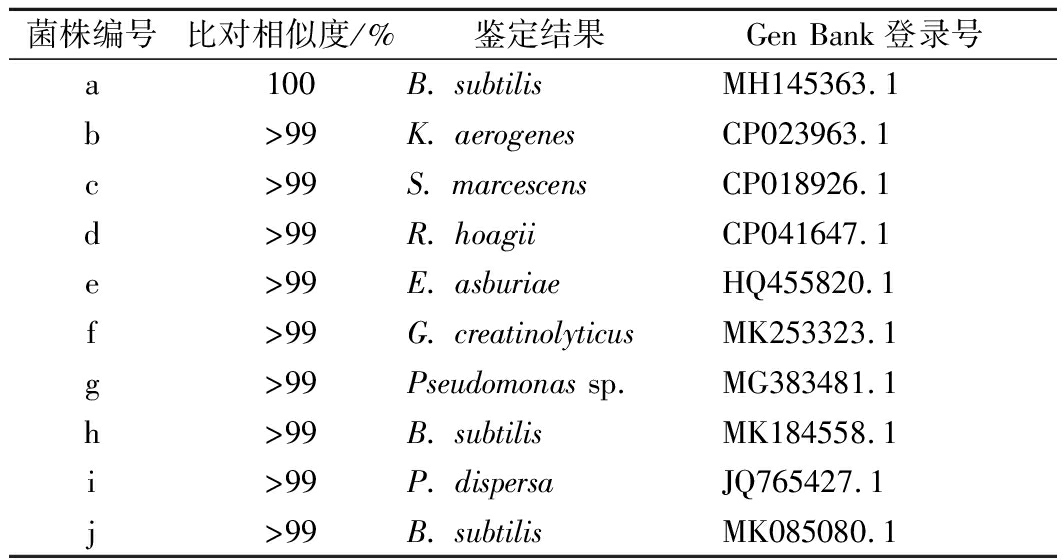
菌株编号比对相似度/%鉴定结果Gen Bank登录号a100B. subtilisMH145363.1b>99K. aerogenesCP023963.1c>99S. marcescensCP018926.1d>99R. hoagiiCP041647.1e>99E. asburiaeHQ455820.1f>99G. creatinolyticusMK253323.1g>99Pseudomonas sp.MG383481.1h>99B. subtilisMK184558.1i>99P. dispersaJQ765427.1j>99B. subtilisMK085080.1
2.2 高通量测序分析结果
2.2.1 测序样本数据统计与OTU分析
对4个样品16S V4区测序,采用Ion S5TMXL测序平台得到295 352条原始数据,经拼接、过滤,得到有效序列279 199条,其平均长度为417 bp,按照97%相似度分类,对有效数据进行聚类,得到678个OTUs,结果如表2所示,测序所得的有效序列达到细菌多样性分析的要求。
表2 测序数据统计结果
Table 2 Statistic results of sequence data

样品名称下机序列数有效序列数有效比例/%序列长度FF184 25380 18695.17414FF283 54680 15295.94407EP168 17264 34994.39426EP259 38154 51291.8422
注:FF1、FF2为佛手鲜果,EP1、EP2为老香黄
稀释性曲线(rarefaction curve)是通过对序列进行随机抽样,从样本中随机抽取一定测序量的数据,统计它们所代表物种数目(即OTUs数目)。以抽取数据量为横坐标,对应的物种数为纵坐标构建曲线,曲线越平坦说明增加更多的测序量只会出现少量的新物种(OTUs),直接反映测序量是否合理,间接反映物种的丰富程度。等级聚类曲线(rank abundance)是将样本中的OTUs按相对丰度由大到小排序得到对应的排序编号,以OTUs的排序编号为横坐标,OTUs中的相对丰度为纵坐标,再将这些点用折线连接,可直观地反映样本中物种的丰富度和均匀度。横向看,丰富度越高曲线跨度越大;纵向看,曲线越平缓,物种分布越均匀[18]。如图3-A所示,随着测序数量的增加,每个样品的稀释曲线趋于平坦,在测序量为30 000上基本趋于平坦,说明增加测序量,不会出现新的OTUs或者很少出现新的OTUs。说明本次测序数据量合理并且测序数据量足够大,可以反映样本中绝大多数的细菌多样性信息[19],由图3-B可知,EP2在横坐标的跨度最大、曲线最平缓,说明在EP2中细菌的丰富度最高且最均匀。
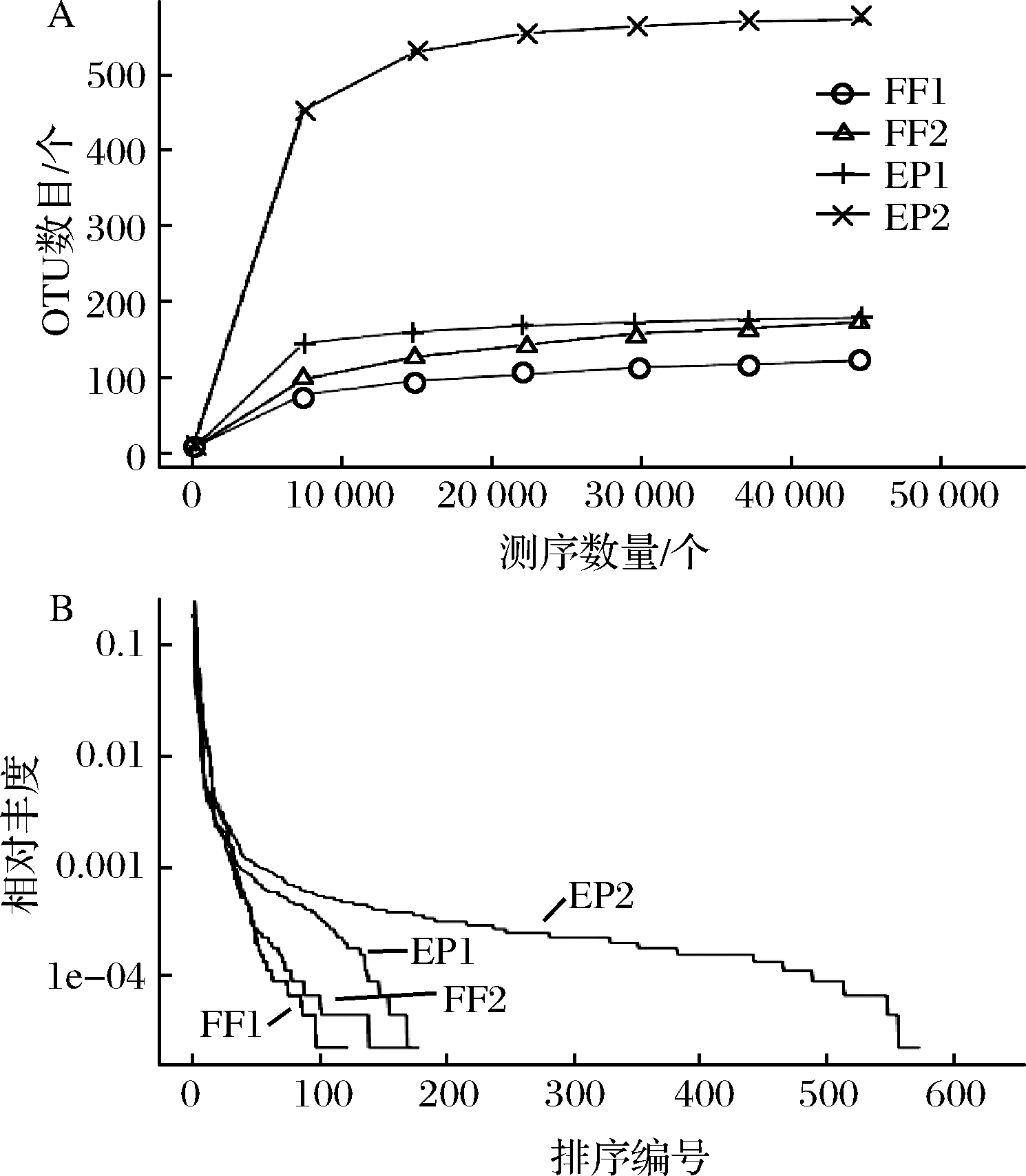
图3 稀释性曲线(A)和等级聚类曲线(B)
Fig.3 Rarefaction curve(A)and rank abundance(B)
2.2.2 样品中所含OTU数目分析
采用Qiime软件,得到每个OTU的分类学信息,通过R软件,绘制维恩图。FF1、FF2、EP1和EP2的OTU数目分别为122、173、179和573,FF1与FF2共有的OTU有97个,EP1与EP2共有的OTU有117个,4种样品中共有的OTU有44个(图4)。EP2中所含的OTU数目最多,按共有OTU的比例来看,FF1与FF2中的OTU共有率比EP1与EP2高,说明FF1与FF2的物种组成更加相近。
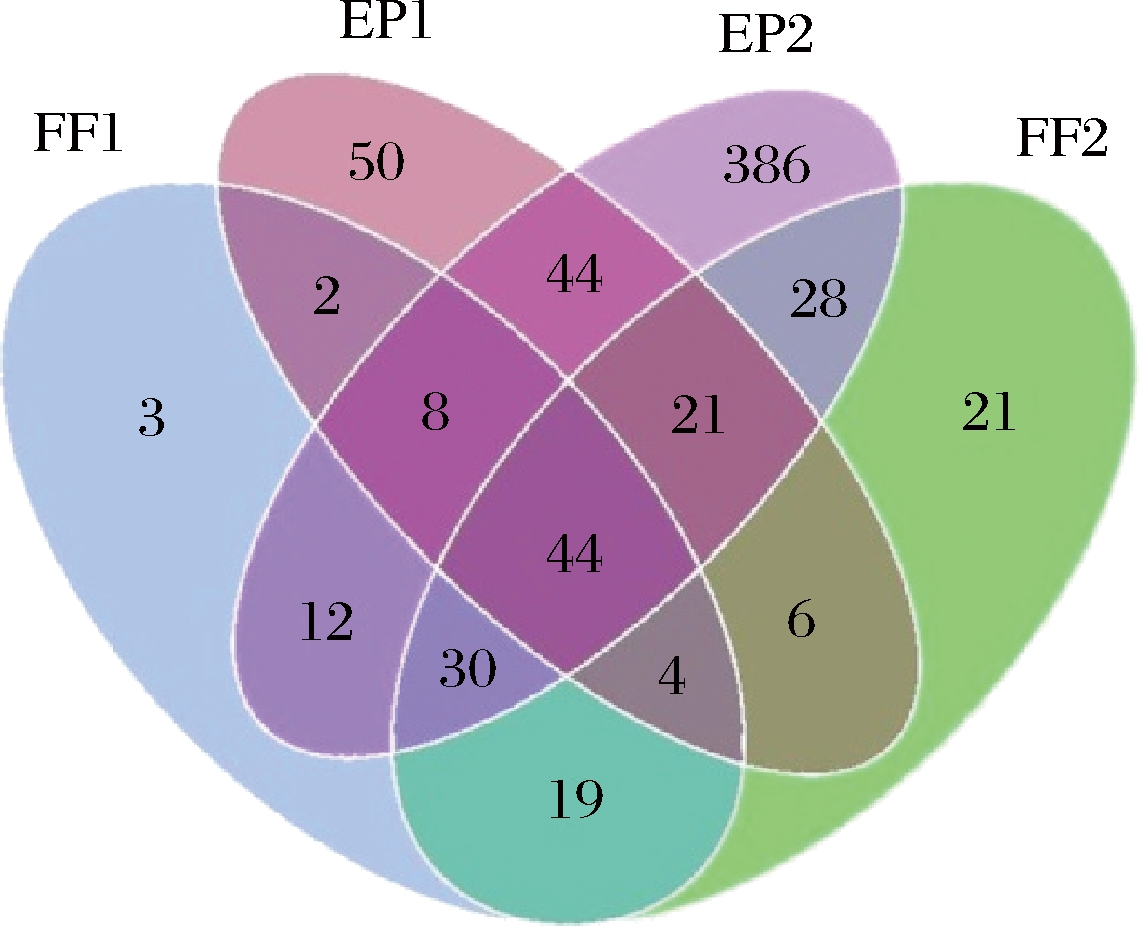
图4 OTU的维恩图
Fig.4 Venn diagram of OTU
2.2.3 Alpha多样性分析
根据97%相似性水平下的OTU信息,采用Alpha多样性指标的Shannon、Simpson、ACE、Chao1及Coverage指数对样品微生物物种的丰富度和多样性进行评估。结果如表3所示,各样本的Coverage都在0.999及以上,说明覆盖面达到99.9%,序列没有被检出的可能性很小,能够提供充足的序列进行微生物多样性分析。样品EP2的Shannon、Chao1、ACE指数明显高于其他3种样品,说明EP2中细菌多样性和丰富度高于其他样品;EP1与EP2同为老香黄,但其细菌的多样性低于EP2,可见老香黄在加工完成后不同的批次存在着差异,其细菌的多样性可能与原料鲜果及生产过程中的环境微生物相关。
表3 Alpha多样性指数
Table 3 Alpha diversity index
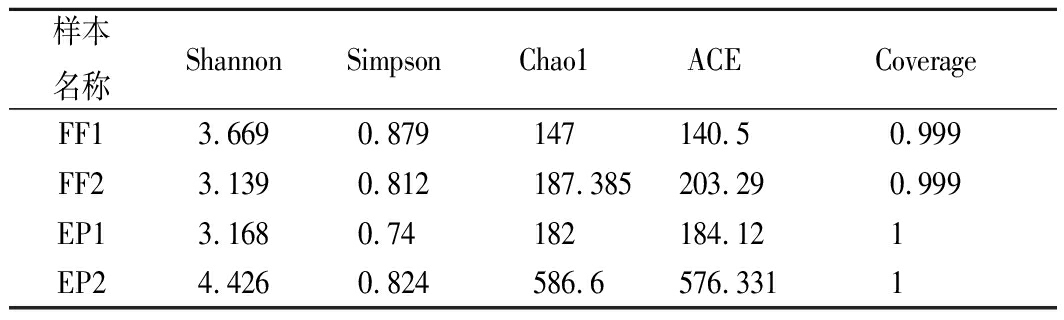
样本名称ShannonSimpsonChao1ACECoverageFF13.6690.879147140.50.999FF23.1390.812187.385203.290.999EP13.1680.74182184.121EP24.4260.824586.6576.3311
2.2.4 细菌菌群结构分析
2.2.4.1 基于门水平的细菌菌群结构分析
门水平上,相对丰度前10的物种如图5所示,FF1中相对丰度比较大的主要有变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria),分别占44.6%、35.9%;FF2中有蓝细菌门、变形菌门,分别占56.0%、9.7%;EP1中有变形菌门、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),分别占61.4%、32.8%、2.7%和1.4%;EP2中有厚壁菌门、变形菌门、蓝细菌门、拟杆菌门、放线菌门和酸杆菌门(Acidobacteria),分别占42.3%、41.7%、5.3%、3.3%、2.4%和1.6%。佛手鲜果在制作老香黄过程中变形菌门、厚壁菌门、拟杆菌门和放线菌门细菌有所增加,蓝细菌门细菌比例减少。变形菌门是细菌中最大的一个门,其均为革兰氏阴性菌,广泛存在于自然界中。许多厚壁菌门的细菌可以产生芽孢,它可以抵抗脱水和极端环境,并且在很多不同环境中都可以找到芽孢,这可能是厚壁菌门细菌能在高盐、高糖的加工品老香黄中仍然出现的原因。KAOUTARI等[20]构建以厚壁菌门、拟杆菌门为主要丰度的微生物区系探究人体肠道微生物内碳水化合物活性酶的分布和丰度,结果显示,厚壁菌门和拟杆菌门微生物基因主要编码碳水化合物活性酶(carbohydrate-active enzymes,CAZyme),而CAZyme的主要功能是分解碳水化合物,因此这可能与老香黄开胃、助消化的功效有关系。酸杆菌门菌株最早由KISHIMOTO等[2]在1991年从酸性矿山环境中分离,并于1995年将酸杆菌划分为新的细菌类群。PANKRATOV等[21]从酸性泥炭土中分离出1株酸杆菌株TPB6017,其具有降解纤维素等多糖类物质的功能。但由于大多数酸杆菌门的微生物处于难以培养的状态,目前学术界对其自然分布以及生态功能还不甚了解,多为借助新型的分子生物学技术对酸杆菌菌群结构和生理学特征进行研究。
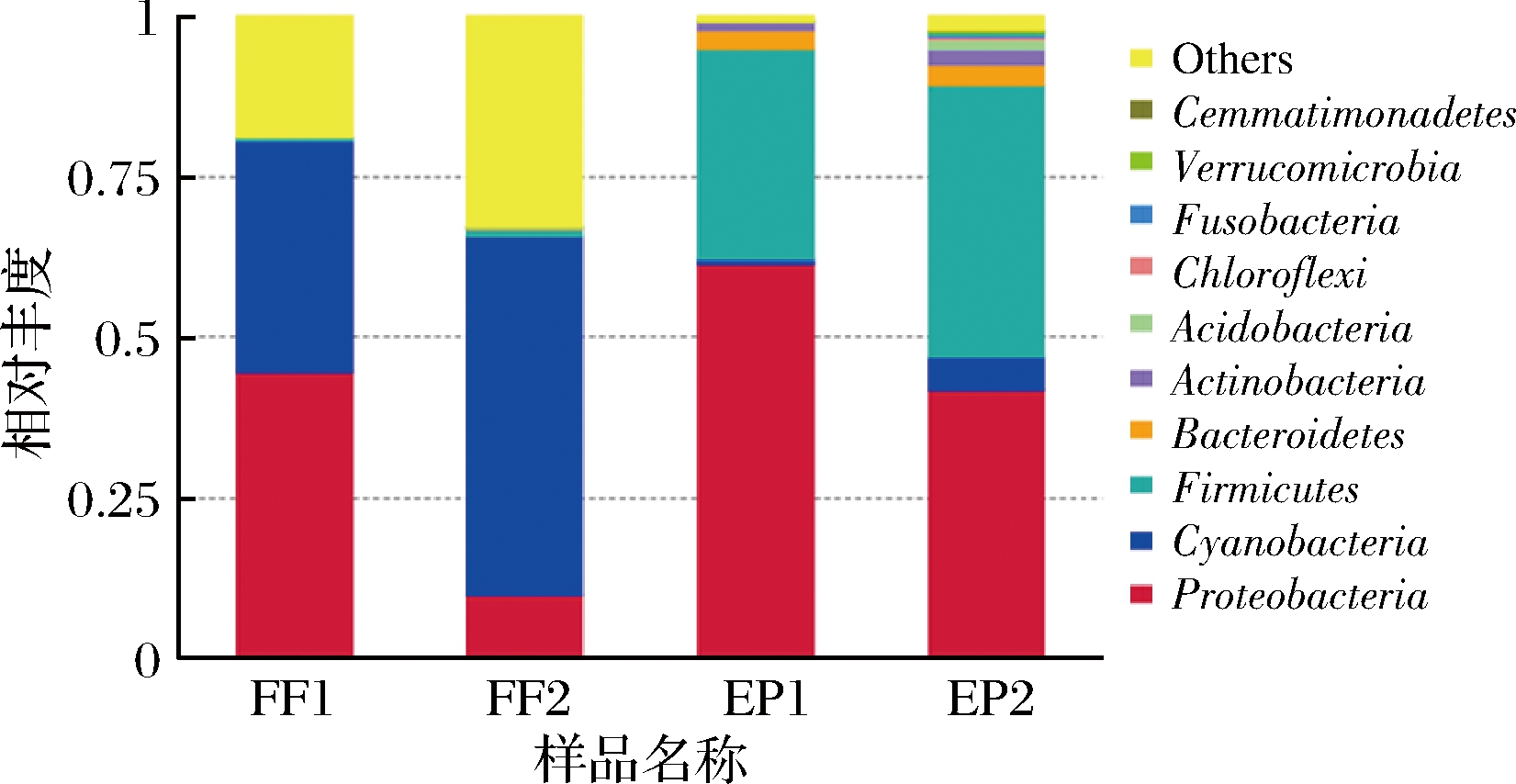
图5 门水平上的菌群相对丰度
Fig.5 Relative abundance of bacteria at phylum level
2.2.4.2 基于科水平的细菌菌群结构分析
科水平上,FF1中相对丰度较大的有肠杆菌科(Enterobacteriaceae)、unidentified Cyanobacteria和unidentified Rickettsiales,分别占40.5%、35.9%和1.2%;FF2中相对丰度较大的有unidentified Cyanobacteria、肠杆菌科、unidentified Rickettsiales,分别占60.0%、6.3%和1.5%;EP1中相对丰度较大的有unidentified Cardiobacteriales、肠球菌科(Enterococcaceae)、unidentified Clostridiales和双歧杆菌科(Bifidobacteriaceae),分别占57.9%、9.0%、2.6%和1.0%;EP2中相对丰度较大的有unidentified Cardiobacteriales、unidentified Cyanobacteria、肠球菌科、乳杆菌科(Lactobacillaceae),分别占34.9%、5.3%、4.4%和2.3%(图6)。经加工后的老香黄,鲜果中原有的优势菌科肠杆菌科和unidentified Rickettsiales未检出,其优势菌科为unidentified Cardiobacteriales、肠球菌科等。此外,老香黄中出现了乳杆菌科和双歧杆菌科等有益菌。
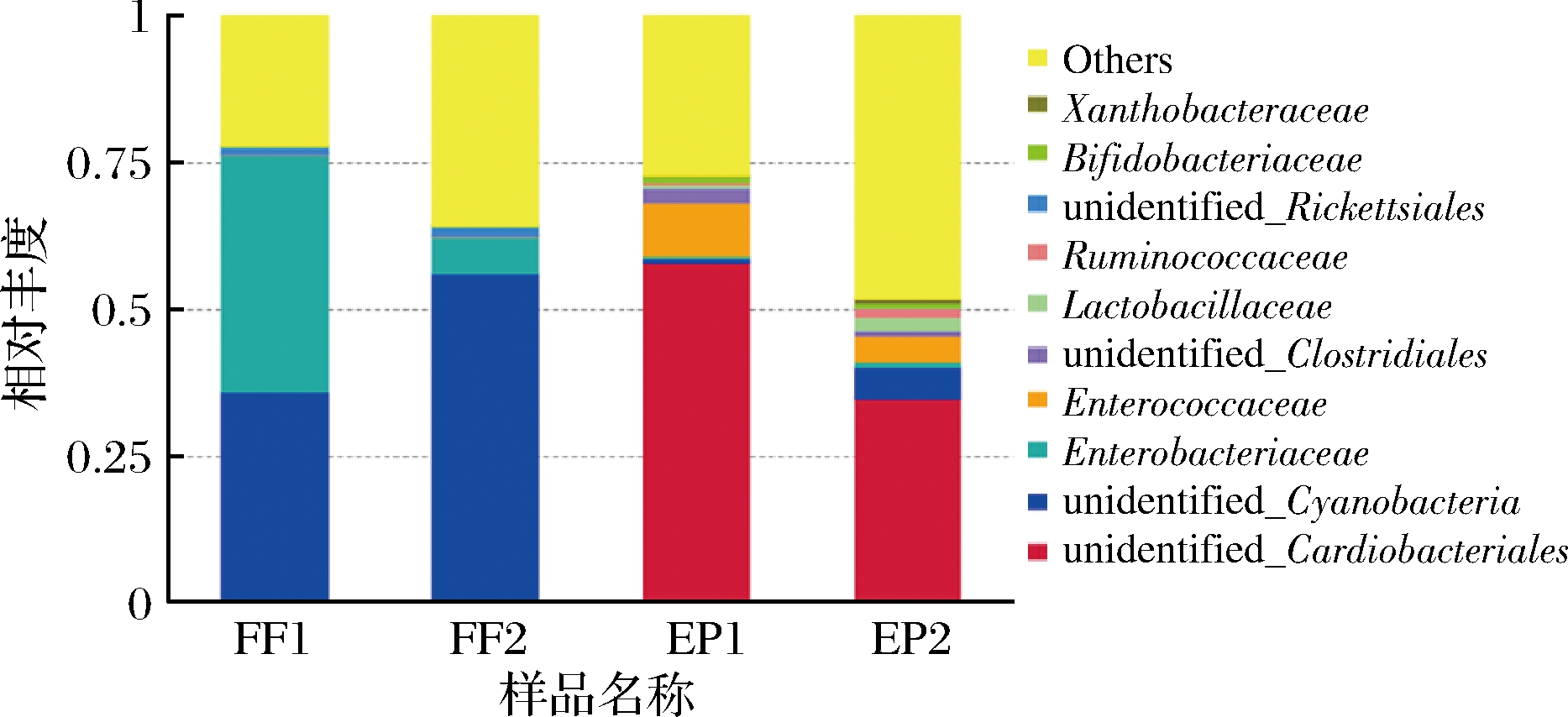
图6 科水平上的菌群相对丰度
Fig.6 Relative abundance of bacteria at family level
2.2.4.3 基于属水平的细菌菌群结构分析
属水平上,相对丰度前30的菌属如图7所示,FF1相对丰度较大的有unidentified Cyanobacteria、泛菌属(Pantoea)、肠杆菌属(Enterobacter)、克雷白氏杆菌属(Klebsiella)、unidentified Rickettsiales、沙雷氏菌属(Serratia)分别占35.9%、24.6%、6.1%、3.1%、1.2%和1.1%;FF2中有unidentified Cyanobacteria(56.0%)、泛菌属(4.4%)、肠杆菌属(1.2%)、unidentified Rickettsiales(1.5%)、芽孢杆菌属(0.3%);EP1中有Ignatzschineria(57.9%)、漫游球菌属(Vagococcus,6.4%)、肠球菌属(Enterococcus,2.6%)、Anaerosalibacter(2.0%)、双歧杆菌属(Bifidobacterium,1.0%)、乳杆菌属(Lactobacillus,0.4%)、芽孢杆菌属(0.2%);EP2中有Ignatzschineria(34.9%)、unidentified Cyanobacteria(5.3%)、漫游球菌属(4.2%)、乳杆菌属(2.2%)、双歧杆菌属(0.5%)、芽孢杆菌属(0.2%)。老香黄的加工中微生物在属水平的变化明显,优势菌从泛菌属、肠杆菌属变成Ignatzschineria、漫游球菌属;而且肠杆菌属未检出,其只在鲜果中出现。COLLINS等[22]于1989年首次对漫游球菌属进行研究分析,发现河流漫游球菌(Vagococcus fluvialis)的来源广泛,其形态特征与肠球菌及其相似。漫游球菌属包括不同的菌种如:河流漫游球菌、鲑鱼漫游球菌(Vagococcus salmoninarum)、水獭漫游球菌(Vagococcus lutrae)和嗜肉漫游球菌(Vagococcus carniphilus)等,其中既有致病菌株又有非致病菌株。SORROZA等[23]研究发现河流漫游球菌具有作为水产养殖中益生菌的潜力。而Ignatzschineria属细菌是一种致病菌[24]。高通量测序结果表明老香黄中的Ignatzschineria与漫游球菌属细菌处于优势地位,这2个菌属可能给老香黄的食用造成安全隐患,虽然纯培养中未发现,但在老香黄的加工过程中仍需要加强安全卫生的管控。此外,老香黄中检测出双歧杆菌属和乳杆菌属,双歧杆菌属是人体肠道菌群中的重要成分,是一类重要的益生菌,它可以调节并维持肠道内正常微生物的菌群平衡[25]。乳杆菌属广泛分布于含有碳水化合物的动植物发酵产品中,也常见于温血动物的口腔和肠道内,具有改善肠道功能的作用。而且双歧杆菌属及乳杆菌属亦有降血脂的作用[26-27]。老香黄具有的“扶正祛邪、改善机体内环境”的功效可能与这2种益生菌相关。

图7 属水平上的菌群相对丰度
Fig.7 Relative abundance of bacteria at genus level
2.2.5 主成分分析(principal component analysis,PCA)
主成分分析是一种基于欧式距离(euclidean distances)的应用方差分解,对多维数据进行降维,从而提取出数据中最主要的元素和结构的方法[28-29]。如果样本的菌群组成越相似,则它们在PCA图中的距离越接近。如图8所示,PC1与PC2的贡献率分别为71.97%和22.34%,说明这2个主成分是解释样品微生物菌群结构差异的主要因素。FF1与FF2很靠近,EP1与EP2彼此之间距离较大并且与FF1、FF2也分隔开,说明加工后的老香黄细菌的菌群结构与鲜果相差较大,而老香黄因加工批次和贮藏年限的不同会导致其中的细菌菌群结构也有所差异。
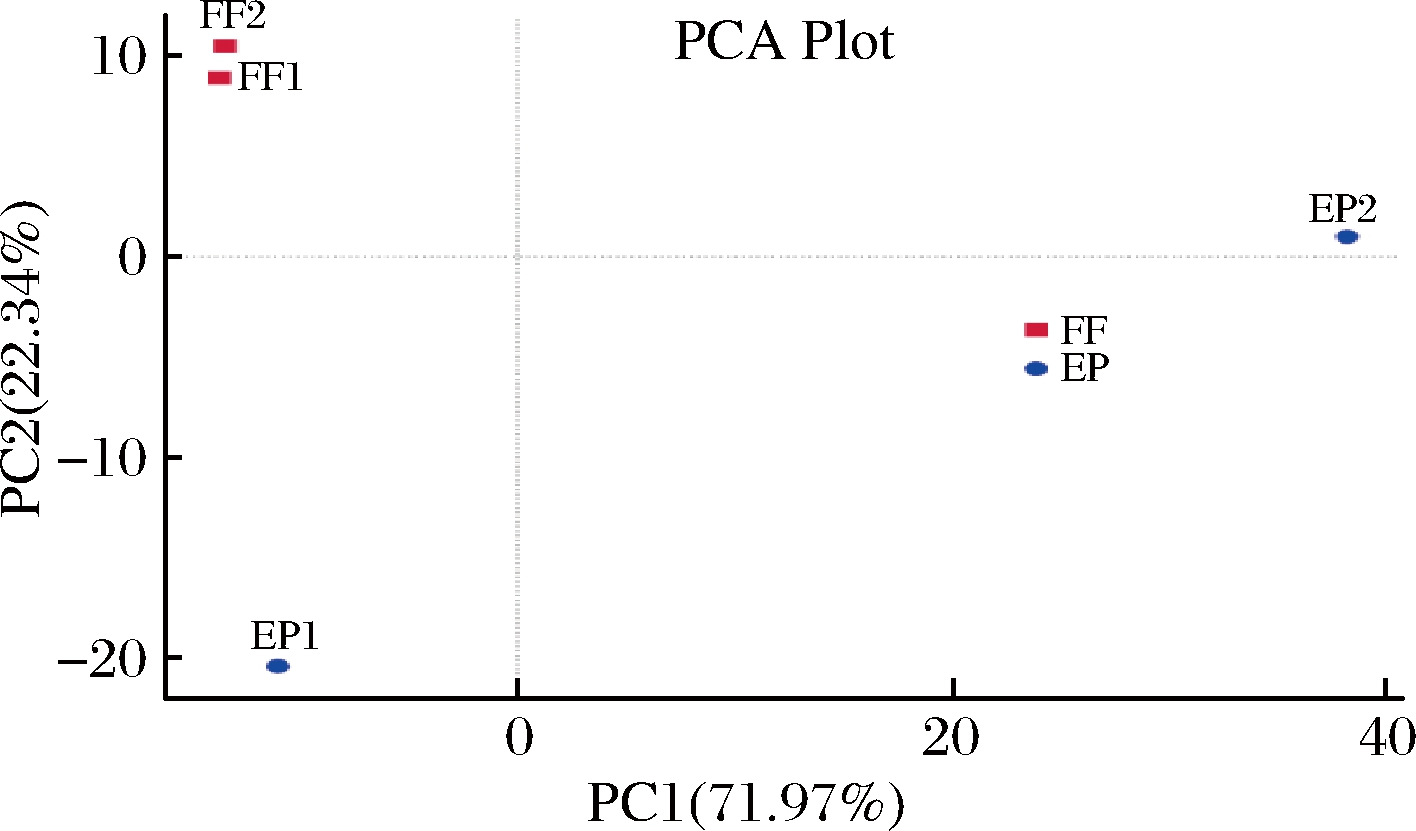
图8 菌群的主成分分析
Fig.8 Principal component analysis
3 结论
本研究采用纯培养方法和基于16S rRNA基因的高通量测序技术对佛手鲜果与佛手加工品老香黄中的微生物进行分析。根据高通量测序结果,佛手鲜果在经过了盐渍、蜜糖渍等一系列的加工工序制作成老香黄的过程中,优势菌群由泛菌属、肠杆菌属等变为Ignatzschineria、漫游球菌属、乳杆菌属和双歧杆菌属等。这表明老香黄中可能存在一定的致病菌,但本研究中通过纯培养方法并未从老香黄中分离出如高通量测序结果所示的大部分细菌菌属,可能是由于在加工成老香黄的过程中,大部分微生物已死亡。另外,高通量测序显示老香黄中还存在双歧杆菌属和乳杆菌属,这2个菌属为益生菌,具有改善肠道功能、维持肠道内正常菌群平衡等多种作用,但本研究中并未分离出活菌,后续还有待于增菌并进一步的分离和研究。结合纯培养和高通量测序结果,虽然以纯培养方法未从老香黄中分离出有害微生物,但是老香黄作为药食两用食品,其生产和食用的微生物安全依然需要人们的重视。
[1] 张思荻,杨海燕,曾俊,等.佛手的研究进展[J].中华中医药杂志,2018,33(8):3 510-3 514.
[2] HIRAISHI A, KISHIMOTO N, KOSAKO Y, et al. Phylogenetic position of the menaquinone-containing acidophilic chemo-organotroph Acidobacterium capsulatum[J]. FEMS Microbiology Letters, 1995,132(1-2):91-94.
[3] 杨继,陆智明,陆国权.金佛手果叶多糖提取工艺的优化[J].食品与发酵工业,2011,37(11):239-242.
[4] 夏雨,王雁胜,张延杰,等.佛手果营养与活性成分测定及降血脂功能研究[J].食品科技, 2016,41(2):70-73.
[5] 杨慧,周爱梅,林敏浩,等.佛手挥发精油提取及其药理研究进展[J].食品安全质量检测学报, 2013,4(5):1 347-1 352.
[6] 赵秀玲.佛手生理活性成分的研究进展[J].食品工业科技,2012,33(21):393-399.
[7] 郑玉忠,郭守军,杨永利,等.药食凉果老香黄制作工艺的研究[J].农产品加工(学刊), 2014(1):44-45.
[8] 张振霞,赖宣,杨启财,等.广佛手与金华佛手理化性质的比较[J].轻工科技, 2017,33(8):22-23.
[9] PENG Mu, ZI Xiaoxue, WANG Qiuyu. Bacterial community diversity of oil-contaminated soils assessed by high throughput sequencing of 16S rRNA genes[J]. International Journal of Environmental Research and Public Health, 2015,12(10):12 002-12 015.
[10] NG T W, HUANG G, WONG P K. Investigation of drinking water bacterial community through high-throughput sequencing[J]. Journal of Environmental Sciences, 2015,37(11):154-156.
[11] XU Yunhe, YE Qing, ZHANG Huajiang, et al. Naturally fermented acid slurry of soy whey: high-throughput sequencing-based characterization of microbial flora and mechanism of tofu coagulation[J]. Frontiers in Microbiology, 2019,10:1 088.
[12] GUO Wei, LI Ying, WANG Lizhi, et al. Evaluation of composition and individual variability of rumen microbiota in yaks by 16S rRNA high-throughput sequencing technology[J]. Anaerobe, 2015,34:74-79.
[13] 阮征,魏力,张延杰,等,高通量测序结合传统培养分析常温油沙冰皮月饼的菌群多样性[J].食品与发酵工业, 2019,45(6):203-208.
[14] 蓝蔚青,谢晶.冷藏带鱼贮藏期间主要微生物动态变化的PCR-DGGE分析[J].食品工业科技, 2012,33(17):118-122.
[15] ZHANG Lihui, WANG Shaojin. Bacterial community diversity on in-shell walnut surfaces from six representative provinces in China[J]. Scientific Reports, 2017,7(1):10 054-10 059.
[16] WARD T, LARSON J, MEULEMANS J, et al. BugBase predicts organism level microbiome phenotypes[J]. BioRxiv, 2017:133 462.
[17] ROGNES T, FLOURI T, NICHOLS B, et al. VSEARCH: a versatile open source tool for metagenomics[J]. Peerj, 2016,4:e2 584.
[18] LUNDBERG D S, YOURSTONE S, MIECZKOWSKI P, et al. Practical innovations for high-throughput amplicon sequencing[J]. Nature Methods, 2013,10(10):999-1 002.
[19] 王伟,冷凯良,刘均忠,等.微生物扩增子高通量测序技术在水产品加工与贮藏中的应用[J].食品与发酵工业,2017,43(10):263-268.
[20] EL KAOUTARI A, ARMOUGOM F, GORDON J I, et al. The abundance and variety of carbohydrate-active enzymes in the human gut microbiota[J]. Nature Reviews Microbiology, 2013,11(7):497-504.
[21] KISHIMOTO N, KOSAKO Y, TANO T. Acidobacterium capsulatum gen. nov., sp. nov.: an acidophilic chemoorganotrophic bacterium containing menaquinone from acidic mineral environment[J]. Current Microbiology, 1991,22(1):1-7.
[22] COLLINS M D, ASH C, FARROW J A, et al. 16S ribosomal ribonucleic acid sequence analyses of lactococci and related taxa. description of Vagococcus fluvialis gen. nov., sp. nov.[J]. The Journal of Applied Bacteriology, 1989,67(4):453-460.
[23] SORROZA L, PADILLA D, ACOSTA F, et al. Characterization of the probiotic strain Vagococcus fluvialis in the protection of European sea bass (Dicentrarchus labrax) against vibriosis by Vibrio anguillarum[J]. Veterinary Microbiology, 2012,155(2-4):369-373.
[24] BARKER H S, SNYDER J W, HICKS A B, et al. First case reports of Ignatzschineria (Schineria) indica Associated with Myiasis[J]. Journal of Clinical Microbiology, 2014,52(12):4 432-4 434.
[25] HADADJI M, BENAMA R, SAIDI N, et al. Identification of cultivable Bifidobacterium species isolated from breast-fed infants feces in West-Algeria[J]. African Journal of Biotechnology, 2005,4(5):422-430.
[26] RERKSUPPAPHOL S, RERKSUPPAPHOL L. A randomized double-blind controlled trial of Lactobacillus acidophilus plus Bifidobacterium bifidum versus placebo in patients with hypercholesterolemia[J]. Journal of Clinical and Diagnostic Research: JCDR, 2015,9(3):C1.
[27] RAJKUMAR H, KUMAR M, DAS N, et al. Effect of probiotic Lactobacillus salivarius UBL S22 and prebiotic fructo-oligosaccharide on serum lipids, inflammatory markers, insulin sensitivity, and gut bacteria in healthy young volunteers: a randomized controlled single-blind pilot study[J]. Journal of Cardiovascular Pharmacology and Therapeutics, 2015,20(3):289-298.
[28] AVERSHINA E, FRISLI T, RUDI K. De novo semi-alignment of 16S rRNA gene sequences for deep phylogenetic characterization of next generation sequencing data[J]. Microbes and Environments, 2013,28(2):211-216.
[29] JOLLIFFE I T. Principal Component Analysis[M]. John Wiley & Sons, Inc, 1986.