四川腊肉是以屠宰后的鲜猪肉为主要原料,用食盐等腌制后,经烘烤、烟熏而成的传统发酵肉制品,因其独有的风味、鲜明的色泽和浓郁的香气,受到广大人民的喜爱[1-3]。在制作与贮藏过程中,微生物作用于腊肉中的蛋白质和脂肪等营养物质,形成了腊肉良好的色泽和风味[4-5]。由于腊肉加工环境相对开放,多种微生物参与了腊肉的成熟过程。
目前关于腊肉中微生物的报道较多。全拓等[6]研究川味腊肉货架期间的主要微生物,得出优势菌是葡萄球菌和微球菌,其次是乳酸菌;刘有晴等[7]采用可培养方法,对农家自制四川腊肉中乳酸菌进行分离筛选,获得1株植物乳杆菌;于华等[8]以四川腊肉为研究对象,采用平皿生化法对其高产脂肪酶的霉菌进行分离筛选,获得1株团青霉(Penicillium commune);田园园等[9]从四川省眉山等5市的传统腌腊肉样品中筛选出8株乳酸菌;陈竞适等[10]以湘西花垣县和凤凰县农户陈年腊肉为原料,研究得出酵母菌、霉菌和微球菌是其主要微生物群落;冯秀娟等[11]研究了湖南怀化地区传统腊肉,得出优势菌为乳酸菌和葡萄球菌;李福荣等[12]对信阳传统发酵腊肉中的细菌进行研究,得出优势菌为葡萄球菌和微球菌;毕旺来等[13]对湖北省神农架林区的腊肉进行研究,得出主要微生物为葡萄球菌。不同地区腊肉的微生物多样性存在差异,以上研究均以传统培养法为基础的微生物学手段进行分析,该方法的缺陷是不能全面、准确地反映样品的微生物信息[14];新兴的Illumina高通量测序技术具有操作简单、成本较低、结果可信度高等优势[15]。因此,通过Illumina高通量测序技术能够全面而准确地了解研究对象中微生物种类组成和结构。
本研究运用Illumina高通量测序技术对采自四川省达州市的腊肉样品中微生物多样性进行分析,以期全面地解析腊肉中的微生物组成及群落结构,为四川传统腊肉的加工及品质控制提供参考依据。
1 材料与方法
1.1 材料与试剂
E.Z.N.A.Soil DNA kit D5626-01试剂盒,美国OMEGA公司;Qubit2.0 DNA检测试剂盒,美国Invitrogen公司;Q5高保真DNA聚合酶,美国New England Biolabs公司;凝胶回收试剂盒,美国AXYGEN公司;TruSeq Nano DNA LT Library Prep Kit,美国Illumina公司。
1.2 仪器与设备
Pico-21型台式离心机,Thermo Fisher;DYY-6C型电泳仪,北京市六一仪器厂;凝胶成像系统,美国UVP公司;Q32866型Qubit 2.0分光光度计,Invitrogen公司;T100TM Thermal Cyeler型PCR仪,BIO-RAD公司;MiSeq System SY-410-1003高通量测序仪,美国Illumina公司。
1.3 实验方法
1.3.1 样品的采集
2019年3月于四川省达州地区采集6份腊肉样品(编号分别为M1、M2、M3、M4、M5、M6),将所有样品装入自封袋,置于冷藏箱中运至实验室以备试验。
1.3.2 腊肉微生物总DNA的提取
腊肉样品中微生物总DNA提取选用E.Z.N.A.Soil DNA Kit D5625-01试剂盒,按照使用说明从样品中提取DNA;然后使用 Qubit 2.0 分光光度计定量提取的 DNA,并通过 0.8%琼脂糖凝胶电泳确定 DNA 提取的完整性。
1.3.3 PCR扩增及测序
以稀释后的基因组总 DNA 为模板,靶向扩增 16S rRNA的 V3-V4区,扩增引物分别为338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTC TAAT-3′)。选用真菌特异性引物对ITS区域进行扩增,扩增引物分别为ITS5F(GGAAGTAAAAGTCGTAACAAGG)和ITS1R(GCTGCGTTCTTCATCGATGC)。PCR条件如下:预变性为95 ℃、5 min,然后95 ℃、30 s,56 ℃、30 s,72 ℃、30 s,共25个循环,72 ℃退火10 min,最后保存在4 ℃条件下。选用2%琼脂糖凝胶电泳对PCR产物进行检测,然后用试剂盒回收并进行质量检测并建库。最后由上海派森诺生物科技股份有限公司在MiSeq测序平台进行双端测序。
1.3.4 高通量测序数据处理
MiSeq测序得到的数据选用Mothur(V.1.31.2)和QIIME(V.1.7.0)软件进行处理及分析[16-17]。首先选用滑动窗口法对FASTQ格式的双端序列逐一进行质量筛选,然后利用FLASH软件(v1.2.7)对质量初筛的双端序列进行配对连接,最后将连接后的序列识别分配对应样本,从而获得有效序列。在测序过程中会产生一些错误或疑问序列,因此选用QIIME软件(v1.8.0)[18]识别疑问序列。随后调用USEARCH(v5.2.236)检查并剔除嵌合体序列[19-22]。最后,调用UCLUST算法进行序列聚类,以97%的序列相似度进行归并和(operational taxonomic units,OTU)[23]划分。
采用Mothur(V.1.31.2)软件进行群落结构的Alpha多样性分析。
1.4 数据分析
采用Excel 2010和Origin 2018软件进行处理分析并作图。
2 结果分析
2.1 四川传统腊肉样品中Alpha多样性分析
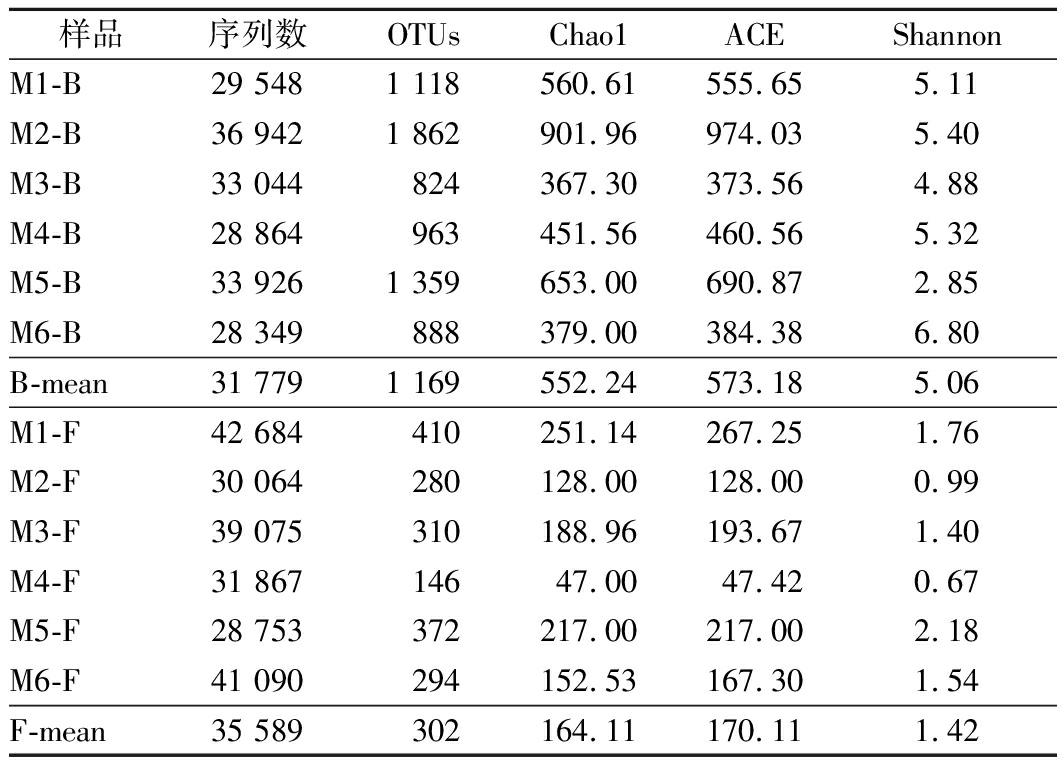
腊肉样品中细菌和真菌的测序结果及Alpha多样性指数如表1所示。通过细菌16S rRNA 的V3-V4区进行测序,6份样品共产生高质量序列190 673条,将所有序列按97%的相似度进行OTU聚类,得到7014个OTU;通过真菌ITS区测序,6份样品共产生高质量序列213 533条,聚类后得到1 812个OTU。
表1 测序结果及Alpha多样性指数表
Table 1 Sequencing results and Alpha diversity index
样品序列数OTUsChao1ACEShannonM1-B29 5481 118560.61555.655.11M2-B36 9421 862901.96974.035.40M3-B33 044824367.30373.564.88M4-B28 864963451.56460.565.32M5-B33 9261 359653.00690.872.85M6-B28 349888379.00384.386.80B-mean31 7791 169552.24573.185.06M1-F42 684410251.14267.251.76M2-F30 064280128.00128.000.99M3-F39 075310188.96193.671.40M4-F31 86714647.0047.420.67M5-F28 753372217.00217.002.18M6-F41 090294152.53167.301.54F-mean35 589302164.11170.111.42
注:表中M1-B到M6-B为样品细菌的Alpha多样性指数,B-mean为细菌样品的Alpha多样性指数的平均值;M1-F到M6-F为细菌样品的Alpha多样性指数,F-mean为样品真菌的Alpha多样性指数的平均值
Shannon指数是衡量物种多样性的指数,Shannon指数值越高,物种多样性越丰富,反之,物种多样性越少。6份样品中细菌的Shannon指数在2.85~6.80之间,最高的为样品M6-B(6.80),表明M6-B中细菌OTU的多样性较高;Shannon指数最低的为样品M5-B(2.85),反映了M5-B中细菌多样性较低;6份样品中真菌的Shannon指数在0.67~2.18之间,最高的为样品M5-F(2.18),表明M5-F真菌OTU的多样性较高;Shannon指数最低的为样品M4-F(0.67),反映了M4-F的真菌多样性较低。
Chao1指数和ACE指数主要体现稀有群落的丰富度。Chao1指数或ACE指数越大,表明群落的丰富度越高。6份样品中细菌的Chao1指数在367.30~901.96之间,ACE指数在373.56~974.03之间;样品M2-B的Chao1指数和ACE指数最高,分别为901.96和974.03,并且OUT数最多(1 862个)。6份样品中真菌的Chao1指数在47.00~2 510.14之间,ACE指数在47.42~267.25之间;样品M1-F的Chao1指数和ACE指数最高,分别为251.14和267.25,并且OUT数最多(410个)。
细菌样品中OUT、Chao1、ACE、Shannon指数的平均值分别为1 169、552.24、573.18、5.06;真菌样品中OUT、Chao1、ACE、Shannon指数的平均值分别为302、164.11、170.11、1.42。细菌的数值均高于真菌,表明细菌的Alpha多样性指数高于真菌。
稀疏曲线是评定每个样品在当前的测序深度下是否能反映该样品中所包含的微生物群落多样性的标准[24-26]。6份腊肉样品中细菌和真菌的香农指数稀疏曲线如图1所示。由图1可以看出,6份腊肉样品的香农指数随着测序量的增大呈现上升趋势,说明在此测序水平下,样品中还有较多的物种没有被检测到;当测序量较高时,香浓曲线逐渐与X轴接近平行,说明在此测序水平下,样品中细菌和真菌的群落多样性已得到充分的展现。

a-细菌;b-真菌
图1 细菌香浓指数稀疏曲线和真菌香浓指数
稀疏曲线
Fig.1 Sparse analysis diagram of bacterial index and
sparse analysis of fungi Shannon index
2.2 四川传统腊肉样品中微生物群落在门水平的比较
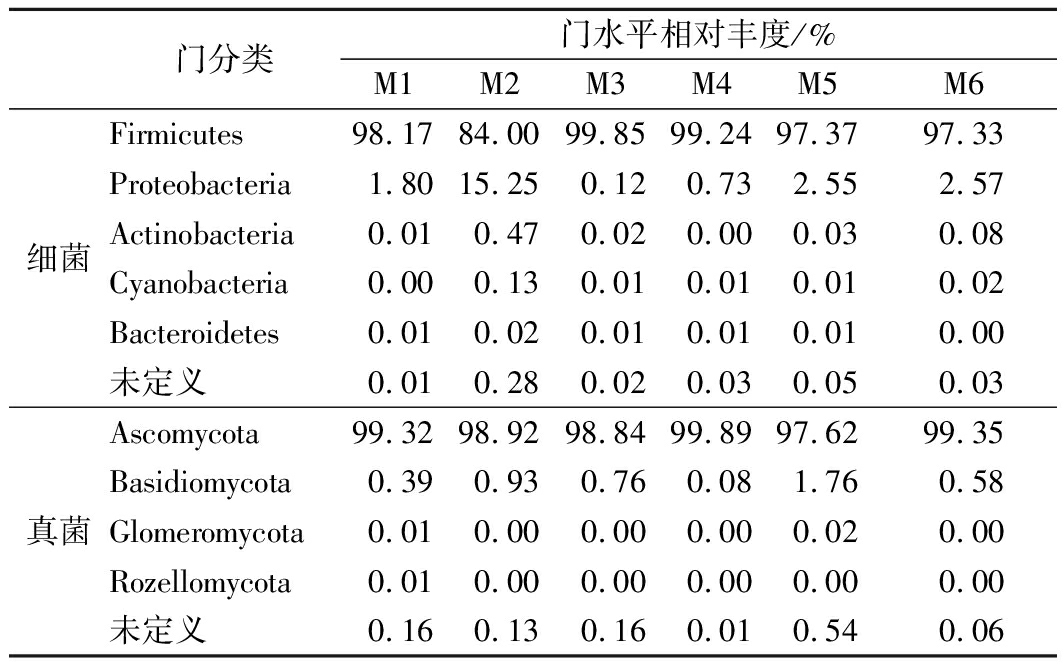
表2为6份腊肉样品从门分类水平进行鉴定的结果。由表2可知,在腊肉样品中检测出5个细菌门,分别是厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、蓝藻门(Cyanobacteria)、拟杆菌门(Bacteroidetes)。其中,所有样品中细菌的第一优势门均为厚壁菌门(相对丰度大于1%),其丰度值在84.00%~99.86%之间,平均相对丰度为95.99%;变形菌门为样品M1、M2、M5、M6的第二优势门;放线菌门、蓝藻门和拟杆菌门平均丰度值均小于1%,为样品中的非优势门。厚壁菌门和变形菌门也是风干肉制品[27]、湖北省恩施腊肉[28]、传统自然发酵酸肉[29]、4 ℃贮藏的冰鲜鸽肉[30]、发酵鳐鱼[31]的优势门,这与本研究结果一致。
表2 各样品门水平菌群分布相对丰度
Table 2 The relative abundance of microbial flora
distribution at phylum level in each sample
门分类门水平相对丰度/%M1M2M3M4M5M6细菌Firmicutes98.1784.0099.8599.2497.3797.33Proteobacteria1.8015.250.120.732.552.57Actinobacteria0.010.470.020.000.030.08Cyanobacteria0.000.130.010.010.010.02Bacteroidetes0.010.020.010.010.010.00未定义0.010.280.020.030.050.03真菌Ascomycota99.3298.9298.8499.8997.6299.35Basidiomycota0.390.930.760.081.760.58Glomeromycota0.010.000.000.000.020.00Rozellomycota0.010.000.000.000.000.00未定义0.160.130.160.010.540.06
在6份腊肉样品中检测出4个真菌门,分别是为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、球囊菌门(Glomeromycota)、罗兹菌门(Rozellomycota);其中子囊菌门为共有优势门(相对丰度大于1%),平均相对丰度为98.99%;担子菌门为样品M5的第二优势门,球囊菌门和罗兹菌门在样品中的丰度都很低,为非优势门。有研究报道,子囊菌门也是发酵酸鱼[32]、传统发酵乳制品[33]的优势门,这与本研究结果一致。
2.3 四川传统腊肉样品的微生物群落在属水平的比较
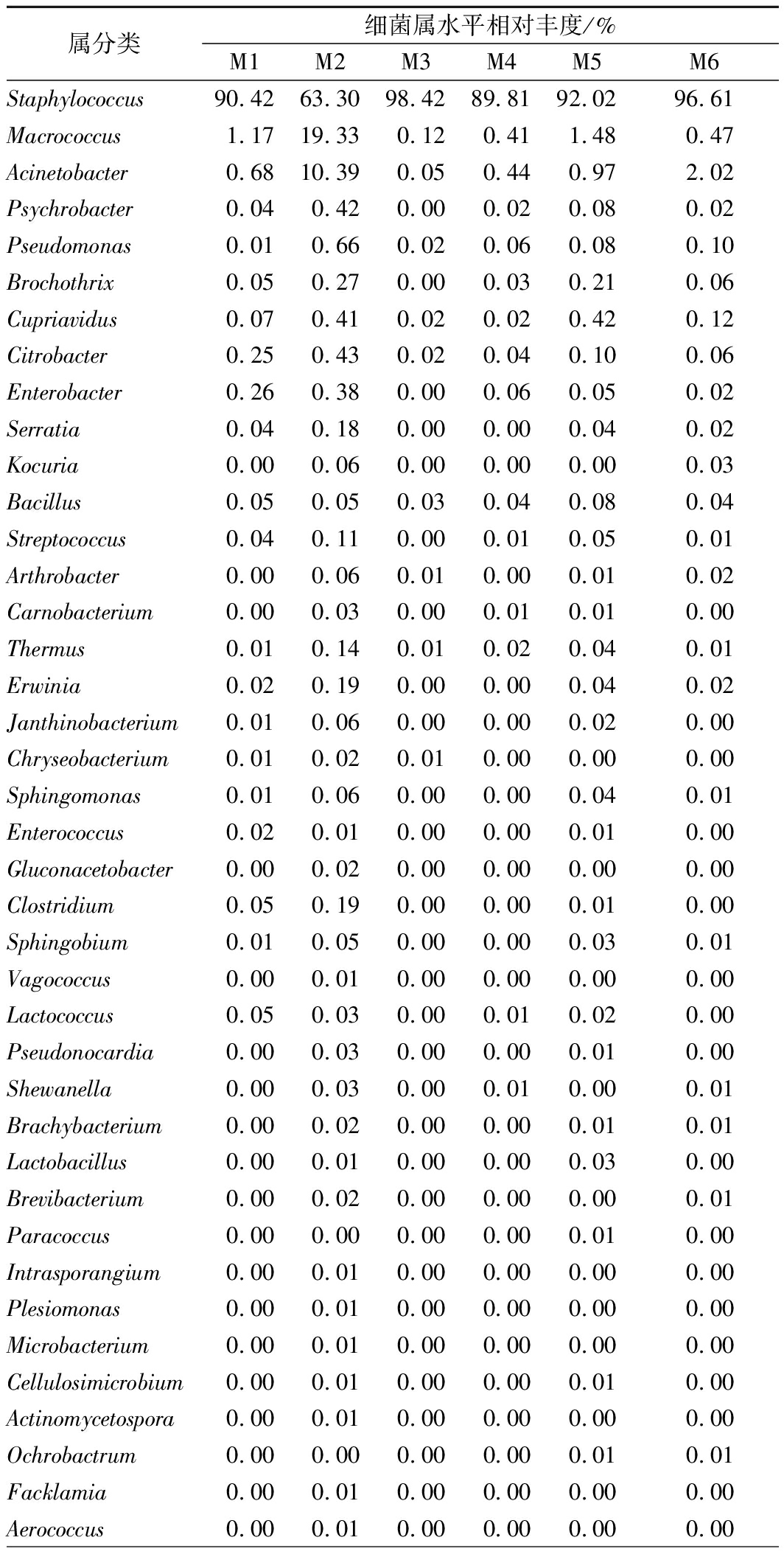
表3为腊肉样品中细菌属水平的分类鉴定结果。由表3可以看出,葡萄球菌属(Staphylococcus)丰度值在 63.30%~98.42%的范围内,平均丰度为88.43%,是所有样品中的第一优势属;巨球菌属(Macrococcus)为M1、M2、M5腊肉样品中的第二优势属;不动杆菌属(Acinetobacter)是M6样品的第二优势属,也是M2样品的第三优势属;嗜冷杆菌属(Psychrobacter)、假单胞菌属(Pseudomonas)、环丝菌属(Brochothrix)、贪铜菌属(Cupriavidus)、柠檬酸杆菌属(Citrobacter)和肠杆菌属(Enterobacter)平均丰度值在0.1%~0.2%之间,为样品中的非优势属;另外,样品中还包含许多在属水平上未鉴定的属(Unclassified),其丰度值在 0.34%~9.03%的范围内。在本研究中葡萄球菌属为四川传统腊肉的绝对优势属,秦丹[34]以四川腊肉和香肠为研究对象,得出葡萄球菌是两者的共同优势菌;陈美春等[1]、全拓等[6]运用纯培养法对四川腊肉的细菌多样性进行解析,发现葡萄球菌是腊肉的优势菌;而LEE等[35]在发酵鱼酱的研究中也发现葡萄球菌是其优势菌,与本研究结果相同。陈美春等[1]和全拓等[6]的研究还表明乳酸菌是四川腊肉的优势菌,而在本研究中检测出的乳球菌属和乳杆菌属丰度较低,这可能与腊肉样品的来源有关;与上述研究不同,本研究中发现巨球菌属在样品M1、M2、M5中的相对丰度大于1%,为优势属。
表3 各样品属水平细菌的相对丰度
Table 3 The relative abundance of bacterial flora at
genus level in each sample
属分类细菌属水平相对丰度/%M1M2M3M4M5M6Staphylococcus90.4263.3098.4289.8192.0296.61Macrococcus1.1719.330.120.411.480.47Acinetobacter0.6810.390.050.440.972.02Psychrobacter0.040.420.000.020.080.02Pseudomonas0.010.660.020.060.080.10Brochothrix0.050.270.000.030.210.06Cupriavidus 0.070.410.020.020.420.12Citrobacter 0.250.430.020.040.100.06Enterobacter0.260.380.000.060.050.02Serratia0.040.180.000.000.040.02Kocuria0.000.060.000.000.000.03Bacillus0.050.050.030.040.080.04Streptococcus 0.040.110.000.010.050.01Arthrobacter0.000.060.010.000.010.02Carnobacterium0.000.030.000.010.010.00Thermus 0.010.140.010.020.040.01Erwinia 0.020.190.000.000.040.02Janthinobacterium0.010.060.000.000.020.00Chryseobacterium 0.010.020.010.000.000.00Sphingomonas 0.010.060.000.000.040.01Enterococcus0.020.010.000.000.010.00Gluconacetobacter 0.000.020.000.000.000.00Clostridium 0.050.190.000.000.010.00Sphingobium 0.010.050.000.000.030.01Vagococcus 0.000.010.000.000.000.00Lactococcus 0.050.030.000.010.020.00Pseudonocardia 0.000.030.000.000.010.00Shewanella 0.000.030.000.010.000.01Brachybacterium 0.000.020.000.000.010.01Lactobacillus 0.000.010.000.000.030.00Brevibacterium 0.000.020.000.000.000.01Paracoccus 0.000.000.000.000.010.00Intrasporangium 0.000.010.000.000.000.00Plesiomonas 0.000.010.000.000.000.00Microbacterium 0.000.010.000.000.000.00Cellulosimicrobium 0.000.010.000.000.010.00Actinomycetospora 0.000.010.000.000.000.00Ochrobactrum 0.000.000.000.000.010.01Facklamia 0.000.010.000.000.000.00Aerococcus 0.000.010.000.000.000.00
续表3
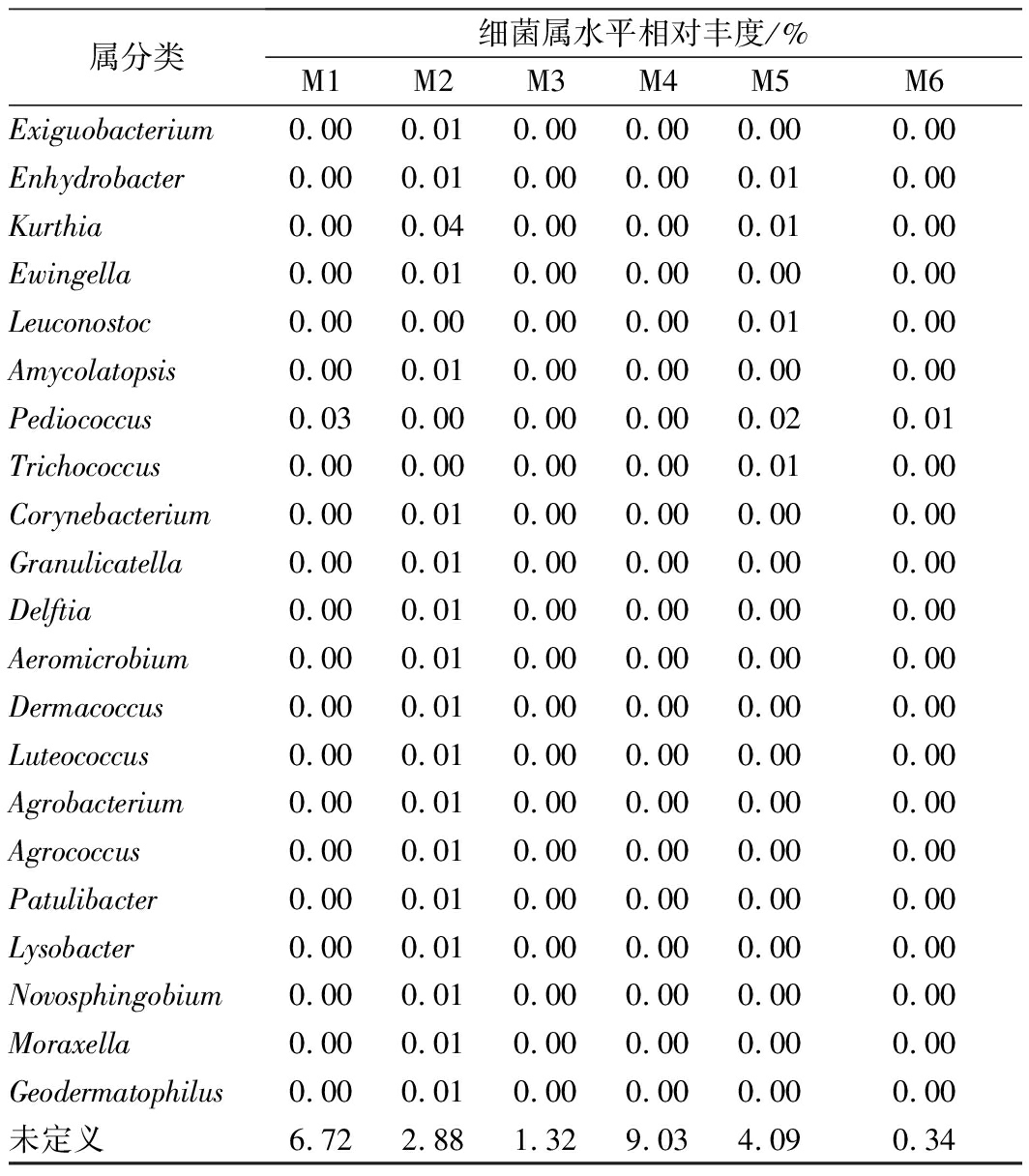
属分类细菌属水平相对丰度/%M1M2M3M4M5M6Exiguobacterium 0.000.010.000.000.000.00Enhydrobacter 0.000.010.000.000.010.00Kurthia 0.000.040.000.000.010.00Ewingella 0.000.010.000.000.000.00Leuconostoc 0.000.000.000.000.010.00Amycolatopsis 0.000.010.000.000.000.00Pediococcus 0.030.000.000.000.020.01Trichococcus 0.000.000.000.000.010.00Corynebacterium 0.000.010.000.000.000.00Granulicatella 0.000.010.000.000.000.00Delftia 0.000.010.000.000.000.00Aeromicrobium 0.000.010.000.000.000.00Dermacoccus 0.000.010.000.000.000.00Luteococcus 0.000.010.000.000.000.00Agrobacterium 0.000.010.000.000.000.00Agrococcus 0.000.010.000.000.000.00Patulibacter 0.000.010.000.000.000.00Lysobacter 0.000.010.000.000.000.00Novosphingobium 0.000.010.000.000.000.00Moraxella 0.000.010.000.000.000.00Geodermatophilus 0.000.010.000.000.000.00未定义6.722.881.329.034.090.34
葡萄球菌可以抑制脂肪氧化,能分解肉制品中乳酸代谢产生的过氧化物,有利于形成风味,改善色泽延缓酸败以及提升产品质安全性[36-37]。因此四川传统腊肉中葡萄球菌的含量可能是颜色和风味形成的主要因素[12]。
表4为腊肉样品中真菌属水平的分类鉴定结果。由表4可以看出,曲霉属 (Aspergillus)丰度值在77.72%~98.42%之间,平均丰度值为88.46%,为腊肉样品的第一优势属;德巴利酵母属(Debaryomyces)为样品M1、M3、M4、M5、M6的第二优势属;假丝酵母属(Candida)为样品M5的第三优势属,但假丝酵母属在所有样品中的平均丰度较低;节菌属(Wallemia)、青霉菌属(Penicillium)、马拉色氏霉菌属(Malassezia)和亚隔孢壳属(Didymella)平均丰度值在0.1%~0.5%之间,为样品的非优势属;另外,样品中还包含许多未鉴定的属(Unclassified),但丰度较低,其丰度值在 0.04%~1.90%的范围内。本研究中发现曲霉属是四川传统腊肉的优势属,谢康庄等[38]采用纯培养法发现曲霉菌是湘西腊肉中的优势菌,与本文研究结果相一致。
霉菌和酵母菌为腊肉中的优势菌可能是因为家庭自制过程中的缺陷和贮藏过程中粗放的环境有关,另外与腊肉的水分含量和湿润气候也有关系,导致霉菌大量增长[10]。霉菌和酵母菌在腊肉形成独特的风味和提升安全性方面发挥了重要作用。霉菌属于好氧菌,往往存在于腊肉表面,能够抑制有害菌生长并且使腊肉具有特殊的香气,添加适量霉菌,还能够延长肉制品的保质期,有利于产品的储存。酵母菌属于兼性菌,在发酵时可吸收掉残存氧气从而能够抑制腊肉氧化酸败,还能阻止有害微生物的滋生,另外对腊肉风味的形成有促进作用[39-41]。
表4 各样品属水平真菌的相对丰度
Table 4 The relative abundance of fungi flora at genus
level in each sample

属分类真菌属水平相对丰度/%M1M2M3M4M5M6Aspergillus 80.8895.9690.0698.4277.7287.74Debaryomyces 16.660.987.401.4014.9010.37Candida 0.370.330.390.021.350.40Wallemia 0.230.580.460.060.650.42Penicillium 0.140.870.290.010.280.41Malassezia 0.080.190.220.020.570.10Didymella 0.020.110.120.000.260.09Trichoderma 0.070.030.020.000.340.02Plectosphaerella0.060.040.090.010.100.03Acremonium 0.160.050.020.000.320.02Guehomyces 0.020.060.010.000.140.04Arachnomyces0.050.010.000.000.040.01Microascus 0.040.000.000.000.140.00Phialemonium0.000.000.000.000.130.00Cosmospora 0.070.010.010.010.130.01Sepedonium 0.070.000.000.000.050.01Talaromyces 0.000.010.000.000.000.00Cystofilobasidium 0.000.000.000.000.010.00Cutaneotrichosporon 0.010.020.010.000.030.00Phaeoacremonium0.020.000.010.010.000.01Cladosporium 0.010.020.010.010.020.01Sarocladium 0.010.050.040.000.040.00Pyrenochaeta 0.020.000.000.000.000.00Neodevriesia0.000.010.000.000.020.00Yarrowia 0.010.000.000.000.010.00Leucosporidium 0.000.000.000.000.050.00Cystobasidium 0.030.010.000.000.090.00Nigrospora 0.010.000.010.000.020.00Microidium0.000.000.000.000.010.00Xerochrysium0.030.000.000.000.120.00Stachybotrys 0.000.010.020.000.020.01Paraglomus 0.010.000.000.000.020.00Colletotrichum 0.000.010.000.000.000.00Alternaria 0.000.010.000.000.000.00Simplicillium 0.000.000.000.000.060.00Vishniacozyma0.000.020.000.000.000.00Trichosporon 0.000.020.010.000.000.00Zasmidium0.010.010.000.000.000.00Sphaerulina 0.010.000.000.000.000.01Melanospora 0.010.000.000.000.000.00
续表4
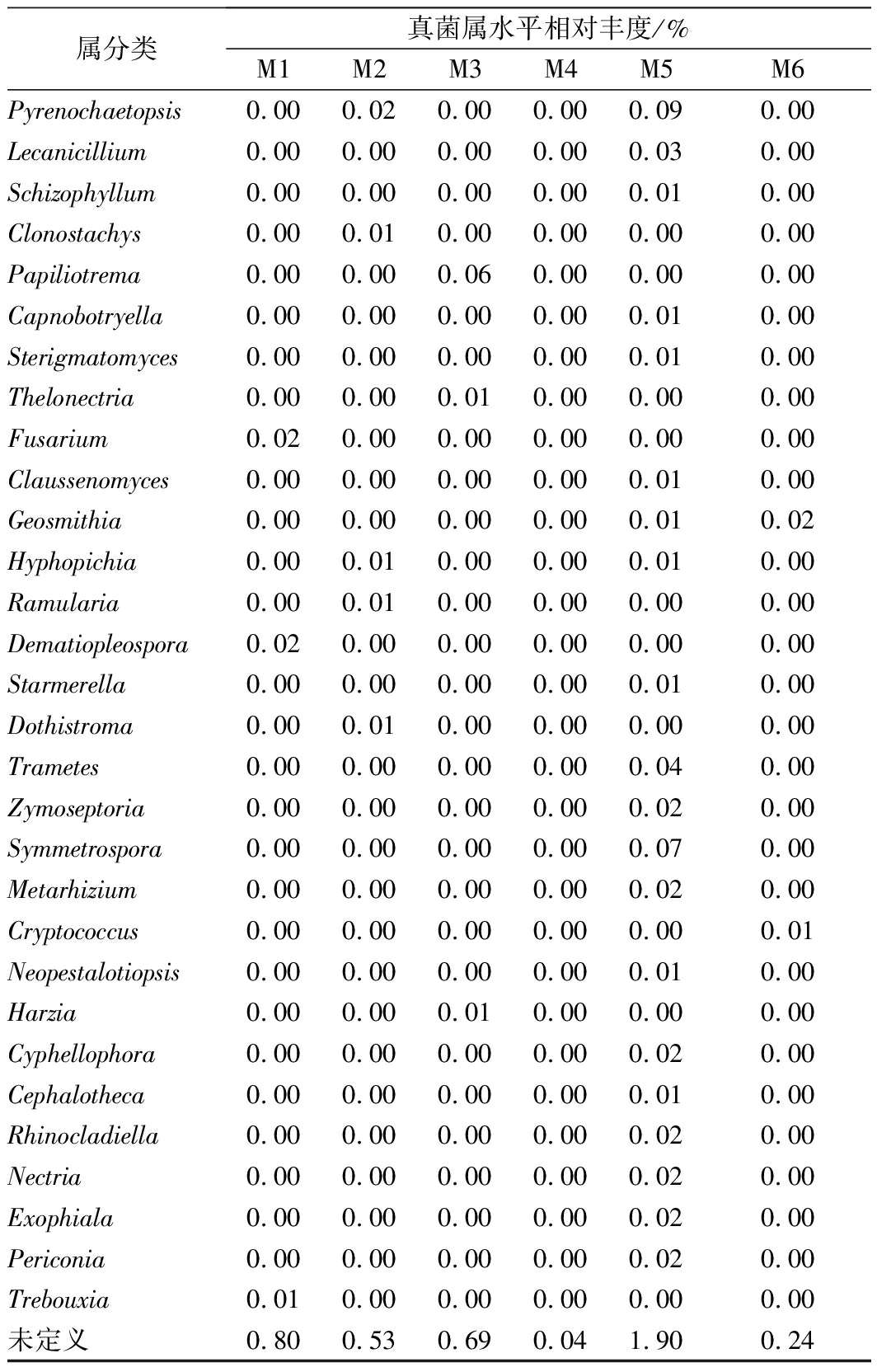
属分类真菌属水平相对丰度/%M1M2M3M4M5M6Pyrenochaetopsis0.000.020.000.000.090.00Lecanicillium 0.000.000.000.000.030.00Schizophyllum 0.000.000.000.000.010.00Clonostachys 0.000.010.000.000.000.00Papiliotrema0.000.000.060.000.000.00Capnobotryella 0.000.000.000.000.010.00Sterigmatomyces 0.000.000.000.000.010.00Thelonectria0.000.000.010.000.000.00Fusarium 0.020.000.000.000.000.00Claussenomyces 0.000.000.000.000.010.00Geosmithia0.000.000.000.000.010.02Hyphopichia 0.000.010.000.000.010.00Ramularia 0.000.010.000.000.000.00Dematiopleospora 0.020.000.000.000.000.00Starmerella 0.000.000.000.000.010.00Dothistroma 0.000.010.000.000.000.00Trametes 0.000.000.000.000.040.00Zymoseptoria 0.000.000.000.000.020.00Symmetrospora0.000.000.000.000.070.00Metarhizium 0.000.000.000.000.020.00Cryptococcus 0.000.000.000.000.000.01Neopestalotiopsis0.000.000.000.000.010.00Harzia 0.000.000.010.000.000.00Cyphellophora 0.000.000.000.000.020.00Cephalotheca 0.000.000.000.000.010.00Rhinocladiella 0.000.000.000.000.020.00Nectria 0.000.000.000.000.020.00Exophiala 0.000.000.000.000.020.00Periconia 0.000.000.000.000.020.00Trebouxia 0.010.000.000.000.000.00未定义0.800.530.690.041.900.24
2.4 四川传统腊肉样品中微生物的主成分分析
在图2中,样品间距远近表示了其差异性大小,2点之间的距离越近,表明2个样本之间的微生物群落结构相似度越高,差异越小。由图2-a可知,样品M1与其他样品差异较大,PC2很好地将M1和其他样品区分开来;由图2-b可知,样品M2、M4与其他样品差异较大,PC2 可以将其与其他样品完全区分开。传统腊肉的加工原料、加工工艺、加工环境都会直接或者间接地影响腊肉中的微生物,使得最终产品中的微生物有一定差异[6]。
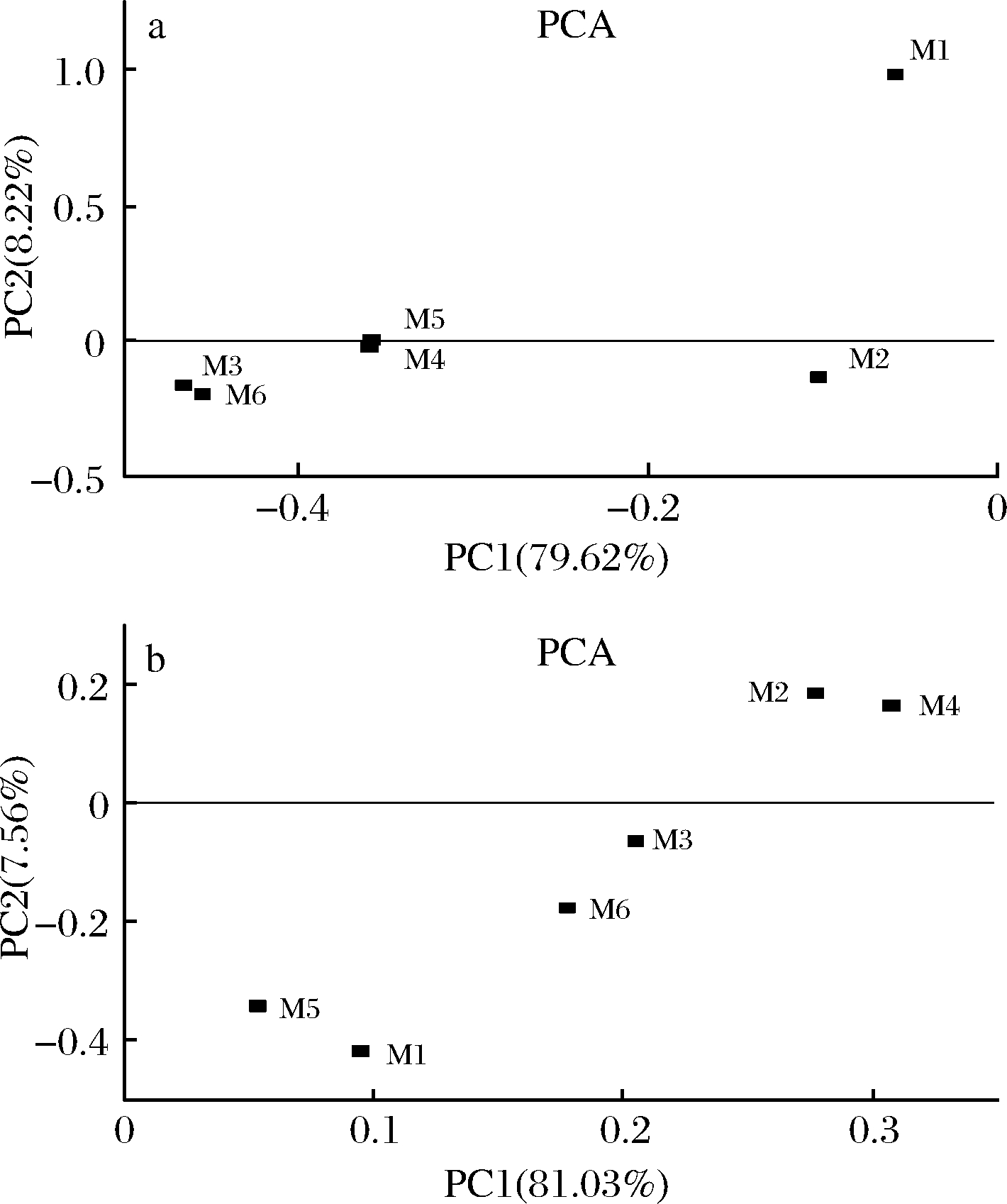
a-细菌;b-真菌
图2 细菌主成分分析图和真菌主成分分析图
Fig.2 principal component analysis for bacteria and
principal component analysis for fungi
3 结论
本研究基于Illumina MiSeq高通量测序平台分析了四川传统腊肉样品中微生物群落结构及多样性。结果表明不同腊肉样品中的微生物多样性存在差异,并且样品中细菌的Alpha多样性指数高于真菌;腊肉中细菌和真菌群落组成分析表明,腊肉样品中共有优势门为厚壁菌门(Firmicutes)和子囊菌门(Ascomycota);样品中的共有优势属为葡萄球菌属(Staphylococcus)和曲霉属(Aspergillus);PCA分析表明,不同来源的样品间微生物组成有一定差异。
[1] 陈美春, 杨勇, 石磊,等. 四川腊肉生产过程中理化及微生物特性的研究[J]. 食品科学, 2008,29(5): 149-152.
[2] 郭昕, 张春江, 胡宏海,等. 不同类型腊肉挥发性风味成分的比较研究[J]. 现代食品科技, 2014, 30(12): 247-254.
[3] GUO X, HUANG F, ZHANG H, et al. Classification of traditional Chinese pork bacon based on physicochemical properties and chemometric techniques[J]. MEAT SCIENCE, 2016,117: 182-186.
[4] 张平, 杨勇, 曹春廷,等. 食盐用量对四川腊肉加工及贮藏过程中肌肉蛋白质降解的影响[J]. 食品科学, 2014, 35(23): 67-72.
[5] 刘洋, 王卫, 王新惠,等. 微生物发酵剂对四川腊肉理化及微生物特性的影响[J]. 食品科技, 2014, 39(6): 124-129.
[6] 全拓, 邓大川, 李洪军,等. 川味腊肉货架期间主要微生物的研究[J]. 西南大学学报(自然科学版), 2017, 39(2): 14-21.
[7] 刘有晴, 黄丹, 于华,等. 四川腊肉中乳酸菌的筛选、产酸特性及其氨基酸产物分析[J]. 食品与机械, 2016, 32(1): 34-37.
[8] 于华, 黄丹, 陈卓,等. 四川腊肉中产脂肪酶霉菌筛选及产酶条件研究[J]. 中国食品添加剂, 2016(12): 78-83.
[9] 田圆圆, 刘绒梅, 耿琦,等. 四川传统腌腊肉制品中乳酸菌的分离鉴定及其抗氧化能力研究[J]. 中国测试, 2018, 44(1): 54-59.
[10] 陈竞适, 刘静, 任海姣,等. 湘西陈年腊肉微生物群落分析及高产脂肪酶细菌的筛选[J]. 肉类研究, 2017, 31(3): 1-6.
[11] 冯秀娟, 刘成国, 娄爱华,等. 湖南腊肉中优势菌种的筛选及初步鉴定[J]. 中国酿造, 2012, 31(5): 127-130.
[12] 李福荣, 袁德峥, 宋淑红. 信阳传统发酵腊肉细菌的分离纯化及鉴定[J]. 信阳师范学院学报(自然科学版), 2009, 22(4): 590-592, 610.
[13] 毕旺来, 胡雷凤, 明亮,等. 烟熏腊肉货架期主要微生物的研究[J]. 武汉轻工大学学报, 2019, 38(2): 16-20.
[14] 曹磊, 梁春御, 曹瑛瑛,等. 甘南地区牦牛曲拉中细菌群落结构研究[J]. 食品科学: 1-12[2019-01-04]. http://kns.cnki.net/kcms/detail/11.2206. ts.20190102.1545.153.html.
[15] LI X F, LI C, YE H, et al. Changes in the microbial communities in vacuum-packaged smoked bacon during storage[J]. Food Microbiology, 2019,77: 26-37.
[16] 聂志强, 韩玥, 郑宇,等. 宏基因组学技术分析传统食醋发酵过程微生物多样性[J]. 食品科学, 2013, 34(15): 198-203.
[17] SCHLOSS P D, WESTCOTT S L, RYABIN T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied & Environmental Microbiology, 2009,75(23):7 537-7 541.
[18] CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-high throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. ISME Journal, 2012, 6(8): 1 621-1 624.
[19] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. Caparose JGKJ, Stombaugh J, Bittinger K, Bushman FD. QIIME allows analysis of high-throughput community sequencing data. Nat Met 7: 335-336[J]. Nature Methods, 2010, 7(5): 335-336.
[20] EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2 194-2 200.
[21] WANG Q, GARRITY G M, TIEDJE J M, et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5 261-5 267.
[22] FU Y, LI X, ZHENG S, et al. Classification and identification of bacteria in the soil treated by AcMNPV using high-throughput sequencing technique[J]. Biotechnology & Bioprocess Engineering, 2015, 20(5): 931-936.
[23] BLAXTER M, MANN J, CHAPMAN T, et al. Defining operational taxonomic units using DNA barcode data[J]. Philosophical Transactions of the Royal Society B Biological Sciences, 2005, 360(1 462): 1 935-1 943.
[24] 郭壮, 沈馨, 董蕴,等. 襄阳大头菜腌制液膜醭细菌群落结构研究[J]. 中国酿造, 2017, 36(7): 143-147.
[25] 张瑶, 白娟, 汪雪瑞,等. 鲊肉粉发酵过程中细菌动态变化研究[J]. 中国酿造, 2018, 37(6): 51-55.
[26] 孙喆, 甄玉国,赵巍,等. 高通量测序分析酵母培养物对肉仔鸡盲肠菌群的影响[J]. 中国畜牧杂志, 2017, 53(12): 67-72.
[27] 田建军, 张开屏, 杨明阳,等. 应用Illumina MiSeq测序技术比较风干肉中细菌多样性和微生物安全性[J]. 食品科学, 2019, 40(8): 33-40.
[28] 董蕴, 王玉荣, 王尧,等. 基于变性梯度凝胶电泳和MiSeq高通量测序技术分析恩施地区腊肉的细菌多样性[J]. 肉类研究, 2018, 32(10): 37-42.
[29] 米瑞芳, 陈曦, 熊苏玥,等. 传统自然发酵酸肉中细菌群落多样性与风味品质分析[J]. 食品科学, 2019, 40(2): 85-92.
[30] 蔡教英, 王小玉, 姚丽锋,等. 冰鲜鸽肉贮藏过程中的微生物菌群多样性[J]. 肉类研究, 2018, 32(9): 41-46.
[31] JANG G I, KIM G, HWANG C Y, et al. Prokaryotic community composition in alkaline-fermented skate (Raja pulchra)[J]. Food Microbiology, 2017,61: 72-82.
[32] ZANG J, XU Y, XIA W, et al. Dynamics and diversity of microbial community succession during fermentation of Suan yu, a Chinese traditional fermented fish, determined by high throughput sequencing [J]. Food Research International, 2018,111: 565-573.
[33] 乌仁图雅. 应用454焦磷酸测序技术对传统发酵乳制品微生物多样性的研究[D]. 呼和浩特:内蒙古农业大学, 2014.
[34] 秦丹. 四川腊肉和香肠加工贮藏过程中微生态系统的RAPD分析[D]. 雅安:四川农业大学, 2011.
[35] LEE S H, JUNG J Y, JEON C O.Bacterial community dynamics and metabolite changes in myeolchi-aekjeot, a Korean traditional fermented fish sauce, during fermentation[J]. International Journal of Food Microbiology, 2015,203: 5-22.
[36] 李晓燕, 王卫, 张佳敏,等. 发酵微生物提升传统腌腊肉制品风味和安全性研究进展[J]. 食品工业, 2018, 39(5): 275-279.
[37] 陈美春. 四川腊肉加工贮藏中理化、微生物特性及产香葡萄球菌筛选的研究[D]. 雅安:四川农业大学, 2008.
[38] 谢康庄, 颜道民, 郑旭,等. 湘西腊肉中高产脂肪酶真菌的筛选[J]. 轻工科技, 2019, 35(4): 1-4.
[39] 祁乐乐. 发酵肉制品中的微生物发酵剂及其作用[J]. 现代食品, 2016,4(7): 127-128.
[40] 林克忠, 杨耀寰, 竺尚武,等. 金华火腿的质量和色香味形成与霉菌关系的研究[J]. 肉类研究, 1992(2): 10-16;21.
[41] 贺稚非, 甄宗圆, 李洪军,等. 金华火腿发酵过程中微生物区系研究[J]. 食品科学, 2008,28(1): 190-195.