甘南牦牛曲拉中真菌群落结构
马江1,文鹏程1,罗俏俏1,曹磊1,朱艳1,杨敏2,张卫兵1*,张忠明1*
1(甘肃农业大学 食品科学与工程学院,甘肃 兰州,730070)2(甘肃农业大学 理学院,甘肃 兰州,730070)
摘 要 提取曲拉样品的DNA,选择合适的通用引物扩增ITS特定区域,采用Illumina高通量测序技术检测目标区域的序列变异和丰度,通过分析曲拉样品中真菌的Alpha多样性、物种分类组成及Beta多样性,以揭示甘南牦牛曲拉中真菌菌群的群落结构。结果表明,曲拉中具有高度的真菌多样性,并且不同来源样品的Shannon指数、Simpson指数、Chao1指数和ACE指数存在差异;在曲拉样品中共检测出6个门和123个属的真菌,所有样品中的共有优势门为子囊菌门(Ascomycota),共有优势属为毕赤酵母属(Pichia)和双足囊菌属(Dipodascus);采用非度量多维尺度分析和非加权组平均法聚类分析可以将曲拉样品分为3类,结果与样品来源一致;置换多元方差分析表明,不同来源的样品间真菌组成差异显著(P=0.001);Spearman关联网络图表明,样品中真菌群落之间正相关占主导地位;研究结果可为牦牛曲拉的品质控制奠定基础。
关键词 牦牛曲拉;Illumina高通量测序;真菌多样性
第一作者:硕士研究生(张卫兵教授和张忠明副教授为共同通讯作者,E-mail:45330301@qq.com,1958711127@qq.com)
基金项目:国家自然科学基金项目(31560442;31960486;31760466);甘肃农业大学学科建设基金项目(GAU-XKJS-2018-247);企业研究转化与产业专项(2018-SF-C29)
收稿日期:2019-09-01,改回日期:2019-11-06
DOI:10.13995/j.cnki.11-1802/ts.022139
引用格式:马江,文鹏程,罗俏俏,等.甘南牦牛曲拉中真菌群落结构[J].食品与发酵工业,2020,46(4):51-56.MA Jiang,WEN Pengcheng,LUO Qiaoqiao, et al. Study on fungi microbiota of Yak Qula in Gannan region[J].Food and Fermentation Industries,2020,46(4):51-56.
Study on fungi microbiota of Yak Qula in Gannan region
MA Jiang1, WEN Pengcheng1, LUO Qiaoqiao1, CAO Lei1, ZHU Yan1, YANG Min2,ZHANG Weibing1*, ZHANG Zhongming1*
1(College of Food Science and Engineering,Gansu Agricultural University,Lanzhou 730070,China)2(College of Science, Gansu Agricultural University, Lanzhou 730070, China)
ABSTRACT This study analysed the fungi microbiota structure of Qula, a kind of fermented Yak milk product, using high throughput sequending technology. After extraction the metagenome DNA of Qula sample, the universal primers of fungi ITS marker gene were amplied and sequenced using Illumina high throughput sequencing technique. And the variation of microbiota structure and abundance were conducted by analyzing the Alpha diversity, species composition and Beta diversity of fungi in yak Qula samples from Gannan. The results showed that there was a high degree of fungal diversity in different Qula samples indicated in Shannon index, Simpson index, Chao1 index and ACE index. A total of 6 phyla and 123 genera of fungi were detected in the Qula samples. The common dominant phylum in all samples was Ascomycota, and the common dominant genera were Pichia and Dipodascus. The non-metric multidimensional scaling analysis and the unweighted group averaging clustering analysis were used to classify the Qula samples into three categories which were consistent with the sample sources. Adonis/PERMANOVA multivariate analysis of variance and replacement test showed that there were significant differences in fungal composition between samples from different sources (P=0.001). The Spearman correlation network diagram showed that there were positive correlation between dominant fungal communities. This study laid the foundation for the quality control of yak Qula.
Key words Yak Qula;Illuminahigh-throughput sequencing;Fungi diversity
牦牛曲拉是藏族牧民将牦牛乳脱脂、自然发酵、脱水、干燥等工艺制备得到的一种发酵乳制品[1-2]。有研究报道,我国曲拉总产量约3万t,其中1/3的留作牧民食用[3-4]。与新鲜牛乳相比,曲拉中蛋白质含量高达75.0%以上,脂肪含量为4.0%~7.0%,营养价值极高[5],同时也具有调节人体肠道微生物和改善人体肠道菌群的作用[6]。甘南地区的曲拉是以牧民家庭自制为主,其制作环境相对开放,其中含有多种真菌资源。
目前,有关乳制品中真菌资源的报道较多。张晓旭[7]通过传统培养法从新疆和内蒙古曲拉中分离出91株酵母菌,对其生物学特性和发酵特性进行比较,结果表明,地域不同的同种酵母亦表现不同的特性;杨俊俊[8]从牦牛曲拉中鉴定出毕赤酵母、酿酒酵母、乳酸克鲁维酵母等为主要优势菌群;李先胜等[9]对西藏地区11份曲拉进行分离筛选,Saccharomyces cerevisiae、Kluyveromycesmarxianus、Debaryomyceshansenii、Candida zeylanoides和Torulasporadelbrueckii为曲拉样品中的优势属,且不同样品之间菌落差异较大;次顿等[10]通过变性梯度凝胶电泳法(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)从拉萨酥油中鉴定出优势真菌菌群为假丝酵母属、亚罗酵母属和毕赤酵母;乌仁图雅[11]和张冬蕾[12]通过焦磷酸测序技术对传统牦牛酸奶中真菌多样性进行研究,结果表明,牦牛酸奶中优势门均为子囊菌门。以上研究采用的方法主要为传统培养法、变性梯度凝胶电泳、454焦磷酸测序等,具有通量低、操作复杂和准确率低等缺陷。新兴的Illumina高通量测序技术[13-14]具有操作简单、成本较低的优势,并且采用边合成边测序原理,结果可信度高。因此,通过Illumina高通量测序技术能够全面而准确地了解研究对象中微生物种类组成和结构。
本研究采用Illumina高通量测序技术对采自甘肃省甘南藏族自治州的曲拉样品中真菌多样性进行分析,以期全面解析曲拉中的真菌组成及群落结构,为曲拉的安全生产提供理论指导,同时为适合曲拉发酵微生物的筛选奠定基础。
1 材料与方法
1.1 材料与试剂
E.Z.N.A.Soil DNA试剂盒,美国OMEGA公司;Qubit2.0 DNA检测试剂盒,美国Invitrogen公司;Q5高保真DNA聚合酶,美国New England Biolabs公司;凝胶回收试剂盒,美国AXYGEN公司;TruSeq Nano DNA LT Library Prep Kit,美国Illumina公司。
1.2 仪器与设备
Pico-21型台式离心机,Thermo Fisher;DYY-6C型电泳仪、DYCZ-21型电泳槽,北京市六一仪器厂;凝胶成像系统,美国UVP公司;Q32866型Qubit 2.0分光光度计,Invitrogen公司;T100TM Thermal Cyeler型PCR仪,BIO-RAD公司;MiSeq System SY-410-1003高通量测序仪,美国Illumina公司。
1.3 实验方法
1.3.1 曲拉样品的采集
曲拉样品于2017年9月采自甘肃省甘南藏族自治州合作市那吾乡塔瓦(S01、S02、S03)、加拉(S61、S62、S63)和玛岗村(S121、S122、S123)3个村庄的9个不同牧民家庭,将所有样品装进自封袋中,置于冷藏箱中运输至实验室以备试验。
1.3.2 曲拉微生物总DNA的提取
曲拉样品中微生物总DNA提取采用E.Z.N.A.Soil DNA Kit D5625-01试剂盒,按照使用说明从样品中提取DNA。
1.3.3 PCR扩增及测序
使用真菌特异性引物对曲拉样品所提取的DNA的ITS区域进行扩增,ITS区扩增引物分别为ITS5F(GGAAGTAAAAGTCGTAACAAGG)和ITS1R(GCTGCGTTCTTCATCGATGC)。PCR条件如下:预变性为95 ℃、5 min,然后95 ℃、30 s、56 ℃、30 s、72 ℃、30 s共25个循环,72 ℃退火10 min,最后保存在4 ℃条件下。通过琼脂糖凝胶电泳对PCR产物进行检测,然后用试剂盒进行回收进行质量检测并建库。最后由上海派森诺生物科技股份有限公司在MiSeq测序平台进行双端测序。
1.3.4 高通量测序数据处理
MiSeq测序得到的数据采用Mothur(V.1.31.2)和QIIME(V.1.7.0)软件进行处理及分析[15-16]。首先采用滑动窗口法对FASTQ格式的双端序列逐一进行质量筛选,然后利用FLASH软件(v1.2.7)对质量初筛的双端序列进行配对连接。将连接后的序列识别分配对应样本,从而获得有效序列。在测序过程中会产生一些错误或疑问序列,因此采用QIIME软件(v1.8.0)[17]识别疑问序列。通过QIIME软件(v1.8.0)调用USEARCH(v5.2.236)检查并剔除嵌合体序列[18-21]。使用QIIME软件,调用UCLUST算法进行序列聚类,以97%的序列相似度进行归并和OTU[22]划分。测序数据在NCBI数据库中的收录编号为PRJNA431342(https://www.ncbi.nlm.nih.gov/bioproject/431342)。
1.3.5 群落多样性和统计分析
利用Mothur(V.1.31.2)软件进行Alpha多样性分析,并在不同的分类水平上对群落结构进行了统计分析;
使用R软件对Weighted的UniFrac距离矩阵分别进行NMDS分析,通过二维排序图描述群落样本的结构分布;
使用QIIME软件进行UPGMA聚类分析和Adonis/PERMANOVA多元方差分析;
使用Mothur软件,计算优势属之间的Spearman等级相关系数,对其中rho>0.6且P<0.01的相关优势属构建关联网络,并导入软件进行可视化。
1.4 数据分析
利用Excel 2010和Origin 2018软件进行数据处理分析并作图。
2 结果与分析
2.1 Alpha多样性分析
9份不同曲拉样品真菌Alpha多样性指数如表1所示,通过真菌的ITS区测序,9份样品共产生高质量序列663 037条,将所有序列按97%的相似度进行OTU聚类,得到1 667个OTU。由序列数及OTU聚类可以看出,曲拉中真菌种类繁多,物种丰富,且不同家庭手工制作的曲拉样品中存在一定差异。
表1 测序结果及真菌Alpha多样性指数表
Table 1 Sequencing results and fungal Alpha diversity index
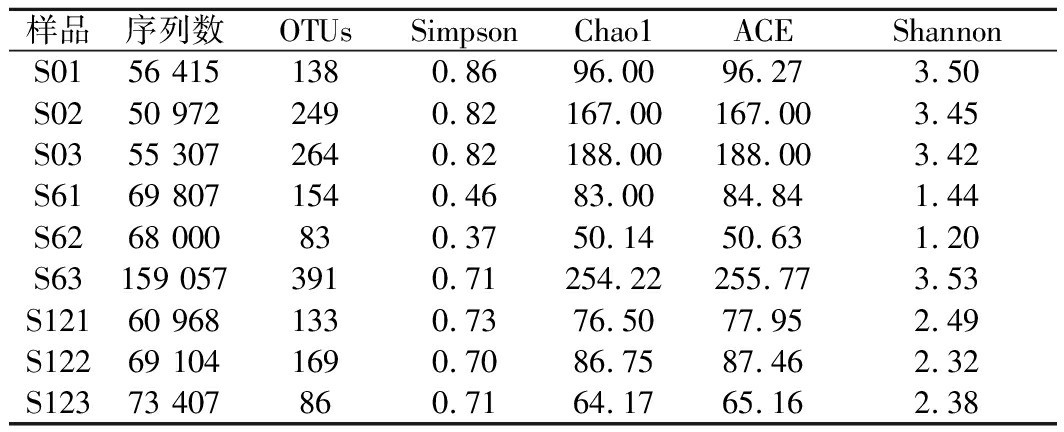
样品序列数OTUsSimpsonChao1ACEShannonS0156 4151380.8696.0096.273.50S0250 9722490.82167.00167.003.45S0355 3072640.82188.00188.003.42S6169 8071540.4683.0084.841.44S6268 000830.3750.1450.631.20S63159 0573910.71254.22255.773.53S12160 9681330.7376.5077.952.49S12269 1041690.7086.7587.462.32S12373 407860.7164.1765.162.38
Shannon指数和Simpson指数是综合衡量物种多样性的指数,其值越高,物种多样性越丰富,反之物种多样性越少。9个样品中指数最高的分别为S63(3.53)和S01(0.86),表明样品S63和S01中真菌OTU的多样性较高;S62的指数值最低,分别为1.20和0.37,反映了S62中真菌多样性较低;Chao1指数和ACE指数主要侧重于体现稀有群落的丰富度,指数越大,表明群落的丰富度越高。而曲拉样品中Chao1指数和ACE指数最高的均为S63,分别为254.22和255.77,并且OTU也是最多的(391)。可以看出,样品的Chao1指数和ACE指数大小与OTU数呈正相关,而群落多样性与群落丰富度之间不存在相关性。
2.2 稀疏曲线
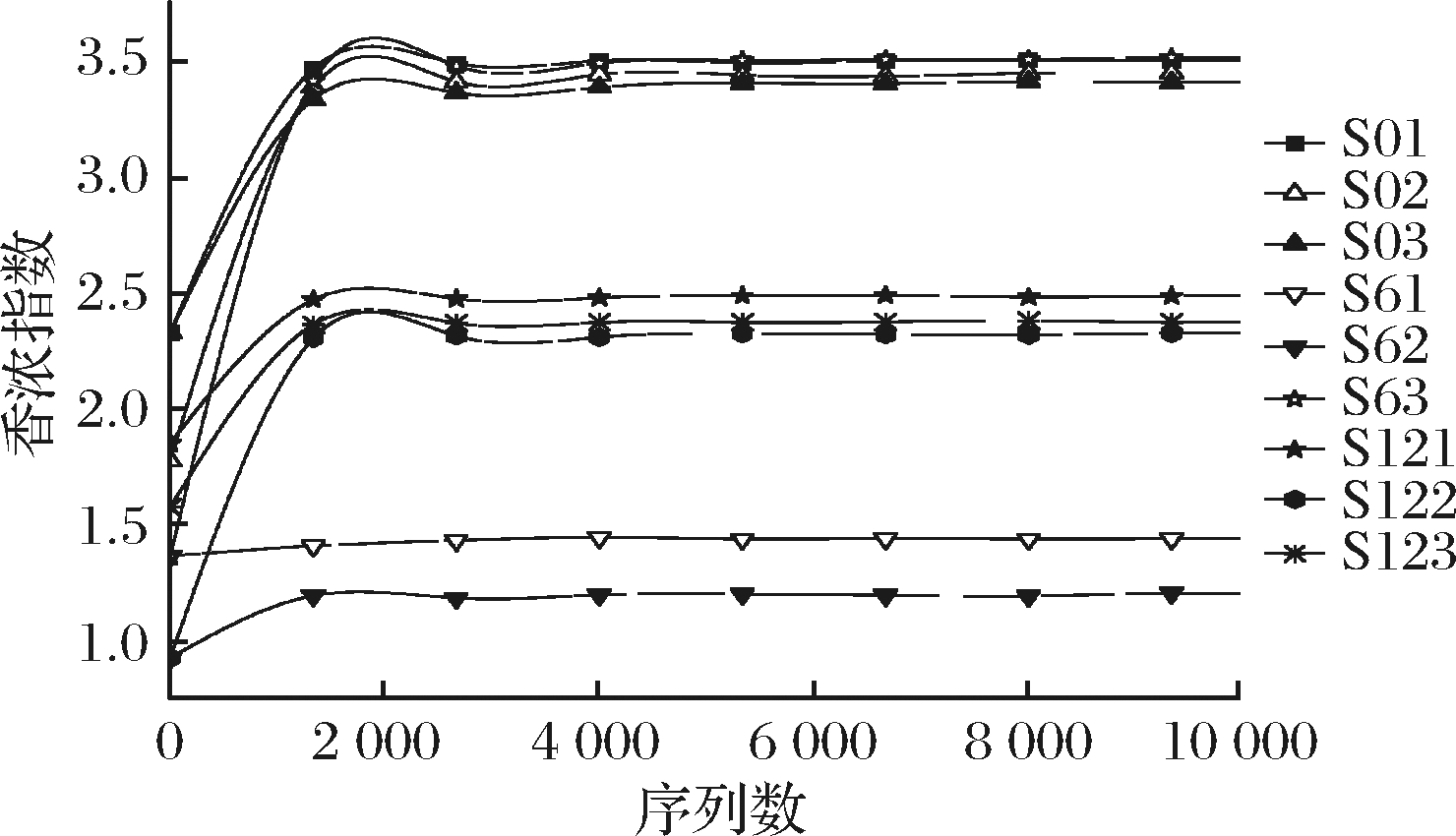
9份不同曲拉样品的香浓指数稀疏曲线如图1所示。由图1可知,不同样品的香浓指数随着测序量的增加而呈现上升趋势,说明在此测序水平下,样品中真菌微生物的多样性较高,并且样品中还有较多的物种还没有被检测到;当测序量较高时,香浓曲线逐渐与X轴接近平行,说明在此测序水平下,样品中真菌的群落多样性已能够充分的展现。
2.3 丰度等级曲线
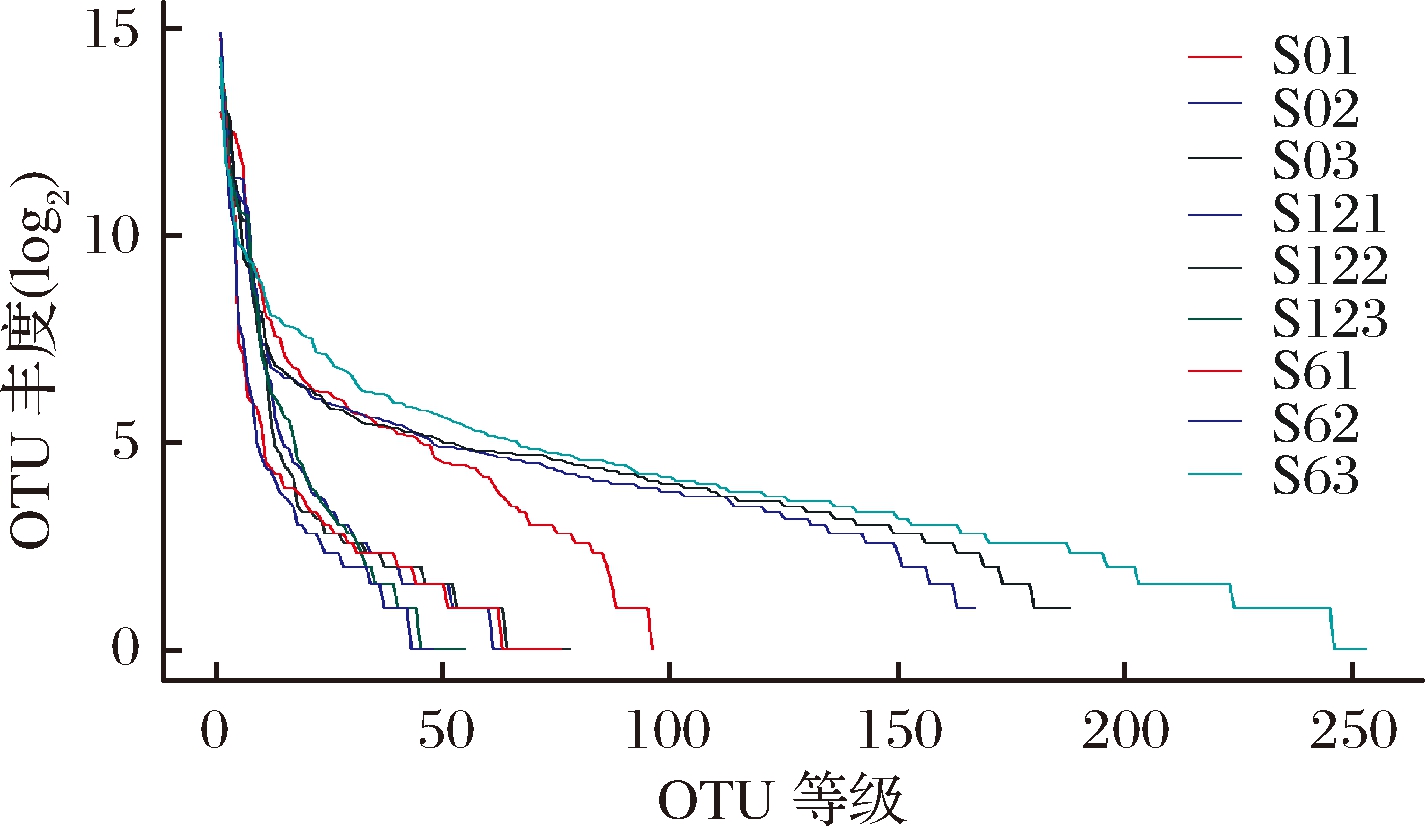
9份不同曲拉样品的丰度等级曲线(rank-abundance curve)如图2所示。由图2可知,9个样品的曲线趋势相似。在水平方向,各样品曲线宽度反映丰富度,在横轴上的宽度,体现出不同样品的物种丰度可能有较大的差异,其中S63丰富度最高,S62、S123丰富度最低。另外,曲线的形状反映样品的均匀度,曲线越平缓,群落组成的均匀度越高,曲线越陡峭,则群落中各OTU间的丰度差异越大,均匀度越低。图2中S63均匀度最高,群落中各OTU间的差异最小。
2.4 样品中真菌群落在门水平的比较
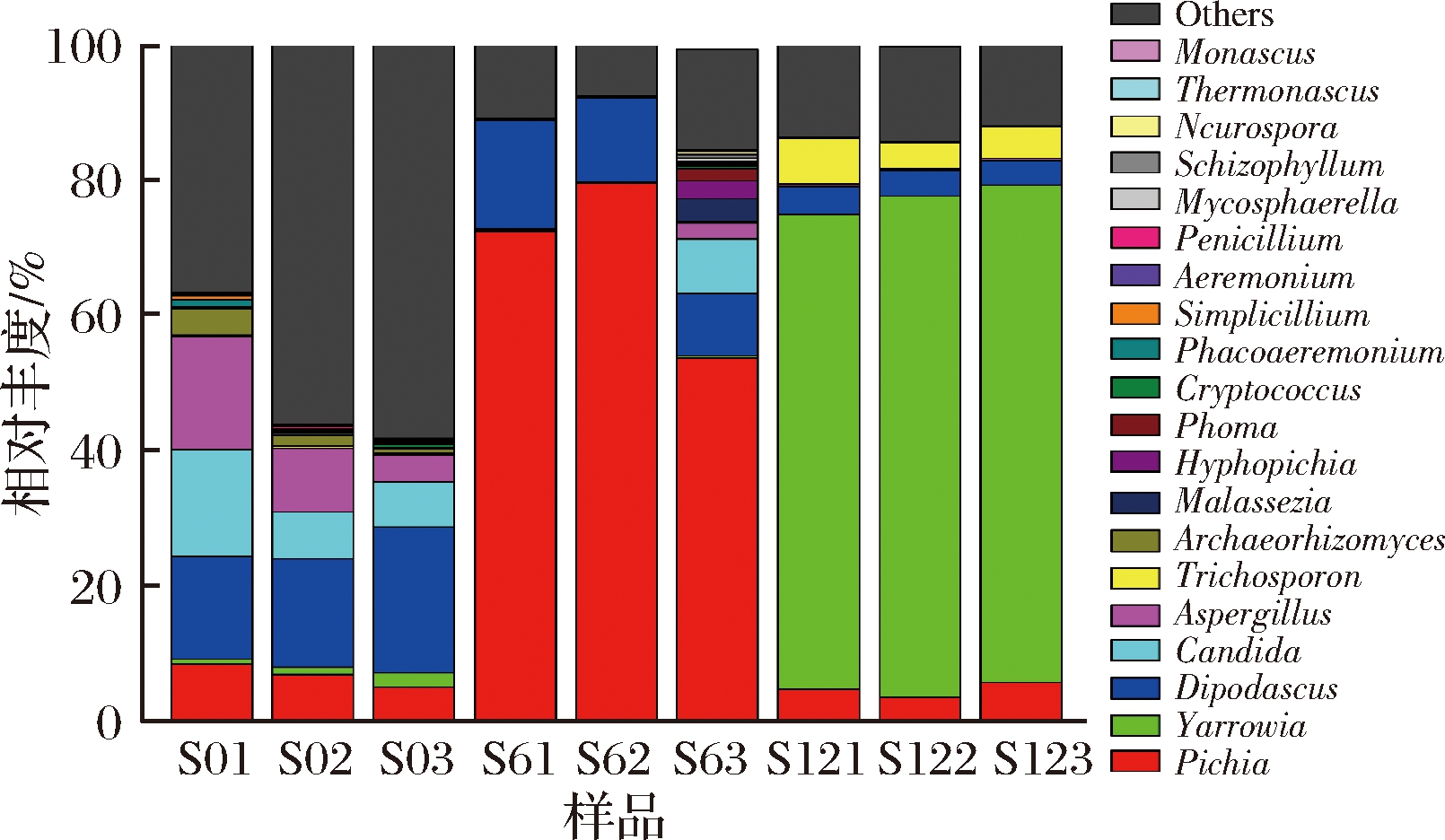
图3为9份曲拉样品从门的分类水平进行鉴定。在曲拉样品中共检测出6个门,分属于子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、接合菌门(Zygomycota)、壶菌门(Chytridiomycota)、球囊菌门(Glomeromycota)和罗兹菌门(Rozellomycota)。由图2可知,子囊菌门为9份样品的共有优势门(相对丰度>1%),平均相对丰度为96.544%;担子菌门在S01、S02、S03、S63、S121、S122和S123样品中为优势属(相对丰度大于1%),平均相对丰度为3.025%;接合菌门、壶菌门、球囊菌门在9份样品中的丰度很低,为样品中非优势门;另外,还有一些在门水平上未鉴定出。
子囊菌门为优势菌门这一结果与新疆地区传统发酵酸牛乳、对韩国酒精饮料和中国白酒中的真菌多样性研究结果一致[23-25]。另外,在新疆阿图什和乌什传统发酵酸奶[26]中均检出担子菌门、接合菌门、壶菌门、球囊菌门和罗兹菌门,并且壶菌门、球囊菌门和罗兹菌门均为非优势门。
2.5 样品中真菌群落在属水平的比较
图4为9份不同曲拉样品从属的分类水平进行鉴定,共检测出123个属。从属分类水平来看,9份曲拉样品中毕赤酵母属(Pichia)和双足囊菌属(Dipodascus)为共有优势属,平均相对丰度分别为26.635%和11.393%;解脂耶式酵母属(Yarrowia)在样品S02、S03、S121、S122和S123中为优势属,平均相对丰度为24.634%;念珠菌属(Candida)和曲霉属(Aspergillus)在样品S01、S02、S03和S63中为优势属,平均相对丰度分别为4.179%和3.640%;毛孢子菌属(Trichosporon)在S121、S122和S123中为优势属,平均相对丰度为1.711%;Archaeorhizomyces在样品S01和S02中占优势,马拉色霉菌属(Malassezia)、丝孢毕赤氏酵母属(Hyphopichia)和茎点霉属(Phoma)仅在S63中占优势:Phaeoacremonium只在S01中为优势属。
这是由于曲拉主要以家庭作坊式生产为主,发酵过程易受奶源、制作方法、海拔、地理环境、气候环境、发酵温度、发酵时间等影响[27]。另外,样品中还包含许多在属水平上未鉴定的属,未鉴定出属也具有较高的丰度,其丰度值在7.363%~55.276%,平均丰度值为23.818%。
毕赤酵母、解耶氏酵母和曲霉属也在西藏曲拉检测到,这是由于这些酵母是酸凝乳[28]干酪中最常见的菌,能够利用发酵乳糖产生乳酸,使凝乳的pH有所升高,有助于发酵乳制品的成熟。
茎点霉属在我国属于检疫性病菌[29],这是由于制作的环境存在安全隐患,并且在发酵豆酱中也检测到该菌[30],目前未有危害报道。
2.6 基于UniFrac距离的NMDS非度量多维尺度分析
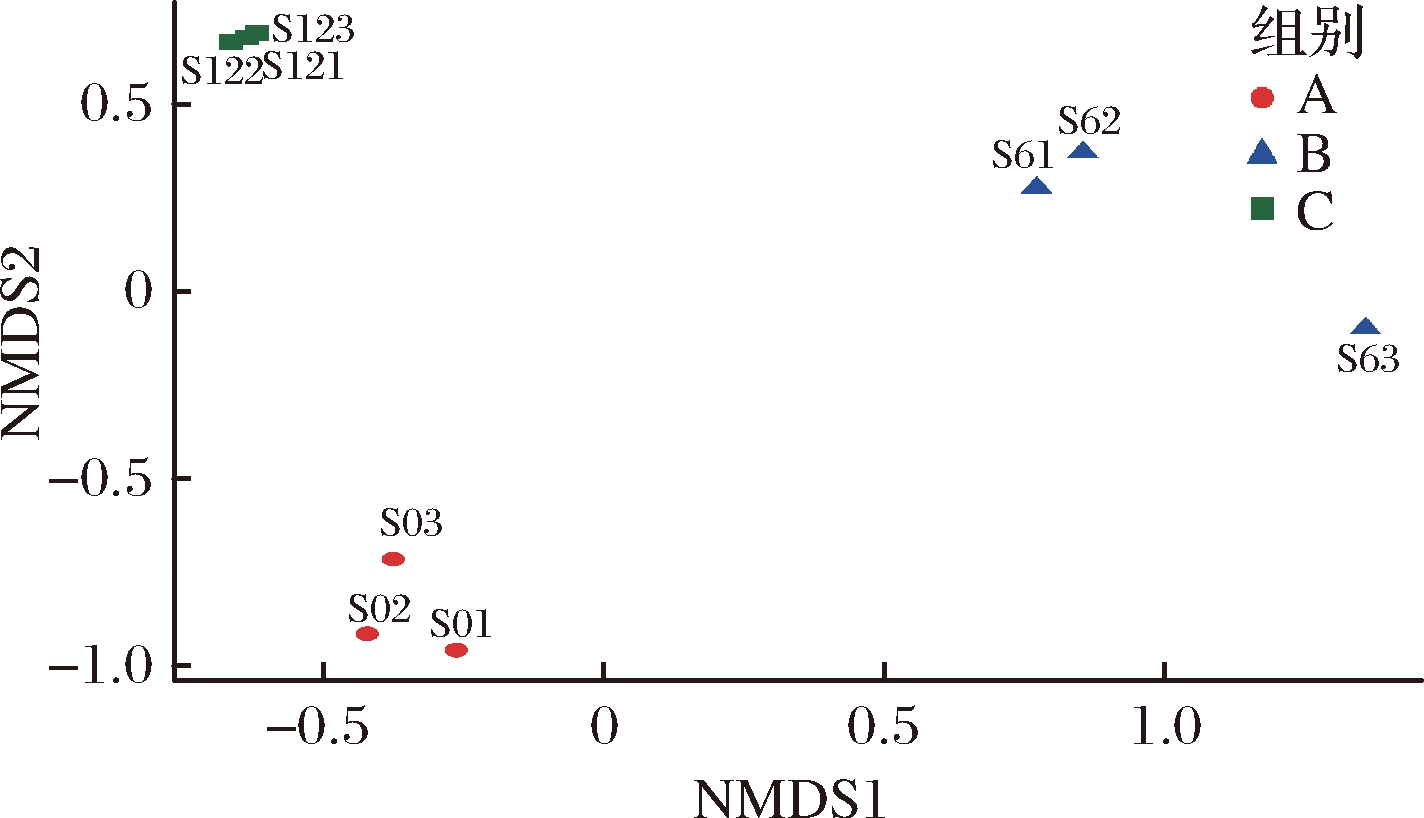
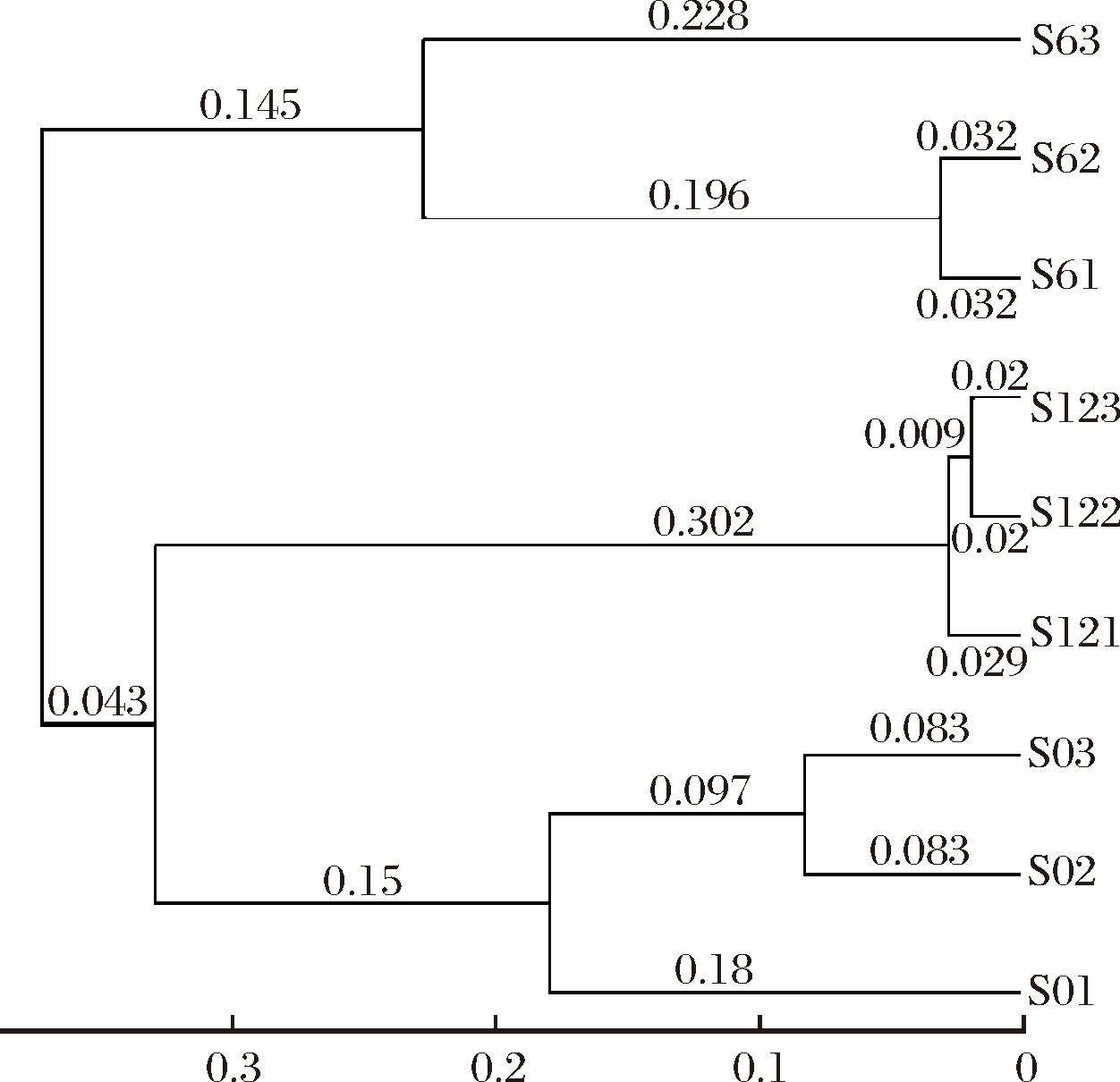
图5是基于加权UniFrac距离的NMDS分析,由图5可知,9份不同来源的曲拉样品在NMDS1和NMDS2维度上有明显的聚集和分离趋势,样品S01、S02和S03聚为第一类(A),S61、S62和S63聚为第二类(B),S121、S122和S123聚为第三类(C)。A和C样本中各点分布比较紧密,表明样品个体间差异性较小,微生物群落结构相似;B样本中各点分布疏散,样品个体间差异性较大。原因可能是曲拉是自然状态下发酵并且制作工艺比较粗放。这与李伟程等对传统发酵乳制品中微生物多样性研究结果一致[31]。
聚类分析主要是以等级树的形式展示样品之间的相似度,通过聚类树的分枝长度衡量聚类效果的好坏。图6为基于加权UniFrac距离的曲拉样品的聚类图,由图6可知,不同来源的样品聚集到不同的类别,说明不同来源的样品微生物多样性存在一定的差异性。在S121、S122和S123曲拉样品之间,其分枝长度最短,样本之间相似度最高;其次为S01、S02和S03,最后为S61、S62和S63曲拉样品。
2.7 置换多元方差分析
基于置换的PERMANOVA(permutational multivariate analysis of variance)[32]分析借鉴了ANOVA方差分析多组间差异的统计检验思路,通过对距离矩阵进行置换检验,从而评价原始样本组间差异的大小及其统计学显著性。表2为基于加权UniFrac距离的Adonis/PERMANOVA分析,由表可知,F=MSt/MSe=16.34,根据df1=2,df2=6查F检验表可得,F0.01(2,6)=10.92,而F>F0.01(2,6),P<0.01,表明不同来源的样品之间差异极显著。而“Pr(>F)”是通过999次置换检验获得的P值,P值越小组间差异性就越强。本研究中不同组间的样品真菌组差异极显著(P=0.001)。
表2 加权UniFrac距离的置换多元方差分析
Table 2 PERMANOVA analysis of Weighted
UniFrac distance

变异来源自由度(df)离差平方和均方差F值R2Pr(>F)处理间21.3080.65416.3400.8440.001∗∗∗处理内60.2400.0400.155总变异81.5491.000
注:“***”代表差异极显著(P<0.001)
2.8 优势物种互作Spearman关联网络分析
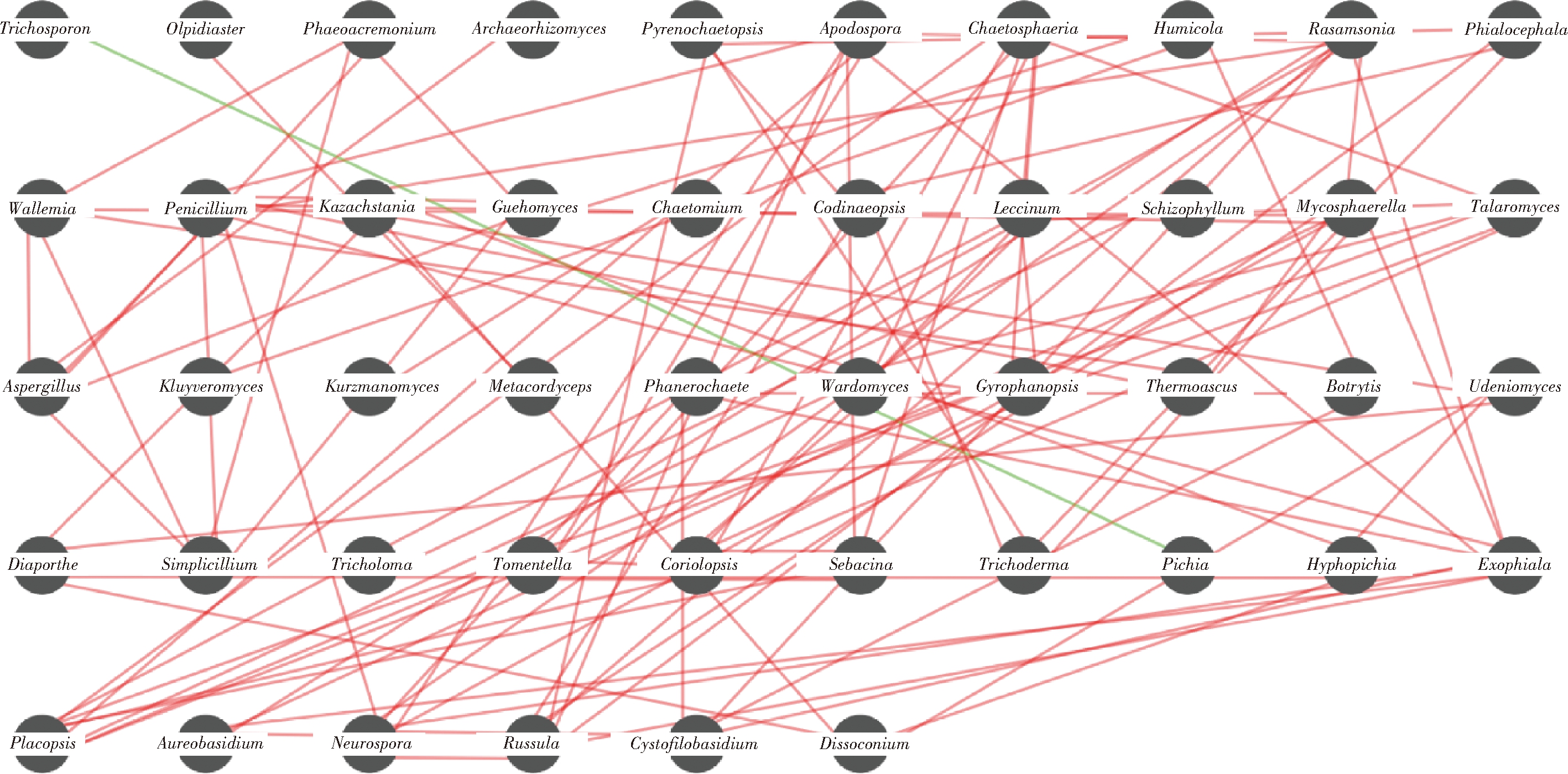
关联网络基于微生物成员之间相互关系,对不同群落成员之间进行分析,推断不同微生物类群之间的的相互作用[33]。本研究使用Spearman等级相关系数计算牦牛曲拉样品中属之间的关系,并通过Cytoscape[34]软件可视化。图7为丰度在前50位的优势属关联网络图,由图7可知,网络图由46个节点和106个边组成。正相关和负相关之比为105∶1,Aspergillus、Penicillium、Neurospora、Rasamsonia、Apodospora、Placopsis、Chaetosphaeria、Leccinum、Mycosphaerella、Talaromyces、Exophiala、Wardomyces、Gyrophanopsis、Coriolopsis和Russula是网络的中枢属(每个节点≥6个边)。第1优势属毕赤酵母属与优势属毛孢子菌属呈负相关,曲霉属与Archaeorhizomyces、Guehomyces、Wallemia、Penicillum、Phaeoacremonium、Simplicillium呈正相关。而解脂耶式酵母、双足囊菌属和念珠菌与其他属之间没有相关性。
3 结论
本研究基于Illumina MiSeq高通量测序平台分析甘南牦牛乳曲拉样品中真菌群落结构及多样性。结果表明,不同来源的曲拉样品中的微生物多样性存在差异性。曲拉中真菌群落组成分析表明,曲拉样品中共有优势菌门为子囊菌门(Ascomycota);共有优势属为毕赤酵母属(Pichia)和双足囊菌属(Dipodascus);Beta多样性结果表明真菌群落组成在不同来源的样品中存在差异。Adonis/PERMANOVA多元方差分析表明,不同组间的样品真菌组差异极显著(P=0.001)。Spearman关联网络图表明,真菌群落之间正相关占主导地位。
参考文献
[1] 陈梦音,王琳琳,韩玲,等.基于主成分和聚类分析的曲拉品质的综合评价[J].食品科学,2017,38(13):102-107.
[2] 刘亚东,张悦,贺银凤,等.西藏曲拉和发酵乳中抗氧化和益生特性乳酸菌的筛选及鉴定[J].食品工业技科技 2019,40(2):142-147.
[3] 王永宁,魏永生.精制干酪素的脱色(中试)研究[J].青海师范大学学报(自然科学版),1997(4):29-31.
[4] 王琳琳,王军,韩玲,等.牧区牦牛“曲拉”优质发酵剂的研究[J].食品工业科技,2014,35(12):152-156.
[5] LIU H N,ZHANG C,ZHANG H,et al.pH treatment as an effective tool to select the functional and structural properties of yak milk caseins[J].Journal of Dairy Science,2013,96(9):5 494-5 500.
[6] 张洋,霍贵成.曲拉的理化特性研究[J].中国乳品工业,2013,41(6):17-19;22.
[7] 张晓旭.西部牧区酸马奶和奶渣中酵母菌分离筛选及其应用研究[D].杨凌:西北农林科技大学,2016.
[8] 杨俊俊.西藏牦牛奶渣中微生物的分离鉴定及优良乳酸菌的筛选[D].杨凌:西北农林科技大学,2014.
[9] 李先胜,姜铁民,陈历俊.西藏曲拉中酵母菌的分离、鉴定及其优势菌分析[J].中国乳品工业,2013,41(5):26-28;3.2
[10] 次顿,潘虎,达娃卓玛,等.拉萨传统乳制品酥油的微生物多样性与开发前景研究[J].西南农业学报,2018,31(1):192-199.
[11] 乌仁图雅.应用454焦磷酸测序技术对传统发酵乳制品微生物多样性的研究[D].呼和浩特:内蒙古农业大学,2014.
[12] 张冬蕾.应用焦磷酸测序技术对新疆地区传统发酵乳制品中微生物多样性的研究[D].呼和浩特:内蒙古农业大学,2016.
[13] 高庆超,常应九,马蓉,等.黑果枸杞酵素自然发酵过程中微生物群落的动态变化[J].食品与发酵工业,2019,45(13):126-133.
[14] 向凡舒,折米娜,何萌,等.基于DGGE和Illumina MiSeq技术解析恩施地区米酒细菌多样性[J].食品与发酵工业,2019,45(14):41-46;62.
[15] 聂志强,韩玥,郑宇,等.宏基因组学技术分析传统食醋发酵过程微生物多样性[J].食品科学,2013,34(15):198-203.
[16] SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur:Open-source,platform-independent,community-supported software for describing and comparing microbial communities[J].Applied &Environmental Microbiology,2009,75(23):7 537-7 541.
[17] CAPORASO J G,LAUBER C L,WALTERS W A,et al.Ultra-high throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J].ISME Journal,2012,6(8):1 621-1 624.
[18] CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7:335-336.
[19] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011, 27(16):2 194-2 200.
[20] WANG Q,GARRITY G M,TIEDJE J M,et al.Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Applied and Environmental Microbiology,2007,73(16): 5 261-5 267.
[21] FU Y,LI X,ZHENG S,et al.Classification and identification of bacteria in the soil treated by AcMNPV using high-throughput sequencing technique[J].Biotechnology & Bioprocess Engineering,2015,20(5):931-936.
[22] BLAXTER M,MANN J,CHAPMAN T,et al.Defining operational taxonomic units using DNA barcode data[J].Philosophical Transactions of The Royal Society B Biological Sciences,2005,360(1 462):1 935-1 943.
[23] 张冬蕾.应用焦磷酸测序技术对新疆地区传统发酵乳制品中微生物多样性的研究[D].呼和浩特:内蒙古农业大学,2016.
[24] JUNG M J,NAM Y D,ROH S W,et al.Unexpected convergence of fungal and bacterial communities during fermentation of traditional Korean alcoholic beverages inoculated with various natural starters[J].Food microbiology,2012,30(1):112-123.
[25] LI X R,MA E B,YAN L Z,et al.Bacterial and fungal diversity in the traditional Chinese liquor fermentation process[J]. International Journal of Food Microbiology,2011,146(1):31-37.
[26] 玛依乐·艾海提,西热娜依·阿布力克木,努尔古丽·热合曼.应用高通量测序法检测南疆传统酸奶中微生物多样性[J].食品科学,2018,39(20):126-131.
[27] 武悦.曲拉传统制备工艺的优化研究及应用[D].兰州:甘肃农业大学,2016.
[28] 卿蔓君,白梅,张勇,等.西藏曲拉和云南乳饼中酵母菌的鉴定及其生物多样性[J].微生物学报,2010,50(9):1 141-1 146.
[29] 杨玲玉,王颖,程颖慧,等.茎点霉生物学特性研究[J].山东农业大学学报(自然科学版),2016,47(4):501-505.
[30] 陈嵘.我国部分地区自然发酵豆酱中真菌的多样性和真菌毒素的检测[D].沈阳:沈阳农业大学,2008.
[31] 李伟程,侯强川,于洁,等.传统发酵乳制品中微生物多样性研究[J].食品工业科技,2018,39(1):131-136.
[32] ANDERSON M A J.Fitting multivariate models to community data:A comment on distance-based redundancy analysis[J]. Ecology,2001,82(1):290-297.
[33] FAUST K,RAES J.Microbial interactions:From networks to models[J].Nature Reviews Microbiology,2012,10(8),538-550.
[34] SHANNON P.Cytoscape:A Software environment for integrated models of biomolecular interaction networks[J].Genome Research,2003,13(11):2 498-2 504.