木质纤维素类生物质是地球上含量最丰富的可再生资源,包括纤维素(40%~50%)、半纤维素(20%~40%)和木质素(20%~30%),在能源转化方面具有广泛的应用前景。纤维素是木质纤维素中含量最多的组分,通过降解后可以生成不同的寡糖,其中可发酵糖可以发酵生成乙醇,缓解石油资源紧张这一现状[1-3]。在一般情况下,纤维素以不溶的晶体状态存在于自然界中。但是目前存在纤维素酶对晶体状态的纤维素降解效率较低的问题[4-8]。然而最近发现了一种裂解性多糖单加氧酶(lytic polysaccharide monooxygenases, LPMOs)可以降解晶体状态的纤维素[9]。LPMOs是一种铜离子依赖型的氧化酶,可以通过氧化断裂糖苷键的方式降解晶体纤维素[9-11]。而且研究表明,当LPMOs与纤维素酶协同作用时,可以显著提高对纤维素的水解效率[10, 12]。
根据LPMOs的序列相似性可将其分为7个家族,分别为AA9-AA11 [13-15]和AA13-AA16 [16-19]。其中AA9家族主要降解纤维素,由于纤维素是木质纤维素中含量最丰富的组分,因此AA9家族是转化木质纤维素生物质为生物燃料的关键[10, 20]。其中来源于真菌嗜热毁丝霉(Myceliophthora thermophile C1)的 MtC1LPMO (GenBank: AKO82493.1)蛋白,以C1和C4两种氧化方式断裂纤维素的糖苷键,同时纤维素存在时,可以断裂木聚糖的糖苷键[21]。因为木聚糖是半纤维素的组成部分,而半纤维素是木质纤维素的重要组分[22]。半纤维素包裹着纤维素极大地阻碍了工业酶制剂对纤维素的降解。因此,对半纤维素的降解也是高效利用生物质的关键。
MtC1LPMO在生物质转化过程中具有重要的作用,这类酶价值高且需求量较大,因此异源表达是一种非常有效的供应手段。大肠杆菌作为人们研究的最深入的微生物之一,是生产重组蛋白最常用的宿主,为许多外源蛋白提供了最经济快速的表达系统[23-25]。但是大肠杆菌中表达的蛋白质常常在细胞质中以包涵体的形式积累[25-27]。因此,不同的信号肽被广泛用于将外源蛋白输出到大肠杆菌胞质空间或者直接分泌到胞外培养基中[26, 28]。如来源于苏云金芽孢杆菌的BtLPMO10A利用载体自身信号肽实现了在大肠杆菌胞内的可溶性表达[29];来源于粘质沙雷氏菌的CBP21(LPMO10)通过不同的信号肽尝试,最终实现了在大肠杆菌的胞外表达[30]。但是目前利用大肠杆菌分泌表达真菌来源LPMO的相关研究较少。实现重组蛋白在大肠杆菌中的胞外表达,可以简化下游处理工艺,因此酶的胞外表达可以降低工业生产的成本,提高工业应用的经济效益。
为了进一步研究MtC1LPMO,该研究利用大肠杆菌将MtC1LPMO进行异源表达,实现MtC1LPMO在大肠杆菌中的分泌表达,提高了可溶性目标蛋白的总含量。最后通过优化培养条件和诱导参数来获得MtC1LPMO在大肠杆菌的最大化生产。
1 材料与方法
1.1 材料与试剂
大肠杆菌JM109、BL21(DE3),pET22b表达载体质粒保存于本实验室;高保真DNA聚合酶、T4 DNA连接酶和限制性内切酶NdeI、XhoI均购自大连宝生物公司;质粒小量提取试剂盒、胶回收试剂盒购自北京索来宝科技有限公司;2,6-二甲氧基苯酚购自上海源叶生物科技有限公司。其他未注明试剂均为分析纯,购自上海生工生物工程股份有限公司。
1.2 仪器与设备
ZXGP-B2080隔水恒温箱、ZHJH-C1106C超净工作台,上海智城分析仪器制造有限公司;Mastercycler nexus PCR仪,德国eppendorf公司;Multifuge X1R高速冷冻离心机、NanoDrop 2000蛋白核酸定量仪,美国ThermoFisher Scientific公司;可见分光光度计,上海精科实业有限公司。
1.3 pET22b-MtC1LPMO不同信号肽载体的构建
首先根据大肠杆菌宿主密码子偏好性进行密码子优化后合成MtC1LPMO基因,然后在C端添加(His)6标签。根据2个不同的信号肽构建2个不同的载体,1个是MtC1LPMO基因本身的信号肽(MLTTTFALLTAALGVSA),另1个是pET22b载体上的PelB信号肽(MKYLLPTAAAGLLLLAAQPAMA)。将MtC1LPMO成熟肽分别与2个不同的信号肽连接,然后插入到pET22b的 NdeI 和XhoI 2个限制性酶切位点之间。最终经测序正确的质粒命名为pET22b- MtC1LPMO。
1.4 信号肽对MtC1LPMO表达的影响
构建好的pET22b-MtC1LPMO质粒转入大肠杆菌Escherichia coli BL21 (DE3)细胞中,然后在LB (lysogeny broth)琼脂平板37 ℃培养过夜。挑取单克隆接入含氨苄霉素的5 mL的LB试管中培养12 h,然后以2%的接种量接种到含有氨苄霉素的LB培养基中进行发酵。当培养基OD600达到0.6时,添加异丙基-β-D-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside, IPTG)至终浓度为0.5 mmol/L,然后置于16 ℃发酵16 h或者30 ℃发酵4 h。发酵结束后,培养基和细胞在4 ℃通过8 000 r/min离心10 min进行分离,获得的细胞重悬于裂解缓冲液buffer A (20 mmol/L Tris-HCl pH 7.4, 20 mmol/L 咪唑, 500 mmol/L NaCl, 1 mmol/L DTT)。添加1%的溶菌酶后在冰上孵育30 min,然后通过超声破碎(功率250 W,开启2.5 s,关闭3 s,总时间15 min)将细胞进行裂解。上清和沉淀通过12 000 r/min离心30 min进行分离。测定不同组分中目标蛋白的含量及纯度,分析不同信号肽对MtC1LPMO表达的影响。
1.5 重组MtC1LPMO表达条件优化
1.5.1 优化诱导时间
将含有PelB信号肽pET22b-MtC1LPMO重组质粒的大肠杆菌表达宿主E. coli BL21 (DE3)在LB培养基发酵。为了获得种子培养基,挑取单菌落于5 mL的含有100 mg/L氨苄霉素的LB试管中220 r/min培养12 h。然后以2%的接种量分别接种到含有100 mg/L氨苄霉素的LB三角瓶培养基中于37 ℃进行发酵。当培养基OD600分别达到0.3、0.6、0.9、1.2和1.5时添加IPTG终浓度至0.5 mmol/L诱导重组MtC1LPMO表达。添加IPTG后将培养基置于30 ℃,200 r/min发酵4 h。发酵结束后,发酵液与细胞通过离心进行分离(8 000 r/min, 4 ℃, 10 min)。
1.5.2 优化IPTG诱导浓度
将上述的种子培养基以2%的接种量分别接种到含有100 mg/L氨苄霉素的LB三角瓶培养基中于37 ℃进行发酵。当培养基OD600达到0.9时,添加IPTG终浓度分别为0.05、0.1、0.25、0.5和1 mmol/L。然后将培养基置于30 ℃,200 r/min发酵4 h。发酵结束后,发酵液与细胞通过离心进行分离(8 000 r/min, 4 ℃, 10 min)。
1.5.3 优化诱导温度及表达时间
将上述的种子培养基以2%的接种量分别接种到含有100 mg/L氨苄霉素的LB三角瓶培养基中于37 ℃进行发酵。当培养基OD600达到0.9时,添加IPTG终浓度为0.5 mmol/L,然后将培养基分别置于16、30、37 ℃下分别发酵4、8和16 h。发酵结束后,发酵液与细胞通过离心进行分离(8 000 r/min, 4 ℃, 10 min)。
1.5.4 优化乙醇浓度
将上述的种子培养基以2%的接种量分别接种到含有100 mg/L氨苄霉素的LB三角瓶培养基中于37 ℃进行发酵。当培养基OD600达到0.9时,添加IPTG终浓度为0.5 mmol/L,同时分别添加乙醇至最终体积分数为0%、1%、2%、3%、4%。然后将培养基在30 ℃下发酵16 h。发酵结束后,发酵液与细胞通过离心进行分离(8 000 r/min, 4 ℃, 10 min)。重组蛋白表达条件优化实验重复了3次。
1.6 重组MtC1LPMO蛋白可溶性表达及纯化
将上述收集的细胞重悬于裂解缓冲液buffer A。添加1%的溶菌酶后在冰上孵育30 min,然后通过超声破碎将细胞进行裂解。上清和沉淀通过12 000 r/min离心30 min进行分离。
将收集的发酵液与细胞可溶性上清分别用0.22 μm滤膜(Millipore, Molsheim, France)过滤后,分别与Ni-NTA树脂(Qiagen, Hilden, Germany)进行结合。结合之前树脂用buffer A进行平衡。然后用清洗液buffer B (20 mmol/L Tris-HCl, pH 7.4, 500 mmol/L NaCl, 50 mmol/L咪唑, 1 mmol/L DTT)进行洗脱,最后用洗脱液buffer C (20 mmol/L Tris-HCl, pH 8.0, 500 mmol/L NaCl, 500 mmol/L 咪唑, 1 mmol/L DTT)洗脱带(His)6标签的重组蛋白MtC1LPMO。收集纯化的蛋白溶液,用Nanodrop ND-2000 spectrophotometer (Thermo Fisher Scientific) 在280 nm下测定蛋白质浓度,用12%的蛋白胶进行SDS-PAGE分析蛋白的纯度[31],洗脱的蛋白用pH 6.0,20 mmol/L的磷酸缓冲液进行透析。
1.7 重组MtC1LPMO酶活力分析
酶活力测定基于2,6-二甲氧基苯酚(2,6-dimethoxyphenol: 2,6-DMP)和H2O2作为共底物[32]。1 mL 反应体系包含860 μL 116 mmol/L pH 7.5磷酸缓冲液,100 μL 10 mmol/L 2,6-DMP溶液,20 μL 5 mmol/L H2O2原液和20 μL纯化的MtC1LPMO。酶活力在469 nm下测定反应300 s的前后吸光值的变化来计算,0.5 μM CuSO4作为空白对照。一个单位酶活力定义为反应条件下每分钟生成1 μmol氧化产物[ε469= 53 200 (M·cm)-1。
2 结果与分析
2.1 重组质粒的构建
由于不同的信号肽对蛋白的表达水平有不同的影响。因此,首先研究了2种不同信号肽对MtC1LPMO表达水平的影响。首先,利用SignalP 4.0[33]网页分析了MtC1LPMO自身的信号肽为MLTTTFALLTAALGVSA,将其作为第1种信号肽构建至质粒中。第2个信号肽是PelB信号肽,其是CBP21 (LPMO10)在大肠杆菌中表达量最高的信号肽,氨基酸序列为MKYLLPTAAAGLLLLAAQPAMA[30]。因此,选择PelB信号肽构建到质粒中来表达MtC1LPMO,以期待提高重组MtC1LPMO的分泌表达水平。因此,以上述2种信号肽分别构建表达载体,质粒图谱如图1-A和图1-B所示。
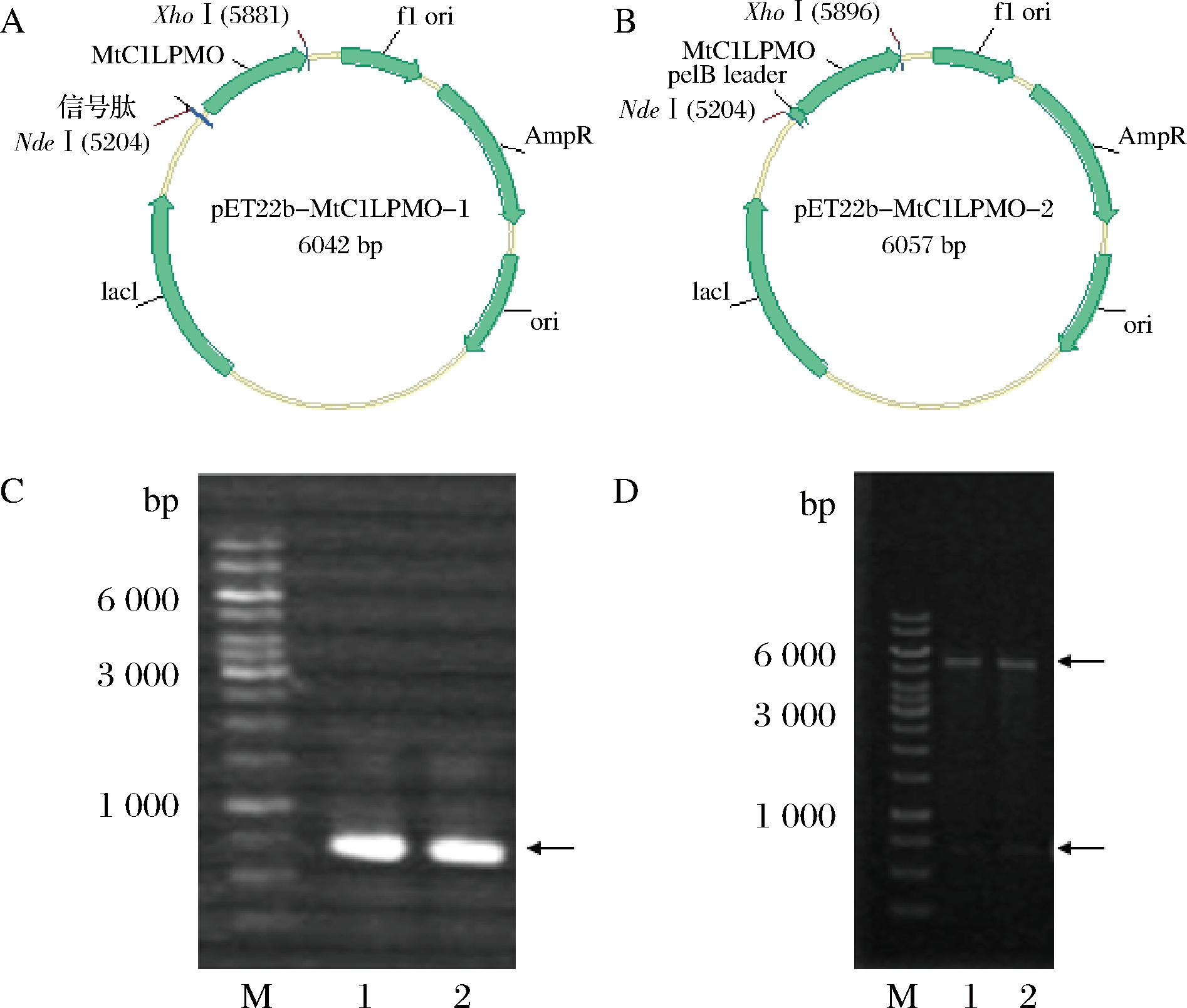
A-含有MtC1LPMO自身信号肽的表达载体; B-含有PelB信号肽的表达载体; C-PCR验证结果; D-NdeI 和XhoI双酶切验证结果;M-1kb Marker; Lane 1-质粒pET22b-MtC1LPMO-1; Lane 2-质粒pET22b-MtC1LPMO-2
图1 不同信号肽表达载体的构建及验证
Fig.1 Construction and identification of the plasmid including different signal peptides
将重组质粒分别用PCR扩增和双酶切鉴定,然后进行琼脂糖凝胶电泳检测,结果如图1-C和图1-D所示,2个质粒的PCR扩增条带均为单一条带,且NdeI 和XhoI双酶切也出现正确的目标条带。测序结果也进一步证实重组载体pET22b-MtC1LPMO构建成功。
2.2 不同信号肽对MtC1LPMO表达的影响
为了分析不同的信号肽及发酵温度对重组MtC1LPMO表达水平的影响,将含有不同信号肽重组质粒的表达宿主分别在16 ℃发酵16 h或30 ℃发酵4 h表达目标蛋白。结果如图2所示,SDS-PAGE结果显示目标蛋白主要存在于包涵体中(图2,泳道6),发酵液和细胞中的可溶性MtC1LPMO比较少。
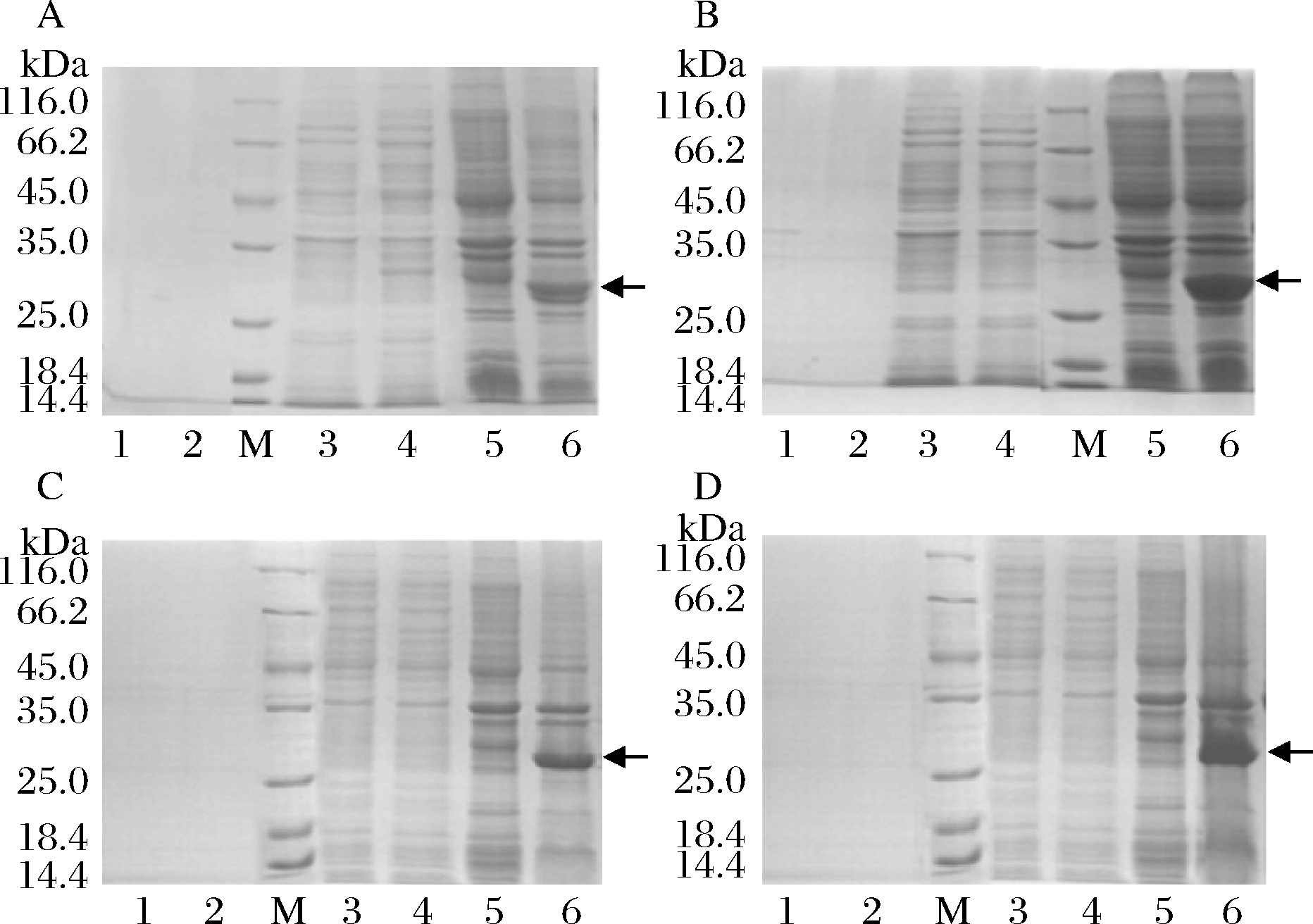
A, B-MtC1LPMO自身信号肽; C, D-PelB信号肽; A, C-16 ℃发酵; B, D-30 ℃发酵;M-标准蛋白; 条带1, 3, 5-BL21原始菌发酵液,菌体上清,菌体沉淀; 条带2, 4, 6-pET22b-MtC1LPMOBL21发酵液,菌体上清液,菌体沉淀
图2 不同信号肽与不同发酵温度对MtC1LPMO表达结果的影响
Fig.2 The effects of signal peptides and temperatures on the production of MtC1LPMO
为了进一步获得有活性的可溶性MtC1LPMO,将培养基发酵液与细胞可溶性上清中的目标蛋白利用Ni树脂进行纯化,然后利用SDS-PAGE进行蛋白纯度检测,结果如图3所示。MtC1LPMO自身信号肽表达的蛋白只存在于细胞可溶性上清中(泳道2和4),而PelB信号肽表达的蛋白存在于发酵液与细胞可溶性上清中(泳道5~8),而且由于发酵液中杂蛋白比较少,所以纯化得到的目标蛋白纯度更高。测定不同体系的蛋白浓度,30 ℃下PelB信号肽表达的发酵液中(图3,泳道7)的目标蛋白含量最高。此外,目标蛋白分泌到细胞外,工业应用中其下游处理则更为简单,可以降低工业生产成本。因此在这种情况下,需要进一步优化E. coli BL21(pET22b-PelB-MtC1LPMO)的发酵条件,来获得重组MtC1LPMO更高的总产量及细胞外比率。

条带1~4-MtC1LPMO自身信号肽; 条带5~8-PelB信号肽;M-标准蛋白;条带1, 5-16 ℃发酵液; 条带2, 6~16 ℃菌体上清液;条带3,7~30 ℃发酵液;条带4,8~30 ℃菌体上清液
图3 不同发酵液与菌体上清的MtC1LPMO的纯化结果
Fig.3 The purified MtC1LPMO of different culture supernatant or soluble fractions of cell
2.3 发酵条件的优化
2.3.1 优化诱导时间
添加诱导剂的时间一般在菌体生长的对数前期或中期[34],为了选择合适的诱导时间点,我们分别在培养基OD600分别达到0.3、0.6、0.9、1.2和 1.5时添加IPTG终浓度至0.5 mmol/L诱导重组蛋白MtC1LPMO的表达。然后将培养基在30 ℃,200 r/min发酵4 h。发酵结束后,测定培养基的菌体浓度(图4-A),然后分别纯化发酵液与细胞可溶性上清中的目标蛋白,测定蛋白浓度(图4-B)。由图4-A可以看出,随着诱导剂添加时的培养基OD600的增高,发酵结束后培养基OD600也是逐步增高。但是图4-B可以看出,重组蛋白MtC1LPMO的总产量在OD600为0.9时达到最高,为7.46 mg/L,细胞内外的可溶性MtC1LPMO含量都得到了一定的提高。
2.3.2 优化IPTG浓度
由于pET22b的启动子是诱导型的T7启动子,因此IPTG诱导浓度也是影响重组蛋白产量的重要原因。为了得到合适的IPTG诱导浓度,我们在培养基OD600达到0.9时,添加IPTG终浓度分别为0.05、0.1、0.25、0.5 和 1 mmol/L。然后将培养基置于30 ℃,200 r/min发酵4 h。然后测定培养基的菌体浓度(图5-A),然后分别纯化发酵液与细胞可溶性上清中的目标蛋白,测定蛋白浓度(图5-B)。由图5-A可以看出,随着IPTG浓度的增高,发酵结束的培养基OD600逐渐降低,这是由于IPTG对细胞有一定的毒性[35-36]。由图5-B可以看出,总可溶性蛋白产量在IPTG为0.5 mmol/L时达到最高,相比于其他IPTG浓度,细胞外比率也明显提高,为51.93%。IPTG为1 mmol/L时总的可溶性蛋白产量下降可能是由于菌体的减少,或者高浓度诱导剂条件下形成更多错误折叠的包涵体。
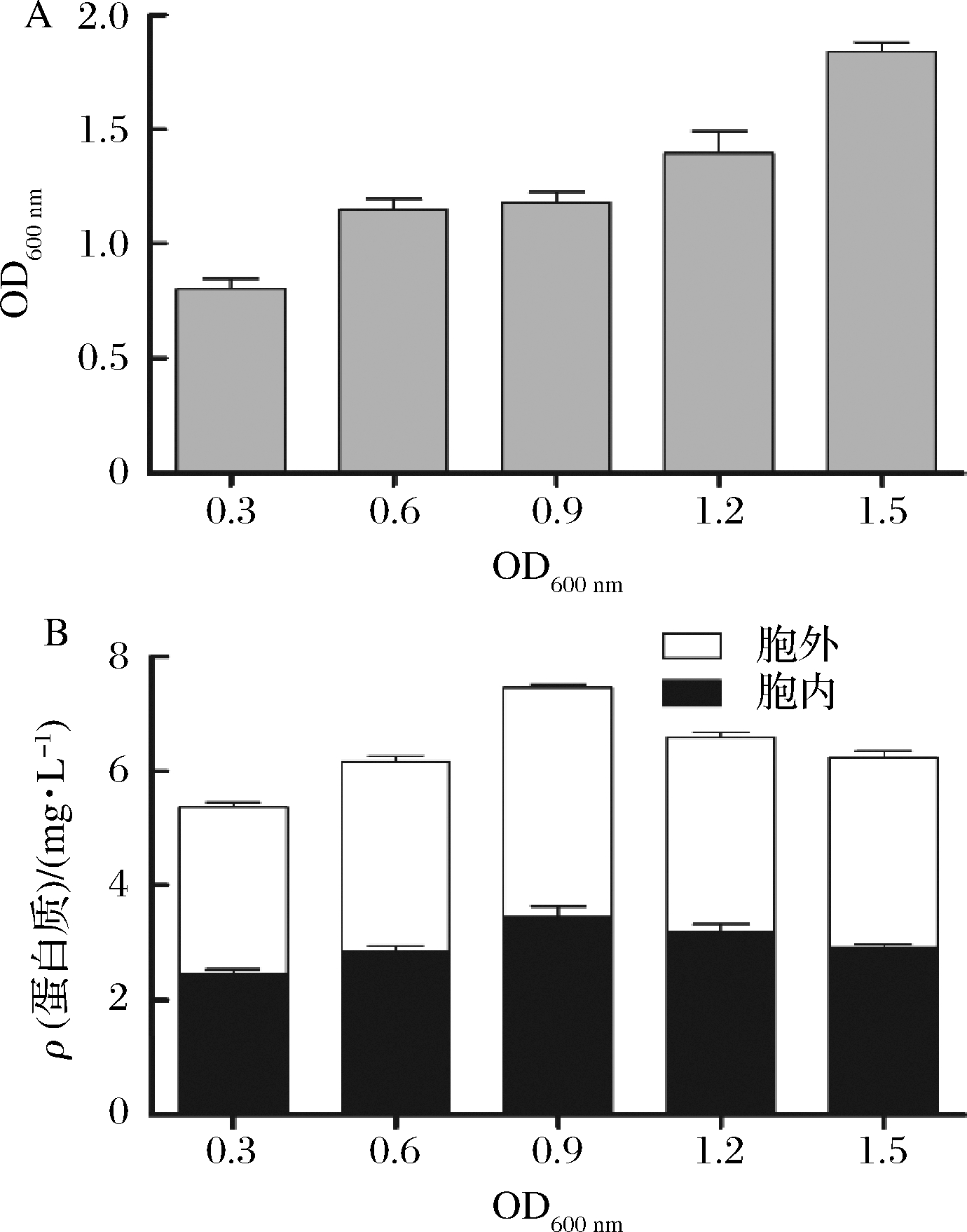
A-发酵结束的菌体浓度; B-可溶性蛋白产量
图4 不同OD600下IPTG诱导对菌体生长及MtC1LPMO蛋白表达的影响
Fig.4 Effect of pre-induction growth on the final growth rate and MtC1LPMO expression
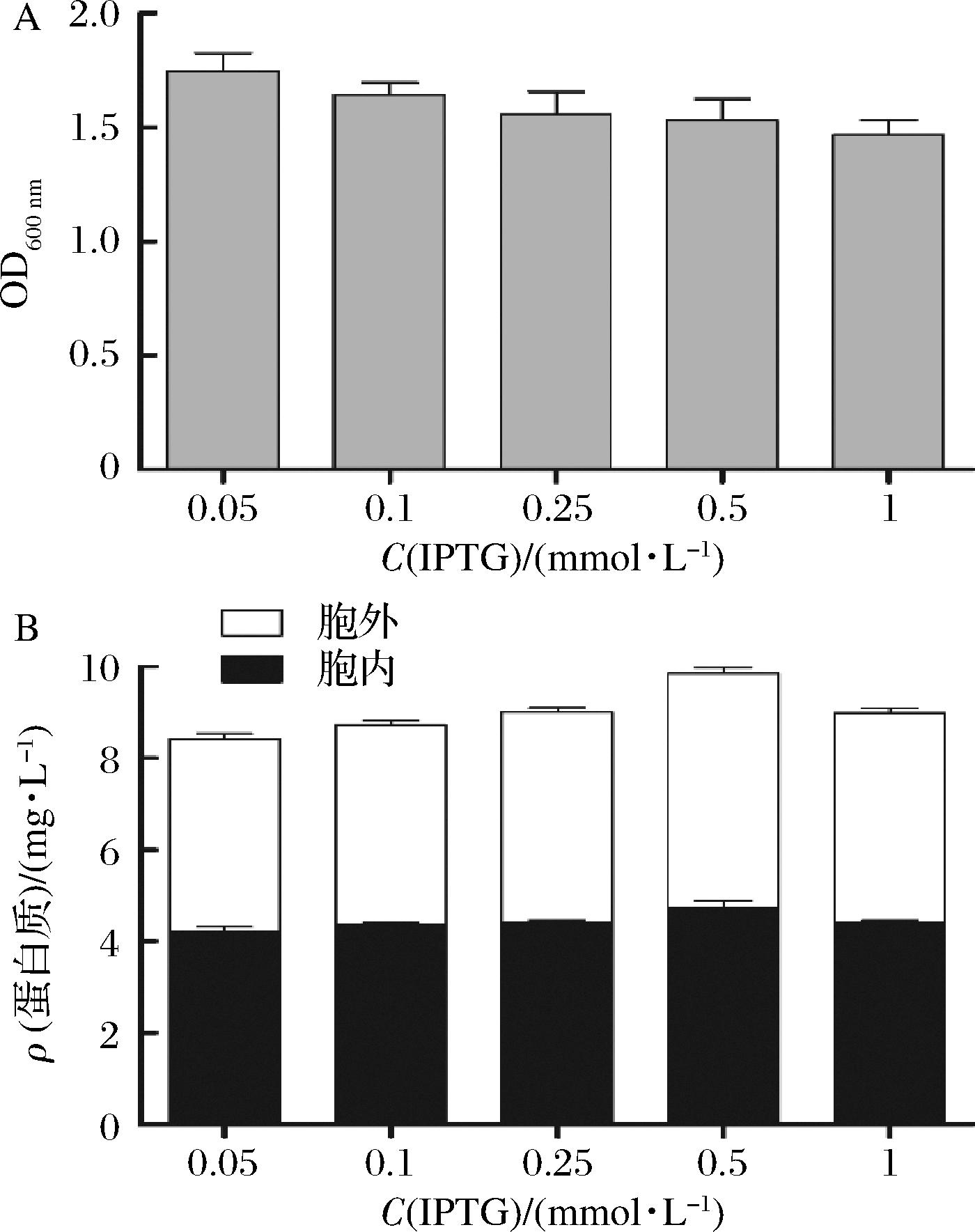
A-发酵结束的菌体浓度; B-可溶性蛋白产量
图5 不同IPTG浓度诱导对菌体生长及蛋白表达的影响
Fig.5 Effect of various IPTG on the final growth rate andMtC1LPMO expression
2.3.3 优化诱导温度及表达时间
为了确定合适的诱导温度及表达重组蛋白的时间,当培养基在OD600达到0.9时,添加IPTG终浓度为0.5 mmol/L后,分别置于16、30、37 ℃下分别发酵4、8和16 h。发酵结束后分别测定培养基的菌体浓度(图6-A),然后分别纯化发酵液与细胞可溶性上清中的目标蛋白,测定蛋白浓度(图6-B)。由图6-A可以看出,同一个的温度下,菌体浓度和蛋白产量都随着时间的增长而增高。但是不同的温度差异明显,其中30 ℃发酵16 h的菌体浓度和蛋白产量是所有优化方案中最高的,总可溶性蛋白产量为10.49 mg/L,其中分泌至细胞外的蛋白产量为6.51 mg/L,细胞外比率为62.00%。

A-发酵结束的菌体浓度; B-可溶性蛋白产量
图6 不同诱导温度及表达时间对菌体生长及蛋白表达的影响
Fig.6 Comparison of the cell growth and MtC1LPMO production at various temperature and post-induction incubation times
2.3.4 氧化应激
利用乙醇氧化应激是以往研究中用于重组蛋白过表达的一种策略[37]。在本研究中,研究了不同体积分数的乙醇对可溶性重组蛋白产量的影响。在添加IPTG的同时分别在不同的培养基添加乙醇至终体积分数为0%、1%、2%、3%、4%。然后在上述最优条件下发酵,发酵结束后的菌体浓度随着乙醇体积分数增高而降低(图7-A),总可溶性蛋白含量在添加乙醇终体积分数为2%时达到最高(图7-B),为12.65 mg/L,其中细胞外蛋白含量也达到了最高,为8.51 mg/L,细胞外比率为67.30%。当乙醇添加更高时蛋白产量的下降可能是由于高浓度乙醇抑制了菌体的生长,导致重组酶的产率下降。
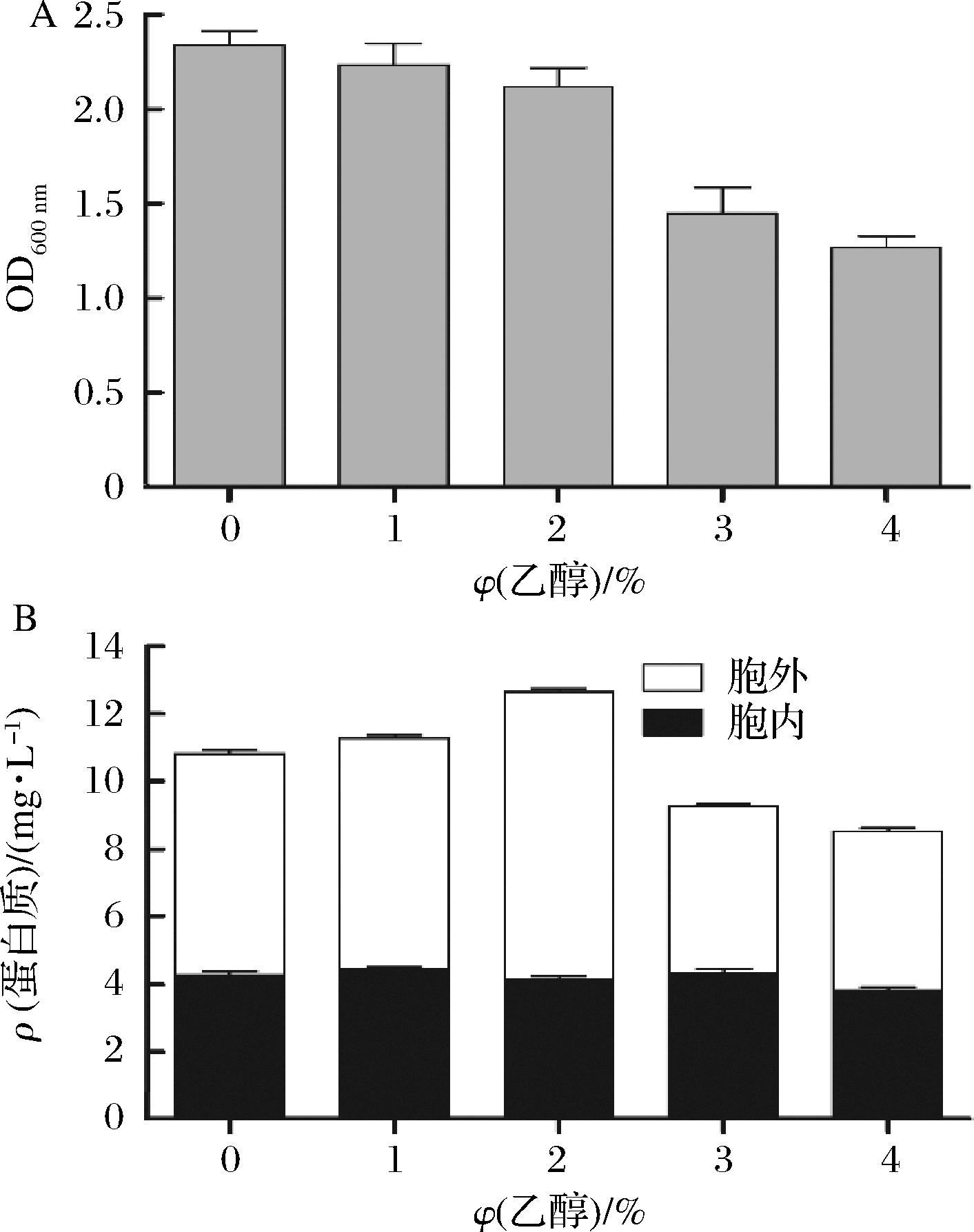
A-发酵结束的菌体浓度; B-可溶性蛋白产量
图7 不同体积分数的乙醇对菌体生长及蛋白表达的影响
Fig.7 Effect of oxidative stress on the cell growth andMtC1LPMO production
2.4 MtC1LPMO酶活力分析
最终优化方案获得的重组MtC1LPMO蛋白,用20 mmol/L, pH 6.0的磷酸缓冲液进行透析后冻干,然后在文献描述的标准条件下测定酶活力[32],即30 ℃,pH7.5条件下以2,6-DMP为底物测定其酶活力,经计算比活力为10.3 U/g。来源于粗糙脉孢菌(Neurospora crassa) 的NcLPMO9F在标准条件下以2,6-DMP为底物的比活力为2.2 U/g[32]。因此,在此标准反应条件下,MtC1LPMO的比活力是NcLPMO9F的4.68倍。
3 结论
本研究异源表达了来源于真菌嗜热毁丝霉的MtC1LPMO,首先构建了不同信号肽的载体,确定了PelB信号肽可以分泌表达MtC1LPMO。为了获得更多的胞外酶,对表达条件进行了优化,确定了E. coli BL21 (DE3)/ pET22b-PelB-MtC1LPMO菌株在37 ℃培养基OD600为0.9时添加终浓度为0.5 mmol/L的IPTG和终体积分数为2%的乙醇,然后30 ℃诱导16 h,重组蛋白的可溶性表达量最高,为12.65 mg/L,为优化前的2.05倍。且细胞外比率也达到最高,为67.30%,其比活力为10.3 U/g。
[1] HEMSWORTH G R,JOHNSTON E M,DAVIES G J,et al. Lytic Polysaccharide Monooxygenases in Biomass Conversion[J]. Trends Biotechnol, 2015, 33(12): 747-761.
[2] LIU Z L. Molecular mechanisms of yeast tolerance and in situ detoxification of lignocellulose hydrolysates[J]. Appl Microbiol Biotechnol, 2011, 90(3): 809-825.
[3] GRONENBERG L S,MARCHESCHI R J,LIAO J C. Next generation biofuel engineering in prokaryotes[J]. Curr Opin Chem Biol, 2013, 17(3): 462-471.
[4] PERCIVAL ZHANG Y H,HIMMEL M E,MIELENZ J R. Outlook for cellulase improvement: screening and selection strategies[J]. Biotechnol Adv, 2006, 24(5): 452-481.
[5] VIIKARI L,ALAPURANEN M,PURANEN T,et al. Thermostable Enzymes in Lignocellulose Hydrolysis[J]. Advances in Biochemical Engineering/biotechnology, 2007, 108(108): 121.
[6] MERINO S T,CHERRY J. Progress and challenges in enzyme development for biomass utilization[J]. Adv Biochem Eng Biotechnol, 2007, 108: 95-120.
[7] KUMAR R,SINGH S,SINGH O V. Bioconversion of lignocellulosic biomass: biochemical and molecular perspectives[J]. J Ind Microbiol Biotechnol, 2008, 35(5): 377-391.
[8] JARVIS M. Chemistry - Cellulose stacks up[J]. Nature, 2003, 426(6 967): 611-612.
[9] VAAJE-KOLSTAD G,WESTERENG B,HORN S J,et al. An oxidative enzyme boosting the enzymatic conversion of recalcitrant polysaccharides[J]. Science, 2010, 330(6 001): 219-222.
[10] HARRIS P V,WELNER D,MCFARLAND K C,et al. Stimulation of lignocellulosic biomass hydrolysis by proteins of glycoside hydrolase family 61: structure and function of a large, enigmatic family[J]. Biochemistry, 2010, 49(15): 3 305-3 316.
[11] LI X,BEESON W T T,PHILLIPS C M,et al. Structural basis for substrate targeting and catalysis by fungal polysaccharide monooxygenases[J]. Structure, 2012, 20(6): 1 051-1 061.
[12] FORSBERG Z,VAAJE-KOLSTAD G,WESTERENG B,et al. Cleavage of cellulose by a CBM33 protein[J]. Protein Sci, 2011, 20(9): 1 479-1 483.
[13] HORN S J,VAAJE-KOLSTAD G,WESTERENG B,et al. Novel enzymes for the degradation of cellulose[J]. Biotechnology for Biofuels, 2012, 5(1): 45.
[14] ANTHONY LEVASSEUR E D, VINCENT LOMBARD, PEDRO M COUTINHO, BERNARD HENRISSAT. Expansion of the enzymatic repertoire of the CAZy database to integrate auxiliary redox enzymes[J]. Biotechnology for Biofuels, 2013, 6: 41.
[15] HEMSWORTH G R,HENRISSAT B,DAVIES G J,et al. Discovery and characterization of a new family of lytic polysaccharide monooxygenases[J]. Nature Chemical Biology, 2013, 10(2): 122-126.
[16] LO L L,SIMMONS T J,POULSEN J C,et al. Structure and boosting activity of a starch-degrading lytic polysaccharide monooxygenase[J]. Nat Commun, 2015, 6: 5 961.
[17] VOSHOL G P,VIJGENBOOM E,PUNT P J. The discovery of novel LPMO families with a new Hidden Markov model[J]. BMC Res Notes, 2017, 10(1): 105.
[18] SABBADIN F,HEMSWORTH G R,CIANO L,et al. An ancient family of lytic polysaccharide monooxygenases with roles in arthropod development and biomass digestion[J]. Nat Commun, 2018, 9(1): 756.
[19] FILIATRAULT-CHASTEL C,NAVARRO D,HAON M,et al. AA16, a new lytic polysaccharide monooxygenase family identified in fungal secretomes[J]. Biotechnology for Biofuels, 2019, 12: 55.
[20] MONCLARO A V,FILHO E X F. Fungal lytic polysaccharide monooxygenases from family AA9: Recent developments and application in lignocelullose breakdown[J]. Int J Biol Macromol, 2017, 102: 771-778.
[21] FROMMHAGEN M,SFORZA S,WESTPHAL A H,et al. Discovery of the combined oxidative cleavage of plant xylan and cellulose by a new fungal polysaccharide monooxygenase[J]. Biotechnol Biofuels, 2015, 8(1): 101.
[22] VOGEL J. Unique aspects of the grass cell wall[J]. Curr Opin Plant Biol, 2008, 11(3): 301-307.
[23] TERPE K. Overview of bacterial expression systems for heterologous protein production: from molecular and biochemical fundamentals to commercial systems[J]. Applied Microbiology and Biotechnology, 2006, 72(2): 211-222.
[24] CHEN R. Bacterial expression systems for recombinant protein production: E. coli and beyond[J]. Biotechnology Advances, 2012, 30(5): 1 102-1 107.
[25] BANEYX F,MUJACIC M. Recombinant protein folding and misfolding in Escherichia coli[J]. Nat Biotechnol, 2004, 22(11): 1 399-1 408.
[26] CHOI J H,LEE S Y. Secretory and extracellular production of recombinant proteins using Escherichia coli[J]. Applied Microbiology and Biotechnology, 2004, 64(5): 625-635.
[27] VENTURA S,VILLAVERDE A. Protein quality in bacterial inclusion bodies[J]. Trends in Biotechnology, 2006, 24(4): 179-185.
[28] LOW K O,MUHAMMAD MAHADI N,MD ILLIAS R. Optimisation of signal peptide for recombinant protein secretion in bacterial hosts[J]. Appl Microbiol Biotechnol, 2013, 97(9): 3 811-3 826.
[29] ZHANG H,ZHAO Y,CAO H,et al. Expression and characterization of a lytic polysaccharide monooxygenase from Bacillus thuringiensis[J]. Int J Biol Macromol, 2015, 79: 72-75.
[30] YANG Y,LI J,LIU X,et al. Improving extracellular production of Serratia marcescens lytic polysaccharide monooxygenase CBP21 and Aeromonas veronii B565 chitinase Chi92 in Escherichia coli and their synergism[J]. AMB Express, 2017, 7(1): 170.
[31] LAEMMLI U K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4[J]. Nature, 1970, 227(5 259): 680-685.
[32] BRESLMAYR E,HANZEK M,HANRAHAN A,et al. A fast and sensitive activity assay for lytic polysaccharide monooxygenase[J]. Biotechnol. Biofuels, 2018, 11: 79.
[33] PETERSEN T N,BRUNAK S,VON HEIJNE G,et al. SignalP 4.0: discriminating signal peptides from transmembrane regions[J]. Nat Methods, 2011, 8(10): 785-786.
[34] MAKRIDES S C. Strategies for achieving high-level expression of genes in Escherichia coli[J]. Microbiol Rev, 1996, 60(3): 512-538.
[35] DVORAK P,CHRAST L,NIKEL P I,et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway[J]. Microb Cell Fact, 2015, 14: 201.
[36] GLICK B R. Metabolic load and heterologous gene expression[J]. Biotechnology Advances, 1995, 13(2): 247-261.
[37] CHHETRI G,KALITA P,TRIPATHI T. An efficient protocol to enhance recombinant protein expression using ethanol in Escherichia coli[J]. MethodsX, 2015, 2: 385-391.