基于转录组学的酿酒酵母耐酸机制解析
田甜甜1,2,3,孙军勇1,2,3,蔡国林1,2,3,杨华1,2,3,吴殿辉1,2,3*,陆健1,2,3*
1(工业生物技术教育部重点实验室(江南大学),江苏 无锡,214122)2(粮食发酵工艺与技术国家工程实验室(江南大学),江苏 无锡,214122)3(江南大学 生物工程学院,江苏 无锡,214122)
摘 要 酿酒酵母的耐酸特性在果酒生产中至关重要,但目前其应对酸胁迫的生物学机制仍不清楚。该研究以2株酿酒酵母(ET008-c54和ET008)为研究对象,分别提取总RNA后进行转录组测序分析,并考察不同pH条件下2菌株的细胞活力及利用不同的分析方法对ET008-c54发酵性能参数(菌体浓度、葡萄糖含量、生物量、乙酸含量、乙醇含量、甘油含量、麦角甾醇含量和H+-ATPase活性)进行测定。转录组学结果表明,688个差异表达基因中,其中364个基因转录水平上调,324个基因转录水平下调。差异表达基因的GO富集和KEGG通路富集表明,这些基因主要涉及细胞膜的组成及生理功能、麦角甾醇合成、亚铁吸收等多条代谢途径。通过对差异表达基因的进一步分析,最终确定了8个与耐酸性有关的重要基因。另外,ET008-c54在细胞活力、生长速率和代谢产物等方面表现出良好的发酵性能。ET008-c54具有很强的耐酸性,为高酸度水果酒的酿造提供了广阔的前景。这些发现为酿酒酵母的的遗传改良提供了方向,同时为果酒的高效发酵提供了重要的理论依据。
关键词 酿酒酵母;转录组学;耐酸性;细胞活性;代谢产物
DOI:10.13995/j.cnki.11-1802/ts.023125
引用格式:田甜甜,孙军勇,蔡国林,等.基于转录组学的酿酒酵母耐酸机制解析[J].食品与发酵工业,2020,46(6):1-7.TIAN Tiantian, SUN Junyong, CAI Guolin, et al. Transcriptomic profiling reveals the acid-tolerance mechanism of Saccharomyces cerevisiae[J].Food and Fermentation Industries,2020,46(6):1-7.
第一作者:博士研究生(吴殿辉助理研究员和陆健教授为共同通讯作者,E-mail: wudianhui@jiangnan.edu.cn,jlu@jiangnan.edu.cn)
基金项目:国家自然科学基金(31701588);江苏省自然科学基金(BK20170178);中央高校基本科研业务费专项资金(JUSRP11965);江苏高校优势学科建设工程资助项目;高等学校学科创新引智计划(111计划)资助项目(111-2-06);江苏省现代工业发酵协同创新中心资助项目
收稿日期:2019-12-17,改回日期:2020-01-10
Transcriptomic profiling reveals the acid-tolerance mechanism of Saccharomyces cerevisiae
TIAN Tiantian1,2,3, SUN Junyong1,2,3, CAI Guolin1,2,3, YANG Hua1,2,3,WU Dianhui1,2,3*, LU Jian1,2,3*
1 (Key Laboratory of Industrial Biotechnology, Ministry of Education (Jiangnan University), Wuxi 214122, China)2 (National Engineering Laboratory for Cereal Fermentation Technology (Jiangnan University), Wuxi 214122, China)3 (School of Biotechnology, Jiangnan University, Wuxi 214122, China)
ABSTRACT Acid tolerance of Saccharomyces cerevisiae is critical to many industrial processes including fruit wine. However, the biological mechanism that causes yeast to adapt to low pH stress is still unclear and remains to be explored. The total RNA was extracted and transcriptomic analysis was performed to investigate the acid-tolerance mechanism of two wine yeasts (ET008-c54 and ET008) at low pH. The cell activity of these two strains were compared at different pH. Different analytical methods were used to measure the fermentation performance parameters of ET008-c54, including OD600, glucose, biomass, acetic acid, ethanol, glycerol, ergosterol and H+-ATPase activity. Based on the transcriptomic results, 688 differentially expressed genes (DEGs) comprising 364 up-regulated genes and 324 down-regulated genes were identified. Gene Ontology (GO) enrichment and Kyoto Encyclopedia of Genes and Genomes (KEGG) pathway enrichment of DEGs indicated that these genes were mainly involved in important biological metabolisms, such as cell membrane composition and physiological functions, ergosterol synthesis and ferrous iron uptake. Eight genes in ET008-c54 were identified as the important genes for acid tolerance. Furthermore, ET008-c54 exhibited outstanding performances in cell activity, growth rate, metabolites, ergosterol and H+-ATPase activity. These results could potentially offer better targets for genetic improvement of wine yeast and efficient fermentation process.
Key words Saccharomyces cerevisiae; transcriptomic; acid-tolerance; cell activity; metabolite
酿酒酵母(Saccharomyces cerevisiae)作为传统生产果酒的主要菌种,在群体基因组水平经历了大量的系统进化、种群分化和适应性驯化等过程,具有清晰的遗传背景[1]。在葡萄酒工业生产中,S. cerevisiae最适pH值通常在3.0~3.6[2],与葡萄相比,青梅和山楂等水果的有机酸含量偏高(15~20 g/L-1),导致发酵pH偏低(2.7~2.9),而目前生产高酸度果酒(青梅酒、山楂酒等)使用的酵母仍是葡萄酒酿酒酵母,最终影响了此类果酒的发酵周期和酒体风味。因此,选育耐酸性能良好的S. cerevisiae成为酿造高酸度果酒育种的新方向。
随着酿酒工业需求的增加和菌种选育技术的提高,S. cerevisiae的耐酸机制引起了人们的广泛关注[3]。目前,关于S. cerevisiae耐酸机制的研究主要集中在燃料乙醇和/或有机酸的工业生产上[4],而关于果酒酿造中与耐酸性相关的研究仍然较少。于玲对筛选出耐酸石榴酒酵母的发酵性能进行初探,但未对耐酸性能进行深入研究[5]。本实验室前期从青梅酒发酵液中分离出1株S. cerevisiae ET008,在此基础上通过等离子诱变技术(atmospheric and room temperature plasma,ARTP)、高通量筛选(high-throughput screening,HTS)和适应性实验室进化(adaptive laboratory evolution,ALE)获得1株耐酸性较好的S. cerevisiae ET008-c54,该菌种发酵生产的青梅酒具有典型性风味。因此,阐明ET008-c54在酸性环境中的生物学机制为提高高酸度果酒品质奠定基础。随着高通量测序技术的发展,基因组学、转录组学等技术为研究S. cerevisiae细胞生理代谢提供了重要手段,使人们对S. cerevisiae的基因背景有了更深入的了解,其中转录组对于解释基因组的功能元件、揭示细胞和组织的分子成分以及对发育和进化的理解具有重要作用[6]。
本研究基于转录组分析S. cerevisiae ET008-c54和ET008的差异表达基因,同时结合生物信息学手段对差异基因进行注释,最终筛选出与耐酸相关的关键基因并进行验证。在此基础上对ET008-c54的发酵特性进行进一步分析,从而为S. cerevisiae的耐酸调控机制的研究提供理论基础,为后续高酸度果酒的生产提供科学依据。
1 材料与方法
1.1 菌株和培养基
S. cerevisiae ET008,从青梅酒发酵过程中分离并保存于本实验室;ET008-c54,是ET008经ARTP、HTS和ALE获得的耐酸菌株。
基础培养基(g/L):葡萄糖20.0、酵母提取物10.0、蛋白胨20.0,用HCl溶液分别将pH调节至3.0和2.5。
发酵培养基(g/L):葡萄糖200.0、酵母提取物12.0、(NH4)2SO4 3.0、KH2PO4 2.0、MgSO4·7H2O 1.0、CaCl2·2H2O 0.1、NaCl 0.1,用HCl溶液将pH调节至2.5[7]。
1.2 试剂和仪器
3,5-二硝基水杨酸、Tris-HCl、EDTA、二硫苏糖醇等,国药集团化学试剂有限公司;标准品乙酸、乙醇、甘油和麦角甾醇,Sigma公司。
RNA提取试剂盒(FastPure Cell/Tissue Total RNA Isolation Kit RC101)、反转录试剂盒(Hiscript Ⅱ Q RT SuperMix for qpcr)和AceQ RT-PCR试剂盒(Universial SYBR qPCR Master Mix),南京诺唯赞生物科技有限公司;H+-ATPase活性测定试剂盒,Sigma公司。
Biostat Q发酵罐,德国Sartorius公司;实时荧光定量PCR仪,德国 Eppendorf 公司;Shimadzu UV-1800分光光度计,德国Shimadzu Europe GmbH公司;6890 N气相色谱,美国Agilent公司。
1.3 总RNA提取及测序
在pH 2.5的条件下,培养S.cerevisiae ET008-c54和ET008生长至对数中期,分别取10 mL菌液于5 000×g离心5 min收集菌体,用无菌水洗涤3次,在液氮中速冻10 min,于-80 ℃保存备用。总RNA提取方法,按照试剂盒说明书进行。利用Nanodrop检测RNA浓度及琼脂糖凝胶检测RNA完整性以及基因组污染情况。检测合格的RNA用于后续Illumina测序仪测序。转录组测序分析,由上海欧易生物医学科技有限公司完成。
将高通量测序获得的每个样本的转录组文库,经过CASAVA碱基识别分析转化为原始测序序列(raw data),再经过去除接头和过滤低质量数据处理,所获得的RNA序列(clean data)作为本研究的基本数据。利用hisat2将过滤后的RNA序列与S. cerevisiae模式菌株S288的参考基因组进行序列比对,获取在参考基因组或基因上的位置信息,以及ET008-c54和ET008特有的序列特征信息。已知的参考基因序列以及注释文件做为数据库,采取序列相似性比对的方法鉴定出各蛋白编码基因在各样本中的表达丰度。使用htseq-count软件获取每个样本中比对到蛋白编码基因上的reads数,cufflinks软件来计算蛋白编码基因的表达量FPKM值。差异表达基因(differentially expressed genes,DEGs)使用DESeq R包功能的EstimateSizeFactors和nbinomTest进行鉴定。将差异表达倍数(fold change,FC)≥2或≤0.5和P<0.05设置为显著差异表达的阈值。
1.4 基因功能注释分析
利用Blast2go在线数据库对ET008-c54和ET008的差异基因分别进行GO(Gene Ontology)数据库(http://www.gEneontology.org/)和KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库(http://www.genome.jp/kegg/)注释分析。利用DESeq2方法对定量结果进行差异表达分析并获得差异表达基因列表,再通过GOSeq方法采取超几何分布样本抽取方法进行差异表达的基因功能的GO富集和KEGG代谢通路富集。
1.5 RT-qPCR验证
利用上述1.3获得的RNA样品及PrimeScript RT试剂和SYBR Premix Ex Taq试剂盒在定量实时PCR系统上进行PCR反应,以测定8个靶基因的mRNA水平。以ACT1基因作为内标,使用2-ΔΔCt算法计算相对基因表达[8]。表1列出了所用的引物。
1.6 不同pH条件下2菌株的生长情况
在pH为3.0和2.5基础培养基条件下,分别对ET008-c54和ET008进行三角瓶培养试验,每间隔10 h 取样测定其OD600并利用美兰染色活细胞计数法计数[9],直至培养结束。
1.7 发酵性能验证及分析方法
分别将ET008-c54和ET008利用配有连续数据采集功能的5 L Biostat Q发酵罐进行发酵试验。初始OD600为0.2,发酵温度25 ℃,搅拌速度为120 r/min,并通过自动添加2 mol/L HCl溶液调节pH 2.5。每间隔24 h取样,直至发酵结束。
菌体浓度的测定:使用分光光度计测定菌液的OD600。
表1 本研究中用到的引物
Table 1 Primers used in this study

引物序列(5′-3′)ERG5F: GGTTTGGGACCAAGTTGCATATCR: CAGCAATCTTTCTGACTTGATCTTCGGFRE1F: GCCATGTACTACCATTGTCACACCCR: CCGTAAACATTCGACAAAGTGCGGOPT2F: CCAGCCCGTGCACTATGGCCAACAGTCATGCCAACR: GGGTCATCATAATCGTCGAGCGCCCTGTAATCGCINO1F: TCGACGTACAAGGACAACGAR: GGCCACTAAAGTGGAGCCATGCN1F: GGGGATCCATGGCCGTTAAGAGCCR: GGCTCGAGGCTGAGGATCATGTCCFIT2F: GACACCGCTGACCCTATCATR: GATGATTCGACGGCTTGAGTFET3F: ACGGTGTGAATTACGCCTTCR: TTGGAAAGCGTGACCATGTAIZH4F: TGG GCC TTC GTCAATAGCR: CCAG CTGAGTCTAAGAACT1F: GCTCCAATGAACCCTAAATCR: GGAAGAGTACAAGGACAAAACG
葡萄糖含量的测定[10]:参照3,5-二硝基水杨酸法(3,5-dinitrosalicylic acid,DNS)进行。
生物量的测定:将5 mL细胞悬浮液转移至预先称重的10 mL离心管中,以5 000×g离心10 min,然后用去离子水洗涤2次并离心,于105 ℃下干燥至恒重。
乙酸、乙醇、甘油含量的检测:样品离心后经0.45 μm PTFE过滤器过滤,并保存于-20 ℃直至分析。使用气相色谱仪通过外标法进行测定。升温程序为进样器和检测器的温度均为260 ℃;柱温在40 ℃保持8 min,然后以10 ℃/min升至260℃并保持5 min;载体为He,流速1.2 mL/min。与标准品的保留时间进行比较以确定该物质,并通过标准曲线进行定量。
麦角甾醇含量的测定:菌体经无菌水洗涤后,在液氮中冷冻并保存在-80 ℃直至分析。将1 g干细胞重悬于100 mL 25%KOH中,加入5 mL甲醇,然后在95 ℃回流2.5 h进行皂化反应。冷却至室温,用70 mL乙醚萃取,并振摇15 min。将混合物转移至分配器装置静置2 h后分离,280 nm处测定上清液在的吸光度,并将其与麦角固醇的标准曲线进行比较。
H+-ATPase活性的测定:通过超声法裂解处于对数期的细胞用5 mL溶液(0.1 mol/L Tris-HCl、0.33 mol/L蔗糖、5 mmol/L EDTA和2 mmol/L二硫苏糖醇)稀释并离心。将沉淀重悬于100 μL溶液(10 mmol/L Tris-HCl、20%甘油、0.1 mmol/L EDTA和0.1 mmol/L二硫苏糖醇)中,并于-80 ℃保存。根据H+-ATPase活性测定试剂盒测定H+-ATPase的活性。
1.8 数据分析
采用OriginPro 7.5进行单因素方差分析(one-way analysis of variance,ANOVA)及Tukey检验来确定数据的显著差异(P<0.05),并利用GraphPad Prism 7.04(GraphPad Software Inc,San Diego,CA)进行绘图。所有实验均重复3次,结果均以x±SD表示。
2 结果与分析
2.1 测序数据的统计与质控报告
测序数据质量的水平直接决定后续测序结果的准确性。采用Illumina HiSeqTM2500高通量测序技术对ET008-c54和ET008进行转录组测序,样品各项检测参数均达到质量要求,结果如表2所示。经质控过滤后,ET008-c54和ET008后分别得到49.48 Mb和 49.27 Mb原始数据,其中过滤后的RNA序列分别占97.51%和96.28%。ET008获得7.40 Gb个碱基,有效数据比例为94.50%;而ET008-c54获得7.41 Gb个碱基,有效数据比例为94.37%。统计显示,低质量(Q<30)碱基比例极少,表明测序效果较好[11]。ET008测序碱基质量值Q30超过94.56%,平均GC含量为42.15%,而ET008测序碱基质量值Q30超过93.89%,平均GC含量为42.24%。ET008-c54和ET008重测序结果Q30都在90%以上,说明基因组重测序原始数据结果可靠,可进行后续信息分析。
表2 测序数据统计
Table 2 Results of sequence data statistics

样品原始数据/MbRNA序列/MbRNA序列/%原始碱基数/Gb纯净碱基数/Gb纯净碱基/%GC含量/%Q30/%ET00849.4848.2597.517.406.9594.5042.1594.56ET008-c5449.2747.7896.987.416.9494.3742.2493.89
注:Q30表示原始数据中Qphred>30的碱基占总体碱基的百分比
2.2 差异表达基因(DEGs)筛选
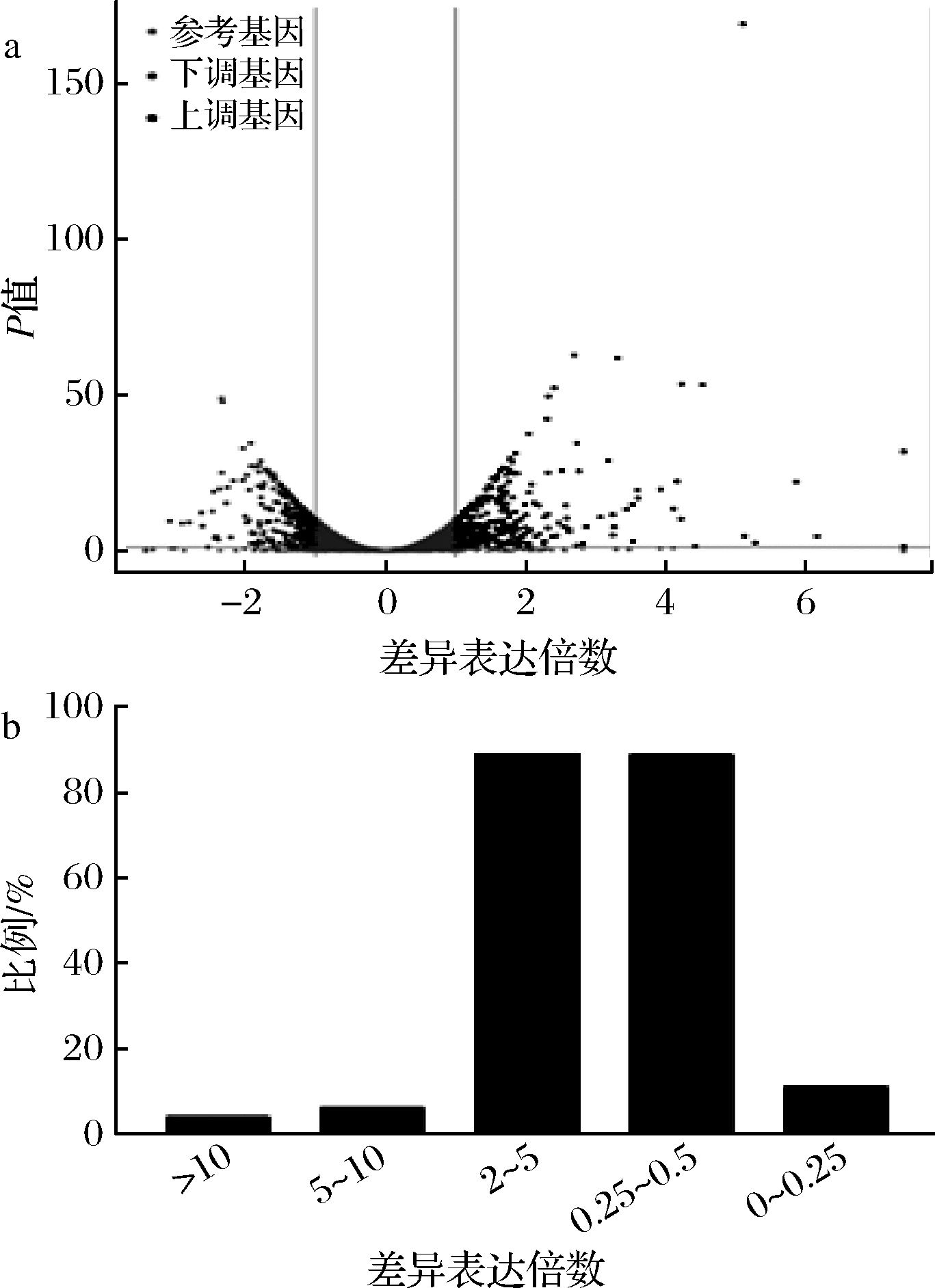
经过数据质控与参考基因组比对,差异表达基因的筛选条件为FC≥2或≤0.5以及P<0.05。差异分析结果如图1-a所示,共鉴定出688个差异表达基因,其中有364个基因显著上调,324个基因显著下调。差异表达上调基因中(图1-b),FC>10倍的基因占上调差异表达基因总数的4.12%,FC 5~10倍的基因占上调差异表达基因总数的6.33%,而FC 2~5 倍的基因占上调差异表达基因总数的89.08%;差异表达下调基因中,FC 0.25~0.5倍的基因占下调差异表达基因总数的88.79%,而FC 0~0.25倍的基因占上调差异表达基因总数的11.21%。相比较而言,菌株ET008-c54的FC在2倍以内和0.25~0.5倍的上调基因和下调基因的数量相当,而FC > 2和0.25~0.5的基因中上调基因数量明显高于下调基因数量,说明这些明显上调的基因可能是潜在的导致ET008-c54具有耐酸性能的关键基因。
通过代谢通路分析以及相关文献相结合[12-13],进一步确定了与耐酸性的相关的关键差异表达基因,其注释情况如表3所示。铁稳态是由复杂的代谢途径的协调运输来调节的,它直接参与关键的生理过程,例如,细胞存活、耐酸和抗氧化应激[14]。在本研究中,与铁吸收有关的基因显著上调,尤其是FET3(167.87)和IZH4(18.89)。INO1(23.02)的显著上调加速了细胞膜的重构,以减少氧化损伤,表明此基因的表达与细胞内的氧化应激密切相关[15]。另外,与麦角甾醇合成有关的基因ERG5(12.21)也显著性上调。总之,确定了8种与耐酸性相关的关键基因。
表3 关键耐酸差异表达基因的注释
Table 3 Annotations of key acid tolerance DEGs

基因名称平均碱基平均碱基(ET008)平均碱基(ET008-c54)差异表达倍数P值上调/下调GeneID描述ERG53 125.871 080.345 171.4012.213.45×10-7上调GeneID:855029C-22固醇去饱和酶FRE1854.34742.58966.105.778.63×10-6上调GeneID:856043Fe/Cu螯合还原酶IZH43 116.88329.995 903.7917.893.79×10-23上调GeneID:854052Zn2+稳态OPT23 305.64656.645 954.643.189.85×10-30上调GeneID:856324与细胞生长有关INO176 381.076 359.87146 402.2823.026.28×10-54上调GeneID:853288肌醇-3-磷酸合酶GCN11 340.86889.461 792.266.341.32×10-9上调GeneID:852680与细胞应对酸性pH有关FET32 512.7129.754 995.67167.871.37×10-32上调GeneID:855080铁转运多铜氧化酶FIT21 479.81279.312 680.309.601.60×10-8上调GeneID:854564与细胞壁合成有关
2.3 差异表达基因功能的GO富集分析
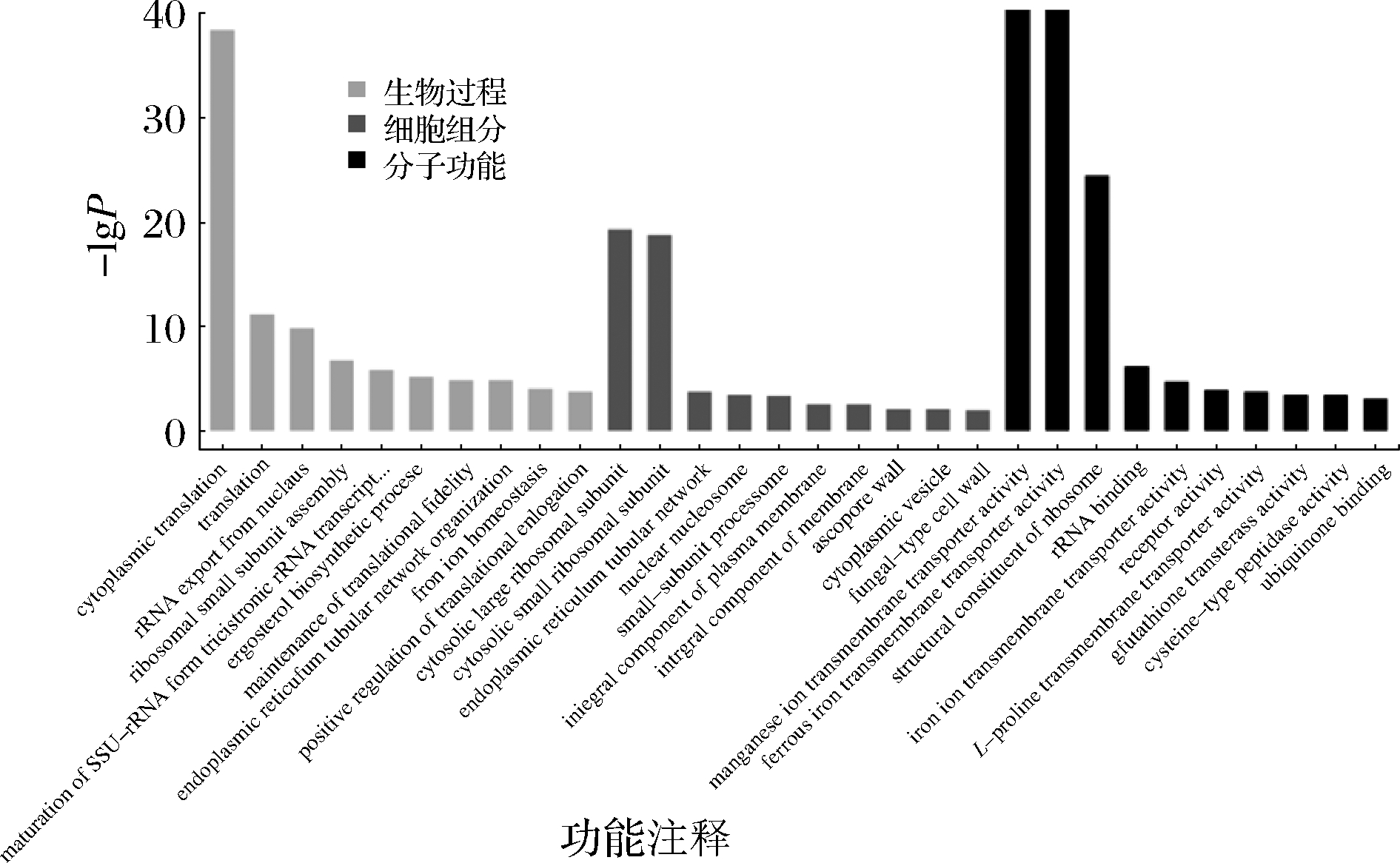
获得差异表达基因之后,与GO数据库进行比对并对差异基因进行GO富集分析,如图2所示。GO数据库把基因的本体分为三类:生物过程(biological process, BP)、细胞组成(cellular component, CC)和分子功能(molecular function, MF)。由图2可知,ET008-c54和ET008的差异基因主要富集在BP过程的细胞质翻译(GO∶0002181)、铁离子稳态(GO∶0055072)和麦角甾醇的生物合成过程(GO∶0006696);差异基因主要集中CC过程的膜的组成部分(GO∶0016021)、质膜的组成部分(GO∶0005887)和真菌型细胞壁(GO∶0009277);差异基因主要富集在MF上的主要是亚铁跨膜转运蛋白活性(GO∶0015093)、铁离子跨膜转运蛋白活性(GO∶0005381)和锰离子跨膜转运蛋白活性(GO∶0005384)。上述结果与关键耐酸基因的结果是一致的,进而说明酸含量过高会影响细胞膜的生理功能进而影响微生物的生长及代谢[16]。
2.4 KEGG富集的差异表达基因的功能
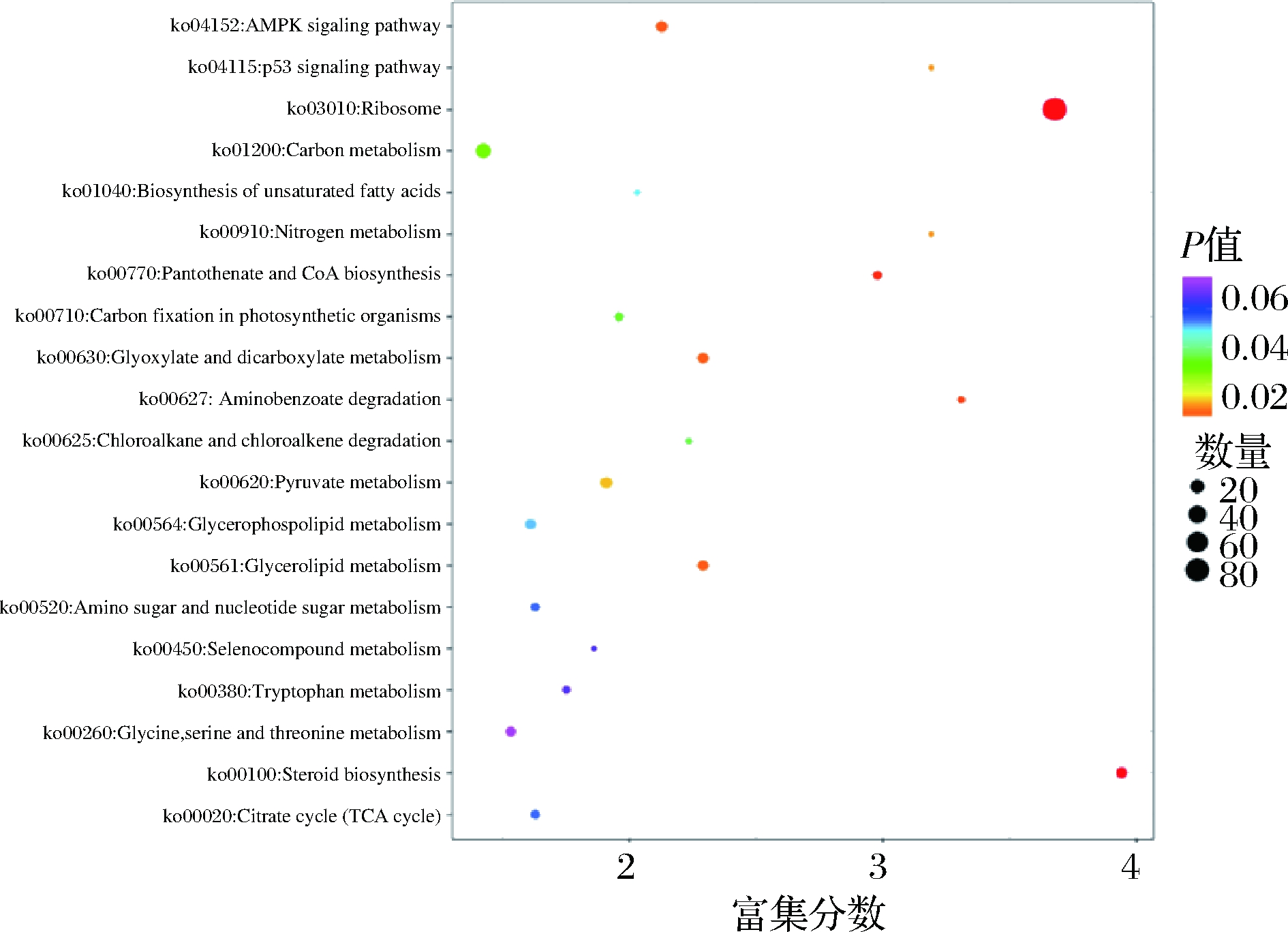
对生物体内差异表达基因的进一步通过KEGG数据库进行功能分析,有助于了解基因的生物学功能[17]。如图3所示,图中横坐标为注释到KEGG通路上的差异基因数与差异基因总数的比值,纵坐标为KEGG通路,点的大小代表注释到KEGG通路上的基因数,颜色从红到紫代表富集的显著性大小。图3富集分析表明,DEGs共涉及30条通路,数量较多的几个被注释的信号通路为核糖体(ko03010)、类固醇生物合成(ko00100)和氨基苯甲酸酯降解(ko00627)等。综上可知,此结果与前期结果是一致的。
2.5 差异表达基因的RT-qPCR验证
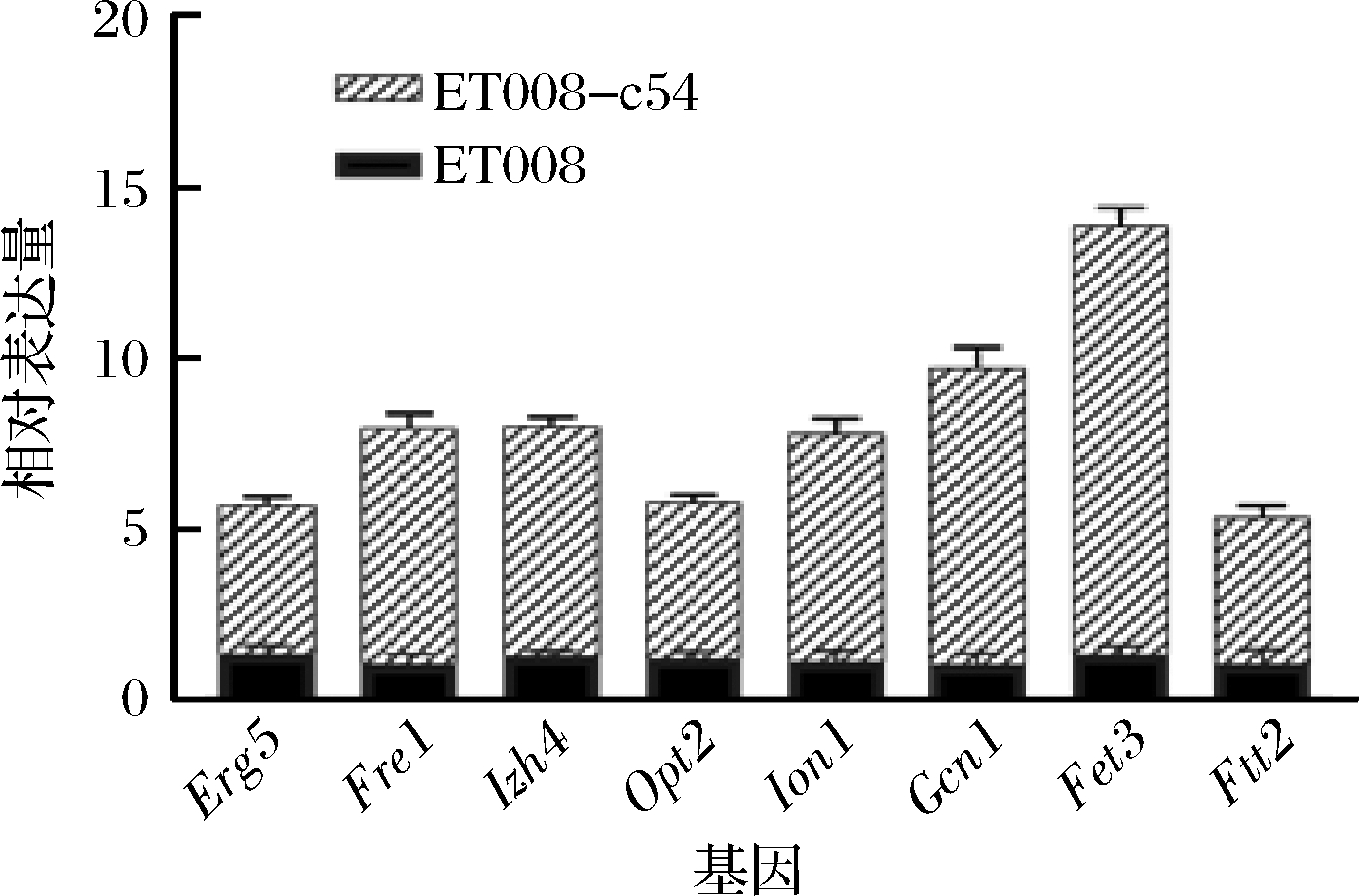
基于转录组数据,确定ET008-c54中与耐酸性相关基因的表达水平发生了显著性变化。为进一步验证ET008-c54中的基因差异表达情况,选取8个与耐酸性相关的关键基因(ERG5、FRE1、IZH4、OPT2、INO1、GCN1、FET3和FIT2),以验证转录组测序结果的可靠性。如图4所示,与ET008相比,ET008-c54中与耐酸性有关的基因的表达水平增强,其中ERG5、FRE1、IZH4,OPT2、INO1、GCN1、FET3和FIT2基因的表达水平是3.20、6.27、5.24、3.79、5.83、7.84、9.55和4.07倍。RT-qPCR验证结果与转录组测序结果中差异表达基因的变化趋势一致,表明本研究结果的有效性和可靠性。
2.6 不同pH条件下2菌株的生长情况
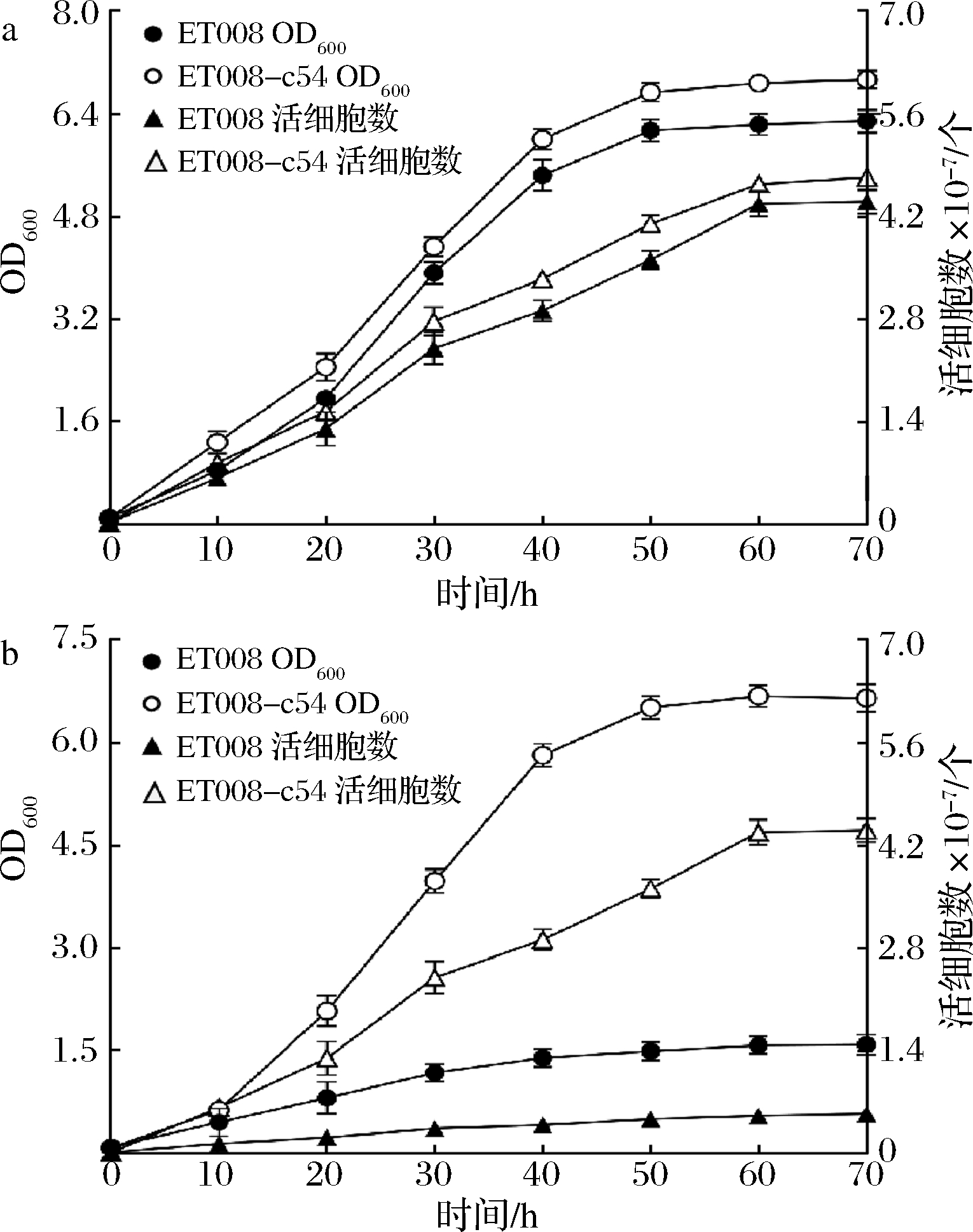
ET008-c54和ET008在pH 3.0和2.5条件下的细胞生长情况如图5所示。由图5可知,在pH值为3.0时,整个培养阶段ET008-c54和ET008的OD600差异都不明显;而pH值为2.5时,整个过程ET008-c54和ET008的OD600差异较大,且在稳定期时ET008-c54的OD600是ET008的4.18倍。对于活细胞个数来说,在pH值为3.0时,ET008-c54和ET008差异依然不明显;而pH值为2.5时,在稳定期时ET008-c54的活细胞数是ET008的8.07倍。这些结果表明,在pH 2.5时,随着培养时间的增加,ET008的活细胞大量死亡,进而说明ET008-c54具有很强的耐酸性。
2.7 发酵实验
ET008-c54和ET008在pH 2.5时的细胞发酵特性如图6所示。由图6可知,在pH值为2.5时,ET008-c54的生长速度更快,最大比生长速率(μmax)是ET008的6.38倍[ET008,μmax=(0.062±0.021) h-1);ET008-c54,μmax=(0.396±0.074) h-1](图6-a)。此外,与ET008相比,ET008-c54最大生物量提高了2.1倍(图6-a)。在发酵结束时,ET008-c54中的葡萄糖几乎耗尽,而在相同的发酵时间内,ET008中只有30.5%的葡萄糖被利用(图6-b)。正如预期,ET008-c54的乙醇产量明显增加,是ET008的150.7%。此外,甘油作为高品质葡萄酒的重要成分[18],与ET008相比,ET008-c54的甘油含量增加218%(图6-c)。乙酸作为不受欢迎的发酵的副产物之一[19],与ET008相比,ET008-c54中乙酸含量降低45.7%。此外,ET008-c54中的麦角甾醇的含量和H+-ATPase活性显著增加,分别比ET008高435%和586%(图6-d)。麦角甾醇是存在于酵母细胞中的主要甾醇,在维持细胞膜的正常功能中起着重要作用[20],例如,较高的麦角甾醇含量可有效保持最佳细胞膜厚度[21]。因此,麦角甾醇的积累可以保护细胞膜免受酸胁迫,并有助于酵母细胞的酸耐受性[22]。低pH和高葡萄糖含量对酵母细胞代谢产生不利的影响,可能导致细胞活力和发酵能力下降,这是由于在极端pH(pH<2.5)下细胞外质子浓缩,负责细胞内质子运输H+-ATPase的压力骤增[23]。这些研究结果表明,具有高活力H+-ATPase的酵母可在低pH和高葡萄糖条件下进行酒精发酵,这与嗜酸酵母的研究一致[24]。总之,ET008-c54的生长速率,葡萄糖消耗速率,乙醇产量和H+-ATPase活性均得到明显改善,表明ET008-c54的发酵能力可有效利用葡萄糖并将其在低pH值下转化为乙醇。
3 结论
本研究通过 Illumina测序平台对耐酸酿酒酵母进行转录组测序分析,共获得688个差异表达基因,其中364个差异基因上调,324个差异基因下调。通过对差异基因的分析,确定8个基因(ERG5、FET3、FIT2、FRE1、GCN1、INO1、IZH4和OPT2)是ET008-c54具有耐酸特性的关键基因。基于差异基因的GO富集和KEGG通路富集结果表明,关键基因与细胞膜的组成及生理功能、麦角固醇合成及亚铁吸收等代谢途径有关。进一步的细胞活力及发酵实验也证实ET008-c54可以耐受较低的pH。本研究结果为进一步筛选高酸度工业果酒酿造菌株提供相关技术参考。
参考文献
[1] DUAN S, HAN P, WANG Q, et al. The origin and adaptive evolution of domesticated populations of yeast from Far East Asia[J]. Nature Communications, 2018, 9(1): 2 690-2 703.
[2] 刘增然. 工业葡萄酒酵母的适应进化和工程菌构建策略[J]. 食品研究与开发, 2011, 32(6):133-138.
[3] VARELA C, SCHMIDT S A, FOULQUIE-BORNEMAN A R, et al. Systems-based approaches enable identification of gene targets which improve the flavour profile of low-ethanol wine yeast strains[J]. Metabolic Engineering, 2018, 49: 178-191.
[4] MEIJNEN J P, RANDAZZO P, MARAIA R, et al. Polygenic analysis and targeted improvement of the complex trait of high acetic acid tolerance in the yeast Saccharomyces cerevisiae[J]. Biotechnology for Biofuels, 2016, 9(1): 5.
[5] 于玲. 耐酸石榴酒酵母的筛选及其耐酸性初步研究[D]. 泰安:山东农业大学, 2008.
[6] 张克俞,张明明,赵心清,等. 关键基因过表达提高酿酒酵母抑制剂耐受性及乙醇发酵性能[J]. 应用与环境生物学报, 2018, 24(3): 541-546.
[7] ROSSIGNOL T, DULAN L, JULIEN A, et al. Genome-wide monitoring of wine yeast gene expression during alcoholic fermentation[J]. Yeast, 2003, 20(16): 1 369-1 385.
[8] 杨宁,王伟明,姚琳,等. 3,5-二硝基水杨酸法测定发酵型果露酒中总糖含量[J]. 中国酿造, 2018, 37(1): 181-184.
[9] 王传荣. 酒精活性干酵母主要质量指标控制的探讨与研究[J]. 江苏调味副食品, 2010(1), 34-37;42.
[10] NYANGA L K, NOUT M J, SMID E J, et al. Fermentation characteristics of yeasts isolated from traditionally fermented masau (Ziziphus mauritiana) fruits[J]. International Journal of Food Microbiology, 2013, 166(3): 426-432.
[11] 白想想. 重排酵母菌转录组注释和基因差异表达分析[D]. 武汉:华中农业大学, 2017.
[12] 赵心清,张明明,徐桂红,等. 酿酒酵母乙酸耐性分子机制的功能基因组进展[J]. 生物工程学报, 2014, 30(3): 368-380.
[13] 陈洪奇,于欣水,张明明,等. 硫酸锌添加对酿酒酵母乙酸胁迫条件下全局基因转录的影响[J]. 微生物学通报, 2017, 44(6): 1 312-1 321.
[14] MIRA N P, HENRIQUES S F, KELLER G, et al. Identification of a DNA-binding site for the transcription factor Haa1, required for Saccharomyces cerevisiae response to acetic acid stress[J]. Nucleic Acids Research, 2011, 39(16): 6 896-6 907.
[15] ZHU X, ZOU S, LI Y, et al. Transcriptomic analysis of Saccharomyces cerevisiae upon honokiol treatment[J]. Research in Microbiology, 2017, 168(7): 626-635.
[16] 郝小明,陈博,安泰. 工业微生物酸胁迫的耐受机制及改造途径[J]. 生物工程学报, 2015, 31(8): 1 151-1 161.
[17] 龙思宇, 严少敏. 酿酒酵母功能基因组学研究进展[J]. 科学技术与工程, 2014, 14(2): 62-66;71.
[18] 王秀菊,杜金华,朱济义,等. 影响果酒酵母甘油产量主要因素的研究[J]. 食品与发酵工业, 2010, 36(2): 80-84.
[19] 屈慧鸽,邓军哲,孙志军,等. 乙酸对葡萄酒不完全发酵的影响[J]. 中外葡萄与葡萄酒, 2002(2): 27-28.
[20] NAVARRO-TAPIA E, NANA R K, QUEROL A, et al. Ethanol cellular defense induce unfolded protein response in yeast[J]. Frontiers in Microbiology, 2016, 7: 189.
[21] AGUILERA F, PEINADO R A, MILLAN C, et al. Relationship membrane in different wine yeast strains[J]. International Journal of Food Microbiology, 2006, 110(1): 34-42.
[22] FLETCHER E, FEIZI A, BISSCHOPS M M M, et al. Evolutionary engineering reveals divergent paths when yeast is adapted to different acidic environments[J]. Metabolic Engineering, 2017, 39: 19-28.
[23] 刘兴艳, 贾博, 赵芳, 等. 酿酒酵母对弱有机酸胁迫的应激机制研究进展[J]. 食品与发酵工业,2013,39(6): 125-129.
[24] FLETCHER E, FEIZI A, KIM S S, et al. RNA-seq analysis of Pichia anomala reveals important mechanisms required for survival at low pH[J]. Microbial Cell Factories, 2015, 14(1): 143.