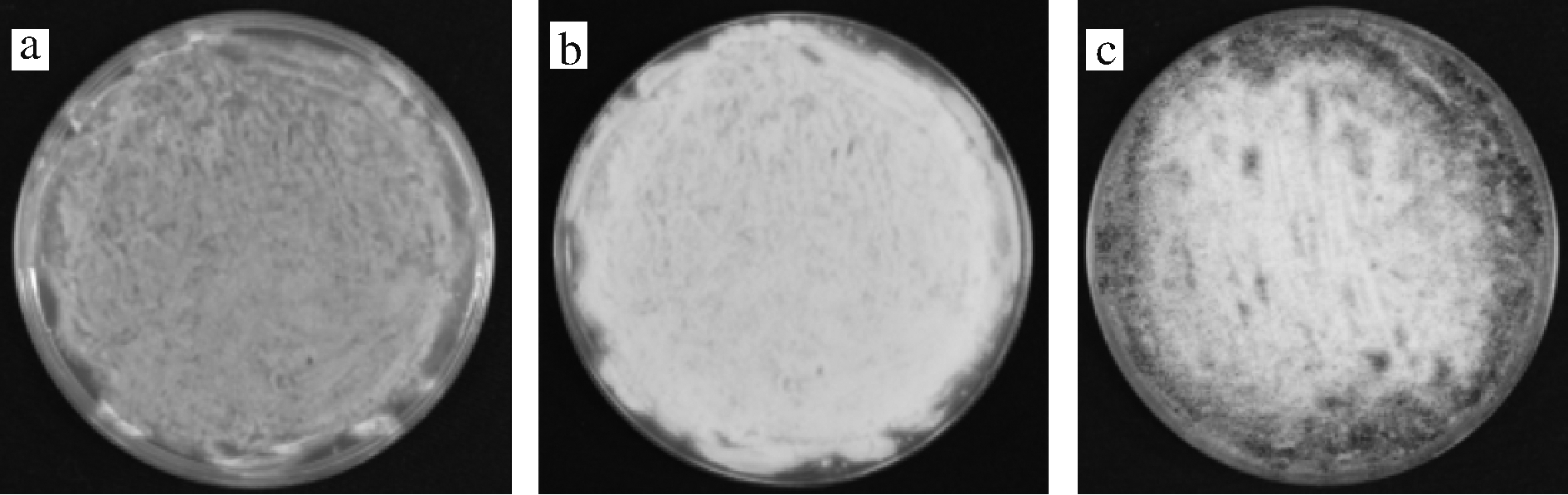
a-培养1 d;b-培养2 d;c-培养3 d
图1 不同时间黑曲霉CICC 40408菌落状态结果
Fig.1 Results of A. niger falling state at different time
黑曲霉(Aspergillus niger)属于子囊菌门Ascomycota,曲霉科Aspergillaceae,曲霉属Aspergillus中的一个常见种,广泛分布于世界各地的粮食、植物性产品和土壤中。美国食品与药品管理局于1987年将黑曲霉列为安全菌种[1],已广泛用于淀粉酶、酸性蛋白酶、纤维素酶、果胶酶、葡萄糖氧化酶、柠檬酸、葡糖酸和没食子酸等的生产。近年,研究发现部分食品工业用黑曲霉菌株具有产伏马菌素能力[2-3],该毒素具有急性毒性和潜在致癌性,影响人类和动物健康[4-8]。辨别产毒菌株和非产毒菌株对我国食品工业用黑曲霉菌株使用安全性十分重要[1]。利用全基因组测序(whole genome sequencing,WGS)的分型方法,能够在全基因组碱基序列的基础上进行生物信息学分析,提高黑曲霉产毒菌株分辨能力。目前,由于黑曲霉细胞壁结构复杂、代谢产生大量黑色色素、胞内多糖、生物酶等原因,存在全基因组DNA提取难度大、可参考的全基因组数据少、全基因组分析方法尚不成熟的问题[9-11]。获得完整性好、纯度高的基因组DNA[12-14],是后续全基因组分析的前提,对黑曲霉产毒菌株分型辨别具有重要意义。
目前关于丝状真菌全基因组DNA提取方法主要有CTAB法、试剂盒法和氯化苄法[15],细胞壁破裂方法主要有石英砂研磨法和液氮研磨法。黑曲霉属丝状真菌,细胞壁结构复杂坚固,富含细胞壁多糖和几丁质,各种成分相互结合,使其具有一定强度[16],在进行全基因组DNA提取时很难将细胞壁破裂,而且细胞中存在大量多糖,使得DNA极少能溶出[17-21],如果长时间研磨会导致溶出的DNA断裂,破坏DNA完整结构[22]。黑曲霉中存在大量色素,使得全基因组DNA的提取更加困难[9-11],常规DNA提取方法得到的DNA样品通常A260/A280值>2.0,A260/A230值<2.0,样品中有色素、RNA、糖类、盐类或有机试剂的污染,虽可成功进行常规PCR扩增,但无基因组主带,不满足全基因组测序要求[23-24]。本文根据目前已有方法,针对黑曲霉菌株特性,优化了黑曲霉全基因组DNA提取方法,为黑曲霉基因组的分析研究奠定了基础。
1.1.1 供试菌株
该研究所用黑曲霉(Aspergillus niger)菌株,来源于20个省市,从23种食品基质中分离而来,目前保藏于中国工业微生物菌种保藏中心,菌株编号为CICC 2286、CICC 2629、CICC 2693、CICC 40040、CICC 40043、CICC 40046、CICC 40068、CICC 40087、CICC 40128、CICC 40130、CICC 40132、CICC 40133、CICC 40137、CICC 40408、CICC 40615、CICC 40755、CICC 40845、CICC 40846、CICC 40847、CICC 40849、CICC 40863、CICC 41115、CICC 41116、CICC 41178、CICC 41194、CICC 41195、CICC 41215、CICC 41255、CICC 41256、CICC 41257、CICC 41258、CICC 41568、CICC 41576、CICC 41586、CICC 41594。
1.1.2 试剂及仪器
溴代十六烷基三甲胺(cetyl trimethyl ammonium bromide,CTAB),Sigma公司;20 g/L RNA酶、20 g/L蛋白酶K,天根生化科技(北京)有限公司;Tris-饱和酚,Solarbio Sigma;氯仿,北京市通广精细化工公司;异戊醇,西陇化工股份有限公司;异丙醇,北京化工厂;乙酸钠,广州化学试剂厂;DNA纯化试剂盒,OMEGA;真菌DNA提取试剂盒,OMEGA;马铃薯葡萄糖琼脂培养基,北京陆桥技术股份有限公司。
IPP 260恒温培养箱,美墨尔特(上海)贸易有限公司;TProfessional PCR仪,北京世贸远东科学仪器有限公司;EC 250-90电泳仪,美国ThermoEC有限公司;Bead Ruptor 12生物样品均质器,奥然科技(北京)有限公司;核酸蛋白分析仪BioDrop, 豪沃生物科技(上海)有限公司;GelDoc EZ全自动免染凝胶成像分析系统,美国伯乐Bio-ra有限公司;AC2-4S1生物安全柜,新加坡艺思高科技有限公司。
将PDA培养基倾倒在培养皿内,待培养基冷却凝固,在培养皿内平整铺放无菌玻璃纸,用无菌棉签蘸取培养好的黑曲霉试管斜面中的孢子,均匀涂布整个培养皿,25 ℃倒置培养。培养结束后,用无菌接种环将玻璃纸上的菌丝体刮至新的培养皿内待用。
将培养平板分别25 ℃倒置培养1、2和3 d,观察黑曲霉菌落生长状态、菌体生物量、产孢子情况,确定黑曲霉全基因组DNA提取的最佳培养时间。
生物均质器菌体破碎法:称取200 mg菌丝体至含0.4 g的0.1 mm石英砂,0.4 g的1.5 mm锆株,0.3 g 的3.0 mm锆珠的无菌研磨管内,加入200 μL 的CTAB裂解液,拧紧管盖,放入生物样品均质器卡槽内,破碎时长2 min,显微观察破碎后的菌体样品。液氮研磨法:将菌丝体加入无菌研钵中,倒入液氮,迅速研磨,在液氮蒸发净前,再次加入液氮,继续迅速研磨菌丝体至粉末状。显微观察细胞壁破裂后的菌丝体样品与生物样品均质器细胞壁破裂法的结果差异。
菌丝体液氮研磨后应用真菌DNA提取试剂盒法、CTAB法和CTAB法结合试剂盒纯化法,分别提取4株黑曲霉CICC 40137、CICC 40130、CICC 40132和CICC 41115全基因组DNA,对比不同方法所提全基因组DNA差异,筛选出适用于全基因组测序的黑曲霉DNA提取方法。
1.5.1 菌丝体液氮研磨后应用真菌DNA提取试剂盒法(方法一)
将菌丝体加入无菌研钵中,倒入液氮,迅速研磨,在液氮蒸发净前,再次加入液氮,继续迅速研磨菌丝体至粉末状,称取液氮研磨好的菌丝体粉末200 mg装入1.5 mL离心管中,后续步骤按照真菌DNA提取试剂盒说明书准确提取。
1.5.2 菌丝体液氮研磨后应用CTAB法(方法二)
菌丝体研磨方法同1.5.1,称取200 mg液氮研磨好的菌丝体粉末至1.5 mL离心管中,加入600 μL的CTAB裂解液,65 ℃水浴45 min,13 000 r/min离心10 min;取上清于一新离心管中,加等体积的V(酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1,12 000 r/min离心10 min;取上清,加等体积的V(氯仿)∶V(异戊醇)=24∶1,12 000 r/min离心10 min;取上清,加等体积的异丙醇试剂,-20 ℃沉淀30 min,12 000 r/min离心20 min;收集沉淀,75%酒精冲洗,超净台真空干燥;100 μL TE溶解DNA,-20 ℃保存备用[25-26]。
1.5.3 菌丝体液氮研磨后应用CTAB法提取结合试剂盒纯化法(方法三)
菌丝体研磨方法同1.5.1,称取200 mg液氮研磨好的菌丝体粉末至1.5 mL离心管中,加入600 μL的CTAB裂解液和少量的聚乙烯吡咯烷酮(polyvingl pyrroliddone,PVP),65 ℃水浴45 min,13 000 r/min离心10 min;取上清于一新离心管中,加等体积的V(酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1,12 000 r/min离心10 min;取上清,加等体积的V(氯仿)∶V(异戊醇)=24∶1,12 000 r/min离心10 min;取上清,加 0.7倍体积的异丙醇,0.1倍体积的3 mol/L NaAc,-20 ℃沉淀30 min,12 000 r/min离心20 min;收集沉淀,75%酒精冲洗,超净台真空干燥;100 μL TE 溶解DNA,加入1 μL的RNA酶,37 ℃水浴 1 h;加入400 μL V(氯仿)∶V(异戊醇)=24∶1,12 000 r/min离心10 min,重复2次。采用OMEGA公司cycle-pure DNA纯化试剂盒进行纯化,得到高纯度基因组DNA,-20 ℃保存备用。
使用BioDrop测定DNA样本浓度,根据A260/A280和A260/A230值判断DNA纯度[27]。
吸取5 μL基因组DNA样品与6×Loading Buffer混合后,点样于质量分数为0.8%的琼脂糖凝胶中,染色剂为GoldView,在电泳缓冲液为 1×TAE,电压为 110 V 的条件下电泳25 min,之后在凝胶成像仪上观察DNA片段的大小、完整性及电泳条带的清晰度,进行拍照。
将31株黑曲霉菌株分别用无菌棉签蘸取培养好的黑曲霉试管斜面中的孢子,接种在铺有玻璃纸的PDA培养皿内,涂抹整个培养皿,25 ℃倒置培养。培养结束后,用无菌接种环将玻璃纸上的菌体刮至研钵内液氮研磨,应用CTAB法提取并结合试剂盒纯化法提取全基因组,对该方法进行应用验证。
黑曲霉培养1 d菌落为灰白色,菌体量少(图1-a);培养2 d菌落为白色,菌落覆盖整个平板,产孢量少(图1-b);黑曲霉培养至第3天,菌落产生大量黑色孢子,菌体老化,细胞壁坚固,收集的菌丝体含有孢子,DNA提取过程中会溶出大量黑色素,影响DNA样品的提取(图1-c),因此选用收集培养2 d的黑曲霉菌丝体作为全基因组DNA提取最佳时间。

a-培养1 d;b-培养2 d;c-培养3 d
图1 不同时间黑曲霉CICC 40408菌落状态结果
Fig.1 Results of A. niger falling state at different time
将收集的菌体采用生物均质器研磨法和液氮研磨法进行显微对比,两种不同细胞破裂方法对菌体破碎情况明显不同。图2-a为研磨前菌体状态,菌丝体结构完整;图2-b为生物均质器研磨法结果显微照片,大部分菌体细胞结构被破坏,但破碎结果不均匀,仍有部分菌体结构未破碎;图2-c为液氮研磨结果显微照片,菌丝体破碎充分、均匀,未观察到完整菌体结构。与研磨前相比,两种破壁方法虽然都有一定程度的破坏,但菌体破碎结果存在明显差异,液氮研磨法研磨更充分均匀,因此选用为黑曲霉全基因组提取细胞壁处理方法。

a-研磨前菌体形态;b-生物均质器破壁结果;c-液氮研磨破壁结果
图2 黑曲霉菌丝体破壁结果显微照片
Fig.2 Micrograph of the broken wall of A. niger mycelium
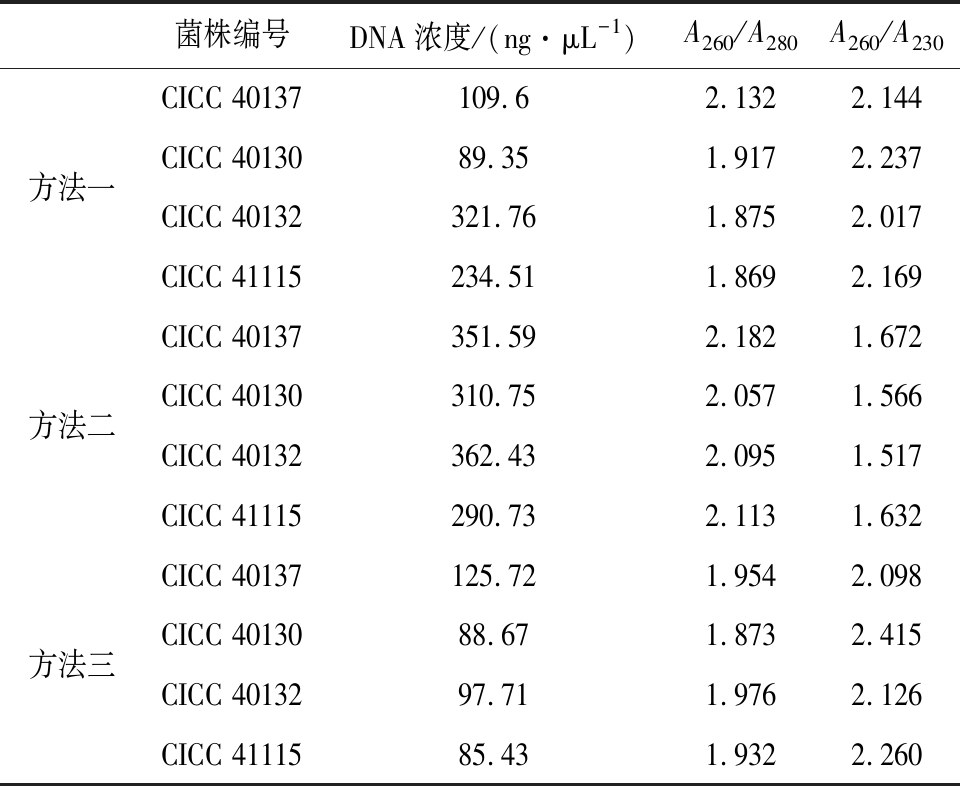
采用BioDrop对3种方法的样品基因组DNA进行测定,结果如表1所示。方法一为液氮研磨后应用真菌DNA提取试剂盒法测定结果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌DNA浓度分别为109.6、89.35、321.76和234.51 ng/μL,菌株CICC 40130、CICC 40132和CICC 41115的A260/A280值在1.8~2.0,A260/A230值均>2,证明DNA纯度较高。但CICC 40137菌株A260/A280值为2.132>2.0,证明RNA含量较高;方法二为菌丝体液氮研磨后应用CTAB法测定结果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌DNA浓度分别为351.59、310.75、362.43和290.73 ng/μL,但4株菌A260/A280值均>2.0,证明DNA样品中有RNA残留,A260/A230值均<2.0,说明样品DNA中含有较多的糖类、盐类或有机试剂的污染;方法三为菌丝体液氮研磨后应用CTAB法提取结合试剂盒纯化法测定结果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌DNA浓度分别为125.72、88.67、97.71和85.43 ng/μL,A260/A280值均在1.8~2.0,A260/A230值均>2.0,说明本方法提取到的基因组DNA样品蛋白质和RNA含量很低,纯度浓度均较好,吸光增色反应明显,在260 nm处吸光度最高,证明基因组纯度很高,优于方法一和方法二所提DNA样品。
分别对3种方法所得到的基因组DNA 进行电泳检测,检测结果如图3所示。图3-a为方法一提取的DNA样品电泳结果,由图可知未见到完整DNA主条带,DNA长度均<5 000 bp。图3-b是应用方法二提取的DNA样品电泳结果,泳道上有黑色阴影,未显示完整基因组DNA主带,多为小分子片段,<5 000 bp;图3-c为方法三所提DNA样品电泳结果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌都有DNA主条带,加样孔附近滞留的物质也很少,基因组大小约为23 kb。方法一和方法二DNA链状结构破坏严重,虽能满足PCR扩增,但未见到全基因组DNA条带。
表1 基因组DNA浓度检测
Table 1 Genomic DNA concentration detection
菌株编号DNA浓度/(ng·μL-1)A260/A280A260/A230方法一CICC 40137109.62.1322.144CICC 4013089.351.9172.237CICC 40132321.761.8752.017CICC 41115234.511.8692.169方法二CICC 40137351.592.1821.672CICC 40130310.752.0571.566CICC 40132362.432.0951.517CICC 41115290.732.1131.632方法三CICC 40137125.721.9542.098CICC 4013088.671.8732.415CICC 4013297.711.9762.126CICC 4111585.431.9322.260
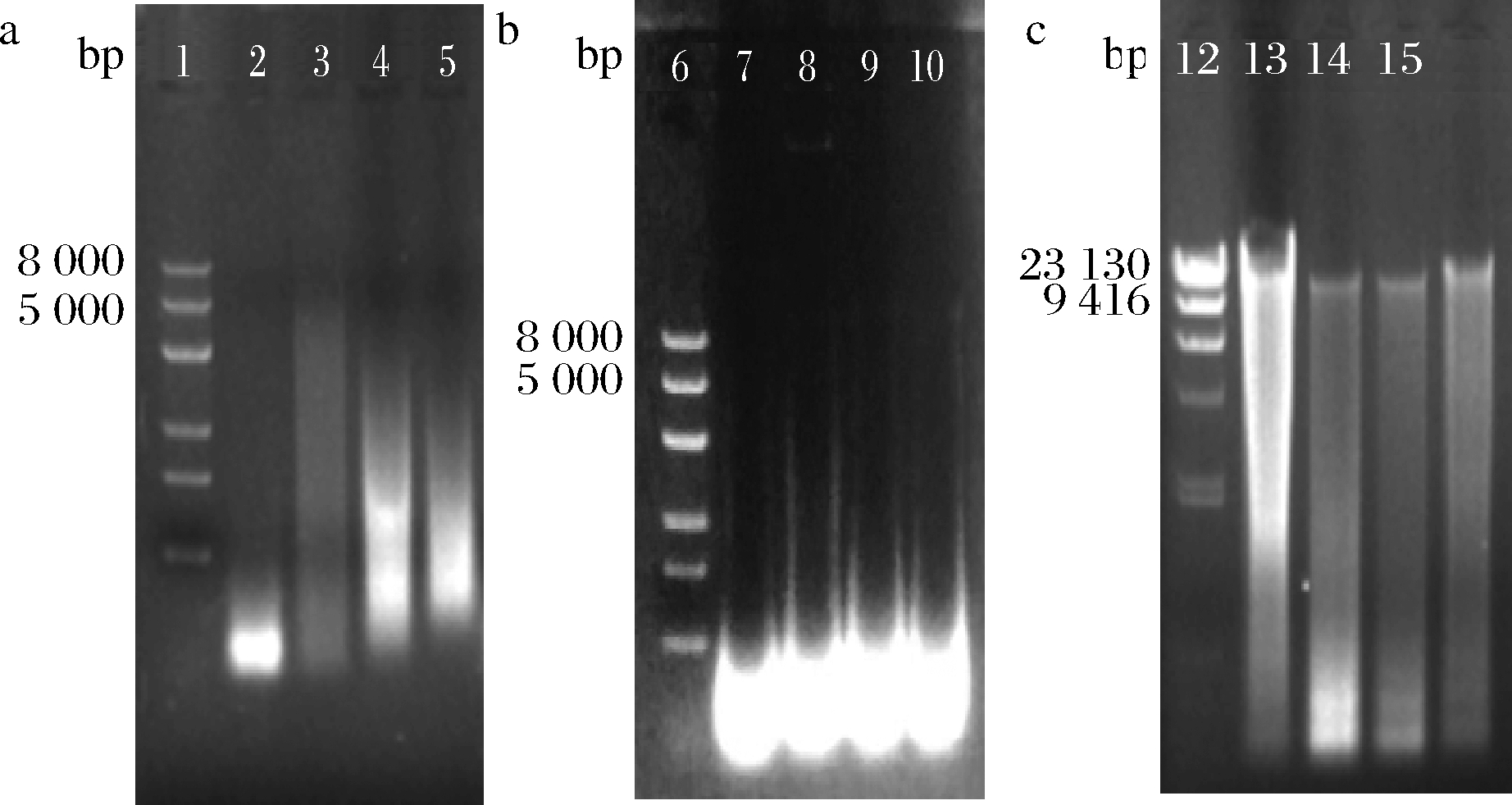
1、6-DL8000 Marker;2、7、11-CICC 40137;3、8、12-CICC 40130;4、9、13-CICC 40132;5、10和14-CICC 41115;15-λDNA/Hind Ⅲ Markera-方法一;b-方法二;c-方法三
图3 不同方法提取黑曲霉全基因组DNA电泳结果
Fig.3 The results of DNA electrophoresis of A. niger whole gene were extracted by different methods
如图4所示,菌丝体液氮研磨后应用CTAB法提取,结合试剂盒纯化法,对31株不同分离源黑曲霉菌株进行方法验证结果,7泳道全基因组DNA条带相对较弱,推测由于液氮研磨步骤研磨不充分,DNA溶出量少。8、11和13泳道的最前端出现明亮条带,RNA酶与样品中RNA反应不彻底,导致样品中含有RNA。但31株黑曲霉DNA样品均有23 kb大小的完整全基因组DNA主条带。对该DNA样品进行浓度测定,实验结果如表2所示,浓度为52.24~212.37 ng/μL。全基因组测序要求DNA浓度>12.5 ng/ μL,DNA总量均在2 μg以上,31株黑曲霉DNA样品满足全基因组测序工作,证明该方法适用于黑曲霉全基因组DNA的提取。
本研究通过对3种常见真菌基因组DNA提取方法比较分析,确定了一种高质量黑曲霉全因组提取方法,具体为菌丝体经液氮研磨后,采用CTAB法提取并结合试剂盒纯化。该方法提取的基因组DNA条带完整、纯度高,可以满足WGS要求,方法优点为:(1)确定了黑曲霉实验菌株的培养条件和细胞壁破裂方法。因为黑曲霉主要成分为几丁质,与细菌或者酵母相比其结构更加坚固。但是丝状真菌培养过程中,细胞壁坚固程度是发生变化的,菌丝体在幼嫩时期细胞壁相对薄弱一些,老熟之后坚固,培养2 d的黑曲霉菌丝体提取全基因组DNA时间最佳[28]。液氮研磨相比生物样品均质器破碎相比,细胞壁更加充分,确保DNA释放。(2)CTAB裂解液中加入PVP、RNA酶和蛋白酶K。PVP可以去除多糖、酚类、醌类等物质[23,29-31],RNA酶去除RNA,蛋白酶K有效除去蛋白。(3)结合cycle-pure DNA纯化试剂盒进行纯化。黑曲霉菌丝体中富含色素、多糖、蛋白质[32],传统CTAB 提取方法尽管对DNA产物进行洗涤除杂,但是色素物质残留较多,在进行凝胶电泳检测时,影响了DNA与染料的结合,因此看不到基因组完整条带。应用试剂盒纯化后虽然基因组DNA有一定损失,但是多糖、色素等杂质可充分除去,避免影响GoldView与基因组结合显色,由电泳结果可知提升了DNA的纯度,有清晰可见的阳性条带。
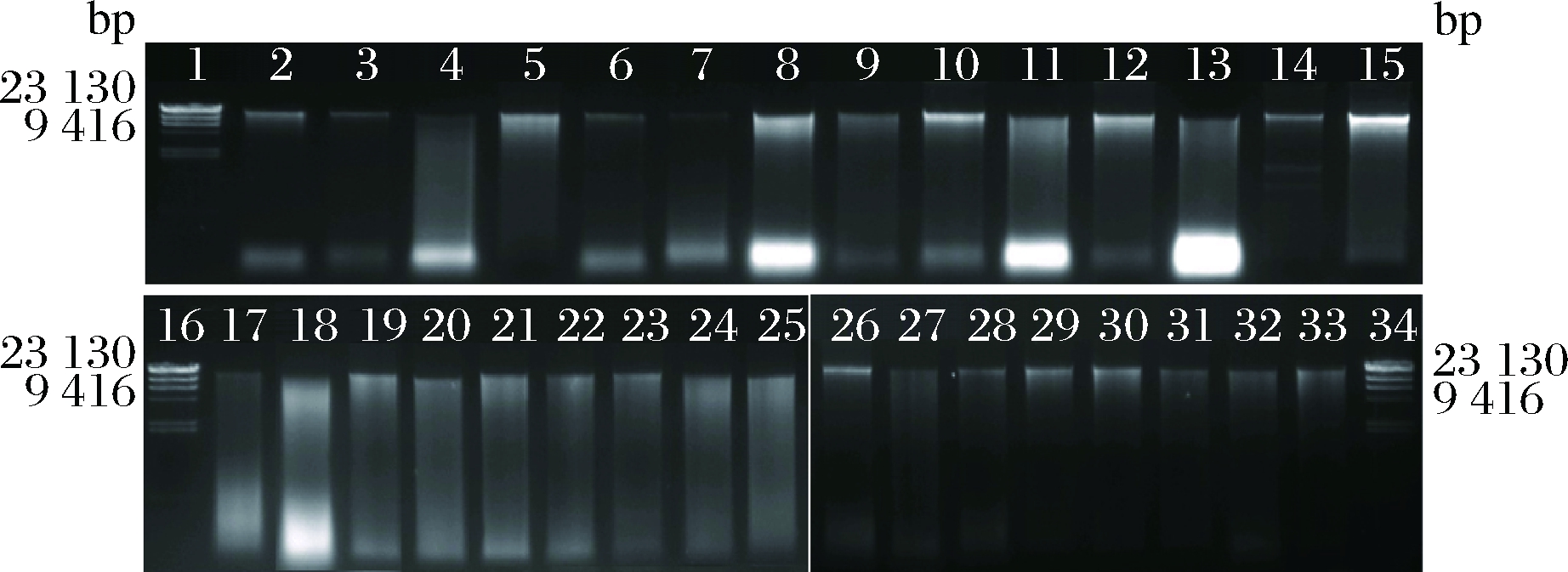
1、16、34-λDNA/Hind Ⅲ Marker;2~15菌株编号分别为CICC 40087、CICC 40863、CICC 40046、CICC 40849、CICC 41586、CICC 40133、CICC 40845、CICC 40128、CICC 40040、CICC 40847、CICC 2286、CICC 41178、CICC 41594、CICC 40068;17~33菌株编号分别为CICC 2693、CICC 40043、CICC 40408、CICC 40615、CICC 40755、CICC 40846、CICC 41116、CICC 41194、CICC 41195、CICC 41215、CICC 41255、CICC 41256、CICC 41257、CICC 41258、CICC 41568、CICC 41576、CICC 2629
图4 不同编号黑曲霉菌株全基因组DNA电泳结果
Fig.4 Results of complete genomic DNA electrophoresis of A. niger strains with different numbers
表2 不同分离源31株黑曲霉基因组DNA浓度
Table 2 Genomic DNA concentrations of 31 strains of A. niger from different isolates

菌株编号 DNA浓度/(ng·μL-1)A260/A280A260/A230菌株编号DNA浓度/(ng·μL-1)A260/A280A260/A230CICC 4008765.42 1.8742.346CICC 40408100.291.8302.312CICC 4086352.24 1.9652.259CICC 40615116.871.8562.232CICC 40046212.37 2.3562.143CICC 4075598.341.8432.155CICC 40849121.911.8452.562CICC 4084692.961.9542.428CICC 4158662.861.9562.375CICC 41116113.291.9112.059CICC 4013369.671.8372.065CICC 41194120.511.9402.320CICC 40845143.54 2.2322.173CICC 41195103.451.8532.145CICC 4012873.931.8952.430CICC 41215107.341.8222.267CICC 40040112.421.9452.358CICC 4125575.781.8972.296CICC 40847190.75 2.2102.268CICC 4125669.531.9432.425CICC 2286112.321.9342.175CICC 4125776.321.8672.342CICC 41178135.132.4322.013CICC 4125897.451.9532.352CICC 4159464.371.8482.436CICC 4156885.781.9252.344CICC 40068211.421.8252.295CICC 4157689.321.9572.167CICC 269371.431.9542.450CICC 262976.231.8522.039CICC 40043132.312.1672.153
本研究确定黑曲霉全基因组DNA提取方法,可满足WGS工作要求,为黑曲霉基因组数据库的构建、菌株溯源、产毒基因分析奠定基础,有助于产毒菌株和非产毒菌株快速准确辨别,对我国食品工业用黑曲霉菌株的使用安全性有重要意义。该法可作为实验室提取黑曲霉高质量基因组DNA的方法推广使用。
[1] 韩小敏,张靖,韩春卉,等.产B类伏马菌素黑曲霉菌株基因水平鉴定研究[J].中国食品卫生杂志, 2014,26(2):105-110.
[2] FRISVAD J C,LARSEN T O,THRANE U,et al.Fumonisin and ochratoxin production in industrial Aspergillus niger strains[J].PLoS One,2011,6(8):e23 496.
[3] VARGA J,KOCSUB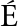 S,SURI K,et al.Fumonisin contamination and fumonisin producing black Aspergilli in dried vine fruits of different origin[J].International Journal of Food Microbiology,2010,143(3):143-149.
S,SURI K,et al.Fumonisin contamination and fumonisin producing black Aspergilli in dried vine fruits of different origin[J].International Journal of Food Microbiology,2010,143(3):143-149.
[4] HARRISON L R,COLVIN B M,GREENE J T,et al.Pulmonary edema and hydrothorax in swine produced by fumonisin B1,a toxic metabolite of Fusarium moniliforme[J].Journal of Veterinary Diagnostic Investigation,1990,2(3):217-221.
[5] GELDERBLOM W,KRIEK N,MARASAS W,et al.Toxicity and carcinogenicity of the Fusanum moniliforme metabolite,fumonisin B1,in rats[J].Carcinogenesis,1991,12(7):1 247-1 251.
[6] CHU F S,LI G Y.Simultaneous occurrence of fumonisin B1 and other mycotoxins in moldy corn collected from the People′s Republic of China in regions with high incidences of esophageal cancer[J].Applied and Environmental Microbiology,1994,60(3):847-852.
[7] ROOS P F,RICE L G,OSWEILER G D,et al.A review and update of animal toxicoses associated with fumonisin-contaminated feeds and production of fumonisins by Fusarium isolates[J].Mycopathologia, 1992,117(1):109-114.
[8] SYDENHAM E W,THIEL P G,MARASAS W F,et al.Natural occurrence of some Fusarium mycotoxins in corn from low and high esophageal cancer prevalence areas of the Transkei,Southern Africa[J].Journal of Agricultural and Food Chemistry,1990,38(10):1 900-1 903.
[9] 周礼红.一种制备富含多糖丝状真菌基因组DNA的方法[J].湖北农业科学, 2008,47(4):379-381.
[10] CAMPOY S,PEREZ F,MARTIN J F,et al.Stable transformants of the azaphilone pigment-producing Monascus purpureus obtained by protoplast transformation and Agrobacterium-mediated DNA transfer[J].Curr Genet,2003,43:447-452.
[11] HAUGLAND R A,HECKMAN L J.Evalution of different methods for the extraction of DNA from fungal conidia by quantiative competitive PCR analysis[J].J Microbiol Methods,1999,37(2):165-176.
[12] 杨艳秋,王丽,贺丹,等.真菌DNA提取方法的建立和比较[J].中国组织工程研究与临床康复,2007,11(50):10 093-10 096.
[13] CHEN S C,HALLIDAY C L,MEYER W.A review of nucleic acid-based diagnostic tests for systemic mycoses with an emphasis on polymerase chain reaction-based assays[J].Med Mycol,2002,40(4):333-357.
[14] 吴发红,黄东益,黄小龙,等.几种真菌DNA提取方法的比较[J].中国农学通报,2009,25(8):62-64.
[15] 邵碧英,王传得,郑晶,等.黄曲霉DNA提取及PCR-RFLP检测方法的建立[J].食品科学,2008,29(12):393-396.
[16] 曹国君,赵缜,彭奕冰,等.真菌基因组DNA的提取和通用PCR检测方法的建立[J].检验医学,2011,26(11):773-778.
[17] 刘艳梅,朱建兰,杨航宇.曲霉基因组DNA提取方法研究[J].西北农业学报,2009,18(2):55-58.
[18] MAAROUFI Y,AHARIZ N,HUSSON M,et al.Comparison of different methods of isolation of DNA of commonly encountered Candida species and its quantitation by using a real time PCR based assay[J].J Clin Microbiol,2004,42(7):3 159-3 163.
[19] HAUGLAND R A,BRINKMAN N,VESPER S J.Evaluation of rapid DNA extraction methods for the quantitative detection of fungi using realtime PCR analysis[J].J Microbiol Methods,2002,50(3):319-323.
[20] 崔丽霞,韩建荣.青霉DNA提取方法比较研究[J].山西大学学报,2004,27(2):185-187.
[21] MIN J, ARGANOZA T, OHMBERGER J, et al. Alternative methods of preparing whole whole-cell DNA from fungi for dot-blot, restriction analysis, and colony filter hybridization[J]. Analytical Biochemistry, 1995,225:94-100.
[22] 薛淑静,岳田利,关健,等.一种真菌DNA提取方法的改进[J].食品研究与开发,2006,27(4):39-40.
[23] 刘丽,张永军,许长征,等.一种改良的CTAB法提取产多糖真菌DNA[J].中国生物工程杂志,2014,34(5):75-79.
[24] 李焕宇,付婷婷,张云,等.5种方法提取真菌基因组DNA作为PCR模板效果的比较[J].中国农学通报,2017,33(16):28-35.
[25] GUO L D,HYDE K D,LIEW E C Y.Identification of endophytic fungi from Livistona chinensis based on morphology and rDNA sequences[J].New Phytologist,2000,147(3):617-630.
[26] SAGHAI-M A ROOF,SOLMAN K M,JORGENSEN R A,et al.Ribosomal DNA spacer-length polymorphisms in barley:Mendelian inheritance,chromosomal location and population dynamics[J].Pnas,1984,81(24):8 014-8 018.
[27] 张颖慧,魏东盛,邢来君,等.一种改进的丝状真菌DNA提取方法[J].微生物学通报,2008,35(3):466-469.
[28] GRIFFIN D W,KELLOGG C A,PEAK K K,et al.A rapid and efficient assay for extracting DNA from fungi[J].Lett Appl Microbiol,2002,34(3):210-214.
[29] 易庆平,罗正荣,张青林.植物总基因组DNA提取纯化方法综述[J].安徽农业科学,2007,35(25):7 789-7 791.
[30] 白飞荣,李辉,刘洋,等.一种冬虫夏草全基因组DNA的提取方法[J].工业微生物, 2014,44(1):19-22.
[31] SHANKAR K,CHAVAN L,SHINDE S,et al.An improved DNA extraction protocol from four in vitro banana cultivars[J].Asian J Biatechnol,2011,3(1):84-90.
[32] 周万青,李芳秋,王立魁,等.不同破壁方法提取真菌DNA的比较[J].临床检验杂志,2009,27(3):212-214.