肉及肉制品的掺假事件是国内外广泛关注的食品安全问题之一。肉类掺假不仅严重干扰肉类市场流通,侵害消费者权益,而且还影响消费者的健康甚至宗教信仰[1]。羊肉产品没有文化和宗教禁忌,因其具有肉质细嫩、味道鲜美,同时又营养丰富而备受消费者青睐。利用价格低廉的原料肉如鸭肉、猪肉、老鼠肉制造假羊肉,已是屡见报道[2]。因此,检测肉制品中羊源性成分含量的方法具有重要意义。
目前,针对肉源性成分的检验方法主要有电子鼻法、红外光谱法、酶联免疫吸附法、聚合酶链式反应(polymerase chain reaction,PCR)法等,其中实时荧光定量PCR法(real-time quantitative PCR,qPCR)的应用较为广泛[3]。但qPCR技术通过Ct值和标准曲线对起始模板只能达到相对定量分析,其检测灵敏度和准确性还达不到精细定量要求[4]。目前我国行业标准采用实时荧光PCR定性检测肉品中某种动物源性成分,但不能精确定量检测样品中所含有的肉源性成分含量,这使得在肉品掺假检测过程中,很难判定是故意掺假,还是生产或取样过程中意外污染所致[5]。因此,数字PCR(digital PCR,dPCR)作为一种对核酸进行精确定量的全新方法应运而生,因为其高通量、高灵敏度、高特异性被广泛运用于生物学、医学和食品科学等领域。dPCR可以分为3种类型,即微孔板数字PCR法、微流控芯片数字PCR 法和微滴数字PCR(droplet digital PCR,ddPCR)法。其中ddPCR法是利用油包水技术将其分割为数万个纳升级大小的微滴,每一个微滴都是一个独立的PCR反应体系,当PCR扩增完成后,利用微滴分析仪逐个对每个微滴进行检测。再根据统计学中的泊松分布原理分析阳性微滴的个数与比例即可得出靶分子的起始拷贝数或浓度,从而实现对低至单个拷贝核酸分子的绝对定量[6]。REN等[7]通过将ddPCR法与rPCR法应用于检测绵羊和山羊肉制品中的鸡源性成分,研究表明ddPCR法在不同热处理和超高的温度下具有良好重复性和稳定性,明显优于qPCR法。因此,对于食品源性成分的鉴定来说,ddPCR在定性和定量检测方面都具有明显的优势。但是,ddPCR应用于肉源性成分定量检测的难点在于如何将PCR扩增得到的基因拷贝数转换成肉的质量。如CAI等[8]利用ddPCR方法检测肉制品中猪肉和鸡肉质量时,发现生肉质量与DNA质量以及DNA质量和DNA拷贝数之间呈线性关系,能够根据DNA拷贝数建立计算生肉质量的公式。NOH等[9]采用ddPCR技术对海产品中阿拉斯加鳕鱼的含量进行测定,通过确立原始样品质量、DNA浓度和DNA拷贝数之间的线性关系,建立了基于DNA拷贝数的样本质量计算公式。由于该方法需要建立2组标准曲线进行两步转化,实验步骤增加,容易导致实验误差加大。因此,如何将基因拷贝数转换成肉的质量是值得深入研究的问题。
目前在有关肉品掺假检测报道中,多是检测某类肉中容易掺假的其他肉类,如在羊肉及其制品中检测掺入的鸭肉含量,但市售掺假肉中多为2种或2种以上肉类的掺杂混合,这就给定量检测带来困难。由于直接对肉品中羊肉ddPCR精准定量的报道较少,因此本研究将通过向样品中添加牛肉作为内标,采用ddPCR对羊肉的单拷贝基因进行定量检测,根据基因拷贝数建立食品中羊肉源性成分ddPCR的定量检测方法,以便为掺假肉的监督管理提供一些技术参考。
1.1.1 实验材料与试剂
主要样品:山羊肉、绵羊肉、鸭肉、水牛肉、黄牛肉、牦牛肉、猪肉、鹿肉、狐狸肉、貂肉、兔肉、鸡肉、鹅肉、鸽肉、火鸡肉、鳕鱼、大黄鱼、虹鳟鱼、银鳕鱼、大菱鲆,以上样品均由四川出入境检验检疫局提供,-70 ℃保存。
Premix Ex Taq(probe qPCR)预混液,TaKaRa公司;蛋白酶K(20 mg/mL),拓样生物有限公司;Droplet Generator DG8 CartndgeDroplet Gene rator,BIO-RAD公司;ddPCR Supermix for Probes (no dutp),BIO-RAD公司;ddPCR Droplet Generation Oil,BIO-RAD公司;DG8 GasketPierceable Foil Heat Seal,BIO-RAD公司。
1.1.2 仪器与设备
FastPrep样品研磨器,印度MP公司;Thermomixer Comfort恒温混匀仪,德国eppendorf公司;X3R高速冷冻离心机,Thermo公司;CFX96 Real-time PCR 仪,Bio-Rad公司;QX100 Droplet Digital PCR系统,BIO-RAD公司;
1.2.1 样品制备
取新鲜羊肉样品的肌肉组织,绞肉机绞碎后于100 ℃烘干至恒重。在研钵中加入液氮,将烘干样品研磨至粉末状,室温条件下保存于干燥皿中。准确称取处理后的肉粉100 mg,采用实验室自制的动物组织基因组磁珠法提取试剂盒进行DNA提取,所得基因组DNA质量通过琼脂糖凝胶电泳和Nanodrop ND-2000核酸蛋白定量仪评价[10]。
1.2.2 羊肉、牛肉源性引物探针筛选设计
1.2.2.1 引物探针的选择与设计
分别选定羊、牛的单拷贝生长激素基因GH(序列号分别为NC-019468、NC-000077)设计引物探针,再根据NCBI等网站中公布的不同物种间各基因序列进行序列比对,设计的引物探针委托成都擎科梓熙生物技术有限公司进行合成。
1.2.2.2 特异性验证
分别提取山羊、绵羊、水牛、黄牛、牦牛、猪、鹿、狐狸、鹅、兔、鸽、鳕鱼、大黄鱼、鸭、火鸡、虹鳟鱼、貂、鸡、银鳕鱼、多宝鱼等动物肌肉DNA作为模板,分别对所设计筛选的羊源性、牛源性引物进行特异性验证。同时设立用ddH2O代替核酸的1个阴性对照验证其对引物的特异性。当阴性对照为阴性时整个试验有效,可进一步判定结果。实验重复3次,每次2个平行。
1.2.2.3 灵敏度验证
将提取的羊肉、牛肉DNA模板分别按梯度进行稀释,均稀释至浓度为100、 10、1、0.1、0.01、0.001 ng/μL,进行qPCR扩增,实验重复3次,每个梯度2个平行,验证该方法灵敏度。
1.2.3 ddPCR定量方法的建立
1.2.3.1 ddPCR操作步骤
具体程序与步骤为:将各物种的反应体系转移至微滴生成卡中,并在反应用油加样孔加入70 μL的微滴成专用油;加样完成后,将微滴生成卡转移至微滴生成器中进行微滴的生成;将生成微滴后的40 μL反应体系转移至96孔板中并封膜进行扩增,PCR扩增程序为95 ℃预变性30 s;95 ℃变性15 s,60 ℃退火延伸60 s,50个循环;98 ℃加热10 min使酶灭活。扩增结束后,将96孔板取出,置于Droplet reader微滴读取仪中进行微滴荧光读取;采用FAM/VIC双通道荧光采集微滴信号,并通过分析热点图确定荧光阈值限以确定阴性微滴和阳性微滴。
以提取的羊DNA(约50 ng/μL)为模板,用ddPCR扩增后的拷贝数确定羊源性特异性基因引物和探针最优浓度,以及最佳退火温度。最后用微滴分析仪对扩增产物进行计数分析,最终以平均值确定羊肉核酸标准品的基因拷贝数。通过比较ddPCR微滴生成数,微滴分布状态、微滴荧光信号强度等来确定优化的结果。
1.2.3.2 ddPCR反应体系和程序
ddPCR扩增体系为:ddPCRTMSupermix for probes,10 μL;上游引物(10 p),1.8 μL;下游引物(10 p),1.8 μL;探针(10 p),0.5 μL;模板(50 ng/μL),2.0 μL;ddH2O,3.9 μL。
ddPCR扩增程序为:95 ℃预变性30 s;95 ℃变性15 s,60 ℃退火延伸60 s,50个循环;98 ℃加热10 min使酶灭活,扩增结束。
1.2.3.3 羊、牛源性引物探针的ddPCR特异性验证
以提取的羊肉基因组DNA为研究对象,牛、猪、驴、马、狐狸、兔DNA为对照模板进行ddPCR扩增,验证引物探针的ddPCR特异性,同时设置阴性对照孔。
以提取的牛基因组DNA为研究对象,山羊、驴、猪、鹿、貂、鸭肉的DNA为对照模板进行ddPCR扩增,验证引物探针的ddPCR特异性,同时设置阴性对照孔。
反应程序及反应体系参照1.2.2.2。
1.2.3.4 ddPCR反应条件的优化
ddPCR体系中羊、牛源性引物浓度优化:羊、牛源性引物的ddPCR体系的引物浓度优化处理方式相同,PCR反应体系20 μL:ddPCRTM Supermix for Probes 10 μL,上、下游引物各1μL,设置上下游引物摩尔浓度0.5、0.6、0.7、0.8、0.9 μmol/L,加入探针0.5 μL(0.4 μmol/L),DNA模板2 μL,加ddH2O补足20 μL体积。在体系充分混匀后加入Bio-Rad公司的微滴生成卡中,油包水微滴生成按照说明书进行。将生成的微滴全部转入96孔PCR反应板中,进行扩增。扩增条件确定为:预变性95 ℃,10 min;变性94 ℃,30 s;60 ℃,1 min,40个循环;固化微滴98 ℃,10 min。
ddPCR体系中羊、牛源性探针浓度优化:羊、牛源性引物的ddPCR体系的引物浓度优化处理方式一致,PCR反应体系20 μL:ddPCRTM Supermix for Probes 10 μL,上、下游引物各1 μL(0.9 μmol/L),探针0.5 μL,设置探针摩尔浓度梯度为100、150、200、250、300 nmol/L,DNA模板2 μL,加ddH2O补足20 μL体积。应在体系充分混匀后加入Bio-Rad公司的微滴生成卡中,油包水微滴生成按照说明书进行。将生成的微滴全部转入96孔PCR反应板中,进行扩增。扩增条件确定为:95 ℃预变性10 min;94 ℃变性30 s;60 ℃退火延伸1 min,40个循环;固化微滴98 ℃,10 min。
ddPCR体系中羊、牛源性引物退火温度优化:羊、牛源性引物的ddPCR反应程序的退火温度优化处理方式一致,设置退火温度为58、59、60、61、62、63、64和65 ℃等8个梯度,最后通过比较分析ddPCR的拷贝数和微滴情况来分别确定羊和牛源性特异性引物ddPCR最佳退火温度。每个样品每个处理做2个平行。
ddPCR灵敏度验证:将提取的羊肉DNA 模板进行10倍梯度稀释,分别稀释至浓度为10、1、0.1、0.01、0.001 ng/μL,进行ddPCR灵敏度的验证。ddPCR实验重复3次,每次2个平行。
ddPCR定量方法的建立:肉的质量与样品DNA溶液中的基因的拷贝数及单位质量的基因拷贝数有关,如公式(1)所示:
Q=M×C
(1)
式中:M,肉的质量;Q,基因拷贝数;C,单位质量肉的基因拷贝数)
不同肉种的质量比按公式(2)计算:
(2)
由于进行固定条件下干燥处理,同一肉种的细胞密度和基因组大小是固定的,因此,Cb/Ca为常数命名为k,只要测得基因拷贝数,即可获得2组分比例[11]。
以羊肉作为研究对象,将羊肉与鸭肉、猪肉按表1配制成含有1%、4%、10%、30%、50%、60%和95%羊肉成分和10 mg内标牛肉的混合样品(共110 mg干物质),分别取5.5 mg干物质提取DNA,进行ddPCR检测,分别测得羊源性成分和内标牛源性成分基因拷贝数,计算K值和相对标准偏差(relative standard deviation,RSD),构建质量与拷贝数的数学关系。每个样品重复3次,每次2个平行,相关系数R2的计算用Excel自动生成。
1.2.4 定量方法灵敏度验证
为验证ddPCR内标定量方法的灵敏度,将羊肉干物质分别按0.5%、0.25%、0.01%与鸭肉、猪肉的干物质混合后加10%内标物质牛肉干物质,进行混合后提取DNA,并对羊源性成分及内标牛源性成分进行双重ddPCR检测。具体混合比例见表2。
1.2.5 定量线性范围和方法的重复性和准确性验证
针对检测体系,按照表3制备不同掺假含量和不同掺假种类的模拟混合样品,提取 DNA后进行ddPCR定量检测,测得的羊与内标拷贝数带入公式(1)计算,得出羊的质量。实验重复3次,每次2个平行。
表1 ddPCR定量方法的建立中各肉样混合比例
Table 1 The proportion of each meat sample in the establishment of ddPCR quantitative method
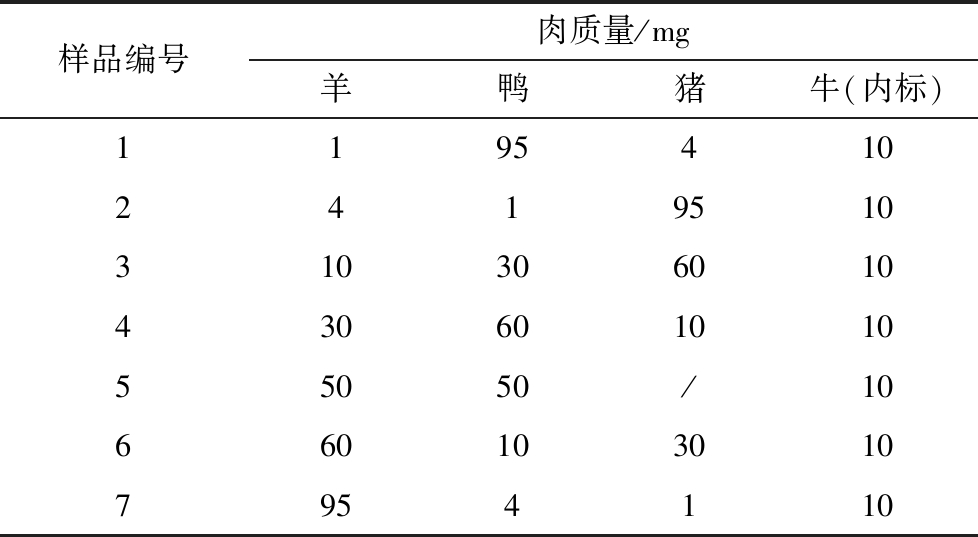
样品编号肉质量/mg羊鸭猪牛(内标)1195410241951031030601043060101055050/106601030107954110
注:“/”表示不含该成分(下同)
表2 ddPCR内标定量方法实际灵敏度验证的样品混合比例
Table 2 ddPCR internal standard quantitation method actual sensitivity verification sample mixing ratio
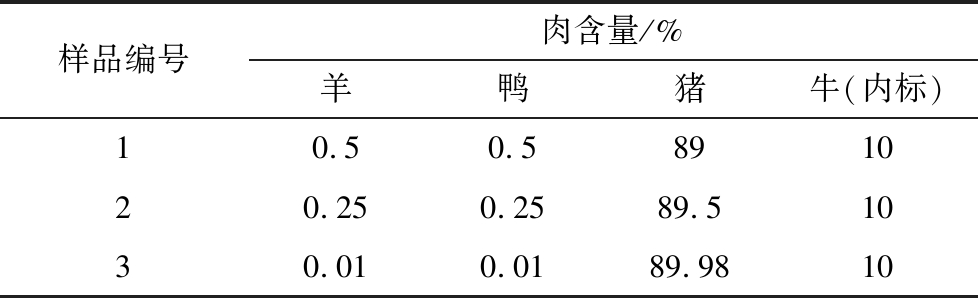
样品编号肉含量/%羊鸭猪牛(内标)10.50.5891020.250.2589.51030.010.0189.9810
表3 模拟混合样品的混合比例(内标法)
Table 3 Simulating the mixing ratio of mixed samples(internal standard method)
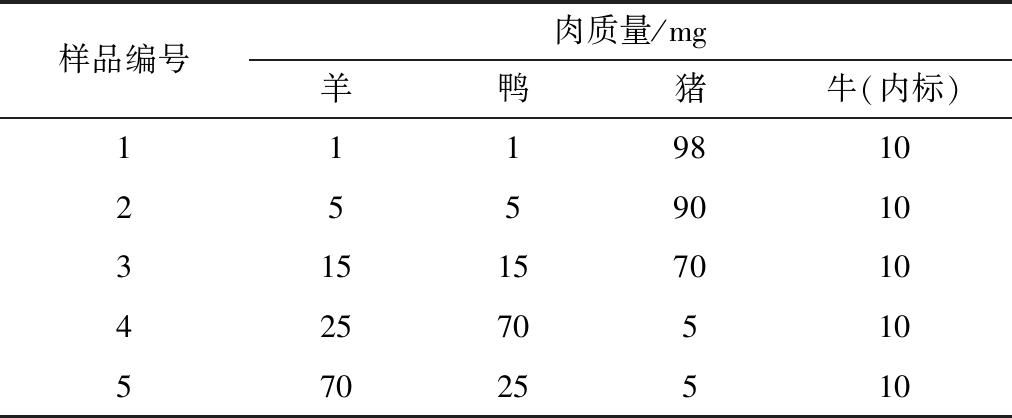
样品编号肉质量/mg羊鸭猪牛(内标)111981025590103151570104257051057025510
1.2.6 ddPCR检测市售样品羊源性成分的含量
为了验证所建立的ddPCR定量方法在实际羊肉样品检测中的可行性,以2018~2019年成都海关技术中心动检实验室保存的四川省内随机抽取的28个羊肉及其制品作为检测对象,样品主要来源于超市及农贸市场,包括13个羊肉片(卷)、8个羊肉串、7个羊肉样品。准确称取经前处理干燥后的1 g待检样品,加入100 mg内标干物质混合均匀后,取7 mg进行DNA提取,采用现行定性检测标准SN/T 2051—2008食品、化妆品和饲料中牛羊源性成分检测方法进行定性检测,检测结果显示为阳性的样品再利用建立的ddPCR内标定量方法进行定量检测,进一步验证所建方法的准确性和应用能力。
2.1.1 单拷贝基因的筛选
参考相关文献,并查询NCBI上基因的相关信息,确定以生长激素基因(GH)为目的基因,分别进行羊、牛源性引物探针的设计,羊肉、牛肉的GH基因在染色体上的位置及数量分别如图1所示。
2.1.2 不同物种GH基因序列比对
利用序列分析软件DNA Man6.0 对山羊、绵羊、水牛、瘤牛、黄牛的生长激素基因序列进行比对分析,选取羊的特异性保守区域进行引物探针的设计,各物种序列比对结果如图2所示。

图1 羊肉、牛肉的单拷贝GH基因
Fig.1 Single copy GH gene of bovine and sheep
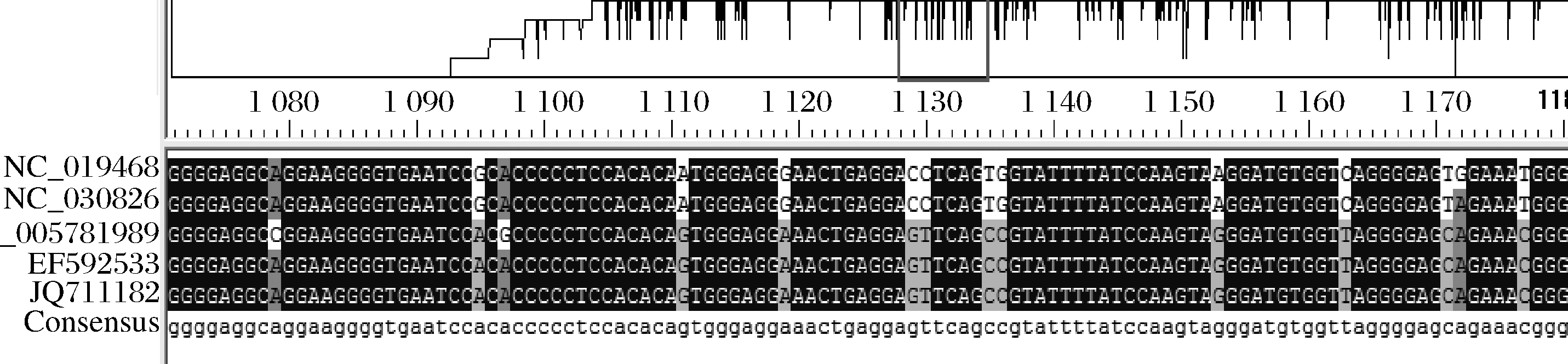
图2 羊与其他物种GH基因序列比对
Fig.2 Comparison of GH gene sequences between sheep and other species
注:左侧序列号从上到下依次为山羊、绵羊、水牛、黄牛、瘤牛。
利用序列分析软件DNA Man6.0 对水牛、瘤牛、黄牛、牦牛、山羊、绵羊的生长激素基因序列进行比对分析,选取牛的特异性保守区域进行引物探针的设计,各物种序列比对结果如图3所示。

图3 牛肉与其他物种GH基因序列比对
Fig.3 Comparison of GH gene sequences between cattle and other species
注:左侧序列号从上到下依次为水牛、黄牛、瘤牛、牦牛、山羊、绵羊
2.1.3 引物探针的设计
利用软件Oligo 6.0、DNAMan对牛的引物探针进行设计与分析,表4、表5分别是设计的羊源性引物探针和牛源性引物探针的基本信息。
表6和表7分别为设计的羊、牛源性GH基因引物探针的序列。合成的引物进行PCR扩增后进行琼脂糖凝胶电泳检测,最后对产物进行测序,测序结果进行Blast同源性数据库比对后结果显示同源性均在96%以上。
2.2.1 羊源性引物探针特异性验证
由图4可以看出,在引物特异性实验中,只有绵羊和山羊的DNA检测到了扩增信号,平均Ct值分别为24.93和25.77,未出现非特异性扩增,说明所设计引物的特异性良好,能同时对绵羊和山羊成分进行检测。
表4 羊源性引物探针基本信息
Table 4 Basic information of sheep primers probe
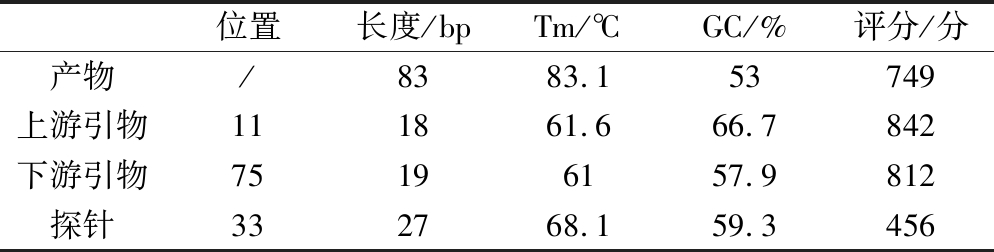
位置长度/bpTm/℃GC/%评分/分产物/8383.153749上游引物111861.666.7842下游引物75196157.9812探针332768.159.3456
表5 牛源性引物探针基本信息
Table 5 Basic information of bovine primers probe
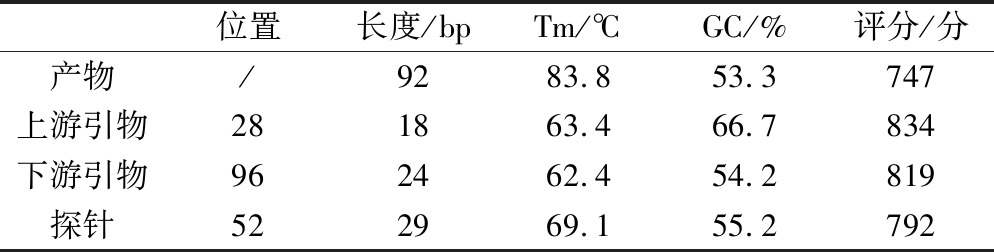
位置长度/bpTm/℃GC/%评分/分产物/9283.853.3747上游引物281863.466.7834下游引物962462.454.2819探针522969.155.2792
表6 羊GH基因引物探针序列
Table 6 Primers probe sequence of GH gene in sheep

物种基因引物和探针序列号GAATCCGCACCCCCTCCA羊GHAGGATGTGGTCAGGGGAGTNC_019468FAM-ATGGGAGGGAACTGAGGACCTCAGTGG-BHQ1
表7 牛肉的GH基因引物探针序列
Table 7 Bovine GH gene primer probe sequence

物种基因引物和探针序列号GTGAATCCACGCCCCCTC牛GHGGATGTGGTTAGGGGAGCAGAAACNW_005781989HEX-AGTGGGAGGAAACTGAGGAGTTCAGCCGT-BHQ1
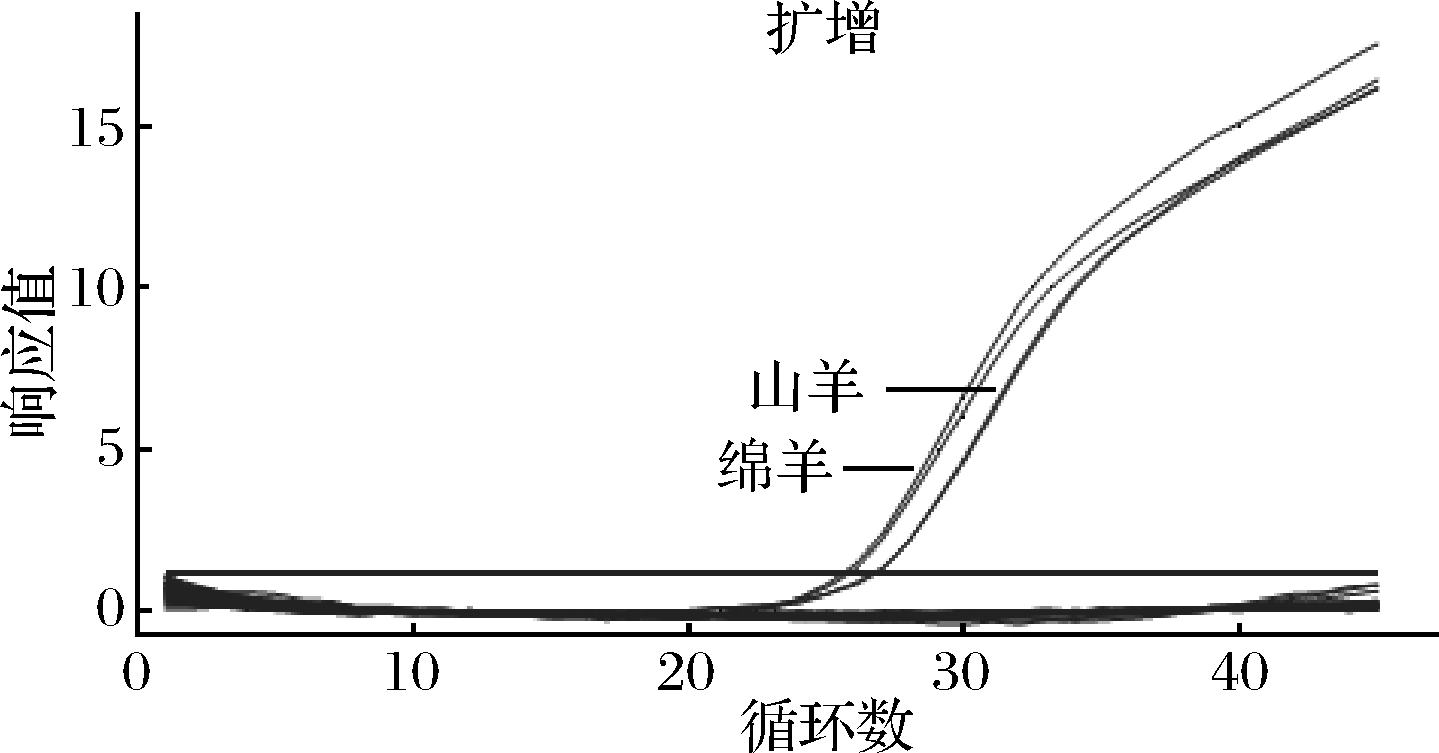
图4 羊源性引物探针qPCR特异性验证
Fig.4 qPCR specificity verification of sheep primers
2.2.2 牛源性引物探针特异性验证
由图5可以看出,牛源性引物的特异性实验中,只有水牛、黄牛和牦牛的DNA检测到了扩增信号,平均Ct值分别为26.63、28.05和30.89,未出现非特异性扩增,说明所设计的引物特异性良好且能同时对水牛、黄牛和牦牛成分进行检测。
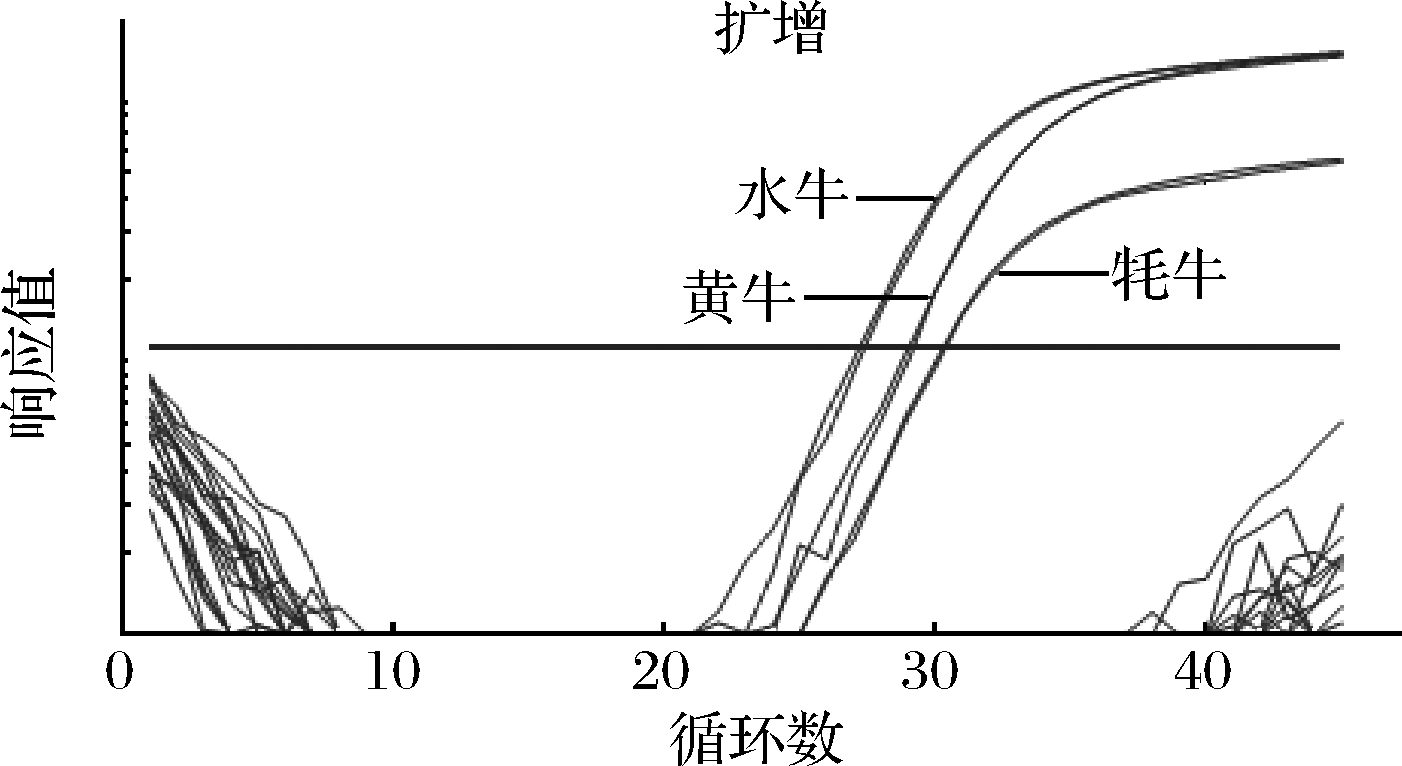
图5 牛源性引物探针qPCR特异性
Fig.5 Bovine primers probe qPCR specificity
羊肉和牛肉DNA的ddPCR扩增结果分别如图6、图7所示,空白对照未出现扩增,说明体系未污染,且每个样品生成微滴数均大于12 000个,符合泊松分布计算需要。羊引物探针特异性良好,只扩增出羊DNA,其他非目标物种肉样均没有出现扩增;牛源性引物探针特异性良好,只有牛DNA发生扩增,未出现非特异性扩增。表明该ddPCR鉴别体系特异性良好,2对引物均可用于后续ddPCR定性和定量检测。

图6 羊源性引物探针ddPCR特异性
Fig.6 Specificity of sheep primers ddPCR
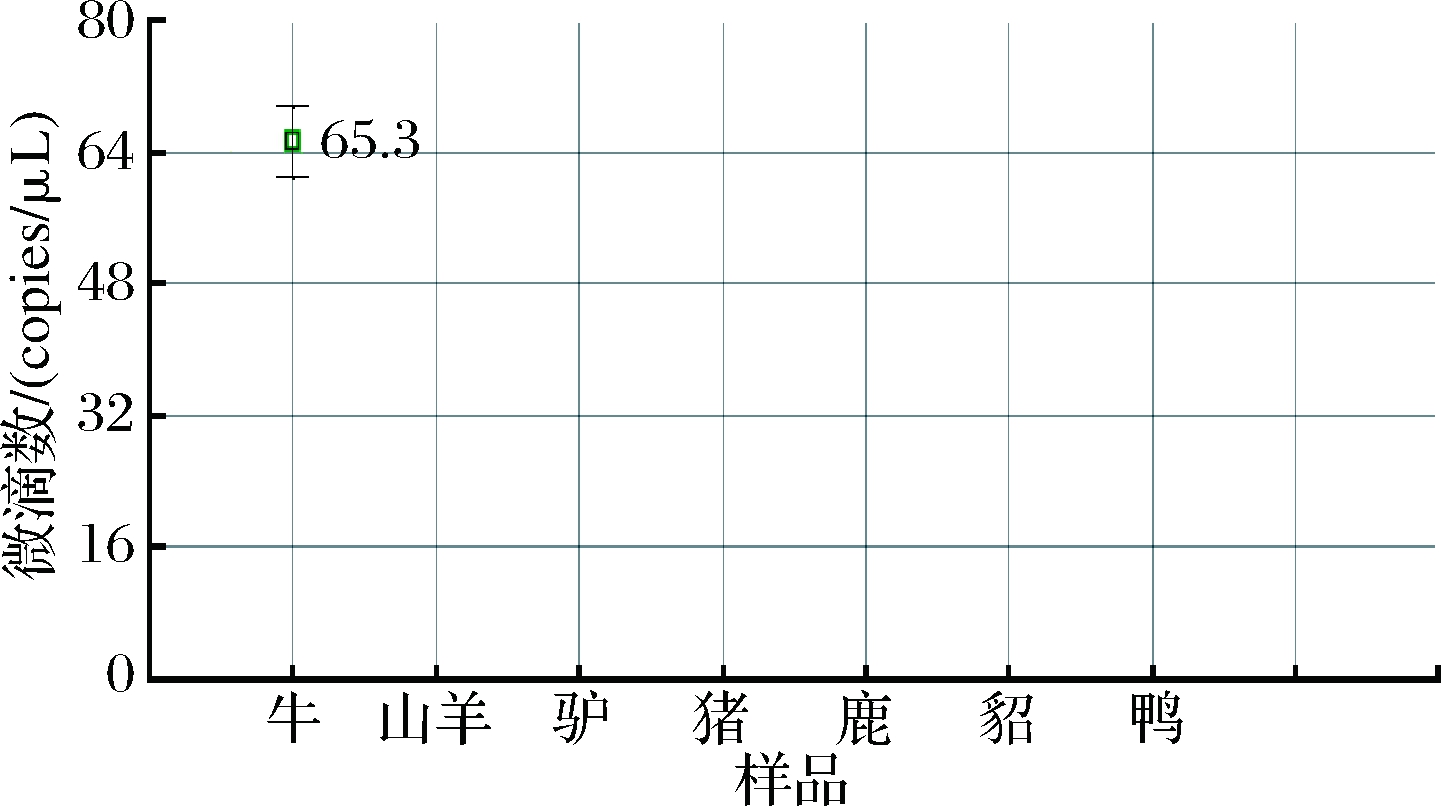
图7 牛源性引物探针ddPCR特异性
Fig.7 Specificity of bovine primer probe ddPCR
ddPCR反应条件的优化结果为:20 μL反应体系中引物体系终摩尔浓度0.9 μmol/L,探针体系终摩尔浓度0.25 μmol/L,ddPCRTM Supermix for Probes 10 μL,DNA模板2 μL,最后用ddH2O补足至20 μL。反应程序最佳退火温度为60 ℃。
由图8可看出,当DNA质量浓度为0.01 ng/μL时,检测为0.05 copies/μL,质量浓度为0.001 ng/μL时,未出现扩增,因此ddPCR检测羊DNA的灵敏度为0.01 ng/μL,灵敏度满足定量检测需要。
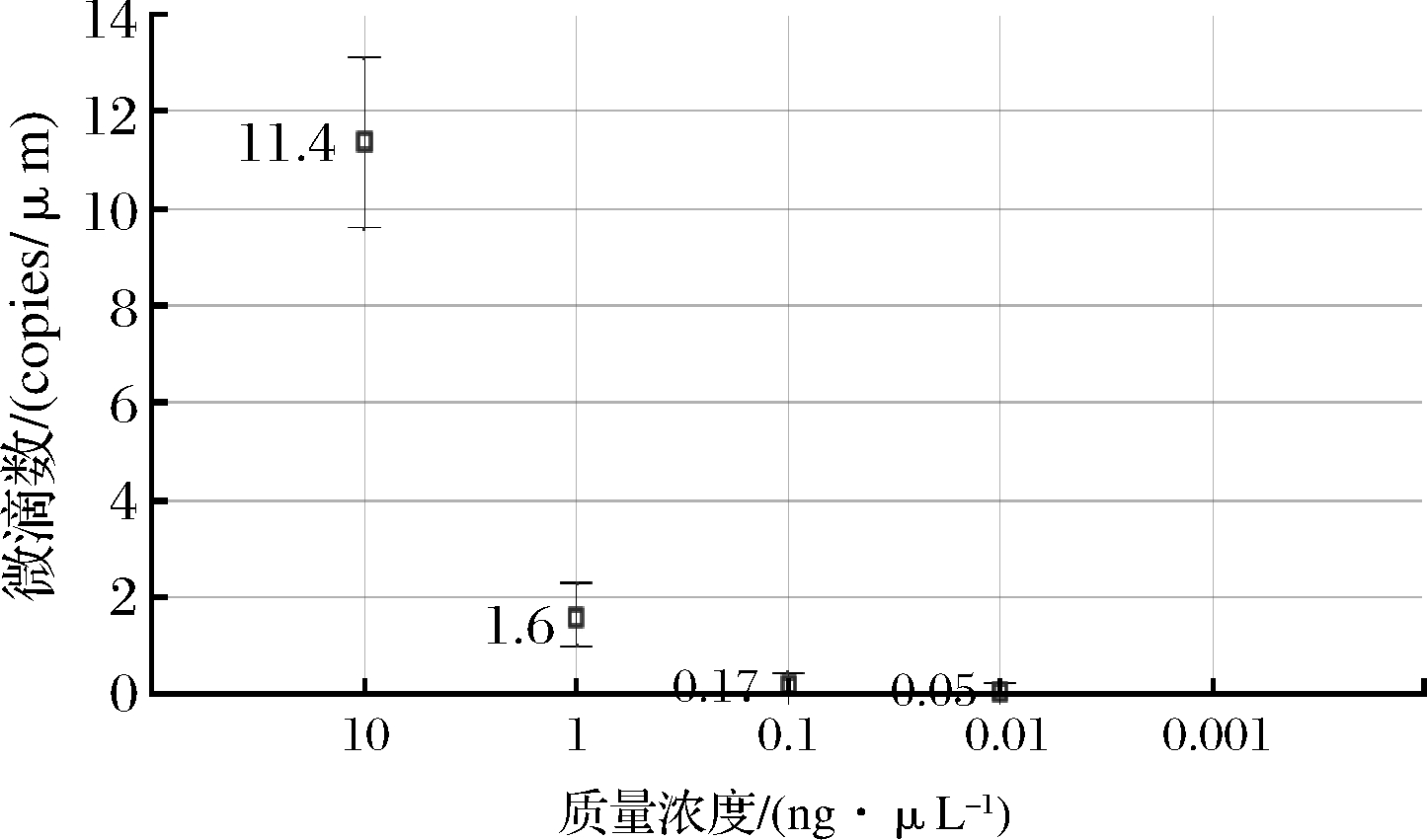
图8 ddPCR检测羊肉的灵敏度
Fig.8 Detecting the sensitivity of mutton by ddPCR
羊源性成分的检测结果如图9所示,羊肉样品含量在1%~95%,羊肉的拷贝数随羊肉含量的增大而增加;内标牛肉的拷贝数检测结果如图10所示,随羊肉含量的增大,牛肉的拷贝数几乎无差异。
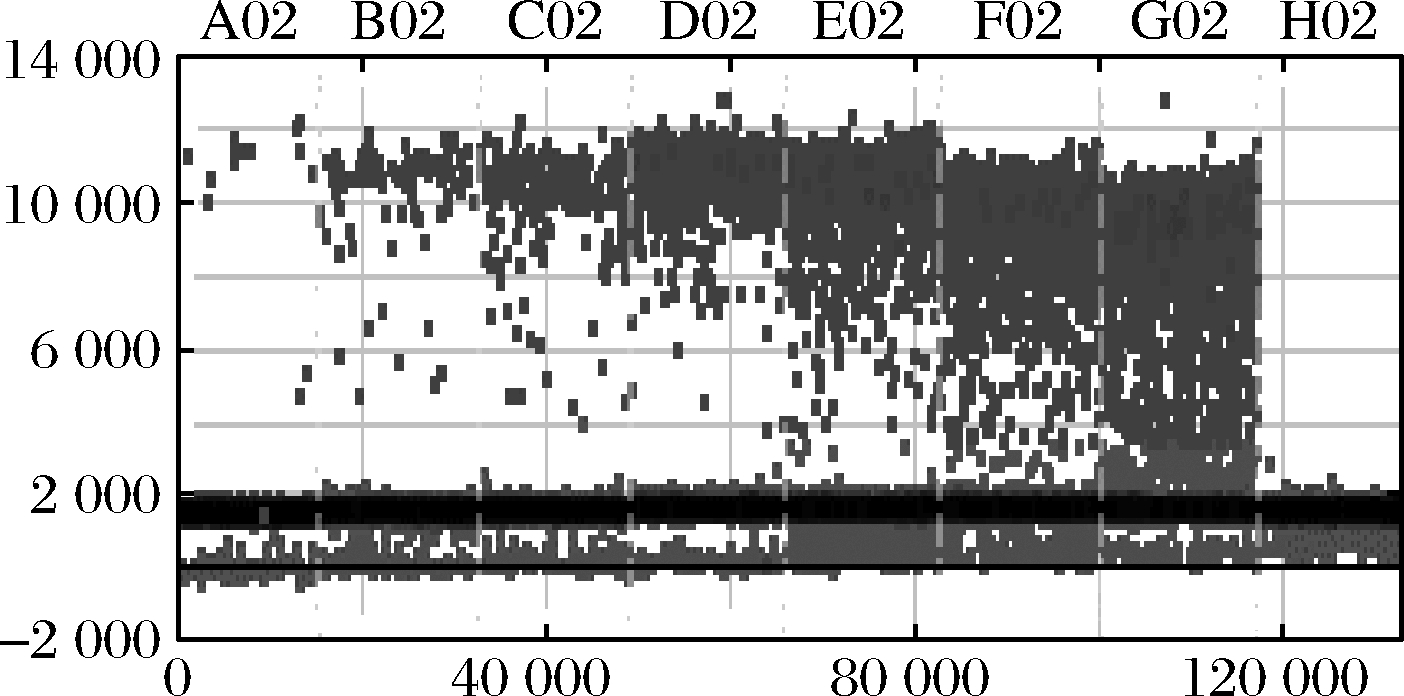
图9 ddPCR内标法检测1%~95%的羊肉拷贝数
Fig.9 ddPCR internal standard method for detecting the copy number of 1%-95% of mutton
注:A02-1%; B02-4%; C02-10%; D02-30%; E02-50%; F02-60%; G02-95%; H02-阴性对照
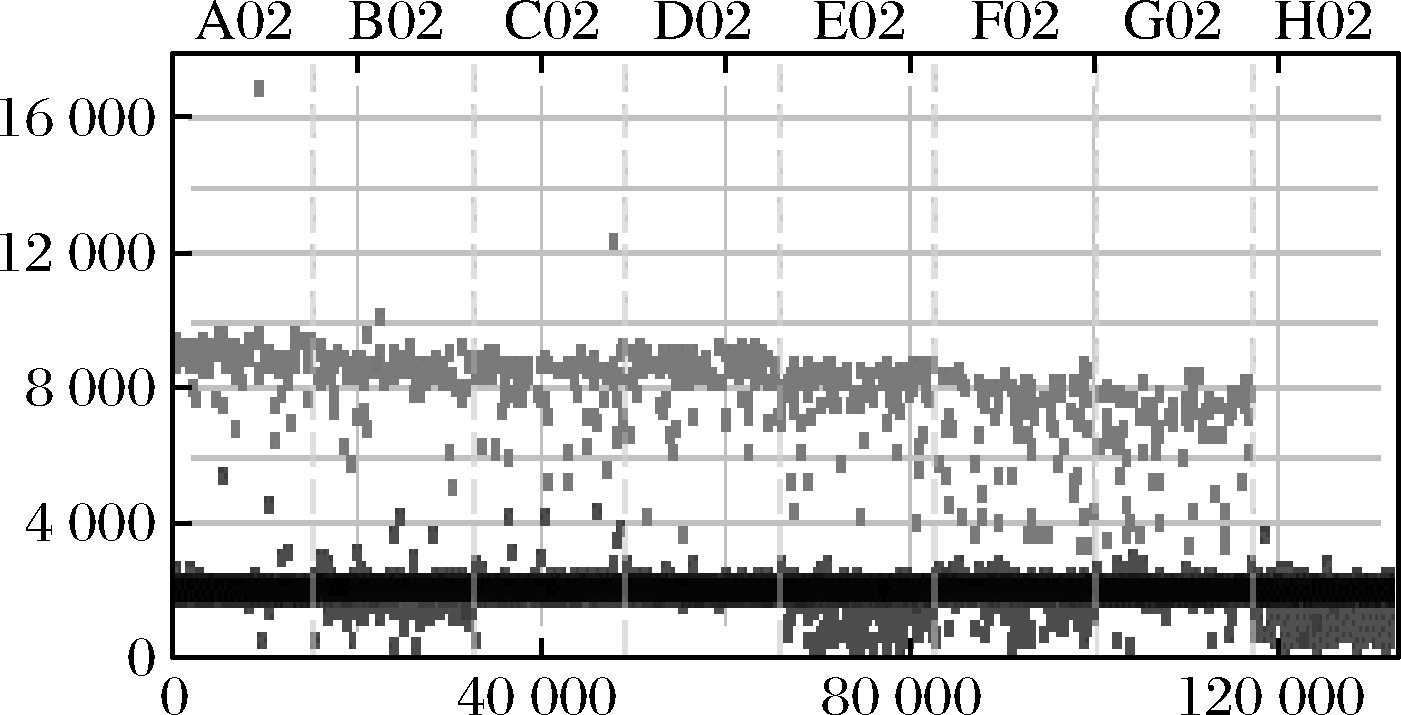
图10 ddPCR检测10%内标牛肉的拷贝数
Fig.10 ddPCR detects 10% of internal standard beef copy number
得到羊肉靶基因拷贝数与内标物种牛靶基因拷贝数后计算2个拷贝数比值,以该比值与对应的羊肉实际质量作为横、纵坐标绘制标准曲线,标准曲线如图11所示。羊肉的含量与羊肉与内标牛肉的基因拷贝数之比呈线性响应,相关系数R2=0.997 3,计算如公式(3)所示:
y= 0.042 5x+0.015 2
(3)
式中:y表示羊肉的质量,x表示羊与内标牛肉的基因拷贝数之比,通过该公式可将羊肉的基因拷贝数转化为质量,该回归方程即可作为羊肉质量的计算方程。
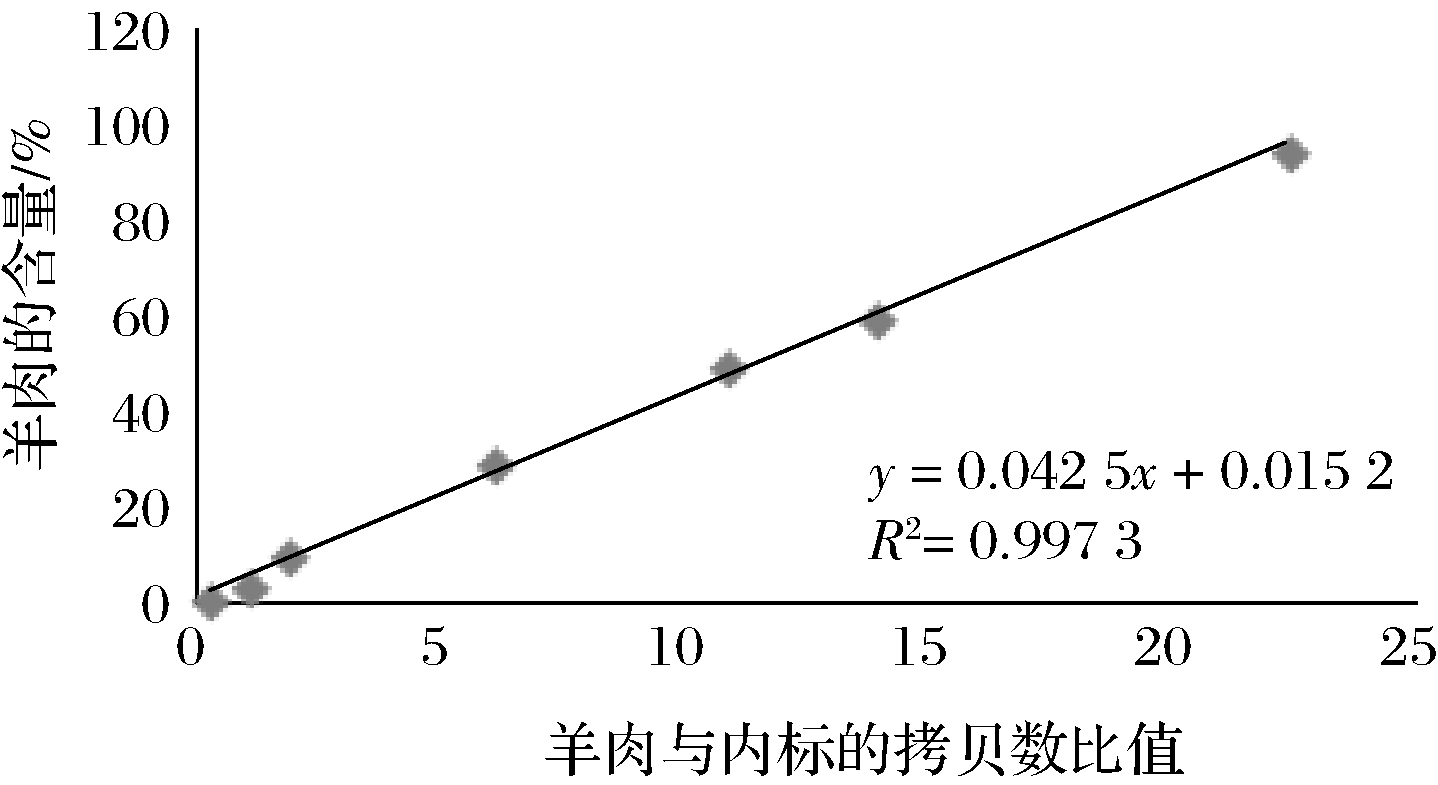
图11 羊肉与内标的拷贝数比值和羊肉含量的函数关系
Fig.11 The relationship between the copy number ratio of mutton and internal standard and the content of mutton
由图12可看出,空白对照未出现扩增,说明体系未污染,且每个样品生成的微滴数均大于15 000个,满足泊松分布统计需要。当羊肉含量为0.01%时,检测结果为0.12 copies/μL。结果表明,该方法具有良好的灵敏度,可以检测出含有0.01%羊肉的样品。

图12 ddPCR内标法检测羊肉的实际灵敏度验证
Fig.12 Verification of actual sensitivity of mutton by ddPCR internal standard method
测得的羊肉拷贝数和牛肉拷贝数分别如图13、图14所示。

图13 ddPCR定量方法的准确性及重复性验证
Fig.13 Accuracy and repeatability verification of ddPCR quantification method
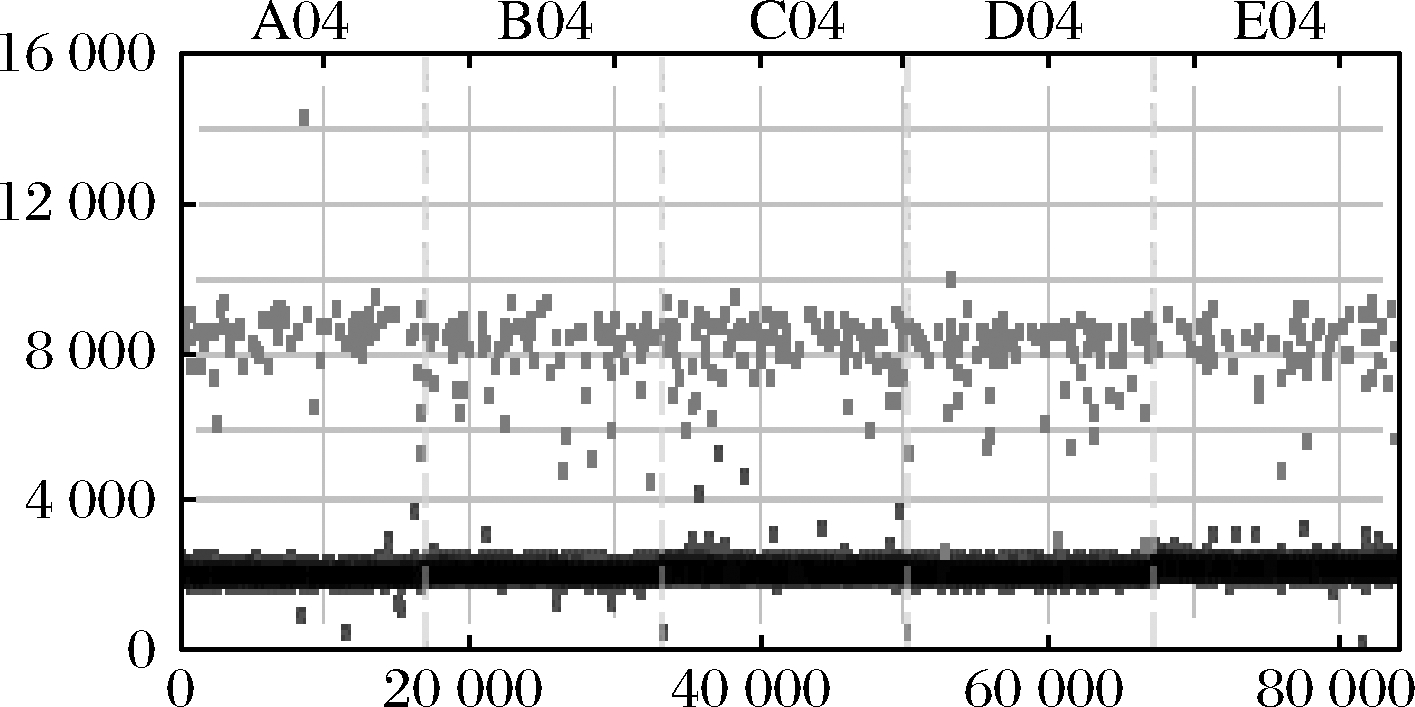
图14 ddPCR检测10%内标牛肉的拷贝数
Fig.14 ddPCR detection of 10% internal standard beef copy number
羊肉与牛肉的拷贝数之比见表8。由表8可看出5个处理的样品6次测量值均有很好的重复性,RSD均小于8.7%。当羊肉含量为1%时,定量的相对误差较大,为155.8%;当羊肉含量大于5%时,测定值与实际值相对误差较小,均在6.6%以内。说明该方法在羊肉含量5%以上时,具有良好的准确性和重复性。
表8 ddPCR定量检测方法检测混合肉中羊肉含量的最低检测限、重复性及准确性
Table 8 Detection of the minimum detection limit, repeatability and accuracy of mutton content in mixed meat by ddPCR quantitative detection method
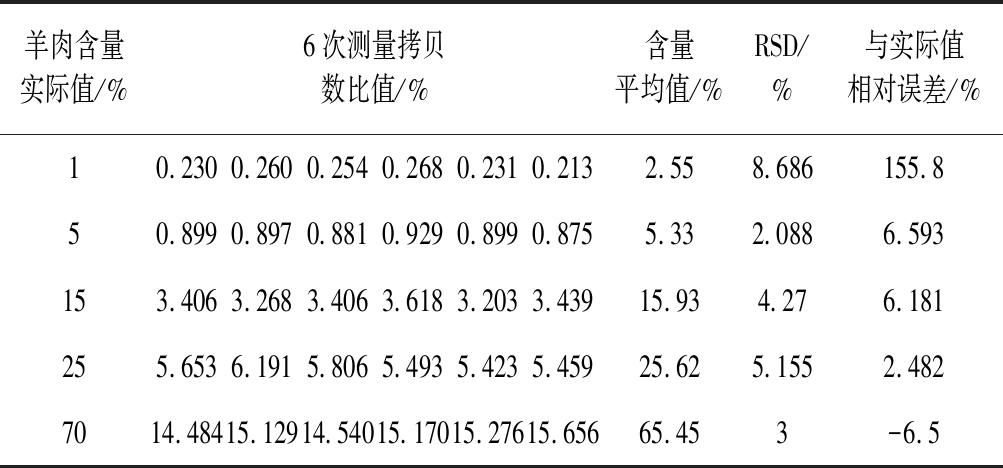
羊肉含量实际值/%6次测量拷贝数比值/%含量平均值/%RSD/%与实际值相对误差/%10.2300.2600.2540.2680.2310.2132.558.686155.850.8990.8970.8810.9290.8990.8755.332.0886.593153.4063.2683.4063.6183.2033.43915.934.276.181255.6536.1915.8065.4935.4235.45925.625.1552.4827014.48415.12914.54015.17015.27615.65665.453-6.5
利用建立的ddPCR绝对定量方法对样品中的羊源性成分进行定量检测,结果表9所示。由表9可知,农贸市场出售的2个羊肉片(卷)和1个羊肉串检测出了非羊源性成分。2个被检出非羊源性成分的羊肉片(卷)样品中羊肉所占比例均<95%,说明样品被其他动物成分污染,这可能是加工环节或者样品流通中出现的无意沾染,而非故意添加。1个被检出非羊源性成分的羊肉串样品中羊肉所占比例仅为65.1%,可确定为掺假,这表明了现有的市售样品存在一定的掺假情况。
近年来,以核酸为标志物的动物种类鉴别检测方法获得了快速发展,尤其dd PCR技术的出现为qPCR检测核酸提供了一种独特的方法,尤其适用于低水平检测。该方法不需要依靠标准曲线或参照基因,可直接得出DNA拷贝数,是对样品的绝对定量[12]。MORISSET等[13]研究了ddPCR在食品和饲料样品常规分析中的适用性,认为ddPCR检测在低靶浓度下显示出更好的重复性,它的定性和定量检测限比传统定量PCR低,能分别达到5个和6个拷贝数,而不需要标准曲线。因此,ddPCR在准确性、特异性和重复性较之于传统定量PCR存在更大的优势,在肉源成分检测方面已经取得一定的成绩。由于ddPCR对待检肉样的精确定量是利用DNA浓度作为中间变量,从而确定肉样质量和微滴拷贝数之间的标准曲线。如SHEHATA等[14]采用ddPCR方法,对食品和饲料样品中牛、猪、鸡和火鸡的DNA进行定量检测,是先将基因拷贝数转换为DNA质量,再从DNA质量换算为肉的质量。苗丽等[5]和陈晨等[15]采用ddPCR方法对肉制品中羊肉和猪肉定量分析,也是以DNA浓度作为中间换算值,得到肉质量和DNA 拷贝数之间的换算关系。但BALLIN等[16]在研究肉类产品的物种标签时认为,相同质量的不同肉种其DNA基因拷贝数不相同,如果直接将基因拷贝数之比作为肉质量之比,结果会出现较大差异。因此,本研究选择直接利用2种肉基因拷贝数之比的固定值,一步实现了基因拷贝数之比与肉质量之比的转化,而实现对羊源性成分的精确定量。
表9 ddPCR对市售样品的检测结果
Table 9 Detection of actual samples by ddPCR

样品编号样品来源样品名称检测结果《SN/T 2051—2008》(阴性-,阳性+)ddPCR内标定量法(含量/%)1羊肉片(卷)1+>952羊肉片(卷)2+>953羊肉片(卷)3+>954超市羊肉片(卷)4+>955羊肉片(卷)5+>956羊肉片(卷)6+>957羊肉片(卷)7+>958羊肉片(卷)8+>959羊肉片(卷)9+92.410羊肉片(卷)10+91.711农贸市场羊肉片(卷)11+>9512羊肉片(卷)12+>9513羊肉片(卷)13+>9514羊肉串1+>9515超市羊肉串2+>9516羊肉串3+>9517羊肉串4+>9518羊肉串5+65.119农贸市场羊肉串6+>9520羊肉串7+>9521羊肉串8+>9522半熟羊肉1+>9523超市净羊后腿肉2+>9524冻分割大块羊肉3+>9525羔羊肉4+>9526法式羊排5+>9527农贸市场分割羊肉6+>9528羊肉7+>95
此外,线粒体基因作为靶基因在物种的定性检测中得到了广泛应用,但线粒体基因在同一物种的不同组织中含量差异巨大,这给定量检测带来困难。FLOREN等[17]认为以线粒体DNA(mtDNA)为靶点进行物种量化具有局限性,因为每个细胞的mtDNA含量有5倍的组织间变异,容易导致物种DNA含量被低估或高估。而单拷贝基因与线粒体基因相比,具有拷贝数少且数量相对恒定等特点,不会因为样品部位的不同导致定量结果的偏差,适合于对肉类产品进行定量检测。KOPPEL等[18]利用单拷贝基因设计特异性引物,定量分析了食品中牛、猪等动物源性成份,取得了满意效果。因此,本研究选择单拷贝持家基因作为靶基因,利用ddPCR技术对肉中羊源性成分进行精确定量。
由于目前市场上肉制品种类繁多,仅从感官上无法识别肉制品的真实性,更无法识别肉制品是人为故意掺假还是无意污染。为了验证本实验建立ddPCR法的准确性和实用性,将ddPCR法与SN/T 2051-2008《食品、化妆品和饲料中牛羊源性成分检测方法》对比进行市售样品的检测,结果表明ddPCR能够对样品中羊源性成分的含量比例进行了准确定量,这有利于食品检测部门对样品中肉源性成分的真伪做出判定。
本研究建立了基于DNA拷贝数与样品质量间线性关系,对羊源性成分进行ddPCR内标定量的方法,实现了从靶基因拷贝数到样品质量间的一步转化。实验证明该方法在检测出0.01%的羊肉源性成分时,检测结果达0.12 copies/μL;对于含量5%以上的羊肉源性成分,具有很好的准确性和重复性。总的来看,本方法在肉及肉制品中羊肉源性成分检测和掺假鉴别方面具有较大的应用潜力。
[1] O’MAHONY P J. Finding horse meat in beef products--a global problem[J]. QJM: Monthly Journal of the Association of Physicians, 2013, 106(6): 595-597.
[2] KUMAR A, KUMAR R R, SHARMA B D, et al. Identification of species origin of meat and meat products on the DNA basis: a review[J]. Critcal Reviews in Food Science and Nutrition, 2015, 55(10): 1 340-1 351.
[3] TIAN X, WANG J, SHEN R, et al. Discrimination of pork/chicken adulteration in minced mutton by electronic taste system[J]. International Journal of Food Science & Technology, 2019, 54(3):670-678.
[4] CAI Y C, HE Y Ping, LYU R, et al. Detection and quantification of beef and pork materials in meat products by duplex droplet digital PCR[J]. Plos One, 2017, 12(8): e091 949.
[5] 苗丽,张秀平,陈静,等.肉制品中羊源性成分微滴数字PCR法定量检测方法的研究[J]. 食品工业科技, 2016, 37(4): 73-76.
[6] DRUML B, MAYER W, CICHNA-MARKL M, et al. Development and validation of a Taq Man real-time PCR assay for the identification and quantification of roe deer (Capreolus) in food to detect food adulteration[J]. Food Chemistry, 2015, 178: 319-326.
[7] REN J A, DENG T T, HUANG W S, et al. A digital PCR method for identifying and quantifying adulteration of meat species in raw and processed food[J]. PLos One, 2017, 12(3): e0 173 567.
[8] CAI Y C, LI X, LYU R, et al. Quantitative analysis of pork and chicken products by droplet digital PCR[J]. Biomed Research International, 2014, 2014: 1-6.
[9] NOH E S, PARK Y J, KIM E M, et al. Quantitative analysis of Alaska pollock in seafood products by droplet digital PCR[J]. Food Chemistry,2019,275:638-643.
[10] 陈传君,金鹭,林华,等.干制羊肉基因组DNA不同提取方法的比较研究[J]. 核农学报,已录用,待刊.
[11] BURHANEITIN YALSINKAYA,YUMBUL E,ERKAN ![]() et al. Comparison of DNA extraction methods for meat analysis[J]. Food Chemistry, 2016, 221: 1 253-1 257.
et al. Comparison of DNA extraction methods for meat analysis[J]. Food Chemistry, 2016, 221: 1 253-1 257.
[12] KÖPPEL R, GANESHAN A, WEBER S, et al. Duplex digital PCR for the determination of meat proportions of sausages containing meat from chicken, turkey, horse, cow, pig and sheep[J]. European Food Research and Technology, 2019, 245(4): 853-862.
[13] MORISSET D, STEBIH D, MILAVEC M, et al. Quantitative analysis of food and feed samples with droplet digital PCR[J]. Plos one, 2013, 8(5): e62 583.
[14] SHEHATA H R, LI J P, CHEN S, et al. Droplet digital polymerase chain reaction (ddPCR) assays integrated with an internal control for quantification of bovine, porcine, chicken and turkey species in food and feed[J]. Plos One, 2013, 12(8): e0 182 872.
[15] 陈晨,张岩,李永波,等.微滴式数字PCR对肉制品中羊肉和猪肉定量分析[J]. 现代食品科技, 2018, 34(1): 221-227.
[16] BALLIN N Z, VOGENSEN F K, KARLSSON A H. Species determination-Can we detect and quantify meat adulteration?[J]. Meat Science, 2009, 83(2): 165-174.
[17] FLOREN C, WIEDEMANN I, BRENIG B, et al. Species identification and quantification in meat and meat products using droplet digital PCR (dd PCR) [J]. Food Chemistry, 2015, 173(173): 1 054-1 058.
[18] KOPPEL R, RUF J, RENTSCH J. Multiplex real-time PCR for the detection and quantification of DNA from beef, pork, horse and sheep[J]. European Food Research and Technology, 2011, 232(1): 151-155.