表1 用于18S rDNA扩增的通用引物
Table 1 Universal primers for 18S rDNA amplification

引物序列(5′-3′)NS1GTA GTC ATA TGC TFG TCT CFungCAT TCC CCG TTA CCC GTT GGC-FungCGC CCG CCG CGC CCC GCG CCC GGC CCG CCG CCC CCG CCC CAT TCC CCG TTA CCC GTT G
宣威火腿是云南省传统名特优产品,其以“身穿绿袍,色香味美,咸淡相宜,风味独特,食而不腻”等特点名扬海内外,距今已有270年的历史[1],它是选用宣威当地传统乌金猪的后腿,利用宣威独特的气候及地理条件在当地经过腌制、发酵、成熟制作而成[2]。云南省宣威市独特的地理环境和气候条件造就了宣威火腿特定的品质[3]。独特的天然环境,促进火腿微生物的大量繁殖,从而使火腿持续发酵[4]。微生物对肉制品的风味形成及品质有着独特的作用,发酵时期,由于微生物的作用会使原料中的蛋白质、脂肪等发生生化变化,进而影响发酵肉制品的品质,促进其产生芳香化合物,提升肉制品的风味[5]。
聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophorsis,PCR-DGGE)是一种分析微生物群落的有效工具,MUZYERS[6]等首次将PCR-DGGE技术应用于研究微生物多样性,近年来该项技术被应用于研究食品微生态、鉴定微生物、评价食品质量如益生菌制品[7]、酸面团[8]、奶酪[9]、肉制品[10]、甜酒曲[11]等。目前,利用PCR-DGGE技术分析不同加工时间宣威火腿中真菌群落结构还未见报道。基于此,本研究通过PCR-DGGE技术分析不同加工年份(1、2和3年)宣威火腿中的真菌群落结构,为进一步探讨宣威火腿品质奠定基础。
宣威火腿样品由云南省宣威市宣泰火腿有限公司提供,并分别在加工1、2和3年火腿的表面和内部(距肉表面5 cm)取约10 g肉样品,标记为:A1、A2(1年腿的表面和内部),B1、B2(2年腿的表面和内部),C1、C2(3年腿的表面和内部)。后将6个样品真空包装,置于-20℃保存以待分析。
乙醇、冰醋酸、硝酸银、37%甲醛、NaOH,国药集团化学试剂有限公司;环境微生物DNA提取试剂盒、50×TAE buffer、ddH2O,北京博友顺有限公司;DNA纯化试剂盒、DH5α感受态细胞,天根生化科技公司;dNTPs、rTaq、pMD18-T Cloning Kit,日本Takara公司;40% Acrylamide/Bis,美国Bio-Rad公司;Formamide (deionized)、Urea、APS,美国Amresco公司;TEMED,美国Sigma公司;Poly-Gel DNA Extraction Kit,美国OMEGA公司。
Centrifuge 5415D 离心机,德国Eppendorf公司;T-gradient PCR仪,德国Biometra公司;SDC-6恒温槽,宁波新芝有限公司;Gel-Doc2000凝胶成像仪,美国伯乐Bio-Rad公司;JY-SPFT CTAB,北京军意东方仪器有限公司;Dcode变形梯度凝胶电泳仪,美国伯乐Bio-Rad公司;DHP-9052恒温培养箱,上海一恒有限公司;ZHWY-100C摇床,上海智城有限公司。
1.3.1 样品中总DNA的提取
根据环境微生物DNA提取试剂盒方法对6个样品进行总DNA提取。具体操作步骤如下,取肉样品0.5 g置于离心管中,加入缓冲液没过离心管,漩涡振荡5 min,后用多层无菌纱布过滤收集滤液,转入2 mL离心管;加入100 mg玻璃珠,在高速振荡器上振荡约5 min;加入蛋白酶K,充分混匀,55 ℃水浴消化30 min;12 000 r/min离心2 min直至没有沉淀;取上清液加入200 μL DNA提取试剂盒溶液B和200 μL无水乙醇,混匀后转移到吸附柱中静置2 min;12 000 r/min离心2 min后,弃废液,然后漂洗吸附柱2次;12 000 r/min离心2 min,将吸附柱放在50 ℃恒温箱一段时间后加洗脱液,室温放置5 min后,12 000 r/min离心1 min;往吸附柱中加入离心所得洗脱液,室温放置2 min,12 000 r/min离心2 min,即可得到基因组DNA,所得DNA置于-20 ℃保存以待分析。
1.3.2 真菌18S rDNA基因的PCR扩增
真菌通用引物[12]如表1所示。
表1 用于18S rDNA扩增的通用引物
Table 1 Universal primers for 18S rDNA amplification

引物序列(5′-3′)NS1GTA GTC ATA TGC TFG TCT CFungCAT TCC CCG TTA CCC GTT GGC-FungCGC CCG CCG CGC CCC GCG CCC GGC CCG CCG CCC CCG CCC CAT TCC CCG TTA CCC GTT G
以样品基因组DNA为模板,以GC-Fung和NS1为引物扩增样品,得到18S rDAN高变区序列。PCR扩增体系(25 μL)为10× Ex Taq buffer(缓冲) 2.5 μL;dNTP(2.5 mmol/L)2 μL;ExTaqPolymerase(5 U/μL)0.25 μL;GC-FungF(10 mmol/L)1 μL;NS1(10 mmol/L)1 μL;模板DNA 0.05 μg;补ddH2O至25 μL。
PCR扩增程序为:94 ℃预变性5 min;94℃变性30 min,60 ℃复性30 s,72 ℃延伸15 s,30个循环;最终72 ℃延伸10 min。PCR产物采用OMEGA公司DNA Gel Extraction Kit纯化回收。
1.3.3 变性梯度凝胶电泳(DGGE)
参考郭继平等[13]的方法,稍作修改。取10 μL PCR的产物进行变性梯度凝胶电泳分析。变性梯度凝胶梯度为20%~40%,胶浓度为6%,电泳温度控制在60 ℃,用220 V电压预电泳5 min,接着在150 V条件下电泳8 h。电泳结束后、采用银染法染色,条带显现后用Gel-Doc2000凝胶成像仪拍照、记录。
1.3.4 条带切胶回收与克隆测序
参考郑艳等[14]的方法,用经过灭菌的手术刀切下待回收DGGE条带,采用OMEGA 公司Poly-Gel DNA Extraction Kit回收目的条带。以回收产物(2 μL)为模板,以Fung/NS1为引物进行PCR扩增。PCR扩增体系(50 μL)为10× PCR buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;Fung(10 mmol/L)1μL;NS1(10 mmol/L)1 μL;模板DNA 1 μL;补ddH2O至50 μL。PCR扩增程序为94 ℃预变性4 min;94 ℃变性30 s,55℃复性30 s,72 ℃延伸30 s,30个循环;最后,72 ℃延伸10 min。将重新扩增的DNA片段切胶回收、纯化后,连接到Pmd18-T载体上,并转化至DH5α感受态细胞中,筛选阳性克隆,进行序列测定。
真菌多样性指数是研究群落物种数和个体数以及均匀度的综合指标。根据电泳图谱中样品条带数目及每个条带的强度(灰度),对各样品中真菌多样性指数(H)、均匀度(E)和丰富度(S)等指标进行分析。利用Quantity one软件分析DGGE图谱,而香农指数(H)、丰度(S)和均衡指数(E)等指标被用来比较不同样品间的多样性情况[15]。其算法参考郑艳等[14]的方法,计算如公式(1)、公式(2)所示:
(1)
(2)
式中:pi为样品中单一条带的强度在该样品所有条带总强度中所占的比率;N为DGGE图谱单一泳道上所有条带的丰度;Ni为第i条带的丰度;S为某样品中总条带数目。
以GC-Fung和NS1为引物,以从不同加工年份宣威火腿的表面和内部6个样品中提取的总DNA为模板进行18S rDNA片段扩增,得到220 bp左右的片段(图1),符合DGGE分析。
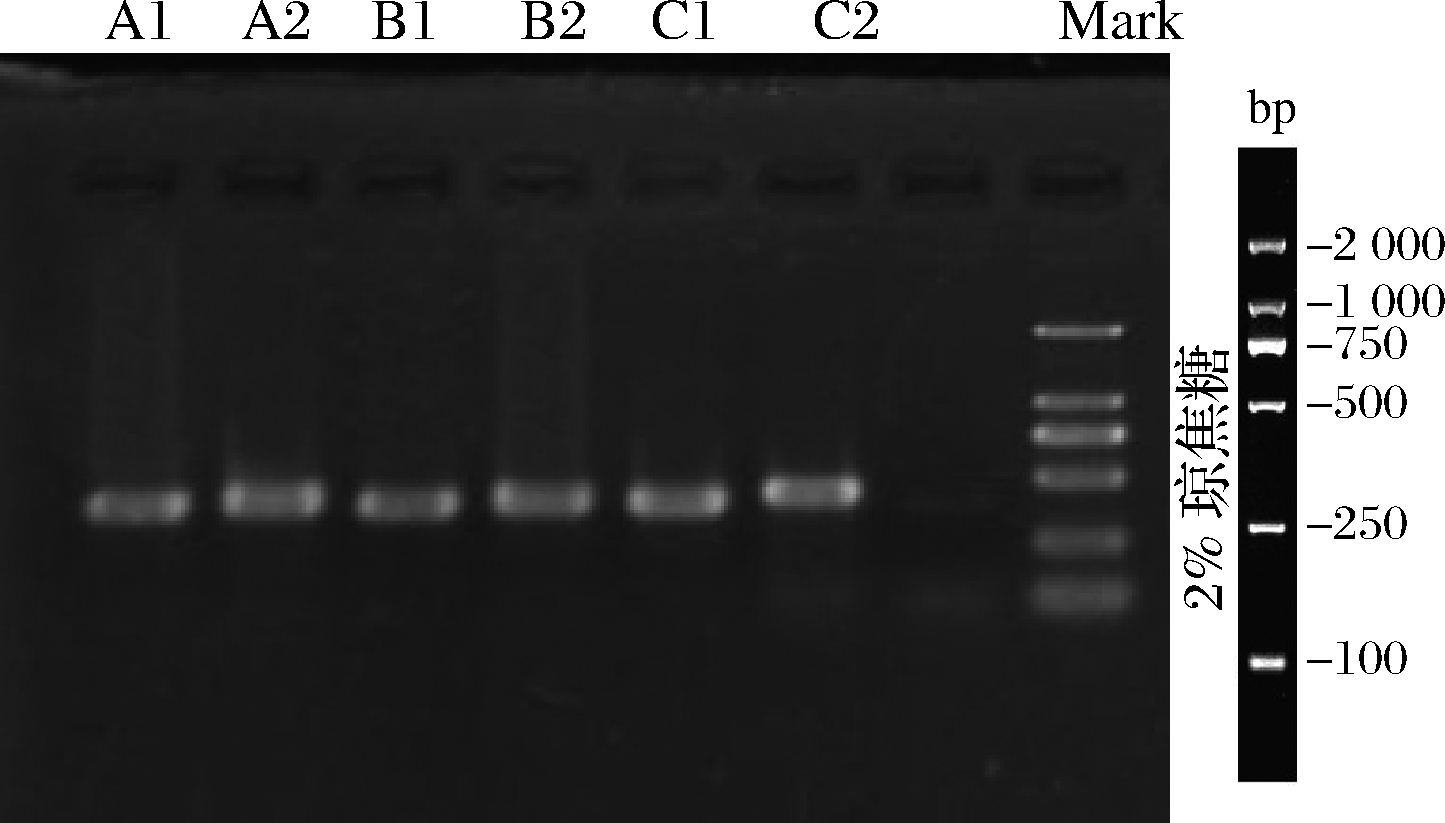
图1 GC-PCR检测
Fig.1 GC-PCR detection
对宣威火腿中的真菌总DNA进行DGGE分析,DGGE图谱如图2所示,泳道中每1条亮带代表1种真菌,不同位置的条带代表不同种属真菌,条带越亮代表相应的种属真菌相对数量越多[16]。由图2和图3可知,共检测到30条不同条带,表明宣威火腿中丰富的真菌种类,不同加工年份宣威火腿样品的电泳条带分离效果较好,条带的亮度也各有不同,其中1年腿表面有22个主要条带,内部有19个;2年腿表面有17个,内部有22个;3年腿表面有23个,内部有17个。

图2 宣威火腿样品的DGGE图谱
Fig.2 DGGE map of Xuanwei ham samples
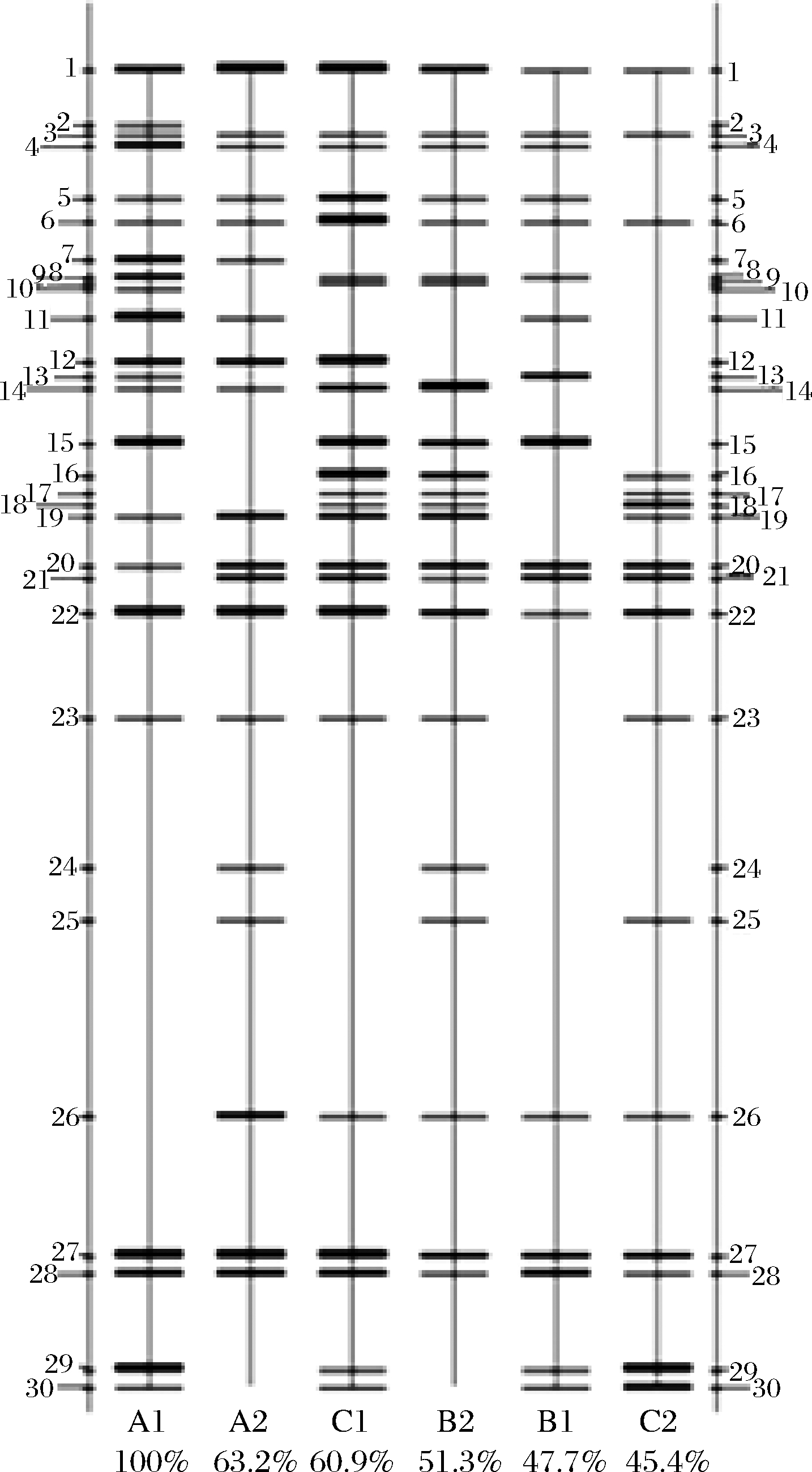
图3 宣威火腿样品的DGGE条带强度示意图
Fig.3 Schematic diagram of DGGE band intensity of Xuanwei ham samples
将宣威火腿样品主要的30条电泳条带进行克隆与测序,测序结果与GenBank中的序列进行比对得到表2。由表2可知,切胶测序的30个条带中有27条可以在GenBank数据库中找到,与其序列的同源性均在96%以上。
结合DGGE图谱与表2可知,加工1年宣威火腿表面最亮的条带为1、11和27,其中条带1为Aspergillus pseudoglaucus(假灰绿曲霉),条带11为Aspergillus oryzae(米曲霉),条带27为Wallemia sebi。加工1年宣威火腿内部最亮的条带为1、22和27,其中条带22为Yamadazyma triangularis。加工2年宣威火腿表面最亮的条带为1和5,其中条带5为Aspergillus penicillioides(帚状曲霉)。2年宣威火腿内部最亮的条带为1和14,其中条带14为Aspergillus glaucus(灰绿曲霉)。加工3年宣威火腿表面最亮的条带为1、3、16、22和27,其中条带3为Phialosimplex caninus,条带16为Aspergillus niger(黑曲霉),条带22为Yamadazyma triangularis。加工3年宣威火腿内部最亮的条带为1、5和27,其条带分别为Aspergillus pseudoglaucus(假灰绿曲霉)、Aspergillus penicillioides(帚状曲霉)和Wallemia sebi。由PCR-DGGE图谱可知,不同加工年份宣威火腿的表面及内部均检测出Aspergillus pseudoglaucus(假灰绿曲霉),由此可以推断,Aspergillus pseudoglaucus(假灰绿曲霉)是宣威火腿的优势菌种。
表2 宣威火腿样品DGGE主要条带的序列比对结果
Table 2 Sequential alignment of the main bands of DGGE of Xuanwei ham samples

条带号最相似菌株名称登录号相似度/%Band 1Aspergillus pseudoglaucus(假灰绿曲霉)AB00208499Band 2Aspergillus candidus(亮白曲霉)EU883597100Band 3Phialosimplex caninusGQ169310100Band 4Aspergillus sydowii(萨氏曲霉)AB01240899Band 5Aspergillus penicillioides(帚状曲霉)DQ985959100Band 6Phialosimplex caninusGQ169310100Band 7Penicillium simplicissimum(简单青霉)JQ00208499Band 8Monascus purpureus(红曲霉)JN94046699Band 9Debaryomyces mycophilus(德巴利氏酵母属)AF44001799Band 10Candida fermenticarens(念珠菌)AB01352599Band 11Aspergillus oryzae(米曲霉)AP00717396Band 12Aspergillus terreus(曲霉)GU57385099Band 13Aspergillus versicolor(杂色曲霉)AB02653999Band 14Aspergillus glaucus(灰绿曲霉)AB10205899Band 15Asoergillus flavipuds(黄柄曲霉)KU06076999Band 16Aspergillus niger(黑曲霉)HQ37985399Band 17Candida zeylanoides(诞沫假丝酵母)AY520262100Band 18Hansenula sydowiorum(赛道威汉逊酵母)AB02708099Band 19Aspergillus wentii(白氏曲霉)KJ12688499Band 20Aspergillus penicillioides(帚状曲霉)DQ985959100Band 21Yamadazyma triangularisJQ69891599Band 22Yamadazyma triangularisJQ69891599Band 23Rhodotorula glutinis(红酵母)AF40209499Band 27Wallemia sebiAY74138097Band 28Candida glucosophilaAB01351999Band 29Uncultured wallemiaFJ39341997Band 30Aspergillus funigatus(烟曲霉)HM61030099
DGGE图谱的相似性是通过Quantity One软件进行分析的,通过分析得到不同加工年份宣威火腿真菌群落相似性指数。由表3可知,加工2年宣威火腿内部与加工3年表面的真菌群落结构相似性最高,为69.3%,加工1年内部与加工2年表面的真菌群落结构相似性最低,为42.7%。从宣威火腿表面看,加工1年的与加工3年表面真菌群落结构相似性为60.9%,分别比加工1年与加工2年表面真菌群落结构相似性、加工2年与加工3年表面真菌群落结构相似性高13.2%、15%。从宣威火腿内部看,加工1年的与加工2年内部真菌群落结构相似性为60.5%,分别比分别比加工1年与加工3年内部真菌群落结构相似性和加工2年与加工3年内部真菌群落结构相似高12.4%、4.9%。
图4为不同加工年份宣威火腿表面及内部的条带相似性聚类分析图,由图4可知,加工2年宣威火腿内部与加工3年火腿表面相似度为69%,说明两者间差异较小。加工2年火腿表面与其他加工年份表面及内部5个样品间的相似度为46%,说明2年火腿表面与其他5个样品间差异较大。从整体看,不同加工年份火腿表面及内部真菌群落结构间存在一定的差异,这可能是随着时间的变化,空气对火腿的氧化,导致真菌群落结构发生了变化。
表3 戴斯系数比较宣威火腿样品PCR-DGGE图谱的相似性 单位:%
Table 3 Comparison of the similarity of PCR-DGGE spectra of Xuanwei ham samples by Dyce coefficient

样品A1A2B1B2C1C2A110063.247.751.360.945.4A263.210042.760.566.548.1B147.742.710047.845.947.9B251.360.547.810069.355.6C160.966.545.969.310049.5C245.448.147.955.649.5100

图4 宣威火腿样本间的聚类图
Fig.4 Cluster diagram between Xuanwei ham samples
微生物多样性指数由于评估微生物群落的丰富度。香农指数(H)用于评估微生物群落的多样性,H值越大,说明微生物的种类和数量越多,微生物群落多样性越高;均匀度指数(E)用于评估微生物分布的均匀程度,E值越大,说明微生物群落分布越均匀;丰富度指数(S)是指一个群落或生境中物种数目的多寡[17]。由表4可知,不同加工年份宣威火腿表面及内部真菌群落多样性有差异。加工3年宣威火腿表面及内部的香农指数分别是最高和最低的,说明其表面及内部真菌群落多样性分别是最高和最低的,加工1年腿表面真菌群落分布最均匀,而加工3年腿表面真菌群落丰富度指数最大。
表4 宣威火腿各样品间的真菌群落多样性分析
Table 4 Analysis of fungal community diversity in different samples of xuanwei ham
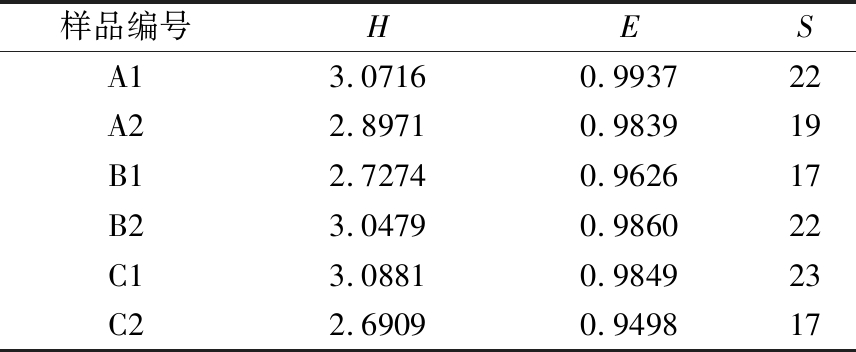
样品编号HESA13.07160.993722A22.89710.983919B12.72740.962617B23.04790.986022C13.08810.984923C22.69090.949817
宣威火腿所具有的特殊风味与其漫长的发酵过程密切相关,微生物的代谢活动以及火腿表面大量霉菌的生长是宣威火腿独特风味形成的基础[18]。本研究发现,假灰绿曲霉、帚状曲霉、假丝酵母属存在于整个发酵期间的表面和内部。霉菌是宣威火腿发酵过程中的优势真菌,这与黄艾祥等[19]对云南干腌火腿和甄宗圆等[20]对金华火腿的研究结果一致。霉菌的生长对火腿风味形成无直接作用,但对防止火腿氧化变质、抑制有害微生物的生长、提高火腿质量有间接作用[21]。酵母菌的存在不但是火腿整个发酵期的优势有益菌群,而且对成熟火腿的VE、脯氨酸、色氨酸等香甜成分的增加及风味的形成起重要作用[22]。NUNEZ等[23]对伊比丽亚干腌火腿中酵母菌的研究发现,诞沫假丝酵母是原料腿的优势菌,德巴利汉逊氏酵母是发酵腿、成熟腿的优势菌。MUHAMED等[24]研究了从香肠中分离出来的微生物菌株抗辐射情况,经过抗辐射处理后,发现诞沫酵母菌(eandidazeylanoides)具有较强的抵抗力。本试验经对云南宣威火腿分离鉴定出的酵母菌数量相对霉菌较少,最主要的有德巴利酵母属和假丝酵母属,这与黄艾祥[25]对云南干腌火腿的研究结果一致。
利用现代分子生物学技术,对微生物种群进行结构分析与鉴定,能克服传统方法的缺点与不足。传统的微生物种群结构分析方法是先进行菌种分离培养再进行单一菌种的鉴定,耗时长,过程繁琐,且自然界中有85%~99%的微生物至今还不可纯培养,而利用DGGE技术可以直接对微生物进行多种种群的鉴定[26]。但由于DGGE只能对微生物群落中数量>1%的优势种群进行分析[27],并且由于嵌合体、共迁移、异源双链体等会导致结果产生偏差,所以该技术也有一定的局限性[28]。因此充分了解该技术的基础背景和局限性,结合其他分子生物学技术如酶学技术、荧光原位杂交技术等,才能从不同方面客观的反映宣威火腿中真菌多样性信息,为宣威火腿品质的研究提供更好的技术支持。
从不同加工年份宣威火腿中共检测到了27种真菌,宣威火腿中主要的真菌为Aspergillus pseudoglaucus(假灰绿曲霉)、Phialosimplex caninus、Aspergillus penicillioides(帚状曲霉)、Yamadazyma triangularis、Wallemia sebi、Candida glucosophila等,其中Aspergillus pseudoglaucus(假灰绿曲霉)是宣威火腿的优势菌种。多样性分析表明,加工3年的宣威火腿表面真菌群落多样性最高,加工2年火腿表面真菌群落多样性最低。加工2年火腿内部真菌群落多样性最高,加工3年火腿内部真菌群落多样性最低。相较于传统的分离培养法,采用PCR-DGGE技术分析宣威火腿的真菌群落结构,结果能够更全面、更直观,也为进一步研究宣威火腿独特风味的形成提供了技术支持。
[1] 董寅初. 中国传统风味肉制品的现代化势在必行(上)[J]. 肉类研究,1998(2):3-6.
[2] 乔发东. 宣威火腿标准化生产与品质改良技术研究[D]. 北京: 中国农业大学,2004, 6:48-58.
[3] 王桥美, 杨瑞娟, 严亮. 微生物多样性与宣威火腿品质关系的研究进展[J]. 食品安全导刊,2016(33X):137-139.
[4] FIDEL TOLDRA, MONICA FLORESl, YOLANDA SANZ. Dry-cured hamflavour: enzymatic generation and process influence[J]. FoodChemistry, 1997, 59 (4): 523-530.
[5] 李晓燕,王卫,张佳敏,等. 发酵微生物提升传统腌腊肉制品风味和安全性研究进展[J]. 食品工业,2018,39(5):275-279.
[6] MUYZER G, WALL D E. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction amplified genes coding for 16SrRNA[J].Appl Environ Microbiol, 1993, 59(3): 695-700
[7] TEMMERMAN R, SCHEIRLINCK I, HUYS G, et al. Culture-independent analysis of probiotic products by denaturing gradient gel electrophoresis[J]. Appl Environ Microbiol, 2003,69(1): 220-226.
[8] ZHANG G, SADIQ F A, ZHU L, et al. Investigation of microbial communities of chinese sourdoughs using culture-dependent and dgge approaches[J]. Journal of Food Science, 2015, 80(11): M2 535-M2 542.
[9] NALEPA B, MARKIEWICZ L H. PCR-DGGE markers for qualitative profiling of microbiota in raw milk and ripened cheeses[J]. LWT - Food Science and Technology, 2017:S0023643817303729.
[10] 孙丹丹,卢士玲,李开雄,等. 贮藏温度对冷鲜羊肉微生物菌群生长变化的影响[J]. 食品工业科技,2017,38(4):327-331;341.
[11] 姚淑敏,陈璐,闫华文. 应用PCR-DGGE方法研究甜酒曲中真菌多样性[J]. 中国酿造,2016,35(11):44-48.
[12] AMPE F, BENOMAR N, MOIZAN C, et al. Polyphasic study of the spatialdistribution ofmicroorganisms in Mexican Pozo,l a fermented maizedough, demonstrates the need for cultivation-independent methods to investigate traditional fermentations[J]. Appl Environ Microbio, 1999, 65(12): 5 464-5 473.
[13] 郭继平,马光,张志强等. 喷施菌剂后葡萄叶片真菌多样性的PCR-DGGE分析[J]. 中国生物防治学报,2018,34(6):937-944.
[14] 郑艳,姚婷. PCR-DGGE分析甘薯酸浆自然发酵过程中细菌多样性[J]. 食品科学,2016,37(7):99-103.
[15] KUANG Y, TANI K, SYNNOTT A J, et al. Characterization of bacterial population of raw milk from bovine mastitis by culture-independent PCR-DGGE method[J]. Biochemical Engineering Journal, 2009, 45(1):76-81.
[16] 刘姝韵,尚思奇,孙灿,等. 基于PCR-DGGE分析云南牛干巴的细菌群落结构[J]. 肉类研究,2017,31(9):44-50.
[17] 龚治翔,马晓寒,任志广,等. 连作烤烟根际土壤细菌群落16S rDNA-PCR-DGGE分析[J]. 中国农业科技导报,2018,20(2):39-47.
[18] 李平兰,沈清武,吕燕妮,等. 宣威火腿成熟产品中主要微生物菌相构成分析[J]. 中国微生物态学杂志, 2003(5):262-263.
[19] 黄艾祥,周利,葛长荣,等. 速制云腿中霉菌的分离及作用[J]. 肉类工业,2002(7):22-23.
[20] 甄宗圆. 金华火腿微生物区系研究[D]. 重庆:西南农业大学,2004,6:33-38.
[21] 林克忠,杨耀寰,竺尚武,等. 金华火腿的质量和色香味形成与霉菌关系的研究[J]. 肉类研究,1992(2):10-16;21.
[22] 马萍,江东福,段若玲,等. 宣威火腿及其微生物作用菌群的研究[J]. 云南大学学报(自然科学版),1990,3(12):64-70.
[23] NUNEZ F, RODRIGUEZ M M, CODROBA J J, et al. Yeas tpopulation during ripening of dry-cured Iberian ham[J]. International journal of food microbiology, l996, 29: 271-280.
[24] MUHAMED L J, ITO H, WATANABE H, et al. The effect of substrate on the radiation resistance of yeasts isolated from sausage meat[J]. Lett App Microbiol, 1986, 22(1): 80-84.
[25] 黄艾祥. 云南干腌火腿品质特征形成与微生物作用研究[D]. 重庆:西南大学,2006,5:87-102.
[26] 夏耕,谢骏,王海英,等. 基于PCR-DGGE技术分析生物絮团的细菌群落结构[N]. 水产学报,2012,36(10):1 563-1 571.
[27] 罗湘南,梁英娟,付红霞,等. PCR-DGGE技术在微生物生态学中的应用[J]. 生物学杂志,2007(6):58-60.
[28] VALLAEYS T, TOPP E, MUYZER G, et al. Evaluation of denaturing gradient gel electrophoresis in the detection of 16S rDNA sequence variation in rhizobia and methanotrophs[J]. FEMS Microbiol Ecol, 1997, 24(3): 279-285.