西藏不同产区曲拉细菌群落结构的比较分析
刘振东1,毕娜1,李哲2,李梁1,罗章1,薛蓓1*,汪雯翰3*
1(西藏农牧学院 食品科学学院,西藏 林芝, 860000)2(西藏自治区藏药审评认证中心,西藏 拉萨,850000)3(上海市农业科学院食用菌研究所,上海, 201403)
摘 要 为探究西藏不同产区传统发酵乳制品曲拉中细菌多样性及其微生物安全性,该研究通过Illumina MiSeq测序平台,对西藏拉萨(LS)、日喀则(RKZ)、山南(SN)、昌都(CD)、林芝(LZ)、那曲(NQ)和阿里(AL)共7个地区的曲拉样品细菌多样性及群落结构进行了系统分析。结果表明,曲拉具有高度的细菌多样性,并且不同地区的样品存在一定的共性和差异。在门的水平上,7个地区的优势菌均集中在厚壁菌门(Firmicutes)和变形菌门(Proteobacteria),其相对丰度分别在(12.82±5.63)%~(96.54±1.39)%和(3.46±1.39)%~(75.09±9.37)%。相比门的种类,拉萨(LS)最多,为24个门,阿里(AL)最少,为2个门。在属的水平上,拉萨地区(LS)曲拉样品中的优势菌属为雷尔氏菌属(Ralstonia)、假单胞菌属(Pseudomonas)和苍白杆菌属(Ochrobactrum),其相对丰度分别为(17.25±7.05)%、(16.05±3.22)% 和(9.52±3.19)%;其他6个产地的曲拉样品中优势菌属主要为乳球菌属(Lactococcus)和乳杆菌属(Lactobacillus),其相对丰度分别为(85.13±2.99)%~(25.82±1.42)%和(4.15±1.38)%~(57.21±3.11)%;而在属的种类上,LS最多,为409个属,AL最少,为15个属。聚类分析显示,不同曲拉样本的菌群与地理位置存在一定关系,同一个采样区的样本具有一定的聚类趋势,同时发现LZ、RKZ和CD可聚为一类,而其他4个地区的样品较为离散,且各自独立。
关键词 曲拉;不同产区;高通量测序;细菌多样性
DOI:10.13995/j.cnki.11-1802/ts.022500
引用格式:刘振东,毕娜,李哲,等.西藏不同产区曲拉细菌群落结构的比较分析[J].食品与发酵工业,2020,46(6):60-66.LIU Zhendong,BI Na, LI Zhe, et al. Comparative analysis of the bacteria community structure of Qula in different producing areas in Tibet[J].Food and Fermentation Industries,2020,46(6):60-66.
第一作者:博士研究生,副教授(薛蓓副教授和汪雯翰研究员为共同通讯作者,E-mail:13658940092@163.com,wangwenhan@saas.sh.cn)
基金项目:科技部重大专项民族特色食品工业化关键技术(2018YFD0400102);西藏农牧学院食品科学与工程重点学科建设(502218009)
收稿日期:2019-10-10,改回日期:2019-11-15
Comparative analysis of the bacteria community structure of Qula in different producing areas in Tibet
LIU Zhendong1,BI Na1, LI Zhe2,LI Liang1,LUO Zhang1,XUE Bei1*,WANG Wenhan3*
1(College of Food Science, Tibet Agriculture and Animal Husbandry University, Nyingchi 860000,China)2(Tibetan medicine evaluation and certification center of the Tibet Autonomous Region, Lasa 850000,China)3(Institute of Edible Fungi, Shanghai Academy of Agricultural Sciences, Shanghai 201403,China)
ABSTRACT In order to investigate the bacterial diversity and microbial safety of Qula (traditional fermented dairy products) in different producing areas of Tibet, high-throughput sequencing technology was used to analyze the bacteria diversity and community structure systematically in Qula samples collected from 7 regions including Lhasa(LS), Shigatse(RKZ), Sannan(SN), Changdu(CD), Nyingchi (LZ), Nagqu (NQ), Ali(AL) in Tibet .The results showed that the Qula samples had a high degree of bacterial diversity, and there were certain commonalities and differences in the samples of different regions. Firmicutes and Proteobacteria were the dominant phylum in different samples at the phylum level, and the relative abundance were (12.82±5.63)% -(96.54±1.39)% and (3.46±1.39)%-(75.09±9.37)%, respectively. The type of phylum was up to 24 phyla in Lhasa (LS) sample and 2 in Ali (AL) sample, which was the least. At the genus level, the dominant bacterial genera in LS were Ralstonia, Pseudomonas and Ochrobactrum, and the relative abundance were (17.25±7.05)%, (16.05±3.22)% and (9.52±3.19)%, respectively. While the dominant bacterial genera in the other six regions were Lactococcus and Lactobacillus at the genus level, and the relative abundance were (85.13±2.99)% -(25.82±1.42)% and (4.15±1.38)%-(57.21±3.11)%, respectively. LS samples had a maximum of 409 genera and AL had the least of 15 genera at the genus level. The cluster analysis showed that the diversity and community structure of bacteria in different Qula samples had a certain correlation with the geographical location. The samples in the same sampling area had a certain clustering trend. At the same time, it was found that LZ, RKZ and CD could be clustered into one category, while the other four regions were discrete and independent.
Key words Qula;different producing areas;high throughput sequencing;bacterial diversity
曲拉(藏语,指奶干渣)是藏区传统发酵乳制品之一,由牦牛奶脱脂后,在自然条件下发酵酪蛋白凝固并风干制成,属于粗奶酪的一种[1-2]。作为传统的藏族食品,曲拉具有蛋白质含量高、保存时间长、携带方便等优点,在整个藏区被广泛食用[3-4]。
曲拉作为一种藏区典型的发酵型乳制品,其制作工艺多采用传统的手工开放式自作,因此不同的制作环境直接关系到曲拉的微生物组成,进而影响曲拉的风味、安全及营养价值[5-7]。
基于微生物分离、纯化、培养及鉴定的传统研究方法难以获得培养困难及痕量的微生物,而且难以展现环境中完整的微生物种群结构及生态关系[8-9]。目前,高通量测序技术已经逐渐成为研究微生物组成及结构的一种重要方法,近年该技术已广泛用于原料乳和各类发酵乳等乳制品生态位的研究[10-11]。
本研究旨在利用高通量测序技术,全面了解西藏7个不同产区牦牛曲拉的细菌群落结构及相互差异,为西藏不同产区曲拉的安全性评价和功能微生物的挖掘奠定一定的理论基础。
1 材料与方法
1.1 材料与试剂
21份牦牛曲拉样品,分别来自西藏拉萨(LS)、日喀则(RKZ)、山南(SN)、昌都(CD)、林芝(LZ)、那曲(NQ)和阿里共7个地区;E.Z.N.A.Soil DNA试剂盒,美国OMEGA公司;Qubit2.0 DNA检测试剂盒,美国Invitrogen公司;Q5® High-Fidelity DNA Polymerase,美国NEB公司;凝胶回收试剂盒,德国Qiagen公司。
1.2 仪器与设备
5424R高速冷冻离心机,德国Eppendorf公司;DYY-6C型电泳仪,北京六一仪器厂;T-100 PCR仪,美国Bio-rad公司;MiSeq高通量测序仪,美国Illumina公司;BG-gdsAUTO(130) 凝胶成像系统,北京百晶生物技术有限公司。
1.3 实验方法
1.3.1 样品的采集
2018年6月采集拉萨市林周县、日喀则市萨迦县、山南市隆子县、昌都市八宿县、林芝市米林县、那曲市申扎县、阿里地区改则县7个地区的曲拉样品,分别编号为LS、RKZ、SN、CD、LZ、NQ、AL。将所有样品置于无菌离心管中,放入冰盒保存并立即运送到实验室,置于-80 ℃冰箱中备用。
1.3.2 DNA提取和PCR扩增
对于不同来源的微生物组样本,根据E.Z.N.A.®soil试剂盒说明书提取各牦牛曲拉样品中的总DNA,然后通过0.8%琼脂糖凝胶电泳的方法检测总DNA的提取质量。本研究采用NEB公司的Q5高保真DNA聚合酶保证结果的可靠性,采用标准细菌V3-V4引物338F(5’-ACTCCTACGGGAGGCAGCA-3’)和806R(5’-GGACTACHVGGGTWTCTAAT-3’)进行PCR扩增。
PCR体系(25 μL):基因组DNA模板2 μL,dNTP 2 μL,ddH2O8.75 μL,缓冲液5 μL,Q5 DNA 0.25 μL,DNA模板2 μL,5×GC缓冲液5 μL。
PCR扩增条件:94 ℃预变性2 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸40 s,扩增40个循环;72 ℃延伸1 min。尽量保证同一批样本的扩增条件一致。
1.3.3 文库建立
采用三代测序技术制备测序文库。把通过PCR扩增得到的产物进行末端修复,将黏性末端修整为平末端;在DNA序列的3′端添加A碱基以防止DNA片段自连;在DNA序列5′端添加含有文库特异性标签的测序接头;对上述连上接头的DNA片段进行PCR扩增,从而富集测序文库模板,并采用BECKMAN AMPure XP Beads再次纯化文库富集产物;最后对文库做最终的片段选择与纯化。
1.3.4 Illumina MiSeq测序
将Illumina MiSeq测序得到的双端序列数据进行质量控制,以97%相似性为分类标准得到OTUs。然后在GenBank数据库进行Blast比对,并提交到美吉生物的云分析平台进行多样性分析(http://www.i-sanger.com/)。
2 结果与分析
2.1 测序数据统计及Alpha多样性分析
采用Illumina MiSeq平台对7个地区曲拉样品测序结果进行分析统计,结果如表1所示。由表1可知21个样品总计测得有效序列条数为(241 148±23 200)条,以97%的序列相似度作为OTU划分阈值,共产生(3 994±78)个OTUs,由序列数及OTUs可以看出,曲拉中细菌种类繁多、物种丰富,且不同来源的曲拉存在明显差异。不同区域曲拉样品间Alpha多样性统计中的Chao1指数、Ace指数差异显著(P<0.05)。
表1 不同样本的测序数据统计
Table 1 Sequence data statistics in different Qula samples

样本名称序列数OTUSSimpson指数Chao1指数ACE指数Shannon指数LS34 505±4 997691±390.93±0.02727.99±54.74c732.66±55.505.84±0.09RKZ30 802±1 986536±240.82±0.04564.14±58.74e564.90±59.484.92±0.24SN30 095±1 759476±90.84±0.01480.05±13.83f487.03±21.044.87±0.14CD37 739±5 158560±390.92±0.02592.23±55.36d606.33±55.085.60±0.29LZ33 605±1 668691±570.90±0.01760.21±109.24a775.91±95.515.46±0.04NQ36 723±1 007698±420.85±0.02752.75±56.67b757.56±47.924.72±0.29AL37 679±7 358342±280.66±0.08352.47±32.77g356.23±32.673.73±0.56合计241 148±23 2003 994±78----
注:“-”表示无需统计;不同小写字母表示差异显著(下同)
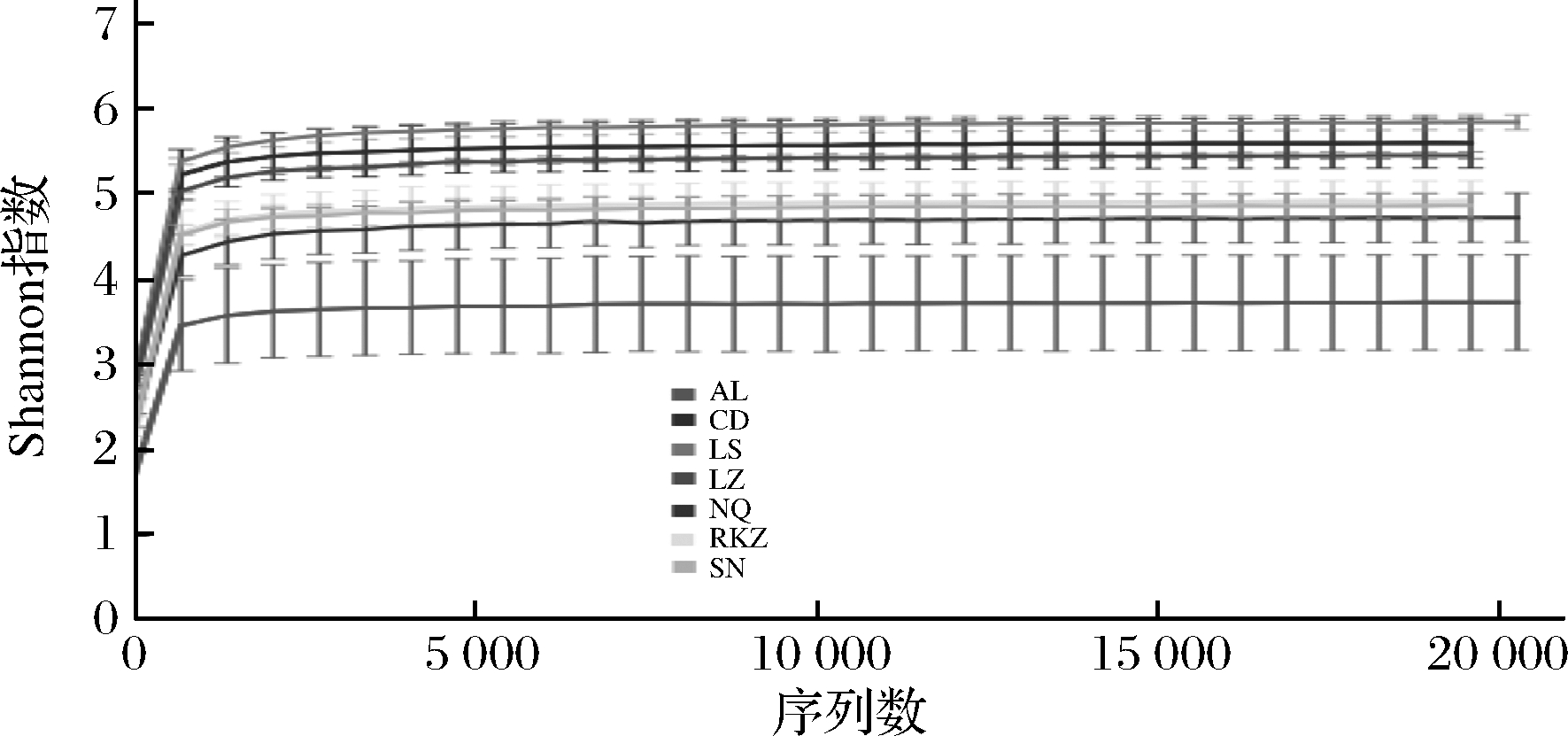
2.2 稀释曲线
由图1可知,当样品测序量较低时,每个样品的Shannon指数随着测序量增加呈显著上升趋势;但随着测序量逐渐增加,各曲拉样品的Shannon指数增加逐渐趋于缓慢;当样品测序量到达5 000后各样品的Shannon指数基本稳定,说明样品测序量达到5 000时已足以反映21个曲拉样品中细菌群落多样性。
2.3 在门水平上各样品菌群分类及比较
由表2可知,7个产地的曲拉样品共检测出24个门,其中LS检测出全部24个菌门,RKZ中检测出6个菌门,SN中检测出7个菌门,CD中检测出6个菌门,LZ中检测出12个菌门,NQ中检测出20个菌门,而AL最少仅检测出2个菌门。7个地区曲拉样品的主要优势菌门均为厚壁菌门和变形菌门,除LS和CD外,其他5个地区曲拉样品的第一优势菌均为Firmicutes,其丰度值在61.01%~96.54%,LS和CD的第一优势菌Proteobacteria,其丰度值在40.60%~75.09%。
2.4 在属水平上各样品菌群分类及比较
如表3所示,对7个地区21份曲拉样品从属水平进行鉴定共鉴定出550个属,其中LS中检测出409个属,RKZ中检测出47个属,SN中检测出69个属,CD中检测出36个属,LZ中检测出99个属,NQ中检测出330个属,AL中检测出15个属。各地区样品在属水平上检出的种类差异较大,LS>NQ>LZ>SN>RKZ>CD>AL。
表2 各样品门水平上菌群分布相对丰度 单位:%
Table 2 The relative abundance of microbial communities in different Qula samples at the phylum level

门分类门水平相对丰度LSRKZSNCDLZNQALFirmicutes12.82±5.63a69.19±9.52b91.18±1.24c5.90±7.41b61.01±2.84b68.47±4.30b96.54±1.39cProteobacteria75.09±9.37d27.13±11.13bc4.73±0.86a40.60±7.36c35.24±2.35bc25.07±4.69b3.46±1.39aBacteroidetes3.27±1.70c0.46±0.26a2.28±0.37bc0.12±0.04a2.89±0.40c1.04±0.38ab0.00±0.00aActinobacteria3.97±0.72b3.17±1.39b3.18±0.18a0.27±0.05a0.65±0.10a3.25±0.80b0.00±0.00aCyanobacteria2.33±0.83b0.03±0.05a0.00±0.00a0.01±0. 01a0.03±0.00a0.31±0.11a0.00±0.00aDeinococcus-Thermus0.69±0.32c0. 02±0.00a0.06±0.01a0.01±0. 00a0.08±0.00a0.41±0.13b0.00±0.00aChloroflexi0.40±0.19b0.00±0.00a0.00±0.00a0.00±0.00a0.01±0.01a0.38±0.10b0.00±0.00aAcidobacteria0.42±0.39b0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.28±0.19ab0.00±0.00aGemmatimonadetes0.16±0.08ab0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.45±0.39a0.00±0.00bPatescibacteria0.10±0.04b0.00±0.00a0.34±0.05c0.00±0.00a0.00±0.00a0.08±0.04b0.00±0.00aPlanctomycetes0.19±0.09b0.00±0.00a0.01±0.01a0.00±0.00a0.01±0.01a0.08±0.05a0.00±0.00aSpirochaetes0.07±0.11a0.00±0.00a0.00±0.00a0.00±0.00a0.02±0.04a0.03±0.03a0.00±0.00aNitrospirae0.08±0.06a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.03±0.04a0.00±0.00aVerrucomicrobia0.08±0.05b0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.01±0.00a0.00±0.00aTenericutes0.05±0.08a0.00±0.00a0.00±0.00a0.00±0.00a0.01±0.02a0.02±0.00a0.00±0.00aEpsilonbacteraeota0.05±0.07a0.00±0.00a0.00±0.00a0.00±0.00a0.01±0.02a0.01±0.02a0.00±0.00aFusobacteria0.04±0.02b0.00±0.00a0.00±0.00a0.00±0.00a0.01±0.00a0.02±0.02ab0.00±0.00aSynergistetes0.05±0.06a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00aArmatimonadetes0.03±0.02b0.00±0.00a0.00±0.00b0.00±0.00a0.00±0.00b0.01±0.01b0.00±0.00aElusimicrobia0.03±0.02b0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.01±0.02ab0.00±0.00aWPS-20.02±0.02b0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.01±0.01ab0.00±0.00aLatescibacteria0.02±0.03a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00aRokubacteria0.01±0.02a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00aBRC10.01±0.01a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a
表3 各样品属水平上菌群分布相对丰度 单位:%
Table 3 The relative abundance of microbial communities in different Qula samples at the genus level

属分类LSRKZSNCDLZNQALLactococcus1.59±0.57a63.73±8.29d25.82±1.42b46.62±4.60c57.60±3.26d0.45±0.07a0.48±0.25aLactobacillus5.52±4.76a4.15±1.38a57.21±3.11c9.20±6.49a1.61±0.10a32.77±3.20b85.13±2.99dAcetobacter1.71±1.22a23.18±10.23b0.23±0.05a16.93±3.38b0.28±0.01a0.16±0.06a3.34±1.36aPseudomonas16.05±3.2d1.12±0.62ab0.84±0.14a9.98±5.38c5.14±0.41c0.26±0.07a0.00±0.00aSerratia0.04±0.03a0.13±0.11a0.08±0.01a3.25±1.73b14.40±0.91c0.02±0.02a0.00±0.00aAnoxybacillus0.06±0.05a0.01±0.01a0.00±0.00a0.00±0.00a0.02±0.01a27.82±3.00b0.00±0.00aRalstonia17.25±7.05c0.07±0.04a0.62±0.30a0.15±0.03a1.15±0.14a8.52±1.96b0.03±0.00aPelomonas11.05±4.89c0.07±0.03a0.41±0.11a0.09±0.04a0.66±0.05a5.47±1.38b0.04±0.01aAcinetobacter1.38±0.47c0.16±0.09a0.91±0.13bc0.53±0.17ab4.08±0.34d0.58±0.27ab0.00±0.00aLeuconostoc0.30±0.10a0.91±0.17ab8.01±1.09c1.79±0.25b1.41±0.28b0.03±0.01a0.01±0.01aOchrobactrum9.52±3.19b0.05±0.05a0.18±0.04a0.07±0.04a0.37±0.06a2.64±0.42a0.01±0.01aStreptococcus0.19±0.09a0.00±0.00a0.01±0.01a0.03±0.02a0.00±0.00a4.19±0.26b8.57±1.33cGluconobacter0.46±0.55a1.12±0.72a0.00±0.00a9.00±2.74a0.00±0.00a0.02±0.01a0.00±0.00aChryseobacterium0.16±0.04a0.41±0.22a1.73±0.28a0.12±0.05b2.54±0.25a0.05±0.03a0.00±0.00aunidentified2.83±2.11a0.08±0.08a0.00±0.00a0.11±0.06a2.64±0.23a0.08±0.04a0.00±0.00aEnterobacter0.45±0.00b0.32±0.25b0.06±0.01a0.01±0.01a2.08±0.16c0.19±0.05ab0.00±0.00aCupriavidus1.82±0.92b0.02±0.00a0.08±0.04a0.01±0.01a0.13±0.01a1.15±0.33b0.01±0.01aStenotrophomonas0.32±0.21b0.01±0.00a0.06±0.02a0.16±0.08ab1.22±0.05c0.09±0.05a0.00±0.00aEnterococcus0.85±0.39a0.31±0.03a0.03±0.01a1.29±1.09b0.00±0.00a0.00±0.00a0.05±0.01aArthrobacter0.42±0.06b1.34±0.43c0.32±0.05ab0.01±0.01a0.00±0.00a0.22±0.04ab0.00±0.00aBifidobacterium0.20±0.11a1.51±0.87b0.01±0.00a0.21±0.05a0.10±0.02a0.06±0.02a0.00±0.00aErwinia1.52±1.11a0.01±0.01a0.00±0.00a0.00±0.00a0.33±0.05a0.01±0.01a0.00±0.00aClostridium_sensu_stricto_10.34±0.42a0.00±0.00a0.00±0.00a0.00±0.00a0.00±0.00a0.22±0.11a1.36±0.09bKocuria0.09±0.04ab0.21±0.10bc0.67±0.10d0.00±0.00a0.27±0.03c0.10±0.05ab0.00±0.00aNasturtium_officinale1.58±0.47b0.02±0.02a0.00±0.00a0.01±0.01a0.00±0.00a0.04±0.02a0.00±0.00aSphingomonas0.83±0.25b0.00±0.00a0.04±0.02a0.00±0.00a0.03±0.02a0.68±0.23b0.00±0.00aunidentified1.10±0.91b0.00±0.00a0.03±0.02a0.00±0.00a0.02±0.03a0.36±0.26ab0.00±0.00aThermus0.65±0.34c0.02±0.01a0.06 ±0.01a0.01±0.00a0.08±0.04ab0.37±0.12bc0.00±0.00aAcidovorax0.61±0.37b0.00±0.00a0.17±0.04a0.00±0.00a0.06±0.02a0.34±0.19ab0.00±0.00aBrevundimonas0.42±0.07c0.00±0.00a0.26±0.01b0.01±0.01a0.09±0.02a0.24±0.10b0.00±0.00a其他/种379236329933294
如表4所示,从属水平分析,多数产地曲拉样品中优势菌属主要为乳球菌属Lactococcus、乳杆菌属Lactobacillus,其中RKZ中Lactococcus为(63.73±8.29)%,Lactobacillus为(4.15±1.38)%;SN中Lactococcus为(25.82±1.42)%;CD中Lactococcus为(46.62±4.60)%,Lactobacillus为(9.20±6.49)%;AL中Lactococcus为(85.13±2.99)%,Lactobacillus为(8.57±1.33)%,与曹磊等[12]研究的青海甘南地区牦牛曲拉群落结构存在共性的结果一致。乳杆菌属和乳球菌属均为乳酸菌,可在曲拉制作的初期产生大量的乳酸、乙酸和一定量的胞外多糖影响曲拉风味[13-14],从优势菌角度分析,以上几个地区的曲拉风味特点存在一定的相似性。而LS中优势菌属与其他地区差异较为明显,其第一优势菌为雷尔氏菌属Ralstonia为(17.25±7.05)%,且致病菌假单胞菌属Pseudomonas相对丰度达(16.05±3.22)%、苍白杆菌Ochrobactrum达(9.52±3.19)%,均明显高于其他地区,从优势菌角度分析其风味及品质特点相对其他地区具有一定特殊性。
2.5 物种丰富度热图
根据OTU的丰度数据,热图可在属水平上将不同的OTU分块聚类,并根据颜色梯度反应不同样品中细菌群落相似性、差异性及物种聚类关系。利用R语言绘制热图,可直观地显示不同产区曲拉样品中细菌的差异。由图2可知,上侧为样品聚类树,左侧为OTU聚类树,西藏7个地市的21个曲拉样品可分为2个大聚类:LS1和LS3可聚为一类,其余19个样品聚为一类;物种丰富度上红色越亮代表该菌属在该样品中的相对含量高于横向的其他样品,绿色越亮则相反,不同地区主要优势菌属存在一定差异。
表4 不同地区曲拉优势菌分布表 单位:%
Table 4 Score table of the dominant bacterial genera in different Qula samples

样品第一优势菌第二优势菌第三优势菌第四优势菌LSRalstonia17.25±7.05Pseudomonas16.05±3.22Pelomonas11.05±4.89Ochrobactrum9.52±3.19RKZLactococcus63.73±8.29Acetobacter23.18±10.23Lactobacillus4.15±1.38Bifidobacterium1.15±0.87SNLactobacillus57.21±3.11Lactococcus25.82±1.42Leuconostoc8.01±1.09Chryseobacterium1.73±0.28CDLactococcus46.62±4.60Acetobacter16.93±3.38Pseudomonas9.98±5.38Lactobacillus9.20±6.49LZLactococcus57.60±3.26Serratia14.40±0.91Pseudomonas5.14±0.41Acinetobacter4.08±0.34NQLactobacillus32.77±3.20Anoxybacillus27.82±3.00Ralstonia8.52±1.96Pelomonas5.47±1.38ALLactococcus85.13±2.99Lactobacillus8.57±1.33Acetobacter3.34±1.36Pseudomonas1.36±0.09
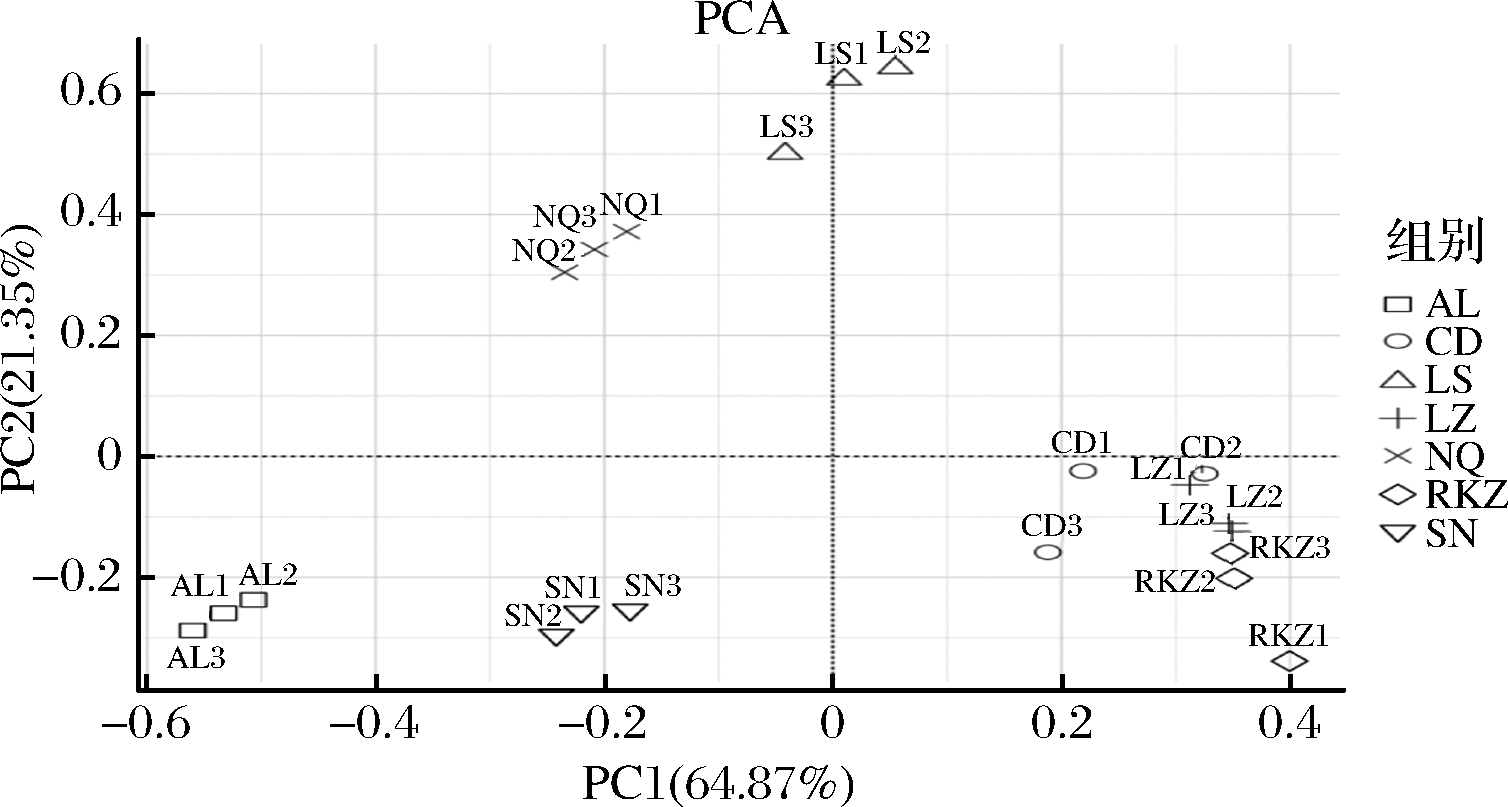
2.6 基于UniFrac距离的样本聚类分析
由图3可知,细菌群落第1主成分(PC1)的贡献率是64.87%,细菌群落第2主成分(PC2)的贡献率是21.35%,两者之和为86.22%,大于70%,表明两个成分较好地代表了曲拉样品中的细菌群落信息。不同曲拉样本的菌群与地理位置存在一定关系,同一个采样区的样本具有一定的聚类趋势,同时发现LZ、RKZ和CD可聚为一类,而其他4个地区的样品较为离散,各自独立。
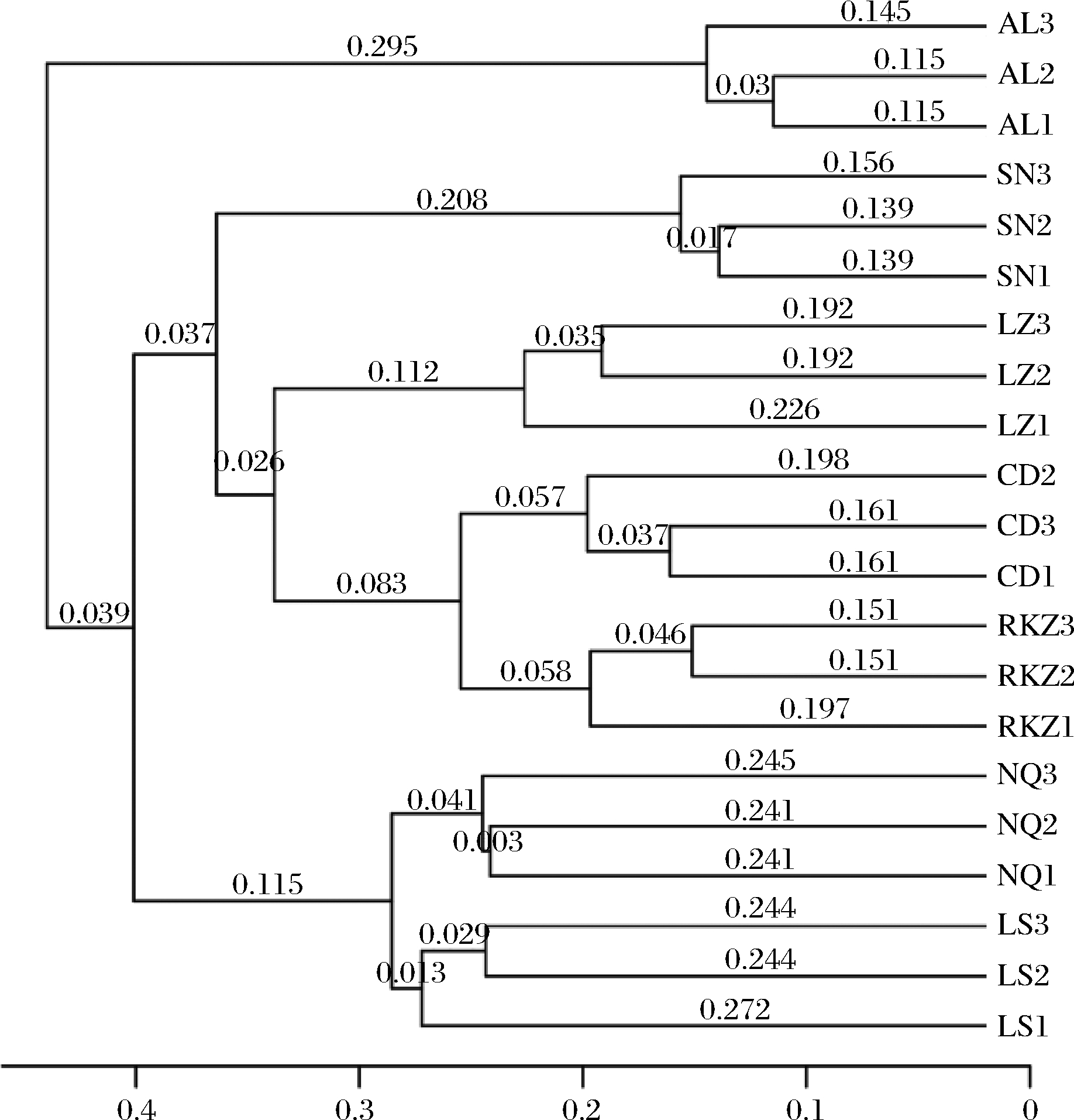
2.7 UPGMA聚类分析图
聚类分析主要指层次聚类(hierarchical clustering)的分析方法,以等级树的形式展示样品之间的相似度,通过聚类树的分枝长度衡量聚类效果的好坏。样本根据彼此之间的相似度聚类,样本间的分枝长度越短,两样本越相似。由图4可知,不同采样地的样品聚集到不同的类别,说明不同来源样品的微生物多样性存在一定的差异性,这与PCA分析的结果一致。
2.8 样品功能类群分布统计
通过将现有的16S rRNA基因测序数据与代谢功能已知的微生物参考基因组数据库相对比,从而实现对细菌和古菌代谢功能的预测。由图5可知,7个样品中细菌群落的代谢功能主要集中在氨基酸代谢(amino acid metabolism)、碳水化合物代谢(carbohydrate metabolism)和能量代谢(energy metabolism),其中AL和SN样品在氨基酸代谢丰度上明显低于其他5个地区样品,而在碳水化合物代谢丰度上LS样品显著地区其它6个地区样品,而在能量代谢丰度上7个地区样品差异不显著。
3 讨论
本研究采用高通量测序技术对西藏7个地市共计21份曲拉样品的细菌群落结构进行分析,从属水平上群落结构分布可知,不同地区曲拉细菌多样性存在一定差异且存在一定的地域独立性,如拉萨(LS)和那曲(NQ)样品的细菌种类远高于其他5个地区,细菌丰度上除拉萨地区革兰氏阴性植物病原细菌雷尔氏菌属(Ralstonia)和致病菌假单胞菌属(Pseudomonas)占主要优势菌群外,其他6个地市的优势菌均主要集中于发酵乳制品常见菌乳球菌属(Lactococcus)和乳杆菌属(Lactobacillus),这6个地区曲拉的主要优势细菌群落结构与SUN等[15]对青海牦牛酸奶进行纯化培养和16S rRNA鉴定的结果一致,同时与其他传统发酵乳制品如酸牛奶、酸马奶、酸驼奶等细菌群落多样性类似[16-17],在门水平上常见原料乳的细菌组成差异不明显[18-19]。而本研究发现拉萨地区样品存在一定的植物病原菌菌属和条件致病菌菌属。通过功能分析发现曲拉中细菌群落有助于人体消化过程中的氨基酸和碳水化合物代谢。
由于本研究采样点较单一及样品量相对较少,并不能直接反映出制备环境直接造成的影响,为进一步探讨拉萨地区牦牛曲拉的细菌多样性及安全性,应增加采样点和采样量,进一步开展细致研究。
参考文献
[1] 王军,王琳琳,韩玲,等.曲拉热风干燥工艺参数筛选及色泽品质评价[J].农业工程学报,2014,30(16):296-304.
[2] 张蓓. 藏族传统曲拉制作过程中乳酸菌群变化及曲拉中益生性乳杆菌的筛选和功能性评价[D].郑州:郑州大学,2017.
[3] 于桂荣,张兰威.提高曲拉精制干酪素色泽质量的研究[J]. 东北农业大学学报,2006,37(6):775-778.
[4] LIU H N, ZHANG C, ZHANG H, et al. pH treatment as an effective tool to select the function and structural properties of yak milk caseins[J]. Journal of Dairy Science, 2013, 96(9): 5 494-5 500.
[5] 罗丽,孙科,鲁迎瑞,等.顶空固相微萃取-气质联用技术分析曲拉干酪素挥发性成分及来源[J].食品工业科技,2016,37(14):83-89.
[6] 卿蔓君,白梅,张勇,等.西藏曲拉和云南乳饼中酵母菌的鉴定及其生物多样性[J].微生物学报,2010,50(9):1 141-1 146.
[7] 郭婷,张健,杨贞耐.基于不同发酵方法制作的曲拉中挥发性风味物质分析[J].食品工业科技,2017,38(8):209-213;238.
[8] GIRAFFA G,NEVIANI E. DNA-based,culture-inde-pendent strategies for evaluating microbial communities in food-associated ecosystems[J].International Journal of Food Microbiology,2001,67(1-2):19-34.
[9] WANG B, PENG Y, GUO Y, et al.Illumina MiSeq sequencing reveals the key microorganisms involved in partial nitritation followed bysimultaneous sludge fermentation, denitrification and anammox process. Bioresource Technology, 2016, 207: 118-125.
[10] NIEMINEN T T, KOSKINEN K, LAINE P, et al. Comparison of microbial communities in marinated and unmarinated broiler meat by metagenomics[J]. Int J Food Microbiol, 2012,157(2):142-149.
[11] 张敏,张艳,黄丽丽,等.基于16S rDNA高通量测序方法比较新疆西北部地区乳品中微生物的多样性[J].食品科学, 2017,38(20): 27-33.
[12] 曹磊,梁春御,曹瑛瑛,等.甘南地区牦牛曲拉中细菌群落结构研究[J]食品科学,2019,40(22):103-109.
[13] BAE K H, SHIN K S, RYU H Y, et al. Identification and fermentation characteristics of lactic acid bacteria isolated from the fermentation broth of Korean traditional liquor Andong-Soju[J]. Korean J Microbiol Biot, 2007, 35(4): 310-315.
[14] PARK Y K, LEE J H, MAH J H. Occurrence and reduction of biogenic amines in traditional Asian fermented soybean foods: A review[J]. Food Chem, 2019,278:1-9.
[15] SUN Z H,LIU W J,GAO W,et al.Identification and characterization of the dom inant lactic acid bacteria from kurut: The naturally ferm ented yak m ilk in Qing-hai,China[J].The Journal of General and Applied M icrobiology,2010,56(1):1-10.
[16] 刘亚华,王宇,徐海燕,等. 基于PacBio SMRT技术的鲜马奶细菌多样性研究[A]. 中国食品科学技术学会.益生菌:技术及产业化——第十三届益生菌与健康国际研讨会摘要集[C].北京:中国食品科学技术学会,2018:2.
[17] 其木格苏都. 自然发酵酸马奶细菌多样性及其基因动态变化研究[D].呼和浩特:内蒙古农业大学,2017.
[18] 高佳媛,邵玉宇,雷飞艳,等.陕西关中地区羊乳中细菌多样性的分析[J].食品与发酵工业,2018,44(1):52-59.
[19] 剧柠,夏淑鸿.原料乳中微生物的多样性[J].食品与发酵工业,2013,39(2):150-155.