表1 MPH突变基因扩增引物序列
Table 1 Oligonucleotides used for MPH mutagenesis

引物名称引物序列(5′-3′)E94A-FGCAACCTCGGTCACCGGTTACCTCGTCAACACCGE94A-RGAGCGGCGCTTTCTGGAAGGACTTGGCCAGN329A-FGTGAACTACTCGGTCGTCGCCCCCAAGTAAGCTTGATCCG-GCTGCTN329A-RGACGACCGAGTAGTTCACCGGCACGAAACG
有机磷农药在当今农业生产中扮演着不可或缺的角色,包括甲基对硫磷、甲异呋喃、单氯磷、乐果等[1]。有机磷农药的大量使用带来了土壤污染、水资源污染及农药残留等问题[1-2]。甲基对硫磷水解酶(methyl parathion hydrolase, MPH)作为一类有机磷降解酶,可有效水解甲基对硫磷等有机磷农药[3-4],在有机磷农药残留检测和清洗剂领域具有良好的应用前景。与色谱检测法[5]、免疫分析法[6]、酶抑制法[7-9]相比,基于MPH的传感器检测方具有简单、快速、成本低廉、检测仪器体积小等优势[7]。以MPH为主要功能成分的“比亚酶”成为2008年北京奥运会和2010年上海世博会的果蔬产品指定的洗涤用品[10]。
研究表明,表面活性剂(Tween-20、Tween-80及十二烷基硫酸钠等)和部分盐离子(Cd2+、Ni2+、Zn2+及Cu2+)能抑制MPH催化活性,而Na+、K+、Ca2+、Co2+及Mn2+对MPH催化有促进作用[11]。一般认为,盐离子通过与催化活性基团的相互作用来抑制MPH的催化活性。例如,添加Cd2+能破坏MPH催化活性中心His与2个Cd2+、1个水分子形成金属中心,从而抑制催化活性[4]。盐离子促进MPH催化活性的机理尚未报道。
近年来,研究者已发展多种理论分析蛋白质-配体相互作用的方法,如分子对接[12-13]、分子动力学(molecular dynamics,MD)[14]、量子力学/分子力学联用[15-16] 等。分子对接可静态的展示蛋白质结构与配体的相对位置,但可靠性不如MD[17-19]。UL-HAQ等[20]利用MD揭示了酪蛋白激活酶Ⅱ与抑制剂的结合模式及抑制机理,并利用此机理设计新的抑制剂。POTTERTON等[21]基于转向分子动力学计算了G蛋白与配体的相对结合时间。POTTERTON 等[21]结合三维结构模拟及MD发现了真核延伸因子-2激酶与其抑制剂的结合机理[21]。上述研究为揭示盐离子调控MPH的机制提供了方法学基础。
在本研究中,首先考察了不同浓度K+对Pseudomonas sp. WBC-3 MPH的催化活性的影响,通过MD分析了MPH与K+结合-分离的动态过程,并基于分布概率及定点突变分析确定了MPH中与K+相互作用的关键氨基酸位点。研究结果将为MPH催化活性的改造提供有效靶点。
编码Pseudomonas sp. WBC-3 MPH基因(PDB:1P9E)的载体pET-20b/MPH,于本实验室前期构建。克隆宿主E.coli JM109和表达宿主E.coli BL21(DE3),购于美国Invitrogen公司。Prime STAR HS DNA聚合酶和胶回收试剂盒,购于Takara公司;甲基对硫磷,购于北京坛墨质检科技有限公司;Ni2+亲和柱、脱盐柱,购于GE Healthcare公司。CloneExpress Ⅱ聚合酶购于南京诺唯赞生物科技有限公司。
1.2.1 定点突变质粒的构建
以pET-20b/MPH为模板,通过全质粒PCR扩增构建得到编码MPH突变体基因的表达质粒。引物对E94A-F/E94A-R和N329A-F/N329A-R,分别对应突变体E94A和N329A。PCR条件为:98 ℃、10 s,64 ℃、5 s,72 ℃、5 min,34个循环。Dpn I消化PCR产物2 h后胶回收,经磷酸化后过夜连接,并转化E.coli JM109,并进行基因测序验证。从E.coli JM109中提取质粒转化至E.coli BL21 (DE3),获取阳性转化子(表1)。
表1 MPH突变基因扩增引物序列
Table 1 Oligonucleotides used for MPH mutagenesis

引物名称引物序列(5′-3′)E94A-FGCAACCTCGGTCACCGGTTACCTCGTCAACACCGE94A-RGAGCGGCGCTTTCTGGAAGGACTTGGCCAGN329A-FGTGAACTACTCGGTCGTCGCCCCCAAGTAAGCTTGATCCG-GCTGCTN329A-RGACGACCGAGTAGTTCACCGGCACGAAACG
注:下划线指突变位点
1.2.2 重组菌的摇瓶发酵
卡那霉素平板上活化12 h后,挑取单菌落接种于LB培养基,37 ℃、220 r/min振荡过夜培养。以1%的接种量将种子液转接与TB培养基,37 ℃、220 r/min振荡培养至OD600为0.6。加入1 mmol/L异丙基硫代半乳糖苷(IPTG)于25 ℃诱导24 h。
1.2.3 蛋白质纯化
离心收集菌体,2倍体积50 mmol/L磷酸缓冲液洗涤菌体3遍,并超声波破碎30 min。细胞破碎液于12 000 r/min、4 ℃离心30 min,取上清液Ni2+亲和柱上样,20~500 mmol/L 咪唑梯度洗脱,将含MPH的洗脱液于脱盐柱脱盐,纯酶保存于50 mmol/L pH为7.0的Tris-HCl缓冲液中。
1.2.4 盐离子对MPH酶活的影响
用50 mmol/L pH为8.9 的Tris-HCl缓冲液分别配制含3 mol/L NaCl和KCl的含盐缓冲液,并将其稀释至0.2~2 mol/L。测定不同盐浓度条件下MPH催化活性。
1.2.5 酶活检测
MPH酶活单位定义:在30 ℃,pH 为8.9的Tris-HCl缓冲体系中,每分钟催化甲基对硫磷生产1 μmol 对硝基苯酚所需要的酶量。建立对硝基苯酚在405 nm吸光度值与浓度的标准曲线,以甲基对硫磷为底物测量酶活。
1.2.6 分子动力学模拟
利用分子动力学分析软件Amber (http://ambermd.org/)模拟K+与MPH相互作用。分析过程如下:基于ff14SB力场构建MPH的溶剂盒子,并添加平衡离子及1 mol/L K+;使用共轭梯度法对整个体系能量最小化;将体系逐步升温至310 K,并于此温度使体系平衡,并持续20 μs。
为考查盐离子对MPH活性的影响,分别测定不同KCl及NaCl浓度下MPH的催化活性。如图1所示,随着KCl浓度提高至2 mol/L,MPH活性迅速提高;KCl浓度达到3 mol/L时,MPH活力较无KCl时提高13.39倍。然而,增加NaCl浓度并未能使MPH活性明显提高。上述结果表明,K+能促进MPH的催化反应。
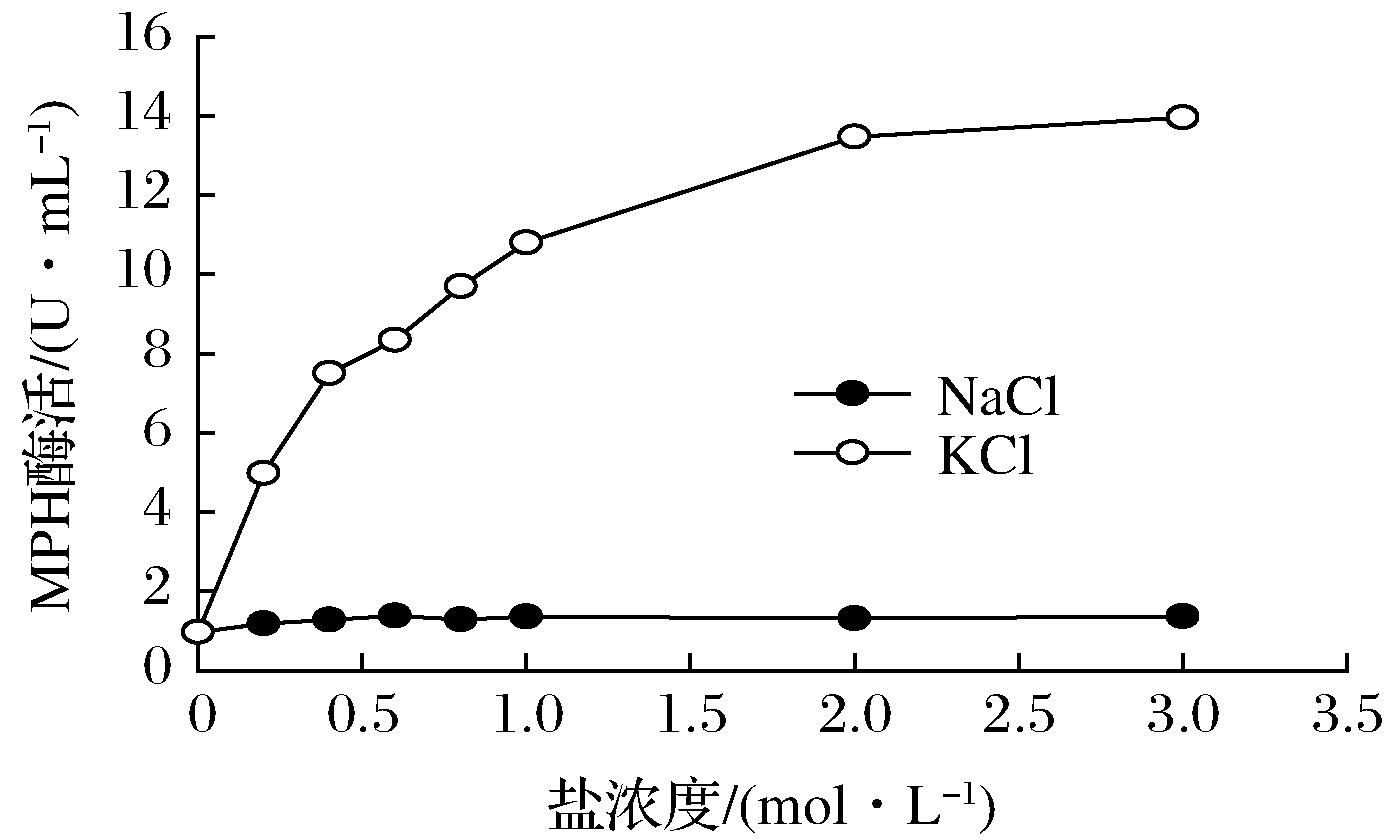
图1 盐离子对MPH酶活的影响
Fig.1 The effect of salts on MPH activity
2.2.1 K+分布概率计算方法的建立
为分析K+与MPH的相互作用,对两者混合体系进行MD分析预测K+与MPH可能的作用位点。为防止K+初始位置对MD分析结果的影响,分别将K+和MPH置于溶剂盒子外围和中央。在300 K下运行MD,6 000 ps 后RMSD趋于平缓,此时K+已分布于整个溶剂盒子,MD体系达到平衡。继续运行20 ns,每2 ps 取1次样,即共10 000次取样。在MD分析过程中,以MPH某原子为中心的圆形区域(半径为R)出现K+的帧数与总帧数的比值定义为原子临近区域K+分布概率,氨基酸残基中原子临近区域K+分布概率的最大值则定义为该氨基酸临近区域K+分布概率。
上述离子分布概率的预测方法可用于表征K+与MPH氨基酸残基的相互作用,但MPH原子邻近区域半径和K+随机运动会影响预测方法的准确性。
2.2.2 MPH原子邻近区域半径对K+分布概率预测的影响
氨基酸残基仅能与一定范围的K+产生有效的相互作用,设定过大的MPH原子邻近区域半径会引入氨基酸与离子相互作用之外的噪音。因此,分别计算3、4、 5 Å K+在氨基酸残基附近的分布概率。不同的分布半径显示了一致的K+分布规律。MPH原子邻近区域半径越大,MPH原子邻近区域K+分布概率越大,但同时引入了大量噪音(图2)。因此,选择3 Å作为MPH原子邻近区域半径。

a-3Å;b-4Å;c-5Å
图2 MPH原子邻近区域不同半径下的K+分布概率
Fig.2 Distribution probability of K+ at different radii around atoms of MPH
2.2.3 水溶剂主体中K+随机运动对K+分布概率预测的影响
K+在水溶剂主体中的随机运动和初始位置可能影响MPH氨基酸残临近区域K+分布概率预测。为消除初始位置对计算结果的影响,在运行MD前使K+远离MPH。MD分析显示,K+随机运动的平均RMSF为60 Å(图3),远大于而溶剂盒子的半径10 Å。上述结果,在20 ns分析范围内,设定的MD分析体系中各区域K+与MPH具有同等的接触机会。
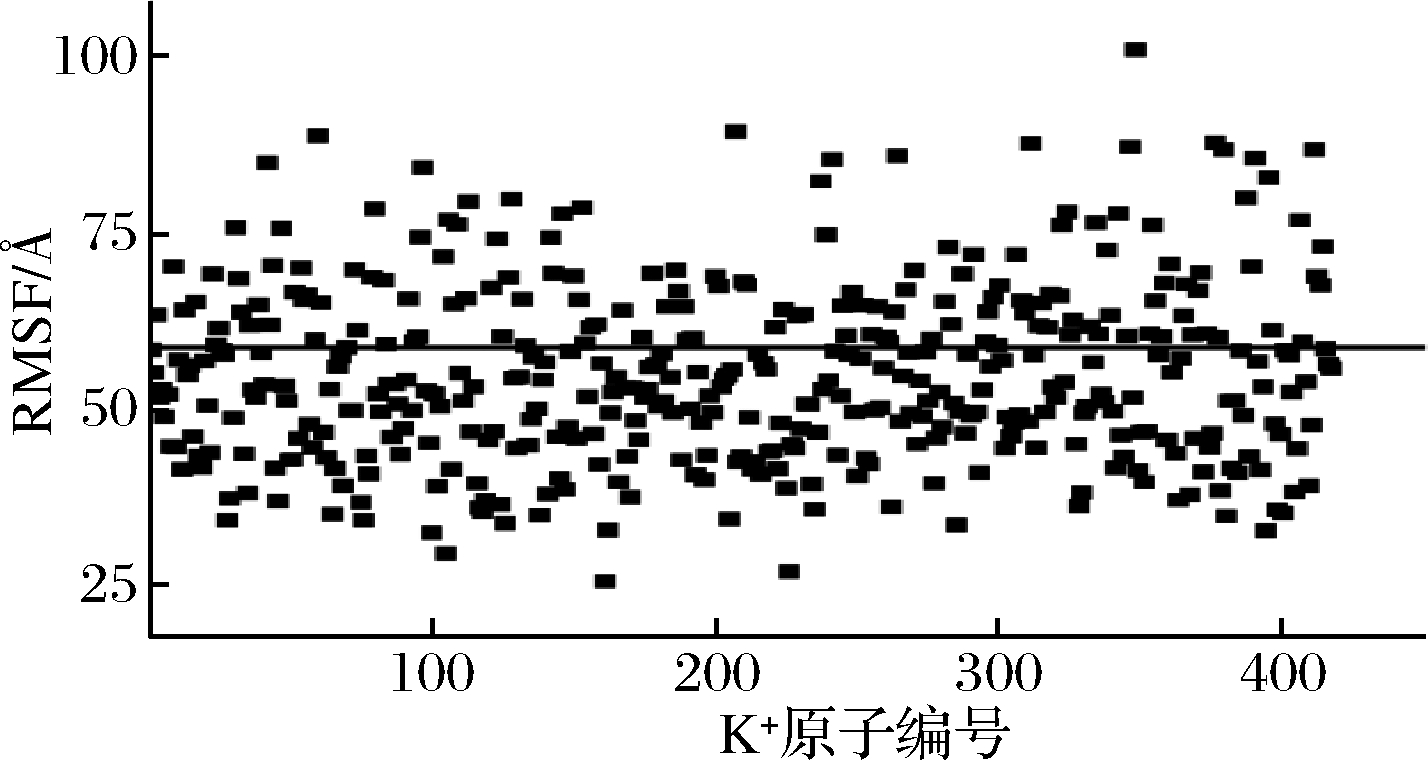
图3 K+在水溶剂中的RMSF分析
Fig.3 RMSF analysis of K+ in the aqueous solvent
2.2.4 MPH氨基酸残基临近区域K+随机运动对K+分布概率预测的影响
基于酶学分析的结果,K+通过与特定氨基酸残的相互作用来改变酶的催化特性。然而,K+迁移至MPH氨基酸残基临近区域时,其随机运动有干扰K+分布概率预测的可能。参照蛋白质分子中“氢键寿命”,定义了氨基酸临近区域K+寿命(MPH氨基酸残基临近区域K+连续出现的帧数所对应的时间)。MD分析显示,氨基酸残基临近区域中单个K+最长寿命与该区域内总体K+最长寿命基本一致(图4)。因此,MPH氨基酸残基临近区域K+随机运动对K+分布概率预测的影响不明显。

a-单个K+;b-总体k+
图4 单个与总体K+最长寿命
Fig.4 The longest life the certain and whole K+
MPH与1 mol/L K+的MD分析显示,临近区域K+分布概率大于5%的原子数仅占MPH总原子数的1.19%(图5-a)。这表明MPH中绝大部分K+分布概率较低。故分析统计临近区域K+分布概率大于5%的氨基酸残基数量,以确定K+作用的氨基酸偏好性。分析结果显示,临近区域K+分布概率大于5%的氨基酸残基数量最多的是Asp,其次是Glu、Gln和Asn(图5-b)。这说明K+主要偏好酸性氨基酸,其次偏好于酰胺类氨基酸。
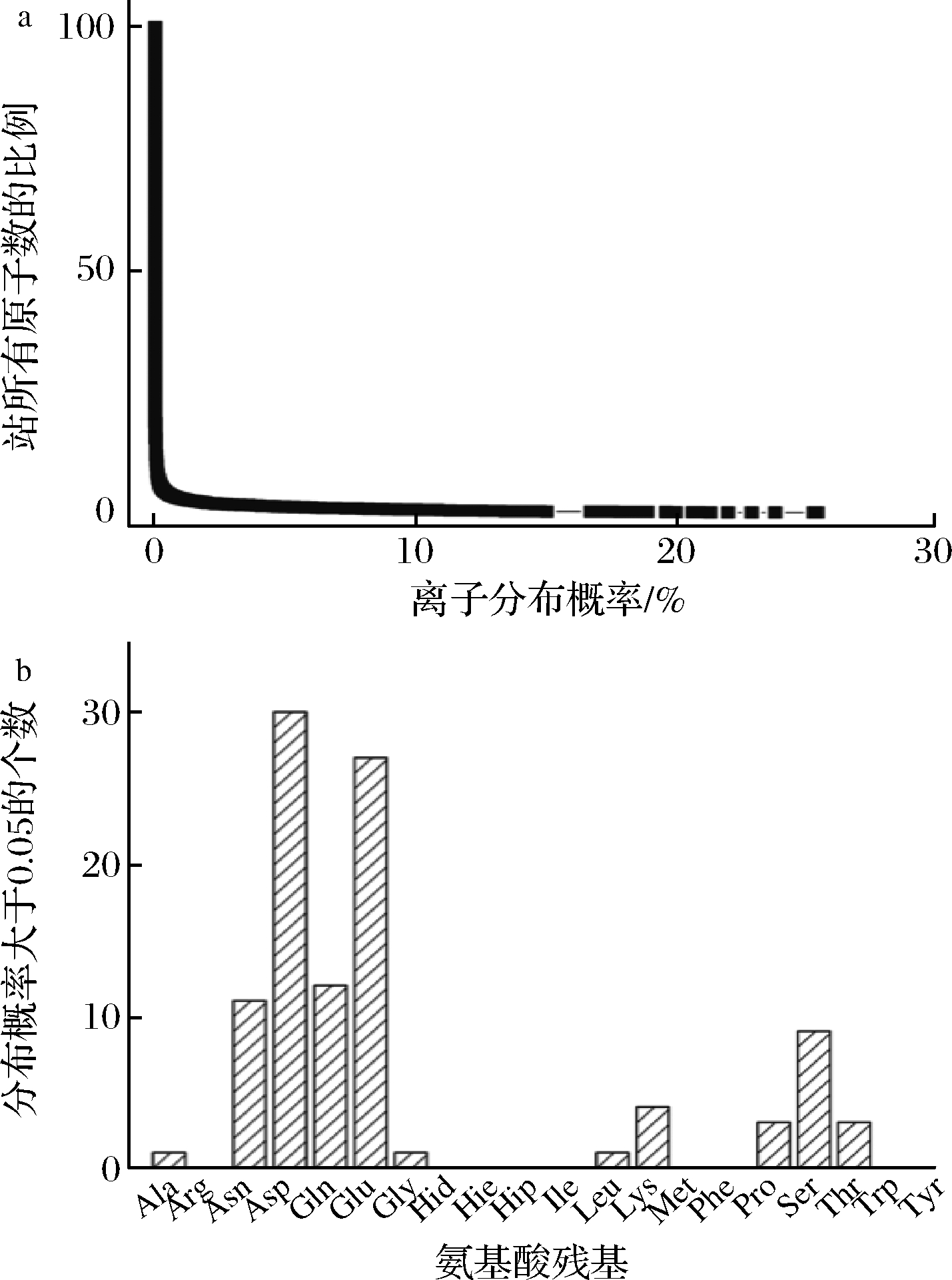
a-高于某分布概率原子的占比;b-氨基酸残基对K+的偏好性
图5 MPH氨基酸残基对K+的偏好性
Fig.5 The preference of MPH amino acid residues to K+
为考察K+对MPH蛋白结构的影响,通过MD分析了MPH结构柔性值RMSF。结果显示,相对于无K+条件下,1 mol/L K+显著降低了MPH 中A85-T95与V323-N329的RMSF,表明2个区域柔性明显下降(图6-a)。在A85-T95中,临近区域K+分布概率大于5%的氨基酸残基为 K90(0.061 8)、P92(0.057 4)、E94(0.1250)。其中,K90与P92临近区域K+分布概率主要受E94影响,故后者可能是该区域内与K+作用的关键氨基酸。在V323-N329,临近区域K+分布概率大于5%的氨基酸残基分别为S326(0.208 5)、V327(0.254)、N329(0.181)。其中,S326与V327临近区域K+分布概率主要受N329影响,故后者可能是该区域内与K+作用的关键氨基酸。

a-MPH氨基酸残基;b-MPH突变体
图6 不同K+浓度下MPH氨基酸残基RMSF和MPH突变体在1 mol/L K+浓度下的相对酶活
Fig.6 RMSF of amino acid residues at different K+ concentrations and the relative enzyme activity of MPH mutant at 1 mol/L K+ concentration
通过丙氨酸取代,进一步验证E94与N329对K+作用的影响。与野生酶相比,E94A与N329A的相对酶活(1 mol/L K+/0 mol/L K+溶液)分别降低26%与33%,组合变体 E94A/N329A的降低53%(图6-b)。上述结果表明,E94与N329是K+调控MPH催化活性的关键位点。
盐离子是影响酶催化性能的重要因素。研究发现,K+浓度对Pseudomonas sp. WBC-3 MPH催化活性有重要影响。基于MPH进行分子动力学分析,建立了MPH氨基酸残基临近区域K+分布概率的预测方法。在1 mol/L K+溶液中,K+倾向分布于MPH的Asp、Glu、Gln及Asn残基附近,并降低了A85-T95与V323-N329的柔性。将A85-T95与V323-N329柔性变化最大的E94与N329分别突变为Ala,使MPH相对催化活性(1 mol/L K+溶液/0 mol/L K+溶液)分别较野生酶降低26%与33%,两者同时突变为Ala,其相对活性降低53%。因此,K+可能通过与E94、N329相互作用来调控MPH催化,为其催化活性的分子改造提供了靶点。研究结果也为其它酶受盐离子调控的机制及确定改造靶点提供参考。
[1] LI Fuhou, WANG Weixia. Research progress of microbial degradation of organophosphorus pesticides[J]. Journal of Anhui Agricultural Sciences, 2009, 37 (22): 10 659-10 661.
[2] CUI Weiwei, ZHANG Qiangbin, ZHU Xianlei. Advance in studies on the harms and exposure of pesticide residues[J]. Journal of Anhui Agricultural Sciences, 2010, 38 (2): 883-884.
[3] JI Xiyan, WANG Qiang, ZHANG Wudi, et al. Research advances in organophosphorus pesticide degradation: A review[J]. Fresenius Environmental Bulletin, 2016, 25(5): 1 556-1 561.
[4] DONG Yanjie, BARTLAM M, SUN Lei, et al. Crystal structure of methyl parathion hydrolase from Pseudomonas sp. WBC-3[J]. Journal of Molecular Biology, 2005, 353 (3): 655-663.
[5] HARSHIT D, CHARMY K, NRUPESH P. Organophosphorus pesticides determination by novel HPLC and spectrophotometric method[J]. Food Chemistry, 2017, 230: 448-453.
[6] BOULANOUAR S, MEZZACHE S, COMB S A, et al. Molecularly imprinted polymers for the determination of organophosphorus pesticides in complex samples[J]. Talanta, 2018, 176: 465-478.
S A, et al. Molecularly imprinted polymers for the determination of organophosphorus pesticides in complex samples[J]. Talanta, 2018, 176: 465-478.
[7] HASSANI S, MOMTAZ S, VAKHSHITEH F, et al. Biosensors and their applications in detection of organophosphorus pesticides in the environment[J]. Archives of toxicology, 2017, 91 (1): 109-130.
[8] MISHRA R K, HUBBLE L J, MARTIN A, et al. Wearable flexible and stretchable glove biosensor for on-site detection of organophosphorus chemical threats[J]. ACS Sensors, 2017, 2 (4): 553-561.
[9] ZHANG Yuanyuan, ARUGULA M A, WALES M, et al. A novel layer-by-layer assembled multi-enzyme/CNT biosensor for discriminative detection between organophosphorus and non-organophosphrus pesticides[J]. Biosensors and Bioelectronics, 2015, 67: 287-295.
[10] 钟欣. “863” 降农残成果比亚酶转民用[J]. 化工管理, 2015(31): 66.
[11] 徐玮. 邻单胞菌M6甲基对硫磷水解酶纯化及性质的研究[D].南京:南京农业大学, 2005.
[12] BENDER B J, CISNEROS Ⅲ A, DURAN A M, et al. Protocols for molecular modeling with Rosetta3 and RosettaScripts[J]. Biochemistry, 2016, 55 (34): 4 748-4 763.
[13] PIRHADI S, SUNSERI J, KOES D R. Open source molecular modeling[J]. Journal of Molecular Graphics and Modelling, 2016, 69: 127-143.
[14] MCCAMMON J A, GELIN B R, KARPLUS M. Dynamics of folded proteins[J]. Nature, 1977, 267 (5612): 585-590.
[15] FRIESNER R A, GUALLAR V. Ab initio quantum chemical and mixed quantum mechanics/molecular mechanics (QM/MM) methods for studying enzymatic catalysis[J]. Annual Review of Physical Chemistry, 2005, 56: 389-427.
[16] SENN H M, THIEL W. QM/MM methods for biomolecular systems[J]. Angewandte Chemie-international Edition, 2009, 48 (7): 1 198-1 229.
[17] FRIEDRICHS M S, EASTMAN P, VAIDYANATHAN V, et al. Accelerating molecular dynamic simulation on graphics processing units[J]. Journal of Computational Chemistry, 2009, 30 (6): 864-872.
[18] LE GRAND S, G TZ A W, WALKER R C. SPFP: Speed without compromise—A mixed precision model for GPU accelerated molecular dynamics simulations[J]. Computer Physics Communications, 2013, 184 (2): 374-380.
TZ A W, WALKER R C. SPFP: Speed without compromise—A mixed precision model for GPU accelerated molecular dynamics simulations[J]. Computer Physics Communications, 2013, 184 (2): 374-380.
[19] STONE J E, PHILLIPS J C, FREDDOLINO P L, et al. Accelerating molecular modeling applications with graphics processors[J]. Journal of Computational Chemistry, 2007, 28 (16): 2 618-2 640.
[20] UL-HAQ Z, ASHRAF S, BKHAITAN M M. Molecular dynamics simulations reveal structural insights into inhibitor binding modes and mechanism of casein kinase II inhibitors[J]. Journal of Biomolecular Structure & Dynamics, 2019, 37 (5): 1 120-1 135.
[21] POTTERTON A, HUSSEINI F S, SOUTHEY M W Y, et al. Ensemble-based steered molecular dynamics predicts relative residence time of A2A receptor binders[J]. Journal of Chemical Theory and Computation, 2019, 15 (5): 3 316-3 330.