沙门氏菌、大肠杆菌和金黄色葡萄球菌是3种主要的食源性病原菌,近年来关于3种病原菌引起食物中毒的报道较多[1-3],大多数中毒与食用受污染的牛奶、碎牛肉、水和乳制品有关。据统计可知,食源性疾病引起的腹泻和侵袭性疾病致死多达23万人[4],因此,对食源性病原菌进行快速准确的监测是控制和预防人类食源性病原菌感染的最有效途径之一。随着食源性病原菌检测技术的快速发展,检测技术从耗时较长的培养基筛选[5],发展到特异性较强的酶联免疫吸附试验(enzyme linked immunosorbent assay, ELISA)[6]和可同时检测多种病原菌的多重PCR[7],再延伸到基因探针[8]和生物传感器[9]等现代常用检测技术,这些检测方法在一定程度上具有快速、有效等优点,但是仍存在一些局限性,比如容易出现假阳性、成本高等。因此,迫切需要开发出一种具有样品预处理简单、成本低、灵敏度高、便于现场检测等优点的检测方法。表面增强拉曼光谱(surface-enhanced Raman scattering, SERS)利用不同菌种细胞壁的结构组成不同,产生独特的拉曼位移,广泛应用于食品病原菌检测鉴定[10]。
普通的拉曼信号非常微弱,在强背景荧光中很难被检测到,为了减少背景荧光的干扰,提高拉曼信号的强度,LAN等[11]采用金纳米颗粒基底的拉曼散射,应用PCA-HCA分析方法准确地区分出鼠伤寒沙门氏菌和大肠杆菌之间的差异。为了寻找增强效果更好的拉曼基底,WANG等[12]制备了金包覆磁性纳米颗粒,与金黄色葡萄球菌抗体结合,捕获细菌的检测限低至10 CFU/mL。此外,为了实现SERS技术在食品安全检测中的广泛应用,CHO等[13]研究了一种银增强技术和膜过滤器辅助的SERS方法用于快速检测碎牛肉中的大肠杆菌,分别在1和3 h内从纯培养物和碎牛肉中检测到极低浓度的大肠杆菌。
随着纳米技术的进步,便携式拉曼光谱仪已应用在食用油、农药和非法添加物等食品检测中[14-15],根据SERS技术的理论和应用实践,有望建立起快速、特异、灵敏的SERS病原菌检测。本研究建立的基于多重PCR和SERS技术的沙门氏菌、大肠杆菌和金黄色葡萄球菌检测方法,对食源性病原菌快速、精准的监测奠定基础。
1 材料与方法
1.1 材料与菌株
AgNO3、柠檬酸三钠,西陇化工股份有限公司;DNA marker,生工生物工程(上海)股份有限公司;2×Taq PCR Master Mix,novoprotein;粪便基因组提取试剂盒,天根生化科技有限公司。
大肠杆菌ATCC 44102、沙门氏菌ATCC 9150、福氏志贺氏菌、巴氏杆菌、变形杆菌、粪肠球菌、苏云金芽孢杆菌和枯草芽孢杆菌,兽医病理生物学与疫病防控安徽省重点实验室保存;金黄色葡萄球菌ATCC 6538,上海兽医研究所馈赠。
1.2 主要仪器设备
拉曼光谱仪,HORIBA堀场贸易有限公司;透射电镜,HT-7700日本日立公司;紫外-可见-近红外分光光度计,日本岛津公司;磁力加热搅拌器,常州普天仪器制造有限公司;PCR仪、超微量核酸蛋白分析仪,BIO-RAD;核酸电泳仪,北京六一生物科技有限公司。
1.3 细菌基因组DNA提取
采用“水煮法”提取细菌DNA,取2 mL在LB肉汤中过夜培养的菌液,12 000 r/min离心5 min,生理盐水洗涤菌体,重悬混匀后置于沸水中煮沸10 min,立即放入-20 ℃冰箱中冰冻30 min,室温溶解后离心取上清用于PCR扩增。
1.4 引物设计及引物特异性验证
根据 GenBank 中已发表的参考序列对沙门氏菌、大肠杆菌和金黄色葡萄球菌的hilA、uid A和clfA靶基因设计特异性引物(表1),引物由北京擎科生物技术有限公司合成。
1.5 多重PCR体系建立及条件优化
以大肠杆菌ATCC 44102、沙门氏菌ATCC 9150和金黄色葡萄球菌ATCC 6538为模板进行PCR扩增,优化引物浓度、退火温度和2×Taq PCR Master Mix用量。多重PCR反应体系:mix 12.5 μL,2 μmol/L的上下游引物各1 μL,模板1 μL,超纯水补足至25 μL。反应程序: 95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共30个循环;72 ℃后延伸7 min。PCR产物上样于2%琼脂糖凝胶,电泳后检测扩增片段大小。
表1 PCR引物序列
Table 1 PCR primer sequences
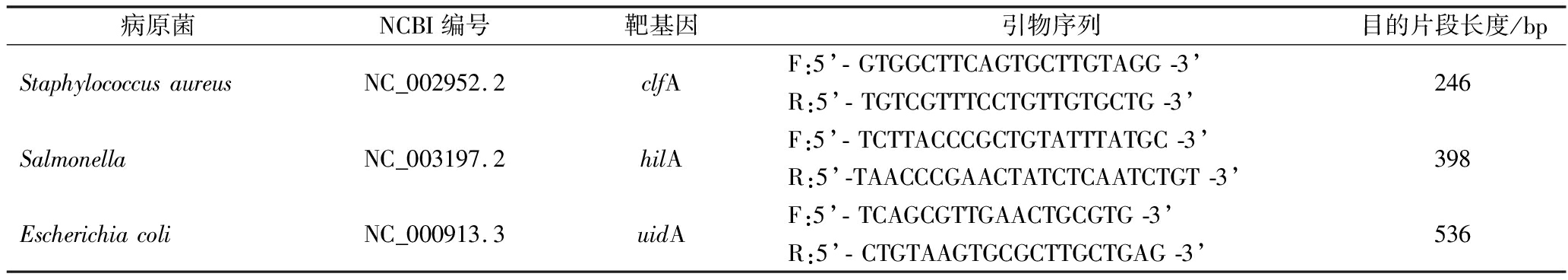
病原菌NCBI编号靶基因引物序列目的片段长度/bpStaphylococcusaureusNC_002952.2clfAF:5’-GTGGCTTCAGTGCTTGTAGG-3’R:5’-TGTCGTTTCCTGTTGTGCTG-3’246SalmonellaNC_003197.2hilAF:5’-TCTTACCCGCTGTATTTATGC-3’R:5’-TAACCCGAACTATCTCAATCTGT-3’398EscherichiacoliNC_000913.3uidAF:5’-TCAGCGTTGAACTGCGTG-3’R:5’-CTGTAAGTGCGCTTGCTGAG-3’536
1.6 多重PCR特异性和灵敏性检测
分别以标准阳性菌株和福氏志贺氏菌、巴氏杆菌、变形杆菌、粪肠球菌、苏云金芽孢杆菌和枯草芽孢杆菌等阴性对照菌株的DNA为模板,每个样品加入3对引物进行PCR扩增,验证多重PCR的特异性。
培养3种阳性菌株混合物至OD600为1.0,涂布平板计数确定细菌浓度后倍比稀释,用于检测多重PCR菌液灵敏度;提取阳性菌株的DNA,稀释为不同浓度的DNA模板,检测多重PCR的DNA敏感性。
1.7 银溶胶制备
称取33.72 mg的AgNO3,倒入200 mL灭菌去离子水的锥形瓶中,充分溶解后放置于磁力搅拌器上搅拌加热15 min直至沸腾,再加入8 mL质量分数为1%的柠檬酸钠,40 min后停止加热,待溶液温度冷却至室温后离心浓缩10倍。
1.8 银纳米颗粒的表征
用碳支持膜(铜网)蘸取浓缩好的银纳米颗粒以及银纳米颗粒包裹的细菌样品,滤纸吸去多余液体,晾干后上样于透射电子显微镜(transmission electron microscope, TEM)观察银胶粒径。
制备的银溶胶原液稀释10倍后,取3 mL加入石英比色皿中,使用紫外-可见-近红外分光光度计对其进行光吸收波谱扫描,观察银胶粒子的特征吸收峰。
1.9 SERS光谱采集
SERS样品制备:取1 mL菌液,8 000 r/min离心5 min得菌体沉淀,生理盐水洗涤2次后加入2 mL银溶胶,混匀置于石英比色皿,在拉曼仪液体池中进行光谱采集。
采用拉曼光谱仪进行检测前使用硅片进行校正,以520.7 cm-1峰作为基准峰,采用785 nm波长的激光器,激光强度设置为最大强度100 mw,拉曼光谱范围为400~1 800 cm-1,分辨率为2 cm-1,单次采谱时间为60 s,循环次数为1次、狭缝孔径为200 μm,PCA及光谱数据分析使用Origin 2019b软件。
1.10 表面增强拉曼光谱的灵敏性检测
菌液制备同1.6多重PCR灵敏度检测,混合银溶胶后进行SERS采集,用于检测SERS的灵敏度。
2 结果与分析
2.1 单重PCR中验证引物的特异性
单重PCR验证引物特异性的加样内容如表2所示,PCR扩增电泳图如图1-A所示,大肠杆菌uid A扩增出536 bp的条带、沙门氏菌hilA扩增出398 bp的条带,金黄色葡萄球菌clfA扩增出246 bp的条带,引物间无交叉反应。
表2 单重PCR验证引物特异性的加样内容
Table 2 Sample addition content of specificity in single PCR validation primer

编号12345678910菌株阴性对照441024410244102915091509150653865386538引物uidA-hilA-clfAuidAhilAclfAhilAuidAclfAclfAuidAhilA
2.2 多重PCR的特异性检测
分别以标准阳性菌株和阴性对照菌株的DNA为模板进行PCR扩增,结果如图1-B所示,阳性菌株均扩增出目的条带,而阴性菌株均未扩增出片段,表明多重PCR方法具有较好的特异性。
2.3 多重PCR的敏感性检测
以稀释后的阳性菌液为模板进行多重PCR扩增,结果如图2-A所示,多重PCR检测大肠杆菌、沙门氏菌、金黄色葡萄球菌的最低检出量为104 CFU/mL。
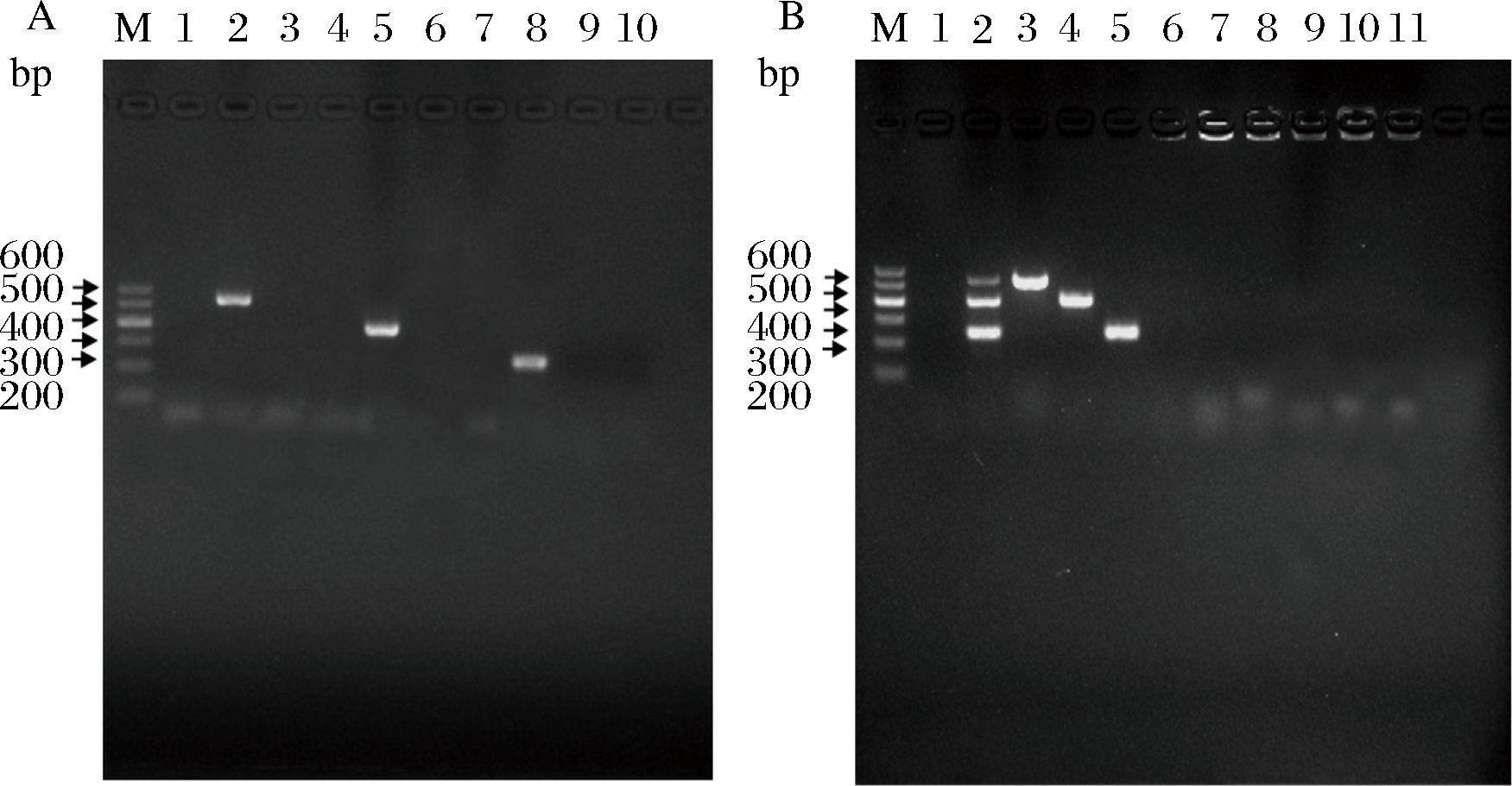
A-单重PCR引物特异性;B-多重PCR特异性检测;M-DL 600;1~5:dd H2O,混合阳性菌株,ATCC 44102,ATCC 9150,ATCC 6538;6~11:阴性菌株
图1 PCR特异性检测
Fig.1 PCR specific detection
以不同浓度的阳性菌株DNA为模板进行多重PCR扩增,结果如图2-B所示,多重PCR检测到3种病原菌的最低检出DNA量为50 pg/μL,具有较高的灵敏度。
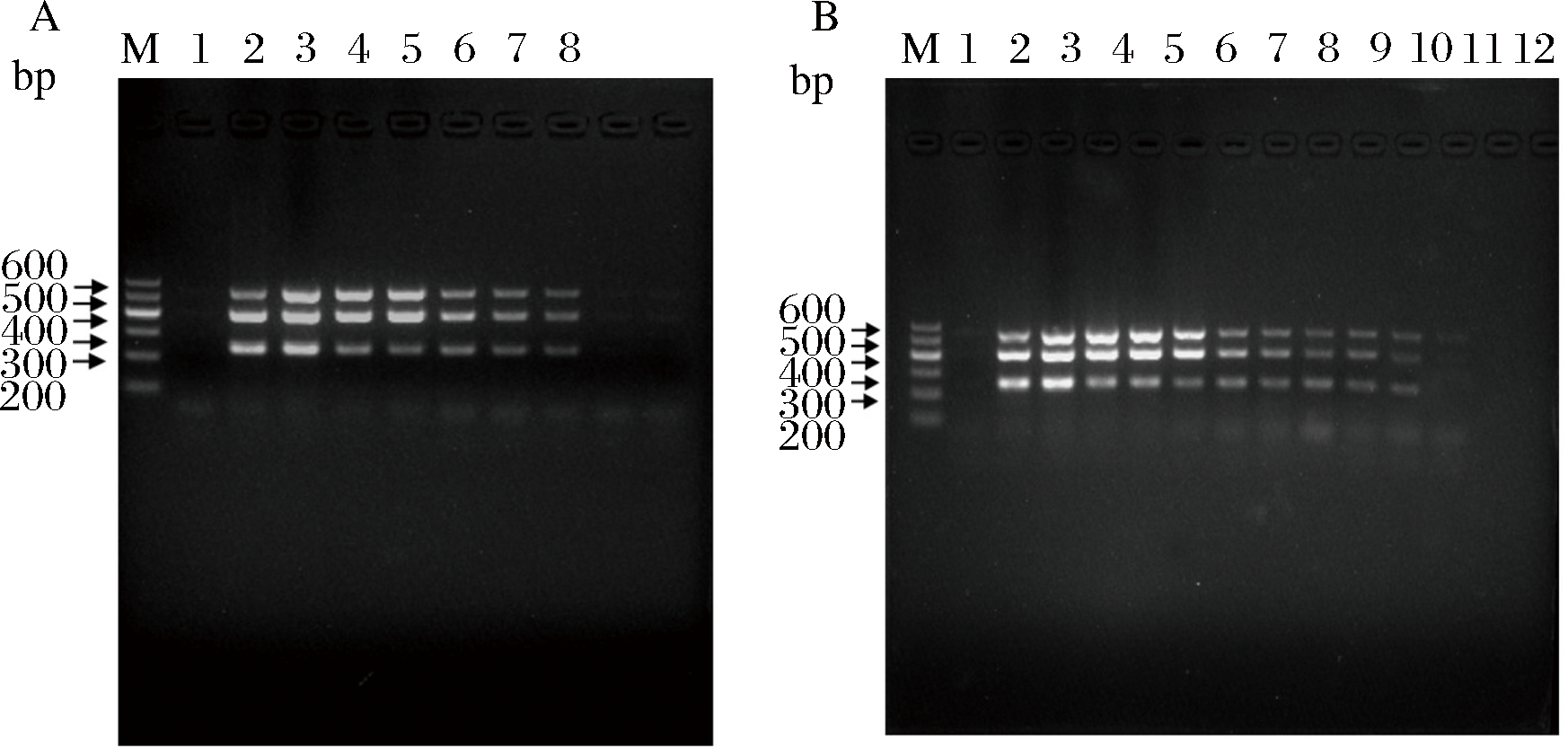
A-菌液敏感性;B-DNA敏感性;M-DL 600;A-1~8:dd H2O,混合阳性菌株,109,108,107,106,105,104 CFU/mL;B-1~12:dd H2O,混合阳性菌株,200 ng,100 ng,50 ng,10 ng,2 ng,1 ng,200 pg,100 pg,50 pg,10 pg
图2 多重PCR的敏感性检测
Fig.2 Sensitivity test of multiplex PCR
2.4 银纳米颗粒的表征
紫外可见光谱法和透射电镜观察是用于AgNPs结构表征的最广泛使用的技术,透射电镜观察到银纳米颗粒呈球状,大小均匀,粒径约40 nm(图3-A)。紫外可见光谱法检测到银胶的吸收峰在410 nm左右(图3-B)。
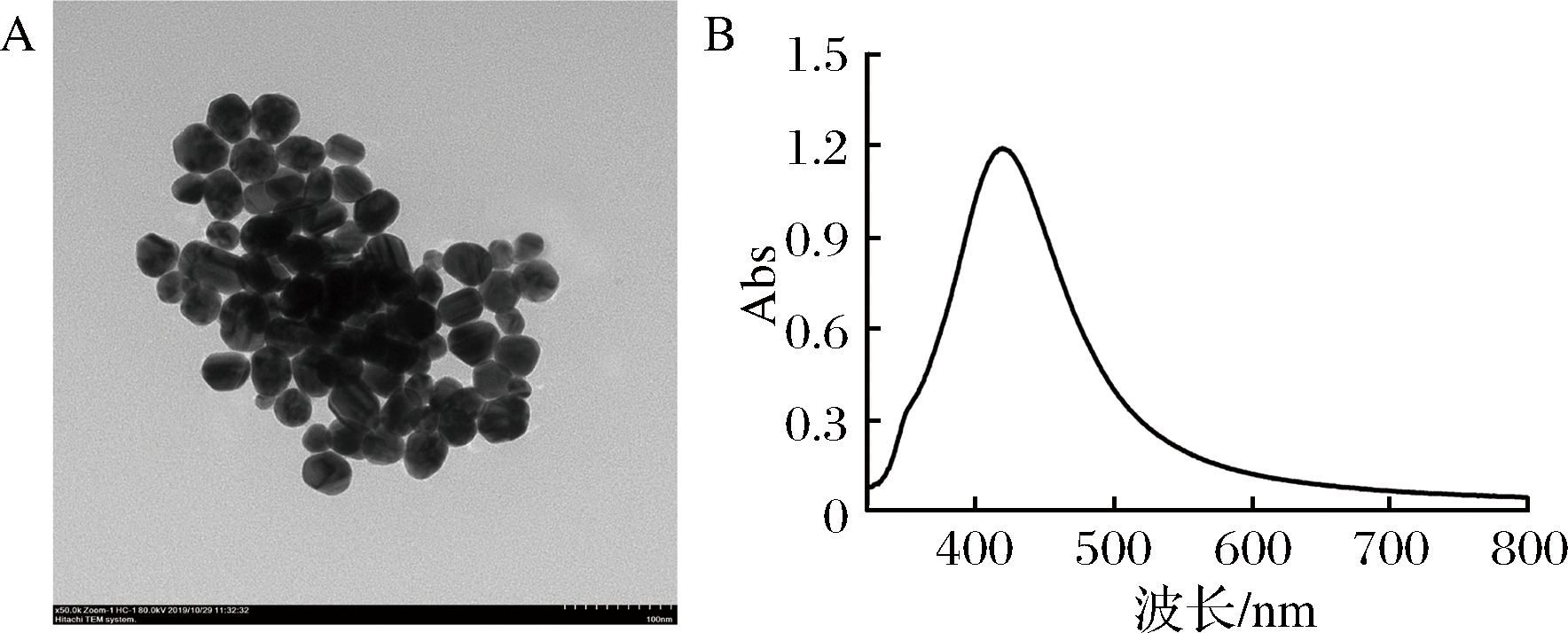
A-银纳米颗粒的TEM图;B-银溶胶的紫外吸收峰
图3 银纳米颗粒的表征
Fig.3 Characterization of silver nanoparticles
2.5 银纳米颗粒包裹细菌的TEM表征
为研究银纳米颗粒是如何增强拉曼信号的,TEM观察菌体与银溶胶混合样品,结果如图4所示,银纳米颗粒均匀包裹在菌体表面,用以增强拉曼信号。
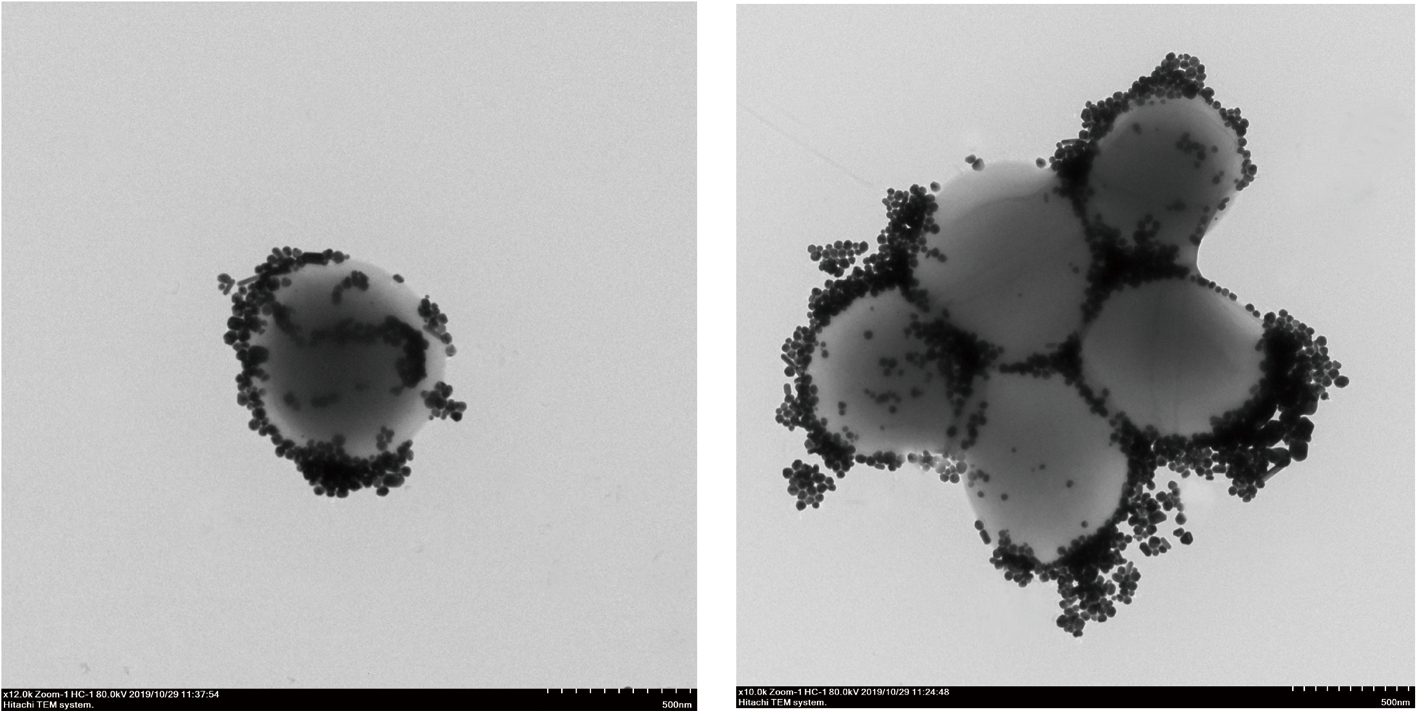
图4 银纳米颗粒包裹细菌的TEM图
Fig.4 TEM image of silver nanoparticle-encapsulated bacteria
2.6 三种菌株SERS谱峰的比较
在400~1 800 cm-1范围内采集拉曼信号,SERS扫描结果如图5所示,比较3种细菌的SERS谱峰,可观察到730和1 330 cm-1左右处的2个强峰为腺嘌呤的典型拉曼特征,此外,大肠杆菌的主要特征峰在626、652、728、956,1 128、1 336、1 461、1 639、1 709 cm-1,沙门氏菌在454、524、643、729、1 009、1 118、1 329、1 395、1 458、1 643 cm-1有明显的拉曼振动谱峰,而金黄色葡萄球菌的拉曼振动谱峰主要在634、732、1 207、1 276、1 340、1 379、1 460、1 547、1 636 cm-1。3者的谱峰特征有很大差异,可以作为区分3种细菌的依据。
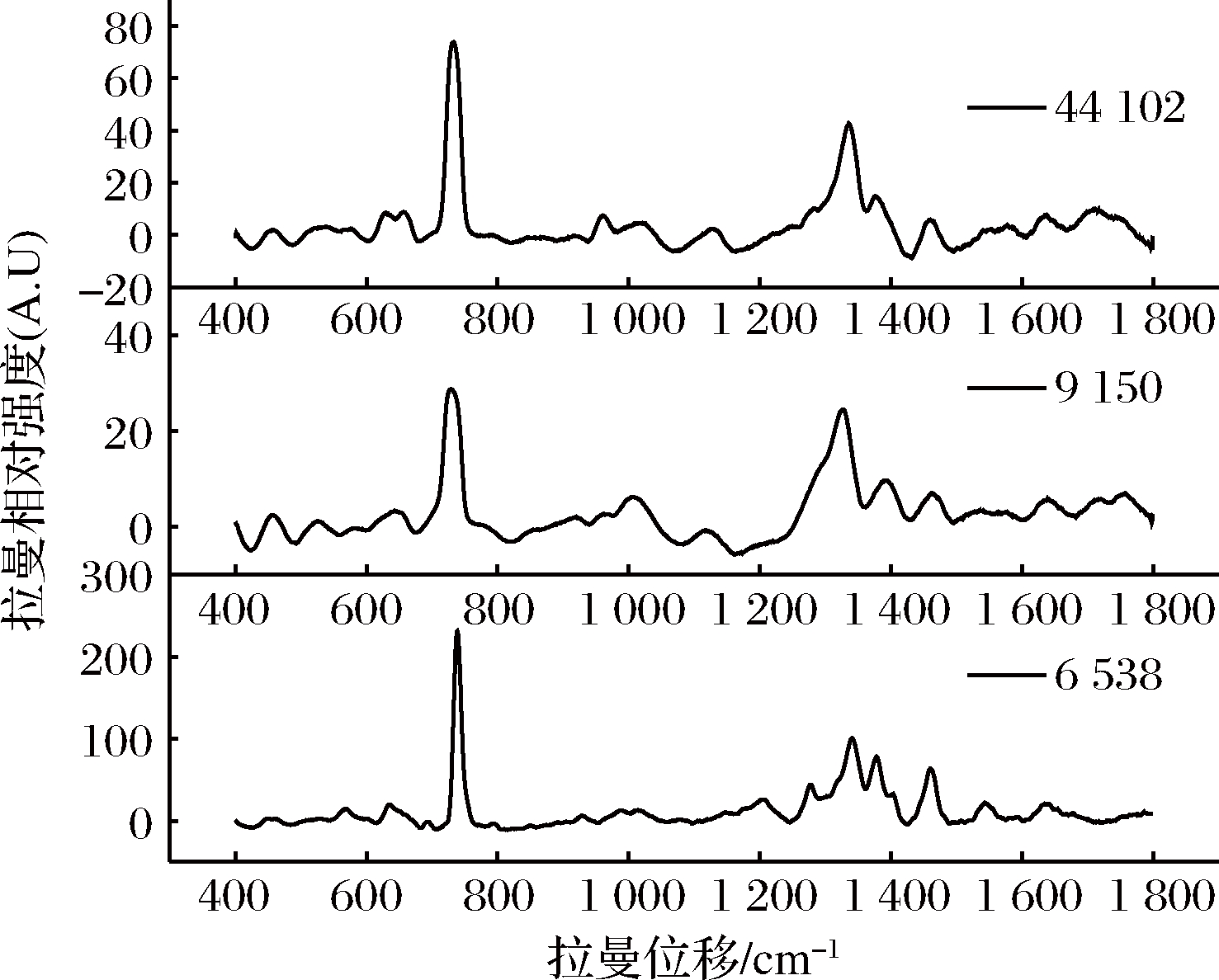
图5 大肠杆菌、沙门氏菌和金黄色葡萄球菌SERS谱峰
Fig.5 SERS peaks of Escherichia coli, Salmonella and Staphylococcus aureus
2.7 PCA模型的建立
3种标准菌株的PCA结果如图6所示,前2个主成分的累积贡献率是93.8%,第一、第二主成分包含了大部分光谱数据所具有的信息,PCA图中可以看出大肠杆菌、沙门氏菌和金黄色葡萄球菌各自聚集在一起,因此,认为3种菌株的谱峰显著不同,可以明显地区分开。
2.8 SERS的灵敏度检测
如图7所示,SERS检测大肠杆菌和金黄色葡萄球菌的最低检出量为104 CFU/mL,沙门氏菌最低检出量为103 CFU/mL。
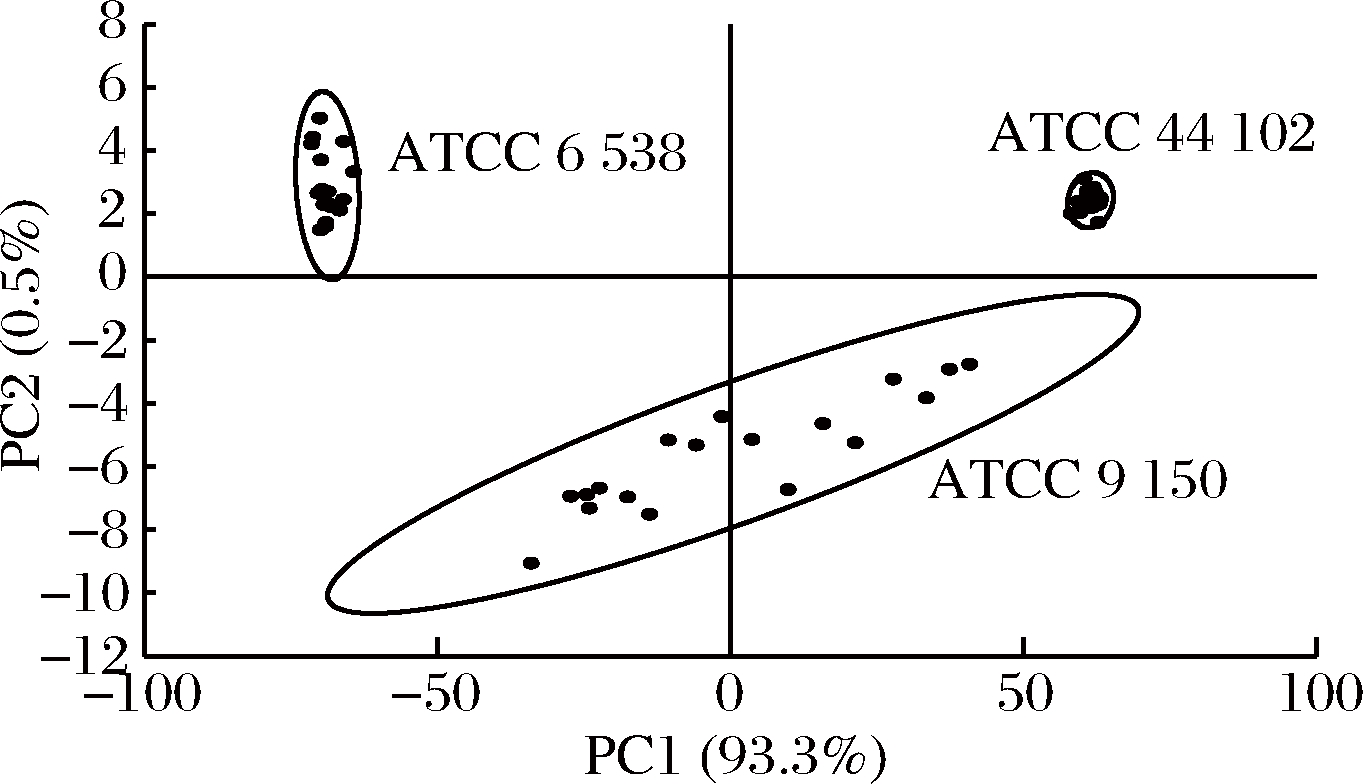
图6 大肠杆菌、沙门氏菌和金黄色葡萄球菌3种标准菌株的PCA分析
Fig.6 PCA analysis of three standard strains of Escherichia coli, Salmonella and Staphylococcus aureus
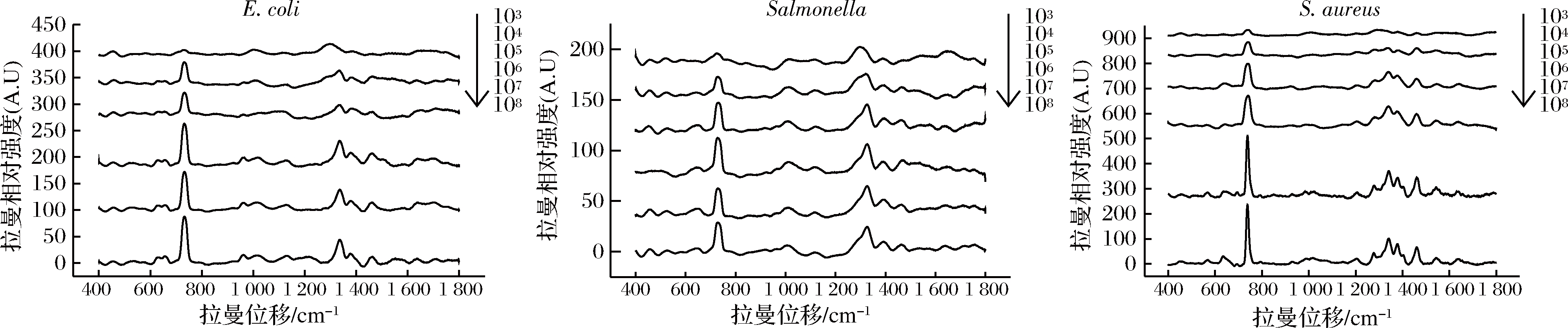
图7 SERS的灵敏度检测
Fig.7 Sensitivity test of SERS
3 结论与讨论
本研究为了同时检测出3种食源性病原菌,建立了基于大肠杆菌、沙门氏菌和金黄色葡萄球菌uidA、hilA和clfA基因的三重PCR检测方法,结果表明,3对引物扩增出的目的片段仅存在于阳性菌株,具有良好的特异性。GONG等[16]检测到金黄色葡萄球菌和沙门氏菌的检测限为100 pg/μL,而本研究的最低检出DNA量为50 pg/μL,说明了本研究的检测效率更高;此外,WEI等[17]在大肠杆菌O157∶H7、金黄色葡萄球菌、沙门氏菌培养液中检出限为103 CFU/mL,在模型食品体系中检出限为104 CFU/mL。本研究建立的多重 PCR 体系检测3种细菌最低检出量为104 CFU/mL,敏感性的差异可能与细菌混合培养的组成或反应体系有关。在实际样品检测中,只有一种细菌存在的情况很少,复杂的检测环境对检测结果有很大的影响,为了进一步接近样品实际检测效果,增加检测的时效性,本研究检测多重PCR敏感性的样品是3个目标细菌混合共培后的菌液。
本研究对银纳米颗粒的表征可知粒径约为40 nm,这与MARWA等[18]的研究结果一致。与以往的研究相比[19],SERS谱峰的分布基本相同,只是在波数上存在一些时相。PCA的前2个主成分的累积贡献率为93.8%,研究认为3种细菌有显著差异。相比SUNDARAM等[20]鉴别食源性病原菌PCA分析的96%,主要区别于在制备银纳米颗粒时未使用聚乙烯醇,而聚乙烯醇主要作用是防止银溶胶发生团聚。杨丹婷[21]以Ag NPs为基底最低检测到105 CFU/mL的大肠杆菌,WANG等[22]检测沙门氏菌的检测限为104 CFU/mL,本研究检测大肠杆菌和金黄色葡萄球菌的最低检出量为104 CFU/mL,沙门氏菌为103 CFU/mL,而在CHONGWEN等[23]的研究中,使用万古霉素修饰的Ag包覆磁性纳米颗粒,检出限低至5×102 CFU/mL,由此可见,特异性基底的制备可以加强SERS检测的灵敏性。
SERS与多重PCR检测相比较,后者需要经过提取模板、PCR扩增、琼脂糖凝胶验证等过程,而SERS只需要进行拉曼扫描和结果比对,检测时间相对节约了3 h。相对于多重PCR而言,SERS方法提高了沙门氏菌的检测限,而且最近开发的各种标签修饰技术,比如特异性抗体修饰[24]、适配体修饰[25]的SERS技术以及万古霉素和适配体的双重识别[26]使得SERS技术检测食源性致病菌的特异性更强,灵敏度更高。随着便携式拉曼光谱仪在食品检测的应用,SERS的现场快速检测成为可能,对食品安全的监测具有重要意义。
[1] WALLACE F M,ANDALORO B,FALLON D,et al.Detection of Salmonella species in a variety of foods by the DuPontTM BAX system Real-Time PCR assay for Salmonella: first action 2013.02[J].Journal of AOAC International,2014,97(3):868-875.
[2] BIRAN D,RON E Z.Extraintestinal pathogenic Escherichia coli[J].Current Topics in Microbiology and Immunology,2018,416:149-161.
[3] ZHAO X,WEI C,ZHONG J,et al.Research advance in rapid detection of foodborne Staphylococcus aureus[J].Biotechnology & Biotechnological Equipment,2016,30(5):1-7.
[4] KUMAGAI Y,GILMOUR S,OTA E,et al.Estimating the burden of foodborne diseases in Japan[J].Bulletin of the World Health Organisation,2015,93(8):540-549.
[5] YENI F, ACAR S, POLAT Ö G, et al.Rapid and standardized methods for detection of foodborne pathogens and mycotoxins on fresh produce[J].Food Control,2014,40(1):359-367.
[6] MIRHOSSEINI S A , FOOLADI A I , AMANI J , et al.Production of recombinant flagellin to develop ELISA-based detection of Salmonella enteritidis[J].Brazilian Journal of Microbiology, 2017,48(4):774-781.
[7] 闫晗,吕国钦,张洋,等.乳品中3种常见细菌多重PCR检测方法的建立[J].食品与发酵工业,2015, 41(7):149-154.
[8] SHIGEMURA K,SHIRAKAWA T,OKADA H,et al.Rapid detection and differentiation of Gram-negative and Gram-positive pathogenic bacteria in urine using TaqMan probe[J].Clinical and Experimental Medicine,2005,4(4):196-201.
[9] CHEN P,LI Y,CUI T,et al.Nanoparticles based sensors for rapid detection of foodborne pathogens[J].International Journal of Agricultural & Biological Engineering,2013,6(1):28-35.
[10] LIN C C, LIN C Y, KAO C J, et al.High efficiency SERS detection of clinical microorganism by AgNPs-decorated filter membrane and pattern recognition techniques[J].Sensors & Actuators B Chemical,2017,241:513-521.
[11] LAN S, PING Z, DAWEI Z, et al.Rapid detection of Escherichia coli and Salmonella typhimurium by surface-enhanced Raman scattering[J].Optoelectronics Letters,2015,11(2):157-160.
[12] WANG J,WU X,WANG C,et al.Facile synthesis of Au-coated magnetic nanoparticles and their application in bacteria detection via a SERS method[J].ACS Applied Materials & Interfaces,2016,8(31):19 958-19 967.
[13] CHO I H, BHANDARI P, PATEL P, et al.Membrane filter-assisted surface enhanced Raman spectroscopy for the rapid detection of E.coli O157:H7 in ground beef[J].Biosensors & Bioelectronics,2015,64:171-176.
[14] 胡玉兰,黄亚伟,王若兰,等.便携式拉曼光谱仪在食品检测中的应用[J].食品工业科技,2017,38(17):319-323.
[15] 段贵娇,张健伟,张志彬,等.表面增强拉曼光谱技术检测食品及农产品中的非法添加剂[J].食品与发酵工业,2019,45(10):272-277.
[16] GONG Q, LI Z,NIU M.A pilot study on PCR-based detection of four foodborne pathogenic microorganisms[J].Food Measure,2018,12(2):675-682.
[17] WEI C,ZHONG J,HU T,et al.Simultaneous detection of Escherichia coli, O157:H7, Staphylococcus aureus, and Salmonella, by multiplex PCR in milk[J].Biotech,2018,8(1):76-82.
[18] MARWA K,MAHMOUD A,REFAAT I H,et al.Antibacterial effect of various shapes of silver nanoparticles monitored by SERS[J].Talanta,2015,138:183-189.
[19] WU X, HAN C, CHEN J, et al.Rapid detection of pathogenic bacteria from fresh produce by filtration and surface-enhanced raman spectroscopy[J].Jom,2016,68(4):1 156-1 162.
[20] SUNDARAM J,PARK B,KWON Y,et al.Surface enhanced Raman scattering(SERS)with biopolymer encapsulated silver nanosubstrates for rapid detection of foodborne pathogens[J].International Journal of Food Microbiology,2013,167(1):67-73.
[21] 杨丹婷.氨基甲酸乙酯和大肠杆菌的表面增强拉曼光谱检测方法[D].杭州:浙江大学,2013.
[22] WANG P, PANG S, CHEN J, et al.Label-free mapping of single bacterial cells using surface-enhanced Raman spectroscopy[J].Analyst,2016,141(4):1 356-1 363.
[23] CHONGWEN W,BING G,QIQI L,et al.Combined use of vancomycin-modified Ag-coated magnetic nanoparticles and secondary enhanced nanoparticles for rapid surface-enhanced Raman scattering detection of bacteria[J].International Journal of Nanomedicine,2018,13:1 159-1 178.
[24] GUVEN B,BASARAN-AKGUL N,TEMUR E,et al.SERS-based sandwich immunoassay using antibody coated magnetic nanoparticles for Escherichia coli enumeration[J].The Analyst,2011,136(4):740-748.
[25] CHEN J,PARK B,HUANG Y W,et al.Label-free SERS detection of Salmonella typhimurium on DNA aptamer modified AgNR substrates[J].Journal of Food Measurement and Characterization,2017,11(4):1-7.
[26] ZHANG C,WANG C,XIAO R,et al.Sensitive and specific detection of clinical bacteria: Via vancomycin-modified Fe3O4@Au nanoparticles and aptamer-functionalized SERS tags[J].Journal of Materials Chemistry B,2018,6(22):3 751-3 761.