氨肽酶(EC 3.4.11)是一种重要的外切蛋白酶,可以高效催化蛋白质N端的肽键,使N端氨基酸游离出来[1]。其中亮氨酸氨肽酶(leucine aminopeptidase)属于氨肽酶家族中重要成员,它可以高效水解蛋白质N端多种疏水性氨基酸例如亮氨酸、精氨酸,赖氨酸等[2],在改善蛋白水解液风味和提高蛋白水解度等食品加工方面有极大的应用价值[1]。目前,来源于Pseudomonas[3], Aspergillus[4], Bacillus licheniformis[1], Streptomyces[5], Lactic acid bacteria[6], Bacillus subtilis[7] 的氨肽酶基因均有报道,其中一些氨肽酶基因已经在E.coli[8], B.subtilis[9]和Pichia pastoris[2]中异源表达。
B. subtilis属于一般安全(generally recognized as safe,GRAS)微生物,为非致病菌[10]。因此B. subtilis常被用作食品加工用酶的宿主菌。目前α-淀粉酶[11]、β-淀粉酶[12]、碱性木聚糖酶[13]、果胶酶[14]、氨肽酶[9]等多种食品加工用酶都已在B. subtilis中高效表达,但是所构建的重组B. subtilis需要添加抗生素以维持质粒稳定性,这为B. subtilis成为食品工业宿主菌带来了巨大阻碍。为解决这一问题,XIA等[15]将目的基因整合到基因组上,阻止目的基因丢失,但这种方法会降低目的基因的拷贝数,减少蛋白产量。YANG等[16]采用EndoA和EndoB的毒性蛋白和解毒蛋白的方法来增加质粒分离稳定性,但是构建方法复杂并且EndoA和EndoB蛋白表达不易控制。与上述方法相比,构建营养缺陷型互补菌株阻止质粒丢失是一种更为有效的方法[17]。
本研究通过Cre/lox系统[18]敲除B. subtilis 168中的dal以及遗留的博来霉系抗性基因(BleR),LAP基因插入质粒pMA5中,并用dal替换质粒中的卡那霉素抗性基因(KanR),继而将pMA5质粒中E. coli的Ori和氨苄霉素抗性基因(AmpR)删除。经过基因敲除的缺陷型菌只能在添加D-丙氨酸的培养基中生长,而在普通培养基中不能生长。质粒中含有的dal可以和缺陷菌形成互补,从而产生筛选压力,阻止质粒丢失现象。为提高所构建缺陷型菌株的氨肽酶发酵产量,在前面研究基础上进一步对该菌株进行发酵过程优化,为工业化生产LAP奠定基础。
1 材料与方法
1.1 材料与试剂
底物L-亮氨酸-4-硝基苯胺,Alfa Aesar公司;蛋白胨、酵母粉,OXID;博来霉素,北京索莱宝公司;基因组提取、质粒提取、胶回收、产物纯化试剂盒,上海生工生物工程公司。核酸内切酶、T4连接酶、磷酸化酶以及ligation solutionⅠ、DNA marker,大连Takara公司;高斯组装试剂盒,南京诺唯赞公司;其他未注明试剂均为分析纯,国药集团化学试剂有限公司。
1.2 仪器与设备
核酸电泳仪和PCR仪,Bio-rad 公司;凝胶成像仪,北京赛智公司;5 L发酵罐,上海迪必尔生物工程有限公司;UH5300双光束紫外分光光度计、高速低温离心机,日本日立HITACH集团。
1.3 实验方法
1.3.1 菌种、质粒、以及培养条件
本研究所用的菌种和质粒,实验室保藏及本研究构建。
种子液在LB培养基(蛋白胨10 g/L,酵母粉5 g/L,NaCl 10 g/L)中37 ℃,220 r/min培养。LB培养基中额外添加氨苄霉素(Amp)100 μg/mL,卡那霉素(Kan)100 μg/mL,博来霉素(Ble)30 μg/mL和200 μg/mL 的D-丙氨酸。工程菌在Super Rich培养基(蛋白胨25 g/L,酵母提取物20 g/L,K2HPO4·3H2O 3.9 g/L培养基中发酵,葡萄糖30 g/L)中pH 7.0、37 ℃、220 r/min中发酵33 h。
1.3.2 引物及PCR扩增
本研究所有引物由SnapGene软件设计,上海生工生物工程公司合成。
1.3.3 基因操作方法及试剂
基因组提取,质粒提取,胶回收,产物纯化,高斯组装均按照试剂盒说明书操作。1%琼脂糖凝胶电泳验证PCR产物,Spizizen法[19]转化B. subtilis感受态。
1.3.4 宿主菌构建
通过Cre/lox系统敲除B. subtilis 168基因组中dal (GenBank no. CAB12271.1)。首先以质粒p7Z6为模板扩增lox71-BleR-lox66,B. subtilis 168基因组为模板分别扩增dal上下游同源臂,以lox71-BleR-lox66和上下游同源臂为模板,通过融合PCR获得敲除片段,即上游同源+lox71-BleR-lox66+下游同源臂。其次直接将纯化好的敲除片段转化进B. subtilis感受态中,在含有Ble和D-丙氨酸的LB平板37 ℃过夜培养,PCR验证正确的即为缺陷型菌株BS168 (dal-, BleR)。最后将温度敏感型质粒pDG148转化至BS168 (dal-, BleR)感受态中,在含有Kan和D-丙氨酸的LB平板上过夜培养,挑取转化子至仅添加D-丙氨酸的LB平板上51 ℃培养12 h除去质粒pDG148,得到无抗性基因且不含pDG148质粒的敲除菌BS168 (dal)-。
1.3.5 质粒的构建
首先以pET-28a-lap为模板扩增LAP基因,通过Ned Ⅰ和BamH Ⅰ双酶切,T4酶连接插入至pMA5质粒中得到质粒pMA5-lap。其次以pMA5-lap为模板,通过高斯组装[20]的方法将质粒中KanR替换为dal构建缺陷互补型表达载体pMA5-lap-dal。最后,为保证食品级表达载体基因来源的安全性,本研究采用定点突变试剂盒的方法删除pMA5中原有的AmpR和E. coli的Ori。具体方法如下:以pMA5-lap-dal质粒为模板,通过反向PCR的方法得到不含AmpR和Ori (E.coli )的线性质粒 pMA5-lap-dal (AmpR, Ori)-;线性质粒骨架依次通过磷酸化处理,ligation solution环化,Dpn Ⅰ消化;将环化质粒转化至缺陷型菌株BS168 (dal)-感受态中,得到食品级工程菌BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-。
1.3.6 重组质粒的分离稳定性
参照HUANG等[21]方法,考察食品级工程菌BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-质粒分离稳定性。具体操作如下:挑取食品级工程菌于LB培养基中培养12 h作为种子液;将种子液分别转接至含有D-丙氨酸的LB和普通LB培养基中培养24 h,吸取菌液稀释105后分别涂布于含有D-丙氨酸的LB平板中过夜培养;从中分别挑取100个菌落转接至含有D-丙氨酸和普通LB平板中,统计两平板菌落数作为传代20代的质粒稳定性。以此类推,传代至第100代。用CC/CD来表示质粒分离稳定性。其中CC代表重组菌在普通LB培养基中的菌落数,CD代表重组菌在LB+D-丙氨酸培养基中生长的菌落数。
1.3.7 酶活测定方法
按照WANG等[22]的方法测定亮氨酸氨肽酶酶活,如公式(1)所示:
(1)
式中:X,样品的酶活,U/mL;N,于405 nm处测得吸光度值;Y,酶液稀释的倍数。
氨肽酶酶活定义为在以上分析测定的基础上,定义一个酶活单位(U)为在特定条件下(50 ℃,pH 8.5),每分钟催化分解L-亮氨酸-硝基苯胺产生1 μmol的对硝基苯胺所需要的酶量。
1.3.8 5 L发酵罐优化
探究不同转速、温度、pH对发酵的产酶的影响,5 L发酵罐初始转速设置为200、300、400、500 r/min,温度设置为30、33、36、39 ℃,pH设置为7.0、7.5和不控制。此外使用摇床优化培养基(蛋白胨15 g/L,酵母提取物30 g/L,葡萄糖25 g/L,K2HPO4·3H2O 3.9 g/L,L-谷氨酸钠5 g/L)发酵。初始pH为7.0,发酵过程中使用30%的H3PO4和适当浓度的氨水控制pH,使用20%有机硅消除泡沫。
1.3.9 数据处理
采用Origin 8.5软件作图,实验数据以平均值±标准差(Mean±SD)表示。
2 结果与分析
2.1 缺陷型宿主菌的构建
在B. subtilis生长代谢中,D-丙氨酸是参与其细胞壁合成的重要物质。然而在普通培养基中,丙氨酸多为L型,需要D-丙氨酸消旋酶将L-丙氨酸酶催化为D-丙氨酸才能保证菌株的正常生长[23]。如若删除B. subtilis基因组中的dal,则B. subtilis会因为缺失D-丙氨酸而无法生长。
在敲除过程中,由于敲除片段与B. subtilis 168基因组dal区域同源性极高,会和dal区域发生双交叉互换。而在含有Ble和D-丙氨酸的平板中,只有发生双交叉互换的菌株才可以正常生长。为保证食品级表达宿主菌基因组不含有抗性基因,需要质粒pDG148进一步删除BS168 (dal-, BleR)基因组中遗留的BleR。pDG148质粒含有Cre重组酶编码基因cre,Cre重组酶可以特异性识别loxP位点并催化lox71和lox66位点使之发生特异性重组[18],从而删除位于lox71和lox66之间的BleR。此外,虽然B.subtilis WB600和B.subtilis WB800删除了部分的蛋白酶基因,更有利于蛋白质的异源表达。但是它们在构建过程引入了其他抗性基因,不符合GRAS菌的要求,因此B. subtilis 168更适合做食品级用酶的宿主菌。
2.2 质粒的构建
质粒图谱如图1所示,首先构建质粒pMA5-lap-dal,质粒含有D-丙氨酸消旋酶的完整基因及其启动子,从而保证含有质粒pMA5-lap-dal的缺陷菌BS168 (dal)-可以在普通培养基中正常生长。为保证基因来源的安全性,在质粒pMA5-lap-dal的基础上删除E.coli的Ori和AmpR,进一步构建食品级质粒pMA5-lap-dal (AmpR, Ori)-。
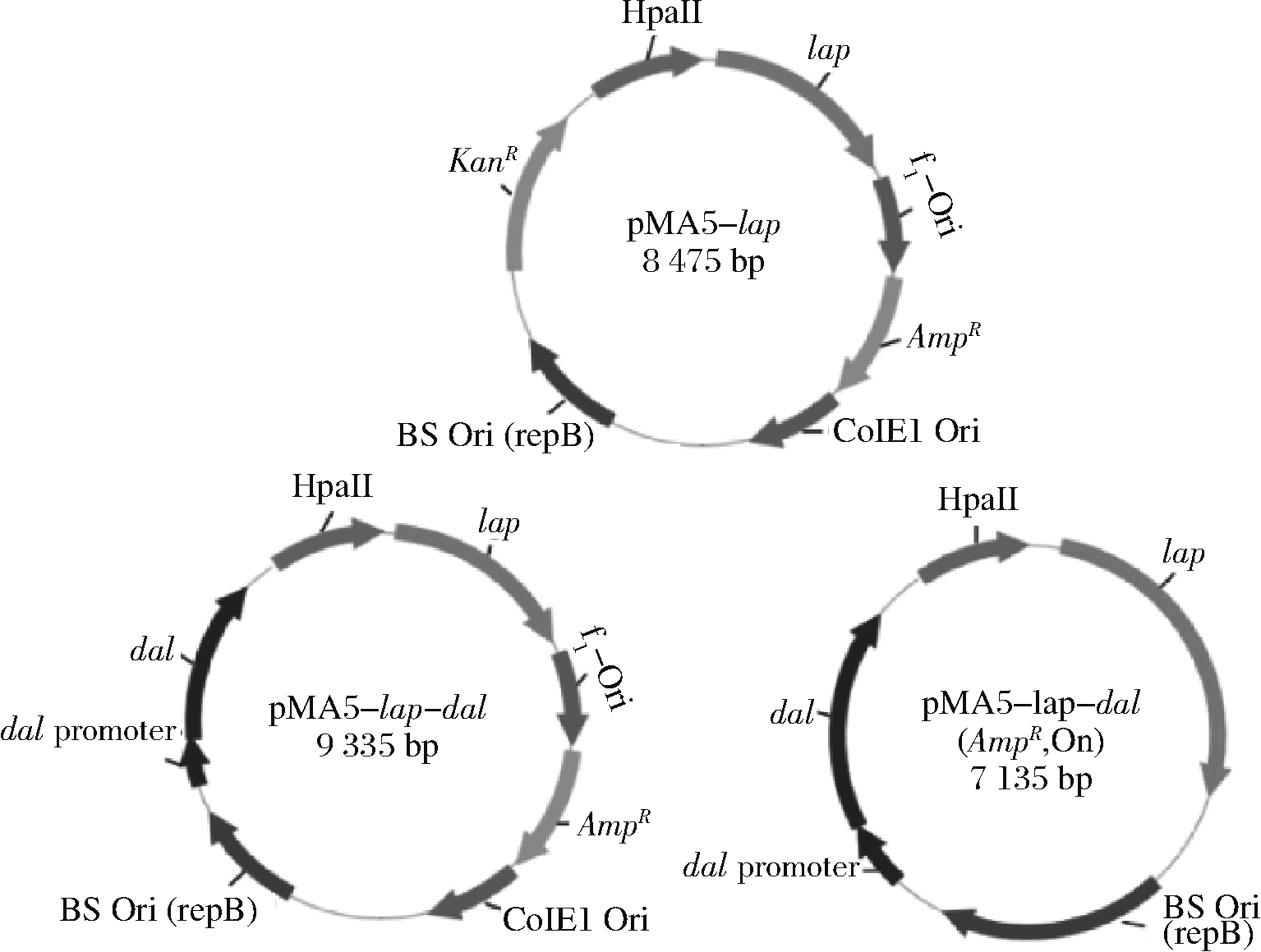
图1 质粒pMA5-lap、pMA5-lap-dal、pMA5-lap-dal (AmpR, Ori)-图谱
Fig.1 Construction of the plasmid pMA5-lap, pMA5-lap-dal, pMA5-lap-dal (AmpR, Ori)-
与传统的抗生素依赖性工程菌相比,食品级工程菌是靠自身对D-丙氨酸的营养缺陷形成筛选压力的。自身的dal代替了原来质粒上的抗性基因,在发酵过程中不需要额外添加抗生素维持质粒稳定性。在源头上杜绝了抗生素的添加,省略了产物提取过程中去除抗生素这一步骤,即节省了发酵生成本,也提高了产物安全性。此外,缺陷型菌株BS168 (dal)-和质粒pMA5-lap-dal (AmpR, Ori)-的构建原理和方法也可以为其他食品用酶表达系统构建提供新思路。
2.3 重组菌的发酵分析
将BS168/pMA5-lap、BS168 (dal)-/pMA5-lap-dal、BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-3株重组菌在Super Rich培养基中发酵33 h。如图2所示,在发酵时间达到30 h时,3株菌OD600均达到最大。其中BS168 (dal)-/pMA5-lap-dal和BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-的OD600曲线相似,最大值分别为9.23和9.58,稍高于BS168/pMA5-lap的最大值。比较3株菌产酶情况可以发现,菌株BS168 (dal)-/pMA5-lap-dal、BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-产酶曲线相似,酶活最高达到60 U/mL,是BS168/pMA5-lap最高酶活的2倍。以上结果表明,BS168 (dal)-/pMA5-lap-dal、BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-在生长、产酶水平方面优于BS168/pMA5-lap。
这可能是由于BS168/pMA5-lap的筛选压力是由卡那霉素提供的,卡那霉素是通过结合核糖体抑制蛋白质合成。虽然KanR在质粒中表达的氨基糖苷磷酸转移酶可以水解卡那霉素以阻止这种结合[24],但会在一定程度上影响细胞生长。对于BS168 (dal)-/pMA5-lap-dal、BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-,筛选压力是基于菌株自身dal的缺失造成的,筛选标记基因来源于宿主菌自身,菌株的生长负荷小,更有利于食品用酶的表达[16]。最后,为保证重组菌达到GRAS菌株标准,本研究选取BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-做为最终产LAP的工程菌。

a-LAP酶活;b-OD600
图2 菌株BS168/pMA5-lap, BS168 (dal)-/pMA5-lap-dal, BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-发酵曲线
Fig.2 The fermentation curve of BS168 /pMA5-lap,BS168 (dal)-/pMA5-lap-dal, BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-
2.4 重组质粒分离稳定性
如果质粒分离稳定性较差,会发生严重的质粒丢失现象,导致发酵后期质粒丢失的菌株成为优势菌种,从而严重影响目的蛋白表达量[21]。由表1可知,BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-在LB+D-丙氨酸培养基中,第60代以前质粒基本可以保证正常的传代,然而到80代时质粒出现严重的丢失现象,传代至100代,质粒保持率仅为57%。而在LB培养基中培养,传代100代,质粒仍保持良好。这可能是由于普通LB培养基中对BS168 (dal)-存在着强有力的筛选压力,质粒一旦丢失,则菌株无法正常生长。而在LB+D-丙氨酸的培养基中不存在筛选压力,无法维持质粒长期分离稳定性。
2.5 重组菌5 L发酵罐不同条件对产酶及生长影响
不同的发酵条件对菌株的生长代谢和产酶水平往往会有重要影响[25-26],在摇床优化发酵培养基及培养条件的基础上进行5 L发酵罐优化。
考察不同转速对BS168(dal)-/pMA5-lap-dal (AmpR, Ori)-产LAP的影响,转速分别设置为200、300、 400和500 r/min,由图3可知,随着转速的增加,菌体OD600和LAP酶活都呈现出先增加后减小的趋势。转速为300 r/min时,OD600和酶活均处于较高水平。
表1 质粒pMA5-lap-dal (AmpR, Ori)-分离稳定性
Table 1 Separationstabilities of the plasmid pMA5-lap-dal (AmpR, Ori)-

传代次数/代质粒分离稳定性(Cc/CD)LBLB+D-丙氨酸20100±0.00100±0.0040100±0.0100±0.006099±0.595±1.228099±0.580±2.39100100±0.0057±3.45
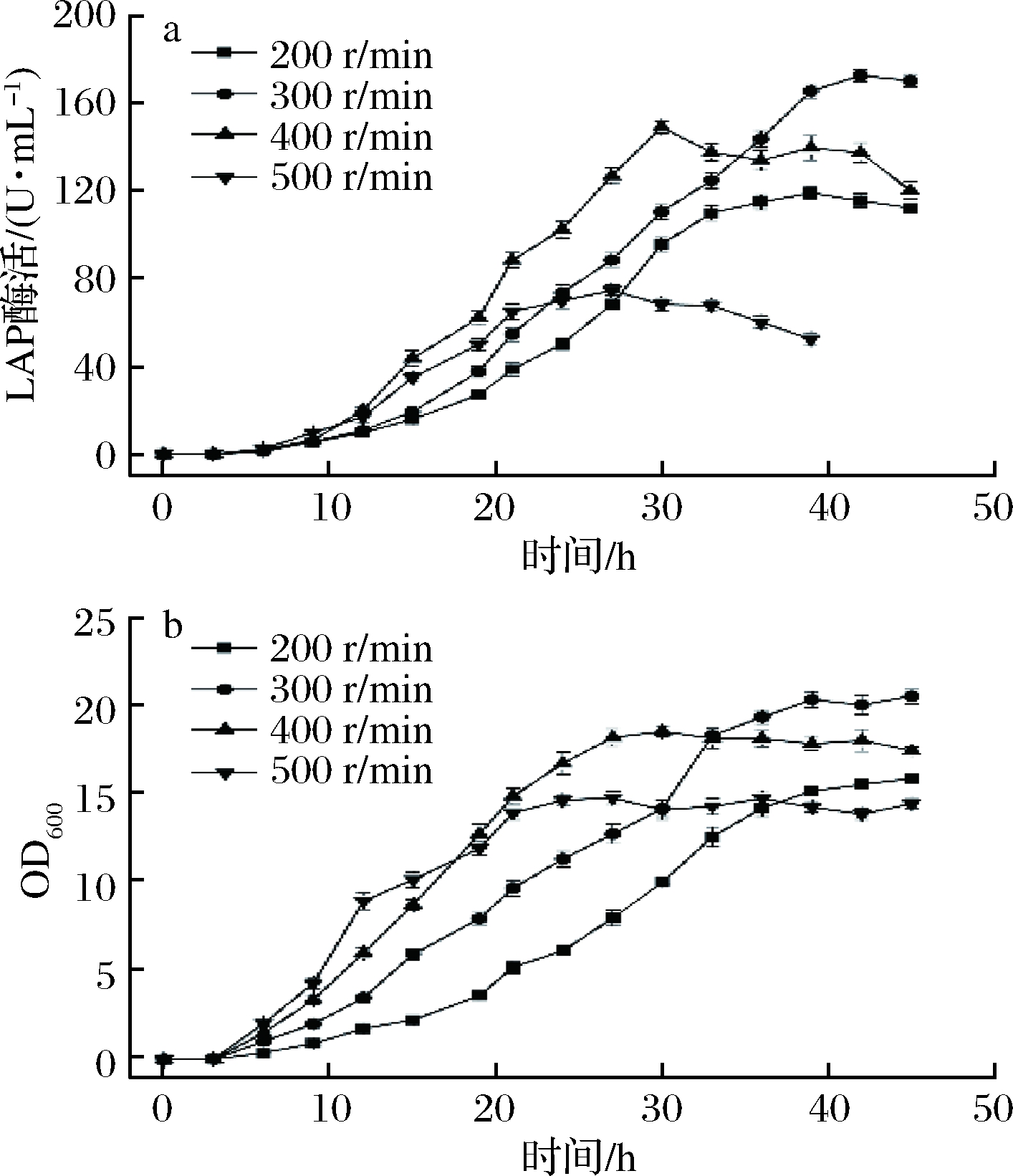
a-LAP酶活;b-OD600
图3 不同转速对BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-发酵的影响
Fig.3 The effects of different stirring speeds on the fermentation of BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-
考察不同温度对BS168(dal)-/pMA5-lap-dal (AmpR, Ori)-产LAP的影响,温度分别设置为30、 33、 36和39 ℃。如图4所示,随着温度的升高,菌株的产酶周期缩短,最高酶活力减少。在温度为33 ℃时酶活达最高为206 U/mL。
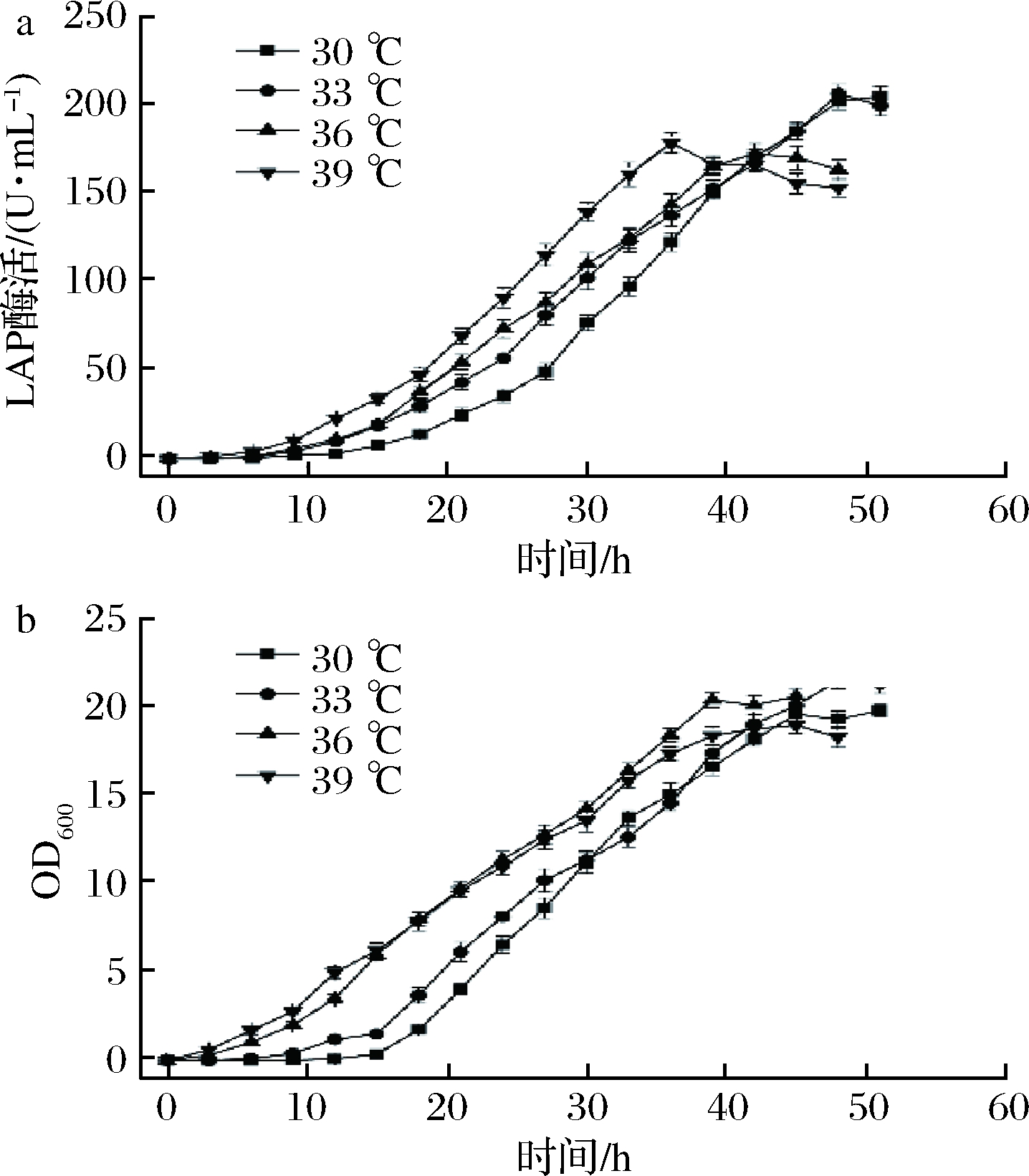
a-LAP酶活;b-OD600
图4 不同温度对BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-发酵的影响
Fig.4 The effects of different temperature on the fermentation of BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-
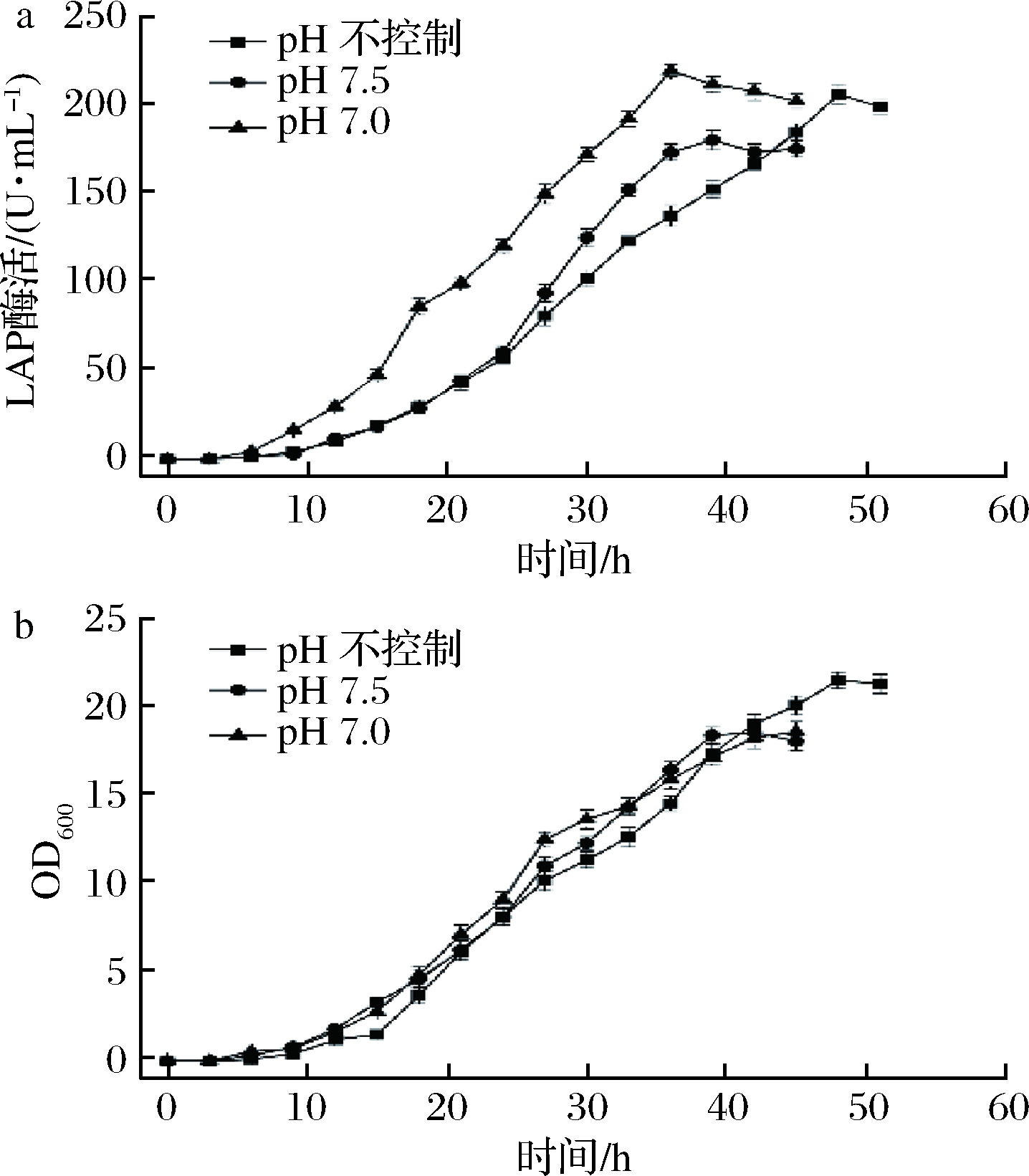
a-LAP酶活;b-OD600
图5 不同pH对BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-发酵的影响
Fig.5 The effects of different pH on the fermentition of BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-
pH可以影响物质分子状态和细胞原生质体所带的电荷,继而使得细胞的代谢通路和细胞膜的通透性发生改变,从而影响发酵产率[27]。考察不同pH对食品级重组菌产LAP的影响,pH分别设置为7.0、7.5、不控制。由图5可知,当pH为7.0时,重组菌在36 h酶活达到最大为220 U/mL;在pH为7.0和pH为7.5的条件下,菌体OD600较为接近,但低于pH不控制条件下的OD600。为使菌体更利于产酶,因此选用恒定pH为7.0的条件进行后续发酵。
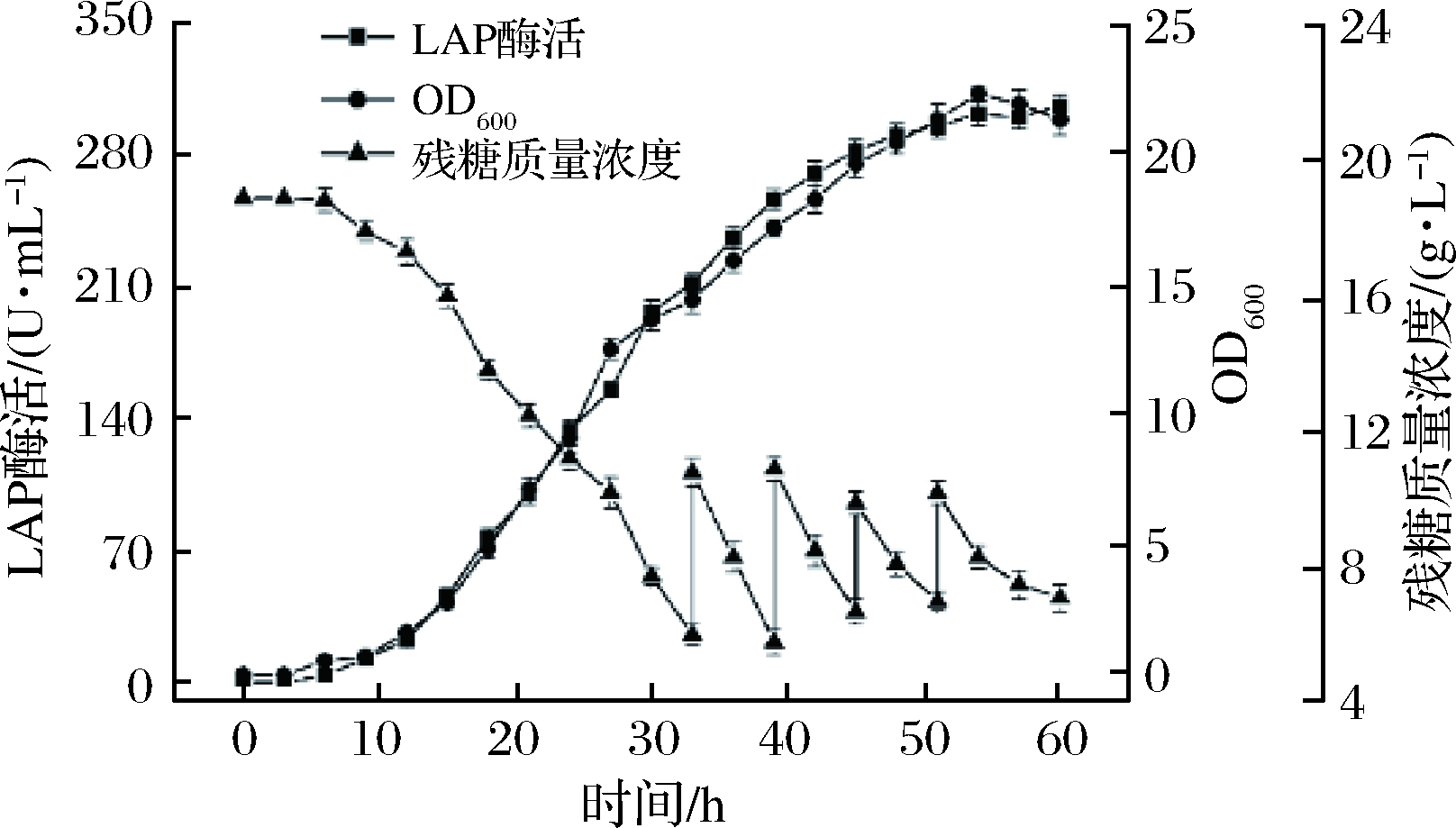
图6 分批补料条件下,BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-发酵过程曲线
Fig.6 Time courses of LAP production by the food-grade recombinant B. subtilis under fed-batch conditions
发酵后期葡萄糖消耗大,残糖浓度低,会影响后期菌体的生长和产酶。合理的补料可以进一步提高产酶[28]。在转速,温度,pH 三条件优化的基础上,采用分批流加补料,结果如图6 所示。在发酵33~61 h,每隔6 h向发酵罐中补充葡萄糖,补料4次。重组菌在发酵至54 h时LAP酶活和OD600 均达到最大。发酵周期虽然延长至54 h,但最高酶活达302 U/mL,相较于未补料时最高酶活220 U/mL,酶活提高37%。
3 结论
本研究基于D-丙氨酸消旋酶缺陷互补,构建了1株不含抗性基因的高产亮氨酸氨肽酶食品级工程菌,解决了B. subtilis在发酵过程中需要添加抗生素这一问题。在本研究中,发现食品级工程菌BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-相较于抗生素依赖型工程菌,更适合LAP的表达。对BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-进行5 L发酵罐优化,在转速300 r/min, 温度33 ℃,pH 7.0,31~61 h、4次分批补料条件下,酶活最高达到302 U/mL,高于目前汪晓东等[29]报道的138 U/mL,XI等[2]报道的28.5 U/mL,孔峰等[30]报道的232.5 U/mL。结果表明,食品级重组菌BS168 (dal)-/pMA5-lap-dal (AmpR, Ori)-具有较好的工业应用前景,且该研究也为其他食品加工用酶的菌株构建提供了一定的借鉴和理论指导。
[1] LEI F,ZHAO Q,SUN W,et al.Characterization of a salt-tolerant aminopeptidase from marine Bacillus licheniformis SWJS33 that improves hydrolysis and debittering efficiency for soy protein isolate[J].Food Chemistry,2017,214:347-353.
[2] XI H,TIAN Y,ZHOU N.Characterization of an N-glycosylated Bacillus subtilis leucine aminopeptidase expressed in Pichia pastoris[J].Journal of Basic Microbiology,2015, 55(2):236-246.
[3] WU Y,ZHOU N,ZHOU Z.A thermo-stable lysine aminopeptidase from Pseudomonas aeruginosa: Isolation, purification, characterization, and sequence analysis[J].Journal Basic Microbiol,2014,54(10):1 110-1 119.
[4] MATSUSHITA M,FURUKAWA I,SUZUKI S,et al.Characterization of recombinant prolyl aminopeptidase from Aspergillus oryzae[J].Journal of Basic Microbiology,2011,109(1):156-165.
[5] WU B,SHI P,LI J,et al.A new aminopeptidase from the keratin-degrading strain Streptomyces fradiae var. k11[J].Appl Biochem Biotechnol,2010,160(3):730-739.
[6] BOZHIDAR T,MARGARITA M,LYDIA G.Debittering of protein hydrolysates by Lactobacillus LBL-4 aminopeptidase[J].Enzyme Res,2011,2011: 538 676-538 683.
[7] 须瑛敏.枯草芽孢杆菌产氨肽酶的研究[D].无锡:江南大学,2005.
[8] HIROYUKI S,KATSUYA D,HIDEKI Y,et al.Efficient production of active Vibrio proteolyticus aminopeptidase in Escherichia coli by co-expression with engineered vibriolysin[J].Applied Microbiology and Biotechnology,2009,84(1):191-198.
[9] GAO X,CUI W,TIAN Y,et al.Over-expression, secretion, biochemical characterisation, and structure analysis of Bacillus subtilis aminopeptidase[J].Journal of the Science of Food and Agriculture,2013,93(11):2 810-2 815.
[10] YANG M,ZHANG W,JI S,et al.Generation of an artificial double promoter for protein expression in Bacillus subtilis through a promoter trap system[J].PloS One,2013,8(2):e56 321.
[11] FU G,LIU J,LI J,et al.Systematic screening of optimal signal peptides for secretory production of heterologous proteins in Bacillus subtilis[J].Journal of Agricultural and Food Chemistry,2018,66(50):13 141-13 151.
[12] 汪薛良,钮成拓,包敏,等.大麦β-淀粉酶在枯草芽孢杆菌中异源表达[J].食品与发酵工业,2019,45(14):34-40.
[13] ZHANG W,YANG M,YANG Y,et al.Optimal secretion of alkali-tolerant xylanase in Bacillus subtilis by signal peptide screening[J].Applied Microbiology and Biotechnology,2016,100(20):8 745-8 756.
[14] LIU Y,CHEN G,WANG J, et al. Efficient expression of an alkaline pectate lyase gene from Bacillus subtilis and the characterization of the recombinant protein[J].Biotechnology Letters,2012,34(1):109-115.
[15] XIA Y,CHEN W,FU X,et al.Construction of an integrative food-grade expression system for Bacillus subtilis[J].Food Research International,2005,38(3):251-256.
[16] YANG S,KANG Z,CAO W,et al.Construction of a novel, stable, food-grade expression system by engineering the endogenous toxin-antitoxin system in Bacillus subtilis[J].Journal of Biotechnology,2016,219:40-47.
[17] HE W,MU W,JIANG B,et al.Construction of a food grade recombinant Bacillus subtilis based on replicative plasmids with an auxotrophic marker for biotransformation of D-fructose to D-allulose[J].Journal of Agricultural and Food Chemistry,2016, 64(16):3 243-3 250.
[18] YAN X,YU H,HONG Q,et al.Cre/lox system and PCR-based genome engineering in Bacillus subtilis[J].Applied and Environmental Microbiology,2008,74(17):5 556-5 562.
[19] ANAGNOSTOPOULOS C,SPIZIZEN J.Requirements for transformation in Bacillus subtilis[J].Journal of Bacteriology,1961, 81(5):741-746.
[20] GIBSON G D,YOUNG L,CHUANG R,et al.Enzymatic assembly of DNA molecules up to several hundred kilobases[J].Nature Methods,2009,6(5):343-347.
[21] HUANG K,GUAN X,JIANG B,et al.Construction of a food-grade arginase expression system and its application in L-ornithine production with whole cell biocatalyst[J].Process Biochemistry,2008,73:94-101.
[22] WANG K,WANG K,ZHOU N,et al.Secretory expression, purification, characterization, and application of an Aspergillus oryzae, prolyl aminopeptidase in Bacillus subtilis[J].Applied Biochemistry & Biotechnology,2016,181:1 611-1 623.
[23] 何伟伟.D-阿洛酮糖3-差向异构酶在枯草芽孢杆菌中的高效表达及应用研究[D].无锡:江南大学,2017.
[24] CARRER H,HOCKENBERRY T N,SVAB Z,et al. Kanamycin resistance as a selectable marker for plastid transformation in tobacco[J].Molecular & General Genetics Mgg,1993,241:1-2.
[25] ZHUANG Z,JIANG C,ZHANG F,et al.Streptomycin-induced ribosome engineering complemented with fermentation optimization for enhanced production of 10-membered enediynes tiancimycin-A and tiancimycin-D[J].Biotechnology and Bioengineering, 2019,116(16):1 304-1 314.
[26] WANG K,TIAN Y,ZHOU N,et al.Studies on fermentation optimization, stability and application of prolyl aminopeptidase from Bacillus subtilis[J].Process Biochemistry,2018,74:10-20.
[27] 王卫,吴耀辉,黎继烈,等.调控pH提高分批发酵赤霉素GA3的产量[J].菌物学报,2019,38(7):1 185-1 190.
[28] YADHU N G,PRAVEEN V V.2,3-Butanediol production using Klebsiella oxytoca ATCC 8724: Evaluation of biomass derived sugars and fed-batch fermentation process[J].Process Biochemistry,2017,28:25-34.
[29] 汪晓东,田亚平.枯草芽孢杆菌ZH-Zj016产亮氨酸氨肽酶的发酵控制研究[J].食品工业科技,2013,34(8):192-195;200.
[30] 孔峰,田亚平.重组枯草芽孢杆菌高产氨肽酶策略与提取工艺优化[J].食品与生物技术学报,2015,34(8):864-872.