目前肉类掺假问题频繁发生,使得肉类食品的安全状况令人担忧[1]。羊肉作为我国传统肉食品,富含优质蛋白质、肉质鲜美,需求量逐年增加。巨大的羊肉消费市场,高昂的价格给羊肉掺假带来了丰厚利润空间。一些不法商家在利益驱使下用低价肉(如马肉、猪肉、鸭肉等)掺入冒充羊肉,这不仅损害了消费者的切身利益,更直接影响消费者的健康甚至会涉及宗教信仰等问题(如在羊肉中掺入猪肉)[2]。因此,为防止羊肉销售市场上造假行为发生,对羊肉的真伪鉴定具有重要意义。
动物品种的鉴定主要是从蛋白质(如等电聚焦电泳、酶联免疫吸附试验等)和核酸(DNA分子杂交、聚合酶链式反应等)两个方面进行。蛋白质鉴定技术在生鲜肉类的品种鉴别方面应用较为成功,但当肉类经过烹调加工后会使肉蛋白质变性,导致蛋白质鉴定技术的可靠性较差[3]。而核酸具有比蛋白质更高的热稳定性,基因可以从组织中提取,以物种间基因差异为基础的分子学鉴定方法成为研究热点[4]。DNA分子杂交法程序复杂,成本昂贵,而且对于近缘物种并不能很好分辨。EBBEHOJ等[5]通过添加未标记杂交物种的DNA,可以减少近缘物种探针和DNA序列之间的杂交,但对密切相关物种如绵羊和山羊之间的分化检测限仅约为10%,应用相对有限。从TARTAGLIA等[6]首先报道了将聚合酶链式反应(polymerase chain reaction,PCR)技术用于检测饲料中的牛源性成分至今,PCR技术经过不断发展与完善,已从只能单一扩增检测,发展到包括实时荧光定量PCR(real-time fluorescence quantitative PCR,rPCR)、多重PCR、微滴式数字PCR(droplet digital PCR,ddPCR)等多种PCR技术的检测分析,其中rPCR具有较高灵敏性和特异性,已成为肉制品种属鉴别的常用方法[7]。目前现有国家、行业标准颁布的动物源性成分检测方法,均是基于PCR扩增的定性筛选,即只能判定样品中是否含有某种动物源DNA成分,结果以阴性或阳性表示,但无法进行精确定量,这也给政府监管部门的打假执法带来一定困难[8]。rPCR在羊肉及其制品中动物源性成分的掺假检测已有一定研究,主要是针对特定的掺假动物成分,如检测羊肉中猪肉、鸡肉、鸭肉、鼠肉等的含量[9]。但对于可能混杂多种其他肉类或尚不明确掺入何种涉假肉类情况下,如果逐个筛查每一种掺假动物性成分,不仅耗时费力,还给rPCR定量检测带来困难。因此,建立对羊肉含量直接定量检测方法,以满足市场监管需求具有广阔前景。
根据NOH等[10]和KÖPPEL等[11]最近研究报道,肉质量与基因拷贝数之间存在着密切的线性关系。而近几年发展起来的dd PCR方法,是结合微流控技术建立在PCR基础上的新兴核酸检测技术,不需要建立标准曲线及标准样本,能够直接测定出检测体系中目的基因的拷贝数[12]。因此,为实现对羊肉的精准定量,本研究尝试将rPCR与ddPCR相结合,利用ddPCR建立拷贝数和羊肉质量的函数关系,建立基于rPCR检测肉中羊肉含量的方法,以便为掺假肉类的监督管理提供技术参考。
1 材料与方法
1.1 材料与设备
1.1.1 实验材料与试剂
实验样品:山羊肉、绵羊肉、鸭肉、水牛肉、黄牛肉、牦牛肉、猪肉、狐狸肉、貂肉、鹿肉、兔肉、鸡肉、鸽肉、火鸡肉、鹅肉、鳕鱼、虹鳟鱼、银鳕鱼、大黄鱼、大菱鲆等,均由成都海关技术中心提供。
引物探针(羊源性引物探针),由成都擎科梓熙生物技术有限公司合成;质粒(pUC57-GH-羊),由北京六合华大基因科技有限公司合成;Droplet Generator DG8 CartndgeDroplet Gene rator,BIO-RAD公司;DG8 GasketPierceable Foil Heat Seal,BIO-RAD公司;Premix Ex Taq(Probe qPCR)预混液,TaKaRa公司;蛋白酶K(20 mg/mL),拓样生物有限公司;质粒小提试剂盒Plasmid Mini Kit,QIAGEN生物科技有限公司;ddPCR Supermix for Probes (no Dutp)、ddPCR Droplet Generation Oil、ddPCR Droplet Reader Oil,BIO-RAD有限公司;
1.1.2 仪器与设备
FastPrep型样品研磨器,印度MP有限公司;X3R型高速冷冻离心机,美国Thermo Fisher Scientific公司;FastPrep-24 5G型匀浆仪,美国MP Biomedicals生物医学公司;ND-1000 NanoDrop型核酸蛋白测定仪、CFX96 Real-time PCR 仪、QX100 Droplet Digital PCR系统(微滴生成仪PX1PCR板热封仪微滴分析仪),美国BIO-RAD公司。
1.2 实验方法
1.2.1 样品DNA的制备
将新鲜羊肉的肌肉组织绞碎后于100 ℃烘干至恒重,再向研钵中加入液氮,将烘干样品研磨至粉状。称取处理后的羊肉粉100 mg,采用实验室自制的动物组织基因组磁珠法提取试剂盒进行DNA提取,所得基因组DNA质量通过琼脂糖凝胶电泳和Nanodrop ND-2000核酸蛋白定量仪评价[13]。
1.2.2 羊源性引物探针筛选设计与条件优化
1.2.2.1 引物探针选择与设计
通过查询NCBI数据库,选定羊的单拷贝生长激素基因GH(序列号为NC-019468)设计引物探针,设计的引物探针由成都擎科梓熙生物技术有限公司进行合成。
1.2.2.2 特异性验证
为了对所设计引物进行特异性验证,采用磁珠法分别提取1.1.1中所述的山羊肉、绵羊肉等20种动物肌肉DNA。同时设置用ddH2O代替核酸的阴性对照(NTC)。若阴性对照结果为阴性,整个试验有效,可进一步判定结果。实验重复3次,每次2个平行。
1.2.2.3 rPCR反应条件的优化
采用TaqMan PCR Master Mix(Bio-Rad)试剂推荐的25 μL体系,对模板DNA(约50 ng/μL)进行rPCR扩增。反应体系为:Dream Taq PCR Master Mix 12.5 μL,上、下游引物各1 μL,DNA模板2 μL,用ddH2O补足25 μL。扩增条件确定为:95 ℃ 5 min;95 ℃ 15 s,60 ℃ 30 s,45个循环。通过比较rPCR的Ct值和扩增曲线等来确定优化结果。每个样品每个处理做2个平行。
退火温度优化:设置退火温度为58、59、60、61、62、63、64和65 ℃共8个梯度,通过比较分析rPCR的Ct值和扩增曲线等来确定羊源性特异性引物和探针的最佳退火温度。每个样品每个处理做2个平行。
引物浓度优化:设置rPCR体系中上下游引物浓度分别为0.1、0.2、0.4、0.6、0.8和1 pmol/μL,Dream Taq PCR Master Mix 12.5 μL,探针浓度0.4 pmol/μL 加1 μL,DNA模板2 μL,用ddH2O补足至25 μL。扩增条件:95 ℃ 5 min;95 ℃ 15 s,60 ℃ 30 s,45个循环。通过比较分析rPCR的Ct值和扩增曲线等来确定优化结果。每个样品每个处理做3个平行。
探针浓度优化:设置rPCR体系中探针浓度分别为0.1、0.2、0.4、0.6、0.8和1 pmol/μL,Dream Taq PCR Master Mix 12.5 μL,上、下游引物(浓度0.4 pmol/μL)各1 μL,DNA模板2 μL,用ddH2O补足25 μL。扩增条件:95 ℃ 5 min;95 ℃ 15 s,60 ℃ 30 s,45个循环。通过比较分析rPCR的Ct值和扩增曲线等来确定优化的结果。每个样品每个处理做3个平行。
1.2.2.4 灵敏度验证
为了验证该方法的灵敏度,将提取的羊的DNA模板按梯度分别稀释为100、10、1、0.1、0.01、0.001 ng/μL,进行qPCR扩增,实验重复3次,每个梯度2个平行。
1.2.3 rPCR定量检测方法的建立
1.2.3.1 质粒的合成与DNA提取
根据设计羊源性引物探针所选取的保守区域合成质粒pUC57-GH-羊,委托北京六合华大基因科技有限公司合成。质粒的制备按照刘麒等[14]的方法进行,质粒DNA采用小量抽提试剂盒提取。
1.2.3.2 基于拷贝数的羊肉质量与Ct值函数关系的建立
(1)质粒拷贝数与Ct值函数关系的建立
用核酸蛋白仪测定提取的pUC57-GH-羊标准质粒DNA质量浓度,并对质粒DNA进行10倍稀释,采用qPCR进行扩增,以lg(重组质粒拷贝数)为横坐标,对应Ct值为纵坐标生成标准曲线,其中重组质粒标准品拷贝数按公式(1)计算[15]:
拷贝数![]()
(1)
式中:样本质量浓度为测定的质粒含量(ng/μL),经核酸蛋白检测仪测定后,羊质粒质量浓度为240.69 ng/μL;样本分子质量=重组质粒长度×660,其中重组质粒长度按公式(2)计算:
重组质粒长度=T载体长度+目的片段长度
(2)
式中:T载体长度为2 710 bp,目的片段长度为300 bp。
(2)羊肉质量与拷贝数函数关系的建立
将羊肉与猪、鸭肉的干物质(总干物质100 mg)按一定比例混合后,准确称取7 mg干物质提取DNA,提取的DNA进行ddPCR扩增,读取羊肉基因拷贝数。以羊肉质量为y轴,质量对应的拷贝数为x轴建立羊肉质量与基因拷贝数的函数关系。实验重复3次,每次2个平行。具体混合比例如表1所示。
1.2.4 rPCR检测方法灵敏度验证
为了验证该方法的实际检测灵敏度,将羊肉干物质与牛肉、鸭肉、猪肉干物质进行混合。实验重复2次,每次2个平行。具体混合比例如表2所示。
表1 混合肉样中的样品混合比例
Table 1 Mixing ratio of samples in mixed meat samples
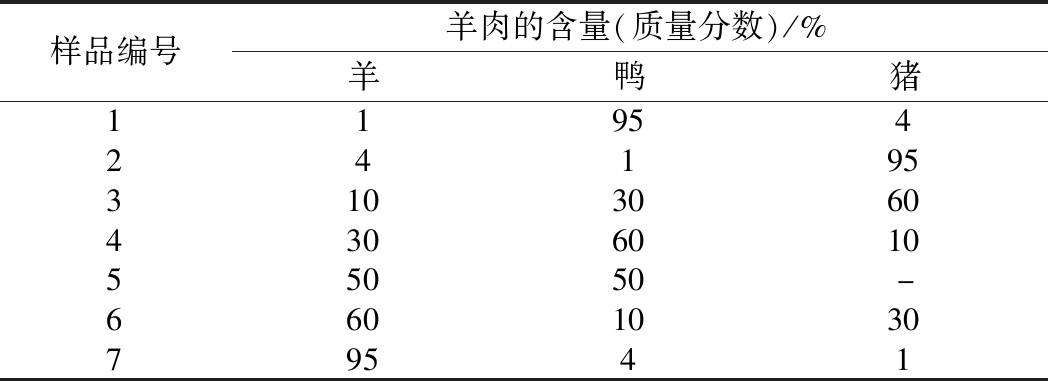
样品编号羊肉的含量(质量分数)/%羊鸭猪11954241953103060430601055050-660103079541
注:-表示不含有(下同)
表2 rPCR检测方法实际灵敏度实验的样品混合比例
Table 2 rPCR detection method sample mixing ratio of actual sensitivity experiment
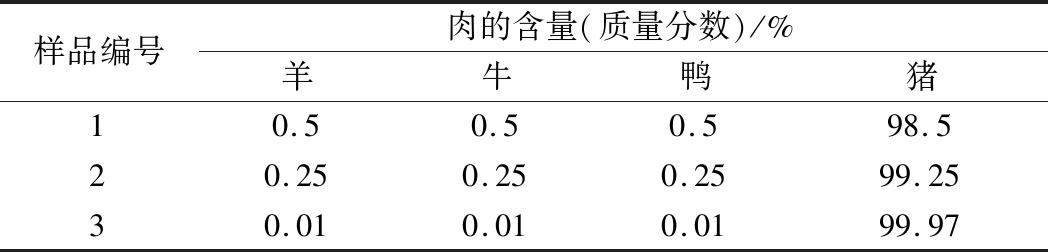
样品编号肉的含量(质量分数)/%羊牛鸭猪10.50.50.598.520.250.250.2599.2530.010.010.0199.97
1.2.5 rPCR检测方法定量线性范围的确定
为确定rPCR定量方法线性范围,将羊肉干物质与猪、鸭肉干物质混合(总干物质10 g),以模拟掺假样品的检测。按照2.2所优化的最佳反应条件进行rPCR扩增。每个比例平行检测3次,通过2.3中得到的函数关系公式计算其含量并与实际样品含量比较,计算平行组内的平均值、相对标准偏差和相对误差,对检测体系的定量准确性进行验证。具体混合比例如表3所示。
表3 rPCR模拟样品混合比例
Table 3 rPCR simulated sample mixing ratio
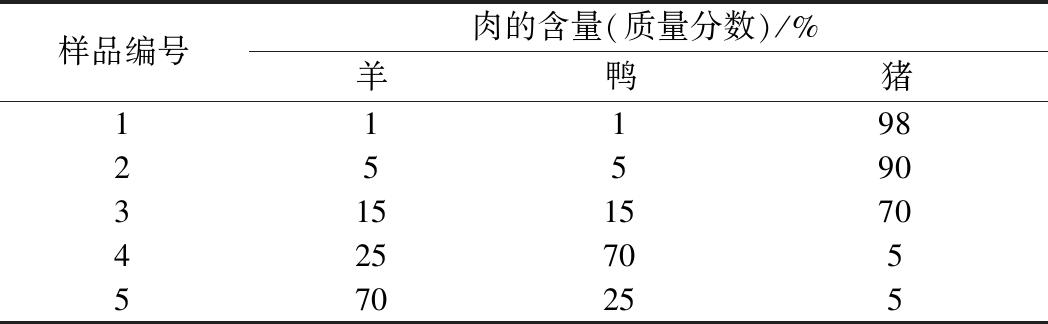
样品编号肉的含量(质量分数)/%羊鸭猪11198255903151570425705570255
1.2.6 市售羊肉样品含量的检测
为了验证本方法在实际羊肉样品检测中的可行性,对2018~2019年成都海关技术中心保存的四川省内超市和农贸市场随机抽取的28个羊肉及其制品进行检测。准确称取经前处理干燥后的1 g待检样品,加入100 mg的内标干物质混合均匀后,取7 mg进行DNA提取,采用现行定性检测标准SN/T 2051—2008食品、化妆品和饲料中牛羊源性成分检测方法进行定性检测,检测结果显示为阳性的样品再利用本实验建立方法进行定量检测,进一步验证所建方法的准确性和实际应用能力。
2 结果与分析
2.1 羊源性引物探针的筛选与设计
2.1.1 单拷贝基因的筛选
GH基因在整个基因组里仅在19号染色体上有1个拷贝,是单拷贝基因。
2.1.2 引物探针的设计
2.1.2.1 序列的筛选
利用序列分析软件DNA Man 6.0 对山羊、绵羊、水牛、黄牛、瘤牛的生长激素基因序列进行比对分析,各物种序列比对结果如图1所示,通过选取羊的特异性保守区域进行引物探针的设计。
2.2.1.2 引物探针的设计
设计的羊源性引物探针基本信息如表4所示。
表4 羊源性引物探针基本信息
Table 4 Basic information of sheep primers probe
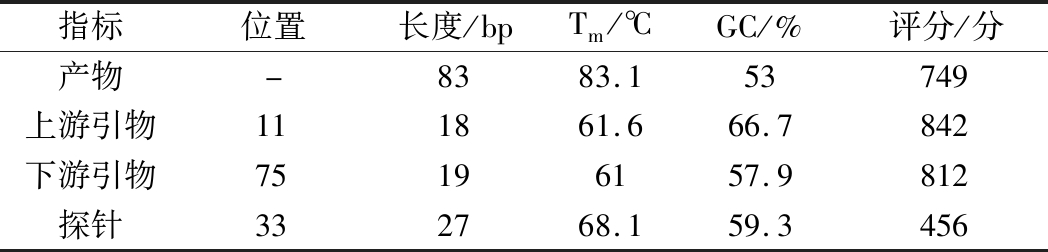
指标位置长度/bpTm/℃GC/%评分/分产物-8383.153749上游引物111861.666.7842下游引物75196157.9812探针332768.159.3456
表5为设计的羊源性GH基因引物探针的序列。合成的引物进行PCR扩增后进一步琼脂糖凝胶电泳检测,最后对产物进行测序,测序结果进行Blast同源性数据库比对后结果显示同源性均在96%以上。

图1 羊与其他物种GH基因序列比对
Fig.1 Comparison of GH gene sequences between sheep and other species
注:左侧序列号从上到下依次为山羊、绵羊、水牛、黄牛、瘤牛
2.1.3 特异性验证
由图2可知,在引物特异性实验中,只有绵羊和山羊的DNA检测到了扩增信号,平均Ct值分别为24.93和25.77,未出现非特异性扩增;而水牛、黄牛、牦牛、猪、鹿、狐狸、鹅、兔、鸽、鳕鱼、大黄鱼、鸭、火鸡、虹鳟鱼、貂、鸡、银鳕鱼、多宝鱼等非目标物种DNA的PCR试验未出现扩增曲线,说明本试验所设计引物特异性良好且能同时对绵羊和山羊成分进行检测。
表5 羊GH基因引物探针序列
Table 5 Primers probe sequence of GH gene in sheep

物种基因引物和探针序列号羊GHGAATCCGCACCCCCTCCAAGGATGTGGTCAGGGGAGTFAM-ATGGGAGGGAACTGAGGACCTCAGTGG-BHQ1NC_019468
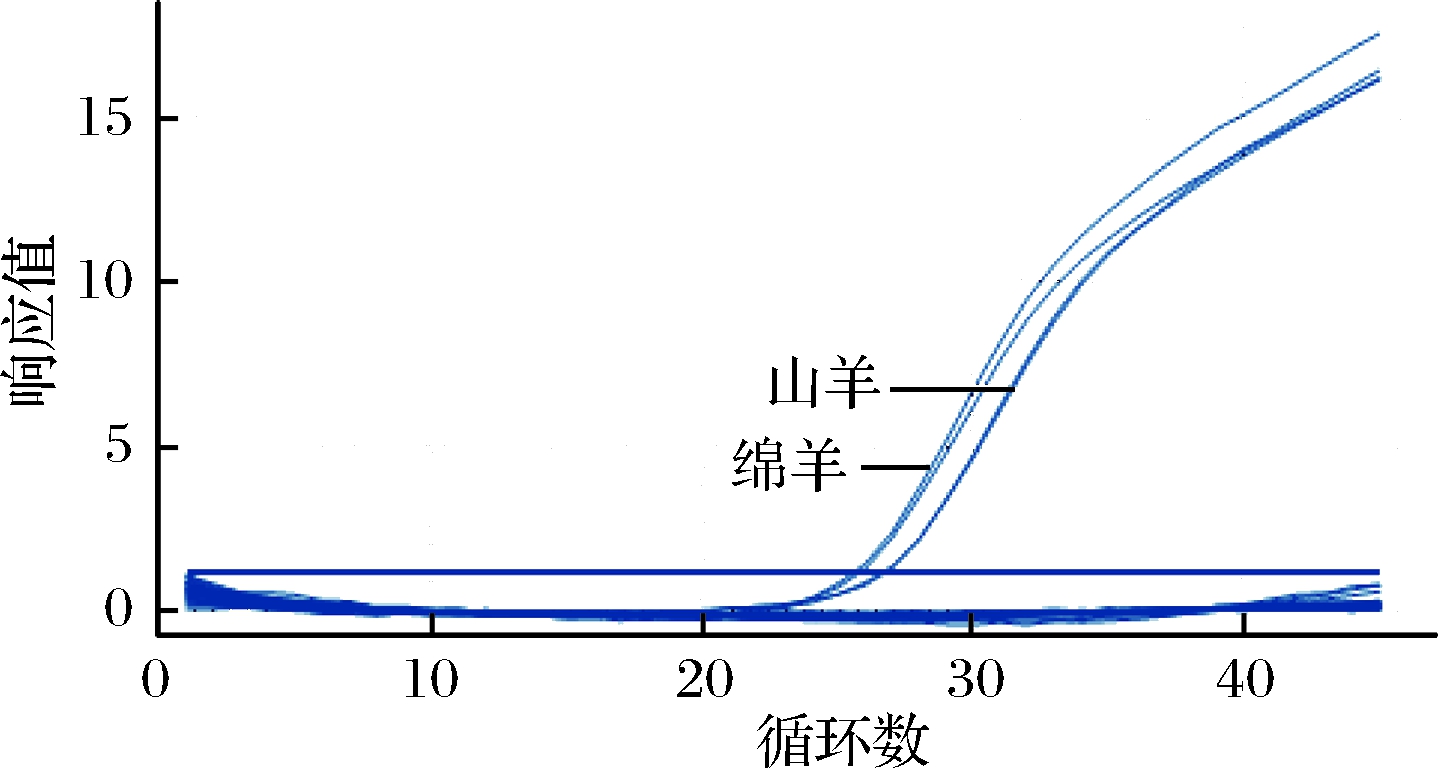
图2 羊源性引物探针rPCR特异性验证
Fig.2 rPCR specificity verification of sheep primers
2.1.4 反应条件的优化
2.1.4.1 rPCR最佳退火温度
由图3可知,将反应的退火温度作为单一变量,当退火温度为60 ℃时,荧光信号强度最强,Ct值为23.01,因此羊源性引物探针rPCR的最佳退火温度为60 ℃。

图3 羊源性引物探针rPCR退火温度优化
Fig.3 rPCR annealing temperature optimization of sheep primers
2.1.4.2 rPCR最佳引物和探针浓度
当rPCR扩增体系中引物终浓度为0.2 pmol/μL时荧光信号最强,Ct值平均值最小为23.286。因此,0.2 pmol/μL为rPCR检测羊源性成分的最佳引物浓度。当rPCR扩增体系中探针终浓度为0.6 pmol/μL时荧光信号最强,Ct值平均值最小为23.07,因此0.6 pmol/μL为rPCR检测羊源性成分的最佳探针浓度。
2.1.5 灵敏度验证
由图4可看出,当DNA质量浓度为0.001 ng/μL时,未发生扩增反应,而当DNA质量浓度为0.01 ng/μL时有扩增,Ct值为38.08。因此,在本实验所建立的方法下,该对引物在rPCR反应体系中检测羊DNA的检测限为0.01 ng/μL,体系总DNA为0.02 ng。
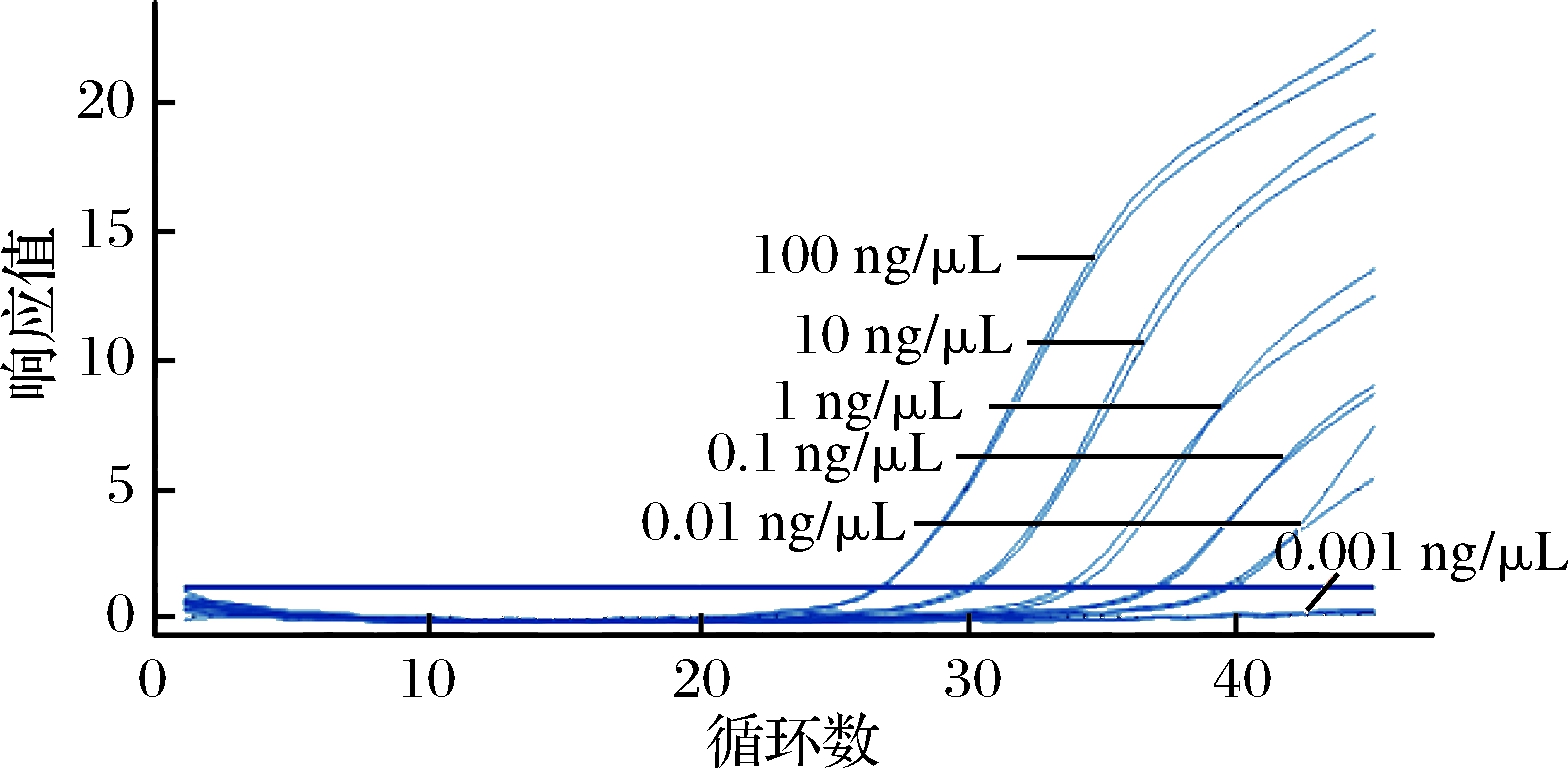
图4 rPCR检测羊肉DNA的灵敏度验证
Fig.4 Sensitivity verification of rPCR for detection of mutton DNA
2.2 rPCR定量检测方法的建立
2.2.1 羊GH基因标准品质粒DNA合成序列与提取结果
2.2.1.1 质粒合成序列
选择用于设计引物的单拷贝基因保守区域作为质粒的合成序列,具体序列如下:
481 cagccatgtc cttgtccggc ctgtttgcca acgctgtgct ccgggctcag cacctgcatc
541 aactggctgc tgacaccttc aaagagtttg taagctcccc agagatgtgt cctagaggtg
601 gggaggcagg aaggggtgaa tccgcacccc ctccacacaa tggga-gggaa ctgaggacct
661 cagtggtatt ttatccaagt aaggatgtgg tcaggggagt ggaaatgggg gtgtgtgggg
721 tggggagggt tccgaataag gcagtgaggg gaaccccgca ccagctgaga cctgggtggg
781 tgtgttctcc ccccaggagc gcacctacat cccggaggga cagagatact ccatccagaa
2.2.1.2 羊GH基因标准品质粒DNA的提取质量
质粒DNA提取的结果如表6所示,提取的pUC57-GH-羊质粒DNA纯度A260/A280均在1.8以上,提取的DNA质量浓度平均值为240.26 ng/μL,说明提取的质粒DNA纯度较好,满足定量检测的要求。
表6 羊GH基因标准品质粒DNA提取质量
Table 6 Sheep GH gene standard plasmid DNA extraction quality

样品类别重复编号A260/A280质量浓度/(ng·μL-1)11.85±0.03242.19±8.06质粒pUC57-GH-羊21.89±0.18239.27±5.3231.89±0.05243.39±6.68
2.2.2 羊肉质量与Ct值函数关系的建立
2.2.2.1 Ct值与拷贝数的函数关系
将经活化的质粒提取DNA,通过10倍稀释后进行rPCR扩增,用核酸蛋白仪对DNA的质量测定,得到羊的质粒质量浓度为240.26 ng/μL。通过拷贝数计算公式得总拷贝数为:7.27×1010。以lg(重组质粒样品稀释度)为横坐标,以对应的Ct值为纵坐标作图得到,羊质粒Ct值和拷贝数线性关系为y=-3.417 7x+72.797,所建标准曲线相关系数(R2)为0.996 4(图5),这表明其线性关系好,可用于准确定量。

图5 羊肉标准质粒的拷贝数与Ct值函数关系
Fig.5 The relationship between the copy number of mutton standard plasmid and Ct value
2.2.2.2 羊肉质量与拷贝数的函数关系
以羊肉质量为y轴,质量对应的拷贝数为x轴建立函数关系。由图6可知,线性方程为y=0.003x+0.097 8,相关系数R2=0.998 4,这表明羊肉的质量与基因拷贝数之间有明显的线性关系。
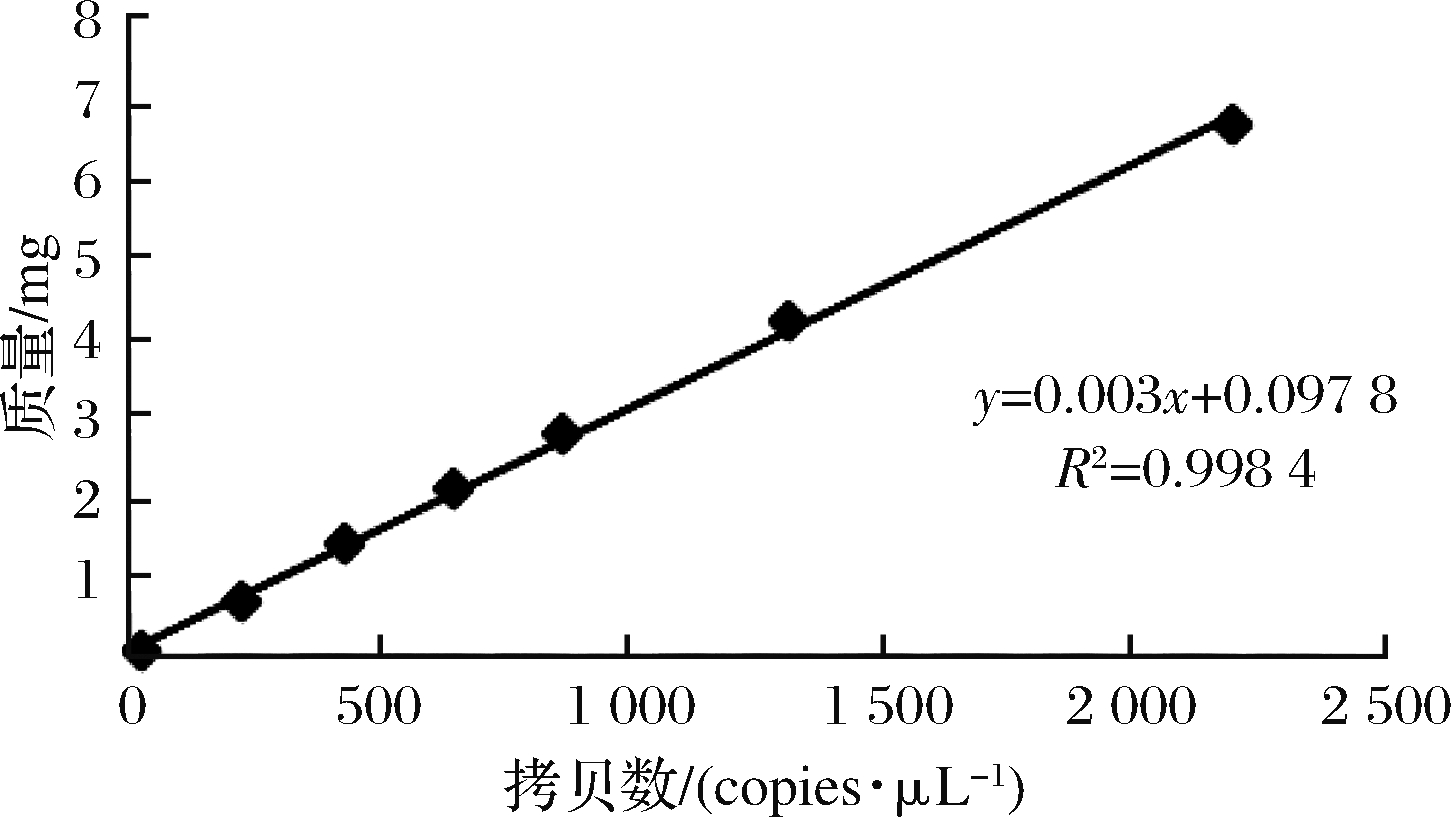
图6 羊肉质量与拷贝数函数关系
Fig.6 Sheep quality and copies number function
2.2.2.3 Ct值与羊肉质量的函数关系
根据2.2.2.1中拷贝数与Ct值的函数关系y=-3.417 7x + 72.797,以及2.2.2.2中羊肉质量与拷贝数的函数关系y=0.003x+0.097 8相结合,以基因拷贝数为中间转化关系,可以由样品rPCR扩增的Ct值计算出样品质量。得出羊肉的质量与Ct值之间的关系如公式(3)所示:
(3)
式中:Mb表示基于基因拷贝数为中间值换算羊肉的质量,A表示羊源性成分扩增的Ct值。
2.3 检测方法灵敏度验证
由图7可知,当羊肉含量为0.25%时,Ct值大于40,视为阴性。即该检测方法实际灵敏度为0.25%,并且随着羊肉比例增加,Ct 值逐渐减小。

图7 rPCR定量方法灵敏度验证
Fig.7 rPCR quantitative method sensitivity verification
2.4 rPCR的定量线性范围及准确性验证
由表7可知,羊肉含量为1%的模拟掺假样品相对标准偏差(RSD)大于25%,不符合国际食品法典委员会关于食品定性和定量检测方法标准中最低定量检测限RSD应小于25%的规定[16]。而其余含羊肉5%~70%的模拟掺假样品RSD值均在14%以内,满足定量检测要求,所以该方法在羊肉含量为5%以上时的准确度和精密度高,因此方法的最低定量检测限为5%。
表7 rPCR定量方法的重复性和准确性验证结果
Table 7 Repeatability and accuracy verification results of rPCR absolute quantification method
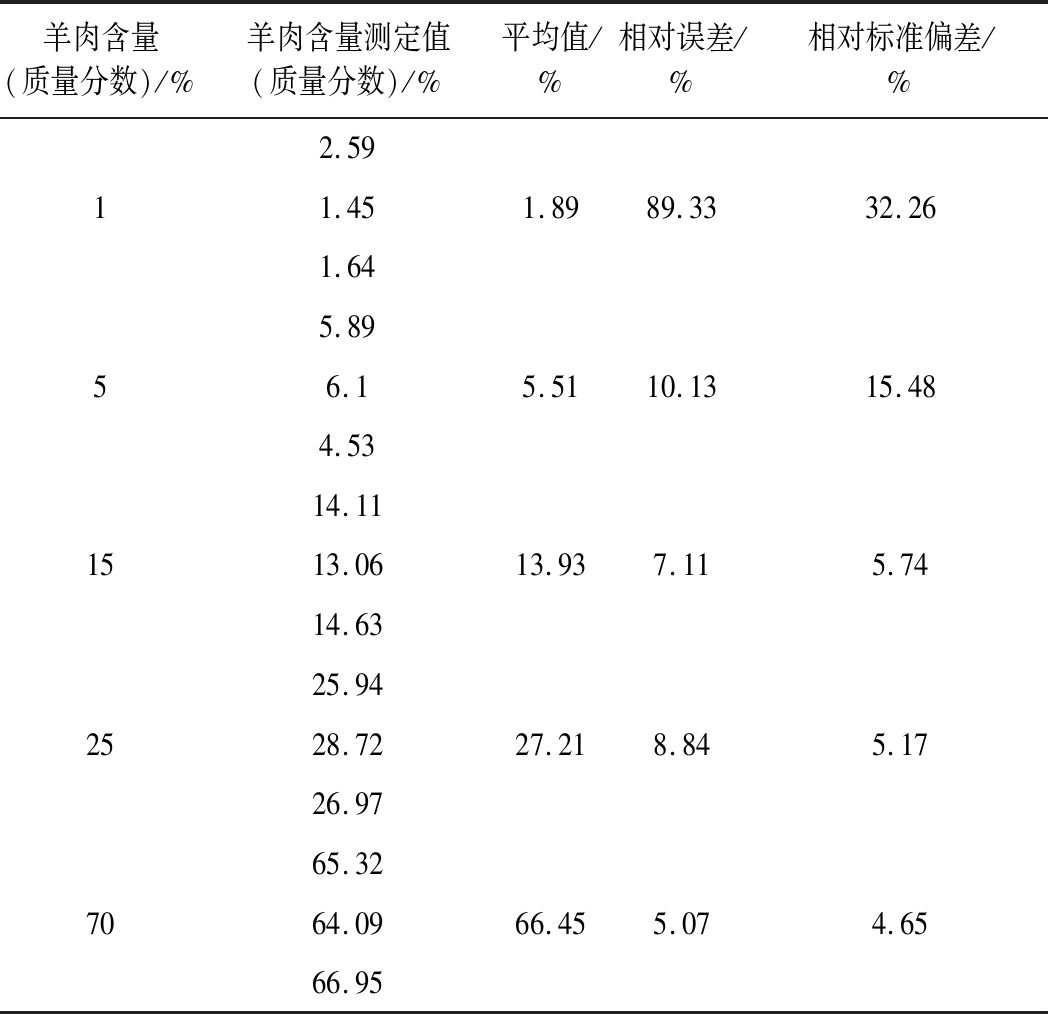
羊肉含量(质量分数)/%羊肉含量测定值(质量分数)/%平均值/%相对误差/%相对标准偏差/%2.5911.451.8989.3332.261.645.8956.15.5110.1315.484.5314.111513.0613.937.115.7414.6325.942528.7227.218.845.1726.9765.327064.0966.455.074.6566.95
2.5 市售羊肉样品含量的检测结果
将本实验建立的定量检测方法和现行检测方法(SN/T 2051—2008)对样品中羊肉含量进行检测,结果如表8所示。
表8 rPCR对市售羊肉样品的检测结果
Table 8 Detection of mutton samples by rPCR

样品编号样品来源样品名称检测结果SN/T2051—2008(阴性-,阳性+)rPCR内标定量法(含量/%)1羊肉片(卷)1+>952羊肉片(卷)2+>953羊肉片(卷)3+>954超市羊肉片(卷)4+>955羊肉片(卷)5+>956羊肉片(卷)6+>957羊肉片(卷)7+>958羊肉片(卷)8+>959羊肉片(卷)9+94.810羊肉片(卷)10+93.511农贸市场羊肉片(卷)11+>9512羊肉片(卷)12+>9513羊肉片(卷)13+>9514羊肉串1+>9515超市羊肉串2+>9516羊肉串3+>9517羊肉串4+>9518羊肉串5+58.519农贸市场羊肉串6+>9520羊肉串7+>9521羊肉串8+>9522半熟羊肉1+>9523超市净羊后腿肉2+>9524冻分割大块羊肉3+>9525羔羊肉4+>9526法式羊排5+>9527农贸市场分割羊肉6+>9528羊肉7+>95
由表8可知,由于现行食品中羊肉含量检测方法(SN/T 2051—2008)仅能够检测出食品中是否含有羊肉成分,但并不能确定羊肉的具体含量。因此,需要进行进一步的检测才能确定其含量。由本实验建立的检测方法可知,来源于农贸市场的3个样品的羊肉含量不足100%,其中2个样品中羊肉含量分别是94.8%和93.5%,这并不能简单判定为掺假,因为掺假的目的是获取更大利润,掺入非羊肉比例太低则无利可图;而且食品在生产加工、流通过程中,不同原料或产品间均存在着交叉污染的可能。因此,综合判定这2个样品可能是加工或流通过程中被其他动物源成分无意污染,而非故意添加。另外一个样品中羊肉所占比例仅为58.5%,可确定为掺假,这表明现有的市售样品存在一定的掺假情况。
3 讨论
为实现rPCR方法检测的可靠性和取样可比性,本研究首先对样品进行干燥处理,以保证检测同种肉样间样品细胞密度趋于一致。rPCR方法应用于肉制品的掺假检验过程中,物种特异基因的正确选择是实现物种鉴定的关键因素。现有鉴定方法多数将研究的基因位点选择在线粒体DNA(mt DNA)上,但THALMANN等[17]认为mt DNA片段可以整合到基因组中,由于进化效率不同,这些整合后的基因可能会和mt DNA同时扩增,对结果分析造成干扰。MURUGAIAH等[18]认为由于不同物种细胞中线粒体的拷贝数各不相同,以线粒体为靶基因的检测结果很难定量分析出食品中动物源性成份的组成比例。FLOREN等[19]研究也表明以线粒体DNA为目标进行物种量化是并不适宜,因为组织中每个细胞mt DNA含量存在至少5倍的差异,这会导致物种DNA含量被低估或高估。与线粒体基因相比,持家基因属于单拷贝基因,具有拷贝数少且数量相对恒定等特点,不会因为取样部位不同导致定量结果出现偏差,更有利于做定量分析。REN 等[20]利用单拷贝基因设计特异性引物,定量检测了食品中牛、猪等动物源性成份,取得了理想效果。因此,为了定量准确性,本文选择单拷贝持家基因生长激素(GH)基因作为靶基因来设计引物。
等[20]利用单拷贝基因设计特异性引物,定量检测了食品中牛、猪等动物源性成份,取得了理想效果。因此,为了定量准确性,本文选择单拷贝持家基因生长激素(GH)基因作为靶基因来设计引物。
此外,对于深加工的肉制品,DNA受到加热影响后容易发生断裂,无法进行PCR的有效扩增[21]。EBBEHOJ等[22]研究发现当肉温加热到100 ℃时DNA片段减少到1 100 bp 左右,而加热到120 ℃时减少至 600 bp 以下。MART N等[23]在采用rPCR检测食品和饲料中猪源性成分时也认为,肉类经过加工后DNA片段会变短,在利用PCR技术进行动物源性成分检测时,引物扩增片段要求尽可能较短。本研究中设计的引物扩增片段长度为83 bp,满足热加工肉制品的分析要求。因此,本研究对18个常见非目标物种DNA进行扩增检测,结果表明引物与探针仅对绵羊和山羊的DNA特异,这降低了其在实际应用中出现非特异性扩增风险。
N等[23]在采用rPCR检测食品和饲料中猪源性成分时也认为,肉类经过加工后DNA片段会变短,在利用PCR技术进行动物源性成分检测时,引物扩增片段要求尽可能较短。本研究中设计的引物扩增片段长度为83 bp,满足热加工肉制品的分析要求。因此,本研究对18个常见非目标物种DNA进行扩增检测,结果表明引物与探针仅对绵羊和山羊的DNA特异,这降低了其在实际应用中出现非特异性扩增风险。
在肉类定量检测中,如何有效得到待检肉类的质量百分比值是研究难点,建立线性回归标准曲线法是目前实现定量检测的有效方法。CAI等[24]利用ddPCR方法检测肉制品中猪肉和鸡肉质量时,发现肉质量与DNA质量以及DNA质量和DNA拷贝数之间呈线性关系,能够通过DNA拷贝数建立计算生肉质量的公式。因此,本研究以羊肉为检测对象,借助ddPCR技术,构建了拷贝数与羊质粒的Ct值、拷贝数与羊肉含量之间的函数关系,以基因拷贝数为中间转化关系,最终构建Ct值与羊肉含量的函数关系。此外,在rPCR的定量研究中,判定Ct值的检测阈值没有统一标准,需根据不同样品、目的基因及引物探针的特异性来综合分析并制定检测阈值[25]。本试验根据样品中羊肉含量为0.25%时Ct值大于40,将Ct值40作为检测阈值,即该检测方法实际灵敏度为0.25%。
为了验证本方法实际的应用能力,将本方法与SN/T 2051—2008食品、化妆品和饲料中牛羊源性成分检测方法对比检测市售样品。结果表明本方法能够对样品中5%以上的羊肉进行精准定量,而且不需要每一次检测都构建标准曲线,方法具有较好重复性和稳定性。对于待检样品,仅需要测定羊源性成分的Ct值就可通过公式得到羊肉含量。
4 结论
本研究将rPCR与ddPCR相结合,建立了基于DNA拷贝数与样品质量间线性关系对羊肉含量进行定量的方法。通过对通过模拟掺假样品和市售样品进行检测,本方法能够对样品中5%以上的羊肉进行精准定量,具有良好的准确性和稳定性,能够为羊肉的掺假鉴别提供参考。
[1] O’MAHONY P J. Finding horse meat in beef products-a global problem[J]. QJM: Monthly Journal of the Association of Physicians, 2013, 106(6): 595-597.
[2] TIAN X, WANG J, SHEN R, et al. Discrimination of pork/chicken adulteration in minced mutton by electronic taste system[J]. International Journal of Food Science & Technology, 2019, 54(3):670-678.
[3] WASINSKI B, OSEK J. New methods of meat species identification and detection of meat adulterations[J]. Medycyna Weterynaryjna, 2013, 69(6): 348-352.
[4] CHENG J H, CHOU H T, Lee M S, et al. Development of qualitative and quantitative PCR analysis for meat adulteration from RNA samples[J]. Food Chemistry, 2016, 192: 336-342.
[5] EBBEHOJ E F, THOMSEN P D. Differentiation of closely related species by DNA hybridization[J]. Meat Science, 1991, 30(4): 359-366.
[6] TARTAGLIA M, SAULLE E, PESTALOZZA S, et al. Detection of bovine mitochondrial DNA in ruminant feeds: a molecular approach to test for the presence of bovine-derived materials[J]. Journal of Food Protection, 1998, 61(5): 513-518.
[7] AMARAL J S, SANTOS G, OLIVEIRA M B P P, et al. Quantitative detection of pork meat by EvaGreen real-time PCR to assess the authenticity of processed meat products[J]. Food Control, 2017, 72: 53-61.
[8] 林彦星, 张彩虹, 阮周曦, 等. 实时荧光定量PCR检测畜禽肉制品中鸭源性成分[J]. 动物医学进展, 2016, 37(11): 48-53.
[9] KÖPPEL R, GANESHAN A, WEBER S, et al. Duplex digital PCR for the determination of meat proportions of sausages containing meat from chicken, turkey, horse, cow, pig and sheep[J]. European Food Research and Technology, 2019, 245: 853-862.
[10] NOH E S, PARK Y J, KIM E M, et al. Quantitative analysis of Alaska pollock in seafood products by droplet digital PCR[J]. Food Chemistry,2019,275:638-643.
[11] KÖPPEL, R, GANESHAN A, WEBER S, et al. Duplex digital PCR for the determination of meat proportions of sausages containing meat from chicken, turkey, horse, cow, pig and sheep[J]. European Food Research and Technology, 2019, 245: 853-862.
[12] FLOREN C, WIEDEMANN I, BRENIG B, et al. Species identification and quantification in meat and meat products using droplet digital PCR (ddPCR)[J]. Food Chemistry, 2015, 173: 1 054-1 058.
[13] 陈传君, 金鹭, 林华, 等. 干制羊肉基因组DNA不同提取方法的比较研究[J]. 核农学报,已录用,待刊.
[14] 刘麒, 王阔鹏, 于凌娇, 等.电转化条件对大肠杆菌TG1转化效率的影响[J]. 吉林农业科技学院学报, 2018, 27(3): 6-9.
[15] SMITH C J, NEDWELL D B, DONG L F, et al. Evaluation of quantitative polymerase chain reaction-based approaches for determining gene copy and gene transcript numbers in environmental samples[J]. Environmental Microbiology, 2006, 8(5):804-815.
[16] Codex Committee on Methods of Analysis and Sampling. Guidelines on performance criteria and validation of methods for detection identification and quantification of specific DNA sequences and specific proteins in foods: CAC/GL740-2010[S]. Rome: Codex Alimentariusliment, 2010.
[17] THALMANN O,HEBLER J,POINAR H N,et al.Unreliable mt DNA data due to nuclear insertions: A cautionary tale from analysis of humans and other great apes[J]. Molecular Ecology, 2004, 13(2): 321-335.
[18] MURUGAIAH C, NOOR Z M, MASTAKIM M, et al. Meat species identification and Halal authentication analysis using mitochondrial DNA[J]. Meat Science, 2009, 83(1): 57-61.
[19] FLOREN C, WIEDEMANN I, BRENIG B, et al. Species identification and quantification in meat and meat products using droplet digital PCR (dd PCR)[J]. Food Chemistry, 2015, 173(173): 1 054-1 058.
[20] REN K, JÜRG R, JÜRG R. Multiplex real-time PCR for the detection and quantification of DNA from beef, pork, horse and sheep[J]. European Food Research and Technology, 2011, 232(1): 151-155.
K, JÜRG R, JÜRG R. Multiplex real-time PCR for the detection and quantification of DNA from beef, pork, horse and sheep[J]. European Food Research and Technology, 2011, 232(1): 151-155.
[21] HIRD H, CHISHOLM J, SANCHEZ A, et al. Effect of heat and pressure processing on DNA fragmentation and implications for the detection of meat using a real-time polymerase chain reaction[J]. Food Additives and Contaminants, 2006, 23(7): 645-650.
[22] EBBEHOJ K F, THOMSEN P D. Species differentiation of heated meat products by DNA hybridization[J]. Meat Science, 1991, 30(3):221-234.
[23] MARTíN I, GARC A T, VIOLETA F, et al. SYBR-Green real-time PCR approach for the detection and quantification of pig DNA in feedstuffs[J]. Meat Science, 2009, 82(2): 252-259.
A T, VIOLETA F, et al. SYBR-Green real-time PCR approach for the detection and quantification of pig DNA in feedstuffs[J]. Meat Science, 2009, 82(2): 252-259.
[24] CAI Y C, HE Y P, LV R, et al. Detection and quantification of beef and pork materials in meat products by duplex droplet digital PCR[J]. Plos one, 2017, 12(8): e091 949.
[25] IZADPANAH M, MOHEBALI N, GORJI Z E, et al. Simple and fast multiplex PCR method for detection of species origin in meat products[J]. Journal of Food Science and Technology, 2018, 55(2): 698-703.