丙酮酸及其盐作为一类重要的平台化合物,在农用化学品、聚合物、化妆品、食品添加剂、医药等领域具有重要的应用[1-2]。例如,丙酮酸及其盐是合成丙氨酸、缬氨酸、亮氨酸、N-乙酰-D-神经氨酸、二羟苯基丙氨酸(用于治疗帕金森疾病)等贵重化合物的关键底物[2-3];最近的临床研究表明,丙酮酸能有效治疗和预防与碳水化合物、脂肪和能量代谢紊乱(压力、肥胖、糖尿病、心血管疾病等)有关的疾病[3]。传统的丙酮酸生产通常采用化学法,该法虽然操作简单,但也存在成本较高,产率低、污染环境等明显缺点[4];相比之下,微生物发酵法因具有以可再生生物质为底物、成本低廉、反应条件温和、产品具有高纯度等优点[5],更具市场竞争优势。
目前,丙酮酸发酵菌株研究最多的主要有多种维生素营养缺陷型光滑念珠菌(Candida glabrata,之前称为Torulopsis glabrata)[6-7]、硫辛酸营养缺陷型大肠杆菌(Escherichia coli)[8-9]工程菌株。然而,由于T. glabrata和E. coli具有条件致病性[10-11],很大程度上限制其应用范围。相比之下,酿酒酵母(Saccharomyces cerevisiae)因具有长期用于食品与酿造等领域,被认证为GRAS(generally regarded as safe)微生物,发酵产品具有安全性;遗传背景清晰,有着较为完善的遗传优化工具,便于遗传改造操作;能耐受低pH条件,可减少酸性产品的中和需求,有利于下游产品处理;抗渗透性强,能耐受高浓度的产物和底物[12]等优点,被认为是丙酮酸生产的潜在最适菌株之一,已引起国内外的广泛关注。
然而,由于S. cerevisiae糖酵解过程产生的丙酮酸在胞质内的丙酮酸脱羧酶(pyruvate decarboxylase,PDC)催化作用下会迅速裂解为CO2和乙醛[13],造成自身无法大量积累丙酮酸。因此,要实现S. cerevisiae大量积累丙酮酸,减弱丙酮酸的降解途径是关键。减弱丙酮酸的降解途径最直接的方法就是失活PDC,该酶受3个结构基因(pdc1、pdc5、pdc6)表达调控。同时敲除这3个结构基因会导致工程菌株无法在以葡萄糖为唯一碳源的培养基上生长,但通过长期进化能获得可利用葡萄糖积累丙酮酸的菌株[14-15]。
适应性进化又称进化工程,是一种通过模拟自然进化中的变异和选择过程,在人工选择的压力下实现微生物的进化,最后从进化菌群中筛选出优良性状的菌种改良技术[16-17]。该技术能够驱使微生物种群或单细胞微生物在较短的时间内,为适应环境的变化,自身的DNA/RNA碱基或基因组结构等自发产生突变[17-18]。适应性进化不像基因工程那样对微生物遗传背景有很强的依赖性,因此该技术尤其适合对生理特性缺乏了解的菌种以及复杂性状的选育。目前实验室最常用的适应进化方法是在特定条件(给予选择压力)下将微生物连续传代培养,通过菌株自发突变的不断积累,获得适应特定条件的表型或生理性能。
本研究以1株工业酿酒酵母XY-49为出发菌株,利用Cre/loxp同源重组方法敲除其丙酮酸脱羧酶基因(pdc1、pdc5、pdc6),削弱或阻断乙醇合成途径,从而实现丙酮酸的积累;同时采用葡萄糖适应性定向进化策略对突变菌株进行传代驯化培养,以期筛选到葡萄糖耐受度高、菌体生长速度快、丙酮酸高效积累的工程菌株。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
本研究所使用的酿酒酵母菌株如表1所示;敲除组件质粒pUG6、筛选标记回收质粒pSH65,均由本实验室自存。
表1 本研究所用的酿酒酵母菌株
Table 1 S. cerevisiae strains used in the present study

菌株基因型/特征来源XY-49MATa/α pdc1/pdc1 pdc5/pdc5 pdc6/pdc6,wild-typestrain实验室保藏XY-6MATa/α pdc1/pdc1 pdc5/pdc5 pdc6::loxp/pdc6::loxp本研究构建XY-56MATa/α pdc1/pdc1 pdc5::loxp/pdc5::loxp pdc6::loxp/pdc6::loxp本研究构建XY-156MATa/α pdc1::loxp/pdc1::loxp pdc5::loxp/pdc5::loxp pdc6::loxp/pdc6::loxp本研究构建XY-156AMATa/α pdc1::loxp/pdc1::loxp pdc5::loxp/pdc5::loxp pdc6::loxp/pdc6::loxp,EvolvedXY-156本研究构建
1.1.2 引物设计与合成
根据NCBI公布的酿酒酵母pdc1、pdc5和pdc6基因碱基序列,设计了表2中的PCR引物,所有引物均由上海Invitrogen公司合成。
表2 PCR引物
Table 2 Primers used in PCR

引物名称引物序列(5′-3′)用途P6-AGAATTGATCTGTATCTGCACCTAGATCGATTTGATTACAGTTCGTACGCTGCAGGTCGA敲除pdc6P6-BGTAACATGCGAATTGCGTAATTCACGGCGATAACGTAGGCATAGGCCACTAGTGGATCTP6-FCATTCTCTAAACAAAAGCCCATGTC验证pdc6P6-RGGCTGCGGAATTTTATAAGACAAGCP5-ACTACATGCGTTTATGGGAAAAGCCTCCATATCCAAAGGTCTTCGTACGCTGCAGGTCGA敲除pdc5P5-BAGGTATGGTTAAAGATCACACCACCCTCTTCAATTAGCGCATAGGCCACTAGTGGATCTP5-FGCGTTTATGCGTTTTGCGCCCCTTG验证pdc5P5-RCAATTGGCGAAGCAGAACAAGTTTTCP1-ACCTTTTTCTGTTAGACGGTGTCTTGATCTACTTGCTATCGTTCGTACGCTGCAGGTCGA敲除pdc1P1-BGCCAATTCAACAGACTGTCGGCAACTTCTTGTCTGGTCGCATAGGCCACTAGTGGATCTP1-FATGTATGCTCTTCTGACTTTTCGTG验证pdc1P1-RGCTGGCAGTTTTGAATTGAG
注:下划线部分代表目标基因的同源臂序列
1.1.3 酶与试剂
LA Taq DNA聚合酶及其相关试剂,TaKaRa公司;乙醇脱氢酶、遗传霉素(G418),索莱宝生物科技有限公司;博莱霉素(Zeocin),invitrogen公司;其余试剂为进口分装或者国产分析纯。
1.1.4 培养基
YPD培养基(g/L):胰蛋白胨20,酵母粉 10,葡萄糖 20,pH自然,121℃ 灭菌20 min。
YPE培养基(g/L):胰蛋白胨 20,酵母粉 10,乙醇 2%(体积分数),pH自然,121 ℃灭菌20 min。
SD培养基(g/L):酵母氮源(YNB) 6.7,(NH4)2SO4 5,葡萄糖 20,无菌0.22 μm滤膜除菌。
发酵培养基(g/L):初始葡萄糖80~100,酵母粉 10,胰蛋白胨 10,K2SO46,KH2PO43,MgSO4 0.5,金属离子母液 10 mL/L,115 ℃,灭菌20 min。其中,糖与其他成分分开灭菌。
金属离子母液(g/L)[19]:EDTA 1.5,ZnSO4·7H2O 0.45,CoCl2·6H2O 0.03,MnCl2·4H2O 0.1,CuSO4·4H2O 0.03,CaCl2·2H2O 0.45,FeSO4·7H2O 0.3 g/L,NaMoO4·2H2O 0.04,H3BO4 0.1,KI 0.01。
1.2 实验方法
1.2.1 基因敲除
以XY-49为出发菌株,参照文献[20]的方法分离制备其MATa和MATα两型单倍体细胞,然后以质粒pUG6为模版,使用引物A和B(表2)进行PCR扩增,PCR条件为 95 ℃、3 min;95 ℃、30 s,55 ℃、30 s,72 ℃、1.8 min,30个循环;72℃、10 min。获得的敲除组件片段含有目的基因上下游同源序列、loxp位点以及kamX抗性筛选标记。PEG/LiAc法分别转化两种配型单倍体细胞(MATa型和MATα型),再分别涂布到含300 μg/mL G418的YPD(或YPE)平板上,30 ℃培养3~5 d,挑取克隆子进行扩培后提取基因组,以此为模版,使用引物F和R(表2)进行验证。PCR条件为95 ℃、3 min;95 ℃、30 s,60 ℃、30 s,72 ℃、2 min,30个循环;72 ℃、10 min。将质粒pSH65转化到阳性克隆子中,半乳糖诱导切除kamX抗性筛选标记,通过至少12次转接培养移除pSH65,获得阳性克隆子。本文遵循pdc6→pdc5→pdc1的次序重复以上过程将上述基因逐个敲除。
1.2.2 MATa型和MATα型单倍体杂交复倍
参照文献[21]的方法,将1.2.1步骤中得到的具有相同基因突变类型的单倍体(MATa型和MATα型)进行杂交复倍,得到相应的二倍体菌株(表1)。
1.2.3 酶活检测
粗酶提取:将酿酒酵母细胞接入100 mL YPD(含80 g/L 葡萄糖)培养基中,30 ℃、220 r/min培养至对数期,然后12 000 r/min离心1 min收集细胞,用10 mmol/L磷酸钾缓冲液(pH 7.0)清洗2次,悬浮并置于-20 ℃备用;检测前用20 mL、100 mmol/L磷酸钾缓冲液(pH 7.4)重悬,并加入1 mmol/L DTT,冰上冷冻5 min后在超声波仪(输出功率为200 W)中超声5 s,冰上间隔冷却1 min,重复过程15次。4 ℃、36 000×g条件下离心20 min,取上清即细胞提取物置于冰上,随即用于酶活的检测[22]。总蛋白量以考马斯亮蓝法[23]测定。
丙酮酸脱羧酶反应混合液每1 mL包含40 mmol/L咪唑盐酸缓冲液,0.2 mmol/L硫胺素焦磷酸,0.15 mmol/L NADH,乙醇脱氢酶88 U/mL,5 mmol/L MgCl2,20 μL细胞破碎上清液,反应起始于50 mmol/L丙酮酸钾的加入,并在30 ℃,pH 7.4条件下,测定OD340处的吸光度变化[19]。每分钟催化1 μmol NADH氧化所需的酶量定义为1个酶活单位(U)。比酶活(U/mg)定义为每mg总蛋白所含有的酶活。
1.2.4 pdc三重缺失突变株的葡萄糖适应性进化
针对pdc缺失突变株无法利用葡萄糖为唯一碳源进行批次发酵的问题,本文参照文献[24]报道的方法对目标突变株进行了葡萄糖适应性定向进化筛选,以改善突变株的细胞生长及葡萄糖利用等生理指标。
1.2.5 突变菌株的生理特性试验
从-80 ℃冰箱取出菌株在YPE(含2%乙醇)平板上划线活化,30 ℃培养箱中培养3 d;挑取活化的单菌落接到5 mL液体YPE(含体积分数为2%乙醇)培养基中,30 ℃、 220 r/min培养2 d; 5 000 r/min、 离心5 min,弃掉上清液,用5 mL无菌水重悬;测定菌体的OD600值,接入装有100 mL发酵培养基(含80 g/L葡萄糖)的250 mL三角瓶中,使发酵初始OD600≈0.2,30 ℃、220 r/min摇床培养96 h。
1.2.6 2 L发酵罐发酵培养
种子培养:将突变菌株XY-156A接入YPD液体培养基中,30 ℃、220 r/min培养48 h,种子连续活化2次。发酵培养:将培养好的种子液按照体积分数为10%的比例接入2 L发酵罐(上海百伦科技),发酵培养基装液量为1 L,发酵温度设为30 ℃,搅拌转速为400 r/min,通气速率为0.75 L/min,在发酵过程中使用10 mol/L KOH自动维持pH=4.5。
1.2.7 分析方法
生物量测定:取0.5 mL菌液,稀释适当倍数,利用Beckman coulter UV 800 紫外可见分光光度计在波长为 600 nm 条件下测定菌体浓度。
代谢产物分析:代谢产物采用高效液相色谱仪(戴安Ultimate 3000)分析,丙酮酸用紫外检测器(ultraviolet absorption detector,UV)检测,检测波长为210 nm;乙醇用视差检测器(refractive index detecor,RI)检测,色谱柱为Phenomenex Rezex ROA-Organic Acid H+(8%)(300 mm×7.8 mm),流动相为2.5 mmol/L H2SO4,流速为0.6 mL/min,柱温50 ℃。葡萄糖测定采用SBA-40D生物传感分析仪。
2 结果与分析
2.1 pdc基因敲除转化子的验证结果
从G418抗性平板上挑取阳性克隆子,扩大培养并提取基因组DNA作为模版,以对应配型的亲本菌株(MATa型和MATα)为对照,分别以P6-F/P6-R、P5-F/P5-R和P1-F/P1-R(表2)为引物进行PCR全长验证,如图1所示,对照扩增出2.4 kb左右大小的pdc6基因片段,转化子则扩增出大小约为2.0 kb的基因片段,与预期结果相符,证明pdc6基因敲除组件片段已成功置换pdc6基因的开放阅读框(open reading frame,ORF),pdc6基因被敲除。同理,如图2和图3所示,证明pdc5和pdc1基因敲除组件片段已成功置换各自的ORF区域,pdc5和pdc1基因被敲除。
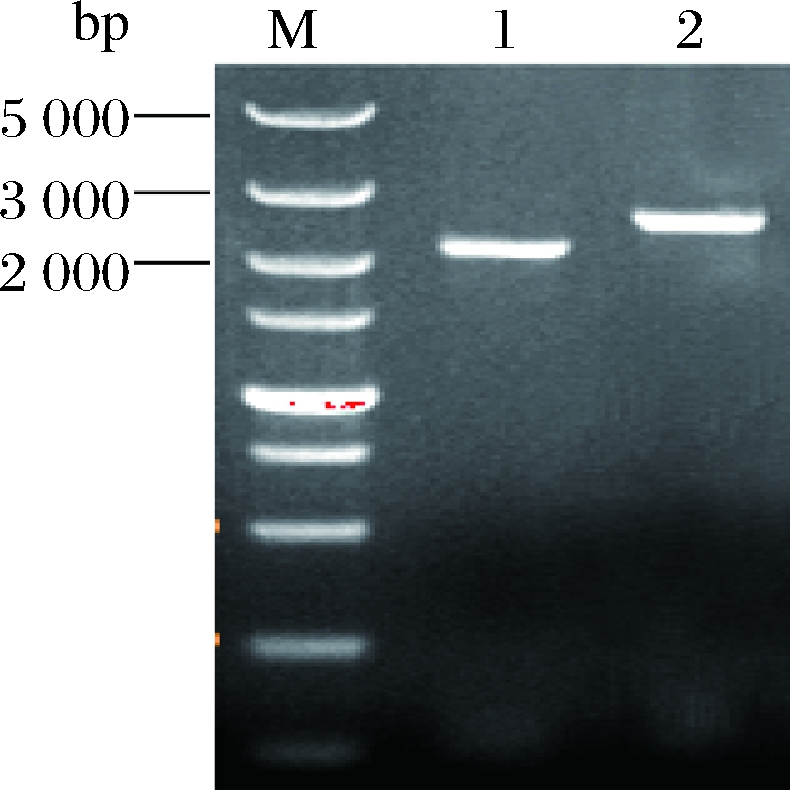
M,DL5000DNA marker;1,缺失开放阅读框的pdc6基因的PCR产物;2,pdc6基因的PCR产物
图1 基因pdc6突变子PCR验证
Fig.1 PCR verification of gene pdc6 mutant

M,DL5000DNA marker;1,缺失开放阅读框的pdc5基因的PCR产物;2,pdc5基因的PCR产物
图2 基因pdc5突变子PCR验证
Fig.2 PCR verification of gene pdc5 mutant
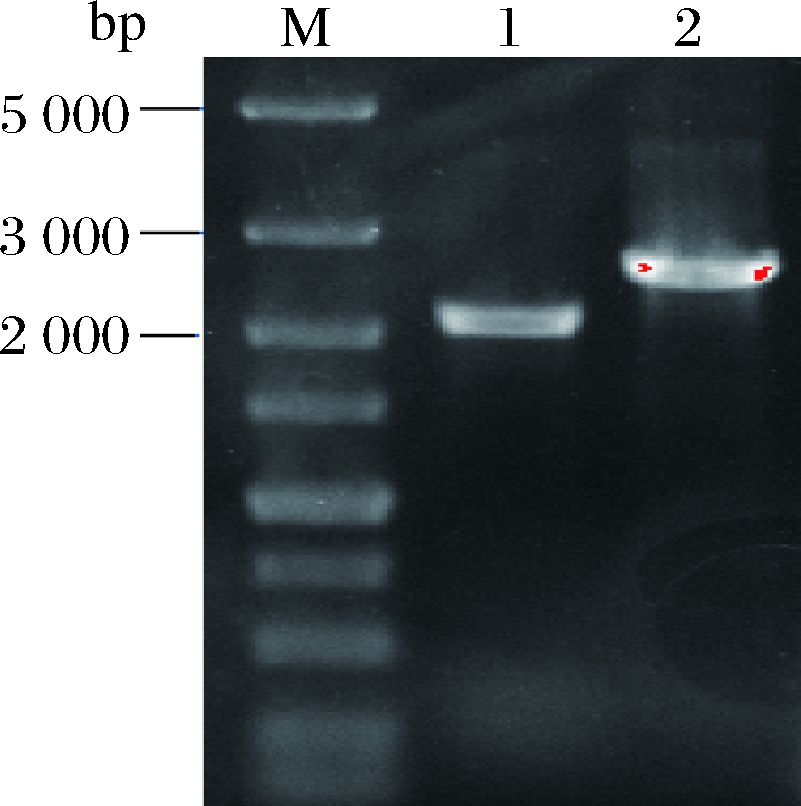
M,DL5000DNA marker;1,缺失开放阅读框的pdc1基因的PCR产物;2,pdc1基因的PCR产物
图3 基因pdc1突变子PCR验证
Fig.3 PCR verification of gene pdc1 mutant
2.2 PDC酶活的检测分析
按照1.2.3的方法,进一步通过测定突变株的PDC酶活,以最终确定pdc三个等位基因被敲除,结果如表3所示。由表3可知,当酿酒酵母的pdc6基因缺失后,其比酶活为原始菌株的92%;当pdc5和pdc6同时缺失之后,其比酶活为原始菌株的86%;当pdc1、pdc5和pdc6同时缺失之后,其比酶活为原来的0.3%,说明pdc 3个等位基因已成功缺失。
表3 突变菌株丙酮酸脱羧酶比酶活
Table 3 Specific pyruvate decarboxylase activity of wild-type and pdc mutant strains

菌株 PDC酶活/(U·mg-1)XY-492.95XY-62.73XY-562.48XY-1560.01XY-156A0.01
2.3 突变菌株的生理特性发酵试验
细胞生长及耗糖情况的测定:摇瓶液体平行培养XY-49、XY-6、XY-56、XY-156和XY-156A菌株,测定整个生长过程的菌体生物量(OD600)及耗糖情况,结果如图4所示。pdc6单独缺失,菌体生长、葡萄糖消耗速率几乎不受影响;pdc5和pdc6双基因缺失对其葡萄糖消耗速率影响很小,但其菌体生物量相比原始菌株稍有降低;pdc1、 pdc5和pdc6三基因缺失后,突变株(XY-156)生长延滞期变长,对数期较短,发酵进行到60 h时,其生物量(OD600)为2.21±0.12,较野生菌株XY-49下降了86%。此时,突变株XY-156葡萄糖消耗了(14.55±1.02) g/L,野生菌株消耗了(79.3±1.2) g/L,表明pdc三个等位基因缺失之后,菌株的生长、葡萄糖消耗受到了较为强烈的影响。

A-细胞生长;B-葡萄糖消耗
图4 不同突变菌株的细胞生长和葡萄糖消耗情况
Fig.4 Cell growth and glucose consumption of different mutant strains
突变菌株产乙醇、丙酮酸情况的测定:在发酵培养过程中,定期取样检测发酵液中乙醇、丙酮酸的含量,考察pdc基因缺失对其主要代谢产物(乙醇、丙酮酸)合成的影响,结果如表4所示。当pdc6单基因缺失或pdc5和pdc6双基因缺失时,获得的突变菌株(XY-6、XY-56)与对照菌株XY-49相比,乙醇合成几乎不受影响,丙酮酸积累量很低;而当pdc1、pdc5和pdc6三基因缺失以后,突变菌株XY-156的丙酮酸积累量较对照菌株XY-49有了大幅提升,发酵过程中没有乙醇产生。
表4 突变菌株主要代谢产物的检测 单位:g/L
Table 4 Determination of main metabolites of different mutant strains
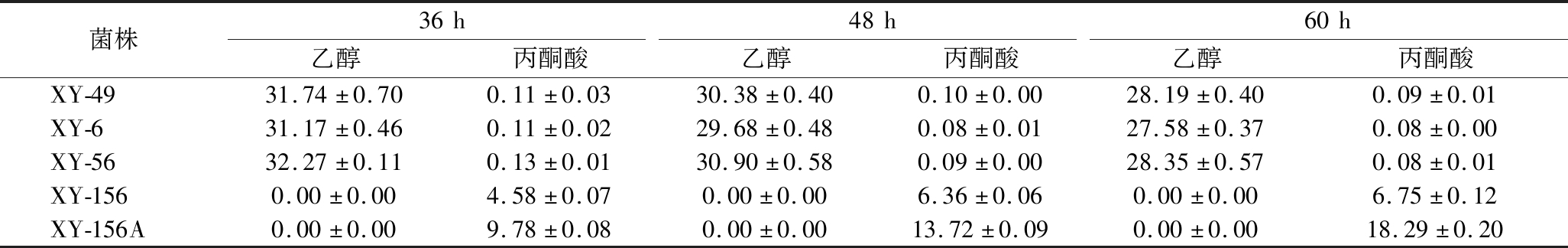
菌株36h48h60h乙醇丙酮酸乙醇丙酮酸乙醇丙酮酸XY-4931.74±0.700.11±0.0330.38±0.400.10±0.0028.19±0.400.09±0.01XY-631.17±0.460.11±0.0229.68±0.480.08±0.0127.58±0.370.08±0.00XY-5632.27±0.110.13±0.0130.90±0.580.09±0.0028.35±0.570.08±0.01XY-1560.00±0.004.58±0.070.00±0.006.36±0.060.00±0.006.75±0.12XY-156A0.00±0.009.78±0.080.00±0.0013.72±0.090.00±0.0018.29±0.20
结合表3、表4和图4可知,pdc6单独缺失或pdc5和pdc6同时缺失对细胞生长、葡萄糖利用、乙醇以及丙酮酸积累影响不大,说明pdc5、pdc6在PDC表达中不起主导作用,这与文献[19]报道一致。同时敲除pdc1、pdc5和pdc6,可完全消除乙醇并实现丙酮酸积累,但同时也导致了突变菌株XY-156对葡萄糖敏感、细胞生长缓慢。这是由于pdc基因缺失以后,胞质内缺少乙酰-CoA(由PDC酶催化丙酮酸脱羧而来)供应,脂质、赖氨酸的生物合成过程受阻所致[15]。此外,由图4和表4可知,虽然进化突变菌株XY-156A的生物量和糖耗速率仍不及原始菌株XY-49,但与进化前菌株XY-156相比,其在细胞生物量、丙酮酸积累、葡萄糖耐受及消耗速率方面均有显著提高。目前,有关pdc-进化突变株耐受葡萄糖机制的解释主要有以下两种:(1)胞质乙酰-CoA替代路径的激活。VAN MARIS 等[25]研究发现,苏氨酸醛缩酶(该酶可以将苏氨酸裂解为甘氨酸和乙醛)的胞质锚定位点,并试图引入一条胞质乙酰-CoA合成替代路径;通过过表达苏氨酸醛缩酶编码基因gly1,pdc-菌株在碳源限制的条件下可利用葡萄糖作为唯一碳源进行生长,表明该酶催化形成的乙醛可作为胞质乙酰-CoA合成的前体,该研究结果较好地论证了这一假设,但仍无法解释gly1过表达的pdc-菌株在批次培养中对高浓度葡萄糖敏感的现象;(2)葡萄糖摄取过程的调控。OUD等[26]对1株不依赖C2化合物的S. cerevisiae TAM进行转录组分析时发现一个葡萄糖摄取负调控因子(mth1)内部碱基发生了突变,并通过后续试验推测MTH1降解减少导致的葡萄糖摄取下调或许是pdc-菌株耐受高浓度葡萄糖的原因。
2.4 2 L发酵罐发酵实验
通过葡萄糖适应性定向进化策略筛选得到突变菌株XY-156A之后,进一步利用2L发酵罐对其积累丙酮酸能力进行放大试验。发酵初始糖质量浓度为98 g/L,当葡萄糖质量浓度降至约10 g/L时,补加800 g/L葡萄糖母液使糖质量浓度恢复至80~90 g/L,整个发酵过程共补料2次。定期取样检测发酵过程中丙酮酸、残糖浓度以及生物量的变化情况,取3次试验的平均值,结果如图5所示。
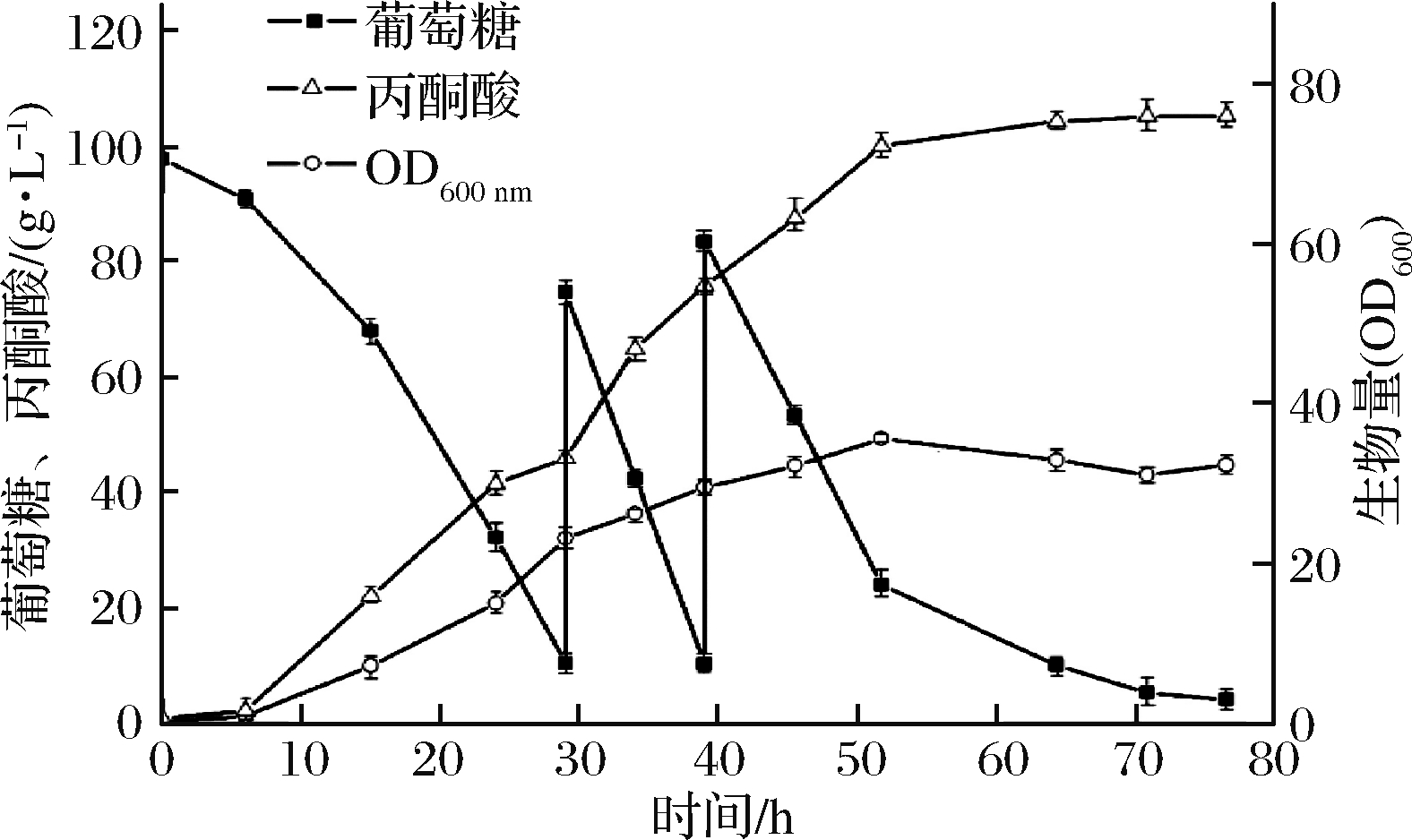
图5 S.cerevisiae XY-156A利用葡萄糖批次补料发酵产丙酮酸过程曲线
Fig.5 Fed-batch fermentation profile of pyruvic acid production from Glucose by S.cerevisiae XY-156A
在0~30 h,随着糖的不断消耗,菌体生物量、丙酮酸浓度不断增长;在30~52 h,期间共进行2次补料,丙酮酸浓度继续攀升,菌体生物量在52 h时达到最大(OD600=35.56);52 h后,糖耗速率放缓,菌体生物量也随之缓慢下降,丙酮酸浓度继续缓慢增加。发酵76 h后,丙酮酸浓度达到105 g/L,得率为0.5 g/g葡萄糖,生产强度为1.38 g/(L·h)。与摇瓶发酵培养相比,发酵罐发酵培养阶段的菌体生长速率、生物量、糖耗速率以及丙酮酸终浓度均有大幅度提升。这可能是由于发酵罐体系的溶氧比摇瓶体系更为充足, 因而更有利于pdc-突变菌株通过线粒体呼吸链将糖酵解过程产生的NADH快速氧化生成NAD+[27],以维持胞内的氧化还原平衡,而NADH的减少则可促使更多的葡萄糖转化为丙酮酸。此外,发酵罐的传质传热效果更好,菌体与物料接触更充分,从而更有利于菌体生长和产物合成。
3 讨论
近年来,国内外针对改造S. cerevisiae生产丙酮酸开展了大量的研究工作,并且取得了不错的进展。例如,WANG等[28]以单倍体菌株Y2为出发菌株,通过同源重组技术敲除PDC的编码基因pdc1和pdc5使PDC酶活下降了98%,但获得的突变株Y2-15需要添加醋酸钠或者其他C2化合物才能正常生长,摇瓶发酵96 h获得24.85 g/L丙酮酸,生产强度为0.26 g/(L·h),丙酮酸得率为0.37 g/g葡萄糖。VAN MARIS等[14]通过敲除PDC的编码基因pdc1、pdc5和pdc6,获得的突变株RWB837同样无法在以葡萄糖为唯一碳源的合成培养基上生长,随后对该菌株进行葡萄糖适应性进化筛选,最终获得1株能利用葡萄糖积累丙酮酸的进化突变株TAM,分批补料发酵100 h获得135 g/L丙酮酸(目前报道的最高产量),生产强度为1.35 g/(L·h),丙酮酸得率为0.54 g/g葡萄糖。WANG等[24]也采用类似进化策略对1株PDC突变株BY5419进行驯化,最终筛选到了1株产丙酮酸的菌株BY5419-A0,通过摇瓶发酵120 h获得66.4 g/L丙酮酸,生产强度为 0.55 g/(L·h),丙酮酸得率为0.55 g/g葡萄糖。上述研究均使用单倍体营养缺陷型S. cerevisiae作为出发菌株,使其不能直接利用简单培养基,需要添加氨基酸或维生素等,增加了发酵过程的控制难度和发酵成本,不利于大规模工业化应用。本研究以二倍体工业酿酒酵母XY-49为出发菌株,通过失活PDC结合适应性定向进化的策略,最终筛选到葡萄糖耐受度高、菌体生长速度快、丙酮酸高效积累的进化突变株XY-156A。通过分批补料发酵76 h,丙酮酸浓度可达105 g/L,生产强度为1.38 g/(L·h),丙酮酸得率为0.5 g/g葡萄糖,试验结果显示该菌株在丙酮酸工业生产中具备一定的应用潜力。但其所获得的丙酮酸得率(0.5 g/g葡萄糖)仍然较低,因此,有必要进一步改造XY-156A以提高其丙酮酸生产指标。首先,继续敲除其余丙酮酸降解途径的相关基因。除了PDC以外,仍然存在一些可降解丙酮酸的酶,例如,丙酮酸脱氢酶(pyrucate dehydrogenase,PDH)和丙酮酸羧化酶(pyruvate carboxylase,PYC),敲除这些酶的编码基因可进一步减少丙酮酸代谢[27]。其次引入辅酶再生系统。PDC缺失后,糖酵解过程产生的大量NADH无法快速转化为NAD+,细胞的氧化还原平衡被破坏,而引入辅酶系统促进NAD+再生,可将更多的葡萄糖转化为丙酮酸,从而提高丙酮酸得率[24]。最后,对发酵条件进行综合优化,为S. cerevisiae工业化生产丙酮酸奠定坚实的基础。
[1] LI Y, CHEN J, LUN S Y. Biotechnological production of pyruvic acid[J]. Appl Microbiol Biotechnol, 2001, 57(4): 451-459.
[2] MALEKI N, EITEMAAN M. Recent progress in the microbial production of pyruvic acid[J]. Fermentation, 2017, 3(1): 8.
[3] SONG Y, LI J, SHIN H, et al. Biotechnological production of alpha-keto acids: Current status and perspectives[J]. Bioresourcetechnology, 2016, 219: 716-724.
[4] XU Ping, QIU Jianhua, GAO Cao, et al. Biotechnological routes to pyruvate production[J]. Journal of Bioscience and Bioengineering, 2008, 105(3): 169-175.
[5] LI Shubo, CHEN Xiulai, LIU Liming, et al. Pyruvate production in Candida glabrata: Manipulation and optimization of physiological function[J]. Critical Reviews in Biotechnology, 2016, 36(1): 1-10.
[6] LIU Liming, XU Qinglong, LI Yin, et al. Enhancement of pyruvate production by osmotic‐tolerant mutant of Torulopsis glabrata[J]. Biotechnology and bioengineering, 2007, 97(4): 825-832.
[7] ZHANG Jian, GAO Nianfa. Application of response surface methodology in medium optimization for pyruvic acid production of Torulopsis glabrata TP19 in batch fermentation[J]. Journal of Zhejiang University SCIENCE B, 2007, 8(2): 98-104.
![]() B, GERHARZ T, BOTT M, et al. Fed-batch process for pyruvate production by recombinant Escherichia coli YYC202 strain[J]. Engineering in Life Sciences, 2003, 3(7): 299-305.
B, GERHARZ T, BOTT M, et al. Fed-batch process for pyruvate production by recombinant Escherichia coli YYC202 strain[J]. Engineering in Life Sciences, 2003, 3(7): 299-305.
![]() S, VUORILEHTO K, et al. Process strategies to enhance pyruvate production with recombinant Escherichia coli: From repetitive fed-batch to in situ product recovery with fully integrated electrodialysis[J]. Biotechnology and Bioengineering, 2004, 85(6): 638-646.
S, VUORILEHTO K, et al. Process strategies to enhance pyruvate production with recombinant Escherichia coli: From repetitive fed-batch to in situ product recovery with fully integrated electrodialysis[J]. Biotechnology and Bioengineering, 2004, 85(6): 638-646.
[10] 韩全乡, 王宗生,刘金淑. 医院真菌感染128例分析[J]. 实用预防科学, 2004, 11(1): 180-181.
[11] XU Da, ZUO Jiakun, CHEN Zhaoguo, et al. Different activated methyl cycle pathways affect the pathogenicity of avian pathogenic Escherichia coli[J]. Veterinary microbiology, 2017, 211: 160-168.
[12] PLEISSNER D, DIETZ D, DUUREN J B J H V, et al. Biotechnological production of organic acids from renewable resources[J]. Advances in Biochemical Engineering/Biotechnology, 2017, 166: 373-410.
[13] BARNETT J A. The utilization of sugars by yeasts[J]. Advances in Carbohydrate Chemistry and Biochemistry, 1976, 32: 125-234.
[14] VAN MARIS A J A, GEERTMAN J M A, VERMEULEN A, et al. Directed evolution of pyruvate decarboxylase-negative Saccharomyces cerevisiae, yielding a C2-Independent, glucose-tolerant, and pyruvate-hyperproducing yeast[J]. Appl Environ Microbiol, 2004, 70(1): 159-166.
[15] FLIKWEERT M T, DE SWAAF M, VAN DIJKEN J P, et al. Growth requirements of pyruvate-decarboxylase-negative Saccharomyces cerevisiae[J]. FEMS Microbiology Letters, 1999, 174(1): 73-79.
[16] 巩继贤, 郑辉杰, 郑宗宝. 微生物进化工程育种技术进展与展望[J]. 生物加工过程, 2010, 8(2): 69-76.
[17] SAUER U. Evolutionary engineering of industrially important microbial phenotypes[J]. Adv Biochem Eng Biotechnol, 2001,73: 129-169.
[18] GUIMARAES P M R, FRANCOIS J, PARROU J L, et al. Adaptive evolution of a lactose-consuming Saccharomyces cerevisiae recombinant[J]. Appl Environ Microbiol, 2008, 74(6): 1 748-1 756.
[19] FLIKWEERT M T, VAN DER ZANDEN L, JANSSEN W M T M, et al. Pyruvate decarboxylase: An indispensable enzyme for growth of Saccharomyces cerevisiae on glucose[J]. Yeast, 1996, 12(3): 247-257.
[20] 肖冬光, 刘青, 李静, 等. 酿酒酵母单倍体制备方法的优化[J]. 酿酒科技, 2004(4): 21-22.
[21] 罗贞贞, 陈小玲, 练梅华, 等. 酿酒酵母Mbp1缺陷型菌株的构建及其乙醇发酵特性[J]. 微生物学通报, 2015, 42(11): 2 108-2 114.
[22] 闫道江, 王彩霞, 周杰民, 等. 酿酒酵母产苹果酸的还原TCA 路径构建及发酵调控[J]. 生物工程学报, 2013, 29(10): 1 484-1 493.
[23] BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1-2): 248-254.
[24] WANG Zhikun, GAO Cuijuan, WANG Qian, et al. Production of pyruvate in Saccharomyces cerevisiae through adaptive evolution and rational cofactor metabolic engineering[J]. Biochemical Engineering Journal, 2012, 67: 126-131.
[25] VAN MARIS A J A, LUTTIK M A H, WINKLER A A, et al. Overproduction of threonine aldolase circumvents the biosynthetic role of pyruvate decarboxylase in glucose-limited chemostat cultures of Saccharomyces cerevisiae[J]. Appl Environ Microbiol, 2003, 69(4): 2 094-2 099.
[26] OUD B, FLORES C L, GANCEDO C, et al. An internal deletion in MTH1 enables growth on glucose of pyruvate-decarboxylase negative, non-fermentative Saccharomyces cerevisiae[J]. Microbial Cell Factories, 2012, 11(1): 131.
[27] PRONK J T, STEENSMA H Y, VAN DIJKEN J P. Pyruvate metabolism in Saccharomyces cerevisiae[J]. Yeast, 1996, 12: 1 607-1 633.
[28] WANG Depei, WANG Lu, HOU Li, et al. Metabolic engineering of Saccharomyces cerevisiae for accumulating pyruvic acid[J]. Annals of Microbiology, 2015, 65(4): 2 323-2 331.