L-苯丙氨酸(L-phenylalanine, L-Phe)作为人体必需的8种氨基酸之一,在医药、食品领域均有广泛应用[1-2]。L-苯丙氨酸生产主要分为化学合成法、酶法和微生物发酵法。由于化学合成和酶法生产存在成本高、原料来源有限及环境污染严重等问题,微生物发酵法已成为主要生产方法[3]。目前,主要通过代谢工程改造大肠杆菌(Escherichia coli)作为细胞工厂合成L-苯丙氨酸[4-6],但是噬菌体污染和食品安全问题限制了它的应用[7]。与大肠杆菌和其他细菌微生物相比,用酿酒酵母(Saccharomyces cerevisiae)作为宿主具有产物安全性、高酸受性、高底物受性和抗噬菌体等诸多优势[8],在现代工业中已得到了广泛的应用。此外,酿酒酵母遗传背景清晰,适合进行多种遗传修饰。
酿酒酵母作为真核模式微生物,经过多年的研究,已成为理解高等生物代谢调控机理的主要模式生物之一[9]。然而,酿酒酵母中芳香族氨基酸代谢部分基因功能和部分调控机理依然缺乏,无法有效指导芳香族氨基酸及其高价值衍生物(柚皮素、白藜芦醇和苯乙烯等)的工业生产。因此,后基因组时代对酿酒酵母芳香族氨基酸代谢网络的注释就显得极其重要。
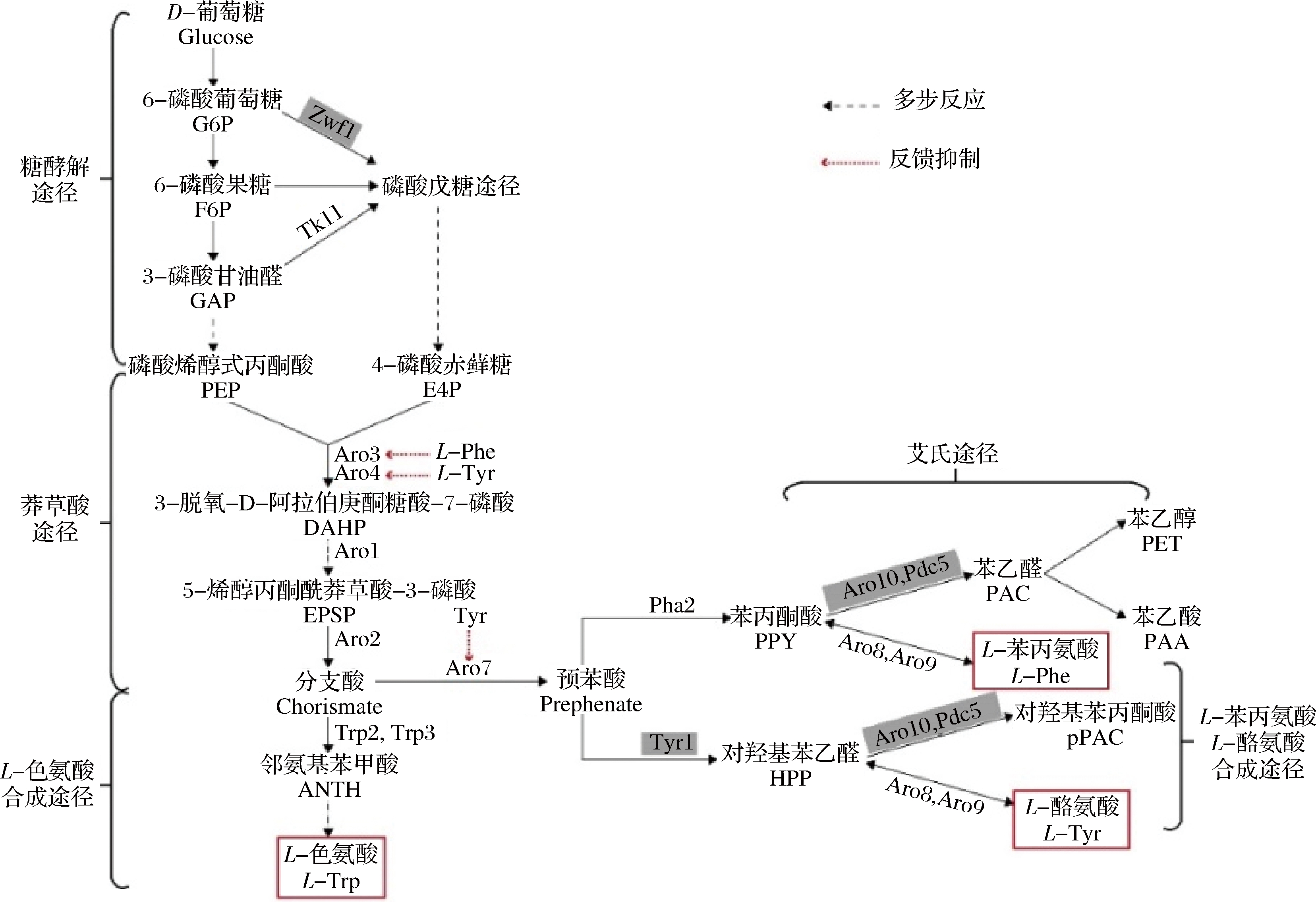
在酿酒酵母中,L-苯丙氨酸合成途径可分为3部分:中心碳代谢、莽草酸和分支酸途径(图1)。研究表明,敲除5-磷酸葡萄糖脱氢酶基因(ZWF1)并过量表达转酮醇酶基因(TKL1),有助于平衡前体物质磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP)和赤藓糖-4-磷酸(erythritol-4-phosphate,E4P)的比例,同时增加E4P进入莽草酸途径的流量[10]。在莽草酸途径中,通过抗反馈亚型Aro4K229L和Aro7G141S取代天然的DAHP合酶同工酶Aro4和分支酸变位酶Aro7,可解除L-酪氨酸对Aro4和Aro7的反馈抑制用以促进芳香族化合物的合成[11],而目前尚无关于抗反馈亚型DAHP合酶同工酶Aro3的确切报道。此外,弱化苯丙酮酸脱羧酶基因ARO10和PDC5,可以减少艾氏途径对L-苯丙氨酸的消耗[12-13]。另一方面,由TYR1基因编码的预苯酸脱氢酶催化碳代谢流向L-酪氨酸合成分支[14]。

图1 酿酒酵母中芳香族氨基酸的生物合成和分解代谢途径
Fig.1 The biosynthesis and catabolism of aromatic amino acids in S. cerevisiae
注:在本研究菌株代谢改造中,过量表达或定点突变的酶用蓝色字体标记;利用CRISPR-Cas9技术敲除的酶用灰色方框表示
L-苯丙氨酸合成途径是酿酒酵母细胞内最为复杂的代谢途径之一, 在野生型细胞中其合成主要用于菌体生长和蛋白质合成等。本研究以酿酒酵母S288c为出发菌株,利用CRISPR-Cas9基因编辑技术分别对中心代谢途径、莽草酸途径、艾氏途径及竞争性L-酪氨酸合成途径进行重构。首先,通过优化中心代谢途径和莽草酸途径,获得1株初步积累L-苯丙氨酸的菌株;在此基础上,弱化艾氏途径,减少L-苯丙氨酸胞内的消耗;最后,阻断竞争性L-酪氨酸合成途径,使更多的碳流向L-苯丙氨酸合成分支。本实验结果为继续利用酿酒酵母解析芳香族氨基酸代谢途径的调控机制,生产芳香族氨基酸及其高价值衍生物提供参考。
表1列出了本研究中使用的菌株和质粒。大肠杆菌菌株JM109用于质粒的亚克隆。
表1 本研究所使用的菌株和质粒
Table 1 Strains and plasmids used in this study
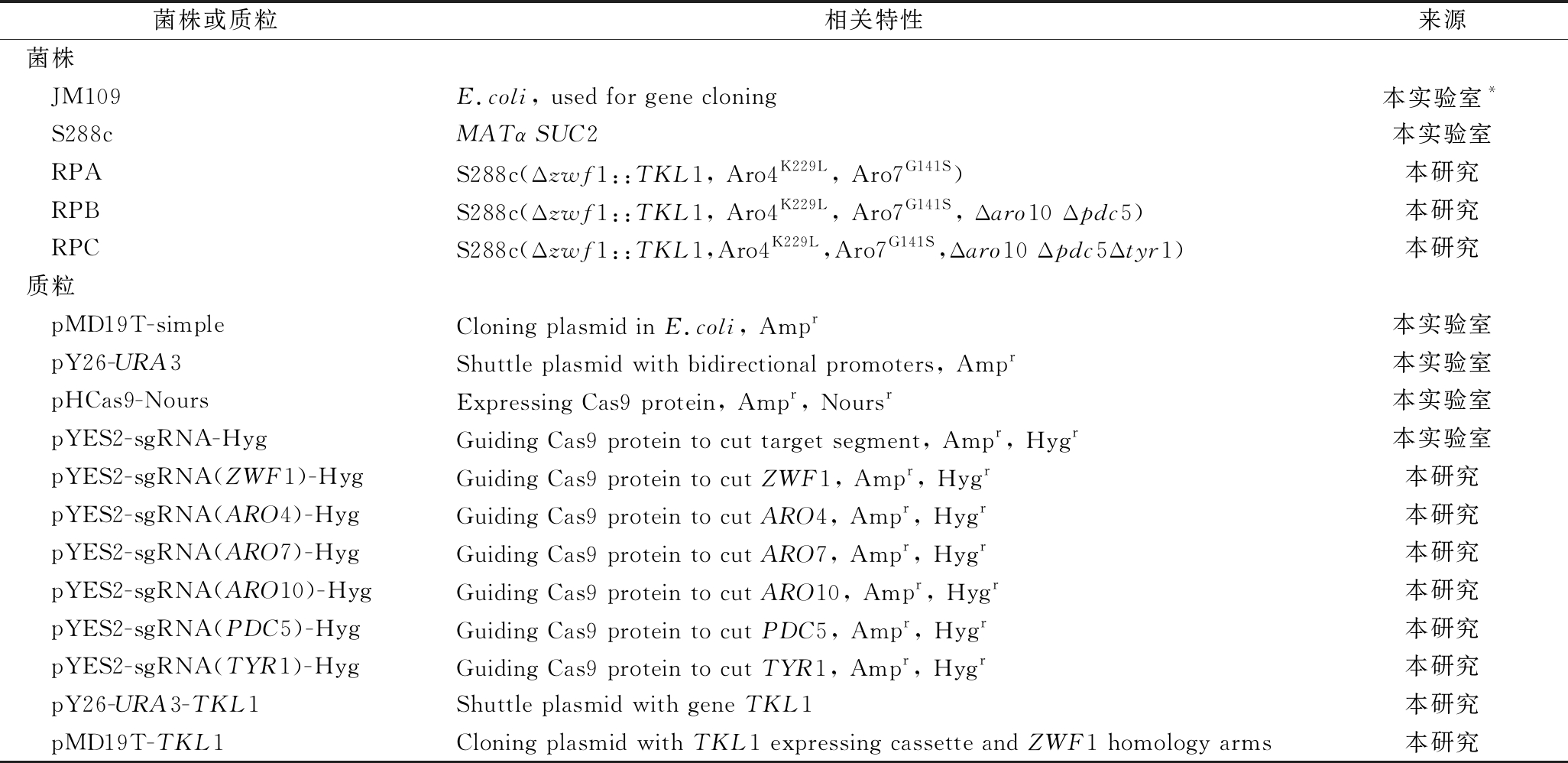
菌株或质粒相关特性来源菌株 JM109E.coli, used for gene cloning本实验室* S288cMATα SUC2本实验室 RPAS288c(Δzwf1::TKL1, Aro4K229L, Aro7G141S)本研究 RPBS288c(Δzwf1::TKL1, Aro4K229L, Aro7G141S, Δaro10 Δpdc5)本研究 RPCS288c(Δzwf1::TKL1,Aro4K229L,Aro7G141S,Δaro10 Δpdc5Δtyr1)本研究质粒 pMD19T-simpleCloning plasmid in E.coli, Ampr本实验室 pY26-URA3Shuttle plasmid with bidirectional promoters, Ampr本实验室 pHCas9-NoursExpressing Cas9 protein, Ampr, Noursr本实验室 pYES2-sgRNA-HygGuiding Cas9 protein to cut target segment, Ampr, Hygr本实验室 pYES2-sgRNA(ZWF1)-HygGuiding Cas9 protein to cut ZWF1, Ampr, Hygr本研究 pYES2-sgRNA(ARO4)-HygGuiding Cas9 protein to cut ARO4, Ampr, Hygr本研究 pYES2-sgRNA(ARO7)-HygGuiding Cas9 protein to cut ARO7, Ampr, Hygr本研究 pYES2-sgRNA(ARO10)-HygGuiding Cas9 protein to cut ARO10, Ampr, Hygr本研究 pYES2-sgRNA(PDC5)-HygGuiding Cas9 protein to cut PDC5, Ampr, Hygr本研究 pYES2-sgRNA(TYR1)-HygGuiding Cas9 protein to cut TYR1, Ampr, Hygr本研究 pY26-URA3-TKL1Shuttle plasmid with gene TKL1本研究 pMD19T-TKL1Cloning plasmid with TKL1 expressing cassette and ZWF1 homology arms本研究
注:粮食发酵工艺与技术国家重点实验室(江南大学)
1.1.2 培养基
大肠杆菌生长所用SOB(super optimal broth)培养基(g/L):蛋白胨20,酵母粉5,NaCl 0.5, KCl 0.186, MgSO4·7H2O 2.203。配制固体SOB时加入20 g/L琼脂粉。
酿酒酵母种子培养基(g/L):酵母粉10,蛋白胨20,葡糖糖20。配制固体时加入20 g/L琼脂粉。
发酵培养基(g/L): 酵母氮源(含(NH4)2SO4 5 g/L) 7,葡糖糖50,蛋氨酸(Met) 0.03。用NaH2PO4和Na2HPO4调节pH至6.5。
1.1.3 主要主要工具酶及试剂
2×Taq PCR Master Mix、2×Pfu PCR Master Mix,杭州宝赛公司;Fast DigestedTM快速限制性内切酶,美国Thermo公司;T4 DNA连接酶和pMD19T-simple,大连TaKaRa公司;L-苯丙氨酸、L-酪氨酸、L-色氨酸、氨苄青霉素、G418、诺尔丝菌素、潮霉素,Sigma-Aldrich公司(上海);质粒DNA小剂量提取试剂盒、DNA 纯化试剂盒和DNA凝胶回收试剂盒,北京博大泰克生物有限公司;蛋白胨、酵母粉、琼脂粉,OXOID公司;其他试剂均为国产或进口分析纯。在菌株构建和摇瓶发酵过程中,培养基中添加的氨苄青霉素、诺尔丝菌素、潮霉素终质粒浓度分别为100、100、500 μg/mL。
1.2.1 CRISPR-Cas9基因编辑系统
此方法为本实验室开发。采用双质粒转化系统,其中一个为含有Cas9编码基因的pHCas9质粒,另一个为含有打靶目的基因sgRNA的pYES2质粒。将pYES2质粒和目的基因上下游同源臂通过CH3COOLi转化法[15]导入含有pHCas9质粒的细胞中,可以达到敲除目的基因的目的。pYES2质粒产生靶向目的基因sgRNA,与pYES2质粒产生的Cas9蛋白结合, sgRNA 20 bp的N20序列与目的基因碱基配对成功后Cas9蛋白会剪短目的基因双链,同时导入酵母的线性同源臂在切口两侧发生同源重组,完成敲除过程。质粒的丢失通过在酿酒酵母无抗性培养基传代完成。本研究所用引物及同源臂见表2。
1.2.2 培养方法
大肠杆菌在补充有100 μg/mL氨苄青霉素的50 mL SOB培养基中,37 ℃、200 r/min条件下培养。酿酒酵母在装有20 mL YPD种子培养基的100 mL三角瓶中30 ℃、200 r/min培养至指数期,用做菌株的转化和发酵前的预培养。预培养物经无菌水洗涤后以初始OD600=0.2接种至装有50 mL YNB发酵培养基的100 mL三角瓶中30 ℃、200 r/min培养,定时取样用于测定芳香族氨基酸及其衍生物。添加L-酪氨酸用于补充营养缺陷型菌株RPC的需求。
表2 本研究所用引物
Table 2 Primers and oligonucleotides used in this study

引物名称引物序列(5'—3')引物长度/bp质粒构建引物 ZWF1-sgRNA-FGGGAGCTGCAAAAAAACCTGGTTTTAGAGCTAGAAATAGCAAG43 ZWF1-sgRNA-RCAGGTTTTTTTGCAGCTCCCGATCATTTATCTTTCACTGCGGAG44 ARO10-sgRNA-FGGCAATTCCAACACCTAGGGGTTTTAGAGCTAGAAATAGCAAG43 ARO10-sgRNA-RCCCTAGGTGTTGGAATTGCCGATCATTTATCTTTCACTGCGGAG44 ARO4-sgRNA-FGCTTAGTAACACCCATGAAAGTTTTAGAGCTAGAAATAGCAAG43 ARO4-sgRNA-RTTTCATGGGTGTTACTAAGCGATCATTTATCTTTCACTGCGGAG44 ARO7-sgRNA-FGGTGATGATAAGAATAACTTGTTTTAGAGCTAGAAATAGCAAG43 ARO7-sgRNA-RAAGTTATTCTTATCATCACCGATCATTTATCTTTCACTGCGGAG44 PDC5-sgRNA-FGATAAGCTTTATGAAGTCAAGTTTTAGAGCTAGAAATAGCAAG43 PDC5-sgRNA-RTTGACTTCATAAAGCTTATCGATCATTTATCTTTCACTGCGGAG44 TYR1-sgRNA-FGTCACTAGAATGACTCAGAAGTTTTAGAGCTAGAAATAGCAAG43 TYR1-sgRNA-RTTCTGAGTCATTCTAGTGACGATCATTTATCTTTCACTGCGGAG44 sgRNA-FAAGGAGGGTATTCTGGGCCTCCATGTC27 sgRNA-RTCTGCAGAATTCGTCGACGAGCTCGGTAC29 sgRNA-v-FGCTAAATGTACGGGCGACAGTCAC24 sgRNA-v-RCGCGTTGGCCGATTCATTAATGCAG25 Donor DNA ARO10TTCTGTAAAGTTTATTTACAAGATAACAAAGAAACTCCCTTAAGCAAACTTGTGGGCGCAATTATAAAACACTGCTACCAATTGTTCGTT90 ARO4TGTTGCTGTGGATGCTTGTCAAGCCGCT-GCTCATTCTCACCATTTCATGGGTGTTACTTTGCATGGTGTTGCTGCTATCACCACTACTAA90 ARO7GAATAACTTCTCTTCTGTTGCCACTAGAGATATAGAAT-GTTTGCAAAGCTTGAGTAGGAGAATCCACTTTGGCAAGTTTGTTGCTGAAGC90 PDC5TCAATCTCAAAGAGAACAACACAATACAA-TAACAAGAAGAACAAAGCTAATTAACATAAAACTCATGATTCAACGTTTGTGTATTTTTTT90 TYR1CCGTAGCACTTGAAGGAAAGAGGACAGCA-TATCCACTTGATAAACTGTTTTTAATGCTGATTTCCTATAATATTAGGCCTTCTTTTGTGA90 TKL1-EcoRI-FCGGAATTCATGACTCAATTCACTGACATTG30 TKL1-SalI-RGCGTCGACTTAGAAAGCTTTTTTCAAAGGAG31 PGAP-TKL1-FTATAGACAGAAAGAGTAAATCCAATAGAATAGAAAACCACATAAGGCAAGTCATTATCAATACTCGCCATTTC73 TKL1-TTEF1-RCACTCGAAAAAAAAAATTTCAGTGACTTAGCCGATAAATGAATGTGCTTGCATTTTTGCAAATTAAAGCCTTCGAGCG78基因缺失或插入的验证 ZWF1-v-FCCGACACATAACACCTAAAGTGGC24 ZWF1-v-RGCCAAGGAGGAAGCTTCTGAAG22 ARO10-v-FACCTTCGGTAAGTGTCGGTATG22 ARO 10- v-RAGTTCTTCAAGTGGTGAGCCGAAAGC26 ARO4-v-FCTCAACTGCACAGAGAATTGGCC23 ARO4-v-RGCCTTAGCTTCTGCAACGGACTTAG25 ARO7-v-FCTACCCACAAATTTTGGCGCC21 ARO7-v-RCCACACCATAGACTTCAGCCTTC23 PDC5-v-FCCTCATTGGCTTAGGCATAATCACCG26 PDC5-v-RCGTTTTTAGGAAGGACAGAGGTCC24 TYR1-v-FGCGGTGTAGAATTGCGTATTTATG24 TYR1-v-RGCATGTGATGATGACAGTATTCATC25 pY26-v-FCACCAGAACTTAGTTTCGACGG22 pY26-v-RCTTTTCGGTTAGAGCGGATGTG22
注:在质粒构建引物中,带下划线部分表示sgRNA N20序列;在供体DNA引物中,带下划线和框格部分分别表示ZWF1同源延伸和碱基突变;产生的限制性位点用粗体标记并在引物名称中表示
1.2.3 菌体生长和碳源代谢物的测定
用722s型分光光度计测定发酵样品的菌体浓度(OD600)。离心收集上清液样品,并保存在-20 ℃直至用于分析。用于碳源代谢(葡糖糖、甘油、乙醇)分析的样品在终体积分数为5%的三氯乙酸(TCA)中沉淀蛋白,4 ℃离心后通过HPLC进行分析。
HPLC条件:色谱柱为Lichrospher C18(250 mm×4.6 mm, 5 μm),示差检测器,流动相为5 mol/L H2SO4,流速为0.8 mL/min,柱温60 ℃,进样量20 μL。
1.2.4 芳香族氨基酸衍生物苯乙酸(PAA)和苯乙醇(PET)的测定
将乙腈添加到样品中至终体积分数为20%用于HPLC分析。色谱柱为Waters C18柱(250 mm×4.6 mm, 5 μm),紫外检测器检测波长为215 nm,以流动相A(8 mmol/L KH2PO4, pH 2.4)和流动相B(纯乙腈)按照参考文献[16]的方法进行梯度洗脱分析样品。
1.2.5 OPA柱前衍生法测定芳香族氨基酸
将体积分数10%TCA添加到样品中至终体积分数为5%用于HPLC分析。色谱柱为Thermo Fisher Hypersil ODS柱(250 mm×4.6 mm, 5 μm),柱温40 ℃,紫外检测器检测波长为338 nm,20 μL。流动相A(pH 7.2)∶60 mmol/L醋酸钠-三乙胺-四氢呋喃(体积比为500∶0.11∶2.5);流动相B(pH 7.2)∶60 mmol/L醋酸钠-甲醇-乙腈(体积比为1∶2∶2)。采用梯度洗脱,流速1.0 mL/min,洗脱程序为:0 min, 8% B; 17 min, 50% B; 20.1 min, 100% B; 24.0 min, 8% B。氨基酸以外标法定量。
利用引物ZWF1-sgRNA-F、sgRNA-R和sgRNA-F、ZWF1-sgRNA-R分别以质粒pYES2-gRNA-Hyg为模板构建质粒pYES2-sgRNA(ZWF1)-Hyg,用引物sgRNA-v-F和sgRNA-v-R进行菌落PCR验证(图3-A),得到大小为600 bp左右的条带,证明质粒pYES2-sgRNA(ZWF1)-Hyg构建成功。用同样的方法构建了质粒pYES2-sgRNA(ARO4)-Hyg、pYES2-sgRNA(ARO7)-Hyg、pYES2-sgRNA(ARO10)-Hyg、pYES2-sgRNA(PDC5)-Hyg、pYES2-sgRNA(TYR1)-Hyg并进行验证。
以酿酒酵母S288c染色体为DNA模板,用引物TKL1-EcoRI-F和TKL1-SalI-R扩增获得1条2.0 kb左右的片段,双酶切纯化后连接pY26-URA3载体,用引物pY26-v-F和 pY26-v-R进行菌落PCR(图3-B),得到大小为2 100 bp左右的条带,证明质粒pY26- URA3-TKL1构建成功。用引物PGAP-TKL1-F和 TKL1-TTEF1-R以构建的质粒pY26-URA3-TKL1为模板,扩增获得带有ZWF1上下游同源臂的TKL1序列,纯化后连接pMD19-T载体送至生工生物工程(上海)股份有限公司进行测序验证,得到无碱基突变的序列。
如图2所示,将质粒pYES2-sgRNA(ZWF1)-Hyg及线性化载体pMD19T-TKL1转化到含有质粒pHCas9-Nours的酿酒酵母S288c细胞,在诺尔丝菌素和潮霉素双抗性YPD平板上筛选转化子,用引物ZWF1-v-F和ZWF1-v-R进行菌落PCR验证(图3-C),得到大小为1 300 bp左右的条带,样品测序和核酸序列比对一致,证明基因ZWF1被成功敲除,并且在敲除位点成功整合TKL1表达盒。

图2 重组菌株构建流程图
Fig.2 The construction flow chart of the recombinant strains
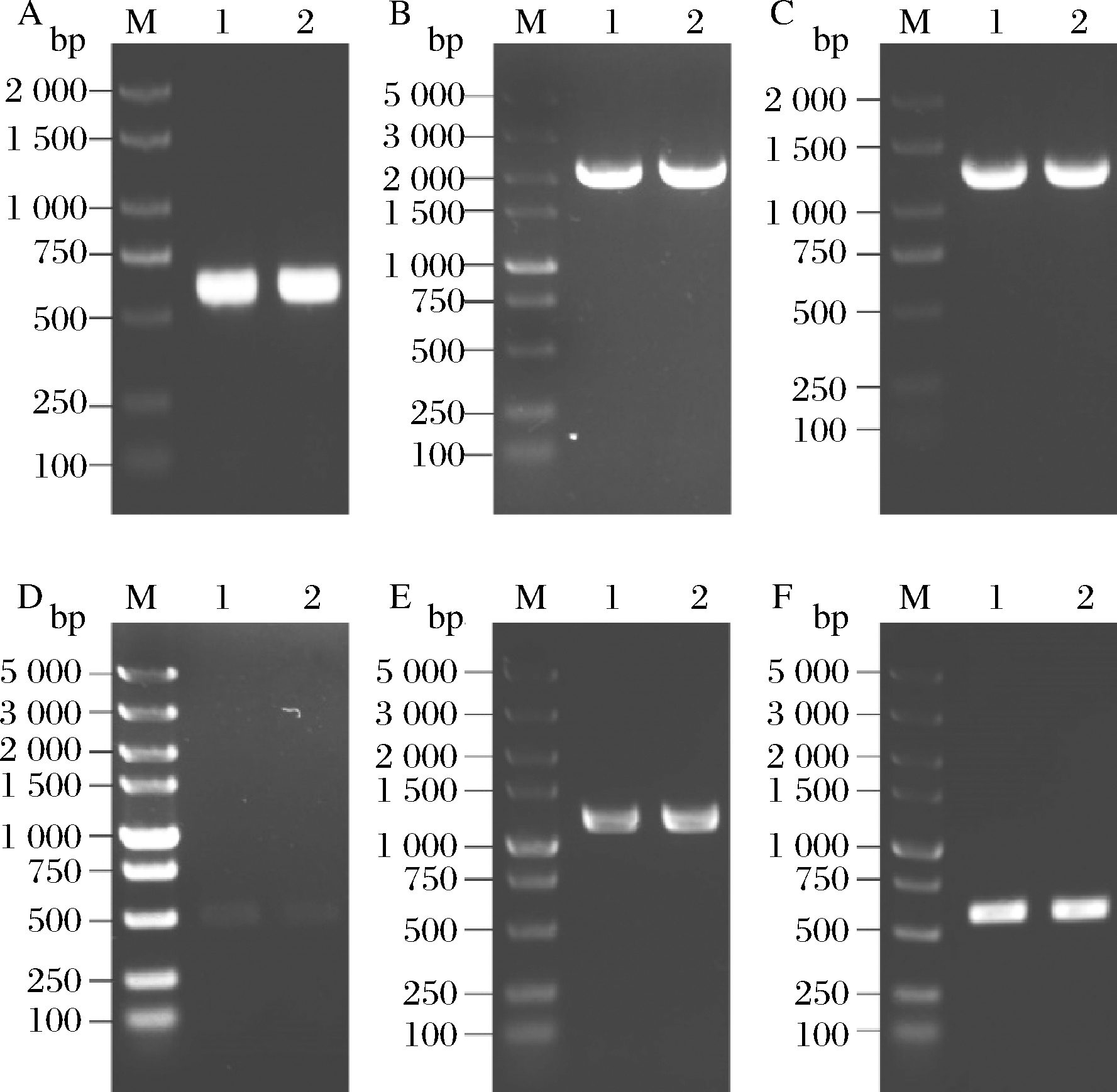
A-pYES2-sgRNA(ZWF1)-Hyg质粒的验证;B-pY26-URA3-TKL1质粒的验证;C-将TKL1插入ZWF1站点的验证;D-ARO10基因缺失
的验证;E-PDC5基因缺失的验证;F-TYR1基因缺失的验证
图3 菌落或基因组PCR验证琼脂糖凝胶电泳图
Fig.3 Agarose gel electrophoresis of colony and genome PCR
由此,成功构建菌株S288c(Δzwf1∶∶TKL1)。将质粒pYES2-sgRNA(ARO4)-Hyg和带有碱基位点突变的90 bp Donor DNA寡核苷酸片段导入菌株S288c(Δzwf1∶∶TKL1),以ARO4-v-F和ARO4-v-R为验证引物经PCR获得带突变位点的目的片段,经测序比对筛选得到菌株S288c(Δzwf1∶∶TKL1, Aro4K229L)。以同样的方法及相应的验证引物构建和验证菌株RPA、S288c(Δzwf1∶∶TKL1, Aro4K229L, Aro7G141S, Δaro10)、RPB及RPC (如表1所示)。菌落或基因组PCR验证如图3-D、3-E、3-F所示。
通过将TKL1整合在ZWF1位点,并定点突变ARO4和ARO7,得到菌株RPA。已有研究表明蛋氨酸补充对于ZWF1缺失的酿酒酵母生长是必需的,可能是蛋氨酸的生物合成需要戊糖磷酸途径释放的碳水化合物[17]。对菌株RPA和出发菌S288c进行了摇瓶发酵比较。如图4-A所示,对照菌S288c胞外L-苯丙氨酸净积累几乎为零,而在菌株RPA胞外检测到2.49 mg/L的L-苯丙氨酸;同时,菌株RPA胞外检测到6.53 mg/L的L-酪氨酸 (图4-B)。在苯乙醇/苯乙酸(PET/PAA)合成方面,菌株RPA胞外苯乙醇/苯乙酸最高产量为30.12 mg/L,而对照菌S288c基本维持本底质粒浓度1.87 mg/L(图4-C)。此外,菌株RPA的最高OD600为8.52,与对照菌S288c相比,降低了37%(图4-D、图4-E)。以上结果表明,中心代谢途径和莽草酸途径的代谢调控同时强化了L-苯丙氨酸和L-酪氨酸的合成;同时,代谢途径的改造影响了菌株生长和对碳源的利用速率(图4-D、图4-E),由于菌株的生长涉及一系列复杂的调控,其原因有待进一步探究。此外,加强L-苯丙氨酸合成带来了苯乙醇/苯乙酸积累的问题。

A-S288c和RPA中细胞外L-苯丙氨酸的产量;B-S288c和RPA中细胞外L-酪氨酸的产量;C-S288c和RPA中胞外PET/PAA的产量;D-S288c中碳源的消耗与菌体生长;E-RPA中碳源的消耗和RPA的增长
图4 重组菌株RPA的摇瓶发酵
Fig.4 Shake flask fermentation of the recombinant strain RPA
艾氏途径消耗了细胞合成的L-苯丙氨酸,其途径的脱羧反应为关键调控位点。研究表明,酿酒酵母脱羧酶基因涉及PDC1、PDC5、PDC6、ARO10[18]。PDC1、PDC5、PDC6基因编码3种丙酮酸脱羧酶(Pdc)同工酶。而在以L-苯丙氨酸为氮源的培养基中只有Aro10和Pdc5显示出苯丙酮酸脱羧酶活性,且Aro10 活性更高[13]。因此,在菌株RPA的基础上,敲除ARO10和PDC5,构建菌株RPB,可以减少艾氏途径对L-苯丙氨酸的利用。
为了考察ARO10和PDC5缺失对L-苯丙氨酸及其降解产物苯乙醇/苯乙酸积累的影响,发酵120 h的结果如图5-A所示。
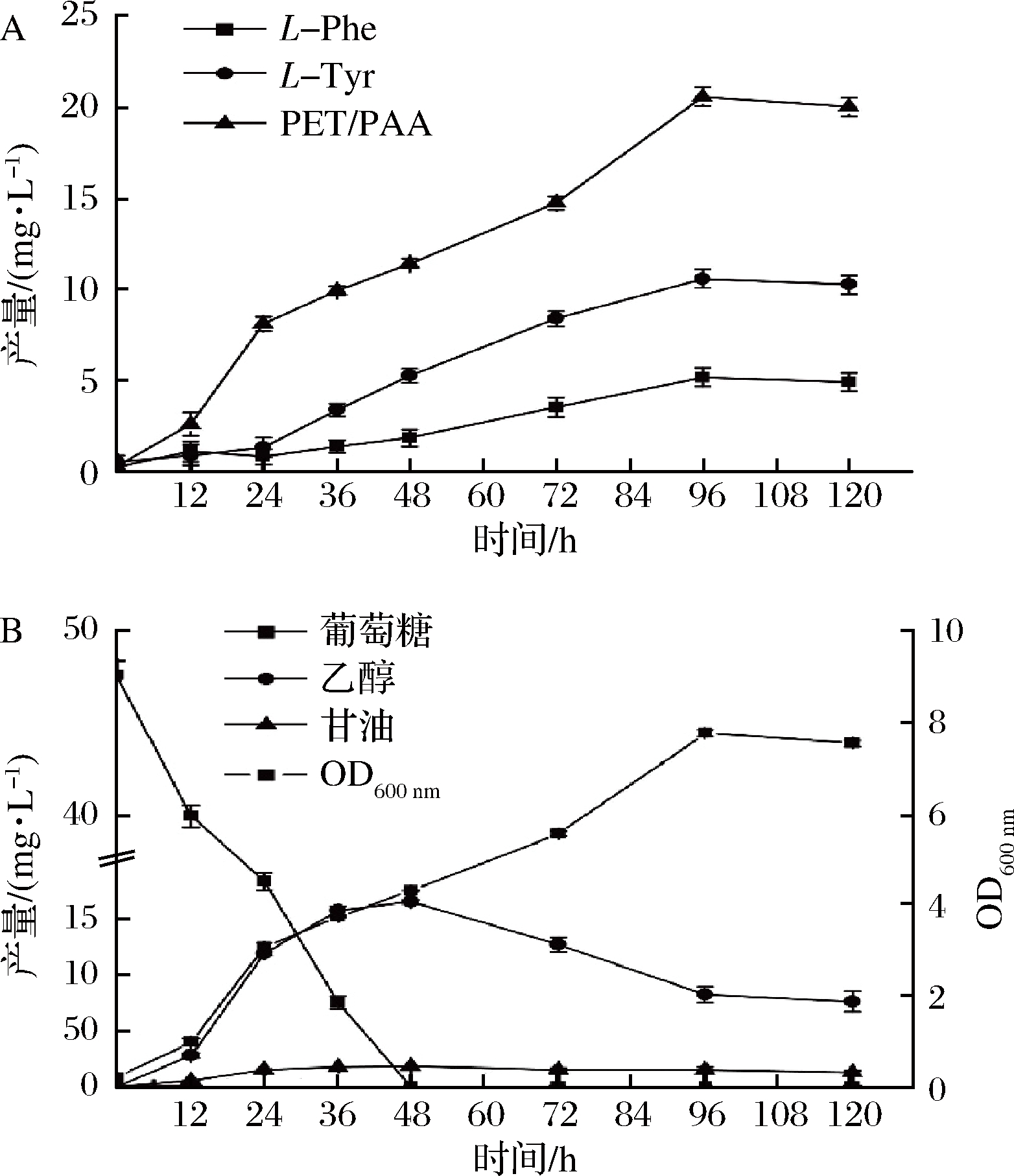
A-RPB中胞外芳香化合物的产量;B-RPB中碳源的消耗与RPB增长
图5 重组菌株RPB的摇瓶发酵
Fig.5 Shake flask fermentation of the recombinant strain RPB
菌株RPB胞外L-苯丙氨酸最高产量为5.16 mg/L,是菌株RPA的2.07倍,与此同时,胞外L-酪氨酸最高产量为10.58 mg/L,是菌株RPA的1.62倍;胞外苯乙醇/苯乙酸最高产量为20.53 mg/L,与菌株RPA相比,减少了31.83%。此外,菌株RPB最高OD600为7.75(图5-B),与菌株RPA相比,降低了9.03%。
以上结果表明,ARO10和PDC5的缺失可以提高L-苯丙氨酸和L-酪氨酸的积累,与已有的研究结果:缺失ARO10和PDC5增加芳香族氨基酸衍生物香豆酸的积累是一致的[19]。此外,虽然目前艾氏途径代谢机制已有研究[20],但各种酸脱羧酶的苯丙酮酸脱羧能力未得到充分的探究[13, 18],苯丙酮酸脱羧酶酶活力调控机制依然模糊,ARO10和PDC5的缺失未能完全阻断苯乙醇/苯乙酸的积累,具体机制仍需进一步研究。
由于L-苯丙氨酸和L-酪氨酸拥有共同前体预苯酸,L-苯丙氨酸合成途径的强化同样增强了L-酪氨酸的合成。此前,研究人员在大肠杆菌中敲除分支酸变位酶/预苯酸脱水酶(tyrA)基因阻断L-酪氨酸合成[21],从而抑制碳向竞争途径的流失[22]。因此,本实验在菌株RPB的基础上,敲除通向L-酪氨酸合成的关键酶——预苯酸脱氢酶基因TYR1[23],构建菌株RPC。
预苯酸脱氢酶Tyr1的缺失,导致菌株RPC L-酪氨酸营养缺陷。本实验在发酵培养基中添加100 mg/L的L-酪氨酸以回补L-酪氨酸营养缺陷进行摇瓶发酵,如图6-A所示。

A-RPB和RPC中细胞外L-苯丙氨酸的产量;B-RPB和RPC中细胞外L-酪氨酸的产量;C-RPB和RPC中细胞外PET/PAA的产量;D-RPB中碳源的消耗与RPB增长;E-RPC中碳源的消耗与菌体生长
图6 重组菌株RPC的摇瓶发酵
Fig.6 Shake flask fermentation of the Recombinant strain RPC
菌株RPC胞外L-苯丙氨酸最高产量为45.40 mg/L,是添加L-酪氨酸发酵的菌株RPB的6.94倍;0~24 h菌株L-酪氨酸消耗速率最高,且由于L-酪氨酸合成途径的阻断,菌株RPC对L-酪氨酸的需求更大,导致L-酪氨酸消耗速率高于RPB。因为菌株RPB L-酪氨酸合成途径并未阻断,所以最终菌株RPB L-酪氨酸残留量大于RPC(图6-B)。
菌株RPC胞外苯乙醇/苯乙酸的最高含量为50.26 mg/L(图6-C),是添加L-酪氨酸发酵的菌株RPB的2.06倍。此外,虽然菌株RPC补充了L-酪氨酸,但TYR1的缺失仍然对菌体生长造成一定影响,最高OD600为7.3,与添加L-酪氨酸发酵的菌株RPB相比,降低了11.83%(图6-D、图6-E)。以上结果表明,TYR1的缺失阻断了L-酪氨酸合成途径及以L-酪氨酸为前体物质的代谢途径(如蛋白质的合成)[24],从而使用于这些代谢途径的碳流向L-苯丙氨酸合成途径,极大促进了L-苯丙氨酸的积累及胞外分泌;同时L-苯丙氨酸的积累也促进了艾氏途径对它的消耗,苯乙醇/苯乙酸产量随之提高。
芳香族氨基酸的生物合成途径在大肠杆菌中已得到广泛的研究。大肠杆菌要消耗芳香族氨基酸合成前体物质PEP的50%参与磷酸转移酶系统用于葡萄糖的吸收[25],使得芳香族氨基酸的合成效率在30%以下。相比于大肠杆菌,酿酒酵母通过己糖转运酶(HXT)采用促进扩散的方式吸收葡萄糖[26],不需要消耗前体PEP,理论上可使芳香族氨基酸的转化率提高近1倍。然而,事实上目前尚无利用酿酒酵母大量积累芳香族氨基酸的先例。包括L-苯丙氨酸在内的芳香族氨基酸的合成途径是酿酒酵母细胞内最为复杂的代谢途径之一。以L-苯丙氨酸为例,其合成需要借助莽草酸途径,后经10步反应完成;与之相关的代谢囊括了糖酵解途径、戊糖磷酸途径、葡糖糖吸收和柠檬酸循环等。然而,已有的酿酒酵母代谢网络模型(Iff708, Ind750) in silico预测准确率不足85%[27-28],芳香族氨基酸代谢途径部分调控基因及调控机制仍十分模糊,无法合理高效指导代谢工程的研究和改造。因此,在后基因组时代对酿酒酵母芳香族氨基酸代谢网络的研究和注释显得尤其重要。
本研究以酿酒酵母模式菌株S288c为研究对象,利用CRISPR-Cas9基因编辑技术,通过代谢工程技术手段,对L-苯丙氨酸合成相关途径进行了初步探究,为酿酒酵母芳香族氨基酸代谢途径的分子调控图谱的绘制和细胞代谢途径及其遗传修饰的合理人工设计提供参考。首先,代谢改造中心代谢途径和莽草酸途径,获得能初步积累L-苯丙氨酸的菌株RPA;而后针对菌株RPA苯乙醇/苯乙酸的积累问题,弱化艾氏途径,减少L-苯丙氨酸胞内的消耗;最后,阻断L-酪氨酸合成途径,成功减少L-酪氨酸的分流,菌株RPC胞外L-苯丙氨酸最高含量为45.40 mg/L。此外,为了降低TYR1的缺失对菌体生长的影响,同时不影响L-苯丙氨酸合成分支的流量,可以参考大肠杆菌中降低分支酸变位酶/预苯酸脱水酶(TyrA)酶活力的思路[29],在酿酒酵母基因TYR1位置添加蛋白酶水解标签,增加预苯酸脱氢酶Tyr1对蛋白水解的敏感性,以此降低Tyr1的总体酶活力。本研究通过对L-苯丙氨酸合成相关途径的探究,增加了对酿酒酵母芳香族氨基酸代谢调控机制的认识,为后续研究酿酒酵母芳香族氨基酸代谢调控提供重要参考价值。
后续将通过深入挖掘酿酒酵母芳香族氨基酸合成及转运机制,从基因和转录水平上阐明芳香族氨基酸合成和转运途径的内在调控机制,以此绘制酿酒酵母芳香族氨基酸代谢途径的分子调控图谱,从而为代谢改造酿酒酵母进行芳香族氨基酸及其高价值衍生物的工业生产提供理论指导。
[1] DANIALI G, JINAP S, SANNY M, et al. Effect of amino acids and frequency of reuse frying oils at different temperature on acrylamide formation in palm olein and soy bean oils via modeling system[J]. Food Chem, 2018, 245: 1-6.
[2] SPRENGER G A. From scratch to value: engineering Escherichia coli wild type cells to the production of L-phenylalanine and other fine chemicals derived from chorismate[J]. Appl Microbiol Biotechnol, 2007, 75(4): 739-49.
[3] 韩建福, 富敏霞, 祝铃钰, 等. L-苯丙氨酸制备的研究进展[J]. 生物加工过程, 2019, 17(2): 117-124.
[4] GU P, YANG F, KANG J, et al. One-step of tryptophan attenuator inactivation and promoter swapping to improve the production of L-tryptophan in Escherichia coli[J]. Microb Cell Fact, 2012, 11: 30.
[5] GU P, SU T, WANG Q, et al. Tunable switch mediated shikimate biosynthesis in an engineered non-auxotrophic Escherichia coli[J]. Sci Rep, 2016, 6: 29 745.
[6] LIU S, ZHANG L, MAO J, te al. Metabolic engineering of Escherichia coli for the production of phenylpyruvate derivatives[J]. Metab Eng, 2015, 32: 55-65.
[7] TANJI Y, HATTORI K, SUZUKI K, et al. Spontaneous deletion of a 209-kilobase-pair fragment from the Escherichia coli genome occurs with acquisition of resistance to an assortment of infectious phages[J]. Appl Environ Microbiol, 2008, 74(14): 4 256-4 263.
[8] LIU Z L. Molecular mechanisms of yeast tolerance and in situ detoxification of lignocellulose hydrolysates[J]. Appl Microbiol Biotechnol, 2011, 90(3): 809-825.
[9] GUO Z, ZHANG L, DING Z, et al. Interruption of glycerol pathway in industrial alcoholic yeasts to improve the ethanol production[J]. Appl Microbiol Biotechnol, 2009, 82(2): 287-92.
[10] DEANER M, ALPER H S. Systematic testing of enzyme perturbation sensitivities via graded dCas9 modulation in Saccharomyces cerevisiae[J]. Metab Eng, 2017, 40: 14-22.
[11] LI M, KILDEGAARD K R, CHEN Y, et al. De novo production of resveratrol from glucose or ethanol by engineered Saccharomyces cerevisiae[J]. Metab Eng, 2015, 32: 1-11.
[12] LI M, SCHNEIDER K, KRISTENSEN M, et al. Engineering yeast for high-level production of stilbenoid antioxidants[J]. Sci Rep, 2016, 6: 36 827.
[13] ROMAGNOLI G, LUTTIK M A, KOTTER P, et al. Substrate specificity of thiamine pyrophosphate-dependent 2-oxo-acid decarboxylases in Saccharomyces cerevisiae[J]. Appl Environ Microbiol, 2012, 78(21): 7 538-7 548.
[14] MANNHAUPT G, STUCKA R, PILZ U, et al. Characterization of the prephenate dehydrogenase-encoding gene, TYR1, from Saccharomyces cerevisiae[J]. Gene, 1989, 85(2): 303-311.
[15] GIETZ R D, WOODS R A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method[J]. Method Enzymol, 2002, 350(152): 87-96.
[16] REIFENRATH M, BOLES E. Engineering of hydroxymandelate synthases and the aromatic amino acid pathway enables de novo biosynthesis of mandelic and 4-hydroxymandelic acid with Saccharomyces cerevisiae[J]. Metab Eng, 2018, 45: 246-254.
[17] THOMAS D, CHEREST H, SURDIN-KERJAN Y. Identification of the structural gene for glucose-6-phosphate dehydrogenase in yeast. Inactivation leads to a nutritional requirement for organic sulfur[J]. The EMBO Journal, 1991, 10(3): 547-553.
[18] VURALHAN Z, MORAIS M A, TAI S L, et al. Identification and characterization of phenylpyruvate decarboxylase genes in Saccharomyces cerevisiae[J]. Appl Environ Microbiol, 2003, 69(8): 4 534-4 541.
[19] RODRIGUEZ A, KILDEGAARD K R, LI M, et al. Establishment of a yeast platform strain for production of p-coumaric acid through metabolic engineering of aromatic amino acid biosynthesis[J]. Metab Eng, 2015, 31: 181-188.
[20] KIM B, CHO B R, HAHN J S. Metabolic engineering of Saccharomyces cerevisiae for the production of 2-phenylethanol via ehrlich pathway[J]. Biotechnol Bioeng, 2014, 111(1): 115-124.
[21] BAEZ-VIVEROS J L, FLORES N, JUAREZ K, et al. Metabolic transcription analysis of engineered Escherichia coli strains that overproduce L-phenylalanine[J]. Microb Cell Fact, 2007, 6: 30.
[22] TYAGI N, SAINI D, GULERIA R, et al. Designing an Escherichia coli strain for phenylalanine overproduction by metabolic engineering[J]. Mol Biotechnol, 2017, 59(4-5): 168-178.
[23] MAO J, LIU Q, SONG X, et al. Combinatorial analysis of enzymatic bottlenecks of L-tyrosine pathway by p-coumaric acid production in Saccharomyces cerevisiae[J]. Biotechnol Lett, 2017, 39(7): 977-982.
[24] BENDER D A. Amino Acid Metabolism[M]. 3nd ed. the United Kingdom: WILEY- BLACKWELL, 2012: 67-104.
[25] POSTMAl P W, LENGELER J W. Phosphoenolpyruvate carbohydrate phosphotransferase system of bacteria[J]. Microbiol Rev, 1985, 49(3): 232-269.
[26] DIDERICH J A, SCHEPPER M, VAN H P, et al. Glucose uptake kinetics and transcription of HXT genes in chemostat cultures of Saccharomyces cerevisiae[J]. J Biol Chem, 1999, 274(22): 15 350-15 359.
[27] SHOBAYASHI M, UKENA E, FUJII T, et al. Genome-wide expression profile of sake brewing yeast under shaking and static conditions[J]. Biosci Biotechnol Biochem, 2007, 71(2): 323-235.
[28] BELTRAN G, NOVO M, LEBERRE V, et al. Integration of transcriptomic and metabolic analyses for understanding the global responses of low-temperature winemaking fermentations[J]. FEMS Yeast Res, 2006, 6(8): 1 167-1 183.
[29] DOROSHENKO V G, SHAKULOV R S, KAZAKOVA S M, et al. Construction of an L-phenylalanine-producing tyrosine-prototrophic Escherichia coli strain using tyrA ssrA-like tagged alleles[J]. Biotechnol Lett, 2010, 32(8): 1 117-1 121.