郫县豆瓣是以红辣椒、蚕豆为主要原料,食用盐、小麦粉等为辅料,经过制曲、甜瓣子发酵、辣椒醅发酵及后发酵生香等阶段酿制而成的传统调味品[1]。郫县豆瓣红棕油亮、味辣香醇、酱香味浓郁,被誉为川菜之魂。郫县豆瓣生产缺乏专用菌种,目前大部分豆瓣酱生产企业采用酱油曲精制曲发酵,然而酱油发酵为熟料发酵,豆瓣酱为生料发酵,两者的发酵方式明显不同,采用酱油发酵用的菌种发酵豆瓣酱存在着制曲效果不佳,发酵速度慢等问题。因此,筛选豆瓣酱发酵专用米曲霉,开发一种豆瓣酱专用的曲精对行业的发展具有重要意义。
α-淀粉酶在酱类食品发酵过程中发挥着重要的作用,它与糖化酶共同作用分解淀粉产葡萄糖,不仅为酱类食品后期发酵的酵母菌及其他微生物提供碳源,还是醇类、有机酸类风味物质形成的前体物质,且能与豆瓣酱中的氨基酸发生美拉德反应,产生令人愉悦的香气成分[2-3]。董丹等[4]从郫县豆瓣中筛选鉴定得到1株芽孢杆菌,所产淀粉酶活力可达80.24 U/mL。周绍琴[5]以贵州传统豆瓣辣酱为研究对象,从中筛选出高酶活力菌株并探究该菌对产品后熟期的促熟性能,进而证明高酶活力菌株可缩短豆瓣发酵时间。因此,高产淀粉酶菌株的筛选及应用对提升豆瓣酱发酵速度,优化产品品质有重要意义。
米曲霉是目前豆瓣酱制曲中普遍应用的菌株,且产α-淀粉酶能力强。DEY等[6]优化米曲霉IFO-30103培养条件后产淀粉酶能力可达1 000 U/mg。SAHNOUN等[7]从一种甜酱中筛选出1株产淀粉酶的米曲霉,优化液态发酵条件后,培养液中α-淀粉酶酶活力可达1 220 U/mL[8]。然而关于郫县豆瓣高产α-淀粉酶米曲霉的筛选及在豆瓣酱发酵中的应用还鲜见报道。本研究以传统发酵郫县豆瓣酱原料筛选高产α-淀粉酶米曲霉,对高效产酶菌株进行鉴定。由于黄曲霉和米曲霉基因相似度很高,因此2者极容易混淆,仅通过ITS(internal transcribed spacer)序列测定很难将二者区分[9]。黄曲霉菌株是代谢黄曲霉毒素B1(aflatoxin B1,AFB1)的重要污染微生物,在食品领域受到极大的重视。由于鉴定方法不完善,有的菌株即使被鉴定为米曲霉,仍有可能会代谢产生黄曲霉毒素[10]。因此,对米曲霉应用菌株进行多相鉴定,彻底区分黄曲霉和米曲霉,菌株产黄曲霉毒素能力的评价对高产淀粉酶菌株的推广应用具有重要意义。本研究从传统的豆瓣曲中筛选α-淀粉酶活力较高的菌株,通过ITS测序,构建系统发育树分析,结合形态观察,菌株产AFB1基因扩增及产AFB1可能性测定对菌株进行多项鉴定,评价其推广应用的可行性。
以传统制曲的豆瓣曲、传统发酵的甜瓣子为菌株筛选原料。
PD培养基:马铃薯削皮,切块,称取200 g,添加500 mL蒸馏水,煮沸30 min,纱布过滤后取滤液;加入20 g葡萄糖,溶解后补加蒸馏水至1 000 mL,121 ℃灭菌20 min。
淀粉酶筛选培养基:可溶性淀粉10 g,蛋白胨2 g,酵母膏5 g,NaCl 5 g,K2HPO4 3 g,琼脂粉20 g,蒸馏水1 000 mL,121 ℃灭菌20 min。
黄豆汁固体培养基:将除杂后的黄豆浸泡4 h以上,煮沸5 h。用纱布过滤后取黄豆汁1 L(调整滤液至5 Bé),20 g可溶性淀粉,1 g KH2PO4,0.5 g MgSO4,0.5 g (NH4)2SO4,20 g琼脂,121 ℃灭菌20 min。
麸皮固体培养基:m(麸皮)∶m(黄豆粉)∶m(水)=4∶1∶4混合,浸润30 min,拌匀。用1 L锥形瓶分装,60 g/瓶,121 ℃灭菌20 min。
产毒培养基(AFPA):蛋白胨 10 g,酵母粉 20 g,柠檬酸铁 0.5 g,四氯醌 0.002 g,氯霉素15 g,琼脂 15 g,蒸馏水1 000 mL,121 ℃灭菌20 min。
可溶性淀粉、KI、I2,成都金山化学试剂有限公司;Na2HPO4、NaH2PO4和甲醛(分析纯),成都市科隆化学品有限公司;H2SO4,四川西陇化工有限公司;Ezup柱式真菌基因组DNA抽提试剂盒,Taq PCR Master Mix,生工生物工程股份有限公司;乳酸酚棉蓝染液,北京索莱宝科技有限公司。黄曲霉毒素检测试剂盒,深圳芬德生物技术有限公司。
752G紫外可见分光光度计,上海仪电分析仪器股份有限公司;DYY-6D电泳仪,北京六一生物科技有限公司;BCD-201MLT冰箱,合肥美菱股份有限公司;H2050R-1离心机,长沙湘仪离心机仪器有限公司;PHSJ-4型pH计,上海仪电分析仪器股份有限公司;T960A智能梯度PCR仪,杭州晶格科学仪器有限公司; 霉菌培养箱,BMJ-250C、Multiskan FC型酶标仪,美国Thermo赛默飞世尔;LDZF-75KB型立体压力蒸汽灭菌器,上海申安医疗器械厂。
2.1.1 初筛
取25 g样品,梯度稀释,涂布于孟加拉红平板,28 ℃培养48 h;挑取菌株划线于PDA培养基进行纯化。将分离纯化后的菌株点植于淀粉酶筛选平板,28 ℃培养72 h,喷洒稀碘液于菌落表面,观察培养基透明圈。用游标卡尺测量菌落直径和透明圈直径,根据菌落直径与透明圈直径的比值初步筛选出产α-淀粉酶能力强的菌株,划线于PDA斜面,28 ℃培养72 h,4 ℃保存。
2.1.2 复筛
将初筛得到产淀粉酶能力强的菌株接种于麸皮培养基,28 ℃培养16 h后,将其摇散继续培养;24 h后进行第2次摇瓶,36 h时曲料结为较紧实的饼状,此时应扣瓶培养至72 h,测定麸皮曲精中α-淀粉酶活力。
α-淀粉酶活力的测定参考徐欢欢[11]的测定方法。淀粉酶活力单位定义为:40 ℃,pH 6.0条件下5 min内水解1 mg淀粉的酶量,以U/g表示。
粗酶液制备:称取麸皮曲精10 g,加入200 mL磷酸缓冲溶液(pH 6.0),40 ℃水浴浸提1 h,间歇搅拌,过滤备用。
测定方法:移取5 mL可溶性淀粉溶液,在40 ℃水浴中预热10 min,加入0.5 mL粗酶液,40 ℃水浴振荡,准确反应5 min,加入5 mL 0.1 mol/L H2SO4终止反应;取0.5 mL反应液与5 mL碘液显色,将反应液加入酶标仪的多孔板中测定OD620 nm值。以0.5 mL蒸馏水替代0.5 mL反应液为空白组,相同体积的缓冲液代替酶液反应为对照组。淀粉酶活力根据公式(1)计算:
淀粉酶活力![]()
(1)
式中:R0,对照组与碘液反应的吸光值;R,反应液与碘液反应的吸光值;D,粗酶液的稀释倍数。
2.2.1 菌株形态学观察
菌落形态:将所选菌株点殖于黄豆汁培养基,28 ℃培养72 h后观察菌株形态。
显微形态:挑取菌株的孢子及菌丝采用乳酸酚棉蓝染液进行染色制片,采用250VX2双目生物显微镜观察菌丝、顶囊及孢子着陆形态。
2.2.2 分子生物学鉴定
将菌株接种于黄豆汁固体培养基中,28 ℃培养72 h。挑取菌丝,采用液氮研磨后,用Ezup柱式真菌基因组DNA抽提试剂盒提取霉菌DNA。以此DNA为模板,以ITS1、ITS4为引物扩增霉菌ITS片段,聚合酶链反应(polymerase chainreaction, PCR)体系为30 μL:ddH2O 12.5 μL、上下引物各1 μL、DNA模板3 μL、Mix酶12.5 μL。扩增反应条件为94 ℃预变性保持4 min;94 ℃变性1 min、60 ℃退火45 s、72 ℃延伸90 s,30次循环;72 ℃延伸10 min。扩增样品送至北京擎科新业生物技术有限公司测序。测序得到的序列通过BLAST比对,采用Mega 5.0建立系统发育树对菌株进行鉴定。
2.2.3 产毒培养基(AFPA)检测方法
将菌株点植于AFPA固体培养基上,28 ℃ 培养2~3 d,观察AFPA培养基上的特征并记录。
2.2.4 产毒基因检测方法
选取4个黄曲霉毒素合成的关键基因进行PCR扩增检测,四者在黄曲霉毒素合成通路中都是必须的,包括调控aflO, aflP和aflQ, tub1 [12],扩增引物序列如表1所示。
表1 AFB1 关键基因扩增引物
Table 1 Amplification primer AFB1 key gene

引物名称编码基因引物序列(5’—3’)PCR产物/bp参考文献mtB (F)-FomtB (F)-RaflOGCCTTGACATGGAAACCATCCCAAGATGGCCTGCTCTTTA1 340SCHERM等[13]omt1-Fomt1-RaflPGCCTTGCAAACACACTTTCAAGTTGTTGAACGCCCCAGT1 440SCHERM等[13]ord-gFord-gRaflQTTAAGGCAGCGGAATACAAGGACGCCCAAAGCCGAACACAAA719SWEENEY等[14]tub1-Ftub1-Rtub1GCTTTCTGGCAAACCATCTCGGTCGTTCATGTTGCTCTCA1 406SCHERM等[13]
aflO、 aflP和aflQ、tub1 PCR扩增条件:94 ℃ 预变性4 min; 94 ℃变性1 min, 60 ℃退火1 min,72 ℃延伸1 min; 5个循环;94 ℃变性1 min,55 ℃ 退火1 min,72 ℃延伸1 min,30个循环,72 ℃延伸6 min。
2.2.5 菌株产黄曲霉毒素能力测定
接种4株菌株于PD液体培养基,28 ℃培养180 h,采用ELISA试剂盒测定发酵液中AFB1含量。具体检测方法如下:
(1)取2 mL发酵液,加入2 mL蒸馏水,7 mL甲醇,振荡5 min,4 000 r/min离心10 min,取上清液进行分析。
(2)取出适量微孔板,加入50 μL标准品/样本。
(3)加入AFB1酶标物50 μL /孔,同时加入抗体试剂50 μL /孔。
(4)盖板膜盖板后,至25 ℃避光环境中反应30 min。
(5)反应完成后将液体甩干,接着用洗涤液300 μL/孔充分洗涤4~5次。
(6)加入底物A液50 μL /孔,并加入底物B液50 μL /孔。
(7)盖板膜盖板后,至25 ℃避光环境中反应15 min。
(8)加入终止液50 μL /孔后用450的酶标仪测定每孔OD值。
采用excel进行数据统计,实验结果中的数据均为3个平行样品均值,相对标准偏差在表和图中分别以数值和误差棒表示,采用origin 9.1作图分析。采用SPASS进行邓肯分析,置信区间为95%。
筛选得到70株霉菌,于淀粉酶筛选培养基培养,其中35株霉菌能在淀粉酶筛选平板产生透明圈,分别测量这35株菌的透明圈直径d及菌落直径D,计算比值,结果如表2所示。菌株30M-1的d/D值最大,达到3.128。其次为BM-2、BM-5、25M-1、14、DM2、16、19、DM1、18M-1、24,d/D值都大于2.000。
表2 菌株菌落直径与透明圈直径测定结果
Table 2 Results of bacterial colony diameter and transparent circle diameter

霉菌编号D/cmd/cmd/D霉菌编号D/cmd/cmd/DMT-30.636±0.0020.962±0.0101.513±0.011BM-50.434±0.0071.080±90.0082.488±0.026MT-20.670±0.0090.992±0.0091.480±0.01224M-10.500±0.0080.642±0.0121.284±0.018CM-20.900±0.0021.310±0.0121.456±0.020DM-90.824±0.0071.080±0.0221.311±0.01422M-10.454±0.0050.844±0.0081.859±0.020BM-70.910±0.0071.180±0.0121.296±0.007160.430±0.0051.010±0.0042.349±0.021DM-121.034±0.0151.326±0.0171.282±0.012BM-50.610±0.0111.246±0.0092.043±0.01821M-20.952±0.0141.136±0.0161.193±0.01318M-10.374±0.0080.754±0.0142.016±0.011IM-50.810±0.0021.190±0.0181.469±0.003MT-70.672±0.0120.844±0.0091.256±0.004DM-31.286±0.0021.472±0.0191.145±0.016190.500±0.0041.108±0.0042.216±0.00841M-11.088±0.0041.314±0.0121.208±0.01130M-10.470±0.0141.470±0.0203.128±0.011MT-50.528±0.0080.962±0.011.822±0.090DM20.452±0.0041.080±0.0182.389±0.017MT-1'0.626±0.0180.932±0.0091.489±0.001CM-10.688±0.0091.258±0.0141.828±0.004MT-10.636±0.0071.060±0.0121.667±0.002240.324±0.0150.654±0.0082.019±0.014MT-80.594±0.0220.790±0.0081.330±0.015AM-10.720±0.0130.910±0.0171.264±0.01540.902±0.0131.138±0.0201.262±0.00225M-60.818±0.0161.088±0.0011.330±0.01024M-60.440±0.0100.634±0.0121.441±0.028BM-20.432±0.0071.102±0.0002.551±0.019140.410±0.0171.012±0.0092.468±0.022AM-60.850±0.0091.154±0.0011.358±0.00321M-10.540±0.0170.854±0.0081.581±0.014BM-50.434±0.0061.080±0.0052.488±0.005DM-90.824±0.0091.080±0.0061.311±0.009FN0.420±0.0071.000±0.0102.381±0.000BM-70.910±0.0091.180±0.0121.296±0.01224M-10.500±0.0120.642±0.0051.284±0.014
对上述35株霉菌于麸皮培养基培养,得到麸曲后测定其α-淀粉酶活力,测定结果如表3所示。供试菌株中,曲霉30M-1为产α-淀粉酶最高的菌株,所产淀粉酶活力高达3 496.915 U/g。此外,菌株DM1、16、DM2、19、14产淀粉酶活力也可达到2 000 U/g以上。筛选菌株淀粉酶活力强,远高于目前已报道的菌株,选取产α-淀粉酶活力最强的30M-1、FN、DM2、16四株霉菌进行鉴定研究,为其推广应用奠定基础。
表3 α-淀粉酶活力测定结果
Table 3 Results of alpha amylase activity determination
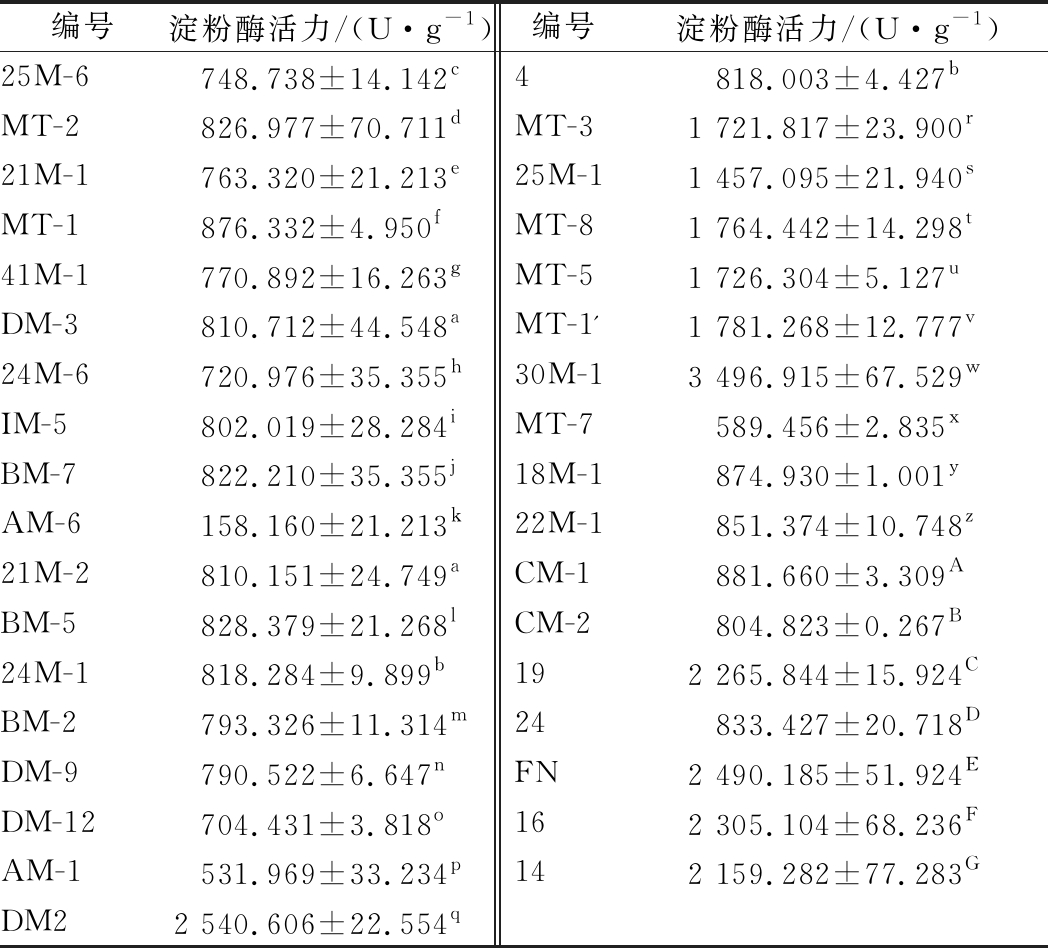
编号淀粉酶活力/(U·g-1)编号淀粉酶活力/(U·g-1)25M-6748.738±14.142c4818.003±4.427bMT-2826.977±70.711dMT-31 721.817±23.900r21M-1763.320±21.213e25M-11 457.095±21.940sMT-1876.332±4.950fMT-81 764.442±14.298t41M-1770.892±16.263gMT-51 726.304±5.127uDM-3810.712±44.548aMT-1'1 781.268±12.777v24M-6720.976±35.355h30M-13 496.915±67.529wIM-5802.019±28.284iMT-7589.456±2.835xBM-7822.210±35.355j18M-1874.930±1.001yAM-6158.160±21.213k22M-1851.374±10.748z21M-2810.151±24.749aCM-1881.660±3.309ABM-5828.379±21.268lCM-2804.823±0.267B24M-1818.284±9.899b192 265.844±15.924CBM-2793.326±11.314m24833.427±20.718DDM-9790.522±6.647nFN2 490.185±51.924EDM-12704.431±3.818o162 305.104±68.236FAM-1531.969±33.234p142 159.282±77.283GDM22 540.606±22.554q
注:t检验P<0.05表示样品间差异显著,不同字母代表差异显著
3.3.1 菌株形态学观察结果
4株菌于28 ℃培养96 h后,菌株形态相似,如图1所示。菌株16菌落呈黄绿色,质地呈丝绒状,中央凸起;菌株30M-1呈现黄绿色,表面平坦;菌株DM2为黄绿色,颜色较深,中央凸起,表面具有放射状褶皱;菌株FN为黄绿色,菌丝较短,疏松。
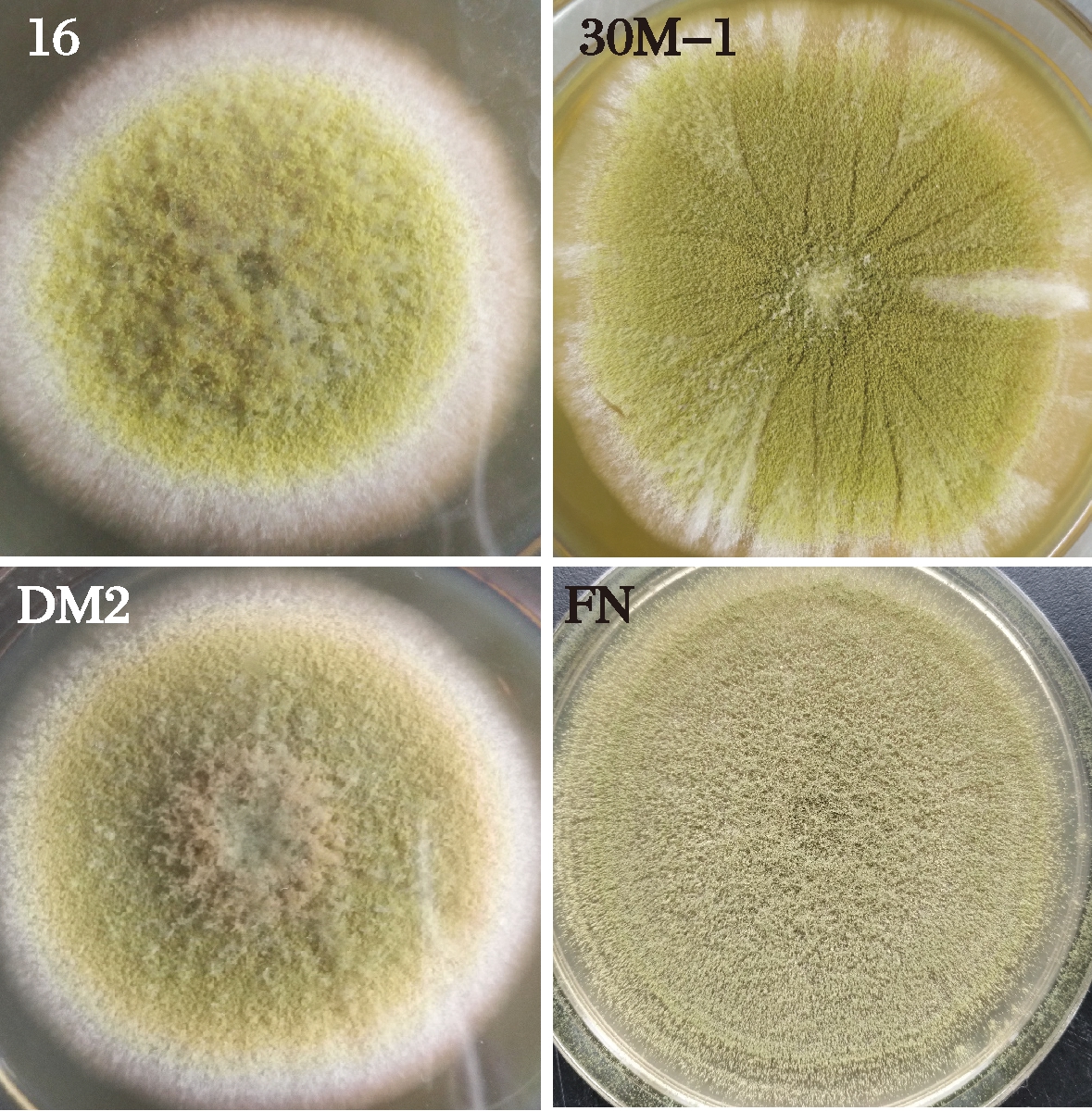
图1 菌株形态特征
Fig.1 Morphological characteristics of strains
显微镜形态观察,如图2所示,所选4株菌的孢梗茎壁薄且长,壁平滑,其分生孢子为圆形。顶囊呈球形,由顶囊的小梗产生孢子,分生孢子头辐射状,未发现菌核的形成。结合白飞荣等[15-16]对米曲霉的鉴定研究结果,初步判断筛选出的4株菌均为米曲霉。
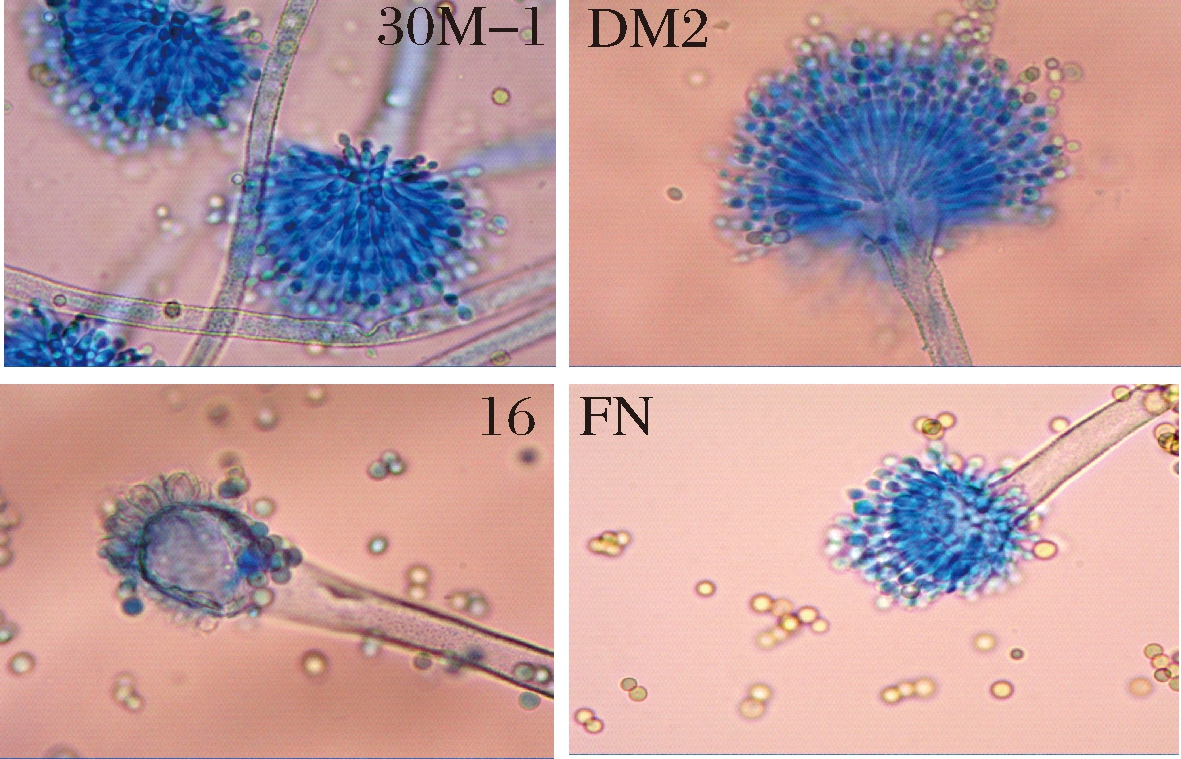
图2 显微产孢结构
Fig.2 Microscopic characteristics of conidia
3.3.2 菌株分子学鉴定结果
测序结果提交NCBI进行BLAST序列同源性比对,采用MEGA 5.0利用邻近法构建进化树,其结果如图3所示。4株菌株ITS序列相似度高,聚为一类;4株菌株均能与米曲霉,黄曲霉模式菌株聚为一类,说明和二者的相似度均较高。

图3 试验菌株系统进化树
Fig.3 Phylogenetic tree of experimental strains
3.3.3 产毒培养基(AFPA)检测结果
PITT等[17]研究发现,产毒的黄曲霉、寄生曲霉在AFPA培养基上,菌落反面呈明显橙黄色,而不产毒的米曲霉AFPA培养基上菌落反面则呈黄褐色或浅黄色。将验菌株三点接种于AFPA固体培养基上培养3 d后,菌株背面形态如图4所示。
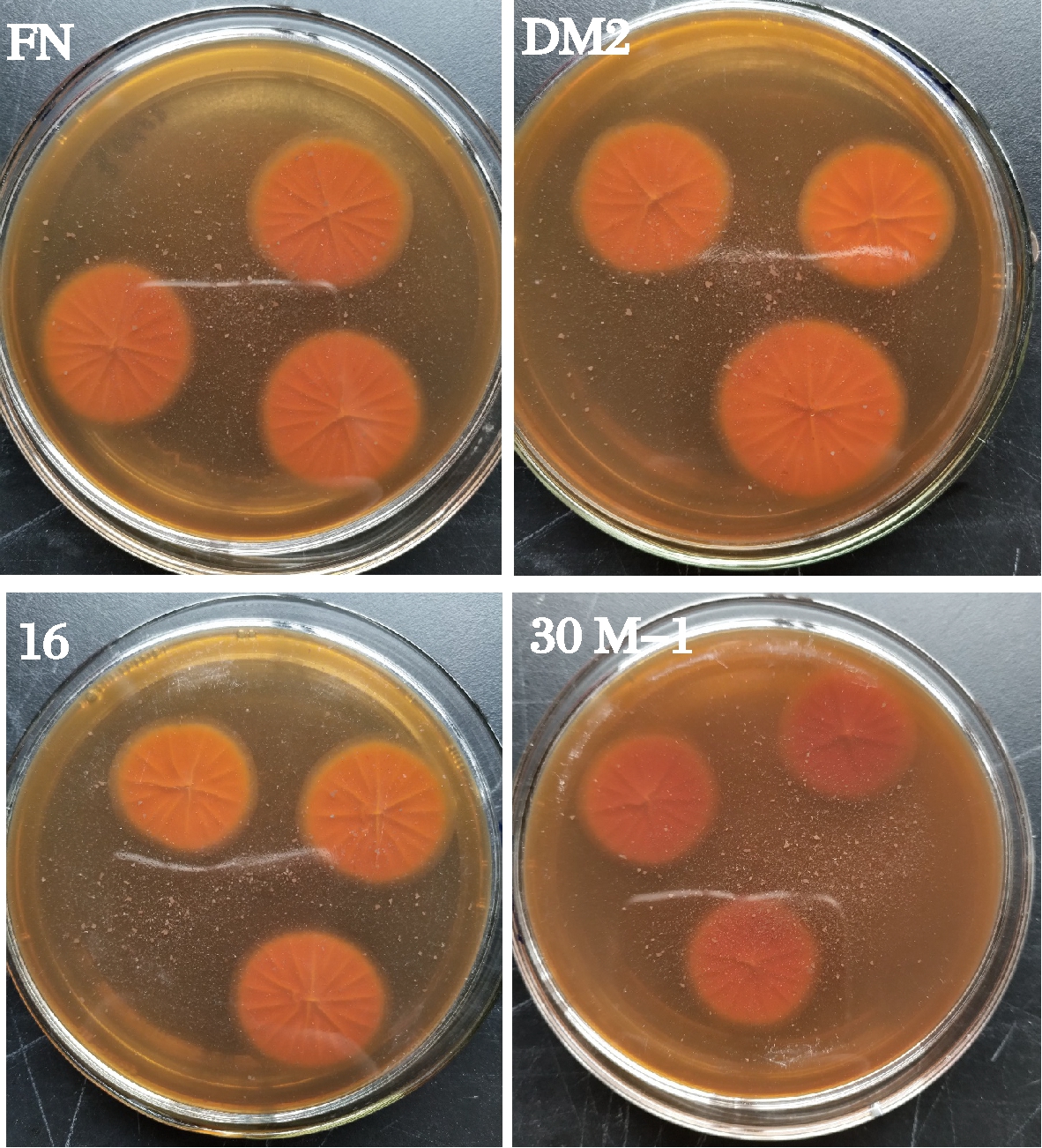
图4 菌株AFPA培养特征
Fig.4 Strain’s characteristic on AFPA
4株菌株于AFPA培养基培养3 d后菌落反面为黄褐色,说明菌株不产黄曲霉毒素。这与白飞荣等[15]研究米曲霉模式菌株在AFPA上培养的结果一致。
3.3.4 产AFB1关键基因
4种关键基因扩增结果如图5所示。4株菌株均扩增出部分产AFB1的关键基因。菌株30M-1为模板未扩增出Aflp 基因,而菌株16、FN、DM2扩增出了试验的4种基因。黄曲霉毒素合成基因簇包含 25 个基因,长度大约 70 kb[18]。部分米曲霉菌株含有产毒基因,但基因发生突变、缺失或碱基替换,不能正常表达。黄曲霉菌株含有产毒基因,大部分正常表达,产黄曲霉毒素;部分菌株因产毒基因发生突变,不能正常表达产生黄曲霉毒素[16,19]。菌株是否产毒需要进一步进行产毒试验验证。
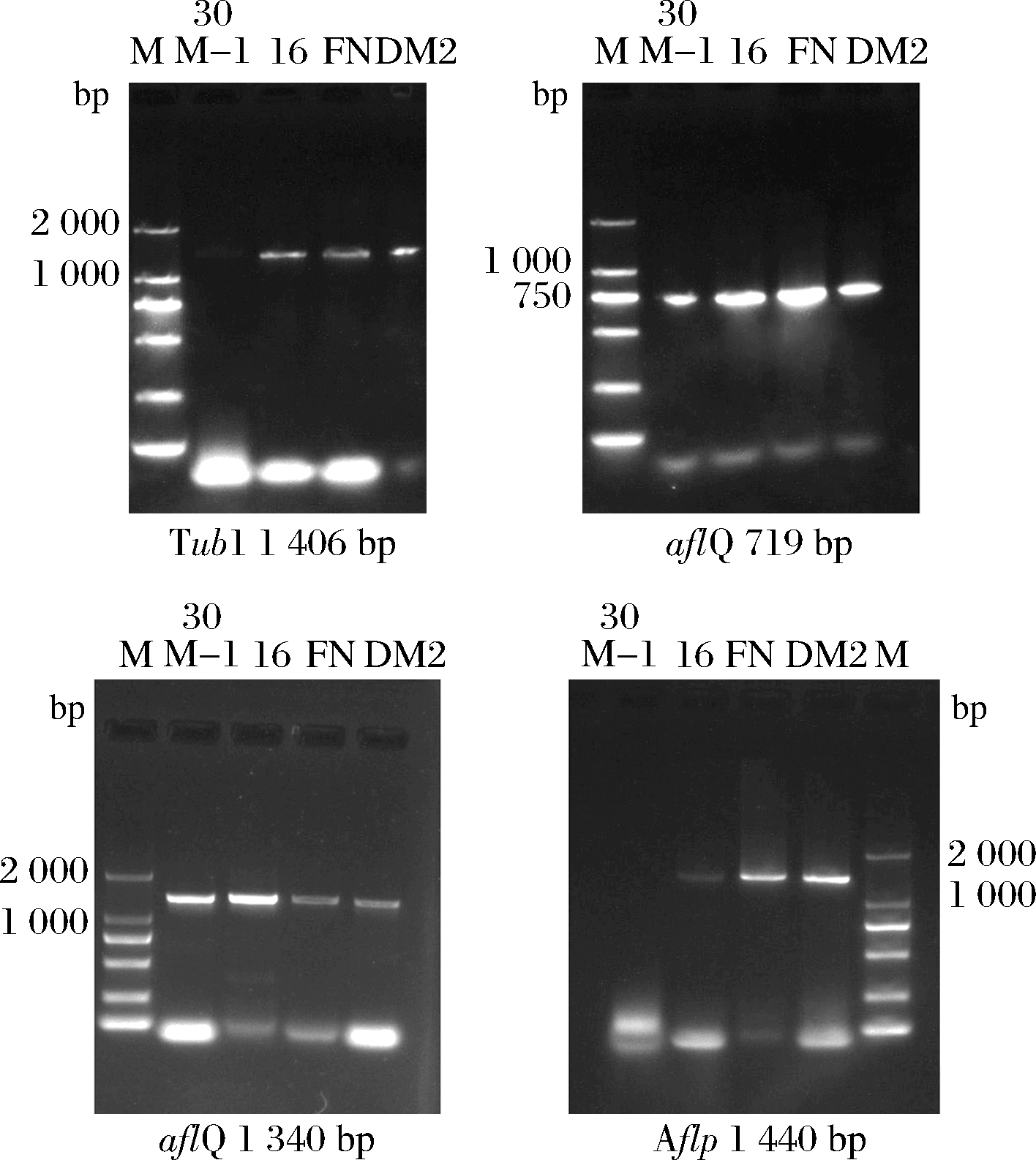
图5 产AFB1关键基因扩增结果
Fig.5 PCR amplicons of AFB1 genes
3.3.5 菌株培养液AFB1检测结果
菌株DM2、FN、16、30 M-1发酵液中未检出AFB1,因此没有黄曲霉毒素污染风险。
本试验中虽然试验的大部分AFB1关键基因均在检测的4株菌株DNA中扩增出,但菌株发酵液中AFB1检测结果显示,4株菌均不产AFB1,说明基因无法正常表达。
米曲霉、酱油曲霉等有益霉菌由于高产淀粉酶、蛋白酶等特性被广泛应用于酱油,大酱等产品的发酵[20]。淀粉酶分解淀粉类物质为小分子物质,对后期的发酵及风味形成具有重要意义。本文筛选的霉菌产淀粉酶能力强,其中菌株30M-1发酵麸皮曲中α-淀粉酶活力为3 496.915 U/g,FN、DM2、16发酵麸皮曲中α-淀粉酶活力分别为2 490.185、2 540.606和2 305.104 U/g。菌株于麸皮培养基中培养均生长良好,产生淀粉酶的能力高于目前报道的菌株。且菌株从传统发酵的豆瓣曲及甜瓣子中筛选得到,适合于豆瓣酱发酵环境中生长,有应用于豆瓣酱发酵的潜力。
目前许多米曲霉(或者类似米曲霉的菌株)仅通过简单的鉴定,未通过安全性评价就应用于食品的发酵。然而米曲霉和黄曲霉菌株在分类学上非常接近,仅通过简单的ITS测序或者形态观测很难将二者进行区分,二者极容易被混淆,所以在应用过程中很容易爆发黄曲霉毒素的污染,导致发酵食品中黄曲霉毒素检出的情况[21-22]。因此,筛选发酵应用的菌株,对菌株进行彻底地鉴定及安全性评价是必须的。CHRISTENSE[23]提出,米曲霉是野生黄曲霉种的驯化种,CHANG等[19]认为米曲霉是黄曲霉的形态学变种,二者同属于曲霉属黄绿组,形态相近、基因组高度相似[24],因此曲霉黄绿组菌株的鉴定需要结合基因测序,形态鉴定及产毒能力进行综合分析。
本文筛选的4株高产淀粉酶的霉菌,通过菌落形态及显微形态鉴定菌株为米曲霉;通过ITS测序构建系统发育树结果显示菌株和米曲霉和黄曲霉相似度高,均能聚为一类。综合菌株产毒能力测定结果分析显示,本文筛选的4株高产淀粉酶的菌株与米曲霉同源性近,不产AFB1。
本研究筛选得到4株高产α-淀粉酶的霉菌,固态发酵麸皮培养基中α-淀粉酶活力均大于2 000 U/g,其中菌株30M-1固态发酵麸皮培养基中α-淀粉酶活达到3 496.915 U/g。4株菌在PDA培养基中菌落均为黄绿色,显微结构与米曲霉结构相似,无菌核产生。AFPA产毒培养基中培养背面为橙黄色,无AFB1产生。ITS测序构建进化树均聚为一类,与黄曲霉和米曲霉模式菌株相似度大于99%。4株菌均能扩增出部分产AFB1关键基因,但发酵液中未检出AFB1,说明基因不能正常表达。多相鉴定结果显示筛选的4株高产淀粉酶的霉菌与米曲霉更相似,没有造成AFB1污染的风险,可以作为豆瓣酱发酵的备用菌株。
[1] 邓维琴,陈功,张其圣,等.传统郫县豆瓣酱中高产蛋白酶曲霉的筛选及应用[J].微生物学通报,2019,46(9):2 272-2 281.
[2] XU N, LIU Y, HU Y, et al. Autolysis of Aspergillus oryzae mycelium and effect on volatile flavor compounds of soy sauce[J]. Journal of Food Science, 2016, 81(8): 1 883-1 890.
[3] LIU S. Soy Sauce Fermentation[M]. Fermented Foods, Part II. CRC Press, 2017: 135-149.
[4] 董丹,陈燕,关统伟, 等.豆瓣发酵瓣子中淀粉酶高产菌株的筛选及其酶活力的测定[J].中国酿造, 2015, 34(2): 68-71.
[5] 周绍琴.传统豆瓣辣酱高酶活菌株筛选及促熟应用研究[D].贵州:贵州大学,2016.
[6] DEY T B, BANERJEE R. Purification, biochemical characterization and application of α-amylase produced by Aspergillus oryzae IFO-30103[J]. Biocatalysis and Agricultural Biotechnology, 2015, 4(1): 83-90.
[7] SAHNOUN M, BEJAR S, SAYARI A, et al. Production purification and characterization of two α-amylase isoforms from a newly isolated Aspergillus oryzae strain S2[J]. Process Biochem,2011,47: 18-25.
[8] NAILI B, SAHNOUN M, BEJAR S, et al. Optimization of submerged Aspergillus oryzae S2 α-amylase production[J]. Food Science and Biotechnology, 2016, 25(1): 185-192.
[9] LEE J H, JO E H, HONG E J, et al. Safety evaluation of filamentous fungi isolated from industrial doenjang koji[J]. Microbiol Biotechnol, 2014, 24(10): 1 397-1 404.
[10] GODET M, MUNAUT F. Molecular strategy for identification in Aspergillus section Flavi[J]. FEMS Microbiol Lett,2010, 304: 157-168.
[11] 徐欢欢. α-淀粉酶在酱油制曲与发酵工艺中的应用研究[D]. 广州:华南理工大学, 2012.
[12] JAMALI M, KARIMIPOUR M, SHAMS-GHAHFAROKHI M, et al. Expression of aflatoxin genes aflO (omtB) and aflQ (ordA) differentiates levels of aflatoxin production by Aspergillus flavus strains from soils of pistachio orchards[J]. Research in Microbiology, 2013, 164(4): 293-299.
[13] SCHERM B, PALOMBA M, SERRA D, et al. Detection of transcripts of the aflatoxin genes aflD, aflO, and aflP by reverse transcription polymerase chain reaction allows differentiation of aflatoxin-producing isolates of Aspergillus flavus and Aspergillus parasiticus[J]. Int J Food Microbiol, 2005, 98: 201-210.
[14] SWEENEY M J, PAMIES P, DOBSON A D W. The use of reverse transcription polymerase chain reaction (RT-PCR) for monitoring aflatoxin production in Aspergillus parasiticus 439[J]. Int J Food Microbiol, 2000, 56:97-103.
[15] 白飞荣,姚粟,凌空,等.黄曲霉和米曲霉的多相鉴定方法[J].微生物学通报,2018,45(01):215-226.
[16] LEE J H, JO E H, HONG E J, et al. Safety evaluation of filamentous fungi isolated from industrial doenjang koji[J]. J Microbiol Biotechnol, 2014, 24(10): 1 397-1 404.
[17] PITT J, HOCHING A D. Fungi and Food Spoilage[M]. 3rd Edition. Dordrecht/Heidelberg: Springer, 2009:305-310.
[18] YU J J, CHANG P K, EHRILICH K C, et al. Clustered pathway genes in aflatoxin biosynthesis[J]. Applied and Environmental Microbiology, 2004, 70(3): 1 253-1 262.
[19] CHANG P K, EHRILICH K C. What does genetic diversity of Aspergillus flavus tell us about Aspergillus oryzae?[J]. International Journal of Food Microbiology, 2010, 138(3): 189-199.
[20] HONG S B, KIM D H, SAMSON R A. Aspergillus associated with Meju, a fermented soybean starting material for traditional soy sauce and soybean paste in Korea[J]. Mycobiology, 2015, 43(3): 218-224.
[21] JUNG YJ, CHUNG SH, LEE HK, et al. Isolation and identification of fungi from a meju contaminated with aflatoxins [J]. J Microbiol Biotechnol, 2012, 22: 1 740-1 748.
[22] IQBAL S Z, ASI M R, MEHMOOD Z, et al. Survey of aflatoxins and ochratoxin A in retail market chilies and chili sauce samples[J]. Food Control, 2017, 81: 218-223.
[23] CHRISTENSEN M. A synoptic key and evaluation of species in the Aspergillus flavus group[J]. Mycologia, 1981, 73(6): 1 056-1 084.
[24] MACHIDA M, ASAI K, SNNO M, et al. Genome sequencing and analysis of Aspergillus oryzae[J]. Nature, 2005, 438(7 071): 1 157-1 161.