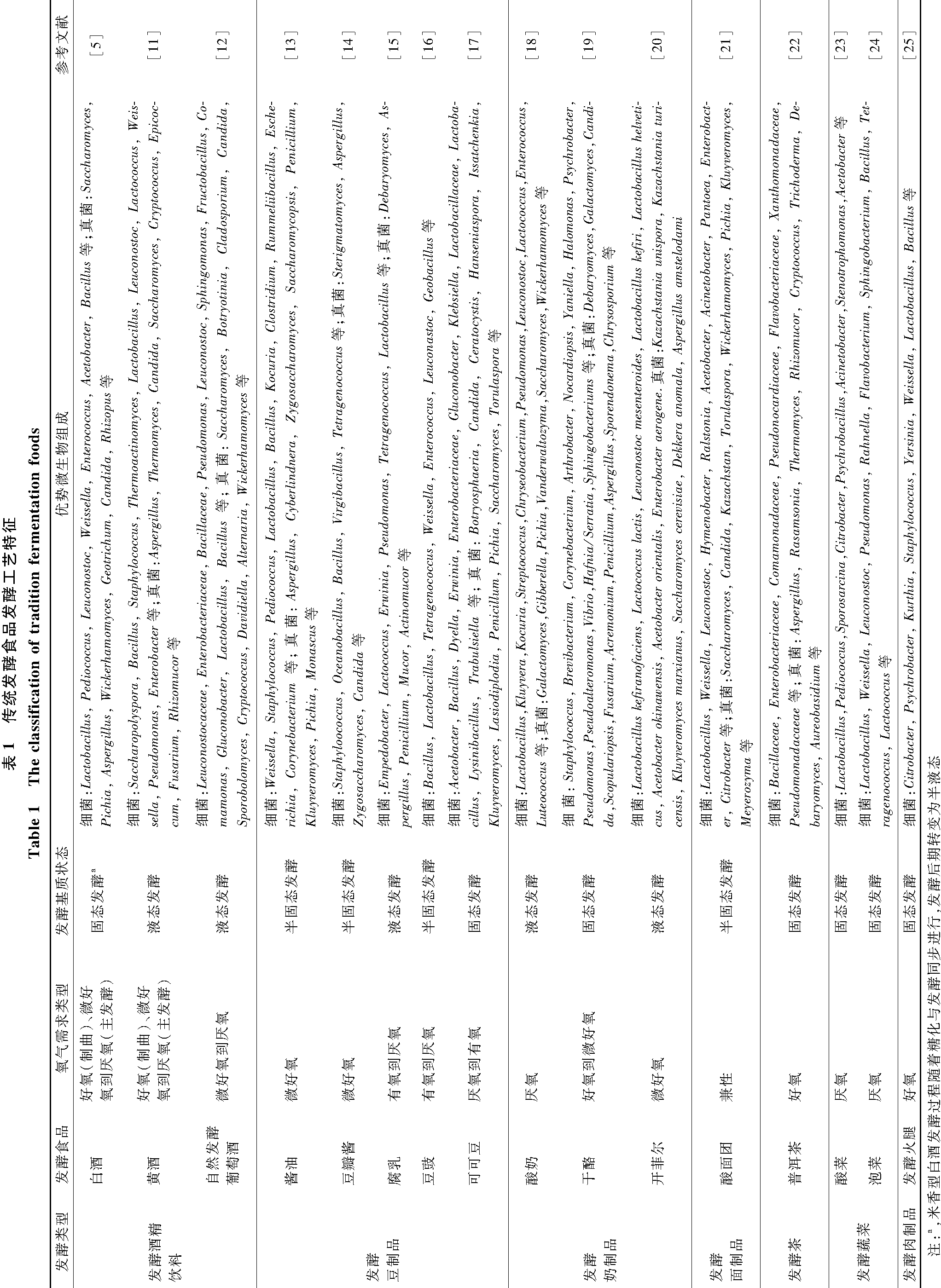
传统发酵食品营养丰富、风味独特,分布在全球不同的国家和地区。据统计,全球发酵食品种类多达200余种,在人类饮食结构中扮演着重要角色。根据原料分类,可将发酵食品分为发酵酒精饮料、发酵豆制品、发酵奶制品、发酵面制品、发酵茶、发酵蔬菜和发酵肉制品(表1)。以高梁、大米、大豆、蔬菜等为原料的发酵食品多来源于以农业为主的东方国家,例如中国的白酒、黄酒和食醋,日本的清酒和纳豆以及韩国的泡菜等;以动物乳、动物肉等为原料的发酵食品,如干酪、火腿等多来源于以畜牧业为主的西方国家[1]。
部分传统发酵食品技艺已经传承数千年,早期用于食品的长期保存,现多用于独特风味食品的加工。虽然发酵食品类别多、分布广,但发酵技艺存在着相似之处:通常将环境中或者原料自身携带的微生物作为发酵剂,在发酵过程中,微生物群落通过降解原料中的多糖、脂质和蛋白质等大分子物质产生葡萄糖、氨基酸等小分子进而代谢产生醛类、醛类、酸类、酯类等丰富的风味物质,赋予产品丰富的营养价值和独特的感官特征。因此,酿造微生物(组)直接影响了发酵产品的品质和质量,但是自然接种的发酵技艺不利于食品生产的稳定、安全和控制[2]。近年来,微生物组学、风味化学、发酵工学等领域的研究让人们对参与食品发酵的微生物组成、结构、功能以及菌群过程控制有了深入的认识,逐步解析了传统发酵食品的酿造机理,为传统食品发酵技术的升级革新提供了充足的理论基础。在此基础上,本综述定义了一种新型的发酵技术——生态发酵技术,系统介绍了生态发酵技术的原理与应用,并阐述了提出该技术的必要性和可行性,期望推动生态技术在不同酿造系统中的应用,加快传统发酵食品的产业转型和升级。
1 传统发酵食品的特征
1.1 传统发酵食品技艺特征
以蒸馏酒(中国白酒和威士忌)为例(图1),中国白酒是典型的传统发酵食品,在制作过程中将高粱等原料经蒸煮、糊化等工艺处理后,以固态形式置于窖池中进行密闭自然发酵[3];威士忌是典型的现代发酵食品,在生产过程中将发芽、存放、糖化后的谷类或者麦类原料捣碎,制成麦芽汁,以液态形式置于发酵罐中进行密闭发酵[4]。传统发酵食品工艺具有如下特征:
(1)传统发酵多采用自然接种的发酵方式。如在白酒发酵过程中,发酵微生物主要来源于“曲”和环境。研究发现,参与发酵过程的63%~91%细菌和20%~39%真菌是由发酵环境中的地面和操作工具所提供[5]。而在威士忌发酵过程中则是以人工添加的纯种酵母作为发酵剂,且对无菌环境要求较高。发酵过程中来源于环境的杂菌污染容易导致生产失败[6]。
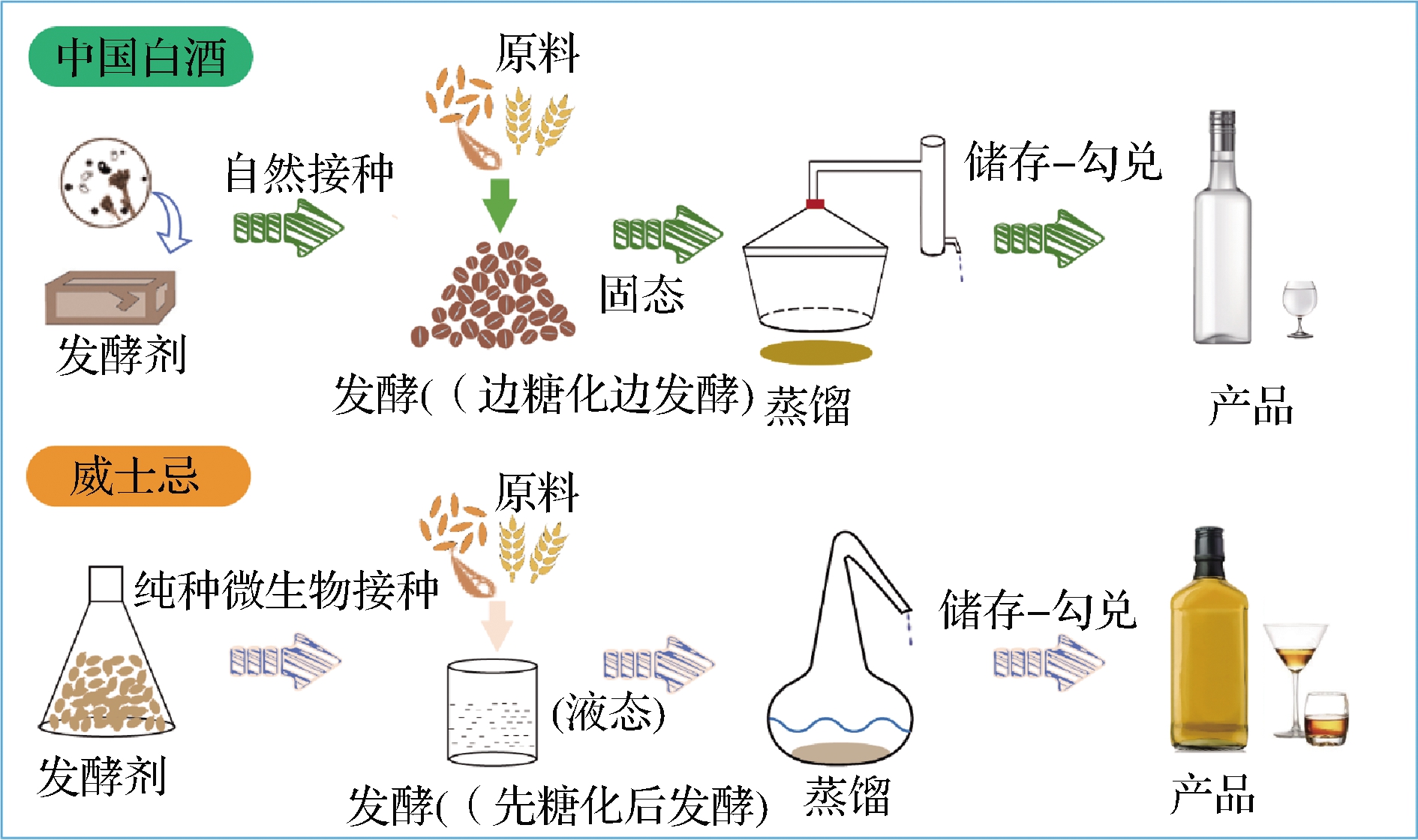
图1 中国白酒发酵传统工艺与威士忌发酵工艺的比较
Fig.1 Comparison of Baijiu and Whisky fermentation technology
(2)传统食品发酵属于多菌种混合发酵体系(表1)。如在白酒发酵过程中,可以检测到丰富的霉菌(Aspergillus、Mucor、Rhizopus和Rhizomucor等)、酵母菌(Saccharomyces、Candida、Issatchenkia、Pichia、Zygosomyces和Schizosaccharomyces等)和细菌(Lactobacillus、Pediococcus、Weissella以及Bacillus等)[7],在上述多种微生物共同作用下完成边糖化边发酵的发酵过程。而威士忌则是在发酵前完成糖化,由单种或多种纯种酵母菌完成。
(3)传统发酵食品品质控制为终端控制,即仅能对最终的产品进行测评。如在白酒发酵过程中,原料入池后无法对发酵过程中的参数以及参与发酵的微生物进行实时调控,控制产品品质。
与现代发酵的威士忌相比,中国白酒由于复杂的发酵工艺和发酵菌群使产品呈现出更丰富的风味、更高的营养和更好的口感。同时,发酵系统与自然环境的长时间交互形成了独特的发酵食品微生态,蕴含着基于酿造环境、生产原料与微生物之间相互作用的复杂酿造机制。
1.2 传统发酵工艺亟需技术革新
目前,由于基于纯种微生物的发酵过程对环境依赖性较小、生产周期较短、操作方式简单,在工业生产上已实现机械化、自动化和智能化,大大提高了生产效率,例如威士忌。而基于自然接种的发酵过程周期长、微生物组成复杂、环境依赖性较强,阻碍了生产效率的提高。现在有些自然发酵食品已尝试使用纯种微生物接种方式取代自然接种,例如日本酱油,将传统自然接种的制曲工艺改为人工接种,提高了生产的可控性。虽然我国发酵食品领域也对自然接种的发酵工艺进行革新例如麸曲白酒生产,但是由于不同发酵产品涉及不同的制作工艺和复杂的微生物群体,目前大多数传统发酵食品产业依然沿袭粗放的传统经验式操作模式进行生产,使得传统发酵食品行业的产品生产稳定性问题始终没有很好地解决。
通过选育源于自然发酵剂的功能微生物,直接代替自然发酵剂并不能保证产品质量,同时需要考虑酿造环境微生物对发酵过程的影响。以中国白酒为例,目前通过扩大生产规模是提高生产效率的主要方式,但实践证明在使用相同发酵剂的条件下,由于新老厂区环境微生物的不同,新厂区的出酒率以及产品品质明显低于老产区[8]。因此,提高新产区产品优质率需要长时间驯化和筛选特定的环境微生物,例如源于地面、操作工具[5]或窖泥等环境的微生物。
由于开放式的操作工艺,产品存在潜在的食品安全隐患。如普洱茶发酵过程中,环境中某些真菌的富集可能造成真菌毒素的污染[9];在白酒发酵过程中同样存在真菌毒素的污染隐患,研究显示大曲制作过程中自然接种上的青霉属、曲霉属等部分微生物存在真菌毒素代谢能力,可能会导致产品中真菌毒素的积累[10]。这些由微生物引起的安全隐患造成了生产过程控制成本增加。因此,以发酵微生物组为核心的发酵技术创新是未来传统酿造食品产业升级的必然趋势。
2 生态发酵技术
2.1 生态发酵技术的定义
基于自然接种的发酵过程一般难以人为控制接种微生物种类,近年来传统发酵微生物生态学与微生物组学技术不断发展,传统发酵科学机理得以解析,更多的酿造功能微生物被分离培养,为克服自然接种的局限性,理性构建新一代多菌种发酵技术奠定了基础。生态发酵技术指运用微生物生态学和微生物组学原理,通过优化发酵工艺,对以最优配比纯种微生物复合菌剂接种并对发酵菌群进行演替定向控制的发酵过程控制技术,实现微生物群落各功能种群协同生长与代谢,以较高效率生产出优质发酵产品。上述定义为狭义的生态发酵技术,强调了复合菌剂在生产中的应用。生态发酵技术的外延还包括:①自然接种的传统发酵体系酿造微生物群落的解构;②发酵微生物菌群的重构。因此,生态发酵技术体系包括了上游
的酿造微生物菌群解构技术、中游的核心酿造微生物菌群重组技术和下游的发酵过程控制技术。需要特别指出,生态发酵技术与以往研究中多菌种发酵技术的重要区别在于生态发酵技术强调了对传统发酵体系机理的解析,以更为理性的方式重构复合多菌种菌剂,在发酵过程中进行发酵参数检测和控制,所生产的发酵产品品质不低于传统自然接种发酵产品的品质。
基于合成微生物组的生态发酵技术可在一定程度上减少对原始环境微生物富集过程的依赖性,生产过程稳定性较高;相对于开放式的传统发酵技术,生态发酵技术中参与发酵过程的微生物均需要进行安全指标测试,保证产品安全。同时,高底物分解能力、高风味代谢能力菌株的组合可以提高发酵性能。因此,基于合成微生物组的生态发酵技术具有高稳定性、高安全性和高效率的特点。
2.2 生态发酵体系的核心技术体系
生态发酵技术体系包括了上游的酿造微生物群落解构技术、中游的核心功能微生物菌群重组技术和下游的发酵过程控制技术(图2)。

图2 生态发酵技术原理
Fig.2 Principle of ecological fermentation technology
(1)上游技术
上游技术的主要目的在于传统发酵体系中微生物菌群解构(组成、结构、功能),菌种的安全、发酵性能的评价。同时分离获取菌种,构建核心功能微生物菌种数据库,为中游核心功能微生物菌群的重构提供组装依据和菌种。
近年来,研究人员对食品发酵微生物菌群的组成及演替规律进行了大量研究。研究发现参与发酵过程的微生物组成较为复杂。如在清香型白酒发酵中,有722个细菌物种分类单元(operational taxonomic units,OTUs)和1 504个真菌OTUs参与发酵,通过物种信息注释,他们属于包括Lactobacillus、Weissella、Lactococcus属在内的49个细菌属和Saccharomyces、Candida、Schizosaccharomyces在内的34个真菌属[26];在虾酱发酵中,研究人员发现有3 593个OTUs参与,平均每个样本中能检测到多达513个OTUs[27]。虽然参与发酵的微生物组成复杂,但在发酵过程中微生物菌群存在一定的演替规律,例如在清香型白酒发酵过程细菌属中,发酵起始微生物是以Pseudomonas和Bacillus为主,随着发酵时间的推移,Lactobacillus逐步演变为绝对优势微生物(相对丰度>80%);真菌属中Pichia是整个发酵过程的优势微生物(平均丰度>10%),其次是Geotrichum、Saccharomyces等酵母[26]。相似的现象同样也在其他香型白酒中发现,芝麻香型白酒发酵过程中发酵起始细菌属以Bacillus、Staphylococcus等非乳杆菌属为优势微生物,随着发酵的进行,Lactobacillus成为绝对优势微生物(相对丰度>80%);真菌属Pichia、Saccharomyces是整个发酵过程中的优势微生物[28]。在特定酿造环境下微生物菌群的演替规律稳定性是发酵产品品质稳定的保证,同时也表明存在潜在的功能微生物组成以及动力学演替规律,这种规律为使用合成微生物组代替自然菌群提供一种可能。
在菌种功能研究中,科研人员在不同的发酵系统中发现了大量的功能菌种或菌群并得到纯培养物。在食醋发酵体系中,乙偶姻是食醋产品的重要风味成分,LU等人[29]发现,Acetobacter pasteurianus和4种Lactobacillus(L.buchneri,L.reuteri,L.fermentum和L.brevis)是乙偶姻合成的功能微生物群。在白酒酿造中,氨基甲酸乙酯是2A类致癌物,对人体存在潜在的致癌性和毒性,广泛存在于酒、醋、酱油和干酪等发酵产品中。CUI等[30]在白酒发酵酒醅中筛选出具有氨基甲酸乙酯及其前体尿素降解能力的Lysinibacillus sphaericus MT33,该菌株可代谢产生940 U/L的氨基甲酸乙酯水解酶和1 580 U/L的脲酶。同时,相关工作人员在研究过程中积累了多种可代谢产生关键风味化合物的功能微生物菌种(菌群),例如,萜烯类化合物产生菌(Saccharomyces cerevisiae)、对甲酚产生菌群(Clostridium butyricum,Clostridium tyrobutyricum,Clostridium aminovalericum和Eubacterium contortum)、己酸产生菌(Clostridium kluyveri N6)、地衣素产生菌(Bacillus licheniformis ATCC 14580)、四甲基吡嗪产生菌(Bacillus subtilis CCTCC M 208157)、蛋白酶产菌(Aspergillus hennebergii HX08)、硫化物产生菌群(Saccharomyces cerevisiae JZ109,Lactobacillus buchneri JZ-JN-2017)、产酱香芽孢杆菌(Bacillus licheniformis MT6,Bacillus licheniformis MT15)、Surfactin产生菌(Bacillus amyloliquefaciens MT45)、土味素控制菌群(Bacillus subtilis 2-16,Bacillus amyloliquefaciens 1-45)等。功能微生物的鉴定和挖掘为中游技术合成微生物组提供可选择的菌种。风味化学研究中,不同特征的风味成分不断被鉴定,近期研究发现酱香型白酒中的咸菜味香气是由8种不同化合物形成[31],然而各种物质的形成机制以及相对应的功能微生物尚不清晰。因此,需要进一步完善功能微生物的数据库。
(2)中游技术
中游技术旨在基于功能微生物构建合成微生物菌群,筛选出最优配比的纯种微生物复合菌剂,包括设计、组装、优化和测试4个模块。
目前,研究人员已在部分自然发酵食品体系尝试利用合成微生物组代替自然发酵菌群,对促进传统发酵生产流程的简化,提高生产机械化程度具有积极的作用。中国白酒通常采用大曲、小曲或麸曲作为发酵剂,经过固态发酵、固态蒸馏、储存勾兑而成。徐岩等[32]建立了以功能微生物Rhizopus oryzae,Saccharomyces cerevisiae,Pichia anomala和Issatchenkia orientalis为基础的最小功能微生物菌群作为发酵剂的小曲清香型生产技术,实现大规模的机械化改造,推动小曲清香型白酒的产业升级。另外,WANG等[26]选择5种微生物作为初始发酵剂(Lactobacillus acetotolerans,Pichia kudriavzevii,Geotrichum candidum,Candida vini和Saccharomyces cerevisiae),在不接触环境的条件下实现了微生物演替规律和风味特征的重现,这项研究结果表明合成微生物菌群可以在一定程度上减小传统食品发酵对酿造环境的依赖。该推测同样在豆酱发酵体系得到验证:近期,JIA等[33]选择Aspergillus oryzae,Bacillus subtilis,Staphylococcus gallinarum,Weissella confusa,Zygosaccharomyces rouxii作为起始发酵剂,重现了原位酿造微生物菌群的功能。另外,针对西方食品发酵,研究人员也进行了使用合成微生物群落替代自然发酵菌群的尝试,并取得较好的效果。在奶酪发酵中,选择参与发酵的6种高丰度微生物(Staphylococcus、Brevibacterium、Brachybacterium、Candida、Penicillium和Scopulariopsis,在超过50%的样本中平均丰度>1%)作为起始发酵剂,成功复制了原始发酵菌群的演替规律[19]。可可豆是全球重要的发酵食品之一,ANDREW等[34]以功能组装为原则构建含有5种微生物的合成微生物组进行模拟发酵(Saccharomyces cerevisiae、Lactobacillus plantarum、Lactobacillus fermentum、Acetobacter pasteurianus和Acetobacter tropicalis),结果显示该5种微生物能够模拟可可豆原位发酵过程中微生物演替规律和化学变化[35]。以上研究表明了用合成微生物组替代自然菌群的可行性。
但是目前合成微生物组技术体系的构建依然处于起步阶段。在实际应用阶段需要考虑以下方面:目前多数发酵食品的质量评价体系是多基于风味特征、口感、色泽等非统一量化的参数为依据,而在设计合成微生物组时需要提出明确的、可量化的质量评价指标作为功能定义,以此为依据设计并评价合成微生物组。因此,需要推行食品领域质量评价指标的精准量化和标准化。虽然传统发酵食品微生物组研究已取得较大的进展,不同发酵食品微生物演替规律被揭示,但是依然存在包括微生物间相互作用以及高阶互作在内的关键风味形成机制的研究空白,这将给合成微生物组的设计带来困难。合成微生物组的研究并不能一蹴而就的,不能期望所设计的合成微生物组一次性达到既定的工作目标。合成微生物组的最终应用需要经过设计、构建、优化、测试多个回合来获取必要的酿造原理,不断对合成微生物组进行改造和优化,最终形成适合应用的合成微生物组。
(3)下游技术
下游技术是以合成微生物组为发酵剂应用于实际生产,包括合成微生物组的放大培养以及发酵过程控制,是科研成果与实际生产结合的重要环节。
目前,在发酵食品生产过程中,存在各种形式的菌种放大和保存方式,例如纯种大曲、菌粉/麸曲、菌液等。最为典型的是已实现工业化生产的酸奶发酵剂(一种商业菌粉),极大地方便了酸奶的制作过程,例如在市场流行的安琪十二菌型酸奶发酵剂、川秀双歧杆菌七菌型发酵剂以及五菌型发酵剂等。但需要注意的是,在应用过程中不同发酵食品应根据微生物的特性以及自身的酿造工艺采取不同的放大策略,同时要综合考虑在放大和储存中,微生物活性和功能的变化。但对于传统发酵食品菌种的培养和放大工艺,目前尚缺少系统的研究,如在白酒酿造领域和黄酒酿造领域,直投式复合菌剂的技术仍然处于初步开发阶段。
目前的研究表明,通过调整发酵参数可实现对产品质量的控制,包括原料和理化参数。食品风味一方面由微生物代谢产生,另一方面由原料直接带入。原料质量的差异会直接影响最终产品的风味,因此生产中对严格把关原料质量是稳定产品品质的基本保证。发酵原料为发酵微生物菌群提供可利用的营养物质,不同特征的原料会影响发酵过程中微生物菌群的结构和功能[36],最终影响产品的特征,例如原料中的还原糖组成[37]以及碳氮比例[38]等。理化参数在一定程度上驱动微生物群落组装和功能演替。以温度为例,在酸面团发酵时,控制发酵温度在21 ℃可使得成熟的酸面团完全由乳酸菌组成,在其繁殖过程中能够抑制其他菌群的生长[39];在干酪发酵过程中,提高干酪成熟温度可促进蛋白水解、脂解和氨基酸分解代谢相关基因的表达,提高干酪的成熟率[40]。除此之外,还包括发酵过程中的pH[41]、水分[19]、微生物代谢产物[42]、盐度[33]等理化参数。因此,生产中原料的选择以及工艺参数的控制,对维持发酵微生物群落功能的稳定性至关重要。
生产参数的检测手段也正随着科学技术的进步不断被开发和优化,并应用于传统发酵食品行业。例如应用无线温度传感器在线检测白酒窖池发酵温度变化、应用低场核磁共振(nuclear magnetic resonance,NMR)在线检测固态发酵体系中的水分含量[43]。同时生产参数的数据已用于构建数学模型,推动生产工艺的进一步优化。例如在白酒生产中,JIN等[44]基于生产参数构建了以Han-Levenspiel方程为基础的数学模型,准确地预测淀粉、乳酸、乙醇的含量变化。另外,很多企业已引入智能化生产线,实现发酵食品智能化和数字化生产,提高发酵过程控制的时效性和有效性。例如,劲牌有限公司较早的进行机械化工艺改革,实现了管理信息化、过程数据化、操作自动化[45]。今世缘酒业采用自动化圆盘制曲系统、自动化生产线、智能装甑、自动分流接酒等现代化生产工艺,实现了白酒的机械化和自动化生产[46]。山东景芝股份有限公司将数字化酿造工艺管理系统应用于浓香型白酒机械化、自动化和智能化酿造生产中,实现对各道工序关键工艺参数的数字化管控,确保了白酒酿造生产的定量化、精细化和标准化操作[47]。
2.3 生态发酵技术的研究方法学体系
(1)微生物组学技术
随着高通量测序、分析、统计学的发展,形成了包括宏组学、分析化学、分子生物学以及统计学在内的微生物组学技术。传统发酵食品领域涉及的微生物组学技术主要用于研究参与发酵过程微生物的组成、结构和功能,服务于生态发酵上游技术。应对复杂的发酵微生物菌群,扩增子测序技术是目前应用最为广泛的微生物群落研究手段。该技术时对特定长度的PCR产物或者捕获的片段进行测序,分析序列中的变异,用于研究微生物菌群的组成和结构。例如通常选择16S rRNA V4、V3-V4区通用引物研究细菌菌群组成,选择ITS 1区、ITS 2的通用引物研究真菌组成。得益于高通量、低成本的优点,扩增子测序技术已逐步取代了基于细胞形态学的可培养方法以及基于分子生物学的DGGE等方法。宏基因组学、宏转录组学和宏蛋白组学主要用于在不同水平上研究微生物菌群功能分布。宏基因组(metagenome)被定义为区域内全部微生物遗传物质的总和,既包括转录活性基因也包括非转录活性基因,属于DNA水平。宏转录组(metatranscriptome)则是测定微生物群落宏基因组中在特定时刻下的具有转录活性基因的存在数量,属于RNA水平。宏蛋白质组(metaproteome)是测定环境样本中全部微生物蛋白质的总和,属于蛋白质水平。在应用中,研究人员通过重构目标产物的合成或分解代谢网络,基于测序结果中的物种以及功能注释,判定相应途径的功能微生物,可在分子水平解析酿造机制。同时,在构建新型化合物或未知代谢途径化合物的代谢途径时,同位素示踪技术以及基因工程等均是可采用的技术手段。统计学分析同样也应用于微生物功能的判定,例如应用较多的双向正交偏最小二乘回归模型(bidirectional orthogonal partial least squares regression,O2PLS)、偏最小二乘回归模型(partial least-squares regression,PLS)、斯皮尔曼相关性等。微生物组学技术的发展为揭示酿造机制及挖掘功能微生物提供了强有力的支撑,允许研究人员针对不同科学需要、实际条件选择可行的技术和分析方法。
(2)合成微生物组工程技术
合成微生物组工程技术包括了合成微生物组的设计、组装、优化和测试。特别指出,合成微生物组工程技术需要对生产目标具有明确的功能定义,即设定可量化和测定的一个或多个参数,作为评价合成微生物组功能的依据。自上而下(top-down design)和自下而上(bottom-up design)是设计合成微生物组的常用原则。自上而下的设计原则是指通过调控环境变量,利用生态位的选择改变现有微生物组组成和功能,达到设定目标,突出了环境选择的重要性。自下而上则是要求以功能为导向构建代谢网络,划分功能模块,并确定相应模块的关键功能物种,从而实现微生物组的功能组合,突出理性设计的重要性。目前,自下而上的设计策略普遍被接受和应用。合成微生物组的组装过程即根据设定的生产目标选择相应的功能微生物,合成功能微生物菌群,并考虑菌群的功能鲁棒性、结构稳定性等问题。合成微生物组的优化过程则是在小试或者中试规模优化微生物组合、接种量以及接种比例,基于正交试验响应面分析、数学模型等统计工具预测最优组合。合成微生物组的测试过程则是对预测最优组合的合成微生物组进行功能测定,并与事先设定的功能定义比较。
(3)菌种放大和发酵过程控制技术
菌种的扩大培养是发酵生产的第一道工序(种子制备)。工业生产规模越大,单次发酵所需的种子就越多。因此,菌种扩大培养的目的就是要为每次发酵投料提供相当数量的代谢旺盛的种子。纯种曲制作技术、麸曲制作技术、发酵菌粉制作技术等固态形式的菌种放大技术,菌株液态培养技术等液态形式的菌种放大技术均是应用于不同传统发酵食品中的菌种扩大培养方法。其中细菌和酵母等既可以使用固态形式的菌种放大技术也可以使用液态形式的菌种放大技术。由于霉菌在液态基质培养条件下的产酶特征不同于固态基质,因此以产酶作为主要功能的霉菌需要选择固态菌种放大技术。
发酵过程控制技术是稳定发酵过程的基本保证。发酵过程控制技术包括发酵参数检测技术和发酵参数控制技术,是实现传统发酵过程智能化、自动化、机械化的基础。技术核心在于学习模型的构建(即解析发酵参数与发酵产品品质的对应关系),通过调控发酵参数实时控制发酵过程,形成发酵参数收集、深度学习、发酵过程控制、发酵生产应用的良性循环。
3 展望:生态发酵技术未来研发方向
(1)生态发酵技术中菌种的选择。确定功能菌种是实现合成微生物组发酵的关键因素。目前,食品发酵过程中微生物的组成研究主要依赖于扩增子测序技术。但是由于扩增长度的限制(<500 bp),该方法较难实现种水平、株水平的微生物组成分析,仅能达到属水平或者OTU水平,这使得无法准确确定功能微生物菌种信息[48]。另外,基于PCR的测序技术存在不可避免的引物偏好性、引物覆盖度不全的问题,这将对解析微生物组成以及后续的分析带来误差。不依赖PCR的鸟枪测序(宏基因组测序)能够有效地避免了PCR过程带来的误差;另外,可用的三代测序延长了测序的长度,提高了物种分辨能力,是未来酿造微生物组研究的发展方向。
(2)未培养微生物的可培养化是实现生态发酵技术应用的关键步骤。大部分自然界中的微生物(~99%)仍然难以培养,如中国白酒发酵系统的窖泥环境中存在大量未培养、功能未知的酿造功能微生物。目前,针对不可培养微生物对培养条件的严格依赖性,研究人员发明了5种可实现原位培养的技术:扩散室、分离芯片(iChip)、Trap技术、I-Tip技术以及胶囊包埋技术(GMDs)。2012年,GREUB 等[49]提出可培养组学的概念,实现微生物筛选条件的多样化、鉴定过程的高通量,解决了微生物筛选鉴定的高成本和低通量的问题。目前,运用可培养组学技术已基本实现了肠道微生物的可培养化[50]。将原位培养技术以及可培养组学技术应用于传统发酵食品领域,有利于完善酿造食品功能微生物数据库。
(3)发酵过程自动化和智能化。首先,需要对发酵设备进行创新。目前,发酵过程中参数收集的困难阻碍了对发酵机制认识以及实验中不同生产方案的应用反馈。例如在白酒发酵过程中,发酵容器为地下的密闭窖池,发酵过程中的理化参数收集需要以多次破坏的封闭体系为代价,多次的开窖取样可能会对产品产生影响。使用具有发酵参数(含氧量、含水量、温度、酸度等)采集和控制功能的集成发酵设备,可以在不破坏原有发酵状态的前提下,快速、方便、密集地对发酵过程中各理化因子指标的变化进行在线跟踪和调控,这将更有利于解析发酵机制,更有利于实时控制发酵过程,进而指导如何提高产品的优质率。其次,需要提高产品生产的靶向性和多样性。生态发酵技术使得传统发酵摆脱环境的限制,可以在实验室规模优化发酵参数,如不同的合成微生物组组成、接种比例、不同的原料等。同时新型的发酵设备方便了发酵过程中参数的收集。因此,可通过整合不同方案中的发酵参数以及对应的产品特征,构建数学模型,编写以发酵参数为输入,产品特征为输出的程序,并搭建用户界面,构建产品靶向设计平台。应用中以可检测、可控制的参数为基础,在线模拟生产过程和预测产品特征,一方面指导更合理地设计生产方案,提高生产效率;另一方面可用来设计更加多样化的产品,以满足消费者多元化的需求。
[1] TAMANG J P,COTTER P D,ENDO A,et al.Fermented foods in a global age:East meets West[J].Comprehensive Reviews in Food Science and Food Safety,2020,19(1):184-217.
[2] 任聪, 杜海,徐岩.中国传统发酵食品微生物组研究进展[J].微生物学报,2017,57(6):885-898.
REN C,DU H,XU Y.Advances in microbiome study of traditional Chinese fermented foods[J].Acta Microbiologica Sinica,2017,57(6):885-898.
[3] 沈怡芳. 白酒生产技术全书[M].北京:中国轻工业出版社,1998.
SHEN Y F.Liquor production technology book[M].Beijing:China Light Industry Press,1998.
[4] WALKER G M,HILL A E.Saccharomyces cerevisiae in the production of whisk(e)y[J].Beverages,2016,2(4):15.
[5] WANG X,DU H,ZHANG Y,XU Y.Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation[J].Applied and Environmental Microbiology,2018,84(4):e02 369-02 317.
[6] DOLAN T C S.Bacteria in whisky production[J].Brewer,1979,65(772):60-64.
[7] ZHENG X W,YAN Z,HAN B Z,et al.Complex microbiota of a Chinese “Fen” liquor fermentation starter (Fen-Daqu),revealed by culture-dependent and culture-independent methods[J].Food Microbiology,2012,31(2):293-300.
[8] 王雪山.不同环境清香类型白酒发酵微生物种群结构比较及溯源分析[D].无锡:江南大学,2018.
WANG X S.Microbial community structure and microbial source tracking of Chinese light-flavor liquor fermentation in different environments[D].Wuxi:Jiangnan University,2018.
[9] 马燕, 张冬莲,苏小琴,等.茶叶中真菌毒素污染的国内外研究概况[J].中国食品卫生杂志,2014,26(6):627-631.
MA Y,ZHANG D L,SU X Q,et al.Review of the studies on the contamination of mycotoxins in tea[J].Chese Journal of Food Hygiene,2014,26(6):627-631.
[10] 朱文优.大曲中赭曲霉毒素A及其产生微生物的分布特征研究[D].无锡:江南大学,2017.
ZHU W Y.The distribution of ochratoxin A in Daqu and its producing microorganisms[D].Wuxi:Jiangnan University,2017.
[11] 刘芸雅.绍兴黄酒发酵中微生物群落结构及其对风味物质影响研究[D].无锡:江南大学,2015.
LIU Y Y.Characterization of microflora and their functions on flavor compounds in Shaoxing rice wine[D].Wuxi:Jiangnan University,2015.
[12] BOKULICH N A,COLLINS T S,MASARWEH C,et al.Associations among wine grape microbiome,metabolome,and fermentation behavior suggest microbial contribution to regional wine characteristics[J].mBio,2016,7(3):e00 631-616.
[13] 胡传旺.酱油发酵过程微生物群落解析及功能研究[D].无锡:江南大学,2017.
HU C W.Dynamics of microbial community during soy sauce fermentation and functional analysis strains isolated from moromi[D].Wuxi:Jiangnan University,2017.
[14] YUNHAO L,XINYI T,YUANPING L,et al.Physicochemical properties and microbial community dynamics during Chinese horse bean-chili-paste fermentation,revealed by culture-dependent and culture-independent approaches[J].Food Microbiology,2020,85:103 309.
[15] 陶康, 吴凌伟,金晓芳,等.基于高通量基因测序分析腐乳微生物多样性[J].食品科学,2020:1-11.
TANG K,WU L W,JIN X F,et al.Microbial diversity of Sufu was analyzed based on high-throughput gene sequencing[J].Food Science,2020:1-11.
[16] 刘晓峰.云南省特色发酵豆豉中微生物群落结构及其元基因组分析[D].昆明:昆明理工大学,2015.
LIU X F.Analysis of microbial community structures and metagenome from traditional fermented soybeans in Yunnan[D].Kunming:Kunming University of Science and Technology,2015.
[17] MOTA-GUTIERREZ J,BOTTA C,FERROCINO I,et al.Dynamics and biodiversity of bacterial and yeast communities during fermentation of cocoa beans[J].Applied and Environmental Microbiology,2018,84(19):17.
[18] 杨超, 丁学智,龙瑞军.青藏高原地区牦牛鲜奶和酸奶营养价值及微生物组成[J].动物营养学报,2018,30(4):1 262-1 270.
YANG C,DING X Z,LONG R J.Nutritional value and microbial flora of yak fresh milk and kurut in qinghai-tibetan plateau[J].Chinese Journal of Animal Nutrition,2018,30(4):1 262-1 270.
[19] WOLFE B,BUTTON J,SANTARELLI M,DUTTON R.Cheese rind communities provide tractable systems for in situ and in vitro studies of microbial diversity[J].Cell,2014,158(2):422-433.
[20] GAO W,ZHANG L W.Comparative analysis of the microbial community composition between Tibetan kefir grains and milks[J].Food Research International,2019,116:137-144.
[21] LIU X J,ZHOU M,JIAXIN C X,et al.Bacterial diversity in traditional sourdough from different regions in China[J].LWT-Food Science Technology,2018,96:251-259.
[22] ZHAO M,SU X Q,NIAN B,et al.Integrated meta-omits approaches to understand the microbiome of spontaneousfermentation of traditional Chinese pu-erh tea[J].mSystems,2019,4(6):e00 680-19.
[23] LIU Z G,LI J Y,WEI B L,et al.Bacterial community and composition in Jiang-shui and Suan-cai revealed by high-throughput sequencing of 16S rRNA[J].International Journal of Food Microbiology,2019,306:9.
[24] MOEUN L,JUNG HEE S,JI MIN P,JI YOON C.Bacterial diversity in Korean temple kimchi fermentation[J].Food Research International,2019,126:108 592-108 592.
[25] 郭明亮.火腿菌群分析及优良菌株对发酵香肠中菌群构成的影响[D].扬州:扬州大学,2015.
GUO M L.Analysis of ham flora and the influence of excellent strains on the flora composition in fermented sausages[D].Yangzhou:Yangzhou University,2015.
[26] WANG S L,WU Q,NIE Y,XU Y.Construction of synthetic microbiota for reproducible flavor metabolism in Chinese light aroma type liquor produced by solid-state fermentation[J].Applied and Environmental Microbiology,2019,85(10):e03 090-18.
[27] 尹超.虾酱发酵过程中菌相变化规律及其对风味形成的影响[D].青岛:青岛科技大学,2019.
YIN C.Bacterial dyanmics during fermentation of shrimp paste and its effect on flavor formation[D].Qingdao:Qingdao University of Science & Technology,2019.
[28] 李小龙, 吴群,徐岩.乙醇与酸度协同作用推动芝麻香型白酒固态发酵过程的微生物群落演替[J].微生物学通报,2019,46(1):1-10.
LI X L,WU Q,XU Y.Synergism between ethanol and acidity drives microbial community succession during roasted sesame-flavor liquor solid-state fermentation[J].Microbiology China,2019,46(1):1-10.
[29] LU Z M,LIU N,WANG L J,et al.Elucidating and regulating the acetoin production pole of microbial functional groups in multispecies acetic acid fermentation[J].Applied and Environmental Microbiology,2016,82(19):5 860-5 868.
[30] CUI K X,WU Q,XU Y.Biodegradation of ethyl carbamate and urea with Lysinibacillus sphaericus MT33 in Chinese liquor fermentation[J].Journal of Agricultural and Food Chemistry,2018,66(6):1 583-1 590.
[31] WANG L,FAN S,YAN Y,et al.Characterization of potent odorants causing a pickle-like off-odor in moutai-aroma type Baijiu by comparative aroma extract dilution analysis,quantitative measurements,aroma addition,and omission studies[J].Journal of Agricultural and Food Chemistry,2020,68(6):1 666-1 677.
[32] 徐岩. 基于风味导向技术的中国白酒微生物及其代谢调控研究[J].酿酒科技,2015(2):1-11;16.
XU Y.Study on liquor-making microbes and the regulation & control of their metabolism based on flavor-oriented technology[J].Liquor-Making Science & Technology,2015(2):1-16.
[33] JIA Y,NIU C T,LU Z M,et al.A bottom-up approach to develop simplified microbial community model with desired functions:Application for efficient fermentation of broad bean paste with low salinity[J].Applied and Environmental Microbiology,2020,86(12):e00 306-320.
[34] LEE A H,NEILSON A P,O’KEEFE S F,et al.A laboratory-scale model cocoa fermentation using dried,unfermented beans and artificial pulp can simulate the microbial and chemical changes of on-farm cocoa fermentation[J].European Food Research and Technology,2019,245(2):511-519.
[35] LEE A H,NEILSON A P,O’KEEFE S F,et al.A laboratory-scale model cocoa fermentation using dried,unfermented beans and artificial pulp can simulate the microbial and chemical changes of on-farm cocoa fermentation[J].European Food Research and Technology,2018,245(2):511-519.
[36] TAN Y,ZHONG H,ZHAO D,DU H,XU Y.Succession rate of microbial community causes flavor difference in strong-aroma Baijiu making process[J].International Journal of Food Microbiology,2019,311:108 350.
[37] LIU C C,FENG S B,WU Q,et al.Raw material regulates flavor formation via driving microbiota in Chinese liquor fermentation[J].Frontiers in microbiology,2019,10:13.
[38] JIANG J,LIU Y C,LI H H,et al.Modeling and regulation of higher alcohol production through the combined effects of the C/N ratio and microbial interaction[J].Journal of Agricultural and Food Chemistry,2019,67(38):10 694-10 701.
[39] MENEZES L A A,SARDARO M L S,DUARTE R T D,et al.Sourdough bacterial dynamics revealed by metagenomic analysis in Brazil[J].Food Microbiology,2020,85:11.
[40] DE F F,GENOVESE A,FERRANTI P,et al.Metatranscriptomics reveals temperature-driven functional changes in microbiome impacting cheese maturation rate[J].Scientific reports,2016,6:11.
[41] BOKULICH N A,BAMFORTH C W,MILLS D A.Brewhouse-resident microbiota are responsible for multi-stage fermentation of American coolship ale[J].PLoS ONE,2012,7(4):e35 507.
[42] YU Z,JUN M,JIWEI N,et al.Succession sequence of lactic acid bacteria driven by environmental factors and substrates throughout the brewing process of Shanxi aged vinegar[J].Applied Microbiology and Biotechnology,2018,102(6):2 645-2 658.
[43] JIN G Y,ZHU Y,RINZEMA A,et al.Water dynamics during solid-state fermentation by Aspergillus oryzae YH6[J].Bioresource Technology,2019,277:68-76.
[44] JIN G,UHL P,ZHU Y,et al.Modeling of industrial-scale anaerobic solid-state fermentation for Chinese liquor production[J].Chemical Engineering Journal,2020,394:124 942.
[45] 杨强. 劲牌公司白酒生产机械化发展历程与体会——在全国第四届清香型白酒高峰论坛上的发言[J].酿酒,2011,38(5):12-13.
YANG Q.The development process and experience of the mechanization of liquor production in Jinpai Company——Speech at the 4th National Fragrance Liquor Summit Forum[J].Liquor Making,2011,38(5):12-13.
[46] 王延才. 今世缘打破白酒香型地域限制的重大意义[J].中国酒,2020(1):49.
WANG Y C.The great aignificance of Jinshiyuan breaking the regional limitation of liquor flavor[J].Chinese liquor,2020,1:49.
[47] 刘选成, 张冬跃,赵德义,等.数字化酿造工艺管理系统在浓香型白酒机械化、自动化和智能化酿造生产[J].酿酒科技,2018(11):70-74-79.
LIU X C,ZHANG D Y,ZHAO D Y,et al.Application of digital process management system in mechanized,automated and intelligent production of Nongxiang Baijiu[J].Liquor-Making Science & Technology,2018(11):70-74-79.
[48] JOHNSON J S,SPAKOWICZ D J,HONG B Y,et al.Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis[J].Nature Community,2019,10(1):5 029.
[49] GREUB G.Culturomics:a new approach to study the human microbiome[J].Clinical Microbiology and Infection,2012,18(12):1 157-1 159.
[50] LAGIER J C,KHELAIFIA S,ALOU M T,et al.Culture of previously uncultured members of the human gut microbiota by culturomics[J].Nature Microbiology,2016,1:16 203.