风干牛肉是新疆哈萨克族传统的发酵肉制品,也是山区牧民过冬必备食品[1]。新疆北疆牧区的牧民通常在11月到来年1月制作风干肉,牛肉经过分割切条、腌制后自然风干,制作时间约为2~8个月不等,其产品类型和我国的火腿[2]、土耳其的pastirma[3]和意大利的bresaola[4]相类似。近年来,随着新疆旅游业的发展,风干肉作为新疆的特色旅游食品,其消费量逐年上升。传统哈萨克族风干肉的发酵主要依靠天然多菌种混合发酵,包括乳酸菌、微球菌、酵母菌、霉菌等各种微生物[5],发酵体系中微生物菌群结构复杂,其中真菌所产生的酶可以对肉中的糖类、脂肪和蛋白质进行分解,生成酮醛酯等风味物质提高产品品质、赋予产品特殊的风味和色泽[6-9]。FLORESM等[10]研究发酵香肠发现德巴利酵母菌(Debaryomyces spp.)对成熟过程中挥发性物质的产生有重要影响,通过抑制脂质氧化产物的产生、促进乙酯的生成,可以产生香肠特有的香气。SIRINE等[11]研究表明青霉菌具有抗氧化、防止酸败和保持色泽的作用,还可以防止香肠表面形成黏性物质。
传统食品中的微生物菌落结构更加复杂[12-14]。为了更加清楚地了解传统发酵食品中微生态体系,可借鉴生态学中对微生物多样性研究技术和方法[15]。高通量测序技术是微生态学研究方法的一种,能够快速全面的分析微生物结构复杂的样品[16],QUIJADA等[17]使用高通量测序技术对西班牙发酵香肠“Chorizo de León”中微生物菌群多样性研究发现,不同地区发酵香肠中微生物在亚属分类等级上有明显差异;REBECCHI等[18]采用Illumina高通量测序研究猪肉、牛肉和羊肉腊肠中微生物的多样性,结果显示不同动物来源的香肠微生物多样性存在显著差异。
由于地理环境的差异,使不同来源的风干肉中微生物多样性有所不同[19]。但目前对新疆风干肉中微生物菌群多样性还没有系统的研究,尤其是真菌,其对环境的要求更低,适应性更强,但在风干肉中的群落结构还不清楚,因此很有必要对新疆风干肉中的真菌多样性进行研究,明确真菌结构组成,为风干肉安全性研究和下一步挖掘专用发酵剂提供理论基础,同时也可为后期产品的工业化生产提供依据。
1 材料与方法
1.1 样品
选取新疆北疆4个哈萨克族聚居的牧区为采样对象,全部为自然发酵。制作时间都为当年11月份制作的新鲜风干肉,且自然风干时间都为40 d,采集的样品单独装入采样袋,放在车载冰箱(0~4 ℃)条件下24 h内运回实验室,存贮在-80 ℃冰箱,备用。详细分组情况如表1所示。其中采样地方相同的用相同的大写字母,数字代表来自于不同的家庭。
表1 样品来源及分组
Table 1 Source and grouping of air-dried meat samples
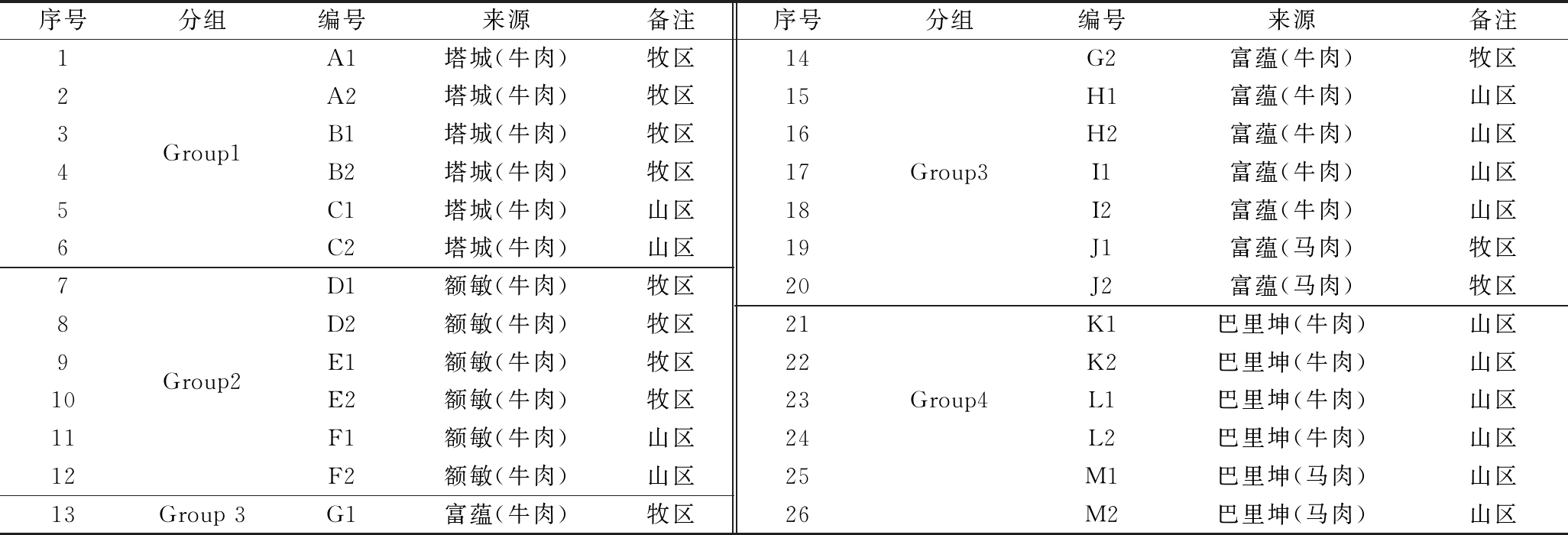
序号分组编号来源备注序号分组编号来源备注123456789101112Group1Group2A1A2B1B2C1C2D1D2E1E2F1F2塔城(牛肉)塔城(牛肉)塔城(牛肉)塔城(牛肉)塔城(牛肉)塔城(牛肉)额敏(牛肉)额敏(牛肉)额敏(牛肉)额敏(牛肉)额敏(牛肉)额敏(牛肉)牧区牧区牧区牧区山区山区牧区牧区牧区牧区山区山区141516171819202122232425Group3Group4G2富蕴(牛肉)牧区H1富蕴(牛肉)山区H2富蕴(牛肉)山区I1富蕴(牛肉)山区I2富蕴(牛肉)山区J1富蕴(马肉)牧区J2富蕴(马肉)牧区K1巴里坤(牛肉)山区K2巴里坤(牛肉)山区L1巴里坤(牛肉)山区L2巴里坤(牛肉)山区M1巴里坤(马肉)山区13Group 3G1富蕴(牛肉)牧区26M2巴里坤(马肉)山区
1.2 试剂
强力土壤®DNA提取试剂盒,美国OMEGA公司;2×Taq Master Mix (Dye Plus),南京诺唯赞生物科技有限公司;Magnetic Soil and Stool DNA Kit试剂盒,天根生化科技(北京)有限公司;Axygen凝胶回收试剂盒,Axygen美国;琼脂糖,西班牙Biowest公司。
1.3 仪器与设备
高速冷冻离心机(Eppendorf 5810R),德国Eppendorf AG公司;精密电子天平(BSM-220.4),上海卓精;Bio-Rad电泳仪、PCR仪(T100)、凝胶成像系统(HP1020),美国Bio-Rad伯乐公司;立式压力蒸汽灭菌器(LDZX-30KBS),上海申安医疗器械厂。
1.4 方法
1.4.1 真菌DNA提取与检测
直接使用DNA提取试剂盒提取样品中真菌的总DNA。提取方案按照试剂盒说明书进行,每个样品提取3次,提取的DNA用乙醇沉淀并提纯。DNA浓度用分光光度法检测,之后在1.2%琼脂糖凝胶电泳上检测DNA的完整性。
1.4.2 真多样性分析
选取真菌的ITS1 区序列进行高通量测序分析。使用PCR仪进行PCR扩增,引物选择ITS1 区通用引物ITS1F:5′-CTTGGTCATTTAGAGGAAGTAA-3′;ITS2:5′-GCTGCGTTCTTCATCGATGC-3′,扩增出的片段用1.2%琼脂糖凝胶电泳检测,使用凝胶成像系统进行观察,效果较好的样本于2%琼脂糖凝胶电泳切胶回收,以回收产物为模板进行1次8 循环的PCR 扩增,将Illumina平台测序所需要的接头、测序引物、标签序列添加到目的片段两端。全部PCR 产物采用Axy Prep DNA 凝胶回收试剂盒进行回收,并用FTC-3000TMReal-Time PCR 仪进行荧光定量,均一化混匀后完成文库构建,在IlluminaMiSeq 2×300 bp平台上完成测序。
PCR反应体系为:4 μL 5×FastPfu Buffer,2 μL 2.5 mmol/L dNTPs,0.8 μL 5 μmol/L Forward Primer,0.8 μL 5 μmol/L Reverse Primer,0.4 μL FastPfu Polymerase,0.2 μL BSA,10 ng Template DNA补dd H2O至20 μL。程序如下:95 ℃预变性5 min,95 ℃变性60 s,50 ℃退火60 s,72 ℃延伸60 s,循环数为25,72 ℃最终延伸7 min。扩增结束后,采用2%琼脂糖凝胶电泳检测扩增产物的纯度。使用凝胶回收试剂盒切胶回收PCR产物进行文库构建后,在IlluminaMiSeq PE 300平台上进行高通量测序分析。
1.5 数据处理及分析
使用QIIME 中的PyNAST[20]对代表性序列进行比对并去除低质量、接头污染和低复杂的序列,用FLASH[21]对有效序列进行拼接,根据序列首尾两端的barcode 和引物序列区分样品。再用UPARSE[22]软件将序列聚类到97%的操作分类单位(operational taxonomic unit,OTU)[23]。筛选重复序列,使用Uchime软件去除嵌合体[24]。所有数据分析使用R语言[25]和Python(https://www.python.org/)进行。结合热图或聚类分析计算物种丰度与环境因子的相关性及其显著性[26]。
2 结果与分析
2.1 样品ITS1基因序列质量评估及Alpha多样性分析
26个样品测序共获得 2 059 240对有效的基因序列条带,平均每个样品产生35 455条有效序列。为了得到更高质量及更准确的生物分析结果,对双端 Reads拼接、过滤后共产生1 936 392条优化序列,每个样品平均产生74 477条优化序列。
根据97%相似性水平下的OTU信息,采用Alpha多样性指标的Chao1、Ace、Shannon、Simpson 指数以及群落覆盖度指数Coverage等[27]对样品中物种丰度和多样性进行评估,结果如表2所示。所有样品的OTU覆盖率为99.98%~100.00%,说明样品中真菌种类几乎都能被检测到,测序结果可以很好的反应样品中真菌的真实情况。OTU数量越多说明样品中的真菌丰度越高[28],由表2可知,样品G1、G2、J1、J2、L1和L2的OTU数量在100以上,其他样品中的OTU数量基本都在100以下。这说明G1、G2、J1、J2、L1和L2这6个样品中真菌种类较多,其中G1、G2、J1、J2源自于富蕴县,L1和L2属于巴里坤地区,这可能是由于当地环境所引起的。来源于富蕴县的4个样品均采自水草丰富、环境相对较好的牧区,微生物更加容易生长,因此真菌的种类相对较高。来源于巴里坤山区的2个风干牛肉样品采自牧民家,这可能是和牧民的制作方式有关,卫生条件直接关系到产品中的微生物情况。图1-a表示真菌群落OTU 数目的Venn
图,该图可用于统计多个样本中共有和独有的OTU 数目,4组共有的OTU 数目84个,26个样品种共有的OUT数目仅有13个(图1-b),其中塔城(Group1)、额敏(Group2)、富蕴(Group3)和巴里坤(Group4)地区样品中OTU数目分别为125、133、261和316, Group1和 Group2之间差异不显著(P>0.05),其他组之间差异显著(P<0.05)。
表2 Alpha多样性指数统计
Table 2 Alpha diversity index statistics
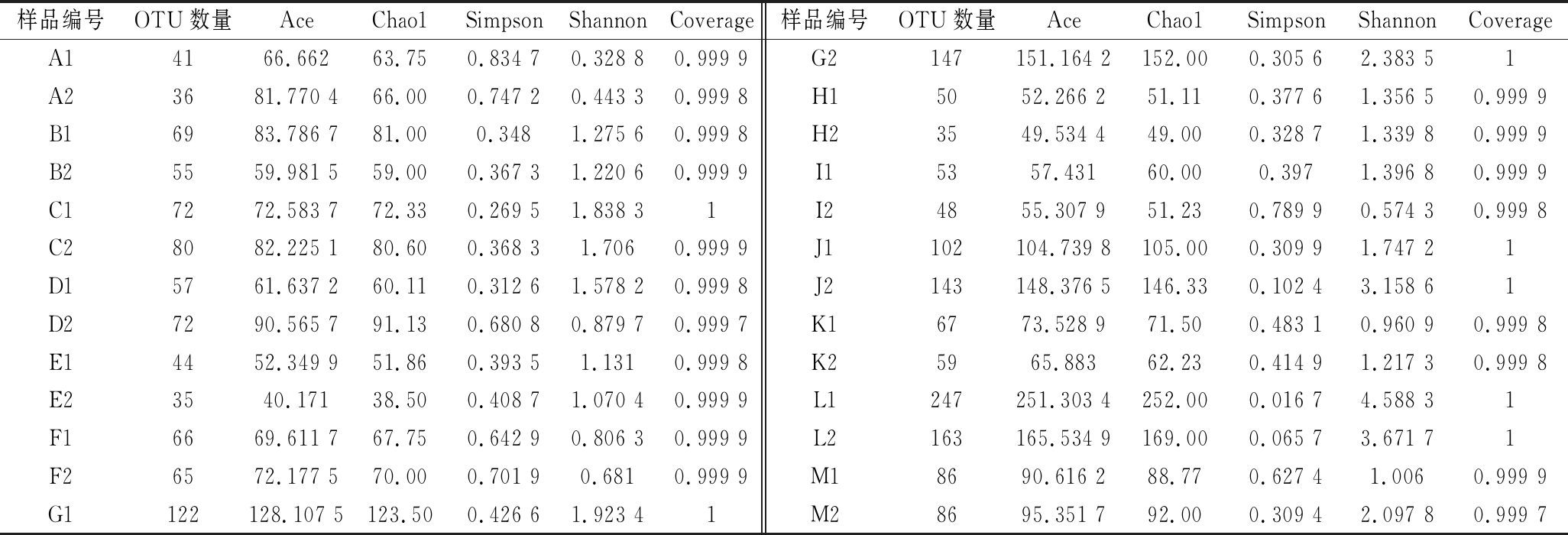
样品编号OTU数量AceChao1SimpsonShannonCoverage样品编号OTU数量AceChao1SimpsonShannonCoverageA14166.66263.750.834 70.328 80.999 9G2147151.164 2152.000.305 62.383 51A23681.770 466.000.747 20.443 30.999 8H15052.266 251.110.377 61.356 50.999 9B16983.786 781.000.3481.275 60.999 8H23549.534 449.000.328 71.339 80.999 9B25559.981 559.000.367 31.220 60.999 9I15357.43160.000.3971.396 80.999 9C17272.583 772.330.269 51.838 31I24855.307 951.230.789 90.574 30.999 8C28082.225 180.600.368 31.7060.999 9J1102104.739 8105.000.309 91.747 21D15761.637 260.110.312 61.578 20.999 8J2143148.376 5146.330.102 43.158 61D27290.565 791.130.680 80.879 70.999 7K16773.528 971.500.483 10.960 90.999 8E14452.349 951.860.393 51.1310.999 8K25965.88362.230.414 91.217 30.999 8E23540.17138.500.408 71.070 40.999 9L1247251.303 4252.000.016 74.588 31F16669.611 767.750.642 90.806 30.999 9L2163165.534 9169.000.065 73.671 71F26572.177 570.000.701 90.6810.999 9M18690.616 288.770.627 41.0060.999 9G1122128.107 5123.500.426 61.923 41M28695.351 792.000.309 42.097 80.999 7
稀释曲线反映了持续抽样下新OTU(新物种)出现的速率;在一定范围内,随着测序条数的加大,若曲线表现为急剧上升则表示群落中有大量物种被发现;当曲线趋于平缓,则表示此环境中的物种并不会随测序数量的增加而显著增多(图2-a)。Shannon指数越大,则表示OTU种类越多,物种越丰富,表明样品中已涵盖绝大多数的微生物信息,当曲线趋于平坦时,说明测序数据量足够大,OTU种类不会再随着测序量的增加而增加。由图2-b 可知,对26种风干肉制品中的真菌进行的高通量测序分析表明,当测序量<40 000时,OTU数呈明显上升趋势,表明此时仍未检测到更多种类的菌种。当测序数量逐渐增加时,样品的OTU数量增加,但趋势趋于平稳。当测序深度达到60 000时,所有样品的OTU变化均趋于平稳。随着测序量的增加,真菌的多样性几乎不变,这表明现有的测序量已经可以反映样品中真菌的丰度信息。采用随机抽样的方法对序列进行测序,以绘制出绘制的序列数和可代表的OTU数的曲线。即可知多数样本在序列数接近60 000时,OTU已经接近饱和,说明现有测序量已经可以反映出样品中真菌丰度信息。

图1 样品中真菌群落OTU的Venn图
Fig.1 Venn diagram of OTU of fungal community in samples
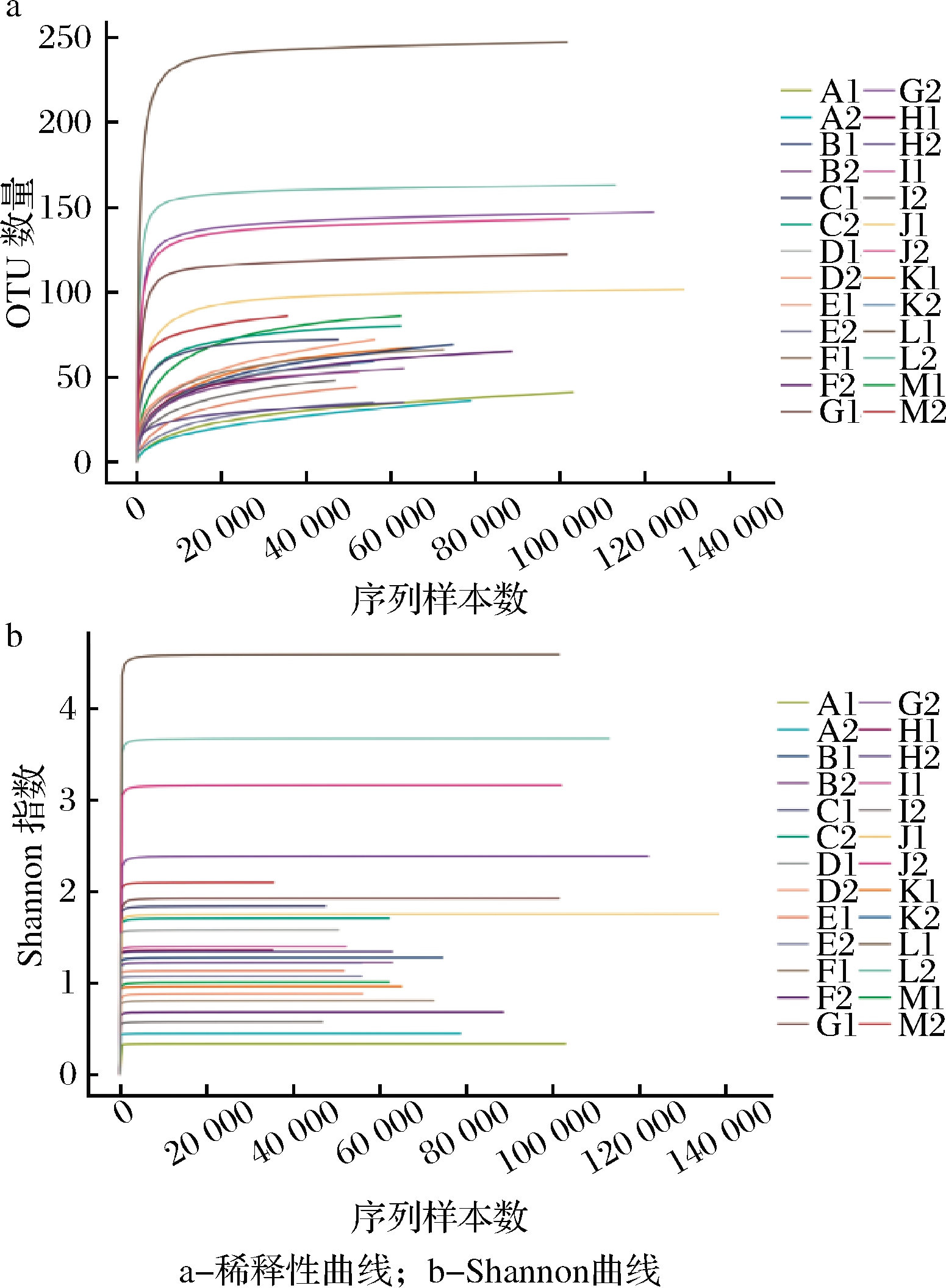
图2 样品稀释性、Shannon指数曲线
Fig.2 Sample dilution and Shannon index curve
2.2 不同来源风干肉中真菌群落组成分析
根据分类学分析结果,可以得到不同样品在不同分类水平上的群落组成情况。图3-a表示的是在门水平下,样品中真菌丰度在0.1%以上的真菌用群落组成表示,丰度<0.1%的真菌用Other表示。由图3-a可知,样品中的优势真菌门有子囊菌门(Ascomycota)73.13%、担子菌门(Basidiomycota)24.77%和被孢霉菌门(Mortierellomycota)2.76%。这3个菌门真菌含量占到测序序列总数的99%。不同地区风干肉中真菌主要优势菌群略有差异,塔城地区优势真菌门为Basidiomycota,占该组真菌菌群的50.65%,而其他3个地区(额敏、富蕴、巴里坤)主要优势真菌门为Ascomycota,分别占该组真菌菌群的70.33%、85.65%和80.47%。高庆超等[29]研究发现,同属于自然发酵的黑果枸杞酵素,通过高通量测序在门分类水平下,子囊菌门(Ascomycota)、担子菌门(Basidiomycota)占整个真菌门水平的99%。陈新[30]在对新疆新疆天山雪岭土壤中微生物种群的研究中也发现,土壤真菌中被孢霉菌门(Mortierellomycota)、担子菌门(Basidiomycota)和子囊菌门(Ascomycota)的相对丰度约占总和的70%,属于优势种群。以上结果可以看出,虽然研究对象不同,但真菌群落结构和优势菌群的差异不大,这是因为自然发酵产品中的微生物主要来自于当地的土壤、空气和水,在门的分类水平上来看,自然发酵食品的真菌类型和土壤当中的真菌基本一致。

图3 各样品在门、属水平下真菌群落结构分布
Fig.3 Relative abundance of the major fungus phylum and genus in samples
不同样品中真菌在分类属水平上的分布情况如图3-b所示。在26份风干肉样品中共鉴定出32个属,相对含量>1%的10个属分别为假丝酵母属(Candida) 30.09%、Mrakia16.26%、Unclassified 15.82%、德巴利酵母属(Debaryomyces)11.14%、球腔菌属(Mycosphaere)6.37%、青霉属(Penicillium)4.23%、曲霉属(Aspergillus)2.39%、(Mrakiella)2.88%、Naganishia1.71%和维希尼克氏酵母(Vishniacozyma)1.55%。其中,塔城牛肉(A1、A2)相较于其他几个样品所含真菌属比较单一,优势真菌属为Mrakia,占到85%左右。额敏牛肉(F1、F2)和托里牛肉(K1、K2)在真菌属组成较为相似。不同地区风干肉中真菌主要优势菌群不同,样品塔城牛肉(C1、C2)中优势真菌属为青霉属(Penicillium),占40%左右;样品富蕴牛肉(G1、G2)中优势真菌属为德巴利酵母属(Debaryomyces),占60%左右;样品巴里坤马肉(M1、M2)中优势真菌属为球腔菌属(Mycosphaere),占65%左右。不同地区真菌在属水平上有着一定差异(图4-b),塔城地区和额敏地区菌群结构及种类比较类似,这可能是由于塔城和额敏都属于塔额盆地,地理环境和气候都比较类似。其他2个地区样品中真菌多样性则更加丰富,可能是由于这2个地区都属于山区牧区,降水量多,物种相对丰富,因此,菌群多样性也更加丰富。
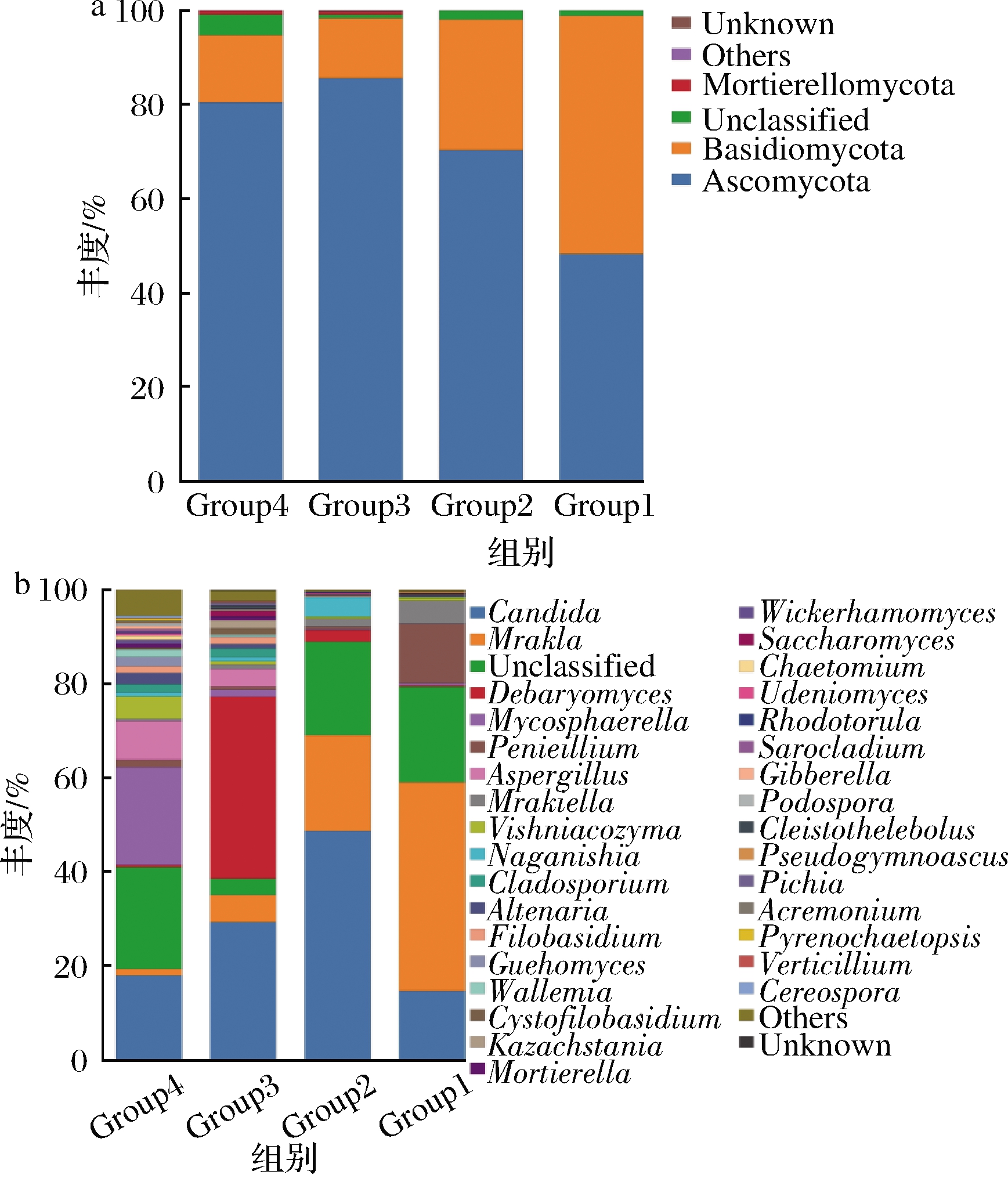
a-门水平;b-属水平
图4 四组样品在门、属水平下真菌群落结构分布
Fig.4 Relative abundance of the major fungus phylum and genus in four groups of samples
有研究表明,汉逊德巴利酵母(Dabaryomyces)和假丝酵母(Candida)是发酵肉制品的生产中最常用的酵母菌[31],因为其可促进发酵香肠的呈色,改善发酵香肠的风味,另外,有些酵母菌还能抑制腐败菌的滋生[32],赵永强等[33]用米曲霉和鲁氏酵母对合浦珠母贝肉进行发酵发现,其可以有效改善产品风味及游离氨基酸含量。另外,文开勇等[34]对四川传统腊肉研究发现,曲霉属(Aspergillus)为四川腊肉中优势真菌属。发酵肉在长时间的发酵过程中,真菌的生长繁殖可以产生多种酶类化合物,将肉中大分子的蛋白质、脂肪分解成小分子的醇酮醛等化合物,并能增加氨基酸的种类和游离脂肪酸的形成,促使亚油酸转化成共轭亚油酸,有利于风干肉中特殊风味的形成,显著提升发酵肉的风味品质和营养价值[35]。另外,酵母菌和霉菌还可以促进发酵肉中乳酸菌的生长,加速肉制品中水分含量的降低,并且能够降低风干肉的酸度,抑制发酵肉中脂质氧化、过氧化物的生成及腐败微生物生长繁殖,从而提高风干肉的安全性能[36-37]。本实验中优势的真菌属有假丝酵母属(Candida,30.09%)、德巴利酵母属(Debaryomyces,11.14%)和曲霉属(Aspergillus,2.39%),说明这3个属的真菌在风干肉的发酵过程中属于优势菌群,对风干肉品质形成有一定的影响。
为了能够更加直观的看出不同来源不同样品之间真菌属水平的相似程度,通过物种丰度聚类热图(Heatmap)表示(图5)。Heatmap是一颜色梯度来代表数据矩阵中数值的大小,并根据物种丰度相似性进行聚类,将高丰度和低丰度的物种分类聚集,通过颜色梯度及相似程度反映多个样品群落组成的相似性和差异性。由图5可知,在真菌属水平,Group1、Group2、Group3和Group4中样品的聚类分析结果相互交叉,并有差异,其中L2、J2和D1样品和其他样品的群落组成差异较大,剩下的其他样品之间菌群聚类结果比较相似。

图5 样品在属水平物种丰度聚类热图
Fig.5 Cluster heatmap of species abundance at genus level
26个样品都属于哈萨克族传统手工制作,其中4个是马肉,22个是牛肉,全部没有添加发酵剂,都属于自然发酵,但由于制作地区环境、风干温度以及制作人员的操作不同等原因导致了不同样品的真菌群落组成差异。实验结果表明,风干马肉和风干牛肉菌群多样性差异不大。从PCoA图可以看出(图6),26 个样品的真菌群落组成大致可以分为2 个类型,其中Group1和Group2样品分布相对较集中,说明这2组样品真菌群落结构比较相近,这与图5的结果相互印证。Group3和Group4组中样品分布在2个不同的位置,且相互之间距离较远,其中Group3组中J1、J2和G1、G2分别来自于富蕴的牧区,而其他样品则是山区,地理环境造成了其真菌菌群的差异。Group4组中K1、K2和其他样品真菌群落差异较大则是因为L1、L2和M1、M2采自于临近村落的2种不同的原料肉,而K1、K2则是采自于另外较远的村庄。
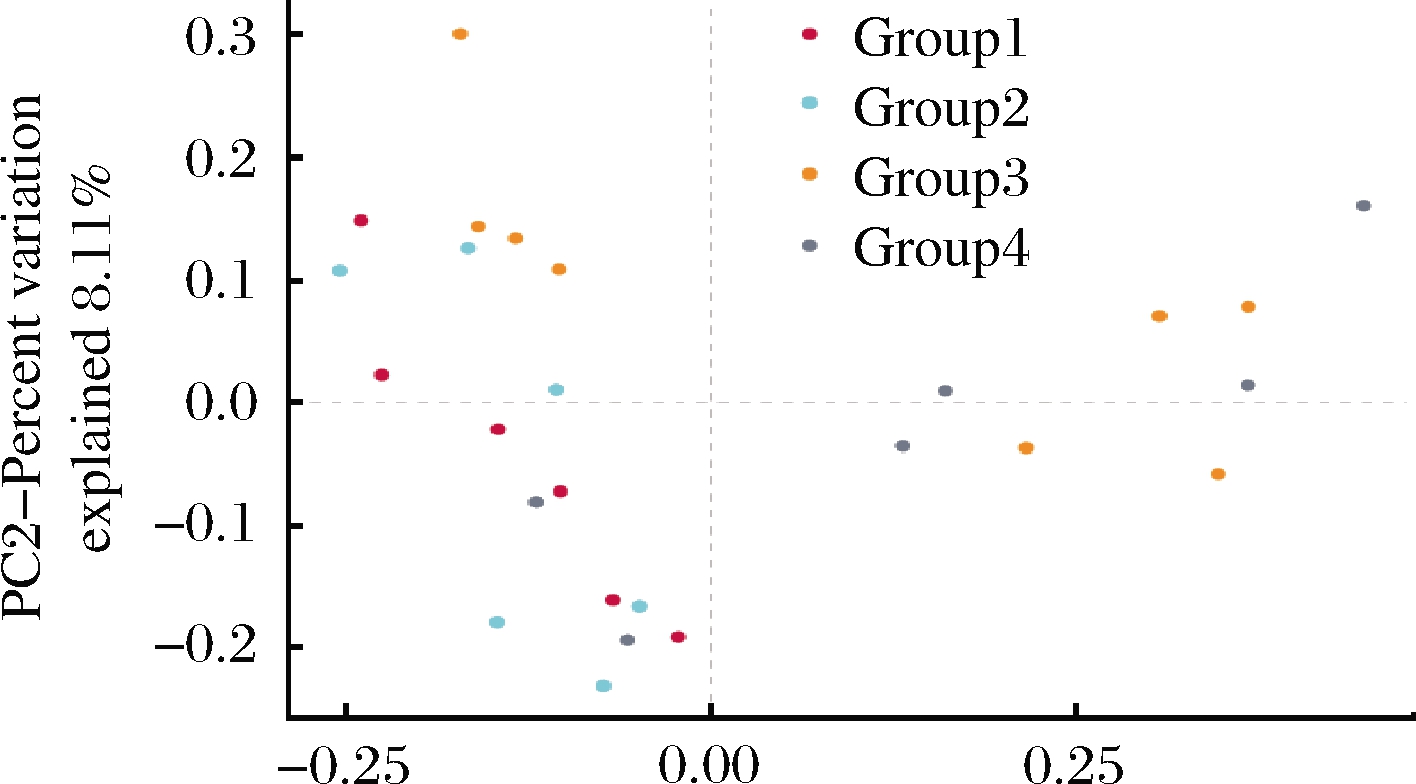
图6 基于weighted UniFrac的不同样品PCoA分析图
Fig.6 PCoA diagram of different samples at genus level based on weighted UniFrac
3 结论
本研究采用IlluminaMiSeq第二代测序技术研究了新疆北疆4个地区26个传统风干肉中真菌多样性,并结合高通量测序结果揭示了风干肉中真菌的多样性,不仅可以检测到可培养的真菌,而且还检测出了大量的不可培养真菌,尤其是对自然发酵肉制品,可以更加全面的反应样品中的菌群多样性。
高通量结果表明,4组26 个样品测序共获得真菌原始序列 2 059 240条,过滤后得到的有效序列为 1 936 392 条。26份风干肉样品中真菌在真菌分类门水平上共鉴定出5个门,平均丰度值>1%的3个真菌门分别为:子囊菌门(Ascomycota,73.13%)、担子菌门(Basidiomycota,24.77%)和被孢霉门(Mortierellomycota,2.76%)。这3个菌门真菌含量占到测序序列总数的99%。不同地区风干肉中真菌主要优势菌群不同,塔城地区优势真菌门为Basidiomycota,而其他3个地区(额敏、富蕴和巴里坤)主要优势真菌门为Ascomycota。另外,在26份风干肉样品中共鉴定出32个属,相对含量>1%的属分别为假丝酵母属(Candida,30.09%)、Mrakia 16.26%、德巴利酵母属(Debaryomyces,11.14%)、球腔菌属(Mycosphaere,6.37%)、青霉属(Penicillium,4.23%)、曲霉属(Aspergillus,2.39%)、(Mrakiella) 2.88%、Naganishia 1.71%和维希尼克氏酵母(Vishniacozyma,1.55%)。丰度聚类热图和Beta多样性分析表明,L2、J2和D1样品和其他样品的群落组成差异较大。
[1] 刘娅,唐慧.浅析新疆传统食品的工业化[J].食品科技,2007,28(10):18-21.
LIU Y, TANG H.Industriaiation of Xinjiang traditiona1 food[J].Food Science and Technology,2007,28(10):18-21.
[2] 竺尚武,杨耀寰,王锡渊,等.金华火腿口味及呈味物质的研究[J].食品科学,1993,14(3):8-11.
ZHU S W,YANG Y H,WANG X Y,et al.Investigation on tastes and taste compounds of jin-huaham[J].Food Science,1993,14(3):8-11.
[3] KABAN G.Changes in the composition of volatile compounds and in microbiological and physicochemical parameters during past rma processing[J].Meat science,2009,82(1):17-23.
rma processing[J].Meat science,2009,82(1):17-23.
[4] PALEARI M A,BERETTA G,COLOMBO F,et al.Buffalo meat as a salted and cured product[J].Meat Science,2000,54(4):365-367.
[5] 王俊钢,李宇辉,郭安民,等.新疆风干牛肉成熟过程中理化及微生物特性分析[J].食品与发酵工业,2016,42(10):129-133.
WANG J G,LI Y H,GUO A M,et al.Physico-chemical and microbial properties of Xinjiang dry-cured beef during the ripening process[J].Food and Fermentation Industries,2016,42(10):129-133.
[6] GAO P,WANG W,JIANG Q,et al.Effect of autochthonous starter cultures on the volatile flavour compounds of Chinese traditional fermented fish (Suan yu)[J].International Journal of Food Science and Technology,2016,51(7):1 630-1 637.
[7] GAO P,WANG W,XIA W,et al.Lipolysis and lipid oxidation caused by Staphylococcus xylosus 135 and Saccharomyces cerevisiae 31 isolated from Suan yu,a traditional Chinese low-salt fermented fish[J].International Journal of Food Science and Technology,2016,51(2):419-426.
[8] 陈东华,范东翠.发酵肉制品风味的研究进展[J].农产品加工,2008(3):63-65.
CHEN D H,FAN D C.Research progress on flavor of fermented meat products[J].Journal of Agricultural Products Processing,2008(3):63-65.
[9] 鄯晋晓,盛占武.发酵肉制品中微生物的作用[J].肉类工业,2007(2):15-18.
SHAN J X,SHENG Z W.The function of microorganisms in fermented meat products[J].Meat Research,2007(2):15-18.
[10] FLORES M,DUR M A,MARCO A,et al.Effect of Debaryomyces spp.on aroma formation and sensory quality of dry-fermented sausages[J].Meat Science,2004,68(3):439-46.
[11] SIRINE,BEN,SLIMA,et al.Effect of partial replacement of nitrite with a novel probiotic Lactobacillus plantarum TN8 on color,physico-chemical,texture and microbiological properties of beef sausages[J].LWT-Food Science and Technology,2017,86:219-226.
[12] NAM Y D,LEE S Y,LIM S I.Microbial community analysis of Korean soybean pastes by next-generation sequencing[J].International Journal of Food Microbiology,2012,155(1-2):36-42.
[13] NAM Y D,PARK S L,LIM S I.Microbial composition of the korean traditional food “kochujang” analyzed by a massive sequencing technique[J].Journal of Food Science,2012,77(4):250-256.
[14] NAM Y D,YI S H,LIM S I.Bacterial diversity of cheonggukjang,a traditional Korean fermented food,analyzed by barcoded pyrosequencing[J].Food Control,2012,28(1):135-142.
[15] HARCOMBE W R,RIEHL W J,DUKOVSKI I,et al.Metabolic resource allocation in individual microbes determines ecosystem interactions and spatial dynamics[J].Cell Reports,2014,7(4):1 104-1 115.
[16] GILLES K,BERNARD T,GEORGES D,et al.Metagenomic insights into the dynamics of microbial communities in food[J].International Journal of Food Microbiology,2015,213:31-39.
[17] QUIJADA N M,FILIPPIS F D,JAVIER S,et al.Different Lactobacillus populations dominate in “Chorizo de León” manufacturing performed in different production plants[J].Food Microbiology,2018,70:94-102.
[18] REBECCHI A,PISACANE V,MIRAGOLI F,et al.High-throughput assessment of bacterial ecology in hog,cow and ovine casings used in sausages production[J].International Journal of Food Microbiology,2015,212(6):49-59.
[19] 田建军,张开屏,杨明阳,等.应用Illumina MiSeq测序技术比较风干肉中细菌多样性和微生物安全性[J].食品科学,2019,40(8):33-40.
TIAN J J,ZHANG K P,YANG M Y,et al.Comparative bacterial diversity analysis and microbial safety assessment of air-dried meat products by Illumina MiSeq sequencing technology[J].Food science,2019,40(8):33-40.
[20] TUOMISTO H.A diversity of beta diversities:Straightening up a concept gone awry.Part 1.Defining beta diversity as a function of alpha and gamma diversity[J].Ecography,2010,33(1):2-22.
[21] TANJA M,SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2 957-2 963.
[22] EDGAR R.C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-1 006.
[23] OBERAUNER L,ZACHOW C,LACKNER S,et al.The ignored diversity:Complex bacterial communities in intensive care units revealed by 16S pyrosequencing[J].Sci Rep,2013,3:1 413-1 425.
[24] BALDAUF S L.Phylogeny for the faint of heart:A tutorial[J].Trends in Genetics,2003,19(6):345-351.
[25] CORE R,R R D C T,TEAM R,et al.A language and environment for statistical computing[J].Computing,2015,1:12-21.
[26] XU J,LIAN F,ZHAO L,et al.Structural modulation of gut microbiota during alleviation of type 2 diabetes with a Chinese herbal formula[J].Isme Journal,2014,9(3):552-562.
[27] QIU Z,LI N,LU X,et al.Characterization of microbial community structure and metabolic potential using Illumina MiSeq platform during the black garlic processing[J].Food Research International,2018,106:428-438.
[28] ZHANG M,ZHANG M,ZHANG C,et al.Pattern extraction of structural responses of gut microbiota to rotavirus infection via multivariate statistical analysis of clone library data[J].FEMS Microbiology Ecology,2009,70(2):21-6.
[29] 高庆超,常应九,马蓉,等.黑果枸杞酵素自然发酵过程中微生物群落的动态变化[J].食品与发酵工业,2019,45(13):126-133.
GAO Q C,CHANG Y J,MA R,et al.Dynamic changes in microbial community during natural fermentation of Lycium Ruthenicum Murr.enzymes[J].Food and Fermentation Industries,2019,45(13):126-133.
[30] 陈新.季节性雪被下新疆天山雪岭云杉林凋落物分解与土壤微生物的关系[D].乌鲁木齐:新疆大学,2019.
CHEN X.Relationship between litter decomposition and soil microbes of Schrenk sprucein forest under seasonal snowfall in Tianshan mountains,Xinjiang[D].Urumqi:Xinjiang University,2019.
[31] BOLUMAR T,SANZ Y,ARISTOY M C,et al.Protease B from Debaryomyces hansenii:Purification and biochemical properties[J].International Journal of Food Microbiology,2005,98(2):167-177.
[32] DURA M A,FLORES M,TOLDRA F.Effect of growth phase and dry-cured sausage processing conditions on Debaryomyces spp.generation of volatile compounds from branched-chain amino acids[J].Food Chemistry,2004,86(3):391-399.
[33] 赵永强,王安凤,陈胜军,等.米曲霉和鲁氏酵母协同发酵优化合浦珠母贝肉酶解液风味[J].食品与发酵工业,2019,45(15):115-120.
ZHAO Y Q,WANG A F,CHEN S J,et al.Synergistic fermentation of Aspergillus oryzae and Saccharomyces rouxii to optimize enzymatic hydrolysate flavor of Pinctada fucata meat[J].Food and fermentation industries,2019,45(15):115-120.
[34] 文开勇,汪月,文鹏程,等.四川传统腊肉中微生物群落结构研究[J].食品与发酵工业,2020,46(3):36-42.
WEN K Y,WANG Y,WEN P C,et al.Study on microbial community structure in Sichuan traditional bacon[J].Food and fermentation industries,2020,46(3):36-42.
[35] CORRAL S, BELLOCH C, L PEZ-D
PEZ-D EZ,et al.Lipolysis and aroma generation as mechanisms involved in masking boar taint in sodium reduced fermented sausages inoculated with Debaryomyces hansenii yeast[J].Journal of the Science of Food and Agriculture,2018,98(4):2 121-2 130.
EZ,et al.Lipolysis and aroma generation as mechanisms involved in masking boar taint in sodium reduced fermented sausages inoculated with Debaryomyces hansenii yeast[J].Journal of the Science of Food and Agriculture,2018,98(4):2 121-2 130.
[36] 韦依侬,张涛,李苗苗,等.即食鱼糜制品贮藏过程中的品质稳定性研究[J].食品工业科技,2017,38(16):285-290.
WEI N N,ZHANG T,LI M M,et al.Study on quality stability of ready-to-eat surimi products during storage process[J].Science and Technology of Food Industry,2017,38(16):285-290.
[37] 史笑娜,黄峰,张良,等.红烧肉加工过程中脂肪降解氧化和挥发性风味物质的变化研究[J].现代食品科技,2017,33(3):257-265.
SHI X N,HUANG F,ZHANG L,et al.Changes in lipolysis,lipid oxidation,and volatile flavor compounds during the processing of red braised pork[J].Modern Food Science and Technology,2017,33(3):257-265.