近年来,近平滑假丝酵母Candida parapsilosis及其来源的羰基还原酶CpCR因广泛的底物谱和优良的催化特性,在香精、功能食品和药物的研发中得到广泛应用[1]。常用于生物催化的近平滑假丝酵母有IFO 0708[2]、CDC317[3]、CCTCC M203011[4-5]和ATCC 7330[6]等优势菌株,均具有自己的底物谱,其中ATCC 7330的底物适应性最强,不仅对4-氯-3-酮基-丁酸乙酯(4-chlor-3-keto-butyrate-ethyl ester,COBE)具有优良的还原能力[7],而且对苯基取代的二酮、酮醛、烯酮及烯胺的立体选择性也非常强[8-9],对芳基烯醇、烯醇酯及吡咯环、噻吩环取代的烯醇酯[10]也具有优良的动力学拆分能力。
羰基还原酶和醇脱氢酶一样,属于氧化还原酶类,广泛存在于细菌、真菌、酵母和动植物体内[11-15],以辅酶NAD(P)+或NAD(P)H作为电子受体和供体,可以特异性地催化酮 (醛) 和醇之间的相互转化,用于合成重要价值的羟基化合物[16-21]。2013年,AGGARWAL等[6]解析了近平滑假丝酵母Candida parapsilosis ATCC 7330的羰基还原酶CpCR的晶体结构,蛋白质数据库为4OAQ,但对该酶的高效表达和酶学性质研究较少[22-24]。
为了系统研究该酶的酶学性质,在本课题组前期研究的基础上[25-27],拟从近平滑假丝酵母C.parapsilosis ATCC 7330克隆出一个编码羰基还原酶CpCR的基因(cpcr),并在大肠杆菌中进行高效表达,利用表达载体中的His标签分离纯化重组酶CpCR,并对其酶学性质进行考察。本研究将为近平滑假丝酵母 C.parapsilosis ATCC 7330羰基还原酶CpCR的分子改造以及催化应用奠定科学基础。
1 材料与方法
1.1 材料与仪器
菌株 Candida parapsilosis ATCC 7330、大肠杆菌Escherichia coil BL21(DE3)由本实验室鉴定及保存;质粒载体 pACYCDuet-1,上海生工生物工程有限公司;Phusion DNA Polymerase、限制性酶切酶 Sal I 和Pst I,New England Biolabs;T4 连接酶、质粒抽提试剂盒、产物纯化试剂盒,Vazyme 生物技术有限公司;DL 5 000 bp Marker、蛋白质电泳 Marker、6×loading Buffer、5×蛋白上样缓冲液,TAKARA有限公司;异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG),Calbiochem 公司,纯度>99.9%;Ni-Agarose His标签蛋白纯化试剂盒,北京康为世纪生物科技有限公司;其他试剂为国产或进口分析纯;PCR 引物合成及测序由上海生工生物技术有限公司完成。
PCR仪(C1000 Touch)、Gel凝胶成像仪(Doc XR+),Bio-Rad公司;紫外可见分光光度计(UV1800),岛津企业管理有限公司;蒸汽灭菌器(SX-700)、落地高速冷冻离心机(MX-307),日本Tomy公司;超微量紫外可见分光光度计(Nano Drop One),Thermo公司;超净工作台(SW-CJ-2FD),苏州净化设备有限公司;恒温培养摇床(ZQLY-180S),上海知楚仪器有限公司;生化培养箱(LRH-250A),泰宏医疗器械有限公司。
1.2 实验方法
1.2.1 高效表达载体的构建
以近平滑假丝酵母ATCC 7330全基因组为模板,用引物:pACYC-F:5′-GCCTGCAG(Pst I)ATGACTAAAGCAGTACCAGACA-3′;pACYC-R2:5′-TGTCGAC(Sal I)TAAGCTTTGAATGCTTTGTCG-3′,扩增CpCR基因。反应体系:0.5 μL基因组 DNA 模板,0.5 μL Phusion DNA polymerase,10 μL Phusion GC Buffer(5×),2.5 μL pACYC-F(10 μmol/L),2.5 μL pACYC-R2(10 μmol/L),1 μL dNTP(10 μmol/L),1.5 μL DMSO,1.5 μL Mg2+,30 μL ddH2O。扩增条件:98 ℃ 预变性 30 s,98℃ 变性 10 s,53 ℃ 退火20 s,72 ℃ 延伸 45 s,30 个循环,最后 75 ℃ 保温5 min。
CpCR扩增产物经限制性内切酶Pst I 和 Sal I 双酶切后定向克隆至表达载体 pACYCDuet-1,并转化 E. coli BL21(DE3)感受态细胞。转化后,取适量菌液涂布在含 100 μg/mL 氯霉素的 LB 平板上,37 ℃避光培养,筛选阳性转化子。培养提取质粒,经测序验证后,获得克隆载体,命名为pACYCDuet-1-cpcr(如图 1)。
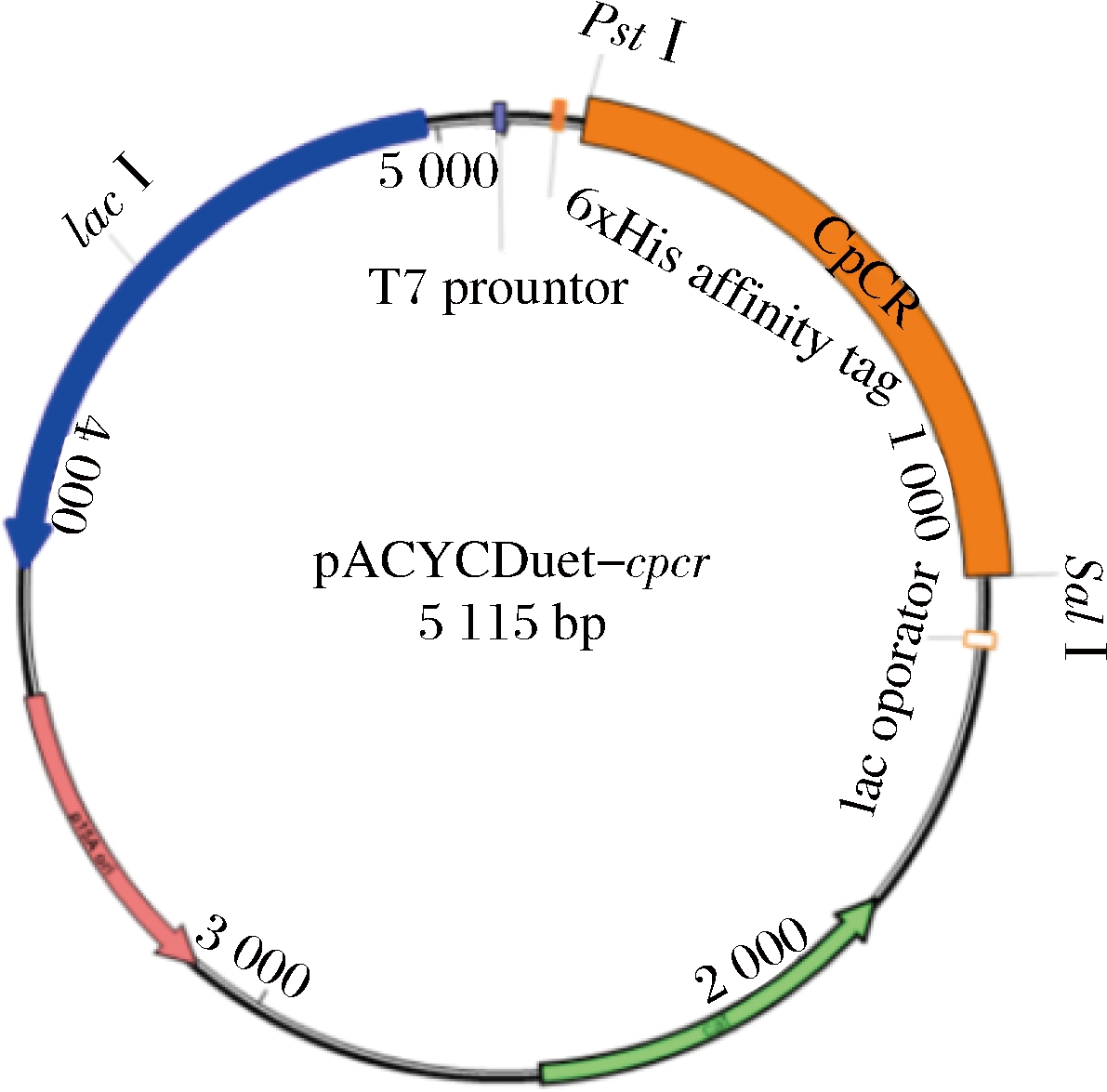
图1 重组表达载体 pACYCDuet-1-cpcr 结构示意图
Fig.1 Structural diagram of recombinant expression vector pACYCDuet-1-cpcr
1.2.2 羰基还原酶CpCR目的基因(cpcr)的诱导表达与纯化
挑取含有重组表达质粒的单菌落于含100 μg/mL氯霉素的 LB 培养基中过夜培养。按OD600值为4的比例接种至100 mL诱导培养基(100 μg/mL氯霉素,1 mmol/L Mg2+、1 mmol/L Zn2+)中,23 ℃、200 r/min 条件下培养至OD600为0.6,加入IPTG使终浓度为 0.4 mmol/L,23 ℃诱导16 h。同时与空载体与未诱导大肠杆菌做对照。离心收集菌体,用适当体积的Buffer A (20 mmol/L Tris,500 mmol/L NaCl,5%甘油,0.5 mmol/L PMSF,pH 7.5) 重悬后,超声破碎25 min(工作2 s,停 6 s),4 ℃,12 000 r/mim离心10 min取上清液。分别以上清液、沉淀为蛋白样品经十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sufate-polyacrylamide gel electrophoresis,SDS-PAGE)检测,记录并分析结果。
粗蛋白使用Ni-Agarose柱分离纯化,将具有His标签的目的蛋白上清液经 0.45 μm 滤膜过滤后上柱,分别用 10 倍柱体积Binding Buffer(20 mmol/L Tris-HCl,10 mmol/L 咪唑,500 mmol/L NaCl,pH 8.0)进行洗杂,20 mL Elution Buffer(20 mmol/L Tris-HCl,500 mmol/L 咪唑,500 mmol/L NaCl,pH 8.0)洗脱,并按每 1 mL 流出液分管收集,保存于 4 ℃备用。蛋白样品经 SDS-PAGE检测,记录并分析结果,得到比酶活力高的重组酶CpCR。
1.2.3 蛋白的分析与测定
重组蛋白分析采用SDS-PAGE[18],浓缩胶 5%,分离胶 12%,考马斯亮蓝 R-250 染色显示蛋白条带;蛋白浓度采用 Bradford 法[28]测定。
1.2.4 重组酶CpCR酶活力测定
羰基还原酶的酶活力检测采用分光光度计法。测定方法:总反应体积为 1 mL,包含750 μL 100 mmol/L PB Buffer(pH 7.5),100 μL 40 mmol/L苯甲醛,100 μL 2 mmol/L NADPH,30 ℃ 温育 3 min,最后加入50 μL酶液,用紫外分光光度计在340 nm条件下测定吸光度值的变化。酶活力定义:在30 ℃、pH 7.5条件下,每1 min催化氧化1 μmol NADPH 所需要的酶量定义为1 U。并按公式(1)计算酶活力:
酶活力![]()
(1)
式中:ΔA为初速度范围内每分钟吸光度值差值,min-1;V总为总反应体积,mL;n为酶液稀释倍数;6.22为NADPH的消光系数,mmol/(L·cm);d为比色皿直径,1 cm;V为酶液添加体积,mL。
1.2.5 重组酶CpCR的酶学性质研究
1.2.5.1 重组酶CpCR的温度稳定性考察和T50的测定
取适量酶液,采用pH 7.5 的缓冲液按照等体积比进行稀释,置于4~50 ℃ 条件下保温2 h,移取部分酶液,按照1.2.4小节的方法测定剩余酶活力,通过计算相对酶活力,考察该酶温度稳定性,测定该酶的T50值。T50的定义:酶的相对酶活力降低至初始酶活力的50%时所对应的温度。
1.2.5.2 重组酶CpCR的pH 稳定性考察
取适量酶液,分别加入pH 5~8的缓冲液中(pH 5~6采用醋酸-醋酸钠体系、pH 6~7.5采用磷酸-磷酸盐体系、pH 7.5~8采用Tris-HCl体系分别配制100 mmol/L缓冲液),30 ℃保温2 h,按照1.2.4小节的方法测定剩余酶活力,计算相对酶活力,考察该酶的 pH 稳定性。
1.2.5.3 金属离子对CpCR重组酶的影响
在反应体系中加入 1 mmol/L 和 5 mmol/L的8种常见金属离子(K+、Mg2+、Ca2+、Fe2+、Ni2+、Cu2+、Zn2+、Mn2+)的氯盐,通过测定相对酶活力研究这些常见金属离子对重组酶CpCR的影响。
1.2.5.4 重组酶CpCR的动力学参数Km、Vmax、Kcat和催化特异性常数Kcat/Km测定
(1)重组酶CpCR对不同底物亲和力和催化特异性常数的考察
分别以苯甲醛、正丁醛、COBE等为底物,选用0.5、1.0、1.5、2.0、3.0和4.0 mmol/L等不同浓度,参考1.2.4小节的方法选用对应的底物测定吸光度变化值,以 Origin 8.0 对数据进行非线性拟合,按照米氏方程计算并确定该条件下的重组酶的动力学参数Km及Vmax值。依据1.2.3小节的方法测定酶的蛋白含量,结合文献[23]中蛋白质数据库信息和测定的相对分子质量,计算出催化常数Kcat及催化特异常数 Kcat/Km。比较重组酶CpCR对上述几种底物的亲和能力和催化效率。
(2)重组酶CpCR对辅酶NADPH和NADH亲和力考察
分别以NADPH和NADH在0.5、1.0、1.5、2.0、3.0和4.0 mmol/L等不同浓度下,固定苯甲醛浓度为4.0 mmol/L,测定催化反应的吸光度变化值,根据米氏方程确定该条件下的重组酶对2种不同辅助底物的动力学参数Km及Vmax值,比较重组酶 CpCR对辅酶NADPH和NADH的亲和力大小。
2 结果与分析
2.1 羰基还原酶CpCR基因的表达与纯化
2.1.1 目的基因PCR扩增产物的鉴定
以近平滑假丝酵母 ATCC 7330为模板,用引物pACYC-F和pACYC-R2扩增cpcr基因,结果如图2 所示,可见约 1 100 bp 的特异性条带,与预期条带一致,为1 107 bp。

M-DL 5 000 DNA Marker;泳道 1~2为 CpCR 酶基因
图2 扩增产物电泳图
Fig.2 Electrophoretic map of the PCR amplification products
2.1.2 重组表达载体pACYCDuet-1-cpcr 的鉴定
对筛选到的阳性克隆重组表达载体 pACYCDuet-1-cpcr 经Pst I 和 Sal I 双酶切,0.8% 琼脂糖凝胶电泳分析,结果如图3所示,可见约 4 kb的载体片段和1.1 kb的目的基因片段。将酶切验证正确的质粒送至上海生工公司测序,结果显示该羰基还原酶基因的序列全长为 1 107 bp,包含 368 个氨基酸的编码序列。
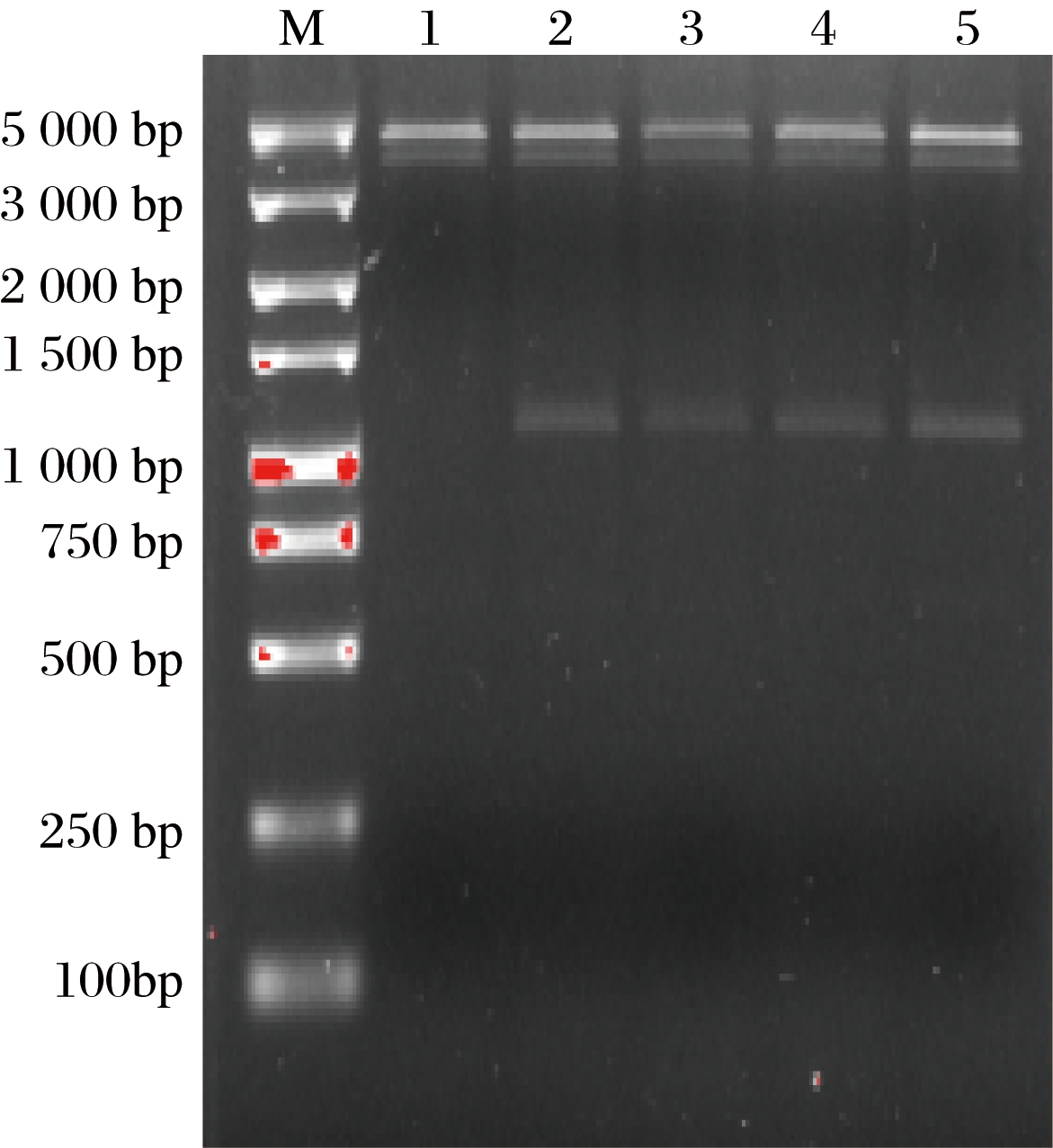
M-DL5 000 DNA Marker;泳道 1-完整重组质粒 pACYCDuet-1-cpcr;泳道 2~5-Pst I 和 Sal I双酶切重组质粒 pACYCDuet-1-cpcr
图3 重组表达载体 pACYCDuet-1-cpcr 的酶切电泳图谱
Fig.3 Digestion electrophoresis of recombinant expression plasmid pACYCDuet-1-cpcr
2.1.3 CpCR目的蛋白诱导表达及分离纯化
将经测序鉴定正确的表达载体 pACYCDuet-1-cpcr转化至宿主菌E.coli BL21(DE3)中诱导表达。收集湿菌体,超声破碎后进行SDS-PAGE电泳检测蛋白表达情况。由图4可知,空载质粒菌、未诱导空载质粒菌破碎液均未见目标蛋白;未诱导的重组菌的破碎上清液和沉淀中有目标蛋白,但含量很少;诱导的重组菌的破碎上清液和沉淀中均出现特异性条带,结合NCBI的CpCR酶的蛋白质数据,确定为 41 kDa左右,与预期目的蛋白分子量相近。由此可知,该羰基还原酶在宿主菌E.coli BL21(DE3)中表达成功,但有部分以包涵体形式不可溶表达。
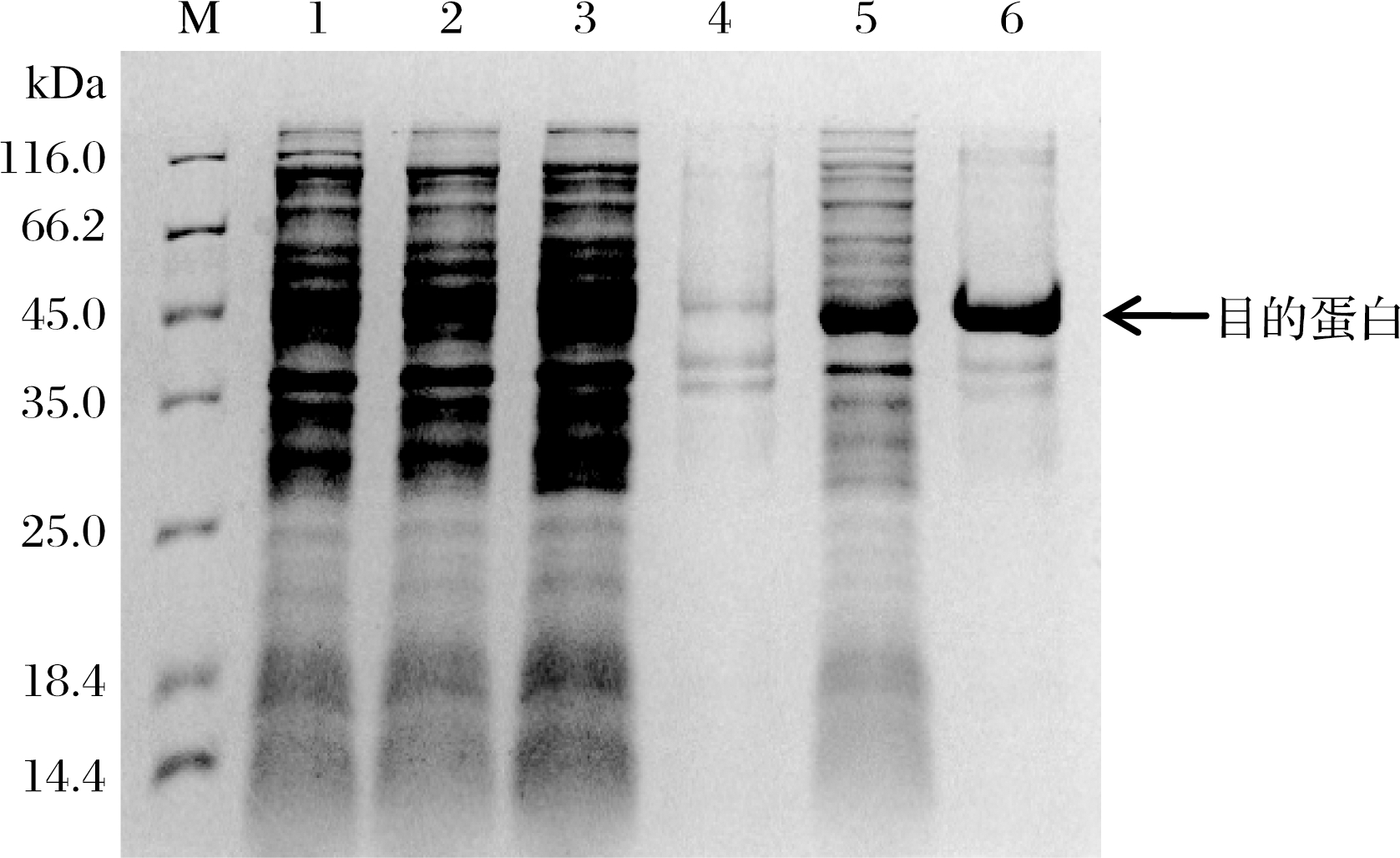
M-蛋白 Marker;1-空载质粒菌;2-未诱导空载质粒菌;3-未诱导重组菌破碎上清液;4-未诱导重组菌破碎沉淀;
5-诱导重组菌破碎上清液;6-诱导重组菌破碎沉淀
图4 目的蛋白CpCR的SDS-PAGE检测结果
Fig.4 SDS-PAGE analysis of the CpCR protein
采用重组菌 E.coli BL21(DE3)-pACYCDuet-1-cpcr 在 IPTG 诱导下进行异源表达含有 His 标签的羰基还原酶 CpCR,按照1.2.2小节的方法经 Ni-Agarose柱亲和层析纯化后,与重组菌菌体破壁上清液相比(图4),纯化后的目的蛋白条带较单一(图5泳道4)。洗脱液总蛋白含量为 8.3 mg,比酶活力为 20 U/mg,总酶活力为 168 U,总回收率为71%,纯化倍数为 5.5(表1)。本研究比文献[6]报道的利用谷胱甘肽(glutathione,GST)作为标签表达的重组酶CpCR分离纯化耗时少,效率高。
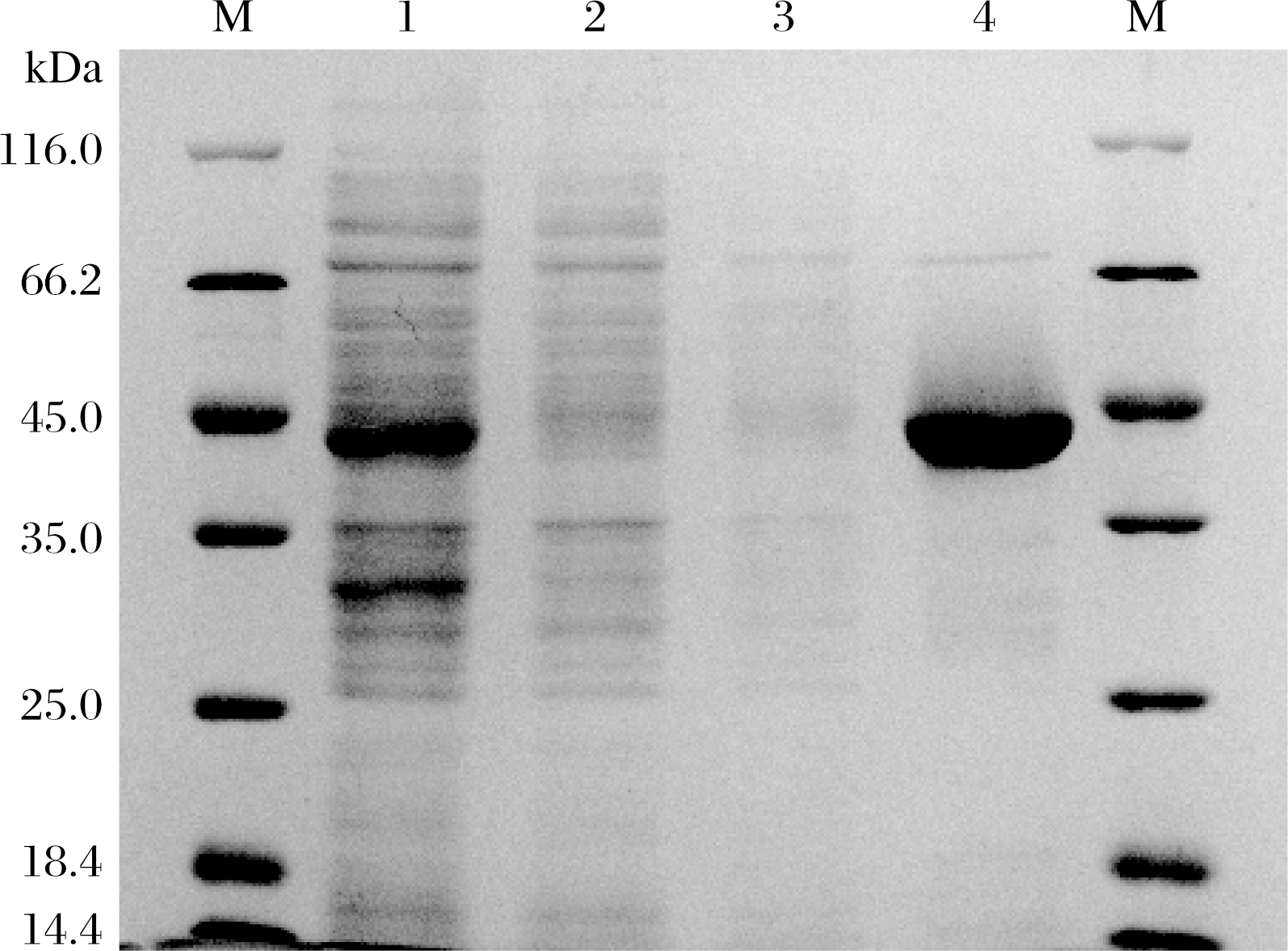
M-蛋白 Marker;1-粗酶液;2-镍柱流穿液;3-镍柱洗涤液;4-洗脱液
图5 重组蛋白纯化 SDS-PAGE 电泳
Fig.5 Purification of SDS-PAGE electrophoresis by recombinant protein
表1 重组酶 CpCR 的纯化
Table 1 Purification of recombinant enzyme CpCR

纯化步骤总酶活力/U总酶蛋白/mg比活力/(U·mg-1)纯化倍数得率/%粗酶液235663.61.0100洗脱液1688.319.85.571
2.2 重组酶CpCR的酶学性质研究
2.2.1 重组酶CpCR的温度稳定性考察
由图6可知,该酶在 4~35 ℃的温度范围稳定性很好,在33 ℃相对酶活力仍可保持在80% 左右。但是当温度超过35 ℃,相对酶活力迅速降低,37 ℃时,该酶的酶活力降低至初始酶的50%,故该酶的T50值为37 ℃。表明该酶在低温或者室温下保藏较好。
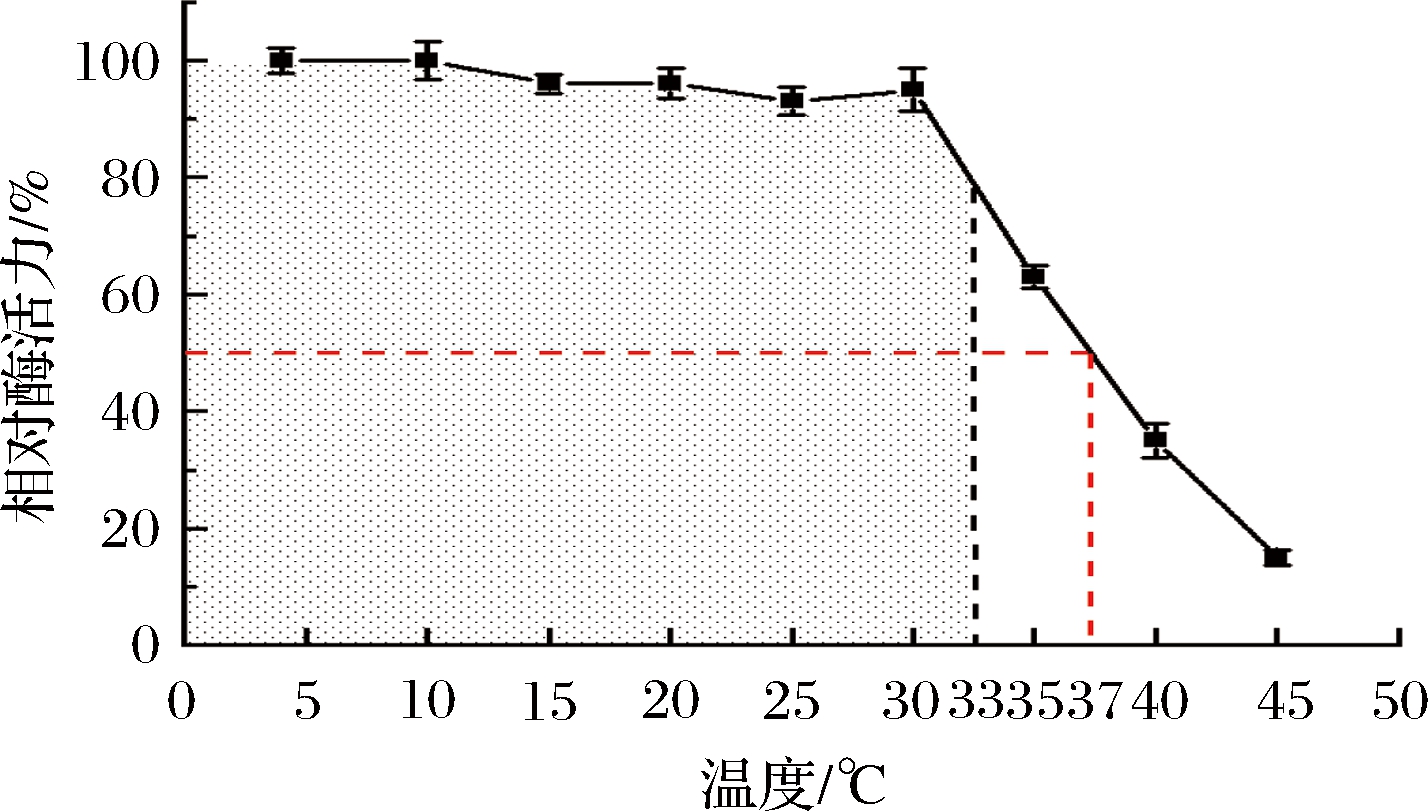
图6 重组羰基还原酶CpCR温度稳定性
Fig.6 Temperature stability of recombinantcarbonyl reductase CpCR
2.2.2 重组酶CpCR的pH稳定性考察
由图7可知,重组酶 CpCR在pH 6.2~7.5稳定性较好,相对酶活力保持在80%以上,中性条件下稳定性最好。但该酶在pH低于6和高于8条件下,其相对酶活力降低较快。因此该酶的pH范围相对较窄,在应用过程中要严格控制其催化体系的酸碱性。SUDAKARA等[22]报道来自该菌株的另一种羰基还原酶SRED,采用GST标签表达重组得到,其酶pH稳定性也较窄,其最稳定的pH值为5。重组酶CpCR耐酸碱范围小的特性为该酶的分子改造提供了依据。

图7 重组酶CpCR的pH稳定性
Fig.7 pH stability of recombinant carbonyl reductase CpCR
2.2.3 8种常见金属离子对重组酶CpCR的影响
很多羰基还原酶在催化氧化还原反应时需要金属离子的参与,有些金属离子对氧化还原酶具有一定的激活或者抑制作用。由图8 可知,羰基还原酶的酶活力在一定浓度下受金属离子的影响较大。在1 mmol/L低浓度下,除Ca2+和Cu2+外,其他6种的金属离子对重组酶CpCR 的酶活力影响较小;但在加入浓度为 5 mmol/L时,抑制作用明显,重组酶CpCR 相对酶活力降低最大的是Cu2+,几乎降低50%,其次是Ca2+、Fe2+的影响较大,相对酶活力降低到 60% 左右,Ni2+和Mn2+相对酶活力降低到70%左右,而Zn2+的相对酶活力仍然在80%以上。因此Cu2+对CpCR 酶的抑制作用最大,分析其原因可能是由于该羰基还原酶的活性中心含有2个半胱氨酸,分别在48位和167位,Cu2+可能会与酶分子中的半胱氨酸侧链上的巯基基团结合,从而导致酶活力降低。这表明该酶的应用环境要严格控制Cu2+的存在。本研究与SUDAKARA等[21]报道来自该菌株的重组酶SRED的金属离子特性基本一致,在1 mmol/L的Cu2+作用下,SRED相对酶活力下降到初始酶活力的67%。
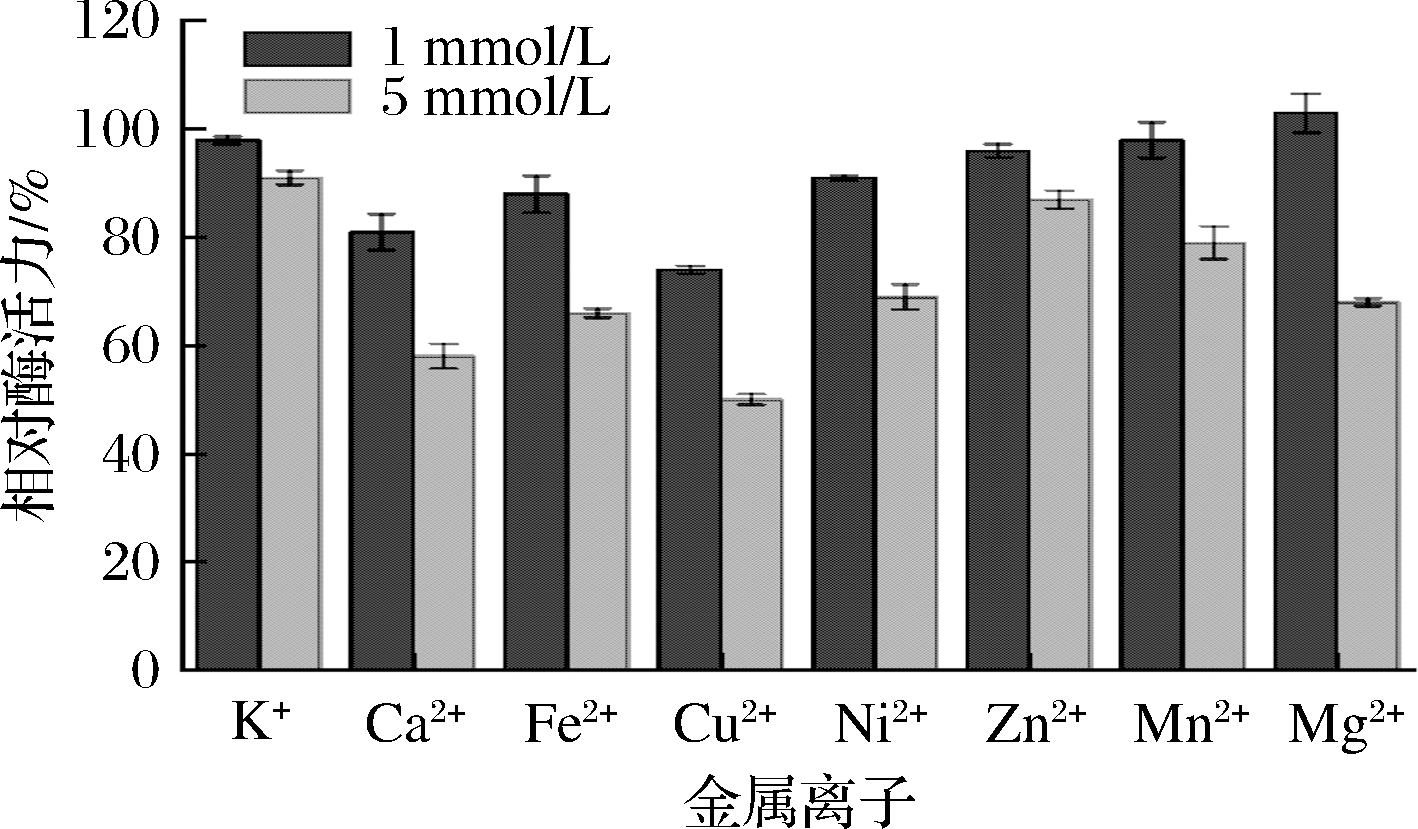
图8 金属离子对重组酶CpCR的酶活影响
Fig.8 Effects of metal ions on the activity of CpCR
2.2.4 重组酶CpCR对不同底物的动力学参数及催化特性参数的考察
由表2可知,重组酶CpCR对2种醛类物质的亲和力均很高,最大反应速度是阿伐他汀药物中间体COBE的7倍。Kcat表示单位时间内每一活性中心或每分子酶所能转换的底物分子数,反映了酶催化特定底物的能力大小。结果显示每个重组酶CpCR分子在1 s可以转化12个分子左右的醛类物质,而对COBE仅能转化近2个分子左右。Kcat/Km是衡量酶的催化效率参数,又称特异性常数,表示不同酶对特定底物的催化效率,或者表示同一种酶对不同底物的催化效率。从表2可以看出,对苯甲醛或正丁醛的催化效率是COBE的20倍左右。文献[23-24]也报道了利用N-端GST标签分离纯化的CpCR酶对苯甲醛、2,4-二氯苯甲醛的米氏常数Km和最大反应速度,和本研究基本一致。重组酶CpCR对苯甲醛、正丁醛亲和力较强,但本研究考察了重组酶CpCR对底物COBE动力学参数和3种底物催化特异性参数,进一步揭示了该重组酶CpCR的催化特性。
表2 重组酶 CpCR 对不同底物 的动力学参数和催化特性参数
Fig.2 The dynamics and catalytic parameters of the recombinant enzyme CpCR

底物Km/(mmol·L-1)Vmax/[μmol·(min·mg protein)-1]Kcat/s-1Kcat/Km[s-1·(mmol·L-1)-1]苯甲醛0.998.312.012.1正丁醛1.078.612.511.7COBE3.001.21.730.58
2.2.5 重组酶CpCR对两种辅酶NADPH和NADH的亲和力考察
按照1.2.5.4小节的方法,选取不同的NADPH和NADH的浓度开展重组酶初速度测定,根据初速度和辅酶浓度变化曲线,采用Origin 8.0 软件进行非线性拟合,得到NADPH的 Km为0.058 mmol/L,而在NADH条件下几乎不反应,说明该重组酶CpCR是NADPH依赖型。与文献[23]报道的GST标签重组CpCR酶依赖辅酶NADPH的结论一致。
3 结论
本实验成功克隆了近平滑假丝酵母 Candida parapsilosis ATCC 7330 中的羰基还原酶 CpCR 基因,并在大肠杆菌BL21(DE3)中实现了异源表达。结果表明,该羰基还原酶 CpCR 基因序列全长 1 107 bp,编码 368 个氨基酸,重组酶分子量大小在 41 kDa 左右。重组酶CpCR分离纯化后对底物苯甲醛的比酶活力为 20 U/mg。His标签的重组酶的酶学性质研究表明:重组酶CpCR在33 ℃以下稳定性较好,相对酶活力在80%以上,但随着温度的逐渐升高,酶活力下降较快;该重组酶CpCR的适宜pH范围在6.2~7.5,该酶在酸性和碱性条件下稳定性不好,可以通过蛋白质分子改造进一步提升其适应范围;该重组酶CpCR在K+、Mg2+、Ca2+、Fe2+、Ni2+、Cu2+、Zn2+和Mn2+常见金属离子中,在5 mmol/L条件下均有一定抑制,其中Cu2+对该酶的抑制作用最强,相对酶活力下降50%左右;通过该酶对底物的亲和力和催化特性的研究表明,该酶对底物苯甲醛、正丁醛亲和能力强于COBE,对苯甲醛或正丁醛的催化效率是对COBE的20倍左右;该重组酶CpCR是一种NADPH依赖型的羰基还原酶。本研究采用His标签的重组表达的羰基还原酶CpCR与GST标签的重组酶SRED相比在耐酸碱特性上有一定的差异,与GST标签的重组酶CpCR的酶学性质基本相同,但His标签的重组酶分离纯化操作更加方便,效率更高。
[1] CHADHA A,VENKATARAMAN S,et al.Candida parapsilosis:A versatile biocatalyst for organic oxidation-reduction reactions[J].Bioorganic Chemistry,2016,68(10):187-213.
[2] QIN H M,TANOKURA M,MIYAKAWA T,et al.Crystal structure of conjugated polyketone reductase (CPR-C1) from Candida parapsilosis IFO 0708 complexed with NADPH[J].Proteins,2013,81(11):2 059-2 063.
[3] WANG Q Y,SHEN L H,YE T T,et al.Overexpression and characterization of a novel (S)-specific extended short-chain dehydrogenase/reductase from Candida parapsilosis[ J].Bioresource Technology,2012,123(7):690-694.
[4] ZHANG RZ,WANG L,XU Y,et al.In situ expression of (R)-carbonyl reductase rebalancing an asymmetric pathway improves stereoconversion efficiency of racemic mixture to (S)-phenyl-1,2-ethanediol in Candida parapsilosis CCTCC M203011[J].Microbial Cell Factories ,2016,15:143-153.
[5] 许娜,王海燕,徐岩,等.近平滑假丝酵母 (R)-专一性羰基还原酶基因的克隆与表达[J].微生物学通报,2006,33(4):112-114.
XU N,WANG H Y XU Y,et al.Cloning and expression of gene encoding (R)-specific carbonyl reductase from Candida parapsilosis CCTCC M203011 in Escherichia coli[J].Microbiology China,2006,33(4):112-114.
[6] AGGARWAL N,MANDAL P K,GAUTHAM N,et al.Expression,purification,crystallization and preliminary X-ray diffraction analysis of carbonyl reductase from Candida parapsilosis ATCC 7330[J].ActaCrystallographica Section F-Structural Biology Communications,2013,69(2):313-315.
[7] BASKAR B,PANDIAN N G,PRIYA K,et al.Deracemisation of aryl substituted a-hydroxy esters using Candida parapsilosis ATCC 7330:Effect of substrate structure and mechanism[J].Tetrahedron,2005,61(52):12 296-12 306.
[8] STELLA S,CHADHA A.Biocatalytic reduction of α-keto amides to (R)-hydroxy amides using Candida parapsilosis ATCC 7330[J].Catalysis Today,2012,198(1):345-352.
[9] YAMAMOTO H,KAWADA N,MATSUYAMA A,et al.Cloning and expression in Escherichia coli of a gene coding for a secondary alcohol dehydrogenase from Candida parapsilosis[J].Bioscience,Biotechnology,and Biochemistry,1999,63(6):1 051-1 055.
[10] LI Z,WU Q Q,ZHU D M,et al.Highly enantioselective double reduction of phenylglyoxal to (R)-1-phenyl-1,2-ethanediol by one NADPH-dependent yeast carbonyl reductase with a broad substrate profile[J].Tetrahedron,2013,69:3 561-3 564.
[11] 聂尧,徐岩.羰基还原酶及其催化不对称合成大基团手性羟基类化合物的研究进展[J].中国科学:生命科学,2019,49(5):595-604.
NIE Y,XU Y.Stereoselective carbonyl reductases and their roles in asymmetric synthesis of bulky chiral hydroxyl compounds[J].Scientia Sinica Vitae,2019,49(5):595-604.
[12] 倪国伟,汤佳伟,邹杰,等.羰基还原酶在动态动力学拆分中的应用进展[J].有机化学,2019,39(2):339-349.
NI G W,TANG J W,ZOU J,et al.Recent advances on carbonyl reductases for dynamic kinetic resolution[J].Chinese Journal of Organic Chemistry,2019,39(2):339-349.
[13] LIU Z Q,DONG S C,YIN H H,et al.Enzymatic synthesis of an ezetimibe intermediate using carbonyl reductase coupled with glucose dehydrogenase in an aqueous-organic solvent system[J].Bioresource Thchnology,2017,229(1):26-32.
[14] 聂尧,徐岩,王海燕,等.重组大肠杆菌不对称还原2-羟基苯乙酮合成(R)-苯基乙二醇[J].化工进展,2006,25(10):1 231-1 236.
NIE Y,XU Y,WANG H Y,et al.Synthesis of (R)-1-phenyl-1,2-ethanediol by stereospecific reduction of 2-hydroxy acetophenone using recombinant Escherichia coli expressing (R)-specific carbonyl reductase[J].Chemical Industry and Engineering Progress,2006,25(10):1 231-1 236.
[15] NING C X,SU E Z,TIANA Y J,et al.Combined cross-linked enzyme aggregates (combi-CLEAs) for efficient integration of a ketoreductase and a cofactor regeneration system[J].Journal of Biotechnology,2014,184(5):7-10.
[16] 张志斌,吴小芳,杨慧林,等.羰基还原酶工程菌构建及其应用于还原制备S-CHBE[J].食品与发酵工业,2013,39(12):24-29.
ZHANG Z B,WU X F,YANG H L,et al.Construction of recombinant strain with carbonyl reductase gene for production of ethyl S-4-chloro-3-hydroxybutanoate[J].Food and Fermentation Industries,2013,39(12):24-29.
[17] WEI P,GAO J X,LOU W Y,et al.Engineering of a novel carbonyl reductase with coenzyme regeneration in E.coli for efficient biosynthesis of enantiopure chiralalcohols[J].Journal of Biotechnology,2016,230(3):54-62.
[18] 薛芳,李红梅,黄艳青,等.响应面满意度函数优化产羰基还原酶工程菌发酵条件[J].食品与发酵工业,2015,41(2):107-114.
XUE F,LI H M,HUANG Y Q,et al.Optimization of carbonyl reductase production by recombinant Escherichia coli using response surface methodology coupled with desirability function[ J ].Food and Fermentation Industries,2015,41(2):107-114.
[19] 王亚军,吴配配,罗希,等.E.coli BL21(DE3)/pET28a(+)-cr羰基还原酶分离纯化及酶学性质研究[J].高校化学工程学报,2015,29(3):607-615.
WANG Y J,WU P P,et al.Purification and enzymatic characterization of E.coli BL21 (DE3)/pET28a(+)-cr carbonyl reductase[J].Journal of Chemical Engineering of Chinese Universities,2015,29(3):607-615.
[20] 聂尧,徐岩.生物催化立体选择性氧化还原中存在问题及其发展策略[J].生物加工过程,2008,6(2):1-9.
NIE Y,XU Y.Biocatalytic systems for stereoselective oxidoreduction existing limitations and development strategies[J].Chinese Joumal of Bioprocess Engineering,2008,6(2):1-9.
[21] 欧玲,谢谚,许建和.还原酶催化羰基不对称还原的应用进展[J].生物加工过程,2011,9(2):72-78.
OU L,XIE Y,XU J H.Advances in application of reductases in carbonyl asymmetric reduction[J].Chinese Journal of Bioprocess Engineering,2011,9(2):72-78.
[22] SUDAKARA S,CHADHA A.A carbonyl reductase from Candida parapsilosis ATCC 7330:Substrate selectivity and enantiospecificity[J].Organic and Biomolecular Chemistry,2017,15(4):4 165-4 171.
[23] KARANAM V K,CHAUDHURY D,CHADHA A.Understanding (R) specific carbonyl reductase from Candida parapsilosis ATCC 7330[CpCR]:Substrate scope,kinetic studies and the role of zinc[J].Catalysts 2019,9(8):702-712.
[24] AGGARWA N,ANANTHATHAMULA R,KARANAM V K,et al.Understanding substrate specificity and enantioselectivity of carbonyl reductase from Candida parapsilosis ATCC 7330 (CpCR):Experimental and modeling studies[J].Molecular Catalysis,2018,460(11):40-45.
[25] 王伟,刘云,余华顺,戴秋红,等.近平滑假丝酵母ATCC 7330中羰基还原酶双水相萃取工艺研究[J].化学与生物工程,2020,37(1):8-12.
WANG W,LIU Y,YU H S,et al.Aqeous two-phase extraction process of carbonyl reductase in Candida parapsilosis ATCC 7330[J].Chemistry & Bioengineering,2020,37(1):8-12.
[26] 王伟,杨锟,梅子龙,等.近平滑假丝酵母ATCC7330均质破碎工艺研究[J].农产品加工,2019,48(9):33-35.
WANG W,YANG K,MEI Z L,et al.Study on the breaking process of Candida parapsilosis ATCC 7330[J].Farm Products Processing,2019,48(9):33-35.
[27] 罗华军,刘云,王亚宁,邱勇,余华顺,戴秋红,姚鹃,龚大春.近平滑假丝酵母ATCC 7330的生长条件优化[J].化学与生物工程,2019,36(2):12-16.
LUO H J,LIU Y,WANG Y N,et al.Optimization in growth conditions of Candida parapsilosis ATCC 7330[J].Chemistry & Bioengineering,2019,36(2):12-16.
[28] 应国清,杨岳微,梅建凤,等.马克斯克鲁维酵母羰基还原酶基因的克隆与表达[J].微生物学通报,2013,40(8):1 393-1 402.
YING G Q,YANG Y W,MEI J F,et al.Cloning and expression of Kluyveromyce marxianus gene encoding carbonyl reductase in Escherichia coli[J].Microbiology China,2013,40(8):1 393-1 402.