苹果酸-乳酸发酵(malolactic fermentation,MLF)是指在葡萄酒酿造过程中,乳酸菌将L-苹果酸分解生成L-乳酸和CO2的过程[1-3]。MLF对干红葡萄酒和陈酿型白葡萄酒的口感、香气及微生物稳定性等的提升具有重要作用。MLF发生在酒精发酵结束后,此时葡萄酒中的乳酸菌受到高酒精度、低pH、高SO2等恶劣环境的胁迫。随着全球气候变暖的加剧,葡萄浆果的潜在酒度不断升高[4]。酒精发酵完成后葡萄酒的高酒精度(可超过16%体积分数)成为很多产区葡萄酒能否顺利进行MLF的最重要因素之一[5]。乙醇胁迫环境下乳酸菌的胁迫应答机制也越来越成为研究人员关注的焦点问题。
在乙醇胁迫条件下,乳酸菌往往表现出细胞膜完整性受损、菌体关键酶活性下降和正常生理代谢调控异常等。同时,乳酸菌也会通过调节细胞膜脂肪酸构成、表达胁迫耐受相关基因和调整细胞生理代谢等作为应对,进而适应乙醇胁迫的环境(图1)。近年来,随着基因组学、转录组学、蛋白质组学等技术和方法的不断发展,人们对乙醇胁迫下乳酸菌应答作用机制的研究逐渐深入。本文主要对MLF中酒球菌属(Oenococcus)、乳杆菌属(Lactobacillus)等常见菌属细菌在乙醇胁迫下的应答机制进行了综述。
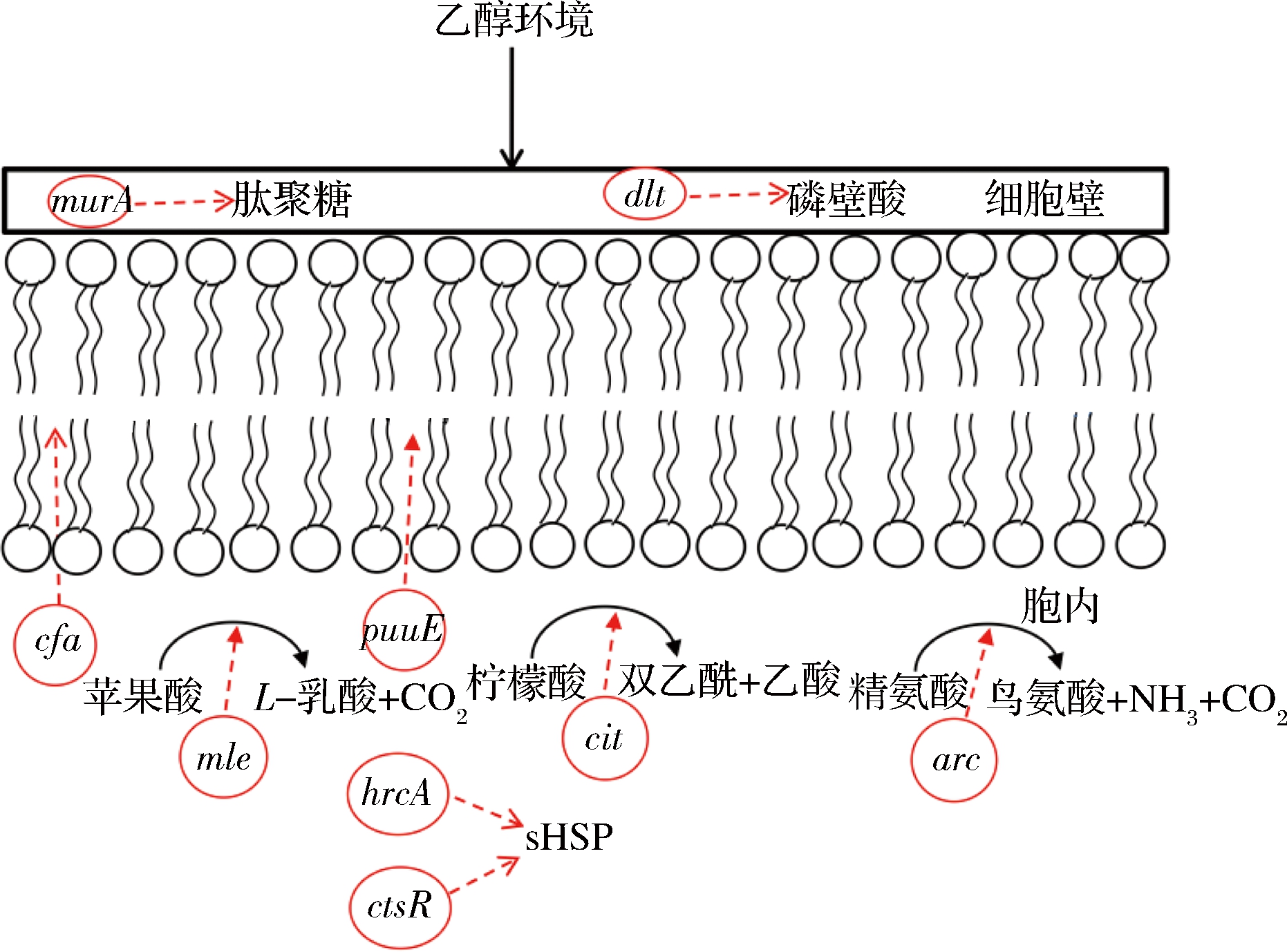
图1 乳酸菌乙醇胁迫适应性机制图
Fig.1 Diagram of the adaptation mechanism of lactic acid bacteria to ethanol stress
注:![]() 表示基因诱导调控表达
表示基因诱导调控表达
1 调节细胞壁和细胞膜功能提高乙醇耐受性
细胞壁和细胞膜作为乳酸菌与乙醇环境接触的屏障,直接影响着乳酸菌存活和MLF的顺利进行。特别是对于缺乏完整细胞结构,直接由细胞膜包被的酒酒球菌(Oenococcus oeni),乙醇胁迫下的细胞膜结构调节显得尤为重要。
1.1 细胞膜功能调节提高乙醇耐受性
细胞膜作为细胞的界面和屏障,在细胞生长、代谢、能量传导和维持稳定的胞内环境中起着重要作用[6]。乙醇的毒害作用往往首先作用于细胞膜,通过破坏细胞膜结构中的疏水核心使极性分子自由地通过渗透屏障,影响细胞关键酶活性,致使细胞膜完整性受损,通透性及流动性增强,细胞生长受到抑制,甚至死亡[7]。细胞膜结构和功能调节是乳酸菌对乙醇胁迫的常见应激防御机制。在受到乙醇胁迫后,细胞膜脂肪酸构成中不饱和脂肪酸(unsaturated fatty acid,UFA)、饱和脂肪酸(saturated fatty acid,SFA)和环丙烷脂肪酸(cyclopropane fatty acid,CFA)含量随着乙醇胁迫浓度和时间发生变化[3, 8]。通常胁迫后,膜脂肪酸平均碳链得到延长,促使酰基链填充,减少游离酰基链末端的运动,使其呈“凝胶状”,从而保持细胞膜稳定性[8]。乙醇胁迫后乳酸菌脂肪酸含量变化相关研究表明,尽管乙醇胁迫后植物乳杆菌(Lactobacillus plantarum)菌株WCFS1编码脂肪酸生物合成(fab)基因簇表达下调,但编码酰基载体蛋白合成酶的相关基因(acpS)和环丙烷脂肪酰基磷脂合成酶相关基因(cfa2)表达上调,通过促进膜脂肪酸碳链的延长和CFA的合成,可抑制乳酸菌细胞流态化作用[9-10]。YANG等[11]在L. plantarum菌株NF92中鉴定到受acrR转录调控的乙醇胁迫抗性基因fabZ1(3-羟基酰基-ACP脱水酶),通过比较敲除和过表达转录调控基因acrR的研究表明,acrR通过正向调控脂肪酸生物合成(fab)基因簇促进脂肪酸的生物合成,过表达acrR后细胞膜中UFA和CFA含量增加,特别是克服乙醇毒性效应重要因子C18:1和cycC19:0,通过调节细胞膜流动性,提高菌株对高浓度乙醇环境的适应性。而O. oeni乙醇胁迫耐受相关研究同样发现,在体积分数为5%的低乙醇条件下菌株膜脂肪酸中SFA含量增加,在体积分数为10%的高乙醇条件下UFA和CFA含量增加[12-13]。据报道,在高于体积分数为12%的乙醇胁迫环境下,O. oeni中香叶基香叶基焦磷酸合酶基因(ggpps)表达水平显著提高,通过促使类异戊二烯前体流向膜稳定剂(如类胡萝卜素途径)相关合成,稳定细胞膜结构,防止细胞死亡[14]。O. oeni和L. plantarum菌株乙醇胁迫后细胞膜调控机制的相似性,也是两者在乙醇胁迫环境下菌株活性较高的重要原因。
1.2 细胞壁功能调节提高乙醇耐受性
由交替的N-乙酰葡萄糖胺、N-乙酰胞壁酸与一种或多种氨基酸短肽交联而成三维网状大分子结构的肽聚糖(peptidoglycan,PG)是乳酸菌重要的细胞壁组分,以维持细胞形状和防止渗透胁迫而著称[15-16]。YUAN等[17]将L.plantarum菌株利用Cre-lox重组技术在大肠杆菌(Escherichia coli)菌株中进行基因整合构建,鉴定插入为催化肽聚糖生物合成第1个重要步骤的编码UDP-N-乙酰氨基葡糖烯醇丙酮酸转移酶基因murA2及其上游基因间973 bp序列,使过表达菌株EC100乙醇耐受性较对照菌株提高4.1倍,证明乙醇胁迫下菌株murA基因表达能够催化PG主要前体合成提高乙醇耐受性[17]。磷壁酸(teichoic acid,TA)是革兰氏阳性菌细胞壁的特殊组分,由甘油磷壁酸和核糖醇磷壁酸组成。转录组分析研究表明,L.plantarum菌株WCFS1在体积分数为8%的乙醇胁迫初期磷壁酸D-丙氨酰化所需的dlt操纵子表达被诱导,随着胁迫时间延长,参与细胞壁磷壁酸生物合成的基因tagE5、tagE6转录表达上调,菌株对乙醇环境的适应性增强[10]。除了表达细胞壁组分合成相关基因外,受乙醇胁迫影响,编码具有自溶素活性的N-乙酰壁酰胺酶基因(oeoe 0735、oeoe 0588、oeoe 1734)受到抑制表达下调,防止细胞壁的自溶作用[18]。有趣的是,乳酸菌代谢在细胞壁外或细胞表面会造成酒体浑浊、增加葡萄酒黏稠感的胞外多糖(exopoly saccharides,EPS)合成,主要是由压力环境所引起的[19]。乙醇胁迫环境下EPS合成相关的胞外多糖生物合成蛋白(oeoe 0071)、UDP-葡萄糖-6-脱氢酶(oeoe 1737)等基因表达上调,通过分解代谢途径中一定量的葡萄糖产生EPS,构成防止渗透压失调的保护因子,提高乳酸菌在葡萄酒中的存活率[20-21]。这也意味着乳酸菌细胞壁和细胞膜的改变是克服乙醇在脂质-水界面的影响所必需的。
2 细胞稳态机制和代谢变化提高细菌乙醇耐受性
除了调节细胞壁和细胞膜结构维持细胞完整性外,乳酸菌往往通过细胞稳态机制和代谢变化来对细胞内部酸化和跨细胞膜质子梯度消散、活性氧积累等乙醇胁迫影响做出应对,表现出增强乙醇抗性的细胞状态[22]。
2.1 质膜结合ATP酶的激活提高乙醇耐受性
随着乙醇浓度的增加,乳酸菌中质膜结合ATP酶激活表达,特别是质膜结合ATP酶中的H+-FoF1-ATP酶,不仅参与细胞内ATP的合成,还与ATP水解偶联,将质子从细胞中挤出到可产生质子梯度的介质中,产生质子驱动力(proton motive force,PMF)施加到质子上,驱动需要膜结合过程的代谢。全基因组分析表明,乳酸菌中普遍存在FoF1-ATP酶基因,这对于乙醇胁迫下乳酸菌存活能量供应而言至关重要[23-24]。CONTRERAS等[25]在体积分数分别为9%和12%的乙醇培养环境下将O.oeni菌株PSU-1和无乙醇胁迫条件进行比较发现,由FoF1-ATP酶满足的非生长相关维持性ATP需求分别提高了10倍和17倍,帮助表达细胞维持相关代谢[25]。除了H+-ATP酶外,Na+-K+-ATP酶、Ca2+-Mg2+-ATP酶也是分布在细胞膜上的典型巯基酶。因乙醇胁迫而消除的Na+-K+、Ca2+-Mg2+巯基会导致细胞膜的通透性增加,因此较高的Na+-K+-ATP酶和Ca2+-Mg2+-ATP酶活性对菌株乙醇菌株胁迫耐受是非常有益的[26-28]。LIN等[29]将O.oeni 菌株SD-2a的puuE基因在L.plantarum菌株WCFS1中进行异源表达,证明puuE基因可以显著提高Na+-K+-ATP酶和Ca2+-Mg2+-ATP酶活性,从而提高细胞膜完整性,在细菌乙醇耐受性中发挥作用。
2.2 苹果酸和柠檬酸代谢调控提高乙醇耐受性
目前已经报道的O.oeni部分蛋白质组胁迫应激反应和代谢机制表明,乳酸菌苹果酸代谢和柠檬酸代谢能够消耗质子,并产生膜电位和pH梯度,在乙醇胁迫环境为菌株能量供应和细胞体内pH稳态机制中发挥重要作用[30-31]。
在葡萄酒中乳酸菌可以摄取环境内的苹果酸和柠檬酸参与相关代谢。苹果酸代谢将L-苹果酸脱羧为L-乳酸和CO2。脱羧过程中质子被消耗,调节细胞内部pH,从而保持质子电位梯度,并在细胞质膜上形成PMF,通过膜结合的FoF1-ATP酶驱动ATP合成,提高乙醇胁迫下菌株存活率[31]。转录组学分析表明,乙醇胁迫下与苹果酸代谢相关mle操纵子(mleA、mleP、mleR)、MLF系统转录激活因子(oeoe 1565)转录表达上调,帮助细菌应对乙醇胁迫[21]。而葡萄酒中柠檬酸由各种膜相关通透酶转运进入细胞后,柠檬酸盐在柠檬酸裂解酶复合物催化下转化为乙酸盐和草酰乙酸盐;然后由草酰乙酸脱羧酶脱羧,最终生成丙酮酸和CO2,或被可溶性细胞质苹果酸酶脱羧,从环境中获得能量,并且产生双乙酰等香气化合物[32]。值得注意的是,菌株柠檬酸相关基因表达水平可能主要受培养环境柠檬酸诱导[21,33]。有研究将O.oeni菌株PSU-1在额外添加0.5 g/L柠檬酸盐的培养基中培养,结果表明柠檬酸代谢激活主要受乙醇影响,当存在乙醇时,柠檬酸途径的基因操纵子cit(citC、citD、citE、citF)表达激活,响应乙醇胁迫代谢产物双乙酰、乙酸的相关基因alsD和ackA的表达量增加,通过利用培养基中柠檬酸盐进行代谢[33]。而将O.oeni菌株Elios在不含柠檬酸的体积分数为8%的乙醇培养基中培养后,则发现柠檬酸操纵子(编码柠檬酸转运蛋白的基因oeoe 0419和编码柠檬酸裂解酶α亚基oeoe 0423)表达下调,并且在蛋白质水平上得到一致结果[21]。
2.3 氨基酸代谢调控提高乙醇耐受性
氨基酸代谢是乳酸菌应对乙醇胁迫的又一重要途径。在乳酸菌中,精氨酸通过脱亚氨酶途径完全降解为鸟氨酸、NH3和CO2,提供一分子ATP,维持细胞内部pH动态平衡。研究表明,乳酸菌生长初期编码精氨酸脱亚氨酶(ArcA)、鸟氨酸氨基甲酰基转移酶(ArcB)和氨基甲酸酯激酶(ArcC)关键酶基因的arc操纵子由碳分解代谢控制蛋白(CcpA)负调控;随着菌株生长(指数生长期或稳定生长期),arc操纵子表达由转录调控因子argR、ahrC调节[34]。在表达arc操纵子的乳酸菌中,精氨酸分解代谢可以为乳酸菌适应乙醇环境和生长提供能量。D EZ等[35]将乳酸乳球菌(Lactococcus lactis)菌株NZ9700在外源添加精氨酸的乙醇培养基中培养,发现与正常株相比argR、ahrC基因敲除株生长速率下降,进一步转录组分析发现,乙醇存在下正常株参与精氨酸降解的arcA、arcB、arcD1等相关基因转录上调20~40倍。精氨酸合成前体之一的谷氨酸在乙醇胁迫应答中同样具有重要作用[36-38]。转录组学和蛋白组学研究结果表明,体积分数为8%的乙醇胁迫后谷氨酸转运蛋白基因(oeoe 1634)的生物合成表达增加,通过脱羧生成γ-氨基丁酸以及谷氨酸-γ-丁酸逆向系统在环境中挤出γ-氨基丁酸作为质子动力,帮助菌株维持胞内pH动态平衡,增加潜在能源可用性[21,39]。
EZ等[35]将乳酸乳球菌(Lactococcus lactis)菌株NZ9700在外源添加精氨酸的乙醇培养基中培养,发现与正常株相比argR、ahrC基因敲除株生长速率下降,进一步转录组分析发现,乙醇存在下正常株参与精氨酸降解的arcA、arcB、arcD1等相关基因转录上调20~40倍。精氨酸合成前体之一的谷氨酸在乙醇胁迫应答中同样具有重要作用[36-38]。转录组学和蛋白组学研究结果表明,体积分数为8%的乙醇胁迫后谷氨酸转运蛋白基因(oeoe 1634)的生物合成表达增加,通过脱羧生成γ-氨基丁酸以及谷氨酸-γ-丁酸逆向系统在环境中挤出γ-氨基丁酸作为质子动力,帮助菌株维持胞内pH动态平衡,增加潜在能源可用性[21,39]。
此外,由于乙醇会引起超氧阴离子![]() 过氧化氢(H2O2)、羟自由基(·OH)和氢过氧化物(ROOH)等活性氧(reactive oxygen species,ROS)自由基积累,造成细菌细胞脂质、蛋白质和核酸氧化损伤。而由L-谷氨酸、L-半胱氨酸和甘氨酸肽键缩合而成的谷胱甘肽(glutathione,GSH)作为一种具有抗氧化特性的三肽化合物(Glu-Cys-Gly),其还原形式通过半胱氨酸巯基中和细胞内ROS,在抵御乙醇胁迫对细胞膜的流态化、减少自由基对DNA的攻击、降低DNA损伤和突变和维持乙醇胁迫后细胞氧化还原平衡等方面具有重要作用(图2)[40]。与其他革兰氏阳性菌类似,乳酸菌通常缺少自主合成GSH的基因,但可以摄取葡萄酒内的GSH,起到抵抗胁迫的保护作用[41]。基因组分析表明,O.oeni中编码GSH氧化还原基因包括谷胱甘肽还原酶(gshR)、谷胱甘肽过氧化物酶(gpo)、假设转运蛋白基因2个亚基(cydC、cydD)以及3个谷氧还蛋白基因(rdx),并且gshR和gpo基因在乙醇应激下普遍表达[42]。MARGALEF-CATAL
过氧化氢(H2O2)、羟自由基(·OH)和氢过氧化物(ROOH)等活性氧(reactive oxygen species,ROS)自由基积累,造成细菌细胞脂质、蛋白质和核酸氧化损伤。而由L-谷氨酸、L-半胱氨酸和甘氨酸肽键缩合而成的谷胱甘肽(glutathione,GSH)作为一种具有抗氧化特性的三肽化合物(Glu-Cys-Gly),其还原形式通过半胱氨酸巯基中和细胞内ROS,在抵御乙醇胁迫对细胞膜的流态化、减少自由基对DNA的攻击、降低DNA损伤和突变和维持乙醇胁迫后细胞氧化还原平衡等方面具有重要作用(图2)[40]。与其他革兰氏阳性菌类似,乳酸菌通常缺少自主合成GSH的基因,但可以摄取葡萄酒内的GSH,起到抵抗胁迫的保护作用[41]。基因组分析表明,O.oeni中编码GSH氧化还原基因包括谷胱甘肽还原酶(gshR)、谷胱甘肽过氧化物酶(gpo)、假设转运蛋白基因2个亚基(cydC、cydD)以及3个谷氧还蛋白基因(rdx),并且gshR和gpo基因在乙醇应激下普遍表达[42]。MARGALEF-CATAL 等[43]外源添加GSH到体积分数为14%的乙醇培养环境中进行O.oeni菌株培养,发现菌株膜脂肪酸中CFA含量显著增加,菌株表现出更高的存活率,证明GSH富集可以提高菌株胁迫耐受性,这对乳酸菌适应葡萄酒环境显然是更有利的[43]。
等[43]外源添加GSH到体积分数为14%的乙醇培养环境中进行O.oeni菌株培养,发现菌株膜脂肪酸中CFA含量显著增加,菌株表现出更高的存活率,证明GSH富集可以提高菌株胁迫耐受性,这对乳酸菌适应葡萄酒环境显然是更有利的[43]。

图2 乙醇环境下谷胱甘肽代谢途径[40]
Fig.2 Glutathione metabolism pathway in ethanol environment[40]
注:Gpo,谷胱甘肽过氧化物酶(glutathione peroxidase);GSH,还原型
谷胱甘肽(glutathione);GSSG,氧化型谷胱甘肽 (glutathione
disulfifide);GshR,谷胱甘肽还原酶(glutathione reductase);
Grx,谷氧还蛋白(glutaredoxin)
3 应激蛋白合成提高乙醇耐受性
当乳酸菌生存环境转变为乙醇胁迫环境后,蛋白质表达由抑制冗余或非必需蛋白表达转变为合成用于抵御胁迫环境的应激蛋白,参与细胞DNA和蛋白质的重组修复。乙醇胁迫后乳酸菌开始诱导应激蛋白特别是热激蛋白(heat shock protein,HSP)表达。
HSP是较早发现的一类应激蛋白质,属于分子伴侣蛋白质。在多种胁迫环境下,HSP都能够阻止蛋白质聚集,并协助蛋白质重新折叠,最终发挥防止蛋白质损伤和提高胁迫耐受性的分子伴侣作用[44]。常见的HSP主要包括分子伴侣蛋白(DnaK、GroESL、DnaJ等)、蛋白酶(Clp、FstH、HtrA等)以及DNA重组修复蛋白(RecA、UvrD解旋酶等)[45]。目前由阻遏蛋白HrcA、CtsR单一或共同调控的DnaK和GroESL较多报道与乳酸菌乙醇胁迫响应相关。在O.oeni中ctsR基因被鉴定并且异源表达验证为逆境反应基因重要表达调控因子,通过调控hsp18、clp等小热休克蛋白基因表达,例如,O.oeni中的hsp18基因合成的Lo18蛋白,对乙醇等胁迫环境做出对应[46-47]。ZHAO等[48]将O.oeni中ctsR基因在L.plantarum菌株WCFS1中异源表达,结果表明菌株WCFS1在酸性乙醇环境下的生长性能得到提高,操纵子下游clp、groESL、dnaK和hsp等基因表达增强。L.plantarum菌株WCFS1在乙醇环境下进行培养后发现,受到乙醇环境影响后菌株蛋白质快速连续展开,仅仅由hrcA转录表达的groELS逐渐不足以维持蛋白质的正确折叠,变性和聚集的蛋白质积累到一定程度后,ctsR调节子开始激活clpP、clpE、hsp18等转录表达,维持正常蛋白质功能[10,49]。HSP中DNA修复蛋白通常在细胞体内外多种因素胁迫时被激活,对受损DNA进行识别和修复。在同为革兰氏阳性菌的简单节杆菌(Arthrobacter simplex)菌株TCCC11037乙醇胁迫报道发现,DNA重组修复过程中关键蛋白基因recA和uvrD转录表达分别提高了2.59倍和2.57倍[50]。而在葡萄酒酿造常见乳酸菌中虽然同样存在DNA修复蛋白,但是乙醇胁迫表达变化研究中却鲜见报道。关于乙醇胁迫后葡萄酒酿造常见乳酸菌DNA修复蛋白表达相关机制可能仍需要进一步探究。
4 结语
对乳酸菌在乙醇胁迫环境下的应答机制研究,不仅有助于更好地了解相关菌种特性,而且为优良菌株构建提供了理论基础。目前,研究人员主要从维持细胞完整性、细胞内部能量供应和代谢调节、胁迫蛋白表达等方面对乳酸菌乙醇胁迫应答和适应性机制进行了研究,以了解葡萄酒中乳酸菌的乙醇胁迫适应性表达机制。然而实际上葡萄酒是多胁迫共存的复杂环境,因此如果能够进一步认识乳酸菌多胁迫因素条件下细胞代谢的内在联系和交叉保护机制,将对全面认识乳酸菌的逆境胁迫应答机制和更好发挥乳酸菌在葡萄酒MLF中的作用具有重要意义。相信随着新技术的发展,人们对葡萄酒胁迫环境中胁迫应答机制的深入研究,乳酸菌胁迫应答适应性机制将得到更全面地揭示。
[1] BRIZUELA S N,BRAVO-FERRADA M B,POZO-BAY N
N  M,et al.Changes in the volatile profile of pinot noir wines caused by patagonian Lactobacillus plantarum and Oenococcus oeni strains[J].Food Research International,2018,106:22-28.
M,et al.Changes in the volatile profile of pinot noir wines caused by patagonian Lactobacillus plantarum and Oenococcus oeni strains[J].Food Research International,2018,106:22-28.
[2] BRIZUELA S N,BRAVO-FERRADA M B,HENS V L D,et al.Comparative vinification assays with selected patagonian strains of Oenococcus oeni and Lactobacillus plantarum[J].LWT-Food Science and Technology,2016,77:348-355.
[3] CAPPELLO M S,ZAPPAROLI G,LOGRIECO A,et al.Linking wine lactic acid bacteria diversity with wine aroma and flavor[J].International Journal of Food Microbiology,2017,243:16-27.
[4] UBEDA C,HORNEDO-ORTEGA R,CEREZO B A,et al.Chemical hazards in grapes and wine,climate change and challenges to face[J].Food Chemistry,2020,314.DOI:100.1016/j.foodchem.2020.126222.
[5] 金刚. 苹果酸-乳酸发酵细菌的多样性及其耐酒精分析[D].杨凌:西北农林科技大学,2015.
JIN G.Study on diversity and ethanol tolerance of malolactic bacteria[D].Ynagling:Northwest A&F University,2015.
[6] ZHANG Y M,ROCK C O.Membrane lipid homeostasis in bacteria[J].Nature Reviews Microbiology,2008,6(3):222-233.
[7] 王婷婷, 李春,李佳栋,等.乙醇胁迫对植物乳杆菌膜生理及黏附性的影响[J].食品科学,2019,40(18):63-69.
Wang T T,LI C,LI J D,et al.Effect of ethanol stress on membrane physiology and adhesion of Lactobacillus plantarum[J].Food Science,2019,40(18):63-69.
[8] LAN C H,MARECHAL P A,MAI L T,et al.Fluorescent probes to evaluate the physiological state and activity of microbial biocatalysts:A guide for prokaryotic and eukaryotic investigation[J].Biotechnology Journal,2008,3(7):890-903.
[9] VAN B D V H,ABEE T,TEMPELAARS M,et al.Short-and long-term adaptation to ethanol stress and its cross-protective consequences in Lactobacillus plantarum[J].Applied and Environmental Microbiology,2011,77(15):5 247-5 256.
[10] BRAVO-FERRADA M B,G MEZ-ZAVAGLIA A,SEMORILE L,et al.Effect of the fatty acid composition of acclimated oenological Lactobacillus plantarumon the resistance to ethanol[J].Letters in Applied Microbiology,2014,60(2):155-161.
MEZ-ZAVAGLIA A,SEMORILE L,et al.Effect of the fatty acid composition of acclimated oenological Lactobacillus plantarumon the resistance to ethanol[J].Letters in Applied Microbiology,2014,60(2):155-161.
[11] YANG X,TENG K,LI L,et al.Transcriptional regulator AcrR increases ethanol tolerance through regulating fatty acid synthesis in Lactobacillus plantarum[J].Applied and Environmental Microbiology,2019,85(22).DOI:10.1128/aem.01690-19.
[12] 赵红玉, 李华,刘龙祥,等.酒酒球菌胁迫适应性机制的研究进展[J].中国食品学报,2019,48(4):556-561.
ZHAO H Y,LI H,LIU L X.The research progress of stress adaptation mechanism of Oenococcus oeni[J].Journal of Chinese Institute of Food Science and Technology,2019,48(4):556-561.
[13] 李中超. 酒酒球菌SD-2a质膜H+-ATPase特性的研究[D].杨凌:西北农林科技大学,2009.
LI Z C.Researches on the characteristics of plasma membrane H+-ATPase of Oenococcus oeni SD-2a[D].Yangling:Northwest A&F University,2009.
[14] CAFARO C,BONOMO M G,SALZANO G.Adaptive changes in geranylgeranyl pyrophosphate synthase gene expression level under ethanol stress conditions in Oenococcus oeni[J].Journal of Applied Microbiology,2013,116(1):71-80.
[15] NICOLAOU S A,GAIDA S M,PAPOUTSAKIS E T.Exploring the combinatorial genomic space in Escherichia coli for ethanol tolerance[J].Biotechnology Journal,2012,7(11):1 337-1 345.
[16] VOLLMER W,BLANOT D,DE PEDRO M A.Peptidoglycan structure and architecture[J].Fems Microbiology Reviews,2008,32(2):149-167.
[17] YUAN Y,BI C,NICOLAOU S A,et al.Overexpression of the Lactobacillus plantarum peptidoglycan biosynthesis murA2 gene increases the tolerance of Escherichia coli to alcohols and enhances ethanol production[J].Applied Microbiology and Biotechnology,2014,98(19):8 399-8 411.
[18] OLGU N N,CHAMPOMIER-VERGES M,ANGLADE P,et al.Transcriptomic and proteomic analysis of Oenococcus oeni PSU-1 response to ethanol shock[J].Food Microbiology,2015,51:87-95.
N N,CHAMPOMIER-VERGES M,ANGLADE P,et al.Transcriptomic and proteomic analysis of Oenococcus oeni PSU-1 response to ethanol shock[J].Food Microbiology,2015,51:87-95.
[19] DIMOPOULOU M,HAZO L,DOLS-LAFARGUE M.Exploration of phenomena contributing to the diversity of Oenococcus oeni exopolysaccharides[J].International Journal of Food Microbiology,2012,153(1-2):114-122.
[20] CIEZACK G,HAZO L,CHAMBAT G,et al.Evidence for exopolysaccharide production by Oenococcus oeni strains isolated from non-ropy wines[J].Journal of Applied Microbiology,2010,108(2):499-509.
[21] COSTANTINI A,RANTSIOU K,MAJUMDER A,et al.Complementing DIGE proteomics and DNA subarray analyses to shed light on Oenococcus oeni adaptation to ethanol in wine-simulated conditions[J].Journal of Proteomics,2015,123:114-127.
[22] SALEMA M,LOLKEMA J S,SAN ROM O M V,et al.The proton motive force generated in Leuconostoc oenos by L-malate fermentation[J].Journal of Bacteriology,1996,178(11):3 127-3 132.
O M V,et al.The proton motive force generated in Leuconostoc oenos by L-malate fermentation[J].Journal of Bacteriology,1996,178(11):3 127-3 132.
[23] GAO Y,LIU Y,SUN M,et al.Physiological function analysis of Lactobacillus plantarum Y44 based on genotypic and phenotypic characteristics[J].Journal of Dairy Science,2020,103:5 916-5 939.
[24] MARGALEF-CATALA M,FELIS G E,REGUANT C,et al.Identification of variable genomic regions related to stress response in Oenococcus oeni[J].Food Research International,2017,102:625-638.
[25] CONTRERAS A,RIBBECK M,GUTIÉRREZ G D,et al.Mapping the physiological response of Oenococcus oeni to ethanol stress using an extended genome-scale metabolic model[J].Frontiers in Microbiology,2018,9:291.
[26] ZHANG J,DU G C,ZHANG Y,et al.Glutathione protects Lactobacillus sanfranciscensis against freeze-thawing,freeze-drying,and cold treatment[J].Applied and Environmental Microbiology,2010,76(9):2 989-2 996.
[27] SAMAD A,JAMES A,WONG J,et al.Insulin protects pancreatic acinar cells from palmitoleic acid-induced cellular injury[J].Journal of Biological Chemistry,2014,289:23 582-23 595.
[28] CHU-KY S,TOURDOT-MARECHAL R,MARECHAL P-A,et al.Combined cold,acid,ethanol shocks in Oenococcus oeni:Effects on membrane fluidity and cell viability[J].Biochimica Et Biophysica Acta,2005,1717(2):118-124.
[29] LIN Y,ZHAO H Y,LIU L X,et al.Heterologous expression of the puuE from Oenococcus oeni SD-2a in Lactobacillus plantarum WCFS1 improves ethanol tolerance[J].Journal of Basic Microbiology,2019,59(11):1-9.
[30] MOHEDANO M D I L,RUSSO P,DE L R V,et al.A proteome reference map of the wine lactic acid bacteria Oenococcus oeni ATCC BAA-1163[J].Open Biology,2014,4(2):130-154.
[31] ZHAO W Y,KANG Z K.Advanced progress on adaptive stress response of Oenococcus oeni[J].Journal of Northeast Agricultural University,2013,20(1),91-96.
[32] PASSERINI D,LAROUTE,VALÉRIE,et al.New insights into Lactococcus lactis diacetyl-and acetoin-producing strains isolated from diverse origins[J].International Journal of Food Microbiology,2013,160(3):329-336.
[33] OLGUIN N,BORDONS A,REGUANT C.Influence of ethanol and pH on the gene expression of the citrate pathway in Oenococcus oeni[J].Food microbiology,2009,26(2):197-203.
[34] ZOMER A L,BUIST G,LARSEN R,et al.Time-resolved determination of the CcpA regulon of Lactococcus lactis subsp.cremoris MG1363[J].Journal of Bacteriology,2006,189(4),1 366-1 381.
[35] D EZ L,SOLOPOVA A,FERN
EZ L,SOLOPOVA A,FERN NDEZ-PÉREZ R,et al.Transcriptome analysis shows activation of the arginine deiminase pathway in Lactococcus lactis as a response to ethanol stress[J].International Journal of Food Microbiology,2017,257:41-48.
NDEZ-PÉREZ R,et al.Transcriptome analysis shows activation of the arginine deiminase pathway in Lactococcus lactis as a response to ethanol stress[J].International Journal of Food Microbiology,2017,257:41-48.
[36] HEBERT E M,RAYA R R,DE GIORI G S.Nutritional requirements and nitrogen-dependent regulation of proteinase activity of Lactobacillus helveticus CRL 1062[J].Applied and Environmental Microbiology,2000,66(12):5 316-5 321.
[37] PAPADIMITRIOU K,ALEGRIA A,BRON P A,et al.Stress physiology of lactic acid bacteria[J].Microbiology and Molecular Biology Reviews,2016,80:837-890.
[38] ZOTTA T,PARENTE E,RICCIARDI A.Aerobic metabolism in the genus Lactobacillus:Impact on stress response and potential applications in the food industry[J].Journal of Applied Microbiology,2017,122:857-869.
[39] MAZZOLI R,PESSIONE E,DUFOUR M,et al.Glutamate induced metabolic changes in Lactococcus lactis NCDO 2118 during GABA production:Combined transcriptomic and proteomic analysis[J].Amino Acids,2010,39(3):727-737.
[40] MASIP L,VEERAVALLI K,GEORGIOU G.The many faces of glutathione in bacteria[J].Antioxidants & Redox Signaling,2006,8(5-6):753-762.
[41] CECCONI D,MILLI A,RINALDUCCI S,et al.Proteomic analysis of Oenococcus oeni freeze-dried culture to assess the importance of cell acclimation to conduct malolactic fermentation in wine[J].Electrophoresis,2009,30(17):2 988-2 995.
[42] MARGALEF-CATAL M,ARAQUE I,BORDONS A,et al.Genetic and transcriptional study of glutathione metabolism in Oenococcus oeni[J].International Journal of Food Microbiology,2017,242:61-69.
M,ARAQUE I,BORDONS A,et al.Genetic and transcriptional study of glutathione metabolism in Oenococcus oeni[J].International Journal of Food Microbiology,2017,242:61-69.
[43] MARGALEF-CATAL M,ARAQUE I,WEIDMANN S,et al.Protective role of glutathione addition against wine-related stress in Oenococcus oeni[J].Food Research International,2016,90:8-15.
M,ARAQUE I,WEIDMANN S,et al.Protective role of glutathione addition against wine-related stress in Oenococcus oeni[J].Food Research International,2016,90:8-15.
[44] CEBRI N G,COND
N G,COND N S,MA
N S,MA AS P.Heat resistance,membrane fluidity and sublethal damage in Staphylococcus aureus cells grown at different temperatures[J].International Journal of Food Microbiology,2019,289:49-56.
AS P.Heat resistance,membrane fluidity and sublethal damage in Staphylococcus aureus cells grown at different temperatures[J].International Journal of Food Microbiology,2019,289:49-56.
[45] VALERIANO V D,PARUNGAO-BALOLONG M,Kang D K.In vitro evaluation of the mucin-adhesion ability and probiotic of Lactobacillus mucosae LM1[J].Journal of pplied Microbiology,2014,117:485-497.
[46] GRANDVALET C,COUCHENEY F,BELTRAMO C,et al.CTSR is the master regulator of stress response gene expression in Oenococcus oeni[J].Journal of Bacteriology,2005,187:5 614-5 623.
[47] DARSONVAL M,JULLIAT F,MSADEK T,et al.CTSR,the master regulator of stress-response in Oenococcus oeni,is a heat sensor interacting with ClpL1[J].Frontiers in Microbiology,2018,9:3 135.
[48] ZHAO H,YUAN L,HU K,et al.Heterologous expression of ctsR from Oenococcus oeni enhances the acid-ethanol resistance of Lactobacillus plantarum[J].FEMS Microbiology Letters,2019,366(15).DOI:10.1093/femsle/fnz192.
[49] FREES D,SAVIJOKI K,VARMANEN P,et al.Clp ATPases and ClpP proteolytic complexes regulate vital biological processes in low GC,Gram-positive bacteria[J].Molecular Microbiology,2010,63(5):1 285-1 295.
[50] 王艳霞. 简单节杆菌乙醇胁迫条件下的抗逆机制研究[D].天津:天津科技大学,2015.
WANG Y X.Study on tolerance mechanism of Arthtrobacter simplex under ethanol stress[D].Tianjin:Tianjin University of Science and Technology,2015.