豆渣是豆腐和腐乳等大豆制品加工过程中的主要副产物,富含膳食纤维、蛋白质、脂肪、维生素和大豆异黄酮等成分[1]。由于豆渣口感粗糙且极易腐败变质,常被用作饲料或直接作为废料丢弃,造成了资源的浪费和环境的污染[2],积极探讨豆渣的加工利用极为重要。霉豆渣又称豆渣粑,是将新鲜的豆渣经微生物自然发酵而成的一类发酵食品,主要分布在湖北省武汉市、洪湖市、荆州市、恩施土家苗族自治州和江西南昌、瑞金等地区[3],使用植物油将其煎炸后常作为火锅的佐料食用。
经实地调研发现,不同农户制作的霉豆渣品质存在较大差异,品质较好的霉豆渣无不良风味,但部分农户制作的霉豆渣则会有一定的臭味,究其原因可能是由于制作环境较为开放,微生物对霉豆渣品质形成的优劣具有重要影响。石威[4]利用纯培养技术对江西南昌地区霉豆渣中的酵母菌、乳酸菌和霉菌进行了分离纯化,发现酵母菌分别为Rhodotorula mucilaginosa(胶红酵母)、Trichosporon montevideense(圆形丝孢酵母)、Aureobasidium pullulans(普鲁兰酵母)和Trichosporon asahii(阿氏丝孢酵母),乳酸细菌分别为Enterococcus faecium(屎肠球菌)和Leuconostoc mesenteroides(肠膜明串珠菌),霉菌为Rhizopus oryzae(米根霉)[4];姚英政[5]采用纯培养技术从湖北武汉地区霉豆渣粑样品中分离出1株真菌,经鉴定为Mucor racemosus Fresenius(总状枝毛霉);张燕鹏等[6]对江西瑞金地区的发酵豆渣进行菌相分析,发现豆渣中真菌主要为Monilia(串珠霉属)、Rhizopus(根霉属)和Saccharomyces(酵母属);细菌主要为Bacillus subtilis(枯草芽孢杆菌)、Enterobacter sp.(肠杆菌属)、Staphylococcus(葡萄球菌属)、Pediococcus(片球菌属)和Deinococcus(异常球菌属)。上述研究均采用了纯培养技术,由于该技术对含量较低、非可培养微生物和某些特殊的微生物会有所遗漏,因而无法对样品中所有微生物进行解析[7]。近年来,随着现代生物技术的不断发展,高通量测序技术不仅成本低、可读量和质量高,为微生物群落结构的全面解析提供了有力工具[8],目前已广泛应用于腐乳[9]、泡菜[10]和豆瓣酱[11]等调味制品中微生物多样性的解析。
本研究采用高通量测序技术解析霉豆渣中细菌多样性并结合PICRUSt软件对细菌类群的基因功能进行了预测,以期为后续更加全面了解霉豆渣中细菌菌群多样性提供理论依据,为特色发酵豆制品的产业化推动提供数据支持。
1 材料与方法
1.1 材料与试剂
霉豆渣:采自湖北省荆州市石首市菜市场(E112°43′~112°47′,N29°67′~29°85′),共采集6 个样品,编号记为M1~M6。
DNA提取试剂盒,德国QIAGEN公司;dNTPs Mix、Fast Pfu Buffer、5×Trans StartTM、Fast Pfu Fly DNA Polymerase,宝生物工程大连有限公司;引物,武汉天一辉远生物科技有限公司。
1.2 仪器与设备
CR21 N高速冷冻离心机,日本HITACHI公司;ND-2000C微量紫外分光光度计,美国Nano Drop公司;Veriti 96孔梯度PCR仪,美国ABI公司;Fluor Chem FC3型化学发光凝胶成像系统,美国ProteinSimple公司;Miseq PE300高通量测序平台,美国Illumina公司;R930机架式服务器,美国DELL公司。
1.3 实验方法
1.3.1 宏基因组提取
参照DNeasy mericon Food Kit试剂盒中方法对霉豆渣样品中的微生物总DNA进行提取。
1.3.2 细菌16S rRNA序列扩增
PCR扩增体系参照沈馨等[12]方法并略有修改。以霉豆渣样品中提取的基因组DNA为模板,利用正向引物338F(5′-ACTCCTACGGGAGGCAGCA-3′)和反向引物806R(5′-GGACTACHVGGGT-3′)进行PCR扩增。
1.3.3 序列质控和生物信息学分析
序列下机,采用尚雪娇等[13]方法选择符合条件的序列进行拼接,然后将拼接好的序列切除7个标签(barcode)序列和引物并进行下一步分析。
使用QIIME(v1.7.0)分析平台[14],对质控后的序列进行分析。采用PyNAST[15]软件将序列对齐,接着进行两步UCLUST[16]划分构建分类操作单元矩阵(operational taxonomic units,OTU),然后利用Greengenes[l7]和RDP[18]数据库进行同源性比对,最后在使用FastTree软件[19]构建系统发育进化树的基础上,计算霉豆渣样品中细菌微生物的Chao1指数和Shannon指数等α多样性指标。在使用Greengenes和RDP数据库进行同源性比对时,若某一序列在2个数据库中的比对结果不相同,则在该分类学地位下将其判定为不可鉴定(unclassfield),同时观察其上一分类学地位比对结果是否相同,若相同则采纳该分类学地位下的比对结果,否则继续将其判定为不可鉴定(unclassfield),并进一步观察其再上一分类学地位结果,直到两个数据库在该分类学地位下判定结果一致为止。
1.3.4 基因功能预测
使用PICRUSt软件对霉豆渣样品中微生物的基因功能进行预测[20],并参照蛋白质直系同源簇数据库(clusters of orthologous groups of proteins,COG)进行功能注释[21]。
1.3.5 图表绘制
本研究主要使用Origin 2017C软件绘制堆积图和柱状图,使用R软件(v3.6.3)的pheatmap软件包绘制热图。
2 结果与分析
2.1 测序深度分析
切除barcode和引物后序列的长度分布情况如图1所示。测序长度在小于425 bp有39 458 条序列,占总序列数的18.54%;测序长度在425~433 bp有173 398 条序列,占总序列数的81.46%;测序长度在434~442 bp、443~451 bp或大于451 bp累计共有8 条序列。由此可知,切除barcode和引物后,81.46%的序列长度在425~451 bp,基本涵盖了16S rRNA V3~V4区,因而后续可在分类学地位“属”水平上对样品细菌多样性进行分析。
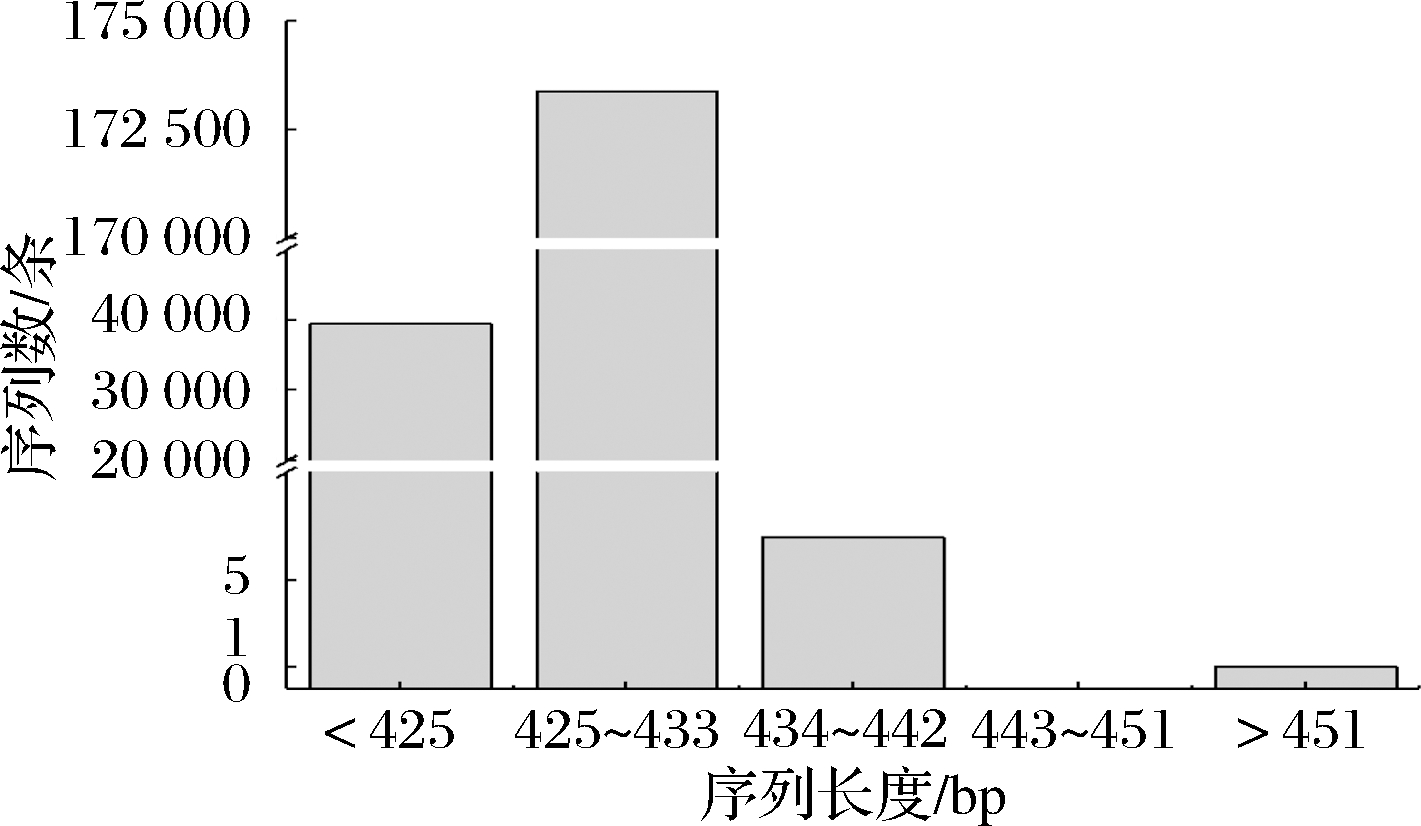
图1 测序长度分布图
Fig.1 Sequencing length distribution
2.2 序列丰富度和多样性分析
由表1可知,6 个霉豆渣样品中共产生212 864 条高质量16S rRNA序列,平均每个样品产生35 477 条,在97%的相似度下划分的OTU数分别为262、526、395、450、473和419 个。样品M5中的序列可鉴定到100 个属,而M6中序列仅鉴定为23 个属;当测序量达到24 010 条时,样品M2的Chao1指数为628,微生物丰度最大,而M1的Chao1指数仅为297;样品M4的Shannon指数为5.77,生物多样性最高,而M1的Shannon指数仅为1.49。由此可见,不同霉豆渣样品的细菌多样性存在较大的差异。
表1 16 rRNA测序情况及各分类地位数量
Table 1 16 rRNA read counts and number of identifiable units at different taxonomical levels
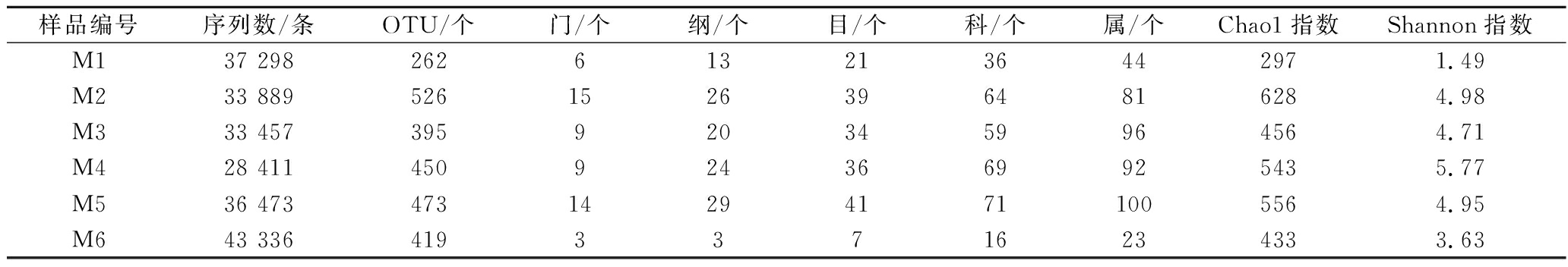
样品编号序列数/条OTU/个门/个纲/个目/个科/个属/个Chao1指数Shannon指数M137 2982626132136442971.49M233 88952615263964816284.98M333 4573959203459964564.71M428 4114509243669925435.77M536 473473142941711005564.95M643 33641933716234333.63
注:计算每个样品Chao1指数和Shannon指数时,样品的测序量均为24 010条序列
2.3 基于门和属水平的细菌群落结构分析
霉豆渣样品中相对含量大于1.0%的优势细菌门构成如图2所示。霉豆渣中平均相对含量大于1.0%的优势菌门为Proteobacteria(变形菌门)、Firmicutes(硬壁菌门)、Bacteroidetes(拟杆菌门)和Actinobacteria(放线菌门),其平均相对含量分别为46.50%、38.80%、12.62%和1.81%。且样品M1和M6中的优势菌门构成较为相似,主要为Firmicutes,而M2、M3、M4和M5中的优势菌门较为相似,主要为Proteobacteria。
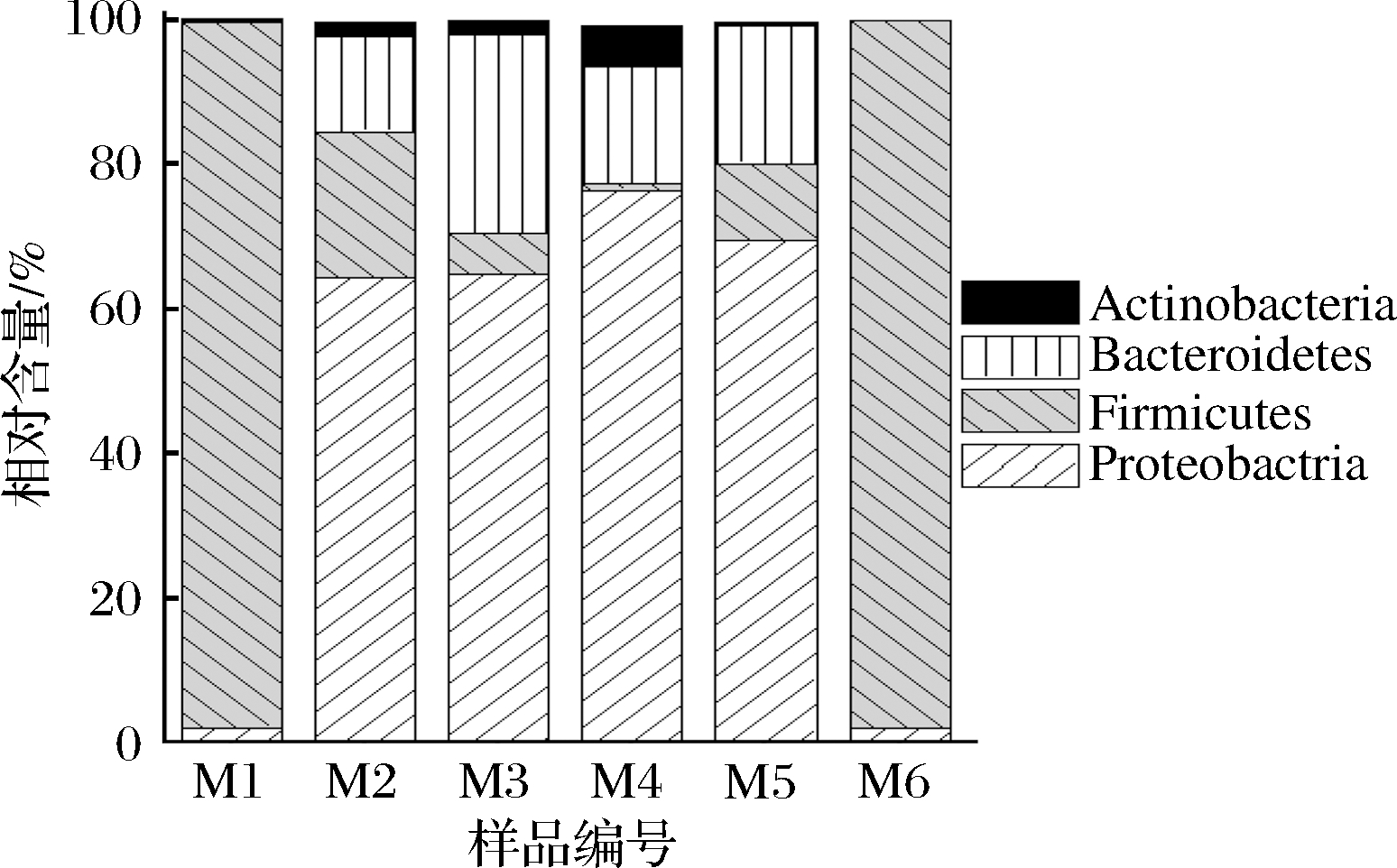
图2 霉豆渣中平均相对含量>1.0%的细菌门
Fig.2 Bacterial phyla with an average relative abundance of >1.0% in Meitauza samples
霉豆渣样品中平均相对含量大于1.0%的优势细菌属构成如图3所示。霉豆渣中平均相对含量大于1.0%的优势菌属包括隶属于Proteobacteria的Acinetobacter(不动杆菌属)、Pseudomonas(假单胞菌属)、Enterobacter(肠杆菌属)、Brevundimonas(短波单胞菌属)、Stenotrophomonas(寡养单胞菌属)和Comamonas(丛毛单胞菌属),其平均相对含量分别为17.27%、10.38%、4.29%、4.03%、3.05%和2.52%,累计相对含量为41.54%;隶属于Firmicutes的Enterococcus(肠球菌属)、Bacillus(芽孢杆菌属)、Brevibacillus(短芽孢杆菌属)和Lysinibacillus(赖氨酸芽胞杆菌属),其平均相对含量分别为1.09%、11.60%、2.41%和21.15%,累计相对含量为36.25%;隶属于Bacteroidetes的Wautersiella(沃特氏菌属)和Sphingobacterium(鞘氨醇杆菌属),其平均相对含量分别为8.68%和2.17%,累计相对含量为10.85%;以及隶属于Actinobacteria的Nocardia(诺片氏菌属),其平均相对含量为1.36%。
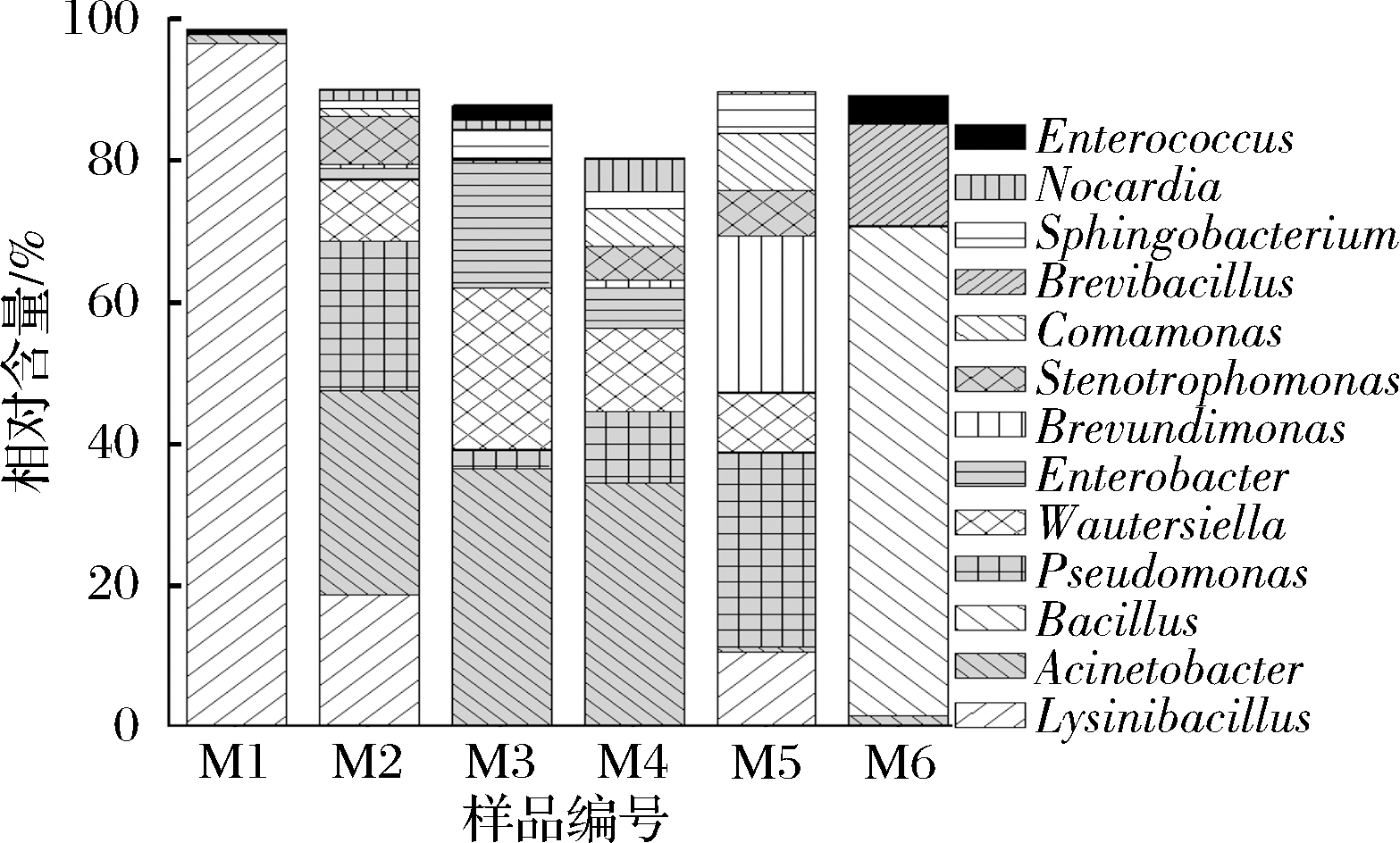
图3 霉豆渣中平均相对含量>1.0%的细菌属
Fig.3 Bacterial genera with an average relative abundance of >1.0% in Meitauza samples
有报道指出隶属于Acinetobacter、Pseudomonas、Brevundimonas、Stenotrophomonas和Comamonas的某些种具有一定的条件致病性,例如隶属于Acinetobacter的A. baumannii(鲍氏不动杆菌)为医院感染的重要病原菌[22],隶属于Pseudomonas的P. aeruginosa(绿脓杆菌)[23]、隶属于Brevundimonas的B. vesicularis(泡囊短波单胞菌)[24]、隶属于Stenotrophomonas的S. maltophilia(嗜麦芽窄食单胞菌)[25]和隶属于Comamonas的C. acidovorans(食酸丛毛单胞菌)可引起肺炎等炎症[26]。Bacillus在发酵过程中产生的次级代谢产物中含多种抗菌物质,其中Lysinibacillus发酵产物有抗真菌活性[27],也可产生蛋白酶,可有效降解豆渣中大分子蛋白为肽类和氨基酸,从而促进人体消化吸收。张燕鹏等[6]在对江西瑞金地区的发酵豆渣进行菌相分析发现豆渣中的细菌主要有Bacillus等,这与本研究发现一致。
由图3亦可知,虽然样品M1和M6在门水平上构成较为相似,但其在属水平上呈现出较大的差异,其中样品M1中占优势的细菌为Lysinibacillus,而样品M6为Bacillus。此外,虽然样品M2、M3、M4和M5中的优势菌门较为相似,但在属水平上亦呈现出较大的差异,样品M2中相对含量占前3位的菌属分别为Acinetobacter、Pseudomonas和Lysinibacillus,其相对含量分别为29.03%、21.22%和18.76%;样品M3为Acinetobacter、Wautersiella和Enterobacter,其相对含量分别为36.50%、23.09%和17.74%;样品M4为Acinetobacter、Wautersiella和Pseudomonas,其相对含量分别为34.62%、11.78%和10.28%;而样品M5为Pseudomonas、Brevundimonas和Lysinibacillus,其相对含量分别为27.73%、22.20%和10.61%。由此可见,在属水平上不同霉豆渣样品的细菌多样性亦呈现出较大的差异。
2.4 基于OTU水平的细菌群落结构分析
本研究共产生了1 355 个OTU,若某一OTU在所有样品中均存在则将其定义为核心OTU[28],本研究共发现了8 个核心OTU,占OTU总数的0.59%,包含的序列数分别占总序列数的9.35%,由此可见,6 个霉豆渣样品中含有的核心细菌类群仅占所有序列数的10%,不同样品间的细菌构成存在较大差异。霉豆渣中核心OTU的相对含量统计结果如表2所示。
表2 霉豆渣中核心OTU的相对含量 单位:%
Table 2 The relative content of core OTU in Meitauza samples
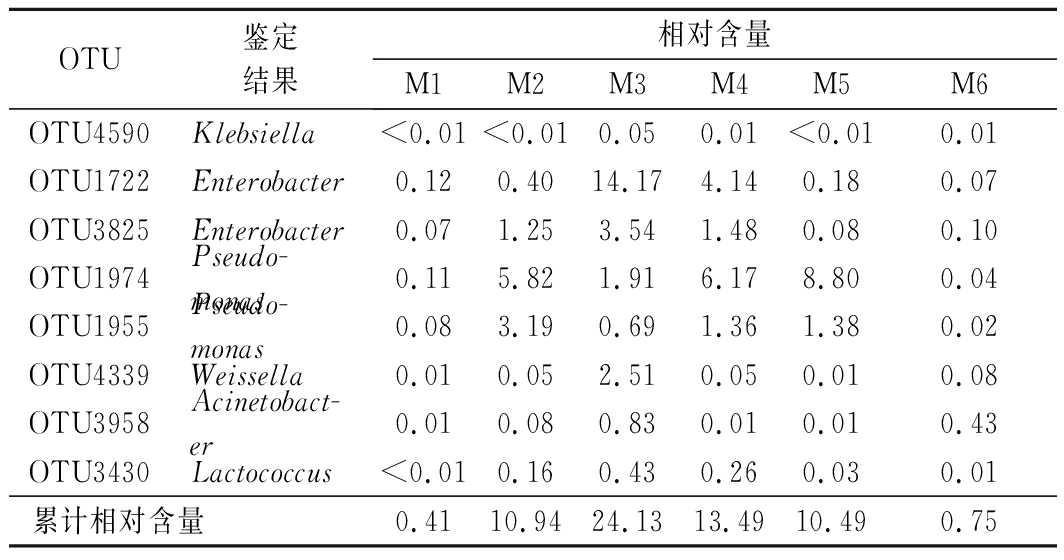
OTU鉴定结果相对含量M1M2M3M4M5M6OTU4590Klebsiella<0.01<0.010.050.01<0.010.01OTU1722Enterobacter0.120.4014.174.140.180.07OTU3825Enterobacter0.071.253.541.480.080.10OTU1974Pseudo-monas0.115.821.916.178.800.04OTU1955Pseudo-monas0.083.190.691.361.380.02OTU4339Weissella0.010.052.510.050.010.08OTU3958Acinetobact-er0.010.080.830.010.010.43OTU3430Lactococcus<0.010.160.430.260.030.01累计相对含量0.4110.9424.1313.4910.490.75
由表2可知,8 个核心OTU,各有2 个被鉴定为Enterobacter和Pseudomonas,其余的4 个各被鉴定为Klebsiella、Weissella、Acinetobacter和Lactococcus。样品M2、M3、M4和M5中核心OTU的累计平均相对含量较高,分别为10.94%、24.13%、13.49%和10.49%,但样品M1和M6中的核心OTU含量仅为0.41%和0.75%。值得一提的是,样品M2和M5中的核心OTU主要隶属于Pseudomonas,累计平均相对含量分别为9.01%和10.18%,样品M3中的核心OTU主要隶属于Enterobacter,累计平均相对含量为17.71%,而M4中的核心OTU主要隶属于Enterobacter和Pseudomonas,累计平均相对含量为5.62%和7.53%。
若某一OTU仅在某一样品中存在,而在其他样品中均不存在,则将其定义为特有OTU[28],霉豆渣样品中特有OTU的相对含量统计结果如表3所示。
表3 霉豆渣中特有OTU的相对含量
Table 3 The relative content of unique OTU in Meitauza samples
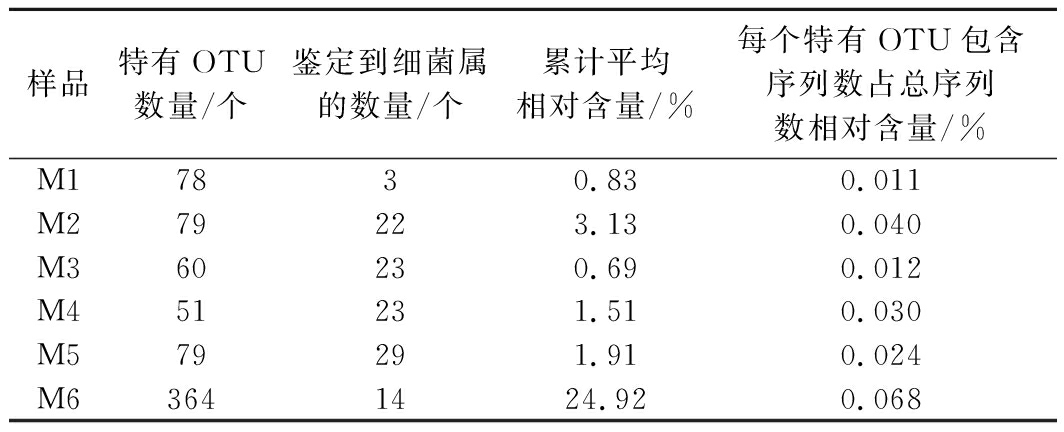
样品特有OTU数量/个鉴定到细菌属的数量/个累计平均相对含量/%每个特有OTU包含序列数占总序列数相对含量/%M17830.830.011M279223.130.040M360230.690.012M451231.510.030M579291.910.024M63641424.920.068
由表3可知,在6 个样品中出现1 次的OTU为711 个,占OTU总数的52.47%,包含的序列数仅占总序列数的6.34%。由此可见,在6 个霉豆渣样品中同时存在,或仅单独存在于某一样品中OTU所包含序列数均较少,可能多数OTU仅存在于2~5 个样品中。
本研究进一步对霉豆渣中OTU出现频率和序列数进行统计,其结果如图4所示。在6 个样品中出现2、3、4和5 次的OTU分别为336、151、104和45 个,分别占OTU总数的24.80%、11.14%、7.68%和3.32%,包含的序列数分别占总序列数的2.17%、24.60%、23.64%和33.90%。由此可见,多数OTU可能存在于3~5个样品中。
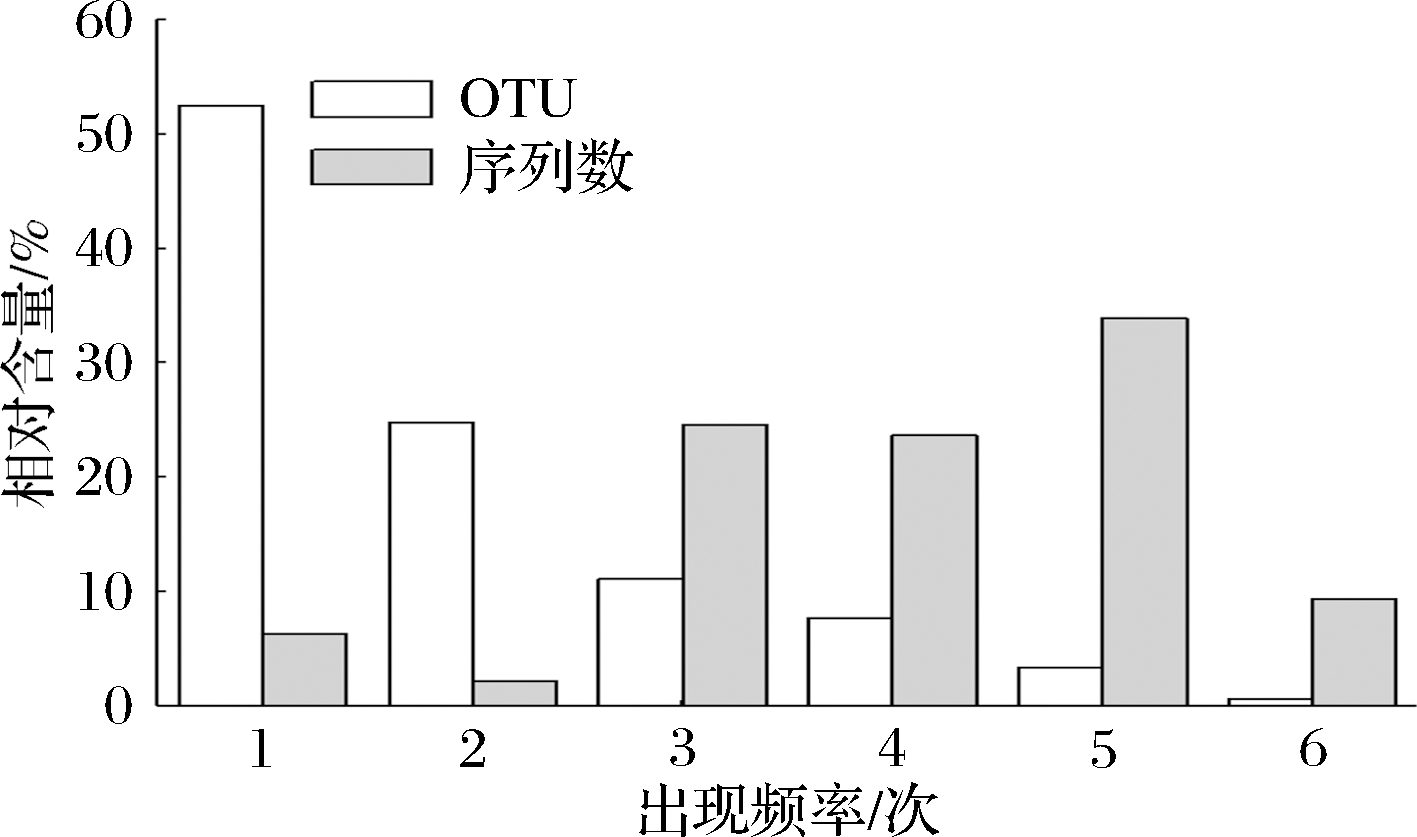
图4 OTU出现频率和序列数统计
Fig.4 Occurrence frequency of OTU and number of sequences within OTU
本课题组前期对米酒曲[29]、辣椒酱[12]和鲊广椒[30]中细菌多样性进行了研究,结果发现上述发酵食品均共有大量的核心细菌菌群,其含量高达80%以上,而本研究中霉豆渣的共有核心细菌菌群仅占10%。米酒曲水分含量较低且添加了凤凰草等中草药,在一定程度上限制了杂菌的生长,使霉菌和戊糖片球菌成为其优势微生物类群[29];而辣椒酱中食盐的添加量高达15%左右,较高的渗透压抑制了多数细菌的生长,仅有芽孢杆菌等耐受性强的细菌得以存活[12];鲊广椒为厌氧发酵,在一定程度上抑制了好氧菌的生长,乳酸菌成为其优势细菌类群[30];而霉豆渣以新鲜豆渣为主要原料,原料含有大量的水分和丰富的营养物质,且发酵方式为开放式发酵,适宜于环境中各类微生物的生长。由此可见,导致不同霉豆渣样品间的细菌构成存在较大差异的根本原因在于其原料渗透压较低、含有丰富营养物质和开放式的发酵方式。
2.5 基因功能预测
在对霉豆渣核心细菌类群解析的基础上,进一步对其基因功能进行了预测,结果如图5所示。
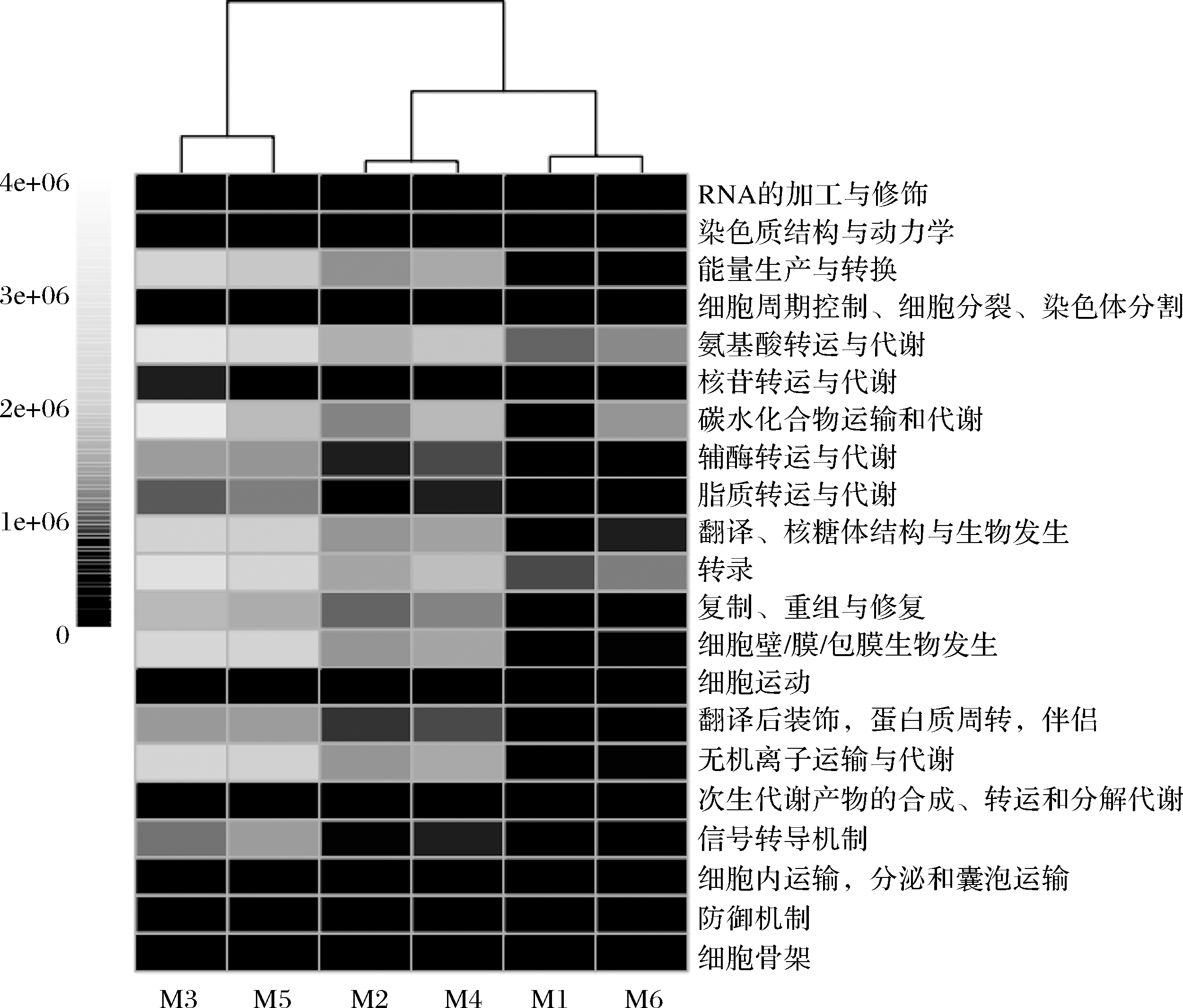
图5 霉豆渣中细菌的基因功能预测
Fig.5 The gene functional prediction of bacteria in Meitauza samples
本研究从霉豆渣细菌中共注释到4 106 个COG,分别隶属于23 个功能类别,样品M1~M6中各有占总序列数10.47%、10.13%、9.58%、9.86%、10.83%和10.77%的序列无法预测其功能。由图5可知,碳水化合物的运输与代谢、氨基酸的转运与代谢在样品中表达较强,RNA的加工与修饰及细胞骨架在样品中表达较低;样品被分为2个聚类,一类是由样品M3和M5构成,另一类是由样品M1、M2、M4和M6构成,表明样品M3与M5细菌功能较为相似,与另外4 个样品细菌功能有部分差异。豆渣中蛋白质含量较高,发酵过程中某些细菌的次级代谢产物可产生蛋白酶,在蛋白酶的作用下蛋白质分解成游离氨基酸,游离氨基酸越多,霉豆渣味道越鲜美[31]。
3 结论
本研究发现霉豆渣中优势细菌门为Proteobacteria、Firmicutes、Bacteroidetes和Actinobacteria;优势细菌属为Acinetobacter、Pseudomonas、Brevundimonas、Stenotrophomonas、Comamonas、Enterococcus、Bacillus、Brevibacillus、Lysinibacillus、Wautersiella、Sphingobacterium和Nocardia;碳水化合物的运输与代谢和氨基酸的转运与代谢在霉豆渣细菌类群中具有较强的表达能力。不同霉豆渣样品间细菌类群存在较大的差异,究其原因可能与制作环境较为开放及其自身营养物质丰富有关。
[1] 姜慧燕, 邹礼根,翁丽萍,等.豆渣营养成分分析及蛋白质营养价值评价[J].食品工业,2020,41(6):325-328.
JIANG H Y,ZOU L G,WENG L P,et al.Nutritional components analyzation and protein nutrition evaluation of soybean residue[J].The Food Industry,2020,41(6):325-328.
[2] 万茵, 王登骁,肖明,等.米根霉NCU1011发酵豆渣开发甜酱生产工艺研究[J].中国调味品,2020,45(4):24-28.
WAN Y,WANG D X,XIAO M,et al.Study on production technology of Rhizopus oryzae NCU1011 fermented soybean residue for developing sweet soybean paste[J].Chinese Condiment,2020,45(4):24-28.
[3] 徐书泽, 黄丽,滕建文,等.传统发酵霉豆渣中产酶优势菌的分离鉴定及性质研究[J].食品与发酵工业,2014,40(3):102-106.
XU S Z,HUANG L,TENG J W,et al.Isolation and identification of dominant microorganism in traditional fermentative Meidouzha and characterization of dominant strain[J].Food and Fermentation Industries,2014,40(3):102-106.
[4] 石威. 自然发酵豆渣中微生物分离、鉴定及混合发酵豆渣产品的研制[D].南昌:南昌大学,2012.
SHI W.Microbes isolation,identification of natural fermented okara and the products development of mixed fermented okara[D].Nanchang:Nanchang University,2012.
[5] 姚英政. 霉豆渣粑发酵过程中营养及风味成分变化研究[D].武汉:华中农业大学,2010.
YAO Y Z.Study on the changes in nutrition and flavor ingredients during soybean residue cakes fermentation[D].Wuhan:Huazhong Agricultural University,2010.
[6] 张燕鹏, 杨瑞金,王贺,等.传统豆渣菌的菌相分析及蛋白酶和纤维素酶主要产生菌株的鉴定[J].食品工业科技,2012,3(1):171-174.
ZHANG Y P,YANG R J,WANG H,et al.Analysis of the microflora and identification of the protease and cellulase producing strains from the traditional fermentative Douzhajun[J].Science and Technology of Food Industry,2012,3(1):171-174.
[7] LITTLEFAIR J E,CLARE E L.Barcoding the food chain:From Sanger to high-throughput sequencing[J].Genome,2016,59(11):946-958.
[8] LU S Y,ZUO T,ZHANG N,et al.High throughput sequencing analysis reveals amelioration of intestinal dysbiosis by squid ink polysaccharide[J].Journal of Functional Foods,2016,20(1):506-515.
[9] WAN H F,LIU T,SU C W,et al.Evaluation of bacterial and fungal communities during the fermentation of Baixi sufu,a traditional spicy fermented bean curd[J].Journal of the Science of Food and Agriculture,2020,100(4):1 448-1 457.
[10] TANG Y,ZHOU X,HUANG S,et al.Microbial community analysis of different qualities of pickled radishes by Illumina MiSeq sequencing[J].Journal of Food Safety,2019,39(2).DOI:10.111/jfs.12596.
[11] 赵馨馨, 崔梦君,董蕴,等.应用Illumina MiSeq高通量测序技术分析巴东地区豆瓣酱中微生物多样性[J].现代食品科技,2019,35(9):297-303.
ZHAO X X,CUI M J,DONG Y,et al.Analysis of microbial diversity in Badong bean paste by Illumina MiSeq high throughput sequencing technology[J].Modern Food Science and Technology,2019,35(9):297-303.
[12] 沈馨, 王艳,代凯文,等.基于Miseq高通量测序技术的辣椒酱核心细菌类群研究[J].食品研究与开发,2018,39(10):151-157.
SHEN X,WANG Y,DAI K W,et al.Characterization of core bacterial microflora in chilli sauce by Miseq high throughput sequencing technologies[J].Food Research and Development,2018,39(10):151-157.
[13] 尚雪娇, 马磊,余海忠,等.基于Miseq测序技术的琚湾酸浆面浆水真菌多样性评价[J].食品研究与开发,2018,39(16):158-163.
SHANG X J,MA L,YU H Z,et al.Study on the diversity of fungal microflora in Suanjiangmian Jiangshui of Juwan by Miseq sequencing[J].Food Research and Development,2018,39(16):158-163.
[14] CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7(4):335-336.
[15] CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST:A flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[16] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2 460-2 461.
[17] DESANTIS T Z,HUGENHOLTZ P,LARSENN,et al.Greengenes,a chimera checked 16S rRNA gene database and workbench compatible with ARB[J].Applied and Environmental Microbiology,2006,72(7):5 069-5 072.
[18] COLE J R,CHAI B,FARRIS R J,et al.The ribosomal database project (RDP-II):Introducing myRDP space and quality controlled public data[J].Nucleic Acids Research,2007,35(1):169-172.
[19] PRICE M N,DEHAL P S,ARKIN A P.Fasttree:Computing large minimum evolution trees with profiles instead of a distance matrix[J].Molecular Biology and Evolution,2009,26(7):1 641-1 650.
[20] WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI:A rumen microbiome focused version of the PICRUSt functional inference software[J].Frontiers in Microbiology,2018,9(5):1095.
[21] GALPERIN M Y,KRISTENSEN D M,MAKAROVA K S,et al.Microbial genome analysis:The COG approach[J].Briefings in Bioinformatics,2019,20(4):1 063-1 070.
[22] SCHOOLEY R T,BISWAS B,GILL J J,et al.Development and use of personalized bacteriophage-based therapeutic cocktails to treat a patient with a disseminated resistant Acinetobacter baumannii infection[J].Antimicrobial Agents and Chemotherapy,2017,61(10):1-14.
[23] DARCH S E,SIMOSKA O,FITZPATRICK M,et al.Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model[J].Proceedings of the National Academy of Sciences of the United States America,2018,115(18):4 779-4 784.
[24] BOLZON C,NGUYEN B H.A rare case of peritonitis due to Brevundimonas vesicularis[J].Journal of Community Hospital Internal Medicine Perspectives,2018,8(3):161-162.
[25] BERDAH L,TAYTARD J,LEYRONNAS S,et al.Stenotrophomonas maltophilia:A marker of lung disease severity[J].Pediatric Pulmonology,2018,53(4):426-430.
[26] RUDAKIYA D M.Metal tolerance assisted antibiotic susceptibility profiling in Comamonas acidovorans[J].BioMetals,2018,31(1):1-5.
[27] 郑雯, 杨兴变,康冀川,等.赖氨酸芽孢杆菌He14发酵产物抗真菌活性的初步研究[J].食品与发酵工业,2019,45(18):22-26.
ZHENG W,YANG X B,KANG J C,et al.Antifungal activity of fermentation broth of Lysinibacillus fusiformis He14[J].Food and Fermentation Industries,2019,45(18):22-26.
[28] ZHANG J,GUO Z,XUE Z,et al.A phylo-functional core of gut microbiota in healthy young Chinese cohorts across lifestyles,geography and ethnicities[J].The ISME Journal,2015,9(9):1 979-1 990.
[29] 向凡舒, 朱媛媛,邓风,等.建始地区米酒曲细菌和真菌多样性研究[J].食品工业科技,2020,42(14):1-10.
XIANG F S,ZHU Y Y,DENG F,et al.Study on bacterial and fungal diversity of rice wine koji in Jianshi Aera[J].Science and Technology of Food Industry,2020,42(14):1-10.
[30] 王玉荣, 沈馨,董蕴,等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械,2018,34(4):25-30.
WANG Y R,SHEN X,DONG Y,et al.Characterization of bacterial microflora and their functions on flavor quality[J].Food and Machinery,2018,34(4):25-30.
[31] 李润生. 霉豆渣生产工艺的研究报告[J].食品科学,1982,3(9):43-46.
LI R S.Research report on the production technology of Meitauza[J].Food Science,1982,3(9):43-46.