鲊广椒是一种以玉米面或大米面或同时2种面粉与鲜辣椒混合作为主料,辅以盐、胡椒粉和花椒等辅料密封发酵制成的一种传统发酵食品,深受我国四川、重庆、贵州、云南以及湖北等省市地区人民的喜爱[1]。洪湖市位于湖北省荆州市,该地区湖泊众多,地理环境独特,出产的鲊广椒口感酸辣,风味较为特别。由于制作的环境较为开放,鲊广椒中的微生物多样性易受到地区环境与制作工艺的影响[2],推测风味独特的洪湖鲊广椒中可能蕴含着一些独特的微生物类群。
近些年,Illumina MiSeq高通量技术由于具有测序通量高、检测速度快和灵敏度高的特点,倍受研究人员的喜爱,被广泛应用于辣椒酱[3]、腊肉[4]、黄酒[5]以及大头菜[6]等食品中微生物群落结构组成研究。第二代测序技术Illumina MiSeq平台不仅降低了测序成本还增加了样品的测序深度,并且具有精确度高和测序量大等特点[7-9],能更加真实地反映发酵食品中微生物群落的结构[10]。既往研究发现多数鲊广椒的乳酸菌相对含量较高,并且添加乳酸菌还可以改善鲊广椒的风味和滋味[11-13]。然而,目前少有对于风味与制作环境较为独特的洪湖鲊广椒的研究报道,对其中蕴含的乳酸菌菌种资源仍不可知。
由于乳酸菌是目前食品领域使用的重要菌种资源,本研究拟同时采用传统可培养方法与MiSeq高通量技术对鲊广椒中乳酸菌多样性进行解析,明确洪湖鲊广椒中的乳酸菌群落组成以及特色菌群,以期为洪湖地区鲊广椒中乳酸菌菌种资源的保存、开发与利用提供实验依据,更可为鲊广椒的产业化提供优质的乳酸菌资源。
1 材料与方法
1.1 材料与试剂
从湖北省荆州市洪湖市农贸市场采集不同农户自制鲊广椒共7份,分别编号为HH1、HH2、HH3、HH4、HH5、HH6和HH7,置于含有冰袋的采样箱中迅速运回实验室进行分装,1份于-70 ℃贮存,用于高通量测序;另1份用于乳酸菌的分离。
dNTPs Mix、FastPfu Fly DNA Polymerase、5×TransStartTM FastPfu Buffer,北京全式金生物技术有限公司;QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒,德国QIAGEN公司;克隆载体pMD18-T,大连宝生物工程有限公司;MRS培养基(酵母膏、牛肉膏、蛋白胨、柠檬酸二胺、KH2PO4、MgSO4·7H2O、MnSO4·H2O、NaAc、葡萄糖和吐温-80,均为分析纯),国药集团化学试剂有限公司;琼脂糖,索来宝生物技术有限公司;琼脂粉,北京奥博星生物技术有限公司;PCR清洁试剂盒,Axygen生物技术(杭州)有限公司。
1.2 仪器与设备
Veriti FAST梯度PCR仪,美国ABI公司;UVPCDS8000凝胶成像分析系统,美国ProteinSimple公司;HR40-IIB2生物安全柜,青岛海尔特种电器有限公司;DG250厌氧工作站,英国DonWhitley公司;LRH-250生化恒温培养箱,上海力辰仪器科技有限公司;Illumina MiSeq高通量测序平台,美国Illumina公司;ND-2000C微量紫外分光光度计,美国Nano Drop公司;离心机,德国Eppendorf公司。
1.3 实验方法
1.3.1 鲊广椒中微生物宏基因组DNA提取
按照基因组DNA提取试剂盒说明书提取鲊广椒样品的宏基因组DNA,将通过琼脂糖凝胶电泳与微量紫外分光光度计检验合格的DNA置于-20 ℃中暂存,备用。
1.3.2 鲊广椒样品的高通量测序
以提取到的鲊广椒样品总DNA为模板,使用加入了成对碱基核苷酸标签的338F/806R引物,参照王玉荣等[14]的PCR扩增体系和扩增条件对鲊广椒样品中的细菌16S rRNA V3~V4区进行PCR扩增。使用的引物为:338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)。将扩增产物用15 g/L的琼脂糖凝胶电泳检测,检测合格后的PCR扩增产物在Illumina MiSeq PE300高通量测序平台进行高通量测序。
1.3.3 序列拼接和质量控制
参照王玉荣等[15]的方法对下机后的数据进行拼接和质控,去除不合格的序列(重叠区碱基数≤10 bp,引物错配数≥2 bp且去除引物后序列长度≤50 bp的序列)后,使用质控后的优质序列进行生物信息学分析。
1.3.4 生物信息学分析
参照郭壮等[16]的方法,对质控后的序列在QIIME分析平台进行生物信息学分析。首先将序列进行标准比对和对齐,分别以100%和97%相似度进行两步UCLUST划分,从而建立分类操作单元(operational taxonomic units,OTU);接下来剔除含有嵌合体的OTU序列,选取代表性序列在GREENGENE和RDP数据库中进行同源性比对,从而在门、纲、目、科和属水平上对样品中的细菌进行解析;计算Chao1指数、Simpson指数和Shannon指数从而对样品中微生物的多样性和丰度进行评价。
1.3.5 鲊广椒中乳酸菌的分离纯化
取10 g鲊广椒样品,移入含90 mL无菌生理盐水的三角瓶,然后充分振荡混匀。将样品做10倍的梯度稀释。完成后,吸取50~100 μL的样品稀释液(稀释度为10-3、10-4、10-5)均匀的涂布于碳酸钙质量浓度为10 g/L的MRS固体平板上,置于30 ℃厌氧工作站[V(N2)∶V(CO2)∶V(H2)=85∶10∶5]中培养72 h。随后挑选大小、颜色、形态均不相同且有透明圈的单菌落进行至少3 次划线纯化,并将革兰氏阳性,过氧化氢酶阴性的乳酸菌使用含有30%(体积分数)甘油的MRS液体培养基重悬,然后冻存于-80 ℃超低温冰箱,待用。
1.3.6 鲊广椒中乳酸菌的鉴定
使用CTAB法提取乳酸菌的DNA[17],参照郭壮等[18]的扩增方法进行PCR扩增,并对扩增产物进行检测、清洁、连接、转化和克隆子的鉴定,挑选出阳性克隆子送至武汉天一辉生物有限公司进行测序。将返回来的测序序列用软件DNAMAN拼接,手动去引物后,在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库中进行BLASTn比对,选取序列相似度≥99%的16S rRNA模式菌株序列,使用软件MEGA 5.0中的邻接法(neighbor joining,NJ)构建系统发育树,确定分离到的乳酸菌的系统分类地位。
1.3.7 数据处理
使用软件MEGA 5.0中的NJ构建系统发育树,并用Adobe Illustrator软件导出系统发育进化树;采用相关性分析法对鲊广椒样品中平均相对含量>1.0%的优势细菌门和属进行相关性研究;使用R软件(v3.5.3)和软件Origin 2017绘图。
2 结果与分析
2.1 洪湖鲊广椒细菌多样性指数分析
本研究采集了7份洪湖鲊广椒样品,通过Illumina Miseq高通量测序技术对洪湖鲊广椒样品细菌多样性进行了解析。通过生物信息学技术对鲊广椒样品的细菌多样性分析结果如表1所示。
表1 洪湖鲊广椒样品的测序数量与细菌多样性分析
Table 1 Sequencing quantity and bacterial diversity analysis of Honghu Zha-chili
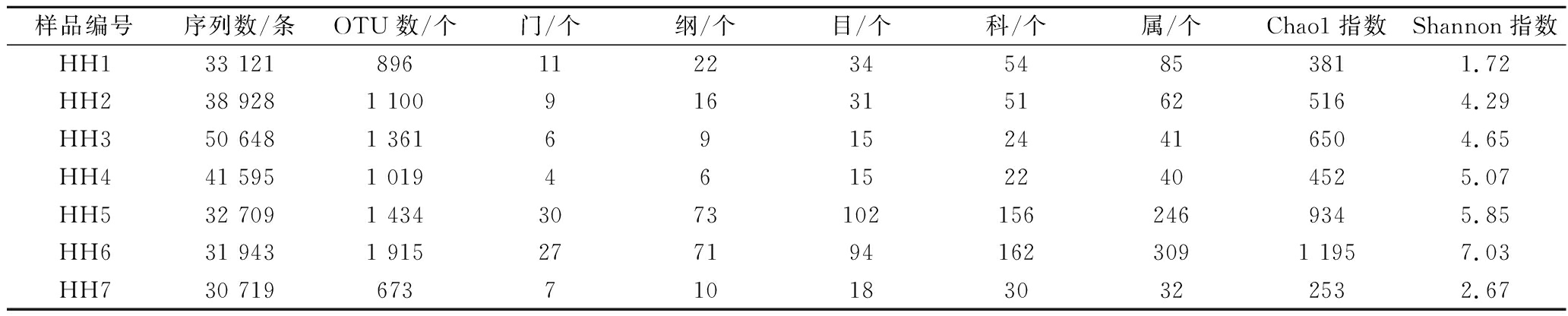
样品编号序列数/条OTU数/个门/个纲/个目/个科/个属/个Chao1指数Shannon指数HH133 12189611223454853811.72HH238 9281 1009163151625164.29HH350 6481 361691524416504.65HH441 5951 019461522404525.07HH532 7091 43430731021562469345.85HH631 9431 9152771941623091 1957.03HH730 7196737101830322532.67
注:Chao1指数和Shannon指数均是在测序量为30 010条序列时所得
由表1可知,高通量测序共产生了259 663 条高质量序列,平均每个样品37 095条。按照100%和97%相似度对序列进行划分且去除嵌合体后共得到8 398个OTU。HH6样品的Chao1指数和Shannon指数均最大,分别为1 195和7.03,由此可见,HH6样品细菌微生物的丰富度最大且多样性最高,细菌群落结构组成可能最为复杂。
2.2 洪湖鲊广椒细菌群落结构分析
在对样品序列丰富度和多样性分析的基础上,进一步对质控合格的序列进行了鉴定,共鉴定出36个门,96个纲,137个目,204个科和403个属,其中仅有0.11%和6.02%的序列鉴定不到门和属水平。将本研究的鲊广椒样品中平均相对含量>1.0%的门和属定义为优势门和优势属,鲊广椒样品中平均相对含量>1.0%的优势细菌门组成如图1所示。

图1 洪湖鲊广椒中的优势细菌门
Fig.1 Dominant phylum in Honghu Zha-chili
由图1可知,鲊广椒样品中细菌平均相对含量>1.0%的细菌门有4个,分别是硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),其平均相对含量分别为77.78%、18.09%、1.74%和1.51%。值得一提的是,硬壁菌门在样品HH3、HH4和HH7中较高,相对含量分别为98.71%、98.40%和99.49%。洪湖鲊广椒中的共有优势菌门为硬壁菌门、变形菌门和放线菌门,累计平均相对含量超过了97.38%。
同样的,本研究在属的分类水平上对鲊广椒样品中的细菌进行了分析,结果如图2所示。

图2 洪湖鲊广椒中的优势细菌属
Fig.2 Dominant bacteria genera in Honghu Zha-chili
由图2可知,洪湖鲊广椒中优势细菌属共有7个,分别是隶属于硬壁菌门的乳杆菌属(Lactobacillus)和海洋芽孢杆菌属(Oceanobacillus),其平均相对含量分别为67.97%和3.88%;隶属于变形菌门的盐单胞菌属(Halomonaas)、不动杆菌属(Acinetobacter)、雷尔氏菌属(Ralstonia)、假单胞菌属(Pseudomonas)和草螺菌属(Herbaspirillum),平均相对含量分别为3.47%、1.83%、1.75%、1.53%和1.02%。乳杆菌属在样品HH1、HH3、HH4和HH7中含量最高,相对含量均超过了85%。由此可见,洪湖鲊广椒样品中主要的细菌是隶属于硬壁菌门的乳杆菌属。
本研究中的高通量测序数据共产生了8 398个OTU,进一步对平均相对含量>1.0%的优势OTU进行了分析。平均相对含量>1.0%的OTU如图3所示。
由图3可知,洪湖鲊广椒样品中含12个平均相对含量>1.0%的OTU,分别是OTU3336、OTU4099、OTU5847、OTU1749、OTU357、OTU2214、OTU5962、OTU1973、OTU7126、OTU7065、OTU6541和OTU4775,其平均相对含量分别为3.30%、1.20%、1.60%、1.66%、1.80%、2.01%、2.89%、3.75%、5.31%、8.12%、8.78%和12.83%。经序列比对分析发现,OTU3336隶属于盐单胞菌属;OTU4099隶属于海洋芽孢杆菌属;OTU5847隶属于雷尔氏菌属;OTU1749、OTU357、OTU2214、OTU5962、OTU1973、OTU7126、OTU7065、OTU6541和OTU4775隶属于乳杆菌属。由此可见,基于属水平及OTU水平的分析均表明,乳杆菌属仍然是洪湖地区鲊广椒样品中的优势细菌。

图3 平均相对含量大于1.0%的OTU方块图
Fig.3 OTU block diagram with average relative content greater than 1.0%
乳酸杆菌最初在1986年被描述,到2019年为止一共建立了289个种水平的分类单元[19]。乳酸杆菌被认为是安全的,可用于食品行业的微生物中,乳酸杆菌占了很大比例。在当阳[20]、恩施[21]、荆州[22]等多个地区的鲊广椒中,乳杆菌属都是优势属,可能对食品的滋味和风味形成具有重要影响[13,23]。洪湖鲊广椒中乳酸杆菌对其特色风味物质的作用仍有待于进一步研究。
2.3 乳酸菌的分离与系统分类
由高通量测序可知,洪湖鲊广椒样品中的乳酸菌以乳酸杆菌为主,而乳酸杆菌在食品等领域具有重要的应用价值,因此接下来对洪湖鲊广椒中的乳酸菌进行了分离与鉴定。本研究使用碳酸钙含量为10 g/L的MRS平板对洪湖鲊广椒样品中的乳酸菌进行了分离,并使用平板划线法进行纯化。将具有透明圈,革兰氏染色为阳性,且过氧化氢酶阴性的菌株初步判定为乳酸菌[24]。从7份洪湖鲊广椒样品中共分离出29 株疑似乳酸菌菌株,均是杆菌,其特征描述见表2。
表2 洪湖鲊广椒样品来源的乳酸菌菌落形态
Table 2 Colony morphology of lactic acid bacteria from Honghu Zha-chili

菌株编号菌落形态革兰氏染色过氧化氢酶菌株登录号HBUAS56256圆形,乳白,不透明,边缘整齐,较小+-MT231745HBUAS56257圆形,乳白,不透明,边缘整齐,较小+-MT231746HBUAS56258圆形,乳白,不透明,边缘整齐,较小+-MT231747HBUAS56259圆形,乳白,不透明,边缘整齐,小+-MT231748HBUAS56260圆形,乳白,半透明,边缘整齐,大+-MT231749HBUAS56261圆形,白色,不透明,边缘整齐,较大+-MT231750HBUAS56262圆形,白色,不透明,边缘粗糙,大+-MT231751HBUAS56263圆形,白色,不透明,边缘粗糙,大+-MT231752HBUAS56264圆形,乳白,不透明,边缘粗糙,较小+-MT231753HBUAS56265圆形,乳白,不透明,边缘整齐,较小+-MT231754HBUAS56266圆形,乳白,不透明,边缘整齐,较小+-MT231755HBUAS56267圆形,乳白,不透明,边缘粗糙,小+-MT231756HBUAS56268圆形,乳白,不透明,边缘整齐,较小+-MT231757HBUAS56269圆形,白色,不透明,边缘整齐,大+-MT231758HBUAS56270圆形,白色,不透明,边缘粗糙,小+-MT231759HBUAS56271圆形,乳白,半透明,边缘整齐,较小+-MT231760HBUAS56272圆形,乳白,半透明,边缘粗糙,较小+-MT231761HBUAS56273圆形,乳白,不透明,边缘整齐,大+-MT231762HBUAS56274圆形,乳白,不透明,边缘整齐,大+-MT231763HBUAS56275圆形,乳白,不透明,边缘整齐,小+-MT231764HBUAS56276圆形,白色,不透明,边缘整齐,大+-MT231816HBUAS56277圆形,乳白,不透明,边缘整齐,大+-MT231765HBUAS56278圆形,白色,不透明,边缘整齐,较大+-MT231766HBUAS56279圆形,白色,不透明,边缘整齐,大+-MT231767HBUAS56280圆形,乳白,半透明,边缘整齐,大+-MT231768HBUAS56282圆形,乳白,不透明,边缘整齐,大+-MT231769HBUAS56283圆形,乳白,不透明,边缘整齐,大+-MT231770HBUAS56284圆形,乳白,不透明,边缘整齐,较小+-MT231771HBUAS56286圆形,乳白,不透明,边缘整齐,较小+-MT231772
注:“+”为阳性反应,“-”为阴性反应
本研究对获得的29 株疑似乳酸菌进行了基因组DNA提取,细菌16S rRNA基因的PCR扩增、TA克隆与测序,将返回来的序列经手动拼接和去引物后,通过NCBI数据库进行BLASTn比对。选取序列相似度达到99%以上的16S rRNA模式序列构建系统发育树,基于16S rRNA基因序列构建的系统发育树如图4所示。
由图4可知,菌株HBUAS56267、HBUAS56268、HBUAS56272、HBUAS56274、HBUAS56276、HBUAS56279、HBUAS56280和HBUAS56282与模式菌株Lactobacillus futsaii JCM 17355的系统发育关系最近,其16S rRNA序列相似度超过了99%,被鉴定为福莱乳杆菌(L.futsaii);菌株HBUAS56258、HBUAS56259和HBUAS56260与模式菌株Lactobacillus zhachilii HBUAS52074的系统发育关系最近,被鉴定为鲊广椒乳杆菌(L.zhachilii);菌株HBUAS56257和HBUAS56275与模式菌株L. paralimentarius JCM 10415的在系统发育树上形成了一个分支,被鉴定为类消化乳杆菌(L.paralimentarius);菌株HBUAS56264、HBUAS56266和HBUAS56286与模式菌株L.alimentarius DSM 20249的系统发育关系最近,被鉴定为消化乳杆菌(L.alimentarius);菌株HBUAS56256、HBUAS56261、HBUAS56262、HBUAS563、HBUAS56269、HBUAS56270、HBUAS56273、HBUAS56277、HBUAS56278和HBUAS56284被鉴定为植物乳杆菌(L.plantarum);菌株HBUAS56265与模式菌株Pediococcus ethanolidurans Z9位于系统发育树的同一分支上,因而被鉴定为耐乙酸片球菌(P.ethanolidurans);菌株HBUAS56271与模式菌株L.acidipiscis NBRC 102163的系统发育关系最近,被鉴定为酸鱼乳杆菌(L.acidipiscis);菌株HBUAS5628被鉴定为绿色魏斯氏菌(Weissella viridescens)。
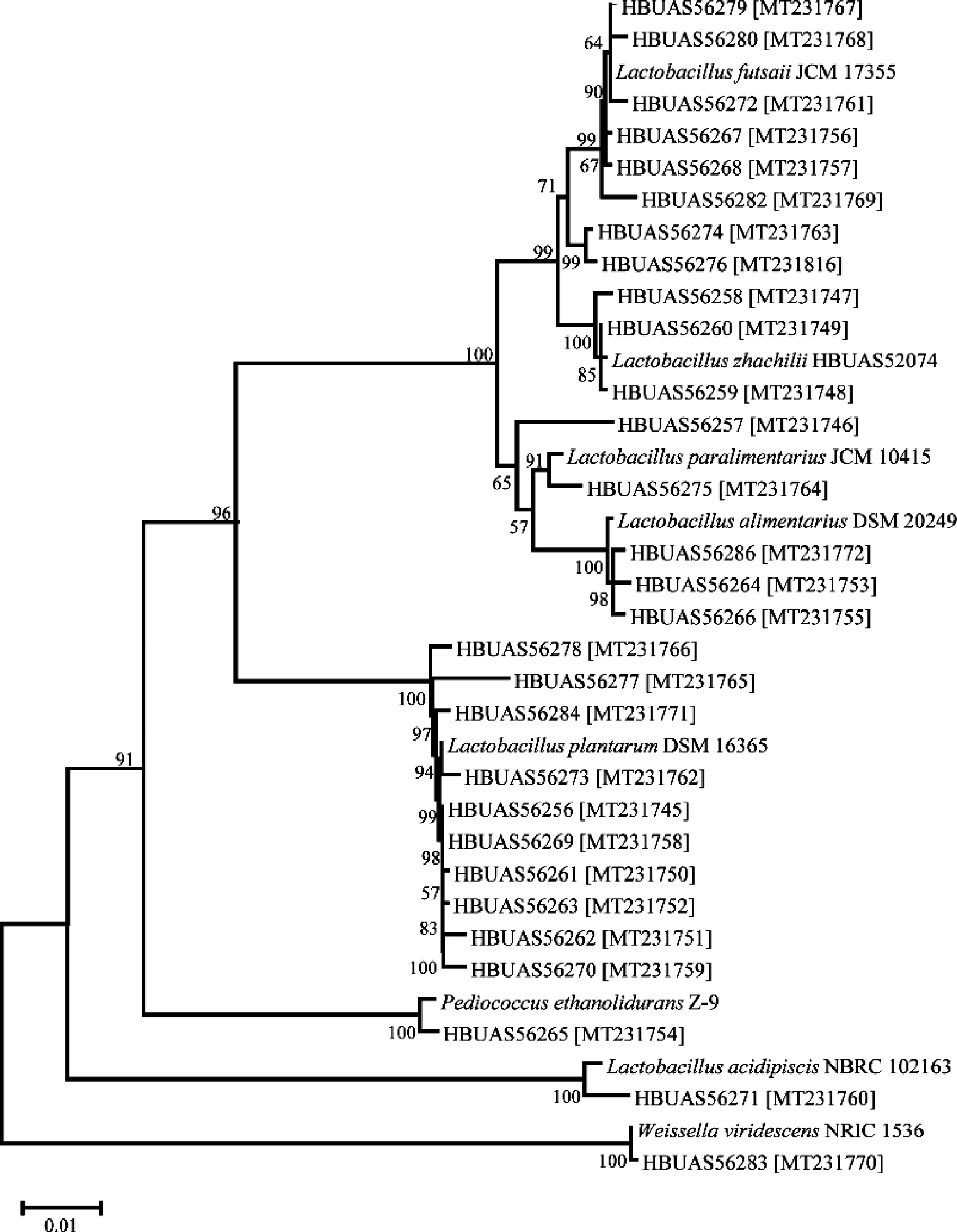
图4 基于16S rRNA序列的洪湖鲊广椒来源的乳酸菌邻接法系统发育树
Fig.4 NJ phylogenetic tree of lactic acid bacteria from Honghu Zha-chili based on 16S rRNA sequence
总的来说,本研究从洪湖鲊广椒样品中共分离出来29株乳酸菌,鉴定为3个属和8个种,其中有植物乳杆菌(L.plantarum)10 株,福莱乳杆菌(L.futsaii)有8株,分别占总分离菌株数的34.48%和27.59%。因此,洪湖地区鲊广椒中除植物乳杆菌外,还含有较高比例的福莱乳杆菌(L.futsaii),可能是洪湖地区鲊广椒的特色细菌类群,而该结果与邓风[25]、雷炎[21]与葛东颖等[22]对鲊广椒的研究结果并不一致。
3 结论
本研究采用Illumina Miseq高通量测序技术结合传统可培养方法对7 份洪湖地区鲊广椒样品中乳酸菌多样性进行了解析。研究发现,硬壁菌门、变形菌门、拟杆菌门和放线菌门是洪湖鲊广椒的优势细菌门;乳杆菌属、海洋芽孢杆菌属、盐单胞菌属、不动杆菌属、雷尔氏菌属、假单胞菌属和草螺菌属是洪湖鲊广椒的优势细菌属,其中乳杆菌属含量最高。此外,研究发现从洪湖鲊广椒样品中分离出来的29 株乳酸菌中植物乳杆菌有10株,福莱乳杆菌有8株,分别占总分离菌株数的32.26%和25.81%,后者可能为洪湖鲊广椒样品中的特色优势菌群。
[1] 任聪, 杜海, 徐岩.中国传统发酵食品微生物组研究进展[J].微生物学报, 2017, 57(6):885-898.
REN C, DU H, XU Y.Advances in microbiome study of traditional Chinese fermented foods[J].Acta Microbiologica Sinica, 2017, 57(6):885-898.
[2] LOIZOU S, MATTAS K, TZOUVELEKAS V, et al.Regional economic development and environmental repercussions:An environmental input-output approach[J].International Advances in Economic Research, 2000, 6(3):373-386.
[3] 宁明, 赵馨馨, 董蕴, 等.基于Illumina MiSeq测序技术不同地区辣椒酱细菌多样性分析[J].中国调味品, 2020, 45(2):58-63.
NING M, ZHAO X X, DONG Y, et al.Analysis of bacterial diversity of chili sauce in different areas based on Illumina MiSeq sequencing technology[J].China Condiment, 2020, 45(2):58-63.
[4] 董蕴, 王玉荣, 王尧, 等.基于变性梯度凝胶电泳和MiSeq高通量测序技术分析恩施地区腊肉的细菌多样性[J].肉类研究, 2018, 32(10):37-42.
DONG Y, WANG Y R, WANG Y, et al. Evaluation of bacterial diversity in Chinese bacon from Enshi by denatured gradient gel electrophoresis and MiSeq high-throughput sequencing[J]. Meat Research, 2018, 32(10): 37-42.
[5] 王洋怡舟, 李柏林, 欧杰, 等.绍兴黄酒酒曲总DNA的提取及其真菌多样性[J].食品工业科技, 2016, 37(15):176-180;186-187.
WANG Y Y Z, LI B L, OU J, et al.Extracting total DNA in Shaoxing rice wine wheat starter and its fungal diversity[J].Science and Technology of Food Industry, 2016, 37(15):176-180;186-187.
[6] 赵慧君, 沈馨, 董蕴, 等.襄阳大头菜腌制液生物膜真菌多样性研究[J].中国调味品, 2017, 42(12):61-65.
ZHAO H J, SHEN X, DONG Y, et al.Study on the diversity of fungal microflora of biomembrane in Xiangyang mustard root bring[J].China Condiment, 2017, 42(12):61-65.
[7] CHEN F, ZHU X L, CUI Q, et al.The partition of regional-specific environment management area for the China pulp & paper industry based on resources and environment carrying capacity[J].Advanced Materials Research, 2013, 726-731:4 163-4 166.
[8] MIKEL B, JOOST H, THOMAS B.The determinants of regional innovation in Europe:A combined factorial and regression knowledge production function approach[J].Research policy, 2010, 39(6):722-735.
[9] LIU X X, YUAN W J, DING T, et al.Analysis of microbial community structure of HuaiYuan, Sichuan special fermented food based on high-throughput sequencing technology[J].Journal of Sichuan University (NATURAL SCIENCE EDITION), 2019, 56(3):537-543.
[10] HIDEKI T, TAKEO H, TOSHIMITSU F, et al.Special articles on global and regional environment and chemistry:Behavior of dissolved organic substances in a shallow lake water, the western part of lake inba-numa[J].Nippon Kagaku Kaishi, 1991(6):852-858.
[11] 向凡舒, 王玉荣, 葛东颖, 等.湖北夷陵地区鲊广椒中乳酸菌的分离鉴定及其对产品品质的影响[J].食品科技, 2019, 44(4):23-29.
XIANG F S, WANG Y R, GE D Y, et al.Isolation and identification of lactic acid bacteria from Zhaguangjiao collected from Yiling of Hubei Province and affection of isolates on product quality[J].Food Science and Technology, 2019, 44(4):23-29.
[12] 张逸舒, 王玉荣, 陈芸曼, 等.保康鲊广椒中乳酸菌的分离鉴定及其发酵特性的评价[J].食品研究与开发, 2019, 40(16):159-165.
ZHANG Y S, WANG Y R, CHEN Y M, et al.Isolation and identification of lactic acid bacteria from Zhaguangjiao in Baokang and evolution of fermentation properties of these isolates[J].Food Research and Develop, 2019, 40(16):159-165.
[13] 李娜, 王玉荣, 葛东颖, 等.当阳地区鲊广椒中乳酸菌的分离鉴定及其应用[J].中国酿造, 2019, 38(2):37-41.
LI N, WANG Y R, GE D Y, et al.Isolation, identification and application of lactic acid bacteria from Zhaguangjiao in Dangyang[J].China Brewing, 2019, 38(2):37-41.
[14] 王玉荣, 杨成聪, 葛东颖, 等.扩增区域对鲊广椒细菌MiSeq测序的影响[J].食品科学, 2019, 40(10):134-140.
WANG Y R, YANG C C, GE D Y, et al.Influence of different amplified regions on results of bacterial diversity in Zhaguangjiao, a Chinese traditional fermented chili product, by MiSeq Sequencing[J].Food Science, 2019, 40(10):134-140.
[15] 王玉荣, 沈馨, 董蕴, 等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械, 2018, 34(4):25-30.
WANG Y R, SHEN X, DONG Y, et al.Characterization of bacterial microflora and their functions on flavor quality[J].Food and Machinery, 2018, 34(4):25-30.
[16] 郭壮, 葛东颖, 尚雪娇, 等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技, 2018, 39(22):93-98;106.
GUO Z, GE D Y, SHANG X J, et al.Comparative of analysis on the diversity of bacterial microflora in degenerated and normal pit mud[J].Science and Technology of Food industry, 2018, 39(22):93-98;106.
[17] 王笋, 吕嘉枥, 辛博, 等.西北地区泡菜中乳酸杆菌的生物学特性[J].中国调味品, 2014, 39(3):15-18.
WANG S, LYU J L, XIN B, et al.Biological characteristics of lactic acid bacteria from pickles in northwest of China[J].China Condiment, 2018, 39(22):93-98.
[18] 郭壮, 蔡宏宇, 杨成聪, 等.六名襄阳地区青年志愿者肠道菌群多样性的研究[J].中国微生态学杂志, 2017, 29(9):998-1 004.
GUO Z, CAI H Y, YANG C C, et al.The diversity of gut microflora in six young people in Xiangyang aera[J].Chinese Journal of Microecology, 2017, 29(9):998-1 004.
[19] 何亮, 熊礼宽, 曾忠铭.乳杆菌的分类与分子鉴定方法研究进展[J].中国微生态学杂志, 2007,19(3):312-313;316.
HE L, XIONG L K, ZENG Z M.Research progress on classification and molecular identification methods of Lactobacillus[J].Chinese Journal of Microecology, 2007,19(3):312-313;316.
[20] 王玉荣, 孙永坤, 代凯文, 等.基于单分子实时测序技术的3个当阳广椒样品细菌多样性研究[J].食品工业科技, 2018, 39(2):108-112;118.
WANG Y R, SUN Y K, DAI K W, et al.Study on the diversity of bacteria microflora in 3 Zhaguangjiao samples of Dangyang aera by single molecule real-time sequencing technique[J].Science and Technology of Food Industry, 2018, 39(2):108-112;118.
[21] 雷炎, 马佳佳, 雷敏, 等.恩施鲊广椒乳酸菌的分离鉴定及其对挥发性风味物质的影响[J].中国酿造, 2019, 38(7):126-130.
LEI Y, MA J J, LEI M, et al.Isolation and identification of lactic acid bacteria from Zhaguangjiao of Enshi and its effect on volatile flavor substances[J].China Brewing, 2019, 38(7):126-130.
[22] 葛东颖, 王玉荣, 向凡舒, 等.荆州地区鲊广椒乳酸菌多样性解析及其分离株发酵特性的评价[J].中国食品添加剂, 2019, 30(4):72-79.
GE D Y, WANG Y R, XIANG F S, et al.Analysis of lactic acid bacterial diversity in Zhaguangjiao from Jingzhou and evaluation of fermentation characteristics of isolated strains[J].China Food Additives, 2019, 30(4):72-79.
[23] SMALL M D, BENDER G, VELDHUIZEN G M, et al.The role of the human orbitofrontal cortex in taste and flavor processing[J].Annals of the New York Academy of Sciences, 2007, 1 121(1):136-151.
[24] 辛星, 宋刚, 周晓杭, 等.传统发酵豆酱中乳酸菌的分离、筛选及鉴定[J].中国食品学报, 2014, 14(9):202-207.
XIN X, SONG G, ZHOU X H, et al.Isolation, screen and identification of lactic acid bacteria from the traditional fermented soybean paste[J].Journal of Chinese Institute of Food Science and Technology, 2014, 14(9):202-207.
[25] 邓风, 张一涵, 罗芳会, 等.咸丰鲊广椒中乳酸菌的分离与鉴定及其泡菜发酵特性评价[J].食品研究与开发, 2019, 40(16):172-177.
DENG F, ZHANG Y H, LUO F H, et al.Isolation and identification of lactic acid bacteria from Xianfeng Zha-Chili and evaluation of their fermenting property[J].Food Research and Development, 2019, 40(16):172-177.