碳水化合物活性酶(carbohydrate active enzymes,CAZymes)参与了复杂碳水化合物和糖复合物的组装和分解,按功能可分为糖苷水解酶(glycoside hydrolases,GHs)、糖基转移酶(glycosyl transferases,GTs)、多糖裂合酶(polysaccharidelyases ,PLs)、碳水化合物酯酶(carbohydrate esterases,CEs)、碳水化合物结合模块(carbohydrate-binding modules,CBMs)和具有辅助活性的酶(auxiliary activities,AAs)等六大类[1-3]。大部分的CAZymes来源于微生物和无脊椎动物,真菌是植物生物质的主要降解微生物,子囊菌和担子菌来源的CAZymes已经广泛应用于工业加工领域[3]。随着高通量测序技术的广泛应用,特别是组学数据的大量产生,系统地揭示微生物的生理代谢途径成为可能,这也为筛选CAZymes基因提供了一个新的思路[4]。
除了霉菌可以产生CAZymes降解植物细胞壁多糖,细菌也可以产生纤维素酶、半纤维素酶和果胶酶来降解植物多糖。近年来,乳杆菌中CAZymes基因发掘成为其功能研究的切入点。研究表明,酸菜中的Paenibacillus peoriae, Bacillus stratosphericus, B.toyonensi和B.cereus能够产生纤维素酶、果胶酶,这是植物组织软化的重要因素[5]。发酵食品中常见的乳酸菌具有胞外蛋白水解酶、β-葡萄糖苷酶、β-甘露糖苷酶和β-甘露聚糖酶活性,筛选自马来西亚发酵食品的Lactobacillus plantarum具有转化棕榈仁饼中生物质的能力[6]。YE等[7]通过全基因组测序表明婴儿粪便中L.pentosus ZFM94含碳水化合物代谢、转运和CAZymes的基因,而CAZymes基因中含有10个GHs和29个GTs,这些基因与其益生功能密切相关。陈臣[8]通过对L.plantarum ST-III全基因组序列分析发现其含有多个潜在的代谢低聚果糖的相关碳水化合物活性酶基因。由此可见,无论从表型还是全基因组水平来看,乳杆菌中存在的CAZymes基因对于其益生功能、生物质转化以及果蔬组织的软化起着关键的作用。
相对于丝状真菌,乳杆菌中降解植物多糖的酶活力较小,对其代谢和产酶过程相关基因的研究还不够深入;虽然乳杆菌的全基因组测序表明存在CAZymes和表型特征的基因,但这些特异基因仍然需要进一步的确认[9]。所以,本研究以酸菜中的乳杆菌为研究对象,利用基因组学对从中筛选出的菌种开展CAZymes相关基因的比较研究,这将有助于我们更好的认识酸菜中乳杆菌降解和软化植物细胞壁的作用机理,对功能性乳杆菌的开发应用提供理论参考。
1 材料与方法
1.1 实验材料
1.1.1 实验菌种及培养基
MRS液体培养基:蛋白胨10 g,牛肉膏10 g,酵母浸膏5 g,K2HPO4 2 g,柠檬酸三铵2 g,CH3COONa 5 g,吐温-80 1 mL,MgSO4 0.58 g,MnSO4 0.25 g,葡萄糖20 g,自然pH;果胶-MRS:MRS中加入2%(质量分数)的果胶(Sigma,P9135)代替葡萄糖;MRS固态培养基:液态培养基加入1.8%(质量分数)琼脂粉。
菌种筛选:从广西酸菜中筛选出的3株具有降解果胶的乳杆菌,分别命名为DC4、GLKK1和GLKK2;筛选培养基为果胶-MRS固态培养基。
1.1.2 仪器与设备
UV760CRT型紫外分光光度计,上海傲谱分析仪器分析有限公司;LDZX-50KBS型立式压力蒸汽灭菌器,上海申安;LRH-250-Z型振荡培养箱,韶关市宏泰医疗器械有限公司;SZGMA3-30K型高速冷冻离心机,德国SZGMA公司;iMark型酶标仪,美国BIO-RAO公司。
1.2 实验方法
1.2.1 菌株活化
将冷冻保藏的乳杆菌菌种接种至MRS液态培养基上,37 ℃恒温培养2 d,活化菌株后备用。
1.2.2 乳杆菌产酶及酶活测定
乳杆菌产酶过程:将乳杆菌分别按10%的接种量接种至以果胶、羧甲基纤维素钠(carboxymethylcellulose sodium,CMC)以及木聚糖代替葡萄糖的MRS液体培养基中,37 ℃,200 r/min,摇床振荡培养5 d,期间每隔12 h取样,10 000 r/min离心10 min,得到的上清液即为样品酶液。
果胶酶、纤维素酶和半纤维素酶活力测定[10-11]:样品组与对照组分别加入0.48 mL果胶、CMC和木聚糖溶液(2 g/L),45 ℃水浴锅预热10 min后,样品组加入0.24 mL酶液,对照组加入0.24 mL经煮沸灭活的酶液,同时在45 ℃的水浴锅中反应20 min后取出,立刻加入0.40 mL的DNS试剂,沸水中显色5 min,冷却后540 nm测吸光度值。酶活单位:1 mL酶液1 min分解果胶、CMC和木聚糖生成1 μmol单糖所需的酶量定义为1个酶活单位U[12]。
1.2.3 乳杆菌的鉴定和基因组测序
将乳杆菌培养至对数生长中期(9 h),冷冻离心获得活菌体样品后进行菌株的鉴定(上海美吉);采用二代测序平台Illumina Hiseq×10对菌株进行全基因组测序,利用SOAPdenovo 2.04[13]进行二代测序数据组装。
1.2.4 功能注释
使用Glimmer 3.02[14]和GeneMarkS 4.3[15]预测编码DNA序列(coding sequences,CDSs),通过EggNOG(Evolutionary Genealogy of Genes: Non-supervised Orthologous Groups)和KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库对预测得到的编码基因进行功能注释;还通过CAZy数据库[16]对CAZymes进行了识别、分类和注释。
1.2.5 三株乳杆菌比较基因组分析
使用OrthoMCL对3株乳杆菌全基因组中编码基因进行同源基因聚类分析,根据KEGG注释结果,针对某一特定的KEGG代谢通路进行每个节点的基因数量统计以及可视化展示,展现3株乳杆菌之间的代谢通路差异。
2 结果与分析
2.1 乳杆菌产CAZy情况
由图1-a可看出,乳杆菌GLKK1和GLKK2的果胶酶酶活力的峰值均出现在72 h;而DC4的峰值最高,出现在48 h,为(0.40±0.01)U/mL。由图1-b和1-c得出,3株乳杆菌中,DC4的纤维素酶与木聚糖酶活力较高,其最高峰均处于72 h,纤维素酶和木聚糖酶活力分别为(0.04±0.01)和(0.19±0.01)U/mL;而GLKK1两种酶的活力最高值为(0.02±0.01)和(0.11±0.01)U/mL;GLKK2产两种酶最高值为(0.03±0.01)和(0.15±0.01) U/mL。由此可见,DC4产果胶酶、纤维素酶与木聚糖酶的能力均较强于另外2株乳杆菌。

a-乳杆菌产果胶酶;b-乳杆菌产纤维素酶;c-乳杆菌产木聚糖酶
图1 乳杆菌产CAZymes情况图
Fig.1 CAZymes production by Lactobacillus spp.
2.2 菌株鉴定结果
用16S rRNA测序技术分别鉴定3株乳杆菌,对得到的序列进行系统进化分析(图2),发现乳杆菌DC4的近缘物种是短乳杆菌 ATCC 14869(NR_044704.2),GLKK1与GLKK2的近缘物种是植物乳杆菌JCM 1149(NR_117813.1)。因此分别命名上述3株乳杆菌为L.brevis DC4,L.plantarum GLKK1和L.plantarum GLKK2。

图2 三株乳杆菌的系统发育树
Fig.2 Phylogenetic trees of three strains of Lactobacillus spp.
2.3 三株乳杆菌的全基因组测序结果分析
2.3.1 全基因组测序和组装结果
利用二代测序平台对3株乳杆菌进行全基因测序,测序组装结果如表1所示,DC4、GLKK1和GLKK2基因全长分别为2 640 916、3 637 522以及3 535 112 bp;总Scaffold数量分别为126、103和49个;GC含量分别为45.66%、46.25%和46.37%;编码基因分别有2 615、3 355以及3 270个;预测出总tRNA数量分别为66、67和63个;预测出总rRNA数量分别为4、7和2个。
表1 三株乳杆菌基因组测序结果概况统计表
Table 1 Summary of the results of genome sequencing of three strains of Lactobacillus spp.
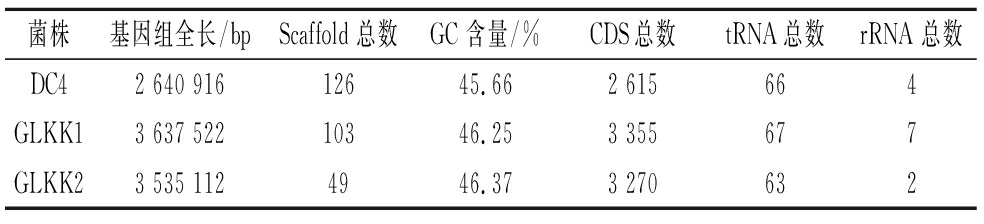
菌株基因组全长/bpScaffold 总数GC 含量/%CDS总数tRNA总数rRNA 总数DC42 640 91612645.662 615664GLKK13 637 52210346.253 355677GLKK23 535 1124946.373 270632
2.3.2 基因注释
2.3.2.1 COG注释
3株乳杆菌全基因组COG注释结果如图3所示,乳杆菌DC4、GLKK1和GLKK2基因组上的全部编码基因中,分别有2 102、2 663以及2 610个蛋白编码基因被注释到19个COG功能类别上。3株乳杆菌的注释结果非常相似,主要集中在碳水化合物转运与代谢(179、294、293个基因),转录(179、236、233个基因),氨基酸转运与代谢(141、214、211个基因),细胞膜壁的合成(109、158、159个基因)等这几个功能分类上。功能注释的多样性表明,3株乳杆菌具有较好的降解多糖的能力。

图3 三株乳杆菌的COG注释功能分类情况图
Fig.3 Classification of COG annotation function of three strains of Lactobacillus spp.
2.3.2.2 KEGG注释
图4对乳杆菌的代谢通路进行注释基因分析,乳杆菌DC4的基因主要集中在其他氨基酸代谢、碳水化合物代谢、多糖合成和代谢,而GLKK1和GLKK2主要集中在碳水化合物代谢、全局和概览通路、氨基酸代谢等。KEGG注释表明,3株乳杆菌的代谢系统都较为完整,碳水化合物代谢和氨基酸代谢通路占据了主导地位,这些重要的功能或富集通路与植物多糖代谢相关的基因表达调控等都有不可或缺的关系。
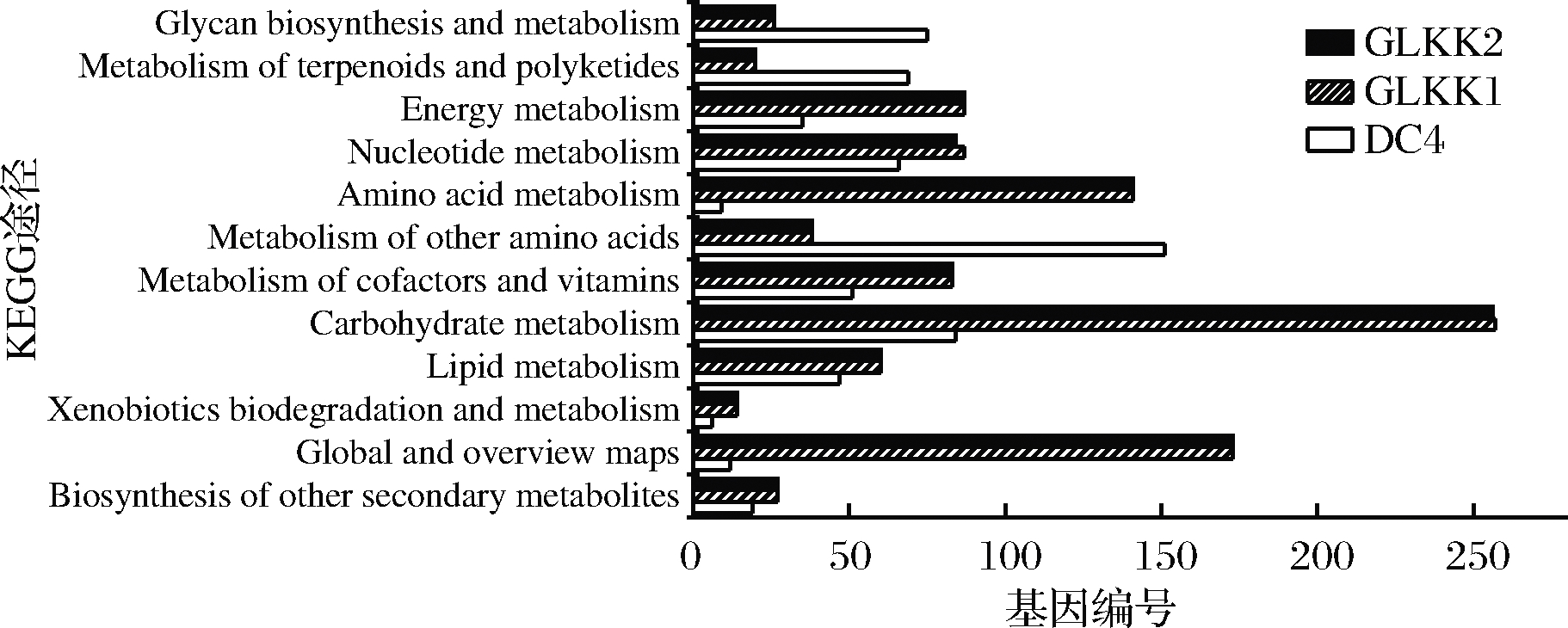
图4 三株乳杆菌KEGG注释中代谢的分类详情图
Fig.4 Classification of metabolism of three strains of Lactobacillus spp.from KEGG annotations
2.3.3 CAZymes功能预测
乳杆菌CAZymes功能预测结果如图5所示,乳杆菌DC4、GLKK1和GLKK2分别有63、114和115个CAZymes编码基因。3株菌的CAZymes注释在GHs基因数量最多;除此之外,L.brevis DC4中的CEs高于GTs,而L.plantarum GLKK1和GLKK2中GTs高于CEs。由此可见,虽然乳杆菌具有降解植物多糖的基因,但水解酶、酯酶和转移酶的比例分布与菌种的种类有直接的联系。

图5 三株乳杆菌的CAZymes功能预测
Fig.5 CAZymes functions prediction of three strains of Lactobacillus spp.
2.3.4 乳杆菌中植物多糖降解相关CAZymes基因
表2是乳杆菌被注释到的CAZymes与其基因家族分布,可见3株乳杆菌均含有较多与降解半纤维素、果胶、纤维素和淀粉等相关的基因家族。
半纤维素的降解需要解聚酶和辅助酶[17],解聚酶包括木聚糖酶、甘露聚糖酶和木葡聚糖酶等。3株乳杆菌中都含有CE1、CE3、CE4、CE7、CE9,GH1、GH2、GH31、GH36和GH42等基因家族,GH1和GH2家族基因编码的β-葡萄糖醛酸酶和α-L-阿拉伯糖呋喃糖苷酶等解聚酶可以从木聚糖上移走葡糖苷侧链[11];CE1、CE2、CE3、CE4和CE7家族基因编码的酯酶可作为半纤维素降解的辅助酶参与木聚糖的降解。果胶降解的主要水解酶属于GH28家族[18],在3株乳杆菌的全基因组中虽然没有发现该基因家族,但出现了GH1、GH2、GH42、GH51和GH78等这几个降解果胶侧链的基因家族,其基因编码的β-半乳糖苷酶(EC 3.2.1.23)水解去除非还原末端的β-D-半乳糖残基,作用于细胞壁中鼠李半乳糖醛酸聚糖 Ⅰ 果胶和半纤维素的木葡聚糖[19],α-L-鼠李糖苷酶与外切β-1,4-葡聚糖酶等负责切割果胶毛发区,从而参与到果胶降解当中。降解纤维素的酶系主要为内切葡聚糖酶、外切葡聚糖酶(纤维二糖水解酶)以及β-葡萄糖苷酶3种[20]。3株乳杆菌中发现了与降解纤维素相关的GH1、GH8、GH30以及GH51家族基因,其中GH1为3株乳杆菌所共有,而其余基因则为DC4所特有,在基因层面进一步揭示了乳杆菌DC4的纤维素降解能力较强的原因。
此外,经CAZy注释分析得出,乳杆菌DC4、GLKK1和GLKK2基因组中含有的与植物多糖降解相关基因总数分别为19、31和31个,在CAZy注释的基因中占比分别为30.16%,27.19%和26.96%。由表2可见,3株乳杆菌包含17种CAZymes共有基因,包括纤维素、半纤维素、果胶和淀粉的水解酶和酯酶。除了共有基因外,DC4菌株有10种纤维素和半纤维素的特有基因,包括GH8、GH30和GH51家族。而GLKK1和GLKK2包含7种与半纤维素、果胶和淀粉相关特有基因,包括GH39、GH13、CE2和GH78。可见,乳杆菌DC4多糖降解基因数量高于其他乳杆菌,此结果与上述乳杆菌发酵产酶情况相吻合(图1)。
表2 三株乳杆菌降解多糖的碳水化合物活性酶注释表
Table 2 CAZymes annotations for polysaccharide degradation from Lactobacillus spp.

底物CAZyEC 编号家族菌株半纤维素,果胶β-半乳糖苷酶3.2.1.23GH1,GH2,GH42DC4,GLKK1,GLKK2半纤维素β-甘露糖苷酶3.2.1.25GH1,GH2DC4,GLKK1,GLKK2半纤维素β-葡萄糖醛酸酶3.2.1.31GH1,GH2DC4,GLKK1,GLKK2GH30DC4半纤维素β-木糖苷酶3.2.1.37GH1DC4,GLKK1,GLKK2GH30,GH51DC4GH39GLKK1,GLKK2半纤维素,果胶α-L-阿拉伯糖呋喃糖苷酶3.2.1.55GH2DC4,GLKK1,GLKK2GH51DC4半纤维素甘露糖糖蛋白 内切-β-甘露糖苷酶3.2.1.152GH2DC4,GLKK1,GLKK2半纤维素内切-1,4-β-木聚糖酶3.2.1.8GH8,GH30,GH51DC4半纤维素,淀粉α-葡萄糖苷酶3.2.1.20GH31DC4,GLKK1,GLKK2GH13GLKK1,GLKK2半纤维素外切β-D-氨基葡萄糖苷酶3.2.1.165GH2DC4,GLKK1,GLKK2半纤维素α-半乳糖苷酶3.2.1.22GH31,GH36DC4,GLKK1,GLKK2半纤维素葡萄糖基神经酰胺酶3.2.1.45GH30DC4半纤维素α-D-岩藻糖苷酶3.2.1.38GH1DC4,GLKK1,GLKK2GH30DC4半纤维素α-木聚糖苷酶3.2.1.177GH31DC4,GLKK1,GLKK2半纤维素乙酰基木聚糖酯酶3.1.1.72CE1,CE3,CE4,CE7DC4,GLKK1,GLKK2CE2GLKK1,GLKK2半纤维素,果胶阿魏酸酯酶3.1.1.73CE1DC4,GLKK1,GLKK2半纤维素乙酰氨基葡萄糖6-磷酸脱乙酰化酶3.5.1.25CE9DC4,GLKK1,GLKK2果胶α-L-鼠李糖苷酶3.2.1.40GH78GLKK1,GLKK2果胶鼠李半乳糖醛酸β-L-鼠李糖水解酶3.2.1.174GH78GLKK1,GLKK2果胶外切β-1,4-葡聚糖酶3.2.1.74GH1DC4,GLKK1,GLKK2纤维素β-葡糖苷酶3.2.1.21GH1DC4,GLKK1,GLKK2GH30DC4纤维素纤维素酶3.2.1.4GH8DC4纤维素地衣激酶3.2.1.73GH8DC4纤维素内切葡聚糖酶3.2.1.4GH51DC4淀粉α-淀粉酶3.2.1.1GH13GLKK1,GLKK2GH31DC4,GLKK1,GLKK2支链淀粉支链淀粉酶3.2.1.41GH13GLKK1,GLKK2
2.4 三株乳杆菌比较基因组分析
2.4.1 同源基因分析
图6是3株乳杆菌的同源基因分析,三者共有的基因数量为1 501个,而DC4、GLKK1和GLKK2特有的基因分别为878、87和49个;GLKK1与GLKK2两者共有1 692个基因家族,而DC4与GLKK1和GLKK2之间共有基因家族分别为46和1个。以上数据说明DC4与GLKK1、GLKK2之间差异性较大,GLKK1和GLKK2的同源性较强。
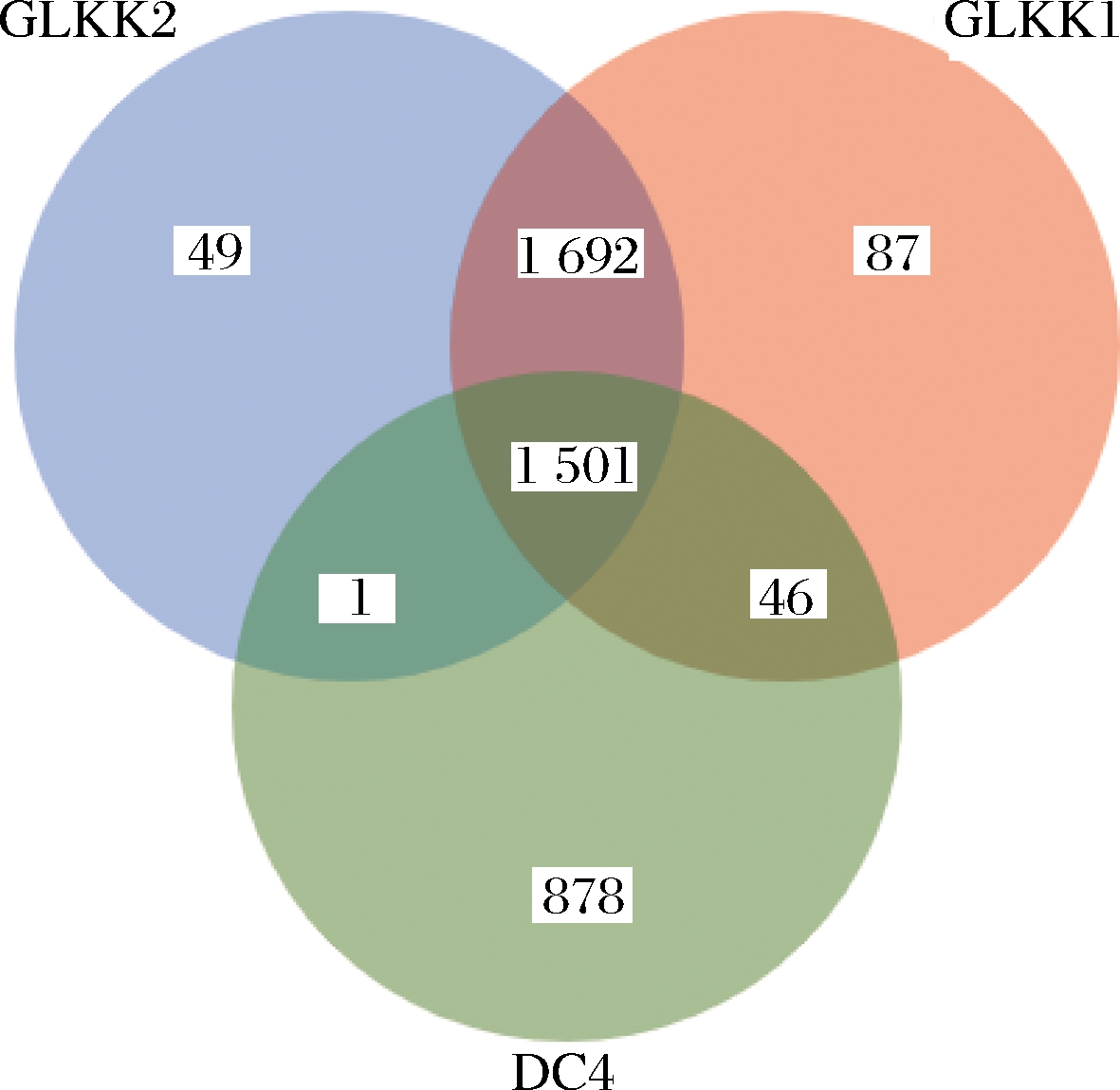
图6 三株乳杆菌的同源基因分析图
Fig.6 Homologous gene analysis of three strains of Lactobacillus spp.
2.4.2 KEGG代谢通路差异分析
3株乳杆菌的KEGG代谢通路差异分析见图7,从图7可看出3株菌株糖酵解/糖异生途径并不完整,均缺少己糖激酶[21](EC 2.7.1.1),而DC4相较于GLKK1和GLKK2还缺少了一些编码酶基因(表3),另外还发现了3株乳杆菌全基因组中都含有eda(2-酮-3-脱氧-6-磷酸葡萄糖醛酸酶)基因,而eda基因是胞内果胶代谢和ED(Entner-Doudoroff)代谢途径的两个关键酶之一[22],说明3株乳杆菌除了利用糖酵解途径代谢糖类外,还可能通过ED途径进行糖代谢产生能量。

图7 糖酵解/糖异生代谢通路差异检验图
Fig.7 Differential test of glycolysis/glycosylated metabolic pathway
注:图中标色的长方框中每一格表示对应菌株注释到该位置的基因个数,从左往右分别为GLKK2、GLKK1、DC4,方框颜色深浅代表基因的多少,颜色越深基因个数越多
表3 三株乳杆菌的糖酵解/糖异生途径差异分析表
Table 3 Analysis of the difference of glycolysis/glycopenia pathway among three Lactobacillus spp.

KO IDKO描述GLKK2GLKK1DC4K01610磷酸烯醇丙酮酸羧激酶(ATP)[EC:4.1.49]110K02777PTS系统,糖特异性II A组分[EC:2.7.1.-]220K20116PTS系统,葡萄糖特异性II A组分[EC:2.7.1.199]110K008506-磷酸果糖激酶1[EC:2.7.1.11]110K01624果糖-二磷酸醛化酶,II类[EC:4.1.2.13]110
3 结论与展望
本研究从广西酸菜中筛选获得了3株产CAZymes的乳杆菌,并通过二代测序技术比较其编码基因。通过鉴定可知3株菌分属于L.brevis DC4,L.plantarum GLKK1和GLKK2,液态发酵L.brevis DC4具有较高果胶酶、纤维素酶和半纤维素酶。通过COG和KEGG对其功能基因和代谢通路进行了注释,CAZymes注释表明其多糖降解功能基因集中在GHs、GTs和CEs。3株乳杆菌包含17种CAZymes共有基因,包括纤维素、半纤维素、果胶和淀粉水解酶和酯酶。而L.brevis DC4有10种特有基因,L. plantarum GLKK1和GLKK2包含7种特有基因。从表型和基因组数据可见上述乳杆菌具有降解植物细胞壁多糖的能力,后续的研究中将对于其产酶的基因进行克隆表达,为开发具有特异功能乳杆菌以及新型CAZymes提供参考。
[1] 王帅, 陈冠军, 张怀强, 等.碳水化合物活性酶数据库(CAZy)及其研究趋势[J].生物加工过程, 2014, 12(1):102-108.
WANG S, CHEN G J, ZHANG H Q, et al.Carbohydrate-active enzyme(CAZy) database and its new prospect[J].Chinese Journal of Bioprocess Engineering, 2014, 12(1):102-108.
[2] NGUYEN S T C, FREUND H L, KASANJIAN J, et al.Function, distribution, and annotation of characterized cellulases, xylanases, and chitinases from CAZy[J].Applied Microbiology and Biotechnology, 2018, 102(4):1 629-1 637.
[3] HUTTNER S, THANH THUY N, GRANCHI Z, et al.Combined genome and transcriptome sequencing to investigate the plant cell wall degrading enzyme system in the thermophilic fungus Malbranchea cinnamomea[J].Biotechnology Biofuels, 2017, 10(1):265.
[4] 《乳业科学与技术》丛书编委会.益生菌[M].北京:化学工业出版社, 2018.
Dairy Science and Technology Series Editorial Board.Probiotics[M].Beijing:Chemical Industry Press, 2018.
[5] ZHANG F, TANG Y, REN Y, et al.Microbial composition of spoiled industrial-scale sichuan paocai and characteristics of the microorganisms responsible for paocai spoilage[J].International Journal of Food Microbiology, 2018, 275:32-38.
[6] LEE F H, WAN S Y, FOO H L, et al.Comparative study of extracellular proteolytic, cellulolytic, and hemicellulolytic enzyme activities and biotransformation of palm kernel cake biomass by lactic acid bacteria isolated from malaysian foods[J].International Journal of Molecular Sciences, 2019, 20(20):4 979.
[7] YE K, LI P, GU Q.Complete genome sequence analysis of a strain Lactobacillus pentosus ZFM94 and its probiotic characteristics[J].Genomics, 2020, 112(5):3 142-3 149.
[8] 陈臣. 植物乳杆菌ST-Ⅲ全基因组序列分析及其对低聚果糖代谢通路的解析[D].无锡:江南大学, 2014.
CHEN C.Characterization of the complete genome sequence of Lactobacillus plantarum ST III and its pathways for fructooligosaccharides metabolism[D].Wuxi: Jiangnan University, 2014.
[9] BURON-MOLES G, CHAILYAN A, DOLEJS I, et al.Uncovering carbohydrate metabolism through a genotype-phenotype association study of 56 lactic acid bacteria genomes[J].Applied Microbiology and Biotechnology, 2019, 103(7):3 135-3 152.
[10] 张飞, 岳田利, 费坚, 等.果胶酶活力的测定方法研究[J].西北农业学报, 2004(4):134-137.
ZHANG F, YUE T L, FEI J, et al.Research on measuring method of PG activity[J].Acta Agriculturae Boreali-occidentalis Sinica, 2004(4):134-137.
[11] 马江山. 解析内生细菌Pantoea ananatis Sd-1的木质纤维素降解体系研究[D].长沙:湖南大学, 2016.
MA J S.Analysis of the lignocellulose degradation system in endophytic bacterium Pantoea ananatis Sd-1[D].Changsha:Hunan University.2016.
[12] 何海燕, 覃拥灵, 陆世则, 等.产果胶酶棘孢曲霉的筛选鉴定及微波诱变育种[J].中国饲料, 2015(2):20-22.
HE H Y, QIN Y L, LU S Z, et al.Screening, identification and microwave mutation breeding of Aspergillus aculeatus of producing pectinase[J].China Feed, 2015(2):20-22.
[13] LUO R B, LIU B H, XIE Y L, et al.Soapdenovo2:An empirically improved memory-efficient short-read de novo assembler[J].GigaScience, 2012, 1(1):18-18.
[14] DELCHER A L, BRATKE K A, POWERS E C, et al.Identifying bacterial genes and endosymbiont DNA with glimmer[J].Bioinformatics, 2007, 23(6):673-679.
[15] BESEMER J, BORODOVSKY M.Genemark:Web software for gene finding in prokaryotes, eukaryotes and viruses[J].Nucleic Acids Research, 2005, 33(Web Server issue):451-454.
[16] LOMBARD V, GOLACONDA R H, DRULA E, et al.The carbohydrate-active enzymes database (cazy) in 2013[J].Nucleic Acids Research, 2014, 42(Database issue):490-495.
[17] S NCHEZ C.Lignocellulosic residues:Biodegradation and bioconversion by fungi[J].Biotechnology Advances, 2009, 27(2):185-194.
NCHEZ C.Lignocellulosic residues:Biodegradation and bioconversion by fungi[J].Biotechnology Advances, 2009, 27(2):185-194.
[18] TANIZAWA Y, TOHNO M, KAMINUMA E, et al.Complete genome sequence and analysis of Lactobacillus hokkaidonensis LOOC260(T), a psychrotrophic lactic acid bacterium isolated from silage[J].Bmc Genomics, 2015, 16:240.
[19] 陈凯莉, 许轲, 张贤聪, 等.果实中果胶代谢相关酶基因的研究进展[J].园艺学报, 2017, 44(10):2 008-2 014.
CHEN K L, XU K, ZHANG X C, et al.Advances in genes information involved in pectin metabolism in fruit[J].Acta Horticulturae Sinica, 2017, 44(10):2 008-2 014.
[20] SINGH A, ADSUL M, VAISHNAV N, et al.Improved cellulase production by Penicillium janthinellum mutant[J].Indian Journal of Experimental Biology, 2017, 55(7):436-440.
[21] 安晓娜, 李伟程, 于洁, 等.比较基因组学分析不同来源罗伊氏乳杆菌基因多样性及生境适应性[J].微生物学报, 2020, 60(5):875-886.
AN X N, LI W C, YU J, et al.Comparative genomics analysis of genetic diversity and habitat adaptability of Lactobacillus reuteri from different sources[J].Acta Microbiologica Sinica, 2020, 60(5):875-886.
[22] 张鹏飞. 胡萝卜软腐果胶杆菌致病关键基因eda的功能研究[D].南京:南京农业大学, 2015.
ZHANG P F.Functional analysis of eda, a crucial gene to pathogenicity of Pectobacterium carotovorum subsp.Carotovorum[D].Nanjing:Nanjing Agricultural University, 2015.