硒(Se)是生物体不可或缺的微量元素,缺乏和过量均引发人体多种疾病。适量的硒摄入具有抗氧化和抗癌作用,可提高人体免疫力,对预防心血管疾病具有积极作用。硒的生物利用率随硒形态种类差别而变化。硒有多种形态,无机态有3种价态,分别为Se(-Ⅱ)、Se(+Ⅳ)和Se(+Ⅵ),硒酸盐和亚硒酸盐可以作为硒源类营养强化剂,但吸收率较低,毒性较大;单质硒包括若干个同素异形体,分别为黑色、红色、灰色和无定形单质硒,当氧化态硒被还原成单质硒,溶液中会出现红色浑浊,红色单质硒具备生物学活性,可以形成纳米硒,用于医疗和营养;有机硒则具有更多形式,包括硒代氨基酸、含硒蛋白、硒多糖、硒核酸和各种甲基化硒,由生物体经各种代谢产生,通常有机硒更易被吸收。本文对各类有机硒的合成途径及不同类别微生物富硒方式进行综述。
1 有机硒的主要形式
1.1 硒代氨基酸、硒蛋白及含硒蛋白
鉴于硒元素与硫元素(S)的化学性质相似,无机硒经生物体的硫代谢途径转变为有机硒,并行使生物学功能。富硒产物中占比较大的是硒代氨基酸,主要为硒代甲硫氨酸(SeMet)和硒代半胱氨酸(SeCys)(图1)。它们分别是甲硫氨酸(Met)和半胱氨酸(Cys)中的硫原子被替换为硒原子,替换前后氨基酸化学性质相近,因此可取代Met和Cys掺入蛋白,形成含硒蛋白。此外,硒蛋白的合成需要特殊的蛋白翻译机制,目前发现人体中共有25种硒蛋白,均利用SeCys为活性中心氨基酸。在某些植物和微生物中还会积累甲基硒代半胱氨酸(SeMeCys)[1]。
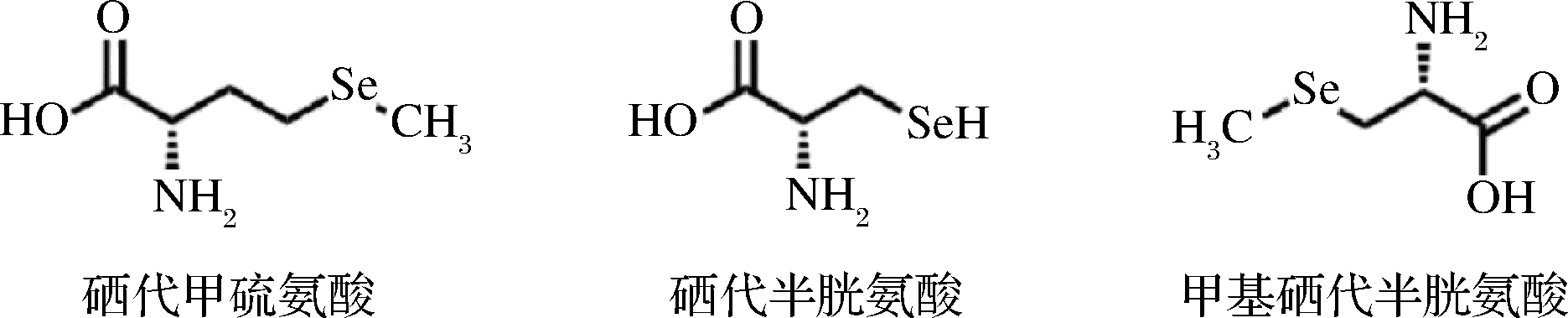
图1 硒代氨基酸的结构式
Fig.1 Structural formula of seleno-amino acids
微生物胞内积累硒始于转运。微生物吸收硒主要依赖于硫酸盐转运蛋白,例如酿酒酵母的Sul1p和Sul2p转运系统。此外,ABC泵、磷酸盐转运系统和单羧酸转运系统也可以转运硒。ABC泵利用水解ATP提供能量来转运含硒离子,大肠杆菌(Escherichia coli)中相关的编码基因为cysAWTP操纵子,转运复合体包括2个CysA结合于ATP,2个膜内蛋白(CysT和CysW)以及在周质空间的硫酸结合蛋白CysP;酿酒酵母的磷酸盐转运蛋白Pho84p和Pho89p在低磷酸盐环境中主导亚硒酸盐的结合。在高磷酸盐环境中,硒转运逐渐依赖于Pho87p、Pho90p和Pho91p;同向转运蛋白Jen1p可转运单羧酸,也能够介导亚硒酸盐的转运,转运原理基于硒酸盐负离子与羧酸负离子在结构上的相似性,并具有相似的解离常数,在生理pH下,它们都是带一个单位负电荷的离子[2]。
硒进入细胞后,硒酸盐由ATP 硫酸化酶催化转变为腺苷酰硒酸(APSe),随后在激酶的作用下形成磷酸腺苷酰硒酸(PAPSe),由PAPSe还原酶催化形成亚硒酸盐,再由亚硫酸还原酶催化亚硒酸盐,形成硒化氢(H2Se)。H2Se是微生物细胞中各种形态硒代谢合成的主要中间代谢物,最终形成各种包括硒代氨基酸在内的有机硒[3]。
H2Se与高半胱氨酸合成酶结合,并与O-琥珀酰高丝氨酸(O-Sul-HSer)反应,生成硒代高半胱氨酸(SeHCys)和琥珀酸。随后SeHCys由高半胱氨酸甲基转移酶催化产生SeMet。在有氧条件下,SeMet可以转变成硒代腺苷甲硫氨酸(SeAM),该步骤由S-腺苷甲硫氨酸合成酶催化。随后SeAM经过酶催化的甲基转移,形成硒代腺苷高半胱氨酸(SeAHCys),再经过水解重新形成硒代高半胱氨酸(SeHCys),最后在胱硫醚-β-裂解酶和胱硫醚-γ-合酶的相继作用下先转化为硒代胱硫醚(SeCysT),随后转化为SeCys[4]。SeCys也可以由半胱氨酸合酶催化,以丝氨酸(Ser)为前体进行合成。
在某些蕈菌中,还能检测到SeMeCys。其合成途径并不明确,但SeMeCys在植物中的合成在于SeCys与S-腺苷甲硫氨酸(SAM)反应,由甲基转移酶催化,转化为SeMeCys和S-腺苷高硒代半胱氨酸。
当微生物从培养基中摄取无机硒,通过上述代谢过程在胞内积累SeMet和SeCys(图2),它们可以在蛋白质合成过程中,随机替代Met和Cys参与翻译过程,形成含硒蛋白。因此相对于其他只存在游离形式的硒代氨基酸,SeMet和SeCys的胞内含量更高。
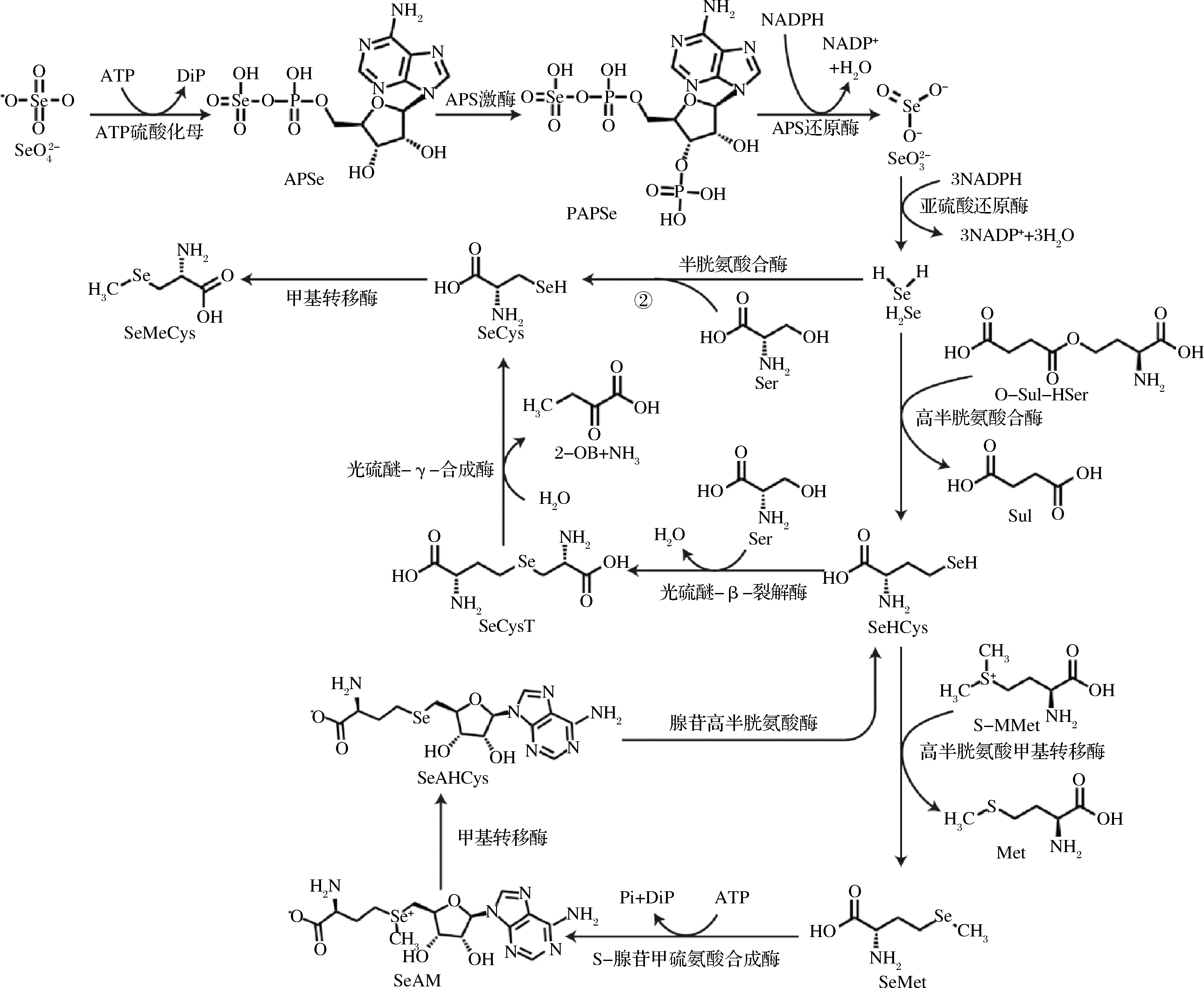
①高半胱氨酸合酶途径;②半胱氨酸合酶途径;
DiP,二磷酸盐;APSe,腺苷酰硒酸;PAPSe,磷酸腺苷酰硒酸;O-Sul-HSer,O-琥珀酰高丝氨酸;Sul,琥珀酸;SeHCys,硒代高半胱氨酸;
SeMet,硒代甲硫氨酸;S-MMet,S-甲基甲硫氨酸;SeAM;Pi,磷酸;硒代腺苷甲硫氨酸;SeAHCys,硒代腺苷高半胱氨酸;Ade,腺嘌呤;
SeCysT,硒代胱硫醚;2-OB,2-氧代丁酸钠;SeCys,硒代半胱氨酸;SeMeCys,甲基硒代半胱氨酸
图2 微生物硒代氨基酸的合成路径
Fig.2 Synthetic pathway for seleno-amino acids by microrganisms
不同生物的硒代氨基酸代谢途径存在差异,酵母的半胱氨酸合酶途径较弱,主要通过高半胱氨酸合酶途径,实现SeCys的合成和SeMet的大量积累[5]。而大肠杆菌主要通过半胱氨酸合酶途径生成SeCys,突变半胱氨酸合酶基因后,游离态Cys和SeCys的合成受阻[6]。
硒蛋白的合成则需要特殊的细胞机制,SeCys特异地掺入蛋白质,形成在所有生命体中均具有重要作用的硒蛋白。携带SeCys的tRNA为硒代半胱氨酰-tRNA(selenocysteyl-tRNASec),是由丝氨酰-tRNA(seryl-tRNASec)与硒磷酸相连接而形成。SeCys利用了终止密码子UGA作为三联体密码,当UGA下游存在SECIS元件(硒代半胱氨酸插入序列)时,该终止密码子可以被特殊的转录因子识别,从而转变为硒代半胱氨酸密码子[7]。硒蛋白都具有很重要的生理功能,所以硒是生命的必需微量元素。
1.2 纳米单质硒和硒多糖
微生物在代谢过程中产生的纳米硒可以作为高生物活性和低毒性的硒产品,具有潜在的生物医药功能,同时可以作为抗菌素,有望成为抗生素的替代产品。化学合成的红色纳米硒并不稳定,在水溶液中易于聚集并转化为其他不活泼的形式,为加强纳米硒的稳定性,多种多糖被用于修饰纳米单质硒。而微生物产生的纳米单质硒由于天然包裹了生物大分子在纳米颗粒外围,因此不易聚集、稳定性好,为此将纳米单质硒也归于有机硒进行介绍。
单质硒在细胞内的合成,主要依赖谷胱甘肽氧化还原系统。四价硒自发与还原态的谷胱甘肽(glutathione,GSH)反应,形成了GS-Se-SG和氧化形式的谷胱甘肽(GSSG)。GSSG是有害的,会氧化含巯基蛋白形成二硫化合物,因此需要迅速被谷胱甘肽还原酶还原成GSH。GS-Se-SG则被转化为GS-Se-H。GS-Se-H的去路有2条,一是转变为H2Se,同时形成GSSG;另一条去路是GS-Se-H由超氧化物歧化酶催化转变为单质硒,该通路被广泛认为是生物还原亚硒酸盐形成单质硒的主要模式[8](图3)。
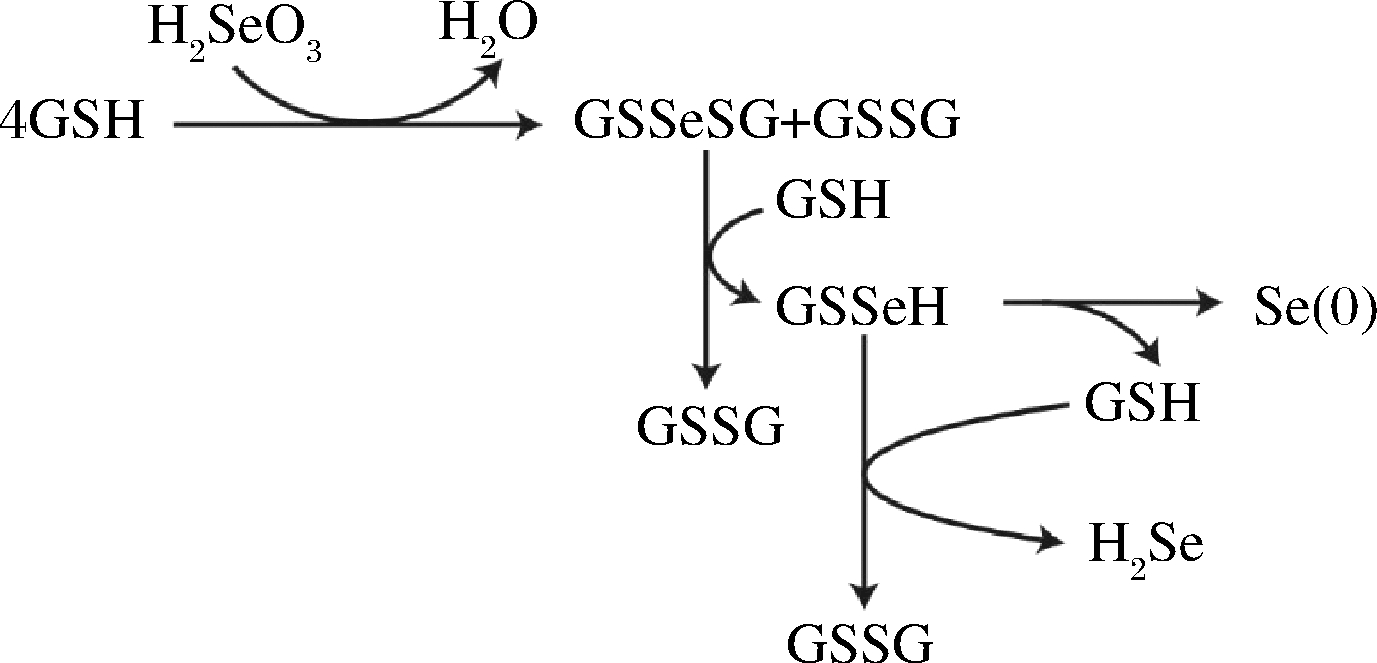
GSH-还原型谷胱甘肽;GSSG-氧化型谷胱甘肽;GSSeSG-硒化谷胱甘肽
图3 谷胱甘肽介导的单质硒合成途径
Fig.3 Glutathione mediated pathway for elemental Se synthesis
另一种单质硒合成系统包括SeCys和来自于棕色固氮菌(Azotobacter vinelandii)的NifS蛋白,该蛋白可以将H2Se转化为硒磷酸。大肠杆菌中也发现了3个NifS类蛋白(CsdB、CSD和lscS),其具有硒代半胱氨酸裂解酶的作用,促进还原SeCys合成单质硒[9]。
在革兰氏阴性菌Thauera selenatis中,无氧呼吸和亚硒酸盐的还原同时进行。由醌池提供电子,经细胞色素C传递给周质延胡索酸还原酶,最终实现亚硒酸盐被还原为单质硒[10]。
在微生物所能适应的较高硒浓度下,大部分微生物均可合成纳米单质硒,合成的纳米硒外围包裹的生物大分子,包括蛋白、多糖、核酸、脂质等。已经鉴定到的纳米硒结合蛋白包括半金属还原酶[11]、硒因子A[8,12]和硒结合蛋白等。多糖-蛋白复合物也可以包裹纳米硒颗粒,这种包裹作用是由纳米颗粒上的多糖羟基和蛋白质氨基之间的物理吸附形成的,帮助纳米颗粒通过细胞的胞吞作用增强吸收。
微生物合成的多糖结合或包裹单质硒,则形成单质硒多糖复合物(图4-a),成为硒多糖。外围结合具有生物活性的多糖形成的纳米单质硒,具备生物兼容性和生物可降解性,而多糖的种类、颗粒直径、硒含量和聚糖的生物活性都对稳定纳米硒有影响。硒多糖的另一种形式是无机硒与多糖共价结合,形成硒化多糖,如硒化卡拉胶(图4-b)。硒多糖作为新的功能性多糖,主要具有抗肿瘤、抗病毒和增强免疫力等作用。
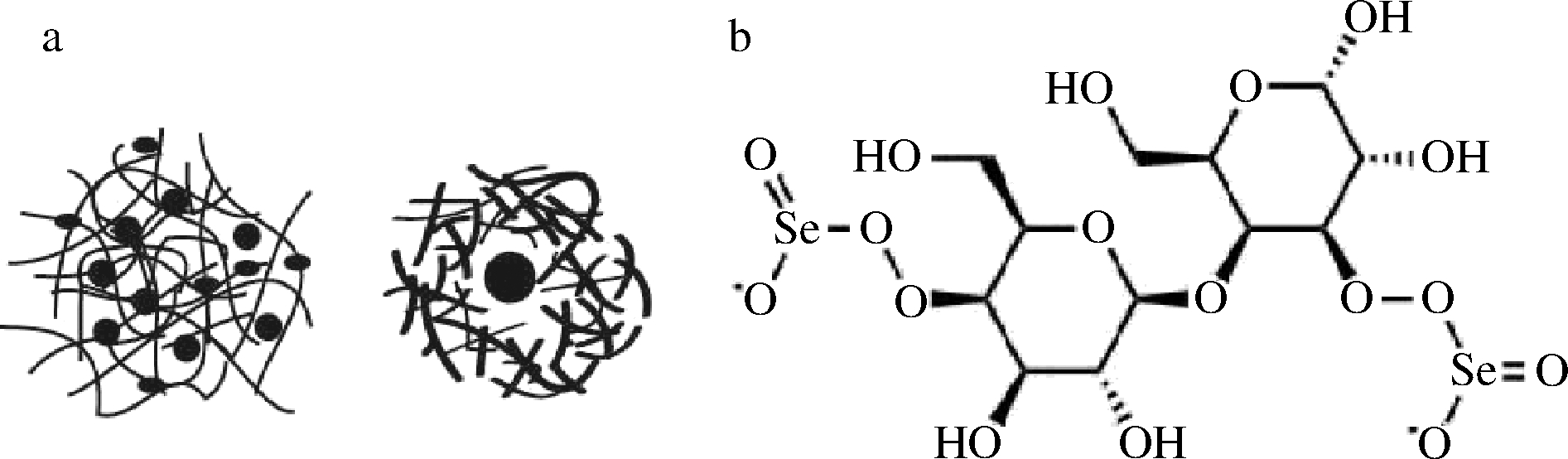
a-单质硒多糖复合物;b-硒化多糖(以硒化卡拉胶为例)
图4 硒多糖的类型
Fig.4 Types of selenium polysaccharides
2 微生物对硒的富集作用
2.1 细菌对硒的富集作用
微生物是人类食物链的重要成员,一方面,微生物在食品发酵和生物合成中作为细胞工厂;另一方面肠道微生物在消化道内定居共生从而对人体健康起重要作用。微生物富硒一直是硒生物转化关注的热点,细菌、酵母、霉菌和蕈菌都可以在无机硒的环境中生长并对其进行同化,而不同的微生物中主要的有机硒形式存在差别。细菌结构简单,具有很强的环境适应能力,各种极端环境都可寻觅到其踪影,在人体和环境的硒循环中具有非常重要的作用,可以通过代谢合成各种形态硒(表1),而其中的富硒益生菌无疑是理想的硒源补充剂。
表1 细菌富硒情况
Table 1 Selenium enrichment of bacteria

菌株硒源耐硒能力底物浓度硒去除率%总硒胞内产物及产量胞外产物及产量文献Enterococcus casseliflavus 82Na2SeO3-20 mg/L104±2-总硒2.4 mg/L总硒1.7 mg/L[13]Fructobacillus tropaeoli CRL 2034Na2SeO3-20 mg/L80±4总硒580 μg Se/g总硒2.5 mg/L SeCys 0.625 mg/L总硒1.5 mg/LLactobacillus brevis CRL 2051Na2SeO3-20 mg/L95±3总硒126 μg Se/g总硒2.2 mg/L SeCys 0.873 mg/L总硒2.2 mg/LLactobacillus plantarum CRL 2030Na2SeO3-20 mg/L98±6-总硒2.0 mg/L SeCys 0.867 mg/L总硒3.0 mg/LLactococcus lactis CRL 2011Na2SeO3-20 mg/L104±5-总硒1.9 mg/L总硒1.8 mg/LWeissella cibaria 10Na2SeO320 mg/L85±4-总硒2.1 mg/L总硒1.4 mg/LBifidobacterium longum DD98----SeMet占总硒的87%-[14]Alcaligenes faecalis Se03Na2SeO3120 mmol/L5 mmol/L100--单质硒[15]Lactobacillus parabuchneri CH135Na2SeO3-150 mg/L-总硒 40.7 mg/g--[16]Lactobacillus delbrueckii ssp.bulgaricusNa2SeO3-80 mg/L94---[17]Streptococcus thermophilusNa2SeO3-80 mg/L97---Pediococcus acidilacticiNa2SeO3-4 mg/L-总硒1.45 mg/g--[18]Lactobacillus reuteri NCDC77Na2SeO3-4.5 mg/L28.8总硒 0.820 mg/g--[19]Bifidobacterium animalis 01Na2SeO3-0.1 mg/L16.7有机硒1.017 mg/g--[20]Bacillus subtilis BSN313Na2SeO3-6 mg/L-总硒 2.123 mg/g--[21]Rhodopseudomonas palustris NNa2SeO38 mmol/L1 mmol/L99.9---[22]Bacillus pumilusNa2SeO320 mg/mL500 mg/L97---[23]Bacillus cereusNa2SeO320 mg/mL500 mg/L----Bacillus licheniformisNa2SeO320 mg/mL-----Exiguobacterium sp.Na2SeO320 mg/mL-----Streptomyces sp.ES2-5Na2SeO350 mmol/L-----[24]
注:-,未检测
2.1.1 益生菌
益生菌在发酵食品领域占据统治地位,乳酸菌和双歧杆菌是受关注最多的益生菌。此外其他革兰氏阳性菌,如粪肠球菌(Enterococcus faecalis)、嗜热链球菌(Streptococcus thermophilus)和枯草芽孢杆菌(Bacillus subtilis)等也在食品工业中有重要应用。已经报道的可以富硒的益生菌包括乳杆菌属、乳球菌属、双歧杆菌属、产碱杆菌属、肠球菌属和芽孢杆菌属等菌株。
亚硒酸盐是益生菌富硒最常用的硒源,益生菌对亚硒酸盐的耐受性在1.4~120 mmol/L之间,粪产碱菌(Alcaligenes faecalis)对硒的耐受力最高[15],此外芽孢杆菌属菌株也有较高的亚硒酸盐耐受性[23]。
富硒量受培养基初始pH、硒添加量、硒添加时间和培养温度的影响。保加利亚乳杆菌(Lactobacillus delbrueckii ssp.bulgaricus)和嗜热链球菌的培养温度分别为33和40 ℃,培养基初始pH接近6,硒添加量约6%时,硒转化率可达94%以上[17]。枯草芽孢杆菌最佳富硒条件则为培养温度37 ℃,培养基pH 7.0,硒添加量为6 mg/L,硒添加时间为接种后3 h[21]。其中,硒的最适添加量差异较大,在1~500 mg/L之间,硒的添加时间会影响总硒的生成,在菌体刚进入对数生长期添加硒可以获得最大富硒量。较高的溶氧也利于提高谷胱甘肽还原酶活性,胞内还原能力的增加利于单质硒的形成,从而增加富硒量。
副干酪乳杆菌(Lactobacillus paracasei)ML13和CH135的富硒能力最高,分别达到38.1和40.7 mg/g[16]。而乳酸片球菌(Pediococcus acidilactici)的富硒能力较低,当硒添加量为4 mg/L时,富硒量仅为1.45 mg/g[18]。
乳酸链球菌(Lactococcus)、魏斯氏菌(Weissella)、明串珠菌(Leuconostoc)、乳杆菌(Lactobacillus)、肠球菌(Enterococcus)和果聚糖杆菌(Fructobacillus)等菌株富硒时,胞内SeCys含量在0.015~0.88 mg/L之间[13]。双歧杆菌从环境中吸收无机硒,胞内SeMet是最主要的硒化物形式,含量占总硒的87%[14],大部分有机硒(50.7%~63.0%)存在于蛋白中,9.62%~18.7%有机硒与多糖结合,0.273%~0.754%有机硒与核酸结合,还有20.8%~30.9%的有机硒以其他形式存在[20]。不同的硒源对代谢产物有重要的影响,唾液链球菌(Streptococcus salivarius)K12、鼠李糖乳杆菌(Lactobacillus rhamnosus)67B、嗜酸乳杆菌(Lacobacillus acidophilus)L10和乳双歧杆菌(Bifidobaterium lactis)LAFTI B94在SeMet充沛的环境中可以形成二甲基二硒和二甲基硒,而在亚硒酸盐环境中主要形成二甲基硒、二甲基二硒和单质硒[25]。
富硒菌株在透射电镜下多能观察到单质纳米硒。干酪乳杆菌(Lactobacillus casei)ATCC 393胞内的纳米硒直径在50~80 nm之间,结合在蛋白和多糖上。粪产碱菌(Alcaligenes faecalis)菌株Se03形成的纳米单质硒结合在胞外,直径为(273.8±16.9) nm,胞内亚硫酸还原酶和硫氧还蛋白可能与亚硒酸盐的还原及硒单质的形成有关[15]。在含硒的条件下,谷胱甘肽还原酶活性普遍升高。富硒后,菌株对胆酸盐的抵抗能力和耐酸能力下降[26]。
2.1.2 其他细菌
大肠杆菌ATCC 35218的纳米硒直径约100~183 nm,颗粒上结合蛋白质[27]。在大肠杆菌中表达亚硒酸还原酶CsrF有助于纳米单质硒的形成,其直径介于60~105 nm,成分包括硒、蛋白质和脂质。阴沟肠杆菌(Enterobacter cloacae)可以还原结合在细胞壁上的硒酸盐,形成单质硒。硒酸盐还原酶结合在细胞膜上,可以对硒进行还原。沼泽红假单孢菌(Rhodopseudomonas palustris)对亚硒酸钠的耐受力为8 mmol/L,无机硒浓度在1~8 mmol/L呈梯度增加时,硒转化率由99.9%下降为2.4%,主要产物为单质硒,颗粒直径约80~200 nm[22]。金黄色葡萄球菌(Staphylococcus aureus)和铜绿假单胞菌(Pseudomonas aeruginosa)可以合成直径90~150 nm的纳米单质硒[28]。
2.2 酵母对硒的富集作用
酵母具有很强的富硒能力,是目前硒源补充剂的主力,其富硒机制和富硒方式均有较多研究。培养条件对酿酒酵母(Saccharomyces cerevisiae)富硒有较大影响,pH和溶氧水平是最重要的影响因素。当培养基pH降至3.5,产朊假丝酵母(Candida utilis)CCTCC M 209298胞内NADH/NAD+和ATP/ADP的比例提高,并且GSH含量提升,促进胞内有机硒含量的增加[4]。此外硒的吸收率和无机硒的种类与浓度有关,有机硒比无机硒更易于吸收,且毒性更低。高浓度硫酸盐、磷酸盐和重金属会使酵母对硒的亲和力降低,从而减弱硒的吸收,硒和硫的摄入比例对胞内硒代谢谱产生较大影响。酿酒酵母富集SeMet的最佳培养条件为27.4 ℃,pH 5.8,在对数生长期前期硒添加量为50 mg/L时,酵母富硒量可达2 354 μg/g[29]。葡萄汁酵母(Saccharomyces uvarum)在添加30~180 mg/L亚硒酸钠的废糖蜜培养基中,可达到与酿酒酵母相当的富硒量[30]。产朊假丝酵母富硒量为4 019 μg/g,水溶性硒为3 306 μg/g,代谢产生的主要的硒化合物为硒代高羊毛氨酸,占总硒的82.3%,而SeMet仅占总硒的11.6% [31]。在酿酒酵母BY106中,检测到的有机硒主要是SeMet,此外还能检测到SeMeCys和2种含硒半乳糖衍生物[32]。胶红酵母(Rhodotorula mucilaginosa)13B可以在菌体内还原硒,细胞内可见单质硒沉淀,该过程可用于治理硒污染的水源,对硒酸盐进行生物修复[33]。
2.3 霉菌对硒的富集作用
霉菌在食品、医药和化工领域是非常重要的工业微生物,其酶系发达且分泌能力强,是多种工业酶制剂的来源,且部分食品安全级菌株可用于食品发酵;霉菌易于在贫瘠的环境中生长,具有底物广谱性,可以有效代谢环境中各种营养成分,减少培养成本,是理想的工业微生物,作为好氧微生物,其代谢过程对硒污染的环境修复也非常重要。霉菌富硒产物主要为纳米单质硒。
黄曲霉(Aspergillus terreus)对硒的吸收率为87%[34]。棘壳孢菌(Pyrenochaeta sp.)、枝顶孢霉(Acremonium strictum)、黄瓜织球壳菌(Plectosphaerella cucumerina)、Stagonospora sp.、链格孢(Alternaria alternate)和Paraconiothyrium sporulosum对0.01 mmol/L 四价硒的吸收率为85%~93%,但对0.01 mmol/L六价硒的吸收率仅为20%~30%,产生的纳米单质硒的直径约50~300 nm[35]。黄孢原毛平革菌(Phanerochaete chrysosporium)则可以同时吸收碲和硒这2种元素,当2种元素的浓度均为10 mg/L,可形成50~600 nm的硒碲颗粒[36]。
不同的霉菌对富硒培养基种类有不同的要求。镰刀霉(Fusarium)和里斯木霉(Trichoderma reesi)在查氏培养基中可以将亚硒酸盐还原成单质硒。黑曲霉(Aspergillus niger)、毛霉(Mucor)SK和少根根霉(Rhizopus arrhizus)只在麦芽提取物培养基中还原亚硒酸盐形成单质硒[37]。米曲霉(Aspergillus orayzae)可以在廉价的羽扇豆提取物培养基中生长并对硒进行转化,合成纳米单质硒,直径约55 nm [38]。
2.4 蕈菌对硒的富集作用
伞菌纲(Agaricomycetes)生物是研究最多的高等丝状真菌,对其富硒方式也进行了大量的研究。因为很多蕈菌多糖结构复杂且具有药用价值,故硒多糖的研究较多。
对市售或野生蕈菌样本的富硒情况进行检测,含硒量最低的是花盖红菇(Russula cyanoxantha)、硬柄小皮伞(Marasmius oreades)和双孢蘑菇(Agaricus bisporus),仅约1 μg/g;含硒量最高的夏牛肝菌(Boletus aestivalis)含硒量48.5 μg/g;而褐红牛肝菌(Boletus pinophilus)、美味牛肝菌(Boletus edulis)和黑牛肝菌(Boletus aereus)含硒量在10~20 μg/g之间[39]。
平菇(Pleurotus ostreatus)、香菇(Lentinus edodes)、灰树花(Grifola frondosa)、灵芝(Ganoderma lucidum)、双孢蘑菇和草原黑蘑(Agaricus arvensis)可以合成纳米单质硒,物种和年龄、培养基种类和成分、培养时间等条件影响纳米单质硒的形成,直径在20~550 nm,在培养基和菌丝体中均存在[40]。在另一项研究中,亚硒酸钠浓度高于0.96 mmol/L可抑制香菇的生长,在含0.64 mmol/L亚硒酸钠和桉树锯末的培养基中,香菇富硒量为0.17 mg/g[41]。平菇在0.5 mmol/L亚硒酸盐的培养基中,子实体可富硒62 μg/g[42]。杏鲍菇(Pleurotus eryngii)的富硒能力稍弱为1.4 μg/g,在体内的硒吸收率为15.8%;平菇的富硒量为20.3 μg/g,硒转化率62.5%,在体内的硒吸收率为36.0%;用作祛风散寒中药成分的桦革裥菌(Lenzites betulina)的硒转化率最低,为8.1%[43]。
香菇的68%富硒产物为水溶性硒,其中,49.8%的水溶性硒结合于高分子上(>40 000 kDa),最主要的含硒代氨基酸是SeMet[44]。双孢蘑菇富硒率可达51.8 μg/g,86%为有机硒[45]。对双孢蘑菇和香菇蛋白中的硒形态进行了检测,水解后的水溶性含硒蛋白利用ICP-MS检测到SeCys、SeMet、SeMeCys和无机硒[46],SeCys和MeSeCys比SeMet更易被消化,消化释放率分别为90%、76%和51%[47]。猴头菇(Hericium erinaceus)中最主要的硒代谢物也是SeMet,占总硒含量63.9%,除了常见的硒代氨基酸,还检测到若干硒代腺苷酸衍生物,其中包括甲基-5硒代腺苷、甲基-5硒代腺苷硒氧化物和二甲基-5-硒代腺苷[48]。对灵芝的富硒产物进行分析,含硒蛋白中的硒约占总硒的56%~61%,硒多糖占11%~18%。此外,水溶和碱溶蛋白是储存有机硒的主要形式,游离的SeMet仅占蛋白硒的8.2%~18.3%。硒含量不超过100 μg/g时,有利于菌体合成蛋白和氨基酸。
利用农业加工副产物或废弃物进行微生物富硒可以降低成本,缓解环境污染。印度丽蘑(Calocybe indica)在富硒的麦秸上培养可以积累硒,随着麦秸上亚硒酸钠含量从2.5 mg/L提高到40 mg/L,子实体的富硒量也直线提升;在亚硒酸盐质量浓度超过5 mg/L时,生物量受到抑制。蛋白中的有机硒占总硒的56%~68%,多糖结合硒占22%~29%,核酸结合硒占1.4%~2.7%[49]。佛州侧耳(Pleurotus florida)在采自富硒地区的麦秸上生长,富硒量为141 μg/g,模拟肠道消化实验显示,其中的75%可被人体吸收,含硒生物大分子会被分解为小分子量的硒化合物。咖啡壳也被用来培养蕈菌,平菇在添加了51 μg/g亚硒酸钠的培养基中生长,硒吸收率最高[50]。
3 总结与展望
本文综述了微生物中主要积累的有机硒的种类和合成机制,以及各类微生物对硒的富集能力及其控制条件。微生物均主要通过高半胱氨酸合酶途径和半胱氨酸合酶途径实现无机硒到有机硒的转化,富硒产物中,游离的硒代氨基酸含量较低,更多以SeCys和SeMet的形式掺入蛋白。硒蛋白为微生物实现生理功能所必需,含硒蛋白则富集了更多的硒代氨基酸,然而基于硒代氨基酸掺入正常蛋白可能造成蛋白结构改变,从而引起蛋白失活,因此表现出硒摄入过量对微生物生长的抑制作用。纳米硒是目前研究的热点,纳米硒是易于吸收且低毒性的硒产品,利用微生物合成的纳米硒,由于通常与生物大分子结合,故不易聚集,稳定性更好。
不同微生物由于代谢途径的差别,富硒能力和有机硒类别存在差异,且由于自身活性物质的差别,具有不同的功效,可以应用于不同领域。酵母普遍具有强大的富硒能力,最高富硒量可达4 mg/g。益生菌的富硒能力参差不齐,副干酪乳杆菌的富硒能力最高可达40 mg/g,是微生物中目前报道的最高富硒量。酵母和益生菌中含量最高的有机硒形式是SeMet,易于消化吸收,通常可用作硒源类营养补充剂。霉菌和蕈菌的富硒研究则更多在纳米硒层面,霉菌的环境适应性强,易于在各种贫瘠和毒性环境中生长,可用于环境修复。蕈菌是可食用的大型丝状真菌,其富硒能力最高仅约100 μg/g,但其多糖通常具有药用价值,故纳米硒多糖的合成和应用有较广泛前景,其结构解析、合成机制和生理功能均需要更明确的阐述。生物体富硒机制的解析及其控制和应用还有很多问题需要解析,比如半胱氨酸合酶受负反馈调控,亟需解析代谢通量对富硒产物的影响。富硒益生菌调节宿主肠道微生物的规律和硒对共生菌群的维持促进作用也值得进一步研究。此外,蕈菌富硒形成的硒单质在菌丝体内部和培养基中均存在,是否存在胞内外转运及相关机制也需要进一步明确。微生物种类繁多,在充分了解菌体生长和富硒方式的情况下,综合利用群落结构实现硒及重金属污染土壤的修复将具有重要的现实意义。
[1] SCHIAVON M,PILON-SMITS E A H.The fascinating facets of plant selenium accumulation-biochemistry,physiology,evolution and ecology[J].New Phytologist,2017,213(4):1 582-1 596.
[2] MCDERMOTT J R,ROSEN B P,LIU Z J.Jen1p:A high affinity selenite transporter in yeast[J].Molecular Biology of the Cell,2010,21(22):3 934-3 941.
[3] ARNAUDGUILHEM C,BIERLA K,OUERDANE L,et al.Selenium metabolomics in yeast using complementary reversed-phase/hydrophilic ion interaction (HILIC) liquid chromatography-electrospray hybrid quadrupole trap/Orbitrap mass spectrometry[J].Analytica Chimica Acta,2012,757:26-38.
[4] ZHANG G C,WANG D H,WANG D H,et al.The mechanism of improved intracellular organic selenium and glutathione contents in selenium-enriched Candida utilis by acid stress[J].Applied Microbiology and Biotechnology,2017,101(5):2 131-2 141.
[5] MAPELLI V,HILLESTROM P R,KAPOLNA E,et al.Metabolic and bioprocess engineering for production of selenized yeast with increased content of seleno-methylselenocysteine[J].Metabolic Engineering,2011,13(3):282-293.
[6] LACOURCIERE G M,LEVINE R L,STADTMAN T C.Direct detection of potential selenium delivery proteins by using an Escherichia coli strain unable to incorporate selenium from selenite into proteins[J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(14):9 150-9 153.
[7] REICH H J,HONDAL R J.Why nature chose selenium[J].Acs Chemical Biology,2016,11(4):821-841.
[8] DEBIEUX C M,DRIDGE E J,MUELLER C M,et al.A bacterial process for selenium nanosphere assembly[J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(33):13 480-13 485.
[9] LACOURCIERE G M,MIHARA H,KURIHARA T,et al.Escherichia coli NifS-like proteins provide selenium in the pathway for the biosynthesis of selenophosphate[J].Journal of Biological Chemistry,2000,275(31):23 769-23 773.
[10] NANCHARAIAH Y V,LENS P N L.Selenium biomineralization for biotechnological applications[J].Trends in Biotechnology,2015,33(6):323-330.
[11] LENZ M,KOLVENBACH B,GYGAX B,et al.Shedding light on selenium biomineralization:Proteins associated with bionanominerals[J].Applied and Environmental Microbiology,2011,77(13):4 676-4 680.
[12] WADHWANI S A,SHEDBALKAR U U,SINGH R,et al.Biogenic selenium nanoparticles:Current status and future prospects[J].Applied Microbiology and Biotechnology,2016,100(6):2 555-2 566.
[13] MARTINEZ F G,MORENO-MARTIN G,PESCUMA M,et al.Biotransformation of selenium by lactic acid bacteria:Formation of seleno-nanoparticles and seleno-amino acids[J].Frontiers in Bioengineering and Biotechnology,2020,8:506.
[14] ZHU H,ZHOU Y,QI Y,et al.Preparation and characterization of selenium enriched-Bifidobacterium longum DD98,and its repairing effects on antibiotic-induced intestinal dysbacteriosis in mice[J].Food and Function,2019,10(8):4 975-4 984.
[15] WANG Y T,SHU X,ZHOU Q,et al.Selenite reduction and the biogenesis of selenium nanoparticles by Alcaligenes faecalis Se03 Isolated from the gut of Monochamus alternatus (Coleoptera:Cerambycidae)[J].International Journal of Molecular Sciences,2018,19(9).DOI:10.3390/ijms19092799.
[16] MORSCHBACHER A P,DULLIUS A,DULLIUS C H,et al.Assessment of selenium bioaccumulation in lactic acid bacteria[J].Journal of Dairy Science,2018,101(12):10 626-10 635.
[17] YANG J P,LI Y,ZHANG L,et al.Response surface design for accumulation of selenium by different lactic acid bacteria[J].3 Biotech,2017,7:52.
[18] KOUSHA M,YEGANEH S,AMIRKOLAIE A K.Effect of sodium selenite on the bacteria growth,selenium accumulation,and selenium biotransformation in Pediococcus acidilactici[J].Food Science and Biotechnology,2017,26(4):1 013-1 018.
[19] SAINI K,TOMAR S K,SANGWAN V,et al.Evaluation of Lactobacilli from human sources for uptake and accumulation of selenium[J].Biological Trace Element Research,2014,160(3):433-436.
[20] ZHANG B W,ZHOU K,ZHANG J L,et al.Accumulation and species distribution of selenium in Se-enriched bacterial cells of the Bifidobacterium animalis 01[J].Food Chemistry,2009,115(2):727-734.
[21] ULLAH A,SUN B,WANG F H,et al.Isolation of selenium-resistant bacteria and advancement under enrichment conditions for selected probiotic Bacillus subtilis (BSN313)[J].Journal of Food Biochemistry,2020,44(6).DOI:10.1111/jfbc.13227.
[22] LI B,LIU N,LI Y,et al.Reduction of selenite to red elemental selenium by Rhodopseudomonas palustris strain N[J].PloS One,2014,9(4).DOI:10.1371/journal.pone.0095955.
[23] IKRAM M,FAISAL M.Comparative assessment of selenite (SeIV) detoxification to elemental selenium (Se0) by Bacillus sp.[J].Biotechnology letters,2010,32(9):1 255-1 259.
[24] TAN Y Q,YAO R,WANG R,et al.Reduction of selenite to Se(0) nanoparticles by filamentous bacterium Streptomyces sp. ES2-5 isolated from a selenium mining soil[J].Microbial Cell Factories,2016,15:157.
[25] KRITTAPHOL W,MCDOWELL A,THOMSON C D,et al.Biotransformation of L-selenomethionine and selenite in rat gut contents[J].Biological Trace Element Research,2011,139(2):188-196.
[26] PESCUMA M,GOMEZ-GOMEZ B,PEREZ-CORONA T,et al.Food prospects of selenium enriched-Lactobacillus acidophilus CRL 636 and Lactobacillus reuteri CRL 1101[J].Journal of Functional Foods,2017,35:466-473.
[27] KORA A J,RASTOGI L.Bacteriogenic synthesis of selenium nanoparticles by Escherichia coli ATCC 35218 and its structural characterisation[J].IET Nanobiotechnology,2017,11(2):179-184.
[28] CRUZ D M,MI G J,WEBSTER T J.Synthesis and characterization of biogenic selenium nanoparticles with antimicrobial properties made by Staphylococcus aureus,methicillin-resistant Staphylococcus aureus (MRSA),Escherichia coli,and Pseudomonas aeruginosa[J].Journal of Biomedical Materials Research Part A,2018,106(5):1 400-1 412.
[29] DE LEON C A P,BAYON M M,PAQUIN C,et al.Selenium incorporation into Saccharomyces cerevisiae cells:A study of different incorporation methods[J].Journal of Applied Microbiology,2002,92(4):602-610.
[30] MARINESCU G,STOICESCU A G,TEODOROF L.Industrial nutrient medium use for yeast selenium preparation[J].The Annals of the University Dunarea de Jos of Galati Facle VI-Food Technology,2011,35(1):45-53.
[31] BIERLA K,SUZUKI N,OGRA Y,et al.Identification and determination of selenohomolanthionine-the major selenium compound in Torula yeast[J].Food Chemistry,2017,237:1 196-1 201.
[32] OGRA Y,SHIMIZU M,TAKAHASHI K,et al.Biotransformation of organic selenium compounds in budding yeast,Saccharomyces cerevisiae[J].Metallomics,2018,10(9):1 257-1 263.
[33] RUOCCO M H W,CHAN C S,HANSON T E,et al.Characterization and distribution of selenite reduction products in cultures of the marine yeast Rhodotorula mucilaginosa-13B[J].Geomicrobiology Journal,2014,31(9):769-778.
[34] RAJA C P,JACOB J M,BALAKRISHNAN R M.Selenium biosorption and recovery by marine Aspergillus terreus in an upflow bioreactor[J].Journal of Environmental Engineering,2016,142(9).DOI:10.1061/(ASCE)EE.1943-7 870.0000999.
[35] ROSENFELD C E,KENYON J A,JAMES B R,et al.Selenium (IV,VI) reduction and tolerance by fungi in an oxic environment[J].Geobiology,2017,15(3):441-452.
[36] ESPINOSA-ORTIZ E J,RENE E R,GUYOT F,et al.Biomineralization of tellurium and selenium-tellurium nanoparticles by the white-rot fungus Phanerochaete chrysosporium[J].International Biodeterioration and Biodegradation,2017,124:258-266.
[37] GHARIEB M M,WILKINSON S C,GADD G M.Reduction of selenium oxyanions by unicellular,polymorphic and filamentous fungi:Cellular location of reduced selenium and implications for tolerance[J].Journal of Industrial Microbiology,1995,14(3-4):300-311.
[38] MOSALLAM F M,EL-SAYYAD G S,FATHY R M,et al.Biomolecules-mediated synthesis of selenium nanoparticles using Aspergillus oryzae fermented Lupin extract and gamma radiation for hindering the growth of some multidrug-resistant bacteria and pathogenic fungi[J].Microbial Pathogenesis,2018,122:108-116.
[39] COSTA-SILVA F,MARQUES G,MATOS C C,et al.Selenium contents of portuguese commercial and wild edible mushrooms[J].Food Chemistry,2011,126(1):91-96.
[40] VETCHINKINA E,LOSHCHININA E,KUPRYASHINA M,et al.Shape and size diversity of gold,silver,selenium,and silica nanoparticles prepared by green synthesis using fungi and bacteria[J].Industrial and Engieering Chemistry Research,2019,58(37):17 207-17 218.
[41] NUNES R G F L,DA LUZ J M R,FREITAS R D,et al.Selenium bioaccumulation in shiitake mushrooms:A nutritional alternative source of this element[J].Journal of Food Science,2012,77(9):C983-C986.
[42] NIEDZIELSKI P,MLECZEK M,SIWULSKI M,et al.Supplementation of cultivated mushroom species with selenium:Bioaccumulation and speciation study[J].European Food Research and Technology,2015,241(3):419-426.
[43] MILOVANOVIC I,BRCESKI I,STAJIC M,et al.Potential enrichment of medicinal mushrooms with selenium to obtain new dietary supplements[J].International Journal of Medicinal Mushrooms,2013,15(5):449-455.
[44] OGRA Y,ISHIWATA K,ENCINAR J R,et al.Speciation of selenium in selenium-enriched shiitake mushroom,Lentinula edodes[J].Analytical and Bioanalytical Chemistry,2004,379(5-6):861-866.
[45] CREMADES O,DIAZ-HERRERO M M,CARBONERO-AGUILAR P,et al.Preparation and characterisation of selenium-enriched mushroom aqueous enzymatic extracts (MAEE) obtained from the white button mushroom (Agaricus bisporus)[J].Food Chemistry,2012,133(4):1 538-1 543.
[46] GERGELY V,KUBACHKA K M,MOUNICOU S,et al.Selenium speciation in Agaricus bisporus and Lentinula edodes mushroom proteins using multi-dimensional chromatography coupled to inductively coupled plasma mass spectrometry[J].Journal of Chromatography A,2006,1101(1-2):94-102.
[47] HU T,HUI G F,LI H F,et al.Selenium biofortification in Hericium erinaceus (Lion’s Mane mushroom) and its in vitro bioaccessibility[J].Food Chemistry,2020,331:127 287.
[48] EGRESSY-MOLNAR O,OUERDANE L,GYORFI J,et al.Analogy in selenium enrichment and selenium speciation between selenized yeast Saccharomyces cerevisiae and Hericium erinaceus (lion’s mane mushroom)[J].LWT-Food Science and Technology,2016,68:306-312.
[49] RATHORE H,SHARMA A,PRASAD S,et al.Selenium bioaccumulation and associated nutraceutical properties in Calocybe indica mushroom cultivated on Se-enriched wheat straw[J].Journal of Bioscience and Bioengineering,2018,126(4):482-487.
[50] DA SILVA M C S,NAOZUKA J,DA LUZ J M R,et al.Enrichment of Pleurotus ostreatus mushrooms with selenium in coffee husks[J].Food Chemistry,2012,131(2):558-563.