豆渣又名豆腐渣,是豆腐、豆奶和腐竹等生产过程中的副产品[1]。新鲜豆渣含水量高、豆腥味重、口感粗糙、难以贮存且干燥成本高,因而应用率较低,造成了一定的环境问题和资源浪费[2]。豆渣中含有丰富的膳食纤维、矿物质和维生素等成分,可为部分微生物菌株的生长提供必要的营养需求,同时微生物的生长亦可改善豆渣的口感,并在发酵过程中产生多种活性物质和酶系[3]。我国是豆制品的生产和消费大国,因而利用微生物发酵开发豆渣产品是极为必要的[4]。
霉豆渣又名豆渣粑,是以豆渣为原料经微生物发酵制作而成的地方特色发酵食品,在我国湖北省、湖南省和江西省具有一定的分布,其中湖北省主要分布在荆州和洪湖地区[5]。近年来,国内研究人员围绕霉豆渣微生物多样性解析开展了系列研究,张燕鹏等[6]对江西省瑞金地区霉豆渣微生物类群解析发现,其真菌主要隶属于串珠霉属(Monilia)、根霉属(Rhizopus)和酵母属(Saccharomyces),细菌主要隶属于微球菌属(Micrococcus)、葡萄球菌属(Staphylococcus)、片球菌属(Pediococcus)和异常球菌属(Deinococcus)。毛欣欣[7]研究发现湖南省邵阳地区霉豆渣细菌类群主要为克罗诺杆菌属(Cronobacter)、不动杆菌属(Acinetobacter)和Staphylococcus,真菌类群主要为Rhizopus和丝孢酵母属(Trichosporon)。由此可见,不同地区制作的霉豆渣其微生物群落结构可能存在一定的差异,然而目前关于湖北省霉豆渣微生物类群的研究鲜见报道。
作为近年来较为流行的第二代高通量测序技术,Illumina MiSeq测序技术具有成本低、通量高和结果准确的优点,目前广泛地应用于豆豉[8]、豆瓣酱[9]、酱油[10]和腐乳[11]等发酵食品的微生物多样性解析中。本研究采用Illumina MiSeq测序技术对湖北省荆州地区下辖的监利市和石首县所产霉豆渣真菌多样性进行了比较分析,明确了2 个县市所产霉豆渣真菌多样性的共性及差异,并对造成差异的真菌类群进行了甄别,以期为后续以霉豆渣为代表的地方特色发酵豆制品的产业化推进提供数据支持。
1 材料与方法
1.1 材料与试剂
霉豆渣:采集自湖北省荆州地区下辖的监利市容城镇(E112°35′~113°19′,N29°26′~30°12′)和石首县高陵镇(E112°43′~112°47′,N29°67′~29°85′),2个采样点间的直线距离约为60 km。
引物SSU0817F/SSU1196R,武汉天一辉远生物科技有限公司;5×FastPfu Buffer、dNTPs Mix、FastPfu Fly DNA Polymerase,宝生物工程大连有限公司;基因组提取试剂盒,德国QIAGEN公司。
1.2 仪器与设备
PE300高通量测序平台,美国Illumina公司;CR21N高速冷冻离心机,日本HITACHI公司;ND-2000C微量紫外分光光度计,美国Nano Drop公司;vetiri梯度基因扩增仪,美国ABI公司;Fluor Chem FC3型化学发光凝胶成像系统,美国ProteinSimple公司;R930机架式服务器,美国DELL公司。
1.3 实验方法
1.3.1 样品采集
2019年11月于湖北省荆州地区下辖的监利市和石首县采集农家自制的霉豆渣样品各4份,编号为JL1~JL4和SS1~SS4。样品采集时,霉豆渣的制作时间在10~15 d,颜色一般为白色不带有黑色或绿色的斑点,略带酱臭味但不刺鼻,其制作工艺大体分为清浆、压榨、蒸料、再次压榨、摊晾、筛匀、成型和霉制等8 个流程。将采集的样品放入采样箱中带回实验室进行分装,置于-20 ℃冰箱中备用。
1.3.2 样品微生物宏基因组DNA提取、PCR扩增及高通量测序
按DNA提取试剂盒中的步骤进行DNA提取,使用分光光度计进行检验,置于-20 ℃暂存备用。以合格的DNA为模板,SSU0817F(5′-TTAGCATGGAATAATRRAATAGGA-3′)和SSU1196R(5′-TCTGGACCTGGTGAGTTTCC-3′)为引物,参照周书楠等[12]的方法对真菌18S rRNA V4~V5区进行PCR扩增。将合格的扩增产物寄往上海美吉生物医药科技有限公司使用PE300高通量测序平台进行测序。
1.3.3 生物信息学分析
序列下机后,参照郭壮等[13]的方法对序列进行质控,剔除错配的序列并进行筛选得到合格的序列集。利用QIIME分析平台[14],对合格的序列进行分析,继而对序列使用PyNAST软件[15]进行标准比对和对齐,之后使用2步UCLUST法在100%和97%的相似度下进行划分[16],构建分类操作单元矩阵(operational taxonomic units,OTU),最后使用SILVA数据库[17]对真菌 OTU 代表性序列进行同源性比对。在绘制系统发育树的基础上,对样品的发现物种数和香浓指数等α多样性指标进行分析,然后基于UniFrac距离使用加权组平均法(weighted pair-group method with arithmetic means,UPGMA)聚类和主坐标分析(principal co-ordinates analysis,PCoA)对2 个县市霉豆渣样品真菌类群进行β多样性解析。
1.3.4 多元统计分析及图表绘制
应用多元方差分析(multivariate analysis of variance,MANOVA)对2 个县市霉豆渣样品真菌群落结构的差异性进行分析,使用LEfSe(linear discriminant analysis effect size)分析对关键真菌类群进行甄别。使用R软件(v4.0.2)的waterfall软件包绘制瀑布图,使用Origin 2017绘制其他图。
2 结果与分析
2.1 α多样性分析
本研究的8 个霉豆渣样品经质控后得到216 309条高质量的序列,平均每个样品27 039条。经2步UCLUST划分后,监利市4 个霉豆渣样品OTU的数量分别为693、753、807 和770 个,石首县的分别为838、847、446和368 个。使用折线图对2 个县市样品的α多样性进行比较分析,进而判断本研究测序深度是否满足后续分析的要求。
由图1-a可知,当测序量达到近40 000 条时,稀释曲线尚未进入平台期,说明随着测序深度的增加霉豆渣中新的真菌物种会进一步被发现。由图1-b可知,当测序量达到5 000 条时,香农指数曲线已经进入平台期,这说明本研究的测序深度已可完全捕获霉豆渣中真菌类群的生物学信息。由此可见,本研究的测序深度是符合后续分析要求的。
本研究中样品的最小测序深度为10 010条序列。在该测序深度下,监利市4个霉豆渣样品的发现物种数分别为390、341、393和391,香浓指数为3.44、3.10、3.28和3.36;石首县各样品的发现物种数分别为357、346、170和348,香浓指数为2.98、2.75、0.90和2.89。经Mann-Whitney检验发现,采集自监利市的霉豆渣真菌类群的香浓指数显著偏高(P<0.01),而两者发现物种数差异不显著(P>0.05)。由此可见,采集自监利市的霉豆渣真菌多样性要显著高于石首县(P<0.01)。
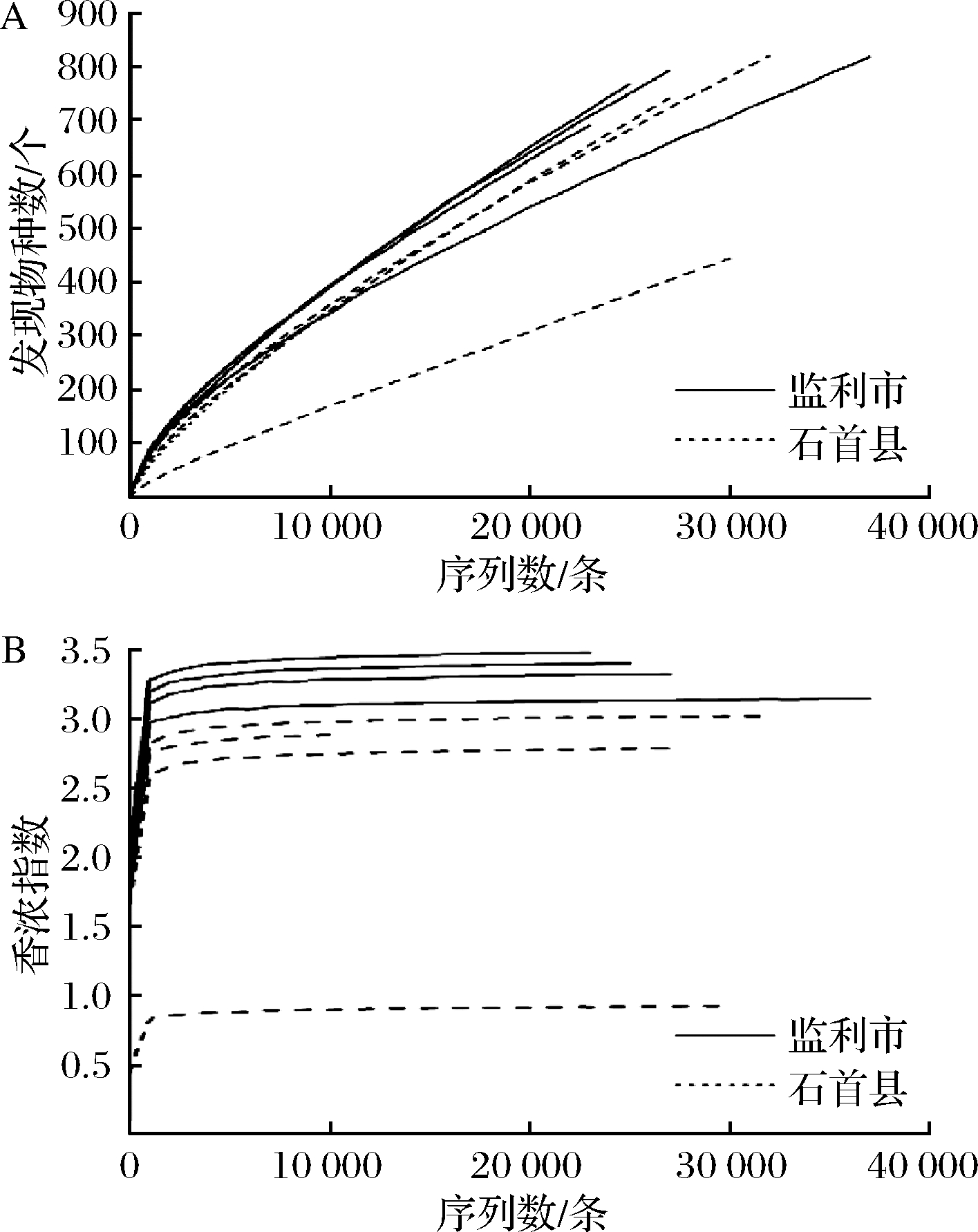
a-稀释曲线;b-香农指数曲线
图1 稀释曲线和香农指数曲线
Fig.1 Rarefaction curves and shannon index curves
2.2 基于门和属分类学地位的真菌类群分析
本研究将平均相对含量>1.00%的真菌门或属定义为优势门或属,将其他<1.00%的门或属归并为“其他”(others),将不能鉴定到门或者属水平的序列归并为“不可鉴定”(unclassified)。基于门水平2 个县市霉豆渣真菌类群的比较分析如图2所示。

图2 基于门水平2 个县市霉豆渣真菌类群的比较分析
Fig.2 Comparative analysis of fungal groups of Meitauza from Jianli city and Shishou county at the phylum level
由图2可知,所有霉豆渣样品中真菌类群可鉴定为3 个门,分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和毛霉门(Mucoromycota),平均相对含量分别为57.77%、33.54%和8.00%,有0.69%的序列无法鉴定到门水平。经Mann-Whitney检验发现,采集自2 个县市的霉豆渣样品在门水平上真菌类群差异均不显著(P>0.05)。
本研究中的所有序列可鉴定为32 个真菌属,其中平均相对含量>1.00%的有5 个,其相对含量如图3所示。

图3 基于属水平2 个县市霉豆渣真菌类群的比较分析
Fig.3 Comparative analysis of fungal groups of Meitauza from Jianli city and Shishou county at the genus level
由图3可知,5 个平均相对含量>1.00%的真菌属分别为镰刀霉属(Fusarium)、Trichosporon、篮状菌属(Talaromyces)、放射毛霉属(Actinomucor)和枝孢属(Cladosporium),平均相对含量分别为34.60%、28.96%、12.51%、7.27%和1.55%。经Mann-Whitney检验发现,2 个县市霉豆渣中仅Fusarium含量差异非常显著(P<0.01),其在监利市和石首县样品中的平均相对含量分别为56.74%和12.45%。
Fusarium和Trichosporon为监利市各样品中的优势真菌属,平均相对含量分别为56.74%和17.40%,样品JL1和JL2中亦存在一定量的Actinomucor,相对含量分别为7.46%和11.02%。采集自石首县的霉豆渣样品存在一定的差异性,其中Fusarium和Trichosporon为样品SS1、SS2和SS4中的优势菌,其平均相对含量分别为16.52%和51.65%,而样品SS3中优势真菌属为Talaromyces,相对含量达到88.39%。此外,较之样品SS2和SS4,样品SS1中还含有一定量的Actinomucor,相对含量为31.83%。由此可见,采集自石首县的霉豆渣真菌类群组间差异性可能要大于监利市。
研究表明,Fusarium和Talaromyces具有一定的致病性,Talaromyces可能引起中枢神经系统感染[18]和肺炎[19]等疾病,而Fusarium可能引起皮肤或呼吸道的感染[20]。有研究表明霉豆渣主要依靠毛霉菌进行发酵,管瑛[21]从湖北省黄石市阳新县霉豆渣中分离出了隶属于毛霉科(Mucoraceae)的雅致放射毛霉(Actinomucor elegans)。本研究发现Actinomucor仅为部分霉豆渣样品中的优势真菌属,其在样品JL3和SS2中的相对含量均<1.00%,而在样品SS3中根本不存在。由此可见,霉豆渣样品的真菌类群整体上是存在较大差异的,究其原因可能在于霉豆渣含水量较高、营养物质丰富且制作方式为开放式发酵,多种真菌均可以其为基质进行生长,因而环境、制作工具及豆渣本身携带的微生物直接决定了最终产品中的真菌类群构成。由于发酵环境的开放性和发酵条件的不可控,霉豆渣的生产受外界影响较大且存在一定的食用安全隐患,因而在后续研究中积极分离、鉴定和筛选具有较高食用安全性和优良发酵特性的Actinomucor菌株,并将其用于霉豆渣的生产具有积极的意义。
2.3 基于OTU水平的真菌类群分析
在97%的相似度下,本研究将序列划分到了3 918 个OTU,若某一OTU在8 个样品中均存在,则定义其为核心OTU,相对含量如图4所示。

图4 基于OTU水平的2 个县市霉豆渣真菌核心类群
Fig.4 Fungal core groups of Meitauza from Jianli city and Shishou county at the OTU level
由图4可知,核心OTU共有10 个,仅占总OTU数量的0.26%,但包含的序列数占总序列的59.56%。OTU1 366被鉴定为Trichosporon,平均相对含量为27.50%;OTU2 599、OTU2 028、OTU1 846、OTU963和OTU1 206被鉴定为Fusarium,其中OTU963的平均相对含量高达29.08%;而其他4 个OTU均鉴定不到属水平,累计平均含量仅为0.29%。这也进一步证实了,Fusarium和Trichosporon为采集自监利市和石首县多数霉豆渣样品中的优势真菌类群。
本研究发现有20 个OTU在监利市4 个霉豆渣样品中均存在,但在石首县样品中均不存在,其累计包含序列仅占总序列数的0.69%,且均鉴定不到属水平。没有发现有OTU仅存在于石首县4个霉豆渣样品中,而在监利市样品中不存在。
2.4 β多样性分析
本研究进一步采用基于UniFrac距离的UPGMA聚类和PCoA对2 个县市霉豆渣真菌类群的β多样性进行了分析,结果如图5所示。

a-基于聚类分析;b-主坐标分析
图5 基于聚类分析和主坐标分析的2个县市
霉豆渣真菌类群β多样性分析
Fig.5 Analysis of beta diversity of fungal groups in Meitauza from Jianli city and Shishou county using cluster analysis and principal coordinate analysis
由图5-a可知,监利市4个样品形成一个聚类,石首县样品SS2和SS4可单独形成一个聚类,虽SS1和SS3与该地区其他2个样品无法形成一个分支,但2个县市霉豆渣形成了明显的聚类趋势。由图5-b可知,监利市样品主要分布在第一和第四象限,而石首县样品主要分布在第二、三和四象限。由此可见,不同县市制作的霉豆渣样品其真菌类群呈现出一定的差异。在提取PCoA前85%因子的基础上,本研究采用MANOVA分析发现,2个县市霉豆渣样品真菌群落结构存在显著差异(P<0.05)。本研究团队对湖南省湘西土家族苗族自治州龙山县和湖北省宜昌市当阳市的豆豉[22]及湖北省恩施土家族苗族自治州咸丰县和湖北省宜昌市当阳市的辣椒酱[23]细菌多样性进行了解析,亦发现了类似的现象,究其原因可能在于生态环境和制作工艺的不同会对发酵食品微生物类群的形成产生一定影响。大量研究表明,不同地区特殊发酵食品中蕴含了较为丰富和独特的微生物类群,对其进行挖掘、保藏和筛选,对我国特色发酵食品的产业化推进和使用安全性提升均具有积极的意义[24]。
由图5-a可知,监利市样品JL1和JL2,JL3和JL4两两形成一个分支且在树的末端,这说明4个样品的真菌类群较为相似;而石首县样品SS3和SS1的分支相对较长,这说明这2个样品真菌类群与其他样品存在较大差异。由图5-b可知,较之监利市样品,石首县样品空间分布更为分散。由此可见,采集自石首县的霉豆渣真菌类群组间差异性可能要大于监利市,这也进一步验证了图3的结论。本研究进一步对2 个县市样品真菌类群的组间差异进行了分析,结果如图6所示。

图6 两个县市霉豆渣真菌类群的组间差异性分析
Fig.6 Intergroup difference analysis of fungal groups in Meitauza from Jianli city and Shishou county
注:*表示两者差异显著(P<0.05)
由图6可知,采集自监利市霉豆渣样品真菌类群的组间距离为0.039±0.002,而石首县为0.107±0.206,且经Mann-Whitney检验发现,两者差异显著(P<0.05)。由此可见,采集自石首县的霉豆渣真菌类群组间差异性大于监利市。
2.5 关键真菌类群甄别
本研究进一步采用LEfSe分析对导致2 个县市霉豆渣真菌群落结构存在差异的类群进行了甄别,结果如图7所示。
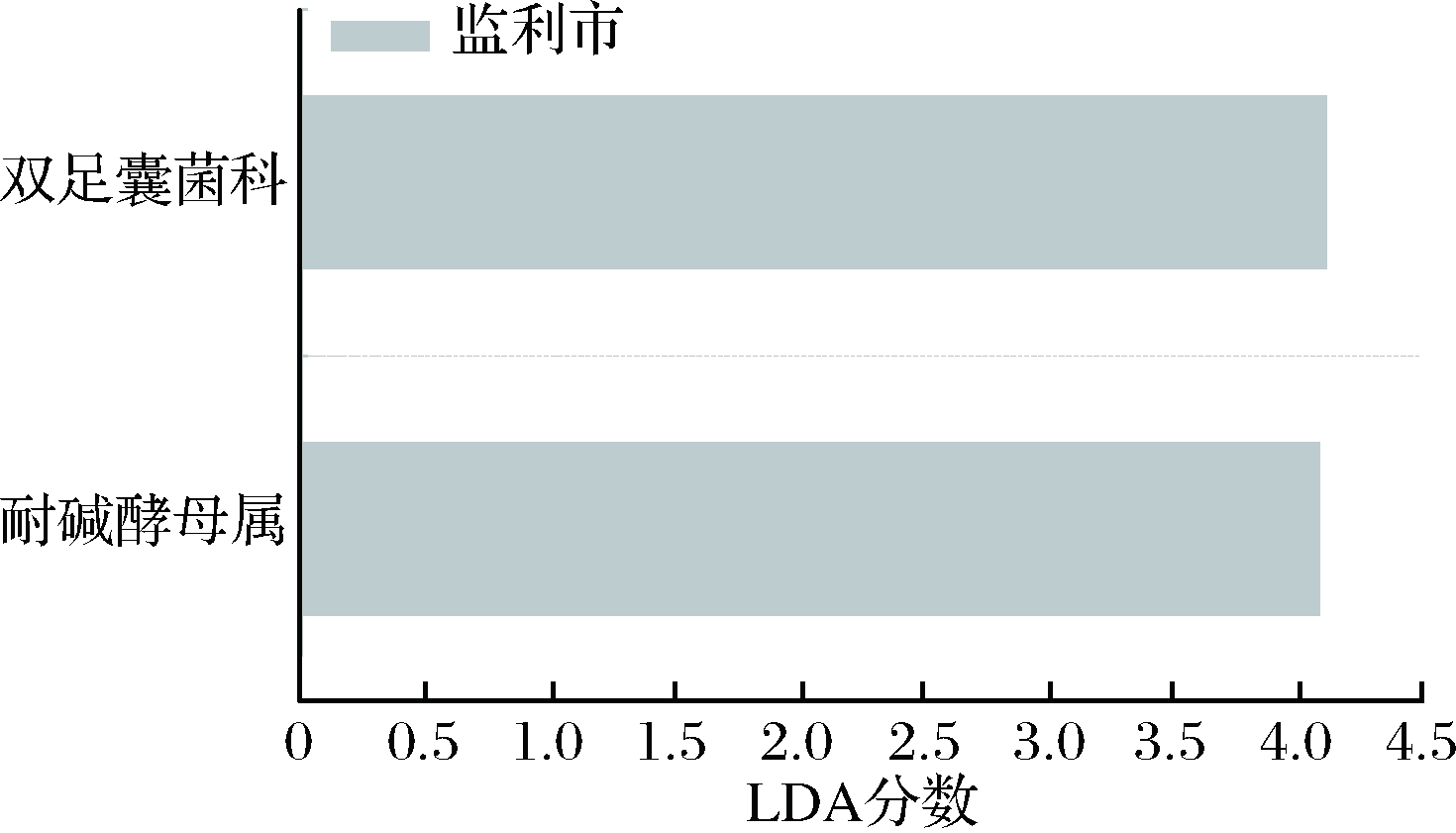
图7 基于LEfSe分析2 个县市霉豆渣关键真菌类群的甄别
Fig.7 Identification of key fungal groups in Meitauza from Jianli city and Shishou county by LEfSe analysis
由图7可知,导致2 个县市霉豆渣真菌群落结构存在差异的类群隶属于双足囊菌科(Dipodascaceae)的耐碱酵母属(Galactomyces),其在采集自监利市样品中的相对含量为1.46%,而在石首县样品中仅为0.62%,为非优势菌属。值得一提的是,除Galactomyces外并未发现其他类群导致2个县市霉豆渣真菌群落结构存在差异。由此可见,正是由于部分相对含量较少的真菌类群存在差异,进而导致了采集自2个县市的霉豆渣真菌群落结构的不同。
3 结论
湖北省荆州地区霉豆渣中的优势真菌类群以Fusarium、Trichosporon、Talaromyces、Actinomucor和Cladosporium为主。虽然荆州地区下辖的监利市和石首县制作的霉豆渣具有大量的核心真菌类群,但采集自2 个地区的样品真菌类群存在显著的差异,且该差异主要是由Galactomyces等非优势真菌类群导致的。
[1] 夏秀东, 刘小莉, 王英, 等.豆渣固定化培养乳酸菌的条件优化及其对乳酸菌的保护作用[J].江西农业学报, 2017, 29(9):89-93.
XIA X D, LIU X L, WANG Y, et al.Optimization of conditions for immobilized culture and protection of lactic acid bacteria on soybean dreg (okara)[J].Acta Agriculturae Jiangxi, 2017, 29(9):89-93.
[2] XIU D X, YING W, XIAO L L, et al.Soymilk residue (okara) as a natural immobilization carrier for Lactobacillus plantarum cells enhances soymilk fermentation, glucosidic isoflavone 59 bioconversion, and cell survival under simulated gastric and intestinal conditions [J].PeerJ, 2016, 4:e2 701.
[3] 李伟伟, 曲俊雅, 周才琼.真菌及乳酸菌联合发酵对豆渣膳食纤维及理化特性的影响[J].食品与发酵工业, 2018, 44(11):159-166.
LI W W, QU J Y, ZHOU C Q.Effects of combined fermentation of fungi and lactic acid bacteria on dietary fiber and physicochemical properties of soybean dregs[J].Food and Fermentation Industries, 2018, 44(11):159-166.
[4] 申春莉, 李曼, 沙见宇, 等.灵芝菌丝体固态发酵豆渣的营养成分变化[J].食品与发酵工业, 2019, 45(12):114-119.
SHEN C L, LI M, SHA J Y, et al.Nutrient changes in solid-state fermented okara with Ganoderma lucidum[J].Food and Fermentation Industries, 2019, 45(12):114-119.
[5] 姚英政. 霉豆渣粑发酵过程中营养及风味成分变化研究[D].武汉:华中农业大学, 2010.
YAO Y Z.Study on the changes in nutrition and flavor ingredients during soybean residue cakes fermentation[D].Wuhan:Huazhong Agricultural University, 2010.
[6] 张燕鹏, 杨瑞金, 王贺, 等.传统豆渣菌的菌相分析及蛋白酶和纤维素酶主要产生菌株的鉴定[J].食品工业科技, 2012, 33(1):171-174.
ZHANG Y P,YANG R J,WANG H, et al.Analysis of the microflora and identification of the protease and cellulase producing strains from the traditional fermentative Douzhajun[J].Science and Technology of Food Industry, 2012, 33(1):171-174.
[7] 毛欣欣. 发酵豆渣微生物多样性与其营养成分变化关系研究[D].广州:华南农业大学, 2019.
MAO X X.Study on the relationship between microbial diversity of fermented soybean dregs and its nutrient composition[D].Guangzhou:South China Agricultural University, 2019.
[8] 倪慧, 王玉荣, 尚雪娇, 等.龙山地区干豆豉真菌多样性研究[J].食品研究与开发, 2020, 41(4):27-31.
NI H, WANG Y R, SHANG X J, et al.Study on fungal diversity of dried Douchi in Longshan[J].Food Research and Development, 2020, 41(4):27-31.
[9] SUN X D, LYU G Z, LUAN Y S, et al.Analyses of microbial community of naturally homemade soybean pastes in Liaoning Province of China by Illumina Miseq Sequencing[J].Food Research International, 2018, 111:50-57.
[10] YAN Y Z, QIAN Y L, JI F D, et al.Microbial composition during Chinese soy sauce koji-making based on culture dependent and independent methods[J].Food Microbiology, 2013, 34(1):189-195.
[11] XU D D, WANG P, ZHANG X, et al.High-throughput sequencing approach to characterize dynamic changes of the fungal and bacterial communities during the production of sufu, a traditional Chinese fermented soybean food[J].Food Microbiology, 2020, 86:103 340.
[12] 周书楠, 王玉荣, 周亚澳, 等.基于MiSeq高通测序技术的米酒真菌多样性分析[J].食品工业科技, 2019, 40(8):85-89;96.
ZHOU S N, WANG Y R, ZHOU Y A, et al.Diversity analysis of fungal microflora in rice wine by MiSeq high throughput sequencing[J].Science and Technology of Food Industry, 2019, 40(8):85-89;96.
[13] 郭壮, 葛东颖, 尚雪娇, 等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技, 2018, 39(22):93-98;106.
GUO Z, GE D Y, SHANG X J, et al.Comparative analysis on the diversity of bacterial microflora in degenerated and normal pit mud[J].Science and Technology of Food Industry, 2018, 39(22):93-98;106.
[14] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[15] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al.PyNAST:A flexible tool for aligning sequences to a template alignment [J].Bioinformatics, 2010, 26(2):266-267.
[16] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics, 2010, 26(19):2 460-2 461.
[17] QUAST C, PRUESSE E, YILMAZ P, et al.The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools [J].Nucleic Acids Research, 2013, 41(1):590-596.
[18] WANG D M, MA H L, TAN M Q, et al.Next-generation sequencing confirmed the diagnosis of isolated central nervous system infection caused by Talaromyces marneffei in an immunocompetent patient[J].Chinese Medical Journal,2020,133(3):374-376.
[19] BOURASSA L, DOPPALAPUDI A, BUTLER-WU S M.Closing the brief case:Pneumonia caused by Talaromyces marneffei[J].Journal of Clinical Microbiology, 2019, 57(5):1 690-1 718.
[20] MOLINA A, CHAVARR A G, ALFARO-CASCANTE M, et al.Mycotoxins at the start of the food chain in Costa Rica:Analysis of six Fusarium toxins and ochratoxin a between 2013 and 2017 in animal feed and aflatoxin M1 in dairy products[J].Toxins, 2019, 11(6):312.
A G, ALFARO-CASCANTE M, et al.Mycotoxins at the start of the food chain in Costa Rica:Analysis of six Fusarium toxins and ochratoxin a between 2013 and 2017 in animal feed and aflatoxin M1 in dairy products[J].Toxins, 2019, 11(6):312.
[21] 管瑛. 雅致放射毛霉DCY-1固态发酵豆渣的营养及功能性研究[D].南京:南京农业大学, 2016.
GUAN Y.Effects on nutrition and bio-functionality of okara by solid-state fermentation with Actinomucor elegans DCY-1[D].Nanjing:Nanjing Agricultural University, 2016.
[22] 王强, 王玉荣, 陈江红, 等.龙山豆豉细菌多样性分析及其与当阳豆豉差异性比较[J].中国酿造, 2020, 39(1):26-30.
WANG Q, WANG Y R, CHEN J H, et al.Bacterial diversity in Longshan Douchi and differences comparison with Dangyang Douchi[J].China Brewing, 2020, 39(1):26-30.
[23] 宁明, 赵馨馨, 董蕴, 等.基于Illumina MiSeq测序技术不同地区辣椒酱细菌多样性分析[J].中国调味品, 2020, 45(2):58-63.
NING M, ZHAO X X, DONG Y, et al.Diversity of bacteria microflora in chili sauce of different area by Illumina MiSeq sequencing technology[J].China Condiment, 2020, 45(2):58-63.
[24] 张娟, 陈坚.中国传统发酵食品产业现状与研究进展[J].生物产业技术, 2015,9(4):11-16.
ZHANG J, CHEN J.Current situation and research progress of Chinese traditional fermented food industry[J].Bioindustry Technology, 2015, 9(4):11-16.