宣恩火腿是湖北省恩施自治州一带特产,其在乾隆时期曾作为贡品,以宣恩县所产火腿最负盛名,甚至被誉为中国“四大名腿”之一[1-2]。宣恩火腿一直沿用传统工艺生产,微生物自然接种,在长达1~2年的加工过程中,形成了独特的质地和风味。关于宣恩火腿发酵过程中蛋白质、脂肪等理化指标的变化规律已有报道[2-4],但宣恩火腿发酵过程中微生物群落的变化规律有待揭示。
研究微生物群落的方法主要包括分离培养法[5]、Biolog-ECO微平板技术[6]、变性梯度凝胶电泳法[7]、16S rRNA/18S rRNA/ITS基因克隆文库测序法[8]和高通量测序法等。高通量测序技术,又称下一代测序技术,通过测定细菌16S rRNA序列、真菌ITS序列等,与对应的物种分类数据库进行比对,从而确定微生物种类和丰度[9-11];其虽然不能获得纯培养菌株,但一次可测得单个样本中数万条菌株序列信息,故能以更高的灵敏度、准确度和相对低廉的价格快速鉴定样本中的微生物种类和丰度,包括丰度很低的种类[12-13]。
微生物在火腿发酵过程中扮演着重要角色,与火腿的风味、品质和安全性密切相关,研究火腿发酵过程中微生物群落组成具有很强的实际意义。本文利用高通量测序技术对不同发酵期宣恩火腿表面的细菌、真菌群落多样性进行研究,以期为宣恩火腿建立工业化生产工艺作铺垫。
1 材料与方法
1.1 材料及取样
不同发酵期宣恩火腿表面(肉面)微生物样本,于2019年11月湖北省思乐牧业集团有限公司采样。宣恩火腿加工工艺流程[4, 14]:选腿(猪后腿)→修胚→摊凉→腌制→洗腿→整形→烘腿→入库发酵→洗霉→修割→验收。自“入库发酵”开始计算发酵时间,取样点为发酵前期(发酵40 d,霉菌生长完全覆盖火腿表面)、发酵中期(发酵90 d,霉菌生长最旺盛阶段)、发酵后期(发酵570 d,霉菌生长消退;发酵180 d以上为发酵后期,但发酵1~2年能更好地保证火腿中病毒失活,增加安全性和品质[15])。采样参照GB 4789.1—2016《食品安全国家标准 食品微生物学检验 总则》规定,各阶段样本分别随机选取20个面积约5 cm×5 cm的取样区(肉面),用灭菌棉球反复擦拭火腿表面(肉面)微生物,并清洗到100 mL无菌水中,而后转移至无菌离心管,6 000 r/min离心15 min,取沉淀备用。
1.2 主要仪器与试剂
GeneAmp® 9700型PCR仪,美国ABI公司;QuantiFluorTM-ST蓝色荧光定量系统,美国Promega公司;Illumina MiSeq测序平台、TruSeqTM DNA Sample Prep Kit,美国Illumina 公司;FastDNA® SPIN Kit for Soil土壤DNA快速提取试剂盒,美国MPbio公司;TransStart Fastpfu DNA 聚合酶,北京TransGen生物;AxyPrepDNA凝胶回收试剂盒,美国Axygen公司。
1.3 实验方法
1.3.1 微生物基因组DNA的提取及高通量测序
取沉淀样本,用土壤DNA快速提取试剂盒提取总DNA,1%琼脂糖凝胶电泳检测后,用NanoDrop 2000 测定DNA纯度和含量。通过PCR扩增细菌16S rRNA V3~V4区,引物为338F(5′-ACTCCTACGGGAGGCAGCAG-3′)、806R(5′-GGACTACHVGGGTWTCTAAT-3′),扩增真菌ITS1-ITS2序列,引物为ITS1F(5′-CTTGGTCATTTAGAGGAAGTAA-3′)、ITS2R(5′-GCTGCGTTC-TTCATCGATGC-3′);PCR产物经电泳、回收、定量分析后进行Illumina MiSeq测序[16],测序公司为上海美吉生物医药科技有限公司。
1.3.2 高通量测序结果分析
MiSeq高通量测序结果借助美吉生物云平台进行分析,主要分析流程为:将测序原始序列进行拼接和质控,按样本进行操作分类单元(operational taxonomic unit,OTU)聚类分析和物种分类学分析,对数据进行抽平处理;基于抽平后的OTU进行Alpha多样性分析;基于分类学信息,在不同分类学水平上进行群落结构统计分析;并对细菌、真菌优势种进行物种相关性网络分析、系统发育分析(即进化分析);并对群落功能进行预测。
OTU分析使用Usearch平台(version 7.0, http://drive5.com/uparse/),相似性在97%以上的序列聚为一类,细菌比对Silva数据库(Release132, http://www.arb-silva.de),真菌比对Unite数据库(Release 7.2, http://unite.ut.ee/index.php);物种相关性网络分析使用Networkx软件;系统发育分析使用FastTree软件(version 2.1.3, http://www.microbesonline.org/fasttree/);功能预测分析使用PICRUSt软件包(version 1.1.0, http://picrust.github.io/picrust/),细菌使用EggNOG(evolutionary genealogy of genes:Non-supervised Orthologous Groups,http://eggnog.embl.de/)数据库,真菌使用MetaCyc(https://metacyc.org/)数据库;样本与物种关系Circos图用Circos-0.67-7(http://circos.ca/)完成,其他图使用Excel软件、R语言工具统计和作图。
1.3.3 高丰度未知物种的进一步注释
高通量测序结果分析中,一些物种的分类无法确定(no_rank/unclassified),挑选其中高丰度OTU代表序列,在线比对NCBI中的16S rRNA或ITS数据库(如果没有高度相似序列,则进一步比对nr数据库),并基于查询的16S rRNA或ITS序列,用MEGA7软件通过最大似然法构建系统发生进化树,以对物种进行更精确的注释(Bootstrap值设置为1 000次重复)[17]。
2 结果与分析
2.1 样本序列统计
宣恩火腿样本测序信息见表1。经质控、拼接,细菌(含古菌)16S rRNA序列V3~V4区共测得134 721条序列,平均长度426 bp;真菌ITS1-ITS2序列共测得111 395条序列,平均长度231 bp。按最小序列数抽平处理[18],获得抽平后的序列数为细菌38 829条/样本、真菌36 268条/样本。
表1 样本信息和Alpha多样性指数
Table 1 Sample information and Alpha diversity index
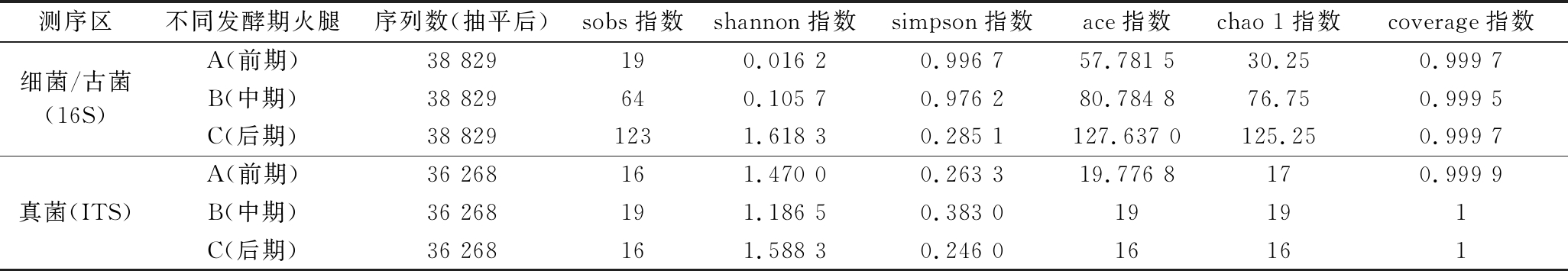
测序区不同发酵期火腿序列数(抽平后)sobs指数shannon指数simpson指数ace指数chao 1指数coverage指数细菌/古菌(16S)A(前期)38 829190.016 20.996 757.781 530.250.999 7B(中期)38 829640.105 70.976 280.784 876.750.999 5C(后期)38 8291231.618 30.285 1127.637 0125.250.999 7真菌(ITS)A(前期)36 268161.470 00.263 319.776 8170.999 9B(中期)36 268191.186 50.383 019191C(后期)36 268161.588 30.246 016161
2.2 宣恩火腿微生物群落Alpha多样性分析
宣恩火腿表面微生物群落Alpha多样性指数见表1。多样性指数中,sobs指数、ace指数和chao1指数可显示群落物种的丰富度,数值越大代表物种丰富度越高;shannon指数和simpson指数综合反映群落多样性,shannon指数越大代表群落多样性越高,simpson指数越小代表群落多样性越高;coverage指数显示物种覆盖度,值越接近1(即100%)表明测序越全面,越能代表样本真实物种组成情况[19-20]。
表1中,所有样本真菌和细菌测序的覆盖率(coverage)指数均大于99.9%,同时样品稀释曲线趋于平缓(图1),说明增加测序量只能发现极少量的物种,即测序结果能很好地反应样本微生物多样性。宣恩火腿发酵过程中,细菌(含古菌)的sobs、ace和chao1指数持续增加,显示物种丰富度不断提高;shannon指数持续显著增加,simpson指数持续显著减小,显示细菌多样性持续提高。发酵过程中,真菌sobs、ace和chao1指数变化较小,显示真菌物种丰富度变化相对较小;真菌的shannon指数先减小后增大,simpson指数先增大后减小,但变化幅度均较小,显示真菌多样性波动较小,呈现微弱的先减小后增大的趋势。另外,比较sobs指数、ace指数和chao 1指数可发现:细菌物种丰富度整体大于真菌。因此,在发酵过程中,宣恩火腿表面细菌丰富度和多样性持续增加,真菌的丰富度和多样性波动较小,且整个发酵过程中细菌物种丰富度大于真菌。
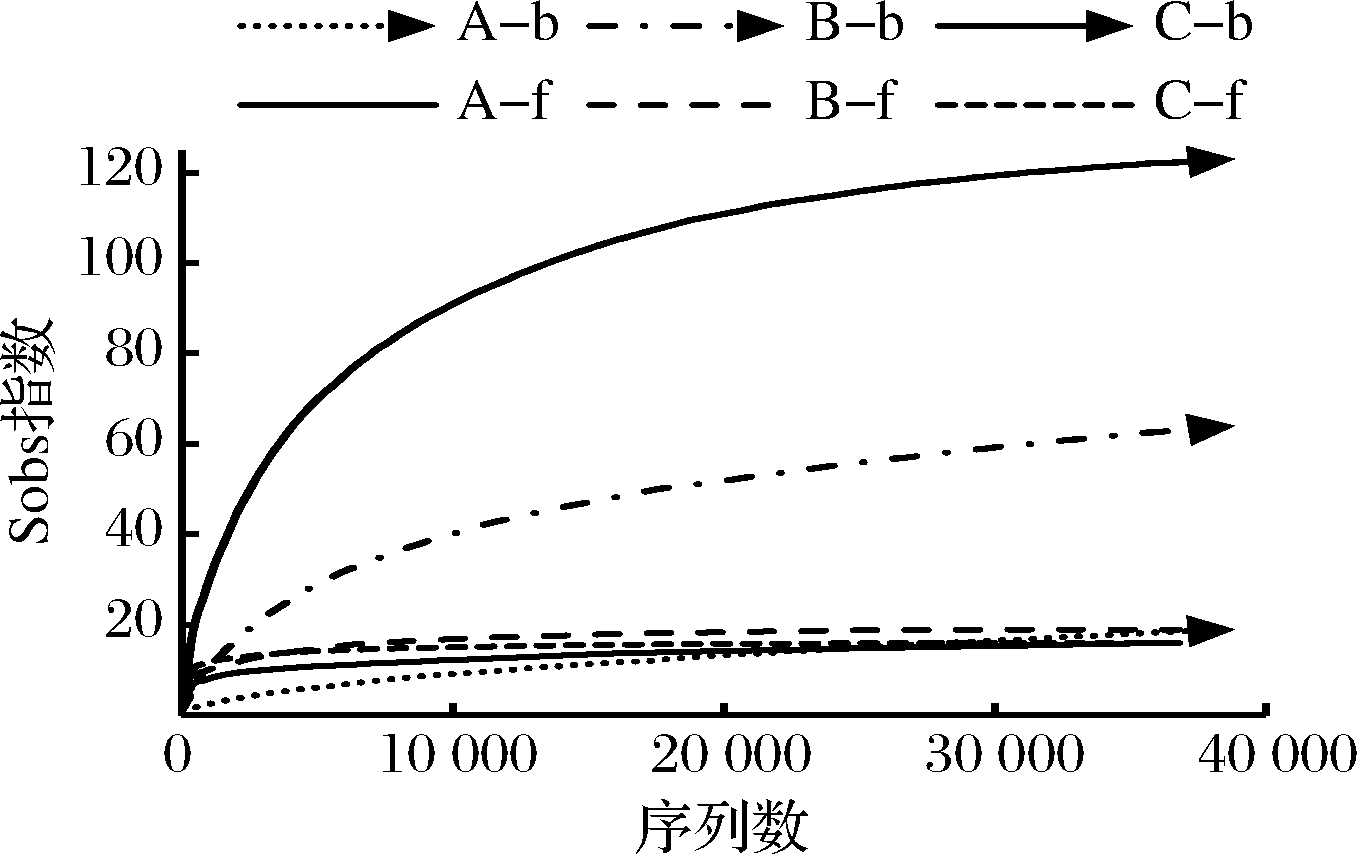
图1 样品稀释曲线
Fig.1 Rarefaction curves of samples
注:A-b、B-b、C-b依次为发酵前期、中期、后期火腿样本的细菌群落;
A-f、B-f、C-f分别为发酵前期、中期、后期火腿样本的真菌群落
2.3 宣恩火腿微生物群落组成分析
2.3.1 细菌(含古菌)群落组成分析
在97%的相似水平上对宣恩火腿表面细菌16S rRNA V3~V4区测序结果进行OTU分析,通过Venn图展示宣恩火腿发酵过程中共有和独有的细菌(含古菌)物种数如图2-a所示。所有样本中共测得133个细菌OTUs,其中发酵前期、中期、后期的火腿样本依次测得19、64、123个OTUs;说明宣恩火腿发酵过程中,细菌群落丰富度逐渐增加,不断有新的细菌种类参与发酵过程。
在门水平上,所有样本共检出11个门的细菌,除1个丰度很低的古细菌门(Euryarchaeota,广古菌门,占比<0.01%)外,其余10个为真细菌门,主要包括厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)等。发酵前、中期,厚壁菌门占绝对优势(>99%);发酵后期,变形菌门占69%,而厚壁菌门下降到27%(图2-b)。宣恩火腿表面细菌在属水平(共96个)、种水平(共125个)上的组成分别见图2-c和图2-d。图2-d为Circos图,是一种描述样本与物种之间对应关系的可视化圈图,其不仅反映了每个样本的优势物种组成比例,同时也反映了各优势物种在不同样本(分组)中的分布比例[21]。发酵前、中期,葡萄球菌属的木糖葡萄球菌(Staphylococcus xylosus)占据绝对优势(>98%);发酵后期,沙雷氏菌属(Serratia)未知种unclassified_g_Serratia(OTU61)占比最大,为42.2%,前期占优势的木糖葡萄球菌下降到24.9%,根瘤菌科未知属未知种s_uncultured_Rhizobiales_bacterium(OTU113)占20.8%,甲基菌属的藤黄亚甲基杆菌(Methylobacterium fujisawaense)占3.9%,Solitalea属未培养菌s_uncultured_bacterium_g_Solitalea占1.9%(OTU72),芽孢杆菌科未知种s_unclassified_f_Bacillaceae占1.6%(OTU37),其他占4.7%。
综上,宣恩火腿发酵中,细菌群落丰富度不断增加,各物种相对丰度呈动态变化,发酵前、中期主要为木糖葡萄球菌,发酵后期的主要优势属为葡萄球菌属、沙雷氏菌属和甲基菌属。
2.3.2 真菌群落组成分析
在97%的相似水平上对真菌ITS1-ITS2测序结果进行OTU分析,通过Venn图展示宣恩火腿发酵过程中共有和独有的真菌物种数目信息(图2-f)。3个发酵期样本中真菌群落共测得21个OTUs,其中发酵前、中、后期的火腿样本依次测得16、19、16个OTUs。所有发酵期共有OTUs为12个,占57%;发酵前期没有独有OTUs,发酵中、后期分别独有1、2个OTUs。因此,宣恩火腿发酵过程中真菌种类较为稳定,57%的种类在整个发酵期始终存在。
宣恩火腿真菌包括子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和未分类的门(unclassified_k_Fungi)。发酵过程中,子囊菌门占比持续增加,担子菌门占比不断减少,未分类门呈先增加后减少的趋势(图2-g)。宣恩火腿表面真菌在属水平(共8个)、种水平(共12个)上的组成分别见图2-h和图2-e。曲霉属(Aspergillus)在发酵过程中较为稳定,发酵前、中、后期占比依次为37.9%、43.2%和41.0%,但在种水平上有物种更替,发酵前、中期Aspergillus cibarius占优势,而发酵后期帚状曲霉(Aspergillus penicillioides)占优势;未分类真菌s_unclassified_k_Fungi(OTU7)在发酵前、中、后期占比依次为24.3%、46.5%和25.6%;节菌属(Wallemia)在发酵过程中逐渐下降,发酵前、中、后期占比依次为37.6%、9.9%和1.5%;未分类酵母unclassified_o_Saccharomycetales(OTU20)由发酵前期未检出、中期占0.01%,到发酵后期占31.1%,说明酵母是发酵后期优势类群之一。

a-细菌OTU水平Venn图;b-细菌门水平相对丰度;c-细菌属水平相对丰度;d-细菌种水平circos图;
e-真菌种水平Circos图;f-真菌OTU水平Venn图;g-真菌门水平相对丰度;h-真菌属水平相对丰度
图2 宣恩火腿的微生物区系
Fig.2 The microbiota of Xuanen ham
注:A、B、C依次为发酵前、中、后期的火腿样本
综上,宣恩火腿发酵过程中,真菌种类相对稳定,但各物种相对丰度存在动态变化,发酵前、中期主要是Aspergillus cibarius、未分类真菌(OTU7)、节菌属占优势,发酵后期主要优势种为帚状曲霉、未分类真菌(OTU7)、未分类酵母(OTU20)等。
2.4 物种相关性网络分析
2.4.1 单因素网络分析
对宣恩火腿表面细菌、真菌优势种分别进行单因素网络分析,当物种之间的相关系数符合某一阈值(P<0.05)时就用线连接,用不同颜色表示物种之间正、负相关性[22],结果见图3-a和图3-b。细菌群落中,木糖葡萄球菌与其他种类呈负相关,其他种之间互为正相关(图3-a)。真菌群落中,曲霉属Aspergillus cibarius和节菌属s_unclassified_g_ Wallemia呈正相关;这两者与发酵后期占优势的帚状曲霉(A.penicillioides)、巴恩曲霉菌(A.baarnensis)、三角山氏酵母(Yamadazyma triangularis)和未分类酵母s_unclassified_o_Saccharomycetales(OTU20)均为负相关;后4种真菌互为正相关(图3-b)。虽然发酵过程中,未分类真菌(OTU7)丰度一直很高,但它与其他种类真菌的丰度没有显著相关性,故未在图3-b中显示。
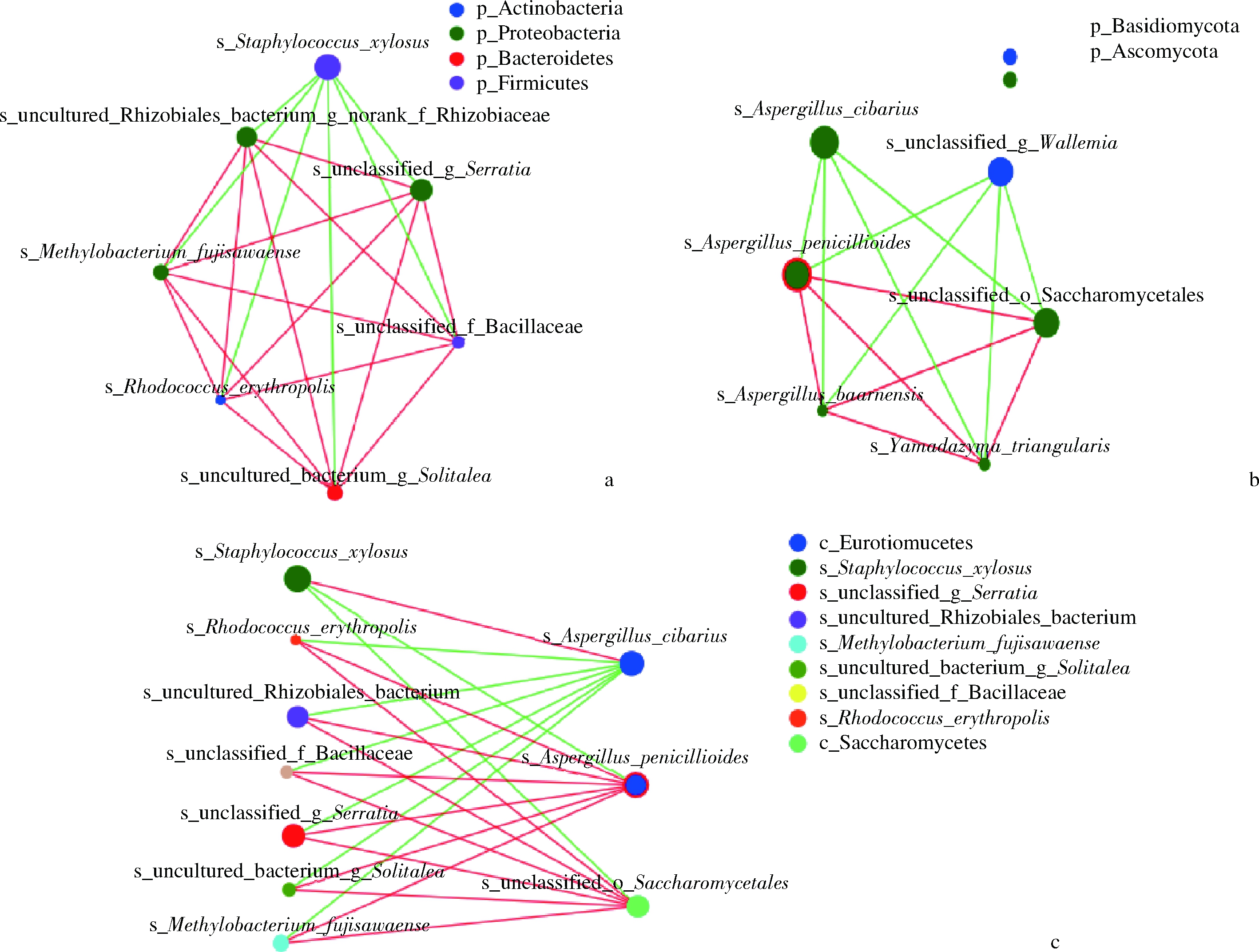
a-细菌种水平单因素网络分析;b-真菌种水平单因素网络分析;c-细菌与真菌种水平双因素网络分析
图3 宣恩火腿微生物相关性网络分析
Fig.3 Network analysis for microbiota of Xuanen ham
注:节点大小与种的丰度成正比,红色连线代表正相关,绿色连线代表负相关;a与b中节点颜色代表门类别,c中节点颜色代表纲类别
2.4.2 双因素网络分析
将主要细菌优势种和真菌优势种(作为其他因子)进行双因素网络分析,结果见图3-c。曲霉属Aspergillus cibarius与木糖葡萄球菌呈正相关,与其他细菌呈负相关;帚状曲霉(A.penicillioides)、未分类酵母(OTU20)与木糖葡萄球菌呈负相关,与其他细菌呈正相关(图3-c);其他主要真菌与细菌种类没有显著相关性,故未在图3-c中显示。
2.5 进化分析
在种水平上对宣恩火腿主要细菌和真菌分别进行进化分析,结果见图4。主要细菌种类来自厚壁菌门、变形菌门、拟杆菌门和放线菌门等4个门(图4-a);主要真菌种类来自子囊菌门、担子菌门和未分类的门(图4-c)。将OTU分析中没有成功注释到种的OTU代表序列,与NCBI数据库中相似度最高的序列一起构建个性化系统发生树(图4-b、4-d),以对物种进行更精确的注释。
细菌(图4-b)中,根瘤菌科未知种uncultured_Rhizobiales_bacterium(OTU113)与Methylobacterium brachiatum和M.phyllostachyos聚为一类,16S rRNA序列一致度为100%,所以OTU113能进一步鉴定到甲基菌属(Methylobacterium),可能为两者之一;同理,芽孢杆菌科未知属未知种unclassified_Bacillaceae(OTU37)可鉴定为Corynebacterium tuberculostearicum,Solitalea属未知种uncultured_bacterium_Solitalea(OTU72)可鉴定为Oceanobacillus picturae,叶杆菌属未知种uncultured_bacterium_Phyllobacterium(OTU84)可鉴定为Phyllobacterium myrsinacearum。沙雷氏菌属未知种unclassified_g_ Serratia(OTU61)与未培养细菌序列KF632521聚为一类,序列一致度100%,其目前还不能鉴定到种;同理,肠杆菌科未知属未知种unclassified_norank_ Enterobacteriaceae(OTU124)不能鉴定到属。
真菌(图4-d)中,未分类真菌unclassified_Fungi(OTU7)与未培养真菌ITS序列KF221761聚为一类,序列一致度99%,其目前还不能进一步鉴定到门。节菌属未知种unclassified_Wallemia(OTU3、OTU4)分别与Wallemia sebi、Wallemia tropicalis一致度达99%,所以unclassified_Wallemia包括这2个种;酵母目未分类种unclassified_Saccharomycetales(OTU20)与嗜糖假丝酵母(Candida glucosophila)、汉逊德巴利酵母(Debaryomyces hansenii)聚在一起,但序列一致度不高(分别为93%、83%),故不能确定该酵母的种属。
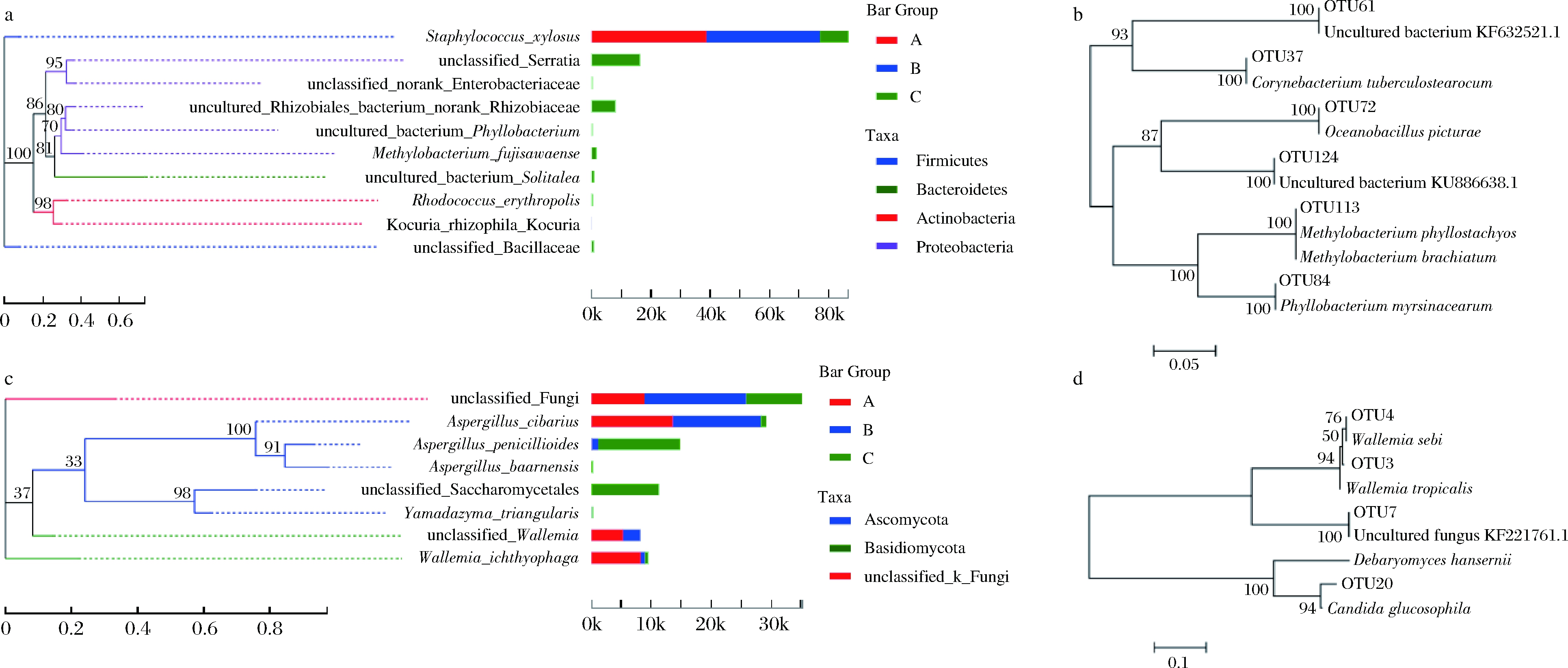
a-细菌种水平系统发生树及物种测序丰度图;b-细菌未分类OTU的进化分析;
c-真菌种水平系统发生树及物种测序丰度图;d-真菌未分类OTU的进化分析
图4 宣恩火腿主要微生物的进化分析
Fig.4 Phylogenetic analysis of major microorganisms of Xuanen ham
注:a~d中,树枝长度为进化距离,分支节点处显示1 000次重复检验的置信值(%);a和c中,进化树树枝颜色代表不同门,右边柱状图展示的是物种在不同样本中的测序数(reads),A、B、C依次为发酵前、中、后期的火腿样本
2.6 功能预测分析
通过PICRUSt对16S rRNA测序结果进行细菌群落功能的预测,获得OTU对应的COG家族相对丰度,结果见图5。忽略未知功能后,宣恩火腿表面细菌功能主要包括氨基酸转运及代谢(相对丰度11.2%~11.6%)、碳水化合物运输及代谢(7.3%~8.0%)、能量产生与转换(6.4%~6.6%)、无机离子转运及代谢(7.1%~7.5%)、脂类运输及代谢(3.4%~3.5%)等方面,这表明细菌群落对火腿蛋白质、脂肪的降解有重要作用。

图5 PICRUSt推断的细菌COG功能相对丰度
Fig.5 Relative abundance of inferred bacterial COG functions by PICRUSt
通过PICRUSt对ITS测序结果进行真菌群落的功能预测,获得真菌MetaCyc pathway丰度(表2)。根据功能预测的丰度值,真菌群落主要功能包括有氧呼吸、乙醛酸循环、脂肪酸β氧化、GDP甘露糖生物合成、三羧酸循环、鸟氨酸降解Ⅱ等途径。
表2 PICRUSt 推断的真菌MetaCyc pathway丰度(前10)
Table 2 Abundance of inferred MetaCyc pathways by PICRUSt
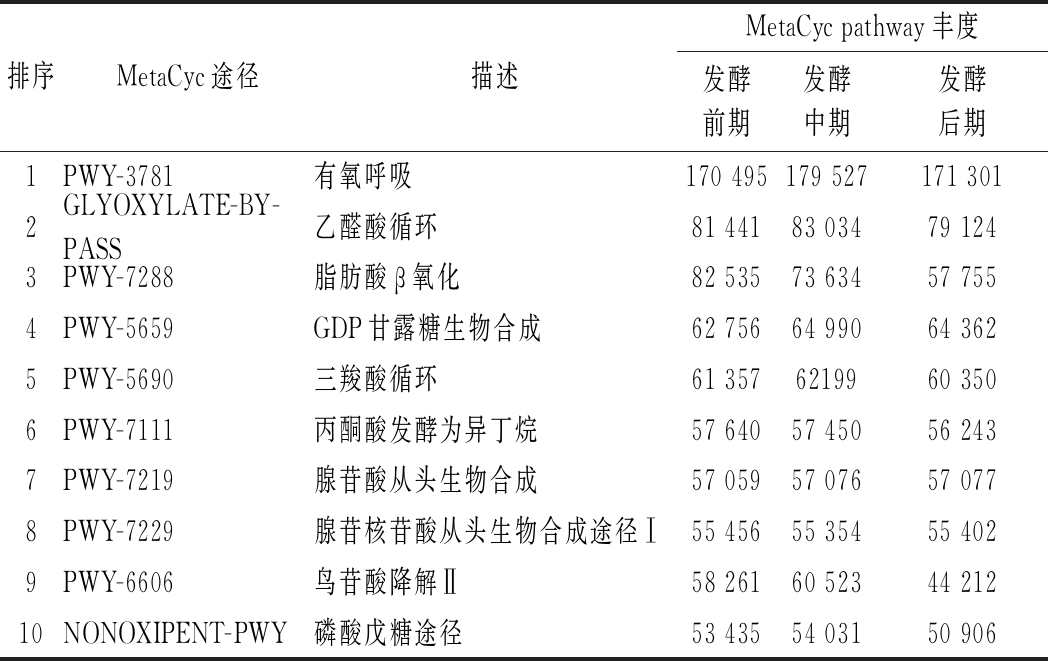
排序MetaCyc途径描述MetaCyc pathway丰度发酵前期发酵中期发酵后期1PWY-3781有氧呼吸170 495179 527171 3012GLYOXYLATE-BY-PASS乙醛酸循环81 44183 03479 1243PWY-7288脂肪酸β氧化82 53573 63457 7554PWY-5659GDP甘露糖生物合成62 75664 99064 3625PWY-5690三羧酸循环61 3576219960 3506PWY-7111丙酮酸发酵为异丁烷57 64057 45056 2437PWY-7219腺苷酸从头生物合成57 05957 07657 0778PWY-7229腺苷核苷酸从头生物合成途径Ⅰ55 45655 35455 4029PWY-6606鸟苷酸降解Ⅱ58 26160 52344 21210NONOXIPENT-PWY磷酸戊糖途径53 43554 03150 906
其中,乙醛酸循环和脂肪酸β氧化都与脂肪降解密切相关,脂肪酸β氧化分解为乙酰CoA之后,在乙醛酸体内生成琥珀酸、乙醛酸和苹果酸,从而进一步代谢;鸟氨酸降解Ⅱ与蛋白质降解途径相关。因此,宣恩火腿表面真菌群落对脂肪、蛋白质的降解有重要作用。
3 结论与讨论
发酵食品虽然种类繁多,但一般都具有各自相对稳定的微生物生态系统[23]。本文通过高通量测序技术研究了宣恩火腿发酵过程中表面细菌和真菌的多样性,揭示了群落演替规律。
实验中发现,宣恩火腿表面细菌主要为葡萄球菌属(Staphylococcus)、沙雷氏菌属(Serratia)、甲基菌属(Methylobacterium)(包括根瘤菌科未知属g_norank_f_Rhizobiaceae,其经进化分析也鉴定为甲基菌属)等;其中,木糖葡萄球菌(S.xylosus)在发酵前、中期占绝对优势(>98%),发酵后期下降到24.9%。有研究表明,金华火腿发酵中葡萄球菌是优势细菌[24-25];云腿主要优势细菌有表皮葡萄球菌(S.epidermidis)、肉葡萄球菌(S.carnosus)、木糖葡萄球菌(S.xylosus)等[26];木糖葡萄球菌能转换氨基酸,使得亮氨酸和其他游离氨基酸含量增加从而提升火腿风味[27],发酵后期细菌群落多样性的增加可能有利于火腿形成更加丰富的风味。
本实验发现宣恩火腿表面真菌主要为曲霉属(Aspergillus)、节菌属(Wallemia)、未分类真菌unclassified_Fungi(OTU7)和酵母;曲霉属在整个发酵过程中均有较高丰度,节菌属在发酵中丰度逐渐降低,而酵母仅在发酵后期占据优势。有研究表明,霉菌和酵母是众多火腿的优势真菌。宣威火腿表面和内部主要真菌为曲霉属、酵母和节菌属[7];云腿中的优势真菌是青霉属(Penicillium)、曲霉属(Aspergillus)和酵母[26];金华火腿发酵前期青霉属占优势,后期曲霉属占优势,酵母菌也发挥了很重要的作用[24];Istrian火腿中分离的真菌主要来自散囊菌属(Eurotium)、曲霉属(Aspergillus)和青霉属(Penicillium)[28]。
以上结果显示,不同产地火腿的微生物群落组成虽然存在差异,但在属水平上具有很高的一致性。这些微生物会分泌蛋白酶、脂肪酶等,促进蛋白质、脂肪水解产生游离氨基酸、脂肪酸,同时代谢产生各种特殊风味成分和保护火腿颜色等[29]。本文通过功能预测也发现,细菌和真菌均对宣恩火腿中蛋白质、脂肪的降解有重要作用。
通过高通量测序研究微生物多样性时,常常会出现因物种数据库序列信息不足而导致物种不能准确注释的情况。本研究中,未分类真菌unclassified_Fungi(OTU7)无法准确注释到门,进一步的进化分析显示其与未培养真菌ITS序列的一致度为99%。随着时间的推移,物种注释的相关数据库会不断更新,这种问题应该会逐渐解决。后续试验中也可尝试利用各种培养基对unclassified_Fungi(OTU7)进行分离培养和进一步鉴定。
本文对宣恩火腿发酵过程中表面细菌和真菌多样性进行了研究,对细菌、真菌优势种之间的相关性进行了分析,有利于了解主要微生物种类及共存关系,可为后续研发宣恩火腿发酵剂和开展人工接种生产火腿作参考,以便进一步改善火腿品质、缩短加工周期和提升安全性。本文没有研究火腿内部微生物,虽然内部微生物会受到表面微生物的抑制,但其也发挥了重要作用[24],后续可以开展相关工作。
[1] 崔莹莹,何乐,王海滨,等.湖北宣恩火腿基本成分分析[J].肉类研究,2015,29(5):6-9.
CUI Y Y,HE L,WANG H B,et al.Proximate composition analysis of Xuanen ham from Hubei province[J].Meat Research,2015,29(5):6-9.
[2] 范露, 冯牛,许嘉验,等.宣恩火腿蛋白质降解规律[J].食品工业科技,2019,40(23):42-46;53.
FAN L,FENG N,XU J Y,et al.Degradation law of protein of Xuanen ham[J].Science and Technology of Food Industry,2019,40(23):42-46;53.
[3] 邱朝坤, 范露,张倩倩.宣恩火腿加工过程中脂肪变化分析[J].中国调味品,2019,44(5):111-114.
QIU C K,FAN L,ZHANG Q Q.Analysis of fat changes during the processing of Xuan’en ham[J].China Condiment,2019,44(5):111-114.
[4] 耿翠竹, 季鑫,王海滨,等.宣恩火腿加工过程中理化指标变化的分析[J].肉类研究,2017,31(2):11-15.
GENG C Z,JI X,WANG H B,et al.Analysis of physicochemical changes during Xuan’en ham processing[J].Meat Research,2017,31(2):11-15.
[5] 陈存坤, 高芙蓉,冯叙桥,等.杏贮藏过程中主要致病菌的分离与鉴定[J].保鲜与加工,2014,14(6):30-33.
CHEN C K,GAO F R,FENG X Q,et al.Isolation and identification of the main pathogenic fungus from apricot during storage[J].Storage and Process,2014,14(6):30-33.
[6] 张婷, 孔云,修伟明,等.施肥措施对华北潮土区小麦-玉米轮作体系土壤微生物群落特征的影响[J].生态环境学报,2019,28(6):1 159-1 167.
ZHANG T,KONG Y,XIU W M,et al.Effects of fertilization treatments on soil microbial community characteristics under the wheat-maize rotation system in fluvo-aquic soil region in North China[J].Ecology and Environmental Sciences,2019,28(6):1 159-1 167.
[7] 邹颖玲,刘姝韵,王桂瑛,等.基于PCR-DGGE技术分析宣威火腿中真菌群落结构[J].食品与发酵工业,2020,46(6):269-274.
ZOU Y L,LIU S Y,WANG G Y,et al.Analysis of fungal community structure in Xuanwei ham by PCR-DGGE[J].Food and Fermentation Industries,2020,46(6):269-274.
[8] 耿道强, 产久林,潘彬彬,等.三种深渊钩虾肠道微生物组成与群落结构[J].上海海洋大学学报,2020,29(1):83-91.
GENG D Q,CHAN J L,PAN B B,et al.Gut microbiome composition and community structure of three hadal amphipod species[J].Journal of Shanghai Ocean University,2020,29(1):83-91.
[9] ZHANG T,WANG N F,ZHANG Y Q,et al.Diversity and distribution of fungal communities in the marine sediments of Kongsfjorden,Svalbard (High Arctic)[J].Scientific Reports,2015,5:14 524.
[10] ROGERS M B,FIREK B,SHI Min et al.Disruption of the microbiota across multiple body sites in critically ill children[J].Microbiome,2016,4:66.
[11] 马冰涛, 范恩帝,李泽霞,等.老白干香型白酒制曲过程中微生物多样性及其与风味成分的关系[J].食品与发酵工业,2020,46(16):7-16.
MA B T,FAN E D,LI Z X,et al.Microbial diversity and its relationship with flavor compounds in the process of Daqu making of Laobaigan-flavor Baijiu[J].Food and Fermentation Industries,2020,46(16):7-16.
[12] 韩齐, 李媛媛,孙方达,等.新一代测序技术在食品微生物学中的应用[J].食品工业,2016,37(1):278-283.
HAN Q,LI Y Y,SUN F D,et al.Application of next generation sequencing techniques in food microbiology[J].The Food Industry,2016,37(1):278-283.
[13] 温崇庆,何瑶瑶,薛明,等.高通量测序分析DNA提取引起的对虾肠道菌群结构偏差[J].微生物学报,2016,56(1):130-142.
WEN C Q,HE Y Y,XUE M,et al.Biases on community structure during DNA extraction of shrimp intestinal microbiota revealed by high-throughput sequencing[J].Acta Microbiologica Sinica,2016,56(1):130-142.
[14] 余红英, 余瑞明.宣恩火腿的加工工艺[J].食品研究与开发,1997,18(3):28-29.
YU H Y,YU R M.Processing technology of Xuan’en ham[J].Food Research and Development,1997,18(3):28-29.
[15] 竺尚武. 干腌火腿中致病微生物和寄生虫的研究进展[J].食品与发酵工业,2006,32(5):101-104.
ZHU S W.Progresses in the research of pathogenic microorganisms and parasites in dry-cured ham[J].Food and Fermentation Industries,2006,32(5):101-104.
[16] 洪洁,康建依,刘一倩,等.生菜连作及生菜-菠菜轮作对土壤细菌群落结构的影响[J].生物技术通报,2019,35(8):17-26.
HONG J,KANG J Y,LIU Y Q,et al.Effects of continuous cropping of lettuce and rotation of lettuce-spinach on soil bacterial community structure[J].Biotechnology Bulletin,2019,35(8):17-26.
[17] CHEN B S,TEH B S,SUN C,et al.Biodiversity and activity of the gut microbiota across the life history of the insect herbivore Spodoptera littoralis[J].Scientific Reports,2016,6:29 505.
[18] SONNENBURG E D,SMITS S A,TIKHONOV M,et al.Diet-induced extinctions in the gut microbiota compound over generations[J].Nature,2016,529(7 585):212-215.
[19] YANG B,XIAO L,LIU S,et al.Exploration of the effect of probiotics supplementation on intestinal microbiota of food allergic mice[J].American Journal of Translational Research,2017,9(2):376-385.
[20] 马岩石, 姜明,李慧,等.基于高通量测序技术分析东北豆酱的微生物多样性[J].食品工业科技,2020,41(12):100-105.
MA Y S,JIANG M,LI H,et al.Analysis of microbial diversity of northeast soy sauce based on high-throughput sequencing technology[J].Science and Technology of Food Industry,2020,41(12):100-105.
[21] SUI Q W,LIU C,ZHANG J Y,et al.Response of nitrite accumulation and microbial community to free ammonia and dissolved oxygen treatment of high ammonium wastewater[J].Applied Microbiology and Biotechnology,2016,100(9):4 177-4 187.
[22] RAMAYO-CALDAS Y,MACH N,LEPAGE P,et al. Phylogenetic network analysis applied to pig gut microbiota identifies an ecosystem structure linked with growth traits[J].The ISME Journal,2016,10:2 973-2 977.
[23] MARCO M L,HEENEY D,BINDA S,et al.Health benefits of fermented foods:Microbiota and beyond[J].Current Opinion in Biotechnology,2017,44:94-102.
[24] 贺稚非, 甄宗圆,李洪军,等.金华火腿发酵过程中微生物区系研究[J].食品科学,2008,29(1):190-195.
HE Z F,ZHEN Z Y,LI H J,et al.Microorganisms flora study on Jinhua ham fermentation[J].Food Science,2008,29(1):190-195.
[25] GE Q F,GU Y B,ZHANG W G,et al.Comparison of microbial communities from different Jinhua ham factories[J].AMB Express,2017,7:37.
[26] 黄艾祥. 云南干腌火腿品质特征形成与微生物作用研究[D].重庆:西南大学,2006.
HUANG A X.Study on the formation of quality traits and microbial roles of Yunnan dry cured ham[D].Chongqing:Southwest University,2006.
[27] BECK H,HANSEN A,LAURITSEN F.Catabolism of leucine to branched-chain fatty acids in Staphylococcus xylosus[J].Journal of Applied Microbiology,2004,96(5):1 185-1 193.
[28] COMI G,ORLIC S,REDZEPOVIC S,et al.Moulds isolated from istrian dried ham at the pre-ripening and ripening level[J].International Journal of Food Microbiology,2004,96(1):29-34.
[29] 黄盼盼, 蒋先芝,田建卿.火腿微生物研究进展[J].生物工程学报,2018,34(9):1 410-1 418.
HUANG P P,JIANG X Z,TIAN J Q.Research advances in ham microorganisms[J].Chinese Journal of Biotechnology,2018,34(9):1 410-1 418.