细菌耐药性的威胁日益严重,寻找新型抗菌药物已迫在眉睫[1]。长期以来,放线菌次级代谢产物一直是微生物药物的重要来源[2]。但随着抗生素开发的“黄金时代”远去,从陆生或土壤微生物中难以寻找到新的抗菌活性物质[3]。于是人们转向研究海洋[4-5]、荒漠[6]或极端环境微生物[7],它们长期生长于特殊环境下,形成了较为独特的生理代谢途径,具有产生多种活性代谢产物的潜力。
我们从香港米埔湿地红树林植物的根际沉积物中,分离到1株放线菌XY-R10,初步鉴定为拟孢囊菌属[8]的Kibdelosporangium phytohabitans(K.phytohabitans),为1株稀有放线菌。该菌公开的基因组大小为11.76 Mb[9],使用antiSMASH 5.0对基因组注释分析[10],预测有49个生物合成基因簇,说明其具有产生多种活性次级代谢产物的潜能。目前已从拟孢囊菌属的多株菌内发现了具有抗菌活性的化合物,如Aridicins[11]、Kibdelins[12]、Azicemicins[13]和Kibdelomycins[14]等,而对K.phytohabitans次级代谢产物的研究尚未见报道。我们对菌株XY-R10发酵粗提物的抑菌谱和稳定性进行了研究,结合液相-串联高分辨质谱(liquid chromatography-tandem high-resolution mass spectrometry,LC-HRMS)分析粗提物中的成分,以期为该菌具有抑菌活性次级代谢产物的挖掘和开发提供研究基础。
1 材料与方法
1.1 材料与试剂
1.1.1 样品采集
红树林植物秋茄[Kandelia candel (L.) Druce],中国香港,米埔内后海湾拉姆萨尔国际重要湿地(E 114.05°,22.49°);以无菌操作法,取根际3~5 cm深的沉积物,装入无菌密封袋中,带回实验室在24 h内完成处理。
1.1.2 抗菌活性测试用菌株
金黄色葡萄球菌(methicillin sensitive Staphylococcus aureus,MSSA)ATCC 25923、耐甲氧西林金黄色葡萄球菌(methicillin-resistant Staphylococcus aureus,MRSA)ATCC 43300、粪肠球菌(Enterococcus faecalis)ATCC 29212、屎肠球菌(Enterococcus faecium)ATCC 35667、枯草芽孢杆菌(Bacillus subtilis)ATCC 6051、表皮葡萄球菌(Staphylococcus epidermidis)ATCC 12228、无乳链球菌(Streptococcus agalactiae)ATCC 13813、单增李斯特菌(Listeria monocytogenes)ATCC 19115、铜绿假单胞菌(Pseudomonas aeruginosa) PAO1、大肠杆菌(Escherichia coli)O157:H7、肠炎沙门氏菌(Salmonella enteritidis)CMCC 50335、副溶血性弧菌(Vibrio parahemolyticus)ATCC 17802,均为本实验室保存。
1.1.3 培养基
TSB、MH、BHI肉汤,广东环凯微生物科技有限公司,肉汤中添加1.8%~2%(质量分数)琼脂为固体培养基;R2A、ISP-2培养基,美国BD DifcoTM公司。
斜面孢子培养基:ISP-2培养基。
种子培养基(g/L):葡萄糖10,玉米淀粉10,黄豆饼粉10,硫酸铵2,玉米浆干粉5,去离子水1 L,pH(7.2±0.1)。
发酵培养基(g/L):可溶性淀粉20,甘油20,Peptone 20,人工海盐20,碳酸钙5(调整pH后加入),去离子水1 L,pH (7.5±0.1)。
1.1.4 主要试剂
Peptone、Yeast Extract,英国OXIOD公司;人工海盐,广州益尔生物工程有限公司;Ezup 柱式细菌基因组 DNA 抽提试剂盒(B518255)、SanPrep 柱式 DNA 胶回收试剂盒(B518131),生工生物工程(上海)股份有限公司;Collismycin A、Maipomycin B和Collismycin X,深圳金普迈生物科技有限公司;C18硅胶填料(50 μm,120 Å),苏州纳微科技股份有限公司;其他化学试剂,北京索莱宝科技有限公司。
1.2 仪器与设备
LC-20AT高效液相色谱仪(DAD检测器),日本岛津公司;Agilent 1260/6545 Q-TOF液质(LC-MS)联用仪,安捷伦科技有限公司;Waters 2695-2996制备液相色谱仪,沃特世科技有限公司;Hei-VAP旋转蒸发仪,德国海道尔夫公司;LRH-70恒温培养箱、THZ-100恒温摇床,上海一恒科学仪器有限公司;1300生物安全柜,赛默飞世尔科技有限公司;SMZ-168体视显微镜,麦克奥迪实业集团有限公司。
1.3 实验方法
1.3.1 放线菌的分离
将沉积物样品浸泡在无菌海水中配制成混悬液,用梯度稀释法将混悬液稀释后,吸取10-2~10-6梯度的稀释液100 μL均匀涂布于R2A培养基固体平板上,平板置于28 ℃恒温培养箱培养4~8 d。对平板上长出的菌落进一步划线分离纯化,获得纯培养物,进行分子生物学鉴定,编号并保藏。
1.3.2 菌株16S rRNA序列的测定和构建系统发育树
DNA抽提试剂盒提取菌株XY-R10的基因组DNA,用通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-TACGGCTACCTTGTTACGACT-T-3′)进行PCR扩增,反应体系为:模板DNA 2 μL,Taq DNA 聚合酶0.2 μL,10×Buffer(含Mg2+)2.5 μL,dNTP 4 μL,27F 1 μL,1492R 1 μL,加双蒸水至25 μL。PCR循环条件:94 ℃预变性5 min;94 ℃ 45 s,55 ℃ 45 s,72 ℃ 90 s,共30个循环;72 ℃再延伸10 min。1%(质量分数)琼脂糖电泳检测PCR产物,切胶回收后送至生工生物工程(上海)股份有限公司完成测序。将测序结果上传到EzBioCloud网站进行比对[15],下载相似性最高的前10株菌的16S rDNA序列。用MEGA 7.0对这些序列进行比对,Phylogeny中选择邻接法构建发育进化树。
1.3.3 菌株形态观察
将菌株XY-R10在ISP-2培养基平板上划线,另取灭菌盖玻片斜插入培养基内,平板在28 ℃培养7~8 d后,观察记录菌落形态,并取出长有菌丝的盖玻片,用结晶紫染色,油镜(1 000×)下观察菌丝形态。
1.3.4 放线菌发酵和产物粗提取
将XY-R10接种于斜面孢子培养基(ISP-2培养基),28 ℃培养7~8 d后接种到种子培养基摇瓶中,在28 ℃、225 r/min条件下培养42~48 h。种子液按5%接种量接种至发酵摇瓶,在28 ℃、225 r/min条件下培养6 d。将发酵液在8 000 r/min条件下离心10 min 取上清液,上清液用等体积的乙酸乙酯萃取3次,合并萃取相减压浓缩得发酵粗提物,二甲基亚砜溶解备用。
1.3.5 抑菌活性检测方法
(1)纸片法测定抑菌活性:参照美国临床和实验室标准协会(Clinical and Laboratory Standards Institute,CLSI)的药敏试验执行标准M02-A11进行。
(2)微量稀释法测定最小抑菌浓度(minimum inhibitory concentration,MIC):参照CLSI的药敏试验执行标准M07-A9和M45-A,用96微孔板测定。
1.3.6 抑菌活性物质的酸碱稳定性和热稳定性考察[16]
取发酵粗提物溶解于水中,分别用0.1 mol/L的NaOH溶液和0.1 mol/L的HCl调节pH值至3、5、7、9、11,未调节pH值的粗提物水溶液为阳性对照,未调节pH值的水、pH 3和pH 11的水为阴性对照,MRSA为活性指示菌,纸片法检测调完pH值后粗提物的抑菌活性,考察不同pH处理对粗提物抑菌活性的影响。另取调节为不同pH值的粗提物溶液,在60 ℃ 水浴处理3 h后检测抑菌活性,考察不同pH处理下粗提物抑菌活性的温度稳定性。
1.3.7 LC-MS分析条件
色谱条件:色谱柱ACE Excel 5 C18-HL,250 mm×4.6 mm;流动相A:乙腈;流动相B:V(10 mmol/L甲酸铵)∶V(乙腈)=9∶1;检测波长200~600 nm;流量1 mL/min;柱温35 ℃。梯度洗脱:0~20 min,A(0~40%);20~35 min,A(40%~80%);35~40 min,A(80%);40.01~45 min,A(0)。
质谱条件:Dual AJS ESI,Auto MS(2) Mode;Gas Temp:325 ℃;Drying gas:10 L/min;Nebulizer:40 psi;Sheath Gas Temp:350 ℃;Sheath Gas Flow:12 L/min;VCap:4 000 V;Nozzle Voltage:500 V。
1.3.8 抑菌活性物质初步分离
取发酵粗提物上样于C18硅胶柱(径高比6∶1),依次用10%~100%(体积分数)甲醇水溶液洗脱,分部分收集洗脱液。各部分洗脱液经旋转蒸发仪浓缩后冷冻干燥为干粉,检测各分离段的MIC值。
将40%甲醇洗脱的部分用制备液相色谱仪制备Maipomycin A,色谱条件:Phenomenex Kinetex XB-C18色谱柱(250 mm×21.2 mm, 5 μm),V(乙腈)∶V(水)=25∶75,流量为4 mL/min。
2 结果与分析
2.1 菌株XY-R10的形态特征
菌株XY-R10在ISP-2培养基上生长良好(图1-a),菌落呈圆形凸起,无光泽,表面干燥、有褶皱,贴紧培养基生长,不易挑起。气生菌丝丰富,为白色或灰白色,体视镜下能观察到致密、绒状的菌丝,具有一般放线菌的特征(图1-b)。菌落周围产生黑褐色可溶性色素,扩散在琼脂中。将其气生菌丝用结晶紫染色后在放大1 000倍的油镜下观察(图1-c),能看到细长、不规则弯曲的菌丝,菌丝形成长、直或弯曲的圆柱形孢子链,另外在菌丝中有许多球形、笼状的结构。
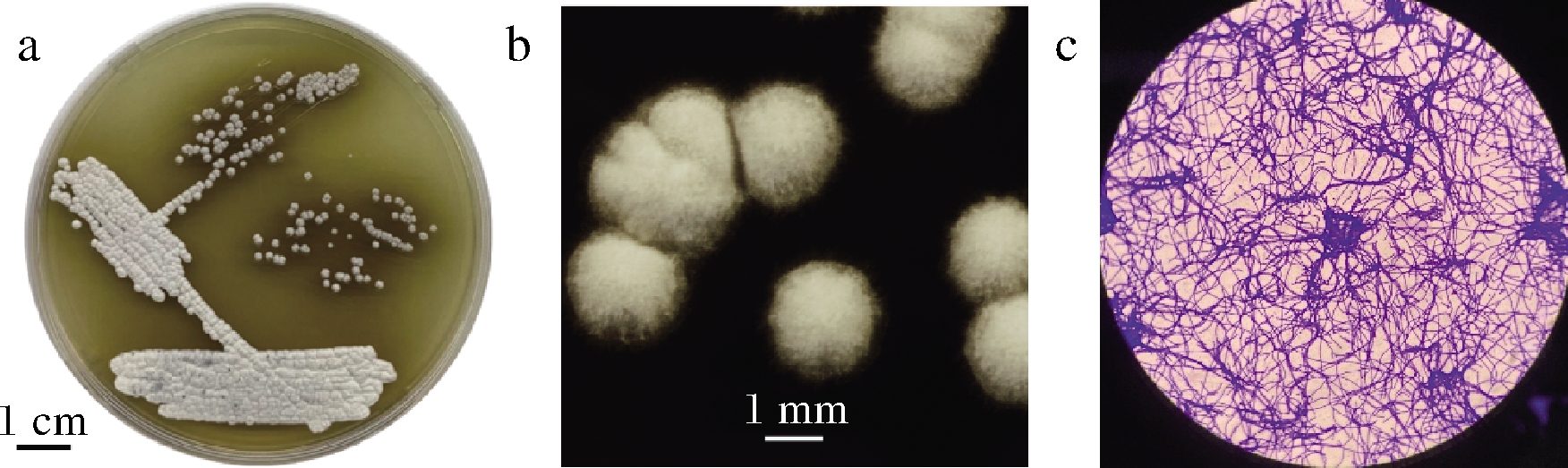
a-菌株XY-R10在ISP-2平板的生长形态;b-体视镜观察菌株XY-R10 的菌落形态;c-油镜观察菌株XY-R10的气生菌丝形态(1 000倍)
图1 菌株XY-R10的形态特征分析
Fig.1 Analysis of morphological characteristics of the strain XY-R10
2.2 菌株XY-R10的系统发育分析
将菌株XY-R10进行16S rRNA测序,序列信息上传至EzBioCloud网站比对,下载相似性较高的序列,用MEGA 7.0采用邻接法构建进化树。结果显示(图2),菌株XY-R10隶属拟孢囊菌属,与Kibdelosporangium phytohabitans KLBMP 1111T同源,相似度达99.79%。结合该菌的形态特征与文献报道相比较,放线菌XY-R10为很可能与Kibdelosporangium phytohabitans KLBMP 1111T为同1个种,是1株稀有放线菌。

图2 基于菌株XY-R10的16S rRNA基因序列构建的系统发育树
Fig.2 Phylogenetic tree based on 16S rRNA gene sequence of the strain XY-R10
2.3 发酵粗提物抑菌活性的研究
菌株XY-R10发酵第6天的上清液对MRSA有较好的抑菌活性,如图3-a所示,载样量为10 μL上清液纸片的抑菌圈直径(约17 mm),略高于载药量10 μg万古霉素纸片的抑菌圈直径(约15 mm)。考察了不同发酵时间发酵液的抑菌活性,抑菌活性物质仅在发酵第6~8天出现(图3-b)。
将发酵液用乙酸乙酯萃取制备粗提物,以多株临床及食源性致病菌为指示菌,测定粗提物的抗菌谱。结果如表1所示,粗提物对MSSA、MRSA、屎肠球菌、枯草芽孢杆菌、表皮葡萄球菌和无乳链球菌等革兰氏阳性菌的MIC≤8 μg/mL,表现出较好的抑菌活性;对粪肠球菌和单增李斯特菌的MIC分别为128、256 μg/mL,抑菌活性微弱。粗提物对铜绿假单胞菌、大肠杆菌和肠炎沙门氏菌等革兰氏阴性菌的MIC>512 μg/mL,无抑菌活性;对副溶血弧菌有微弱的抑菌作用(MIC=256 μg/mL)。因此,该粗提物对革兰氏阳性菌有较广谱的抑菌活性。
2.4 粗提物中抑菌活性物质的稳定性
对粗提物抑菌活性成分的稳定性考察结果见表2。室温条件下,粗提物水溶液pH值调节为3、5、7时,抑菌圈大小与对照(未调节pH,原始pH值为6.6左右)基本一致,说明抑菌活性物质在中性和弱酸性条件下较稳定。当pH值提高至9和11时,抑菌圈变小,说明碱处理使抑菌活性物质不稳定因而活性降低。
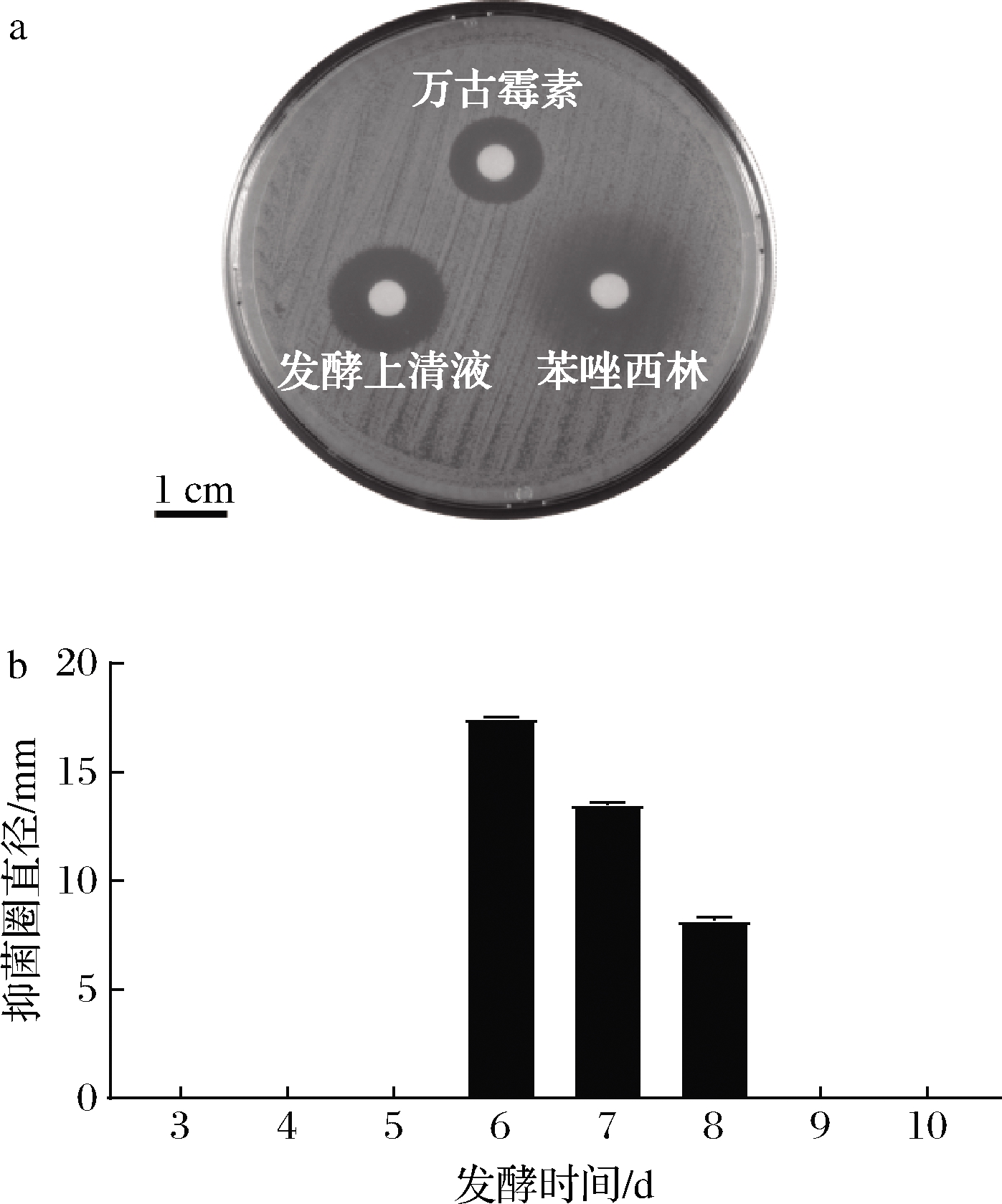
a-发酵7 d的上清液对MRSA的抑菌活性;b-发酵上清液抑菌 活性随发酵时间的变化
图3 发酵液上清抑菌活性的发现
Fig.3 Antibacterial activity of fermentation broth
注:万古霉素10 μg,苯唑西林10 μg,发酵上清液10 μL
表1 发酵粗提物MIC的测定 单位:μg/mL
Table 1 MIC of fermentation extracts

测试菌株发酵粗提物ClsVan革兰氏阳性菌金黄色葡萄球菌 (MSSA)4-1金黄色葡萄球菌 (MRSA)8-2粪肠球菌128-2屎肠球菌8-1枯草芽孢杆菌4-0.5表皮葡萄球菌4-2无乳链球菌4-0.5单增李斯特菌256-4革兰氏阴性菌铜绿假单胞菌>5121-大肠杆菌>5122-肠炎沙门氏菌>5122-副溶血弧菌2562-
注:“-”代表未进行测试;“Cls”为多黏菌素E,“Van”为万古霉素(表4同)
研究了酸碱和加热同时处理对抑菌活性物质的影响。粗提物水溶液在pH值为3时,经60 ℃热处理后抑菌圈略微减小,活性略微降低;而pH越高,热处理后粗提物抑菌活性越差甚至失去活性。因此,粗提物中的抑菌活性物质在弱酸性环境下稳定性较好,不耐受碱性环境,热稳定性较差。
表2 pH和温度对抑菌活性物质稳定性的影响
Table 2 Effects of pH and temperature on the stability of antibacterial active substances
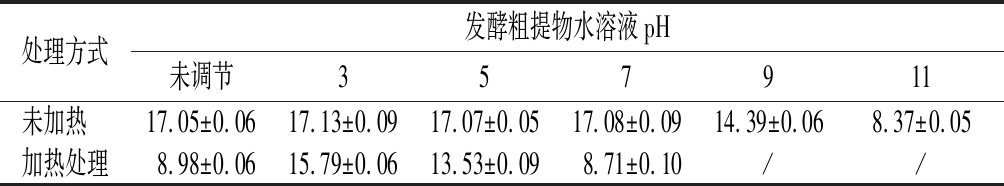
处理方式发酵粗提物水溶液pH未调节357911未加热 17.05±0.0617.13±0.0917.07±0.0517.08±0.0914.39±0.068.37±0.05加热处理8.98±0.0615.79±0.0613.53±0.098.71±0.10//
注:“/”代表无抑菌圈
2.5 粗提物分段分离后各部分的抑菌活性
使用C18柱对粗提物进行分段分离,并检测各分离段的抑菌活性,结果如表3所示。用20%~60%甲醇洗脱的各分离段对MRSA具有不同程度抑菌活性,但它们的MIC值均高于粗提物、抑菌活性减弱,在MSSA、粪肠球菌、屎肠球菌、表皮葡萄球菌、枯草芽孢杆菌、无乳链球菌中也有类似的结果。使用HPLC分析,各分离段的峰各不相同。推测粗提物中可能存在多个活性成分,粗提物广谱抗革兰氏阳性菌的作用可能为多个成分联合的效果,因此经分离后,各分离段的单独作用的抑菌活性减弱。70%~100%甲醇洗脱的各分离段对所有菌株无抑菌活性(MIC>512 μg/mL);各分离段对铜绿假单胞菌、大肠杆菌和肠炎沙门氏菌均无抑菌活性(MIC>512 μg/mL)。
值得注意的是,与粗提物相比,40%甲醇洗脱的分离段对单增李斯特菌和副溶血弧菌的MIC值降低,抑菌活性有所增强,我们首先对这一分离段的成分进行研究。
表3 粗提物经初步分离后各分离段MIC的测定 单位:μg/mL
Table 3 MIC of fractions of extracts after separated

测试菌株粗提物甲醇的洗脱液体积分数/%102030405060革兰氏阳性菌金黄色葡萄球菌(MRSA)8> 5121286412864256单增李斯特菌256>512>512>51264256>512革兰氏阴性菌副溶血弧菌256>512>512>512128>512>512
2.6 LC-HRMS分析发酵粗提物成分
由图4分析可知,粗提物中有超过9个较为明显的组分,其中峰5~峰9来源于培养基。峰1和峰2的一级质谱[M+H]+m/z分别为308.075 9和308.071 0,经Mass Hunter定性分析软件计算分子式均为C13H13N3O4S,二者有一些相同的二级质谱碎片m/z 290.05、m/z 210.06、m/z 181.06等,但紫外吸收峰略有不同,推测二者可能为同分异构体。峰1为Maipomycin A,是本实验室之前从菌株XY-R10筛选到的具有强效杀藻活性的化合物(图4-h,原命名为Kibdelomycin A,后发现与已报道化合物重名,现改为Maipomycin A)[17-18]。结合HPLC-DAD检测粗提物用40%甲醇分离的部分,该部分的主要成分是Maipomycin A,因此下一步对Maipomycin A的抑菌活性进行研究。
峰3在正离子模式下无质谱响应,负离子模式的一级质谱[M-H]-m/z为394.163 4,经计算分子式为C23H25NO5。峰3紫外吸收峰在240、268、299、336 nm,与Azicemicin A(图4-i,分子式C23H25NO9)有类似的紫外吸收(234、277、322、335 nm)[13]。Azicemicin A分离自拟孢囊菌属的另一株菌Kibdelosporangium sp.MJ126-NF4[19],具有抗金黄色葡萄球菌活性(MIC ≥ 100 μg/mL)[13],而峰3的分子式比Azicemicin A少4个氧原子,推测峰3可能为Azicemicins类化合物。
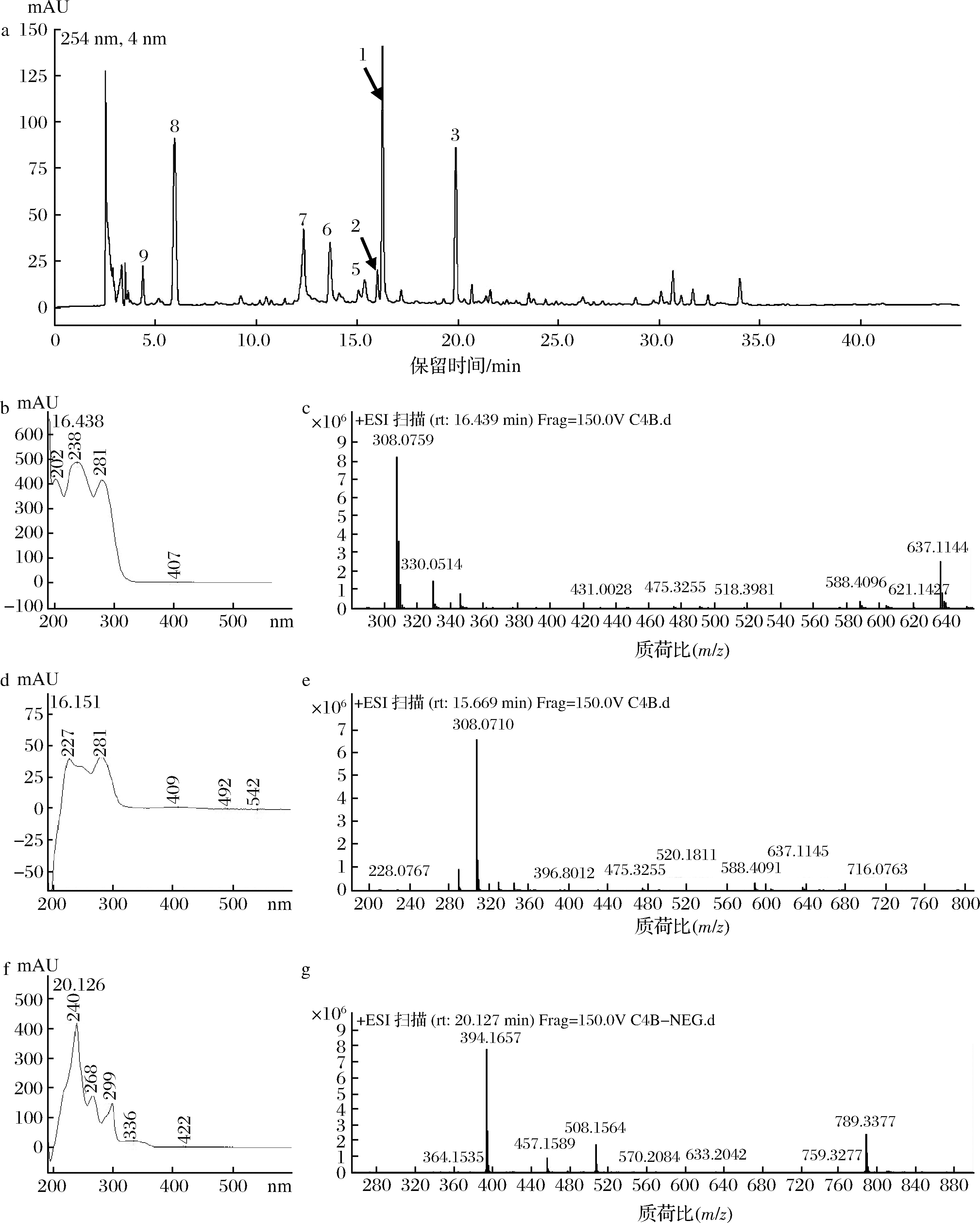
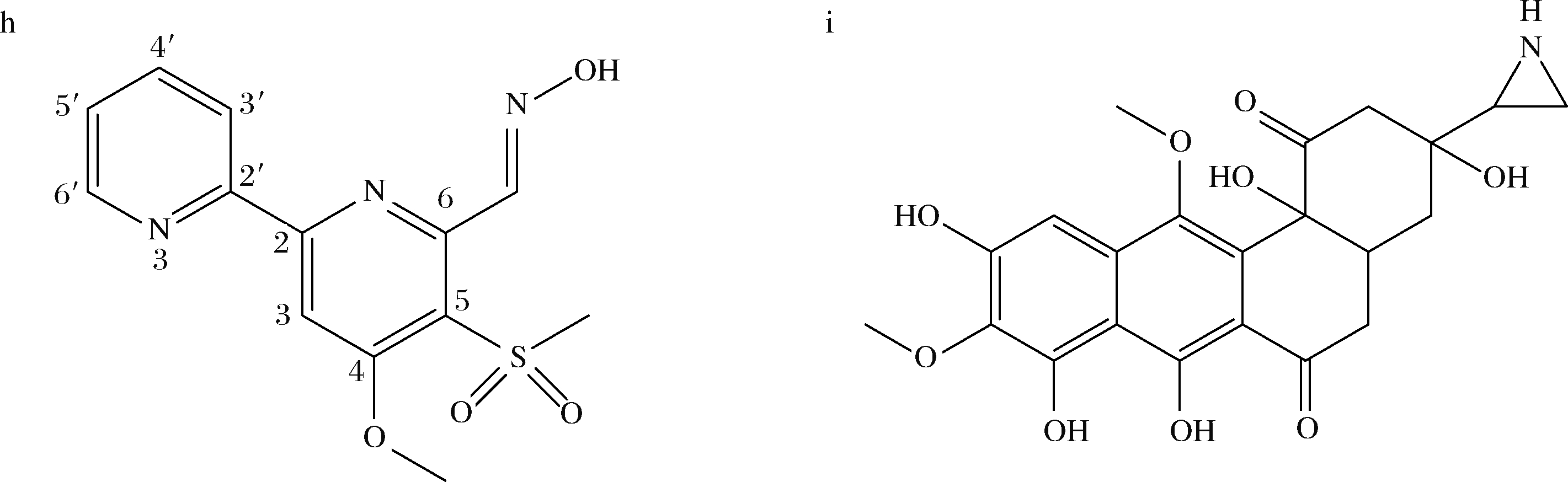
a-发酵粗提物HPLC图;b-峰1紫外吸收图;c-峰1一级质谱图;d-峰2紫外吸收图;e-峰2一级质谱图;f-峰3紫外吸收图; g-峰3一级质谱图;h-Maipomycin A结构;i-Azicemicin A结构
图4 LC-HRMS分析发酵粗提物成分
Fig.4 LC-HRMS analysis the fermentation extracts
2.7 Maipomycin A抑菌活性的研究
Maipomycin A具有2,2′-联吡啶的结构,对大多数革兰氏阳性菌和革兰氏阴性菌无抑菌活性,仅对表皮葡萄球菌、单增李斯特菌和副溶血弧菌有抑菌作用,MIC分别为32、16、32 μg/mL(表4)。含有2,2′-联吡啶结构的天然产物还有Caerulomycins[20]、Collismycins[21]和Pyrisulfoxins[22]等,这类化合物具有抗菌、细胞毒活性和免疫调节作用[23],我们比较了Maipomycin A的3个结构类似物(图5)的抑菌活性。如表4所示,Collismycin A同样对表皮葡萄球菌、单增李斯特菌和副溶血弧菌有抑菌作用,活性与Maipomycin A相当,但Maipomycin B和Collismycin X的抑菌活性就相对较弱。从结构上看,Maipomycin A和Maipomycin B仅在6位的基团有差异(图5),前者是醛肟基,而后者是醛基,Collismycin A和Collismycin X结构上的差异也是如此。因此推测Maipomycin A和Collismycin A的抑菌活性很可能与6位的醛肟基有关,这与文献报道的结论基本一致[25]。
表4 Maipomycin A及其类似物MIC的测定 单位:μg/mL
Table 4 MIC of Maipomycin A and its analogues

测试菌株Maipomycin ACollismycin AMaipomycin BCollismycin XClsVan革兰氏阳性菌表皮葡萄球菌3232128128-2单增李斯特菌16166464-4革兰氏阴性菌副溶血弧菌32322561282-
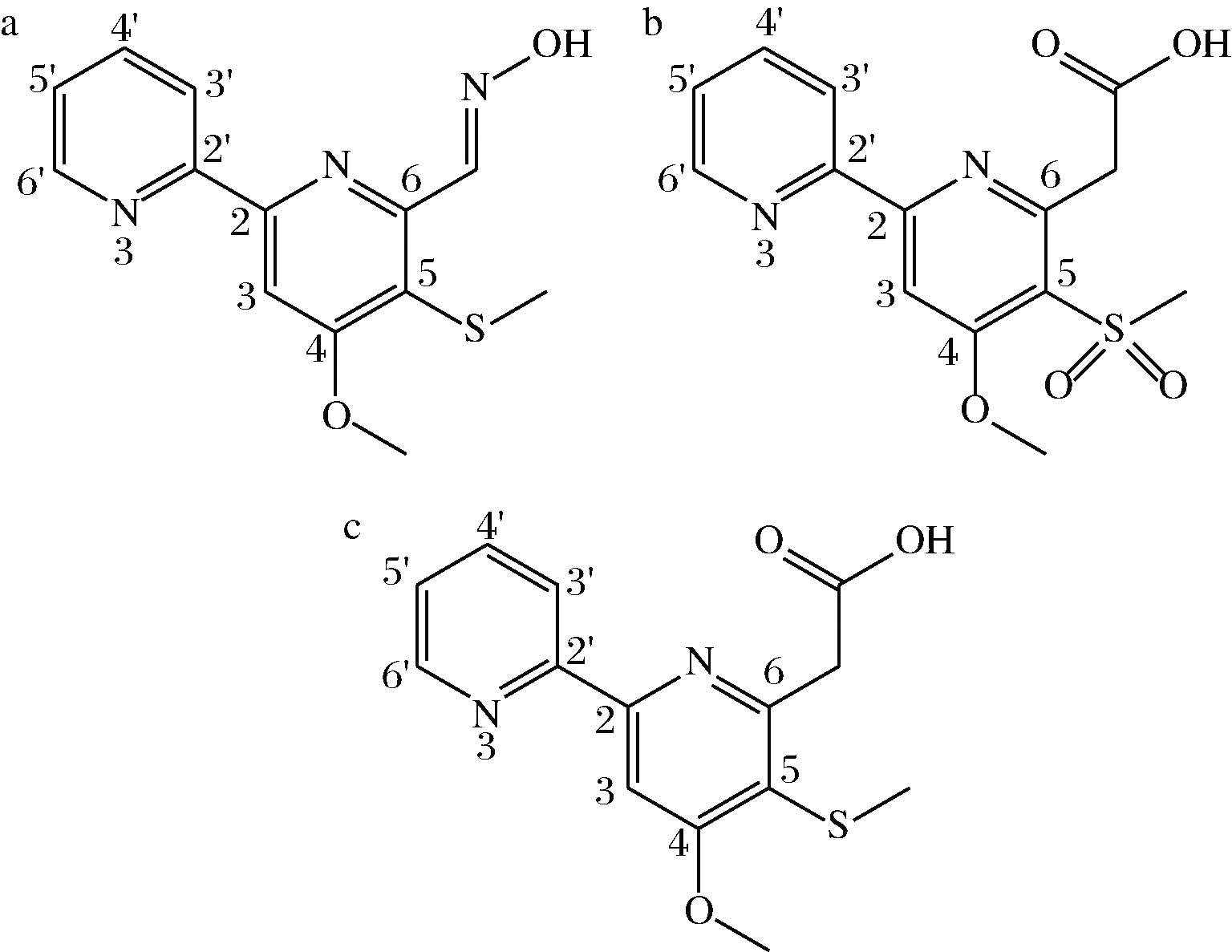
a-Collismycin A结构;b-Maipomycin B结构;c-Collismycin X结构
图5 Maipomycin A类似物结构式
Fig.5 Structures of Maipomycin A analogues
3 结论
红树林潮间带是一种特殊的生态系统,孕育着丰富而新颖的微生物资源,是研究微生物天然产物的热点之一[24]。本实验室长期从事海洋和红树林来源微生物的分离及活性次级代谢产物的研究,菌株XY-R10即为红树林植物根际沉积物中分离得到的其中1株放线菌,本文对该菌进行了鉴定和抑菌活性的研究。经菌落特征识别、显微镜观察,测序比对和构建进化树分析,初步鉴定为拟孢囊菌属的K.phytohabitans,是1株稀有放线菌。
放线菌XY-R10的发酵粗提物对多种常见的临床和食源性致病菌有较好抑菌作用,具有广谱的抗革兰氏阳性菌活性。抑菌活性物质在弱酸性条件下较稳定,耐热性差。粗提物经分段分离后,各分离段的抑菌活性均减弱,推测粗提物中可能含有多个活性成分,其广谱抗革兰氏阳性菌的的作用可能为多个成分联合的效果。利用LC-HRMS鉴定出粗提物中一个成分为Maipomycin A,此外还可能存在Maipomycin A和Azicemicin A的类似物。Maipomycin A对表皮葡萄球菌、单增李斯特菌和副溶血弧菌有抑菌作用,其抑菌作用可能与6位的醛肟基相关,可为后续作用机制探索、构效关系研究提供参考。
综上,本文对红树林来源稀有放线菌XY-R10进行了分离和鉴定,并对其具有抑菌活性的次级代谢产物进行了挖掘和初步研究,有望为临床抗菌药物的研发,以及食品工业进行抑菌防腐、生物防治提供一定的理论参考和实践依据。
[1] BROWN E D, WRIGHT G D.Antibacterial drug discovery in the resistance era[J].Nature,2016,529(7 586):336-343.
[2] GENILLOUD O.Actinomycetes:Still a source of novel antibiotics[J].Natural Product Reports,2017,34(10):1 203-1 232.
[3] MOHR K I.History of antibiotics research[J].Current Topics in Microbiology and Immunology,2016,398:237-272.
[4] SUBRAMANI R, SIPKEMA D.Marine rare actinomycetes:A promising source of structurally diverse and unique novel natural products[J].Marine Drugs,2019,17(5):249-289.
[5] LU Q P, YE J J, HUANG Y M, et al.Exploitation of potentially new antibiotics from mangrove actinobacteria in maowei sea by combination of multiple discovery strategies[J].Antibiotics,2019,8(4):236-254.
[6] MOHAMMADIPANAH F, WINK J.Actinobacteria from arid and desert habitats:Diversity and biological activity[J].Frontiers in Microbiology,2015,6:1 541.
[7] SIVALINGAM P, HONG K, POTE J, et al.Extreme environment streptomyces:Potential sources for new antibacterial and anticancer drug leads?[J].International Journal of Microbiology,2019.DOI:10.1155/2019/5283948.
[8] 熊有威, 蒋继宏, 秦盛.拟孢囊菌属放线菌的研究进展[J].微生物学报,2019,59(5):799-808.
XIONG Y W, JIANG J H, QIN S.Recent advances on the genus Kibdelosporangium[J].Acta Microbiologica Sinica, 2019,59(5):799-808.
[9] QIN S, FENG W W, XING K, et al.Complete genome sequence of Kibdelosporangium phytohabitans KLBMP 1111(T), a plant growth promoting endophytic actinomycete isolated from oil-seed plant Jatropha curcas L.[J].Journal of Biotechnology,2015,216:129-130.
[10] BLIN K, SHAW S, STEINKE K, et al.AntiSMASH 5.0:Updates to the secondary metabolite genome mining pipeline[J].Nucleic Acids Research,2019,47(1):81-87.
[11] SHEARER M C, ACTOR P, BOWIE B A, et al.Aridicins, novel glycopeptide antibiotics.I.Taxonomy, production and biological activity[J].The Journal of Antibiotics,1985,38(5):555-560.
[12] SHEARER M C, GIOVENELLA A J, GRAPPEL S F, et al.Kibdelins, novel glycopeptide antibiotics.I.Discovery, production, and biological evaluation[J].The Journal of Antibiotics,1986,39(10):1 386-1 394.
[13] TSUCHIDA T, IINUMA H, KINOSHITA N, et al.Azicemicin A, a new antimicrobial antibiotic from Amycolatopsis[J].The Journal of Antibiotics,1993,46(11):1 772-1 774.
[14] PHILLIPS J W, GOETZ M A, SMITH S K, et al.Discovery of kibdelomycin, a potent new class of bacterial type II topoisomerase inhibitor by chemical-genetic profiling in Staphylococcus aureus[J].Chemistry & Biology,2011;18(8):955-965.
[15] YOON S H, HA S M, KWON S, et al.Introducing EzBioCloud:A taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J].International Journal of Systematic & Evolutionary Microbiology,2017,67(5):1 613-1 617.
[16] 李晓然,叶德晓,付鸣佳 等.枯草芽孢杆菌SX3411产羊毛硫细菌素subtilomycin的初步鉴定与理化特性分析[J].食品与发酵工业,2019,45(12):46-54.
LI X R, YE D X, FU M J, et al.Identification and physicochemical characterization of lantibiotic subtilomycin produced by Bacillus subtilis SX3411[J].Food and Fermentation Industries, 2019,45(12):46-54.
[17] 梁晓彦.两株红树林来源细菌的新种鉴定和溶藻活性物质初步研究[D].青岛:中国海洋大学,2017.
LIANG X Y.Study on the identification of one novel bacteria species and the algicidal substance of one bacteria derived from mangrove[D].Qingdao:Ocean University of China, 2017.
[18] XU Y, HU Z L, WANG C Y, et al.Novel natural algicide with low toxicity to non-target organisms:China, WO/2018/133114[P].2018-07-26.
[19] OGASAWARA Y, TORREZ-MARTINEZ N, ARAGON A D, et al.High-quality draft genome sequence of actinobacterium Kibdelosporangium sp.MJ126-NF4, producer of type II polyketide azicemicins, using Illumina and PacBio technologies[J].Genome Announcements,2015,3(2):14-15.
[20] FUNK A, DIVEKAR P V.Caerulomycin, a new antibiotic from Streptomyces caeruleus Baldacci.I.Production, isolation, assay, and biological properties[J].Canadian Journal of Microbiology, 1959, 5(4):317-321.
[21] GOMI S, AMANO S, SATO E, et al.Novel antibiotics SF2738A, B and C, and their analogs produced by Streptomyces sp.[J].the Journal of Antibiotics, 1994, 47(12):1 385-1 394.
[22] TSUGE N, FURIHATA K, SHIN-YA K, et al.Novel antibiotics pyrisulfoxin A and B produced by Streptomyces californicus[J].the Journal of Antibiotics,1999,52(5):505-507.
[23] 梅显贵,蓝萌萌,金言正,等.天然来源的浅蓝霉素类化合物[J].中国海洋药物,2017,36(5):83-92.
MEI X G, LAN M M, JIN Y Z, et al..Natural caerulomycins and the analogues[J].Chinese Journal of Marine Drugs, 2017,36(5):83-92.
[24] XU D B, YE W W, HAN Y, et al.Natural products from mangrove actinomycetes[J].Marine Drugs, 2014, 12(5):2 590-2 613.
[25] FU P, WANG S X, HONG K, et al.Cytotoxic bipyridines from the marine-derived actinomycete Actinoalloteichus cyanogriseus WH1-2216-6[J].Journal of Natural Products, 2011, 74(8):1 751-1 756.