发酵乳是以生牛(羊)乳或乳粉为原料,经杀菌、发酵后制成的pH值降低的产品(GB 19302—2010《食品安全国家标准发酵乳》)。发酵乳行业的规模和质量飞速发展,符合“健康中国”、“国民营养计划”等国家战略,是关系国计民生的重要产业。嗜热链球菌(Streptococcus thermophilus)和德氏乳杆菌保加利亚亚种(Lactobacillus delbrueckii subsp.bulgaricus)是发酵乳常用发酵剂菌种,二者之间存在共生关系,德氏乳杆菌保加利亚亚种代谢生成的氨基酸可刺激嗜热链球菌的生长,而嗜热链球菌在代谢过程中产生的甲酸可以促进德氏乳杆菌保加利亚亚种的生长[1]。发酵乳通常添加对人体有益的益生菌,如:动物双歧杆菌乳亚种(Bifidobacterium animalis subsp.lactis)和干酪乳酪杆菌(Lacticaseibacillus casei),有助于促进肠道微生物的定殖,治疗和预防腹泻、缓解便秘、增强人体免疫力等[2],某一株益生菌在产品中需要达到一定数量才能起到保健作用,因此发酵乳中菌种的鉴定和定量尤其重要。
目前我国对于发酵乳中乳酸菌的使用和监管,标准主要为GB 4789.34—2016《食品安全国家标准 食品微生物学检验 双歧杆菌检验》和GB 4789.35—2016《食品安全国家标准 食品微生物学检验 乳酸菌检验》,采用传统培养方法和生理生化鉴定方法进行发酵乳中乳酸菌的鉴定和定量,具有成本低,易于操作等优点,但需要获得菌种纯培养物,且存在检测结果不准确等问题。建立快速、准确的复合乳酸菌发酵乳菌种鉴定和定量检测方法,成为发酵乳行业亟待解决的热点、难点问题之一[3]。近几年来,高通量测序技术迅速发展,采用宏基因组测序分箱分析可将包含不同生物体的序列进行组装,重现复合样品中的微生物种类,解析产品组成。该方法以复合样品总基因组DNA为分析对象,有效避免了菌种分离培养步骤,目前已被应用于复合益生菌产品的物种组成分析[4]。平均核苷酸一致性(average nucleotide identity,ANI)是通过全基因组序列鉴定细菌物种的常用方法之一,其域值95%~96%与DNA-DNA杂交值70%相对应,可作为细菌种水平界定的标准[5]。ANI分析具有方便、快捷、分辨率高的优点[5],其与宏基因组分箱分析相结合,为复合乳酸菌发酵乳菌种的种水平精准鉴定提供了一种可靠方法。
复合乳酸菌发酵乳产品的菌种组成与定量分析是产品质量的关键。本文以3款市售复合乳酸菌发酵乳产品为研究对象,利用宏基因组测序和分箱技术、ANI分析等技术,研究复合乳酸菌发酵乳产品中菌种种水平鉴定及相对定量。对复合菌发酵乳产品的质量控制和行业监管具有重要参考意义。
1 材料与方法
1.1 实验材料
1.1.1 复合乳酸菌发酵乳产品
本研究选取3款不同品牌的复合乳酸菌发酵乳产品A、B、C,均购自北京大型超市。产品标签标识菌株类别及活菌数如下:
样品1:产品A(菌种名称:动物双歧杆菌BB-12、嗜热链球菌、德氏乳杆菌保加利亚亚种;保加利亚乳杆菌和嗜热链球菌总数1×106 CFU/g,双歧杆菌1×107 CFU/g);
样品2:产品B(菌种名称:保加利亚乳杆菌、嗜热链球菌、干酪乳杆菌,乳酸菌活菌数≥1×106 CFU/g);
样品3:产品C(菌种名称:乳双歧杆菌BL-99、嗜热链球菌、保加利亚乳杆菌;乳双歧杆菌总数≥1×106 CFU/g)。
1.1.2 试剂与仪器
DNA提取试剂盒:E.Z.N.A.® Bacterial DNA Kit,美国OMEGA公司;文库构建试剂盒NEXTflex Rapid DNA-Seq Kit,美国Bioo Scientific公司。
Eppendorf微量可调节移液器、5424R小型台式冷冻离心机,德国Eppendorf公司;HG-50高压灭菌锅,日本HIRAYAMA公司;AC2-4S1生物安全柜,新加坡Esco公司;BioDrop核酸蛋白分析仪,豪沃生物科技(上海)有限公司;Qubit 3.0 核酸荧光定量仪,美国ThermoFisher Scientific公司;Illumina HiSeq 3000基因测序平台,美国Illumina公司。
1.2 实验方法
1.2.1 基因组DNA的提取
取20 mL发酵乳样品,4 000 r/min离心5 min,吸取上清液,12 000 r/min离心10 min,取沉淀按照试剂盒E.Z.N.A.® Bacterial DNA Kit的说明书提取基因组DNA。使用Qubit 3.0核酸荧光定量仪、BioDrop核酸蛋白分析仪分别检测基因组DNA的浓度和纯度,使用1%琼脂糖凝胶电泳检测基因组DNA的完整度。
1.2.2 宏基因组文库构建与测序
通过物理方法将质检合格的基因组DNA随机打断成300~400 bp的片段,使用NEXTflex Rapid DNA-SeqKit按照说明书进行文库制备。使用Illumina HiSeq 3000测序平台(上海美吉生物医药科技有限公司)进行宏基因组测序,宏基因组序列NCBI登录号分别为:SAMN22013808,SAMN22013809,SAMN22013810。
1.2.3 下机数据质控与组装
采用MetaWRAP流程[6]对宏基因组下机数据进行质控和组装,采用Trim Galore(http://www.bioinformat- ics.babraham.ac.uk/projects/trim_galore/)去除reads 的adapter、过滤质量值低于20的reads,参数采用默认设置。使用Megahit[7]软件进行序列组装,采用succinct de Bruijn graph方法进行拼接,k-mer参数从小至大迭代,选择长度≥1 000 bp的contigs用于后续分析。
1.2.4 分箱分析
采用MetaWRAP流程对拼接后的contig开展分箱分析,获得各样品的分箱结果(bin)。利用checkM[8]评估各bin的完整度和污染度,选取完整度>70%和污染度<10%的bin[9],利用blobology模块绘制各样品中bin的丰度散点图。通过Circos[10](版本0.69-8)软件对各样品bins 进行可视化分析,每个bin的contigs重新基于模式菌株有参组装,展示最终的基因组圈图。
1.2.5 ANI分析
从NCBI genome 数据库下载相关种模式菌株的基因组序列,使用fastANI[11](版本:1.32)计算各bin与相关物种模式株基因组序列之间的ANI值。以ANI值为95%~96%作为细菌物种界定的标准[5],对各样品的分箱结果进行物种鉴定。
1.2.6 物种定量分析
采用Quant_bins模块分析各bin的相对丰度。
2 结果与分析
2.1 测序数据质控和组装
针对样品的宏基因组测序下机数据,采用Fastp[12]软件,碱基质量值≥20,对raw data进行质控。质控后A、B、C 3个产品的clean data数分别为13 667 335 968、12 529 436 956和12 225 941 409 bp,碱基质量值Q30均大于90%,测序数据质量能够满足后续分析(表1)。
表1 样品宏基因组测序数据统计
Table 1 Data statistics for metagenomic sequencing data
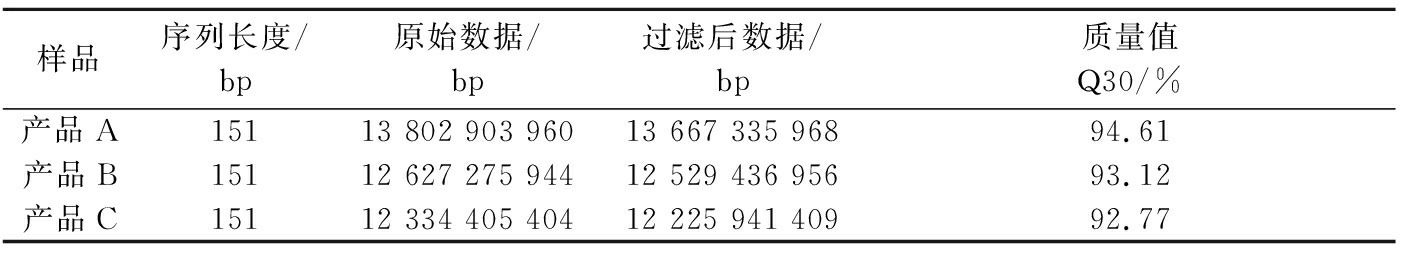
样品序列长度/bp原始数据/bp过滤后数据/bp质量值Q30/%产品A15113 802 903 96013 667 335 96894.61产品B15112 627 275 94412 529 436 95693.12产品C15112 334 405 40412 225 941 40992.77
对clean data进行组装,各产品获得的contigs数目及相关参数见表2。
表2 样品宏基因组数据组装结果
Table 2 Assembly result of the metagenomic sequencing data
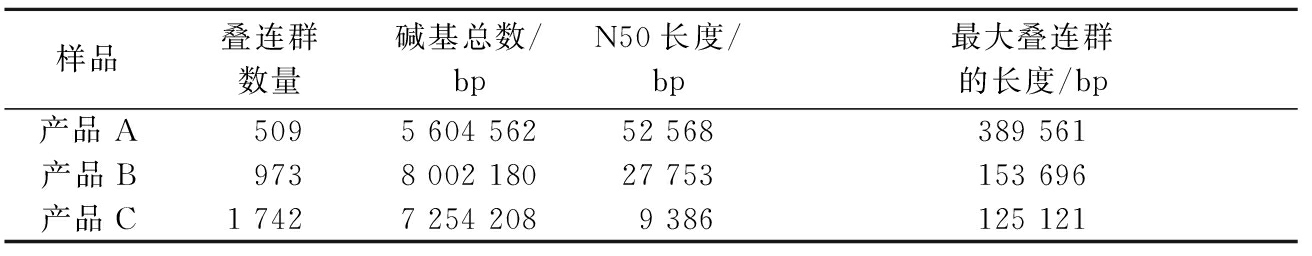
样品叠连群数量碱基总数/bpN50长度/bp最大叠连群的长度/bp产品A5095 604 56252 568389 561产品B9738 002 18027 753153 696产品C1 7427 254 2089 386125 121
2.2 复合乳酸菌产品中物种基因组重建
使用MetaWRAP宏基因组分析流程对组装后的contigs进行分箱分析,获得分箱结果(bin),即重建的基因组。产品A、B、C的contigs 均分别分箱成3个bin(表3),与各产品标识所声称添加的菌种数量一致。通过checkM来评估分箱后bin的完整度和污染度,结果表明,各样品分箱产生的bin的完整度均高于85%,污染度均低于5%(表3)。各Bin丰度散点图见图1。
表3 宏基因组分箱后各bin的参数
Table 3 The results of metagenomics binning
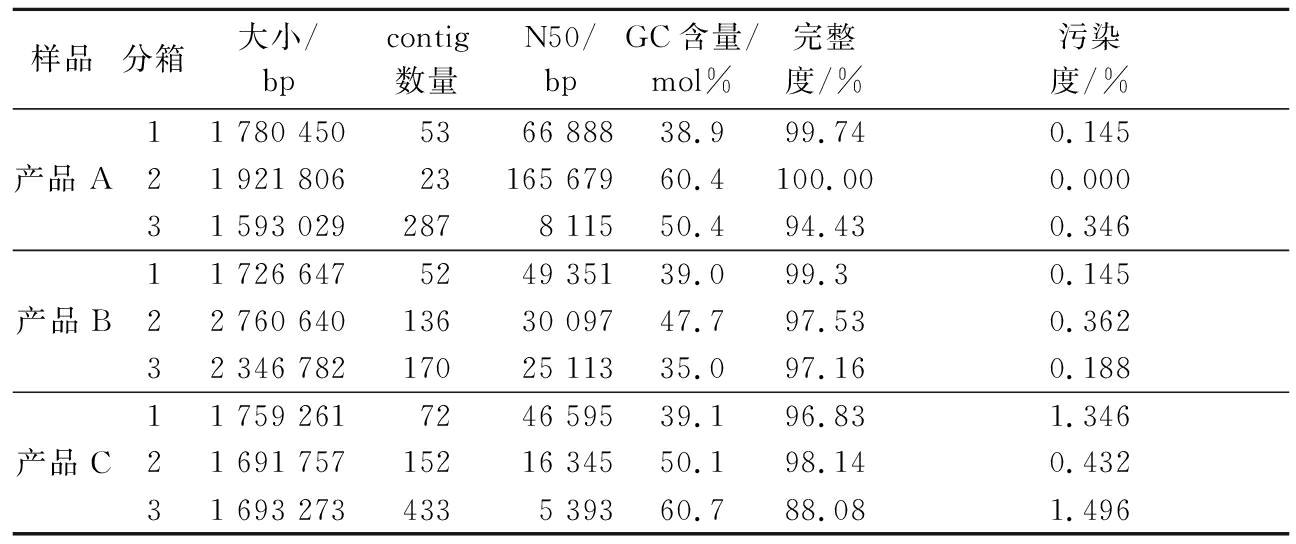
样品分箱大小/bpcontig数量N50/bpGC含量/mol%完整度/%污染度/%产品A11 780 4505366 88838.999.740.14521 921 80623165 67960.4100.000.00031 593 0292878 11550.494.430.346产品B11 726 6475249 35139.099.3 0.14522 760 64013630 09747.797.530.36232 346 78217025 11335.097.160.188产品C11 759 2617246 59539.196.831.34621 691 75715216 34550.198.140.43231 693 2734335 39360.788.081.496
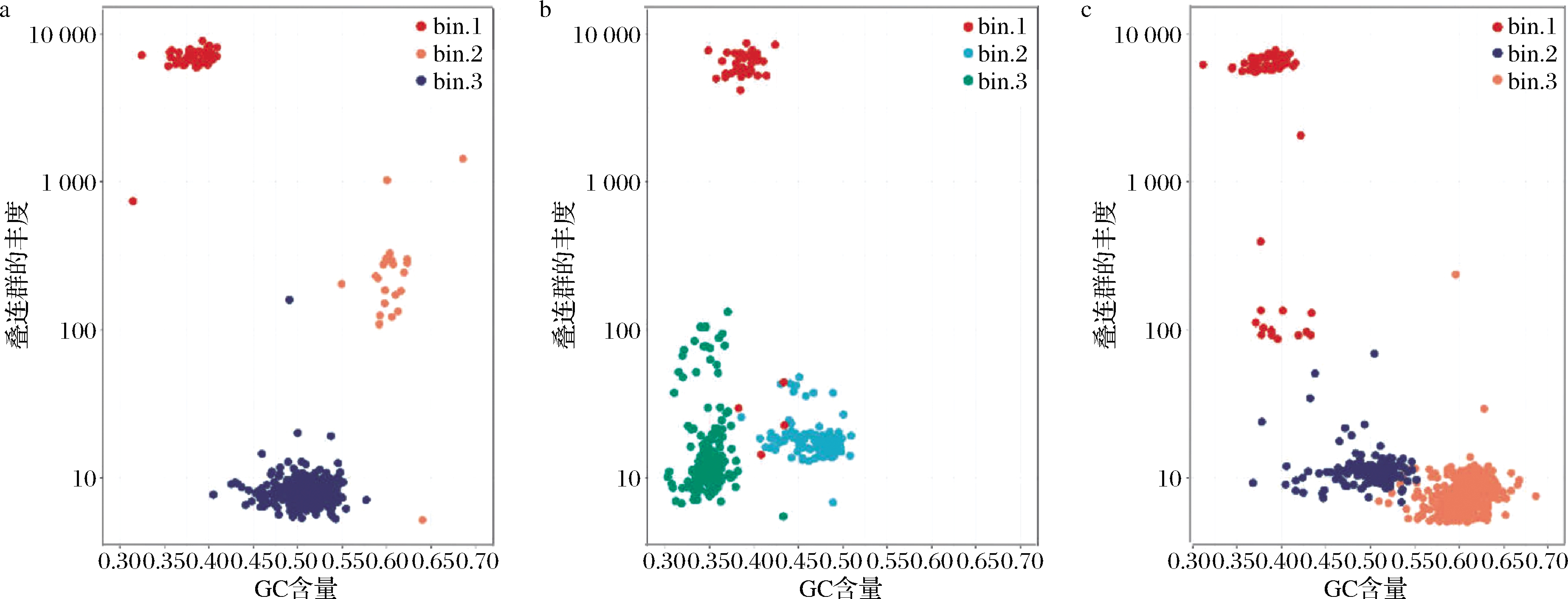
a-产品A;b-产品B;c-产品C
图1 Bin丰度散点图
Fig.1 Scatter plots of bins abundance
注:一个点代表一个contig,相同颜色的contig来自同一个bin
2.3 复合乳酸菌产品中物种的鉴定
将分箱获得的各bin通过NCBI-Blast进行初步比对,确定其近缘物种,利用fastANI软件计算各bin与近缘物种模式株之间的ANI值(表4)。每个样品中的bin与最近缘物种模式菌株基因组ANI分析的值均>95%,表明每个bin 代表的菌种与模式菌株为同一个种(表4)。
表4 分箱结果与参考模式菌基因组ANI 分析
Table 4 The analysis results of ANI between bins and genomes of type strains
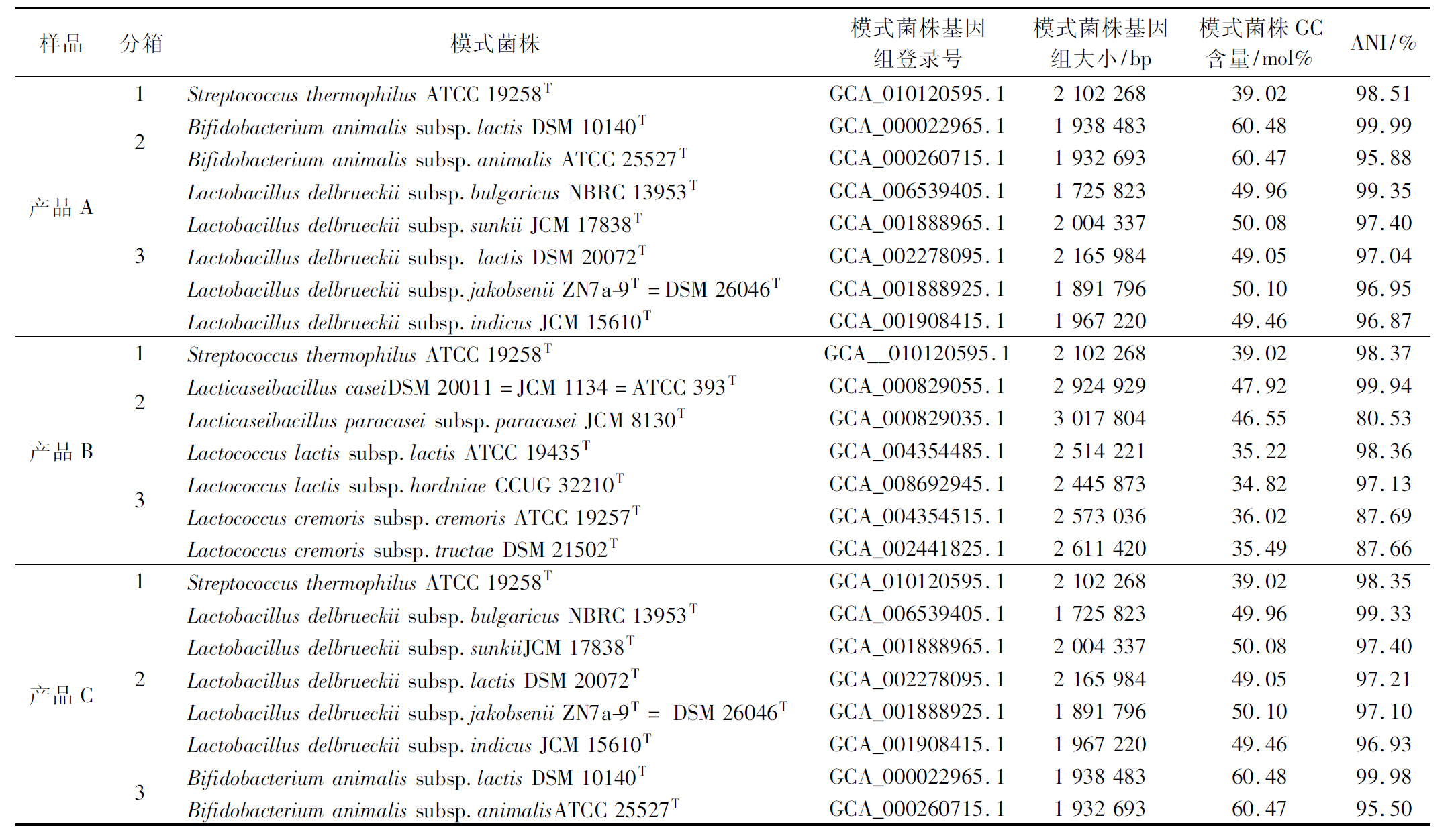
样品分箱模式菌株模式菌株基因组登录号模式菌株基因组大小/bp模式菌株GC含量/mol%ANI/%产品A123Streptococcus thermophilus ATCC 19258TGCA_010120595.12 102 26839.0298.51Bifidobacterium animalis subsp.lactis DSM 10140TGCA_000022965.11 938 48360.4899.99Bifidobacterium animalis subsp.animalis ATCC 25527TGCA_000260715.11 932 69360.4795.88Lactobacillus delbrueckii subsp.bulgaricus NBRC 13953TGCA_006539405.11 725 82349.9699.35Lactobacillus delbrueckii subsp.sunkii JCM 17838TGCA_001888965.12 004 33750.0897.40Lactobacillus delbrueckii subsp. lactis DSM 20072TGCA_002278095.12 165 98449.0597.04Lactobacillus delbrueckii subsp.jakobsenii ZN7a-9T=DSM 26046TGCA_001888925.11 891 79650.1096.95Lactobacillus delbrueckii subsp.indicus JCM 15610TGCA_001908415.11 967 22049.4696.87产品B123Streptococcus thermophilus ATCC 19258TGCA__010120595.12 102 26839.0298.37Lacticaseibacillus caseiDSM 20011=JCM 1134=ATCC 393TGCA_000829055.12 924 92947.9299.94Lacticaseibacillus paracasei subsp.paracasei JCM 8130TGCA_000829035.13 017 80446.5580.53Lactococcus lactis subsp.lactis ATCC 19435TGCA_004354485.12 514 22135.2298.36Lactococcus lactis subsp.hordniae CCUG 32210TGCA_008692945.12 445 87334.8297.13Lactococcus cremoris subsp.cremoris ATCC 19257TGCA_004354515.12 573 03636.0287.69Lactococcus cremoris subsp.tructae DSM 21502TGCA_002441825.12 611 42035.4987.66产品C123Streptococcus thermophilus ATCC 19258TGCA_010120595.12 102 26839.0298.35Lactobacillus delbrueckii subsp.bulgaricus NBRC 13953TGCA_006539405.11 725 82349.9699.33Lactobacillus delbrueckii subsp.sunkiiJCM 17838TGCA_001888965.12 004 33750.0897.40Lactobacillus delbrueckii subsp.lactis DSM 20072TGCA_002278095.12 165 98449.0597.21Lactobacillus delbrueckii subsp.jakobsenii ZN7a-9T = DSM 26046TGCA_001888925.11 891 79650.1097.10Lactobacillus delbrueckii subsp.indicus JCM 15610TGCA_001908415.11 967 22049.4696.93Bifidobacterium animalis subsp.lactis DSM 10140TGCA_000022965.11 938 48360.4899.98Bifidobacterium animalis subsp.animalisATCC 25527TGCA_000260715.11 932 69360.4795.50
产品A和产品C中3个bin通过ANI分析鉴定为嗜热链球菌、动物双歧杆菌和德氏乳杆菌,与该产品声称添加的菌种一致。产品B中3个bin通过ANI分析鉴定为嗜热链球菌、干酪乳酪杆菌和乳酸乳球菌(Lactococcus lactis),其中,嗜热链球菌和干酪乳酪杆菌与该产品声称添加的菌种一致,未检测到该产品声称添加的德氏乳杆菌,检测到的乳酸乳球菌未出现在该产品声称中。
Contig利用模式基因组进行有参组装后,通过Circos v 0.69-8软件对各样品bin进行可视化,基因组圈图见图2。

图2 产品宏基因组菌种基因组重建
Fig.2 Re-constructed genomes from samples by means of the metagenomics binning
注:每一个产品的bins 都由横向排列的遗传图谱来表示,相同物种用同一种颜色显示,绿色“√”表示分箱得到的物种与产品声称菌种一致
动物双歧杆菌和干酪乳杆菌在我国《可用于食品的菌种名单(卫办监督发〔2010〕65号)》之列,是
发酵乳常用的益生菌,在A、B和C 3个产品中也均有添加。其中,动物双歧杆菌乳亚种曾用名为乳双歧杆菌(Bifidobacterium lactis)[13];德氏乳杆菌保加利亚亚种的曾用名为保加利亚乳杆菌(Lactobacillus bulgaricus)[14]。干酪乳杆菌(Lactobacillu casei)在2020年其分类学地位变更为干酪乳酪杆菌[15]。
德氏乳杆菌在产品B中未被检测到,进一步分析发现,在利用完整度>70%、污染度<10%参数对产品B宏基因组分箱初步结果的过滤前,检测到1个完整度仅为6.61%的bin,通过ANI分析显示为德氏乳杆菌,定量结果显示相对丰度为0.09%,由于bin完整度较低,可能会引起该定量检测结果的偏差,该菌种按照宏基因组数据过滤参数未形成完整bin,说明产品中菌种的相对丰度较低时,可能引起分箱bin的完整度不够而没有被检测到。产品B中检测到的乳酸乳球菌,推测可能为原料、环境、生产过程等环节引入,或未经菌种鉴定误按嗜热链球菌添加进产品,尚需进一步的验证确认。
2.4 复合乳酸菌产品中各物种的定量分析
利用Quant_bins模块计算各样品中bin的相对丰度,结果如表5所示。
表5 各分箱结果的定量分析
Table 5 Quantitative analysis of bins

样品声称名称声称含量鉴定结果定量结果产品A嗜热链球菌德氏乳杆菌动物双歧杆菌BB-12保加利亚乳杆菌和嗜热链球菌总数1×106 CFU/g双歧杆菌1×107 CFU/g嗜热链球菌(S.thermophilus)96.85%德氏乳杆菌(L.delbrueckii)0.12%动物双歧杆菌(B.animals)3.02%产品B嗜热链球菌干酪乳杆菌保加利亚乳杆菌/乳酸菌活菌数≥1×106 CFU/g/嗜热链球菌(S.thermophilus)99.43%干酪乳酪杆菌(L.casei)0.27%未检出0.00%乳酸乳球菌(L.lactis)0.21%产品C嗜热链球菌/嗜热链球菌(S.thermophilus)99.55%保加利亚乳杆菌/德氏乳杆菌(L.delbrueckii)0.18%乳双歧杆菌BL-99乳双歧杆菌≥1×106 CFU/g动物双歧杆菌(B.animals)0.14%
对于产品A,其分箱分析所检测到的嗜热链球菌、德氏乳杆菌和动物双歧杆菌3个物种的相对丰度分别为96.85%、0.12%和3.02%,嗜热链球菌为绝对优势物种。该产品标识声称嗜热链球菌和德氏乳杆菌总量为1×106 CFU/g,定量结果表明,产品中二者的相对比例约为807∶1。产品B标识声称活菌总数为1×106 CFU/g,通过定量分析显示,分箱分析所检测到的嗜热链球菌、干酪乳酪杆菌和乳酸乳球菌3个物种的相对丰度分别为99.43%、0.27%和0.21%,嗜热链球菌为绝对优势物种。产品C声称添加的物种类别与产品A相同,且其中动物双歧杆菌的添加量为1×106 CFU/g。定量结果表明,嗜热链球菌的相对丰度为99.55%,德氏乳杆菌和动物双歧杆菌的相对丰度分别为0.18%和0.14%。复合乳酸菌发酵乳产品中对所添加各菌种定量主要是以乳酸菌活菌数或某一种/株益生菌活菌数为指标,本研究中基于宏基因组的定量分析为相对定量,以活菌数和死菌数总和的百分比表示,随着后生元(postbiotics)[16]概念逐渐被大众所熟悉及相应产品的开发,相较于传统定量检测方法,本文提供了新的思路。
3 结论
本研究以3款市售复合乳酸菌发酵乳产品为研究对象,利用宏基因组测序和分箱分析、ANI分析等技术,研究复合乳酸菌发酵乳产品中菌种种水平鉴定及相对定量。产品A和产品C分箱获得的3个bin通过ANI分析鉴定为嗜热链球菌、德氏乳杆菌和动物双歧杆菌,与产品声称添加菌种一致,产品A 3个物种的相对丰度分别为96.85%、0.12%和3.02%,产品C 3个物种的相对丰度分别为99.55%、0.18%和0.14%。产品B分箱获得的3个bin通过ANI分析鉴定为嗜热链球菌、干酪乳酪杆菌和乳酸乳球菌,与产品声称添加菌种部分一致,3个物种的相对丰度分别为99.43%、0.27%和0.21%。本研究为复合乳酸菌发酵乳产品的菌种鉴定与定量提供了一种可行的分析方法,与传统基于可培养的分析方法相比,具有准确、可靠等优点,且不受菌种死活状态的影响。随着测序技术的不断革新,高通量测序在成本与周期方面必将进一步下降和缩短,宏基因测序及分箱作为一项新兴技术在食品检测领域将进一步推广应用,有望发展成为一种常规分析方法。本研究对复合乳酸菌发酵乳产品的质量控制和行业监管具有重要参考意义。
[1] VERINGA H A, GALESLOOT T E, DAVELAAR H.Symbiosis in yoghurt (II).Isolation and identification of a growth factor for Lactobacillus bulgaricus procuced by Streptococcus thermophilus[J].Netherlands Milk and Dairy Journal, 1968, 22:114-120.
[2] MORIYA J, FACHIN L, G NDARA A L N, et al.Evaluation of culture media for counts of Bifidobacterium animalis in the presence of yoghurt bacteria[J].Brazilian Journal of Microbiology, 2006, 37(4):516-520.
NDARA A L N, et al.Evaluation of culture media for counts of Bifidobacterium animalis in the presence of yoghurt bacteria[J].Brazilian Journal of Microbiology, 2006, 37(4):516-520.
[3] 中国食品科学技术学会益生菌分会. 益生菌的科学共识(2020年版)[J].中国食品学报, 2020, 20(5):303-307.
Probiotics Society of the Chinese Institute of Food Science and Technology.Scientific consensus on probiotics (2020)[J].Journal of Chinese Institute of Food Science and Technology, 2020, 20(5):303-307.
[4] LUGLI G A, MANGIFESTA M, MANCABELLI L, et al.Compositional assessment of bacterial communities in probiotic supplements by means of metagenomic techniques[J].International Journal of Food Microbiology, 2019, 294:1-9.
[5] RICHTER M, ROSSELL -M
-M RA R.Shifting the genomic gold standard for the prokaryotic species definition[J].Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(45):19 126-19 131.
RA R.Shifting the genomic gold standard for the prokaryotic species definition[J].Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(45):19 126-19 131.
[6] URITSKIY G V, DIRUGGIERO J, TAYLOR J.MetaWRAP-a flexible pipeline for genome-resolved metagenomic data analysis[J].Microbiome, 2018, 6(1):158.
[7] LI D H, LIU C M, LUO R B, et al.MEGAHIT:an ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph[J].Bioinformatics, 2015, 31(10):1 674-1 676.
[8] PARKS D H, IMELFORT M, SKENNERTON C T, et al.CheckM:Assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes[J].Genome Research, 2015, 25(7):1 043-1 055.
[9] BANDLA A, PAVAGADHI S, SRIDHAR SUDARSHAN A, et al.910 Metagenome-assembled genomes from the phytobiomes of three urban-farmed leafy Asian greens[J].Scientific Data, 2020, 7(1):278.
[10] KRZYWINSKI M, SCHEIN J, BIROL I, et al.Circos:An information aesthetic for comparative genomics[J].Genome Research, 2009, 19(9):1 639-1 645.
[11] JAIN C, RODRIGUEZ-R L M, PHILLIPPY A M, et al.High throughput ANI analysis Of 90K prokaryotic genomes reveals clear species boundaries[J].Nature Communications,2018, 9 (1):1-6.
[12] CHEN S F, ZHOU Y Q, CHEN Y R, et al.Fastp:an ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics, 2018, 34(17):i884-i890.
[13] ZHENG J S, WITTOUCK S, SALVETTI E, et al.A taxonomic note on the genus Lactobacillus:Description of 23 novel genera, emended description of the genus Lactobacillus Beijerinck 1901, and union of Lactobacillaceae and Leuconostocaceae[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(4):2 782-2 858.
[14] MASCO L, VENTURA M, ZINK R, et al.Polyphasic taxonomic analysis of Bifidobacterium animalis and Bifidobacterium lactis reveals relatedness at the sub species level:Reclassification of Bifidobacterium animalis as Bifidobacterium animalis subsp.animalis subsp.nov.and Bifidobacterium lactis as Bifidobacterium animalis subsp.lactis subsp.nov[J].International Journal of Systematic and Evolutionary Microbiology, 2004, 54(Pt 4):1 137-1 143.
[15] WEISS N, SCHILLINGER U, KANDLER O.Lactobacillus lactis, Lactobacillus leichmannii and Lactobacillus bulgaricus, subjective synonyms of Lactobacillus delbrueckii, and description of Lactobacillus delbrueckii subsp.lactis comb.nov.and Lactobacillus delbrueckii subsp.bulgaricus comb.nov[J].Systematic and Applied Microbiology, 1983, 4(4):552-557.
[16] SWANSON K S, GIBSON G R, HUTKINS R W, et al.The international scientific association for probiotics and prebiotics (ISAPP) consensus statement on the definition and scope of synbiotics[J].Nature Reviews Gastroenterology & Hepatology,2020, 17(11), 1-15.