腐乳,以大豆制成的豆腐为原料,辅以辣椒、食盐、花椒、红曲(红腐乳)等,通过固态发酵制成,其质地细腻,醇香可口,营养丰富,含有丰富的游离氨基酸,尤其是谷氨酸含量较高,还含有B族维生素、矿物质等,易于消化,对人体有益[1-2]。
腐乳按照发酵的微生物类型可以分为细菌型腐乳和毛霉型腐乳,按照颜色可以分为红方腐乳、青方腐乳、白方腐乳和花色腐乳等[3]。红方腐乳由于添加了红曲,因此成品为红色,而花色腐乳则是除了主要原料豆腐外,还添加了芝麻、玫瑰、虾仔、香油中的一种或几种制成。腐乳的制作往往通过开放式的发酵方式,因此不同腐乳的菌群可能存在差异,导致代谢产生的风味物质含量与种类也有不同。目前对于传统发酵食品的研究多集中在传统发酵食品的菌群结构及演替、滋味、气味和挥发性风味物质[4]。
以MiSeq等为代表的第二代测序技术具有通量高、成本低、准确度高,且不依赖于培养条件等特点,能对样品的低丰度微生物进行检测与分析,还能与其他组学方法进行联合分析[5-6]。近年来用于探索菌群结构的扩增子测序的分析方案不断完善,出现了多种分析平台,如QIIME,mothur和usearch,以及相应的注释数据库如RDP,GreenGene以及SILVA[7-9],因此该技术被广泛应用到传统发酵食品的微生物区系研究。多组学分析方法的发展,不仅能获得食品的菌群特征,还能结合食品挥发性有机物(volatile organic compound, VOC)进行关联分析。
红方腐乳和湖北省谷城市茨河地区的花色腐乳均呈红色,但是气味迥异[1]。为分析其菌群与风味的差异,本研究采集了市售的不同厂家的花色和红方腐乳样品,进行高通量测序及VOC物质分析,旨在明确并比较花色腐乳与红方腐乳的微生物菌群特征,总体感官及挥发性风味物质特征,并分析两种腐乳风味特征与菌群的关联,以期为后续腐乳品质改良与安全性评价提供数据支撑。
1 材料与方法
1.1 材料与试剂
Pfu高保真DNA聚合酶、DNA Marker DL2000、dNTP混合物,宝生物工程(大连)有限公司;琼脂糖,西班牙Biowest公司;E.Z.N.A.®食品DNA提取试剂盒,美国Omega生物科技公司;PCR产物清洁试剂盒,艾思进生物技术(杭州)公司;用于电子舌的内部溶液、参比溶液、阴离子溶液、阳离子溶液和味觉标准溶液,日本Insent公司。
1.2 仪器与设备
Vetiri梯度基因扩增仪,美国AB公司;ND-2000C型微量紫外分光光度计,美国Nano Drop公司;PEN3电子鼻,德国Airsense公司;SA-402B型电子舌,日本Insent公司;FlavourSpec®气相色谱-离子迁移谱联用仪,德国G.A.S公司;DYY-6C型电泳仪,中国北京六一生物科技有限公司;VD-1320-U洁净工作台,中国苏净安泰公司;LDZX-50KBS蒸汽压力灭菌器,中国上海申安医疗器械厂。
1.3 实验方法
1.3.1 样品采集
花色腐乳(n=3,样品编号为C1-C3),湖北省襄阳市谷城县茨河镇农贸市场3家不同厂家;红方腐乳(n=4,样品编号为R1-R4),上海、浙江绍兴、河北张家口和河北衡水不同厂家。样品运回实验室后,立刻装入带有螺旋盖子的无菌玻璃样品瓶,在实验室迅速用液氮冷冻,随后放入-70 ℃冰箱备用。
1.3.2 高通量测序
样品处理过程参照赵恒等[10]的方法进行,使用E.Z.N.A.®食品DNA试剂盒对7份腐乳样品进行总DNA的提取。然后使用338F和806R获取腐乳样品细菌16S rRNA的V3-V4区,另外使用ITS3F和ITS4R扩增样品真菌ITS间区片段[11]。PCR产物使用Illumina HiSeq测序平台进行高通量测序。
1.3.3 生物信息学分析
生物信息学分析参照赵恒等[10]的方法进行,主要使用QIIME v1.9.1平台[12]。使用cutadapt v1.9(https://cutadapt.readthedocs.io/en/stable/)对数据进行过滤,然后按照97%的阈值划分分类操作单元(operational taxonomic unit,OTU),使用UCHIME去除嵌合体序列[13],并通过Ribosomal Database Project (RDP) v11.5,GreenGene v13.5和SILVA v132数据库进行细菌物种注释[7-9],使用UNITE v7.2进行真菌物种注释[14]。样品的α多样性与β多样性及测序深度通过QIIME平台自带程序进行计算。
1.3.4 样品的VOCs分析
对VOCs测定使用FlavourSpec®气相色谱-离子迁移谱仪(gas chromatography-ion mobility spectrometry,GC-IMS):首先准确称取腐乳样品1 g,装入20 mL顶空瓶中,设置进样针温度为85 ℃。顶空进样条件:50 ℃孵育15 min(转速500 r/min),自动顶空进样,进样量100 μL。GC条件:色谱柱为极性WAX柱(30 m×0.53 mm,1 μm),柱温60 ℃,分析时间30 min,载气N2,载气流速在0~10 min为2 mL/min,10~20 min升至10 mL/min,20~30 min升至100 mL/min。IMS条件:45 ℃,漂移气流速150 mL/min。获取初步数据后,使用NIST数据库和IMS数据库可对物质进行定性分析,通过峰面积归一化法确定各种成分的相对含量。
1.3.5 统计分析
使用SPSS 19.0对数据进行单因素方差分析,基于t检验进行组间差异显著性检验,结果以平均值±标准差表示。主坐标分析(principal co-ordinates analysis,PCoA)、普氏分析和置换多元方差分析(permutational multivariate analysis of variance,perMANOVA)使用R包vegan v2.5-7(https://cran.r-project.org/web/packages/vegan/index.html);使用R自带的corr.test函数进行Pearson相关性分析;不同样品间的生物标志物通过LEfSe(The linear discriminant analysis Effect Size)通过在线服务器(http://huttenhower.sph.harvard.edu/galaxy/)进行分析。文中所有图使用Origin v8.5或者R包ggplot2完成。
2 结果与分析
2.1 腐乳微生物多样性分析
2.1.1 微生物多样性
基于MiSeq测序平台进行腐乳样品的高通量测序,共获得了420 944条细菌序列,花色腐乳平均65 664条序列每个样品,红方腐乳平均43 258条序列。共聚类得到5 860个OTUs:花色腐乳每个样品平均1 928个OTUs,红方腐乳平均1 238个OTUs。当测序深度达到32 010条序列时,稀释曲线变得平缓,表明测序量足够反映样品菌群多样性。α多样性分析表明,花色腐乳的Chao1指数与Shannon指数菌低于红方腐乳,但是仅Chao1指数的差异显著(P<0.5),表明花色腐乳的细菌丰富度显著低于红方腐乳(图1)。
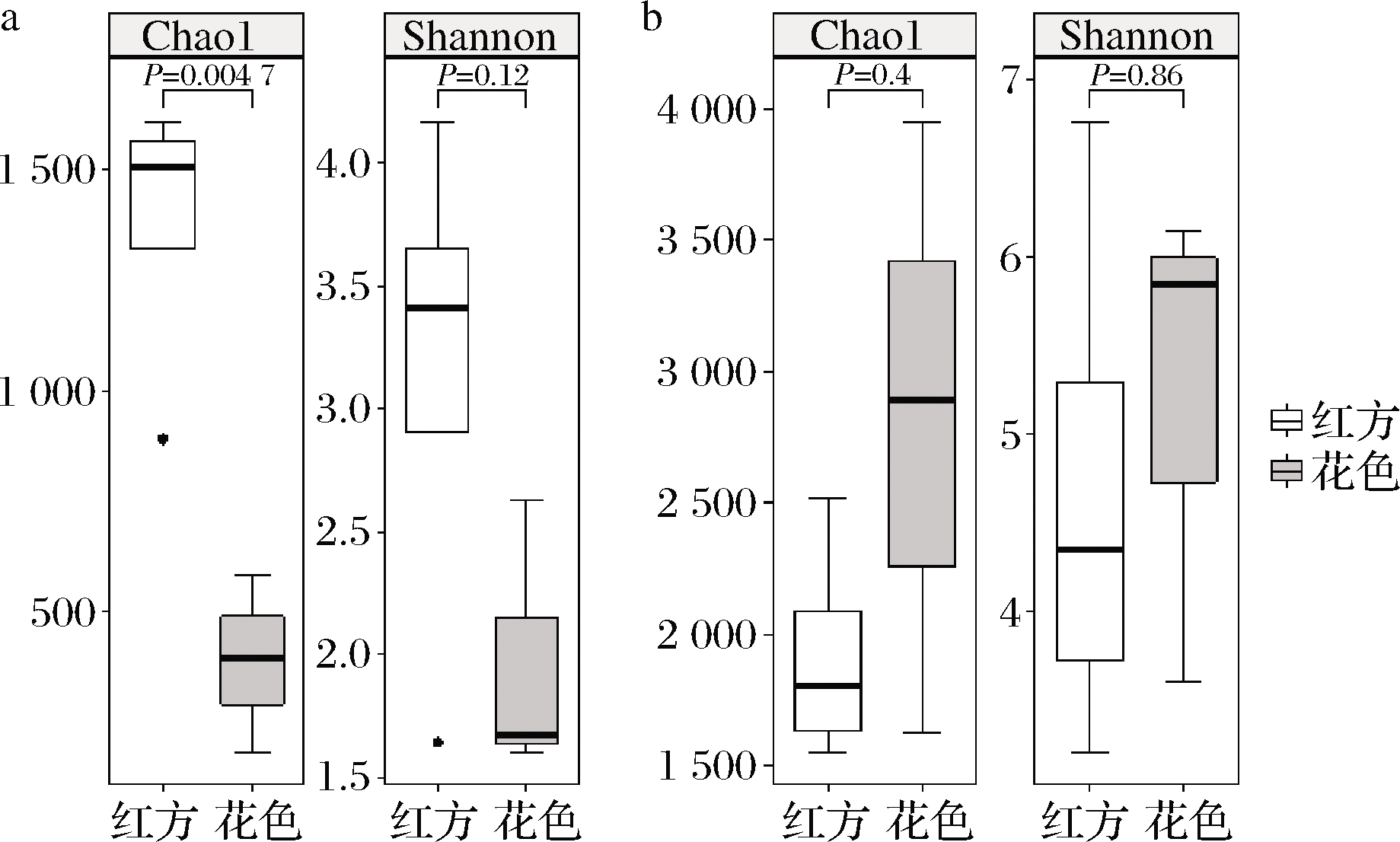
a-细菌;b-真菌
图1 花色腐乳和红方腐乳α多样性
Fig.1 α Diversity of Huase and red sufu
扩增子测序获得了436 925条真菌高质量扩增子序列,按照97%的阈值,划分出1 413个OTUs,花色腐乳每个样品平均309个OTUs,红方腐乳平均314 OTUs,远低于细菌的OTU数量。在测序深度达到35 010的基础上对腐乳真菌α多样性进行分析,显示真菌的Chao1指数和Shannon指数均无显著差异(P>0.05)。
基于UniFrac距离矩阵的PCoA分析表明(图2),红方腐乳与花色腐乳的细菌与真菌菌群在坐标轴上分布在不同的位置;perMANOVA分析进一步证实了红方腐乳与花色腐乳在细菌与真菌菌群结构上具有显著差异(P<0.05)。为进一步分析腐乳样品的菌群特征,计算了腐乳中相对含量大于1%的优势菌群组成。

a-真菌;b-细菌
图2 基于UniFrac距离的腐乳微生物PCoA图
Fig.2 PCoA plots of microbial community in sufu samples based on UniFrac distance
2.1.2 微生物菌群结构
如图3-a所示,红方腐乳的优势真菌门(相对含量>1%)有:子囊菌门(Ascomycota,57.04%),担子菌门(Basidiomycota,39.68%)和毛霉门(Mucoromycota)(1.81%);而在花色腐乳中,子囊菌门平均相对含量最高(94.24%),担子菌门(0.08%)和毛霉门(4.33%),接合菌门(1.32%)相对含量最低。
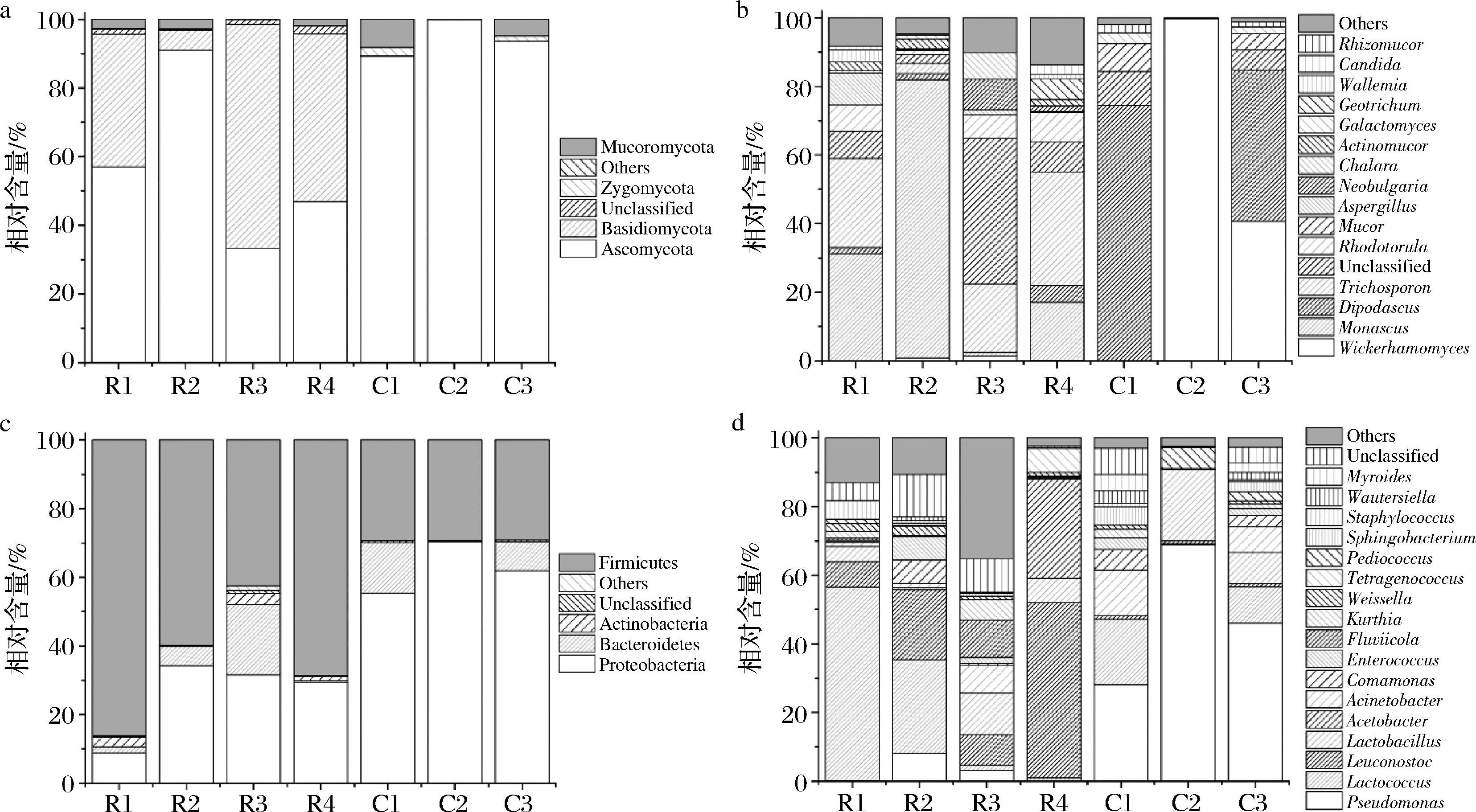
a-真菌优势菌门;b-真菌优势菌属;c-细菌优势菌门;d-细菌优势菌属
图3 腐乳样品优势微生物类群组成
Fig.3 Dominant microbial taxa of sufu samples
在属水平上(图3-b),红方腐乳共11个优势真菌属:红曲霉属(Monascus,32.55%),双足囊菌属(Dipodascus,2.13%),丝孢酵母属(Trichosporon,20.44%),红酵母属(Rhodotorula,6.12%),曲霉属(Aspergillus,2.83%),Neobulgaria(2.64%),Chalara(2.16%),放射毛霉属(Actinomucor,1.81%),地霉属(Geotrichum,1.53%),节担菌属(Wallemia,1.20%),假丝酵母属(Candida,1.06%),仅红曲霉属和丝孢酵母属的相对含量超过了5%;花色腐乳仅有5个优势真菌属:威克汉姆酵母属(Wickerhamomyces,46.83%),双足囊菌属(39.44%),毛霉属(Mucor,4.30%),半乳糖酵母属(Galactomyces,1.68%),根毛霉属(Rhizomucor,1.32%),而其中4个(威克汉姆酵母属、毛霉属、半乳糖酵母属和根毛霉属)均为特有优势真菌属。花色腐乳和红方腐乳的共有优势菌属仅有双足囊菌属,该属酵母菌能能产生小分子酯和含硫化合物,异丁醇、醛类物质和有机酸等代谢产物[15]。另外,红腐乳的优势属—红曲霉属(32.55%),在花色腐乳中未检出,可能是在红方腐乳生产时使用了原料红曲呈色导致。
腐乳的细菌菌群组成如图3-c所示。红方腐乳中有4个优势菌门:厚壁菌门(Firmicutes,64.25%),变形菌门(Proteobacteria,26.04%),拟杆菌门(Bacteroidetes,7.09%)和放线菌门(Actinobacteria,1.88%);花色腐乳则有3个:变形菌门(62.58%),厚壁菌门(29.28%),拟杆菌门(7.76%)。在属水平上(图3-d),红方腐乳样品有4个菌属的相对含量超过了5%,分别为:乳球菌属(Lactococcus,21.52%),明串珠菌属(Leuconostoc,21.98%),乳杆菌属(Lactobacillus,6.05%)和醋酸杆菌属(Acetobacter,7.21%),其他优势菌属还有:假单胞菌属(Pseudomonas,2.86%),不动杆菌属(Acinetobacter,2.68%),从毛单胞菌属(Comamonas,1.83%),肠球菌属(Enterococcus,2.36%),库特氏菌属(Kurthia,2.11%),Fluviicola(2.91%),魏斯氏菌属(Weissella,1.77%),葡萄球菌属(Staphylococcus,1.55%)和四联球菌属(Tetragenococcus,1.77%)。花色腐乳中相对含量超过5%的优势细菌属为:假单胞菌属(47.66%),乳球菌属(9.97%),乳杆菌属(9.94%),不动杆菌属(6.90%),其他优势属还有从毛单胞菌属(3.11%),肠球菌属(1.84%),库特氏菌属(1.27%),片球菌属(Pediococcus,2.92%),鞘氨醇杆菌属(Sphingobacterium,2.77%),类香味杆菌属(Myroides,2.49%),Wautersiella(1.94%)。花色腐乳与红方腐乳的共有优势菌属为假单胞菌属、乳球菌属、乳杆菌属、不动杆菌属、从毛单胞菌属和肠球菌属。
花色腐乳中的4个特有优势菌属为片球菌属、鞘氨醇杆菌属、类香味杆菌属、Wautersiella,其中的鞘氨醇杆菌属还出现在毛霉型腐乳中[16],而片球菌出现在白溪腐乳中[17],类香味杆菌属和Wautersiella仅出现在农家自制的花色腐乳中[18],但在市售的酱腐乳、白菜腐乳、青腐乳、红方腐乳和白方腐乳以及细菌型腐乳中均未检测到[16-22],因此可以判断类香味杆菌属和Wautersiella是花色腐乳特有的优势细菌属。
值得注意的是,花色腐乳与红方腐乳均无比例超过50%的绝对优势菌属,该结果与前人研究一致[19,21-22]。另外,本研究通过多元统计分析表明,花色腐乳与红方腐乳无论是真菌还是细菌的菌群结构都具有显著差异(图2,P<0.05)。但是2种类型腐乳仍然有6个共有优势细菌属,它们也作为优势细菌属出现在白方腐乳及农家自制的花色腐乳中[18,20]。分析已报道的不同类型腐乳发现,仅乳球菌属是普遍存在于各种腐乳中的优势细菌属[16,20-21]。WAN等[17]通过可培养方法发现,腐乳中的乳球菌为乳酸乳球菌(Lactococcus lactis)。乳酸乳球菌能促进食品风味[23],被美国食品和药物管理局(Food and Drug Administration,FDA)认为是安全的(Generally Recognized As Safe,GRAS)微生物,常用于食品发酵、药物生产和饲料添加等,因此该菌可能是在腐乳生产中具有潜在应用价值的乳酸菌种。
进一步通过LEfSe来分析不同类型红色腐乳中的生物标志物,图4-a和图4-b中的柱长度显示了线性判别值,该值越大,表明其富集程度越高。由图4可知,有3个优势真菌属在红方腐乳中富集,分别为红酵母属、曲霉属和红曲霉属,而毛霉属在花色腐乳中富集。此外,优势细菌属—明串珠菌属在红方腐乳中富集,2个非优势细菌属—糖芽孢杆菌(Saccharibacillus)和克吕沃尔氏菌属(Kluyvera)与优势细菌属—假单胞菌属在花色腐乳中富集。由于糖芽孢杆菌和克吕沃尔氏菌属在花色腐乳中的相对含量低于1%,而毛霉在多种毛霉型腐乳中均有出现,因此红酵母属、曲霉属和明串珠菌属为红方腐乳的生物标志属,而假单胞菌属为花色腐乳的生物标志属。这些市售花色腐乳中出现的生物标志属同样也出现在农家自制的茨河花色腐乳中[18]。
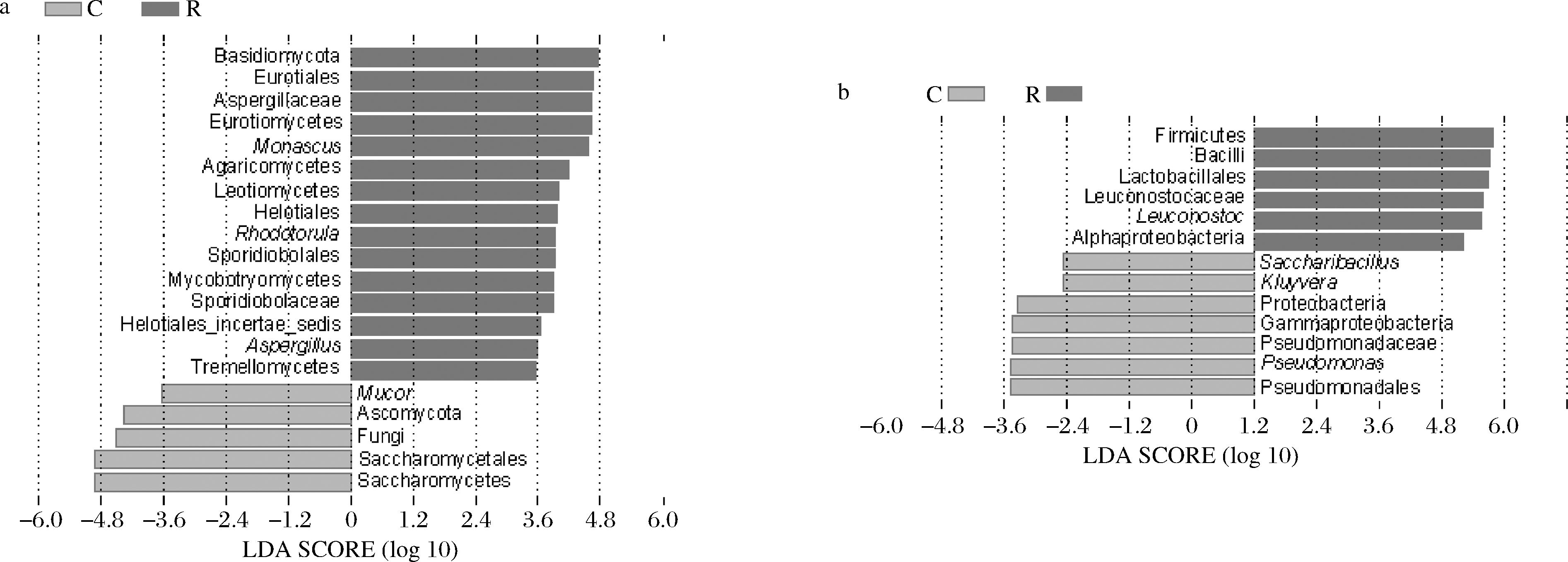
a-真菌;b-细菌
图4 基于LEfSe分析的腐乳微生物LDA分析
Fig.4 Microbial LDA scores plot based on LEfSe analysis
2.2 腐乳VOC分析
花色腐乳与红方腐乳在菌群结构上差异很大,因此进一步使用GC-IMS定性分析2种腐乳的风味化合物,并建立了菌群与气味的关联。由表1可知,共鉴定出30种VOC,包括7种醇类、4种酮类、5种醛类、11种酯类物质,其中酯类物质种类最丰富。
表1 花色腐乳和红方腐乳的VOC鉴定
Table 1 Identification of VOC for red sufu and Huase sufu
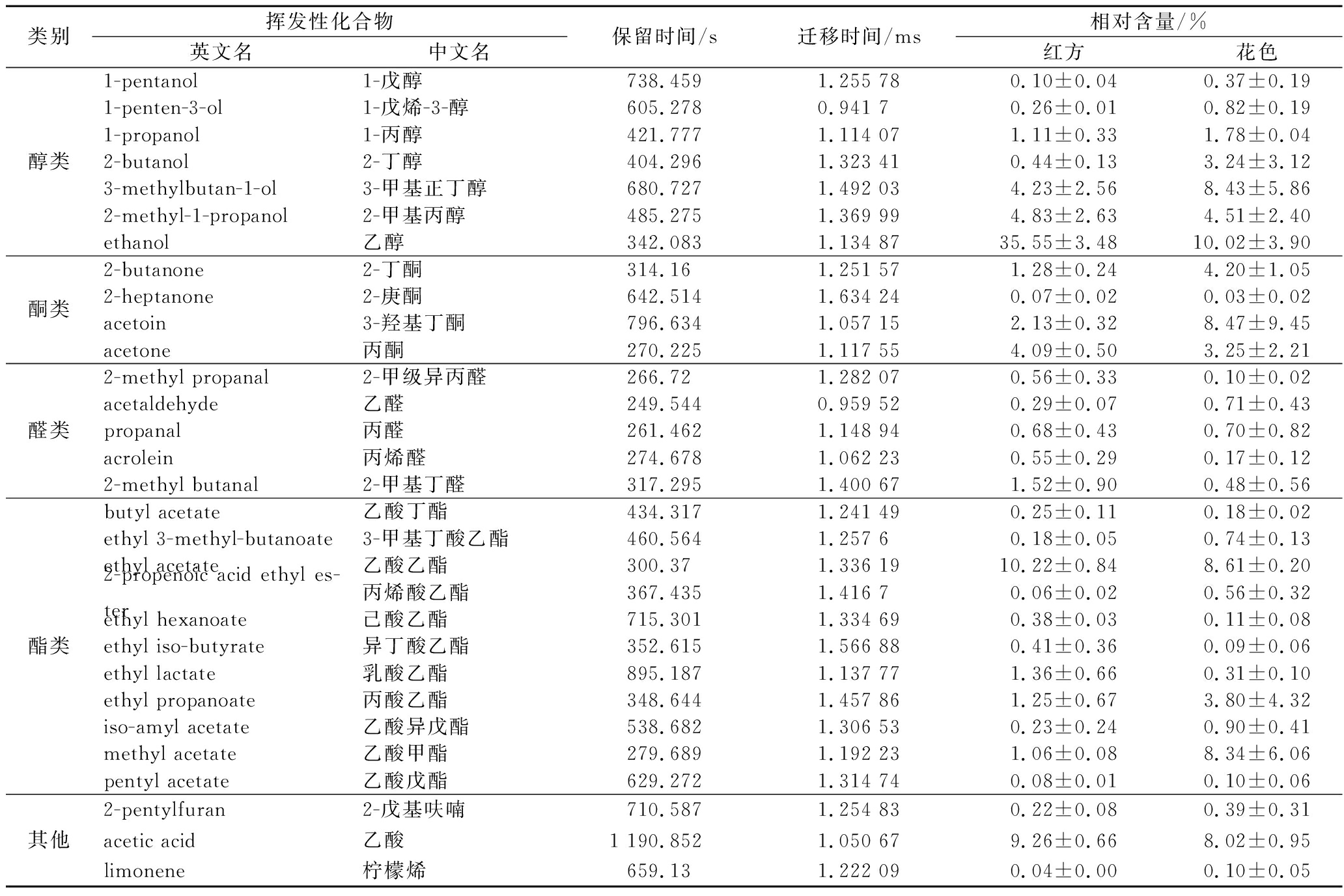
类别挥发性化合物英文名中文名保留时间/s迁移时间/ms相对含量/%红方花色醇类1-pentanol1-戊醇738.4591.255 780.10±0.040.37±0.191-penten-3-ol1-戊烯-3-醇605.2780.941 70.26±0.010.82±0.191-propanol1-丙醇421.7771.114 071.11±0.331.78±0.042-butanol2-丁醇404.2961.323 410.44±0.133.24±3.123-methylbutan-1-ol3-甲基正丁醇680.7271.492 034.23±2.568.43±5.862-methyl-1-propanol2-甲基丙醇485.2751.369 994.83±2.634.51±2.40ethanol乙醇342.0831.134 8735.55±3.4810.02±3.90酮类2-butanone2-丁酮314.16 1.251 571.28±0.244.20±1.052-heptanone2-庚酮642.5141.634 240.07±0.020.03±0.02acetoin3-羟基丁酮796.6341.057 152.13±0.328.47±9.45acetone丙酮270.2251.117 554.09±0.503.25±2.21醛类2-methyl propanal2-甲级异丙醛266.72 1.282 070.56±0.330.10±0.02acetaldehyde乙醛249.5440.959 520.29±0.070.71±0.43propanal丙醛261.4621.148 940.68±0.430.70±0.82acrolein丙烯醛274.6781.062 230.55±0.290.17±0.122-methyl butanal2-甲基丁醛317.2951.400 671.52±0.900.48±0.56酯类butyl acetate乙酸丁酯434.3171.241 490.25±0.110.18±0.02ethyl 3-methyl-butanoate3-甲基丁酸乙酯460.5641.257 60.18±0.050.74±0.13ethyl acetate乙酸乙酯300.371.336 1910.22±0.848.61±0.202-propenoic acid ethyl es-ter丙烯酸乙酯367.4351.416 70.06±0.020.56±0.32ethyl hexanoate己酸乙酯715.3011.334 690.38±0.030.11±0.08ethyl iso-butyrate异丁酸乙酯352.6151.566 880.41±0.360.09±0.06ethyl lactate乳酸乙酯895.1871.137 771.36±0.660.31±0.10ethyl propanoate丙酸乙酯348.6441.457 861.25±0.673.80±4.32iso-amyl acetate乙酸异戊酯538.6821.306 530.23±0.240.90±0.41methyl acetate乙酸甲酯279.6891.192 231.06±0.088.34±6.06pentyl acetate乙酸戊酯629.2721.314 740.08±0.010.10±0.06其他2-pentylfuran2-戊基呋喃710.5871.254 830.22±0.080.39±0.31acetic acid乙酸1 190.8521.050 679.26±0.668.02±0.95limonene柠檬烯659.131.222 090.04±0.000.10±0.05
进一步通过LEfSe分析发现(图5),醛类物质总体上在红方腐乳中富集,而酮类物质在花色腐乳中含量显著较高(P<0.05)。具体来说,这些VOC中的乙醇、2-甲基丁醛、2-甲基异丙醛、乙酸甲酯、异丁酸乙酯和丙烯酸在红方腐乳中显著富集;另外,乙醛、丙烯酸乙酯、柠檬烯、1-丙醇在花色腐乳中显著富集,为这2种腐乳各自的特征VOCs。
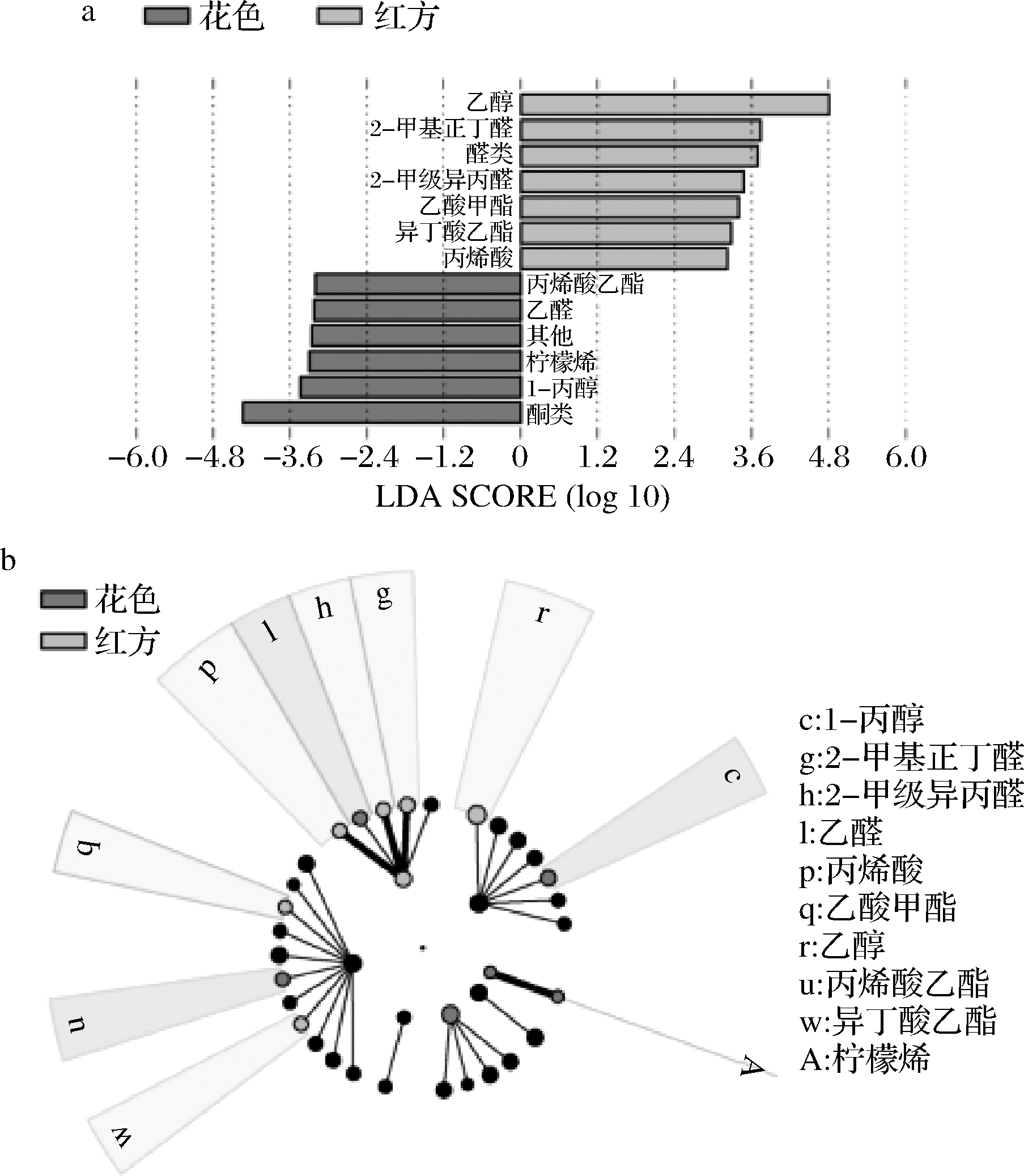
a-LDA得分图;b-LDA分支图
图5 腐乳样品挥发性有机物的LEfSe分析
Fig.5 LEfSe analysis for evaluation of VOCs of sufu samples
2.3 腐乳VOC特征与菌群的关联分析
普氏分析显示,腐乳VOC与真菌菌群显著相关(M2=0.472 4,P=0.041 7),而与细菌不显著相关(P=0.417)。因此,我们进一步建立了真菌与风味物质的关联。
通过图6分析共找到了28对显著相关的菌群-VOC关联(P<0.05)。其中与酯类物质显著相关的真菌属有8个:Chalara,Neobulgaria与异丁酸乙酯显著正相关(P<0.05);丝孢酵母属和地霉属与乳酸乙酯显著正相关(P<0.05);曲霉属与乙酸乙酯显著正相关(P<0.05),同时也与丙烯醛极显著正相关(P<0.01);威克汉姆酵母属与丙烯酸乙酯、乙酸甲酯、乙酸戊酯、丙酸乙酯均呈极显著正相关(P<0.01),而与丙酮显著负相关(P<0.05),还与2-丁酮显著正相关(P<0.05);毛霉和双足囊菌均与乙酸异戊酯呈正相关(P<0.05),还与1-戊醇、3-甲基正丁醇、2-戊基呋喃显著正相关(P<0.05);此外,毛霉还与1-戊烯-3-醇和乙醛均呈显著正相关(P<0.05)。因此,真菌与VOC显示出复杂的关联,特别是VOC的酯类关联程度较强。
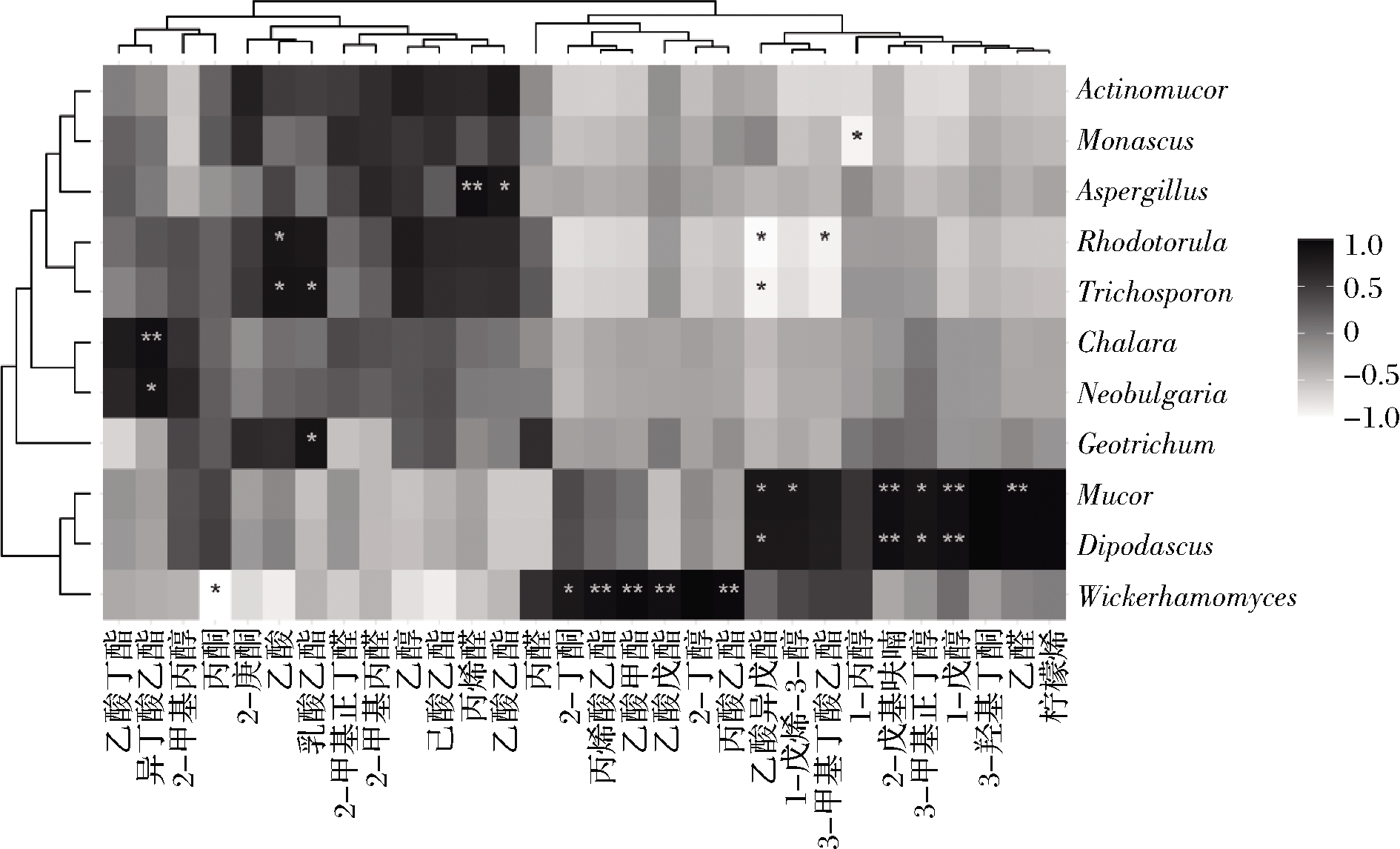
图6 腐乳菌群与VOC关联分析
Fig.6 Correlation analysis between VOC and Huase sufu
酯类物质是发酵类豆制品风味物质的主体成分,小分子的酯类物质具有果香与清香特征[24]。本研究使用GC-IMS检测到的酯类物质种类最多,与以往报道一致[19,25]。乙酸甲酯、异丁酸乙酯在红方腐乳中显著较高(P<0.05),为红方腐乳的特征性风味酯,而丙烯酸乙酯为花色腐乳中的特性风味酯。值得注意的是,威克汉姆酵母属在红方腐乳中为优势属,而在花色腐乳中相对含量低于1%,且该菌属与乙酸甲酯、丙烯酸乙酯显著正相关(P<0.05)。研究显示,威克汉姆酵母属能从啤酒、酸面团、水果、清酒、葡萄酒等发酵食品中分离出,能合成小分子酯类物质,因此推测,威克汉姆酵母属可能与这2种风味物质有直接关系,或者间接地促进了它们的形成。此外,威克汉姆酵母还能产生糖苷酶,从风味物质前体中释放小分子风味物质,产生蛋白酶,有助于防止葡萄酒等酿造酒浑浊,还能控制果酒腐败[26-27]。红方腐乳特征风味物质异丁酸乙酯的形成可能与优势属Chalara和Neobulgaria相关联。总的来看,与威克汉姆酵母属以及腐乳初发酵阶段的重要微生物毛霉呈正相关的酯类风味物质最多,因此推测它们在腐乳生产中具有较大的开发和应用潜力,可以进一步筛选,用于腐乳生产。
3 结论
茨河花色腐乳的细菌与真菌菌群组成差异显著,前者的生物标志属为假单胞菌属,而红方腐乳的为红酵母属、曲霉属和明串珠菌属。尽管2种腐乳的菌群差异较大,但仍然有假单胞菌属、乳球菌属、乳杆菌属、不动杆菌属、从毛单胞菌属和肠球菌属为共有优势细菌属,其中的乳球菌不仅出现在乳制品、花色与红方腐乳中,在其他类型的腐乳中也有检测到,是极具潜在开发能力的菌种资源。茨河花色腐乳与红方腐乳的共有优势真菌属仅有双足囊菌属。2种腐乳均含有丰富的VOC,其中小分子酯类VOC种类最多。进一步分析发现,醛类物质在红方腐乳中富集,而酮类物质在花色腐乳中富集,红方腐乳的特征风味物质为乙醇、2-甲基丁醛、2-甲基异丙醛等,而花色腐乳的特征风味物质为乙醛、丙烯酸乙酯、柠檬烯等。另外还发现威克汉姆酵母属和毛霉属可能参与并促进了腐乳的酯类风味物质的产生,具有进一步在腐乳生产中应用的潜力。
[1] HAN B Z, ROMBOUTS F M, NOUT M J.A Chinese fermented soybean food[J].International Journal of Food Microbiology, 2001, 65(1-2):1-10.
[2] WANG P, WANG W P, XU D D, et al.Analysis of taste compounds in red sufu during fermentation and discrimination with electronic tongue[J].食品科学, 2021, 42(14):170-179.
WANG P, WANG W P, XU D D, et al.Analysis of taste compounds in red sufu during fermentation and discrimination with electronic tongue[J].Food Science, 2021, 42(14):170-179.
[3] 张兰威. 发酵食品工艺学[M].北京:中国轻工业出版社, 2011.
ZHANG L W.Fermented Food Technology[M].Beijing:China Light Industry Press, 2011.
[4] 周袁璐, 师苑, 陆跃乐, 等.传统发酵过程微生物互作研究进展[J].中国食品学报, 2021, 21(11):349-358.
ZHOU Y L, SHI Y, LU Y L, et al.Research progress on microbial interaction in traditional fermentation[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(11):349-358.
[5] 夏梦雷, 杨帆, 陆锴, 等.高通量测序技术在传统发酵食品微生物群落中的应用研究[J].中国酿造, 2021, 40(5):1-7.
XIA M L, YANG F, LU K, et al.Advances in the application of high-throughput sequencing technology in traditional fermented foods[J].China Brewing, 2021, 40(5):1-7.
[6] JAGADEESAN B, GERNER-SMIDT P, ALLARD M W, et al.The use of next generation sequencing for improving food safety:Translation into practice[J].Food Microbiology, 2019, 79:96-115.
[7] COLE J R, CHAI B, FARRIS R J, et al.The ribosomal database project (RDP-II):Introducing myRDP space and quality controlled public data[J].Nucleic Acids Research, 2006, 35(suppl_1):D169-D172.
[8] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al.Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Applied and Environmental Microbiology, 2006, 72(7):5 069-5 072.
[9] QUAST C, PRUESSE E, YILMAZ P, et al.The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools[J].Nucleic Acids Research, 2012, 41(D1):D590-D596.
[10] 赵恒,陈富安,赵慧君,等.茨河腐乳细菌与真菌多样性及其功能关联分析[J].食品与发酵工业,2022,48(10):213-219.
ZHAO H, CHEN F A, ZHAO H J, et al.Diversity and functional correlation analysis of bacteria and fungi in Huase sufu[J].Food and Fermentation Industries, 2022,48(10):213-219.
[11] QIU X Y, YU L L, WANG W Y, et al.Comparative evaluation of microbiota dynamics and metabolites correlation between spontaneous and inoculated fermentations of Nanfeng tangerine wine[J].Frontiers in Microbiology, 2021, 12:649978.
[12] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[13] EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics, 2011, 27(16):2 194-2 200.
[14] K LJALG U, NILSSON R H, ABARENKOV K, et al.Towards a unified paradigm for sequence-based identification of fungi[J].Molecular Ecology, 2013, 22(21):5 271-5 277.
LJALG U, NILSSON R H, ABARENKOV K, et al.Towards a unified paradigm for sequence-based identification of fungi[J].Molecular Ecology, 2013, 22(21):5 271-5 277.
[15] KURYLENKO O O, RUCHALA J, DMYTRUK K V, et al.Multinuclear yeast Magnusiomyces (Dipodascus, Endomyces) magnusii is a promising isobutanol producer[J].Biotechnology Journal, 2020, 15(7):1900490.
[16] XIE C, ZENG H, WANG C, et al.Volatile flavour components, microbiota and their correlations in different sufu, a Chinese fermented soybean food[J].Journal of Applied Microbiology, 2018, 125(6):1 761-1 773.
[17] WAN H F, LIU T, SU C W, et al.Evaluation of bacterial and fungal communities during the fermentation of Baixi sufu, a traditional spicy fermented bean curd[J].Journal of the Science of Food and Agriculture, 2020, 100(4):1 448-1 457.
[18] ZHANG Z D, WANG Y R, XIANG F S, et al.Distinct bacterial community of a solid-state fermented Chinese traditional food huase sufu revealed by high-throughput sequencing[J].Food Science and Biotechnology, 2021, 30(9):1 233-1 241.
[19] 陈卓, 吴学凤, 穆冬冬, 等.红腐乳后酵期风味物质与细菌菌群分析[J].食品科学, 2021, 42(6):118-125.
CHEN Z, WU X F, MU D D, et al.Determination and correlation analysis of flavor components with bacterial community in post-fermented red sufu[J].Food Science, 2021, 42(6):118-125.
[20] FEI Y T, LI L, CHEN L Y, et al.High-throughput sequencing and culture-based approaches to analyze microbial diversity associated with chemical changes in naturally fermented tofu whey, a traditional Chinese tofu-coagulant[J].Food Microbiology, 2018, 76:69-77.
[21] TAN G L, HU M, LI X Y, et al.High-throughput sequencing and metabolomics reveal differences in bacterial diversity and metabolites between red and white sufu[J].Frontiers in Microbiology, 2020, 11:758.
[22] 徐琼, 刘洋, 曲勤凤, 等.高通量测序分析不同地区红腐乳细菌多样性[J].食品科学, 2020, 41(10):110-116.
XU Q, LIU Y, QU Q F, et al.High-throughput sequencing analysis of bacterial diversity in red sufu from different regions[J].Food Science, 2020, 41(10):110-116.
[23] WHETSTINE M E C, DRAKE M A, BROADBENT J R, et al.Enhanced nutty flavor formation in cheddar cheese made with a malty Lactococcus lactis adjunct culture[J].Journal of Dairy Science, 2006, 89(9):3 277-3 284.
[24] 李俊娥, 毛亚玲, 祝霞, 等.苹果酸-乳酸发酵接种方式对赤霞珠干红葡萄酒香气品质的影响[J].食品与发酵工业, 2021, 47(19):126-136.
LI J E, MAO Y L, ZHU X, et al.Effects of different inoculation on aroma quality of Cabernet Sauvignon dry red wine[J].Food and Fermentation Industries, 2021, 47(19):126-136.
[25] 张雅婷, 孙娜, 于寒松, 等.红腐乳发酵过程中菌群结构与风味相关性研究进展[J].食品与发酵工业, 2020, 46(1):287-294.
ZHANG Y T, SUN N, YU H S, et al.Advances in research on correlation between flora structure and product flavor during red sufu fermentation[J].Food and Fermentation Industries, 2020, 46(1):287-294.
[26] KUMAR VERMA D, THYAB GDDOA AL-SAHLANY S, KAREEM NIAMAH A, et al.Recent trends in microbial flavour compounds:A review on chemistry, synthesis mechanism and their application in food[J].Saudi Journal of Biological Sciences, 2022, 29(3):1 565-1 576.
[27] PADILLA B, GIL J, MANZANARES P.Challenges of the non-conventional yeast Wickerhamomyces anomalus in winemaking[J].Fermentation, 2018, 4(3):68.