酸笋是以新鲜竹笋为主要原料,经过发酵而成的一类传统泡菜,富含纤维素和多种矿物质元素[1]。目前,除了对其风味物质有研究外,部分学者还针对其微生物类群方面展开了研究。孙宁等[2]使用MRS培养基从自然发酵的酸笋中分离得到了69 株乳酸菌,分别隶属于乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)和链球菌属(Streptococcus);陈正培等[3]对柳州酸笋中具有降解亚硝酸盐特性的乳酸菌展开了筛选工作,得到了1 株亚硝酸盐降解率达76.58%(亚硝酸盐质量浓度为625 mg/mL)的植物乳杆菌(Lactobacillus plantarum);周金沙等[4]采用Illumina MiSeq测序技术解析广西无盐发酵酸笋的细菌多样性,发现乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)和魏斯氏菌属(Weissella)为优势菌属。上述研究均表明酸笋中乳酸菌类群含量较高。然而目前的研究大多仅从纯培养方式或测序技术单方面来进行解析,而更全面地揭示酸笋中细菌及乳酸菌类群的研究较为少见。
酸笋是南宁最知名的小吃“老友粉”中必不可少的特色调味料,因而解析南宁地区酸笋的细菌类群具有一定的代表性。当地传统酸笋的做法通常是将新鲜竹笋切成块状、条状或丝状后,用凉白开或泉水直接浸泡,不添加食盐及其他调味料,经过15~30 d密封发酵即可[5]。本研究在采用Illumina MiSeq高通量测序技术对南宁地区酸笋细菌类群结构进行解析的基础上,进一步预测其细菌类群的功能特性,并结合纯培养方式对酸笋中的乳酸菌菌株进行分离、鉴定和保存。以期在相关产业对酸笋发酵条件进行优化的过程中提供参考依据和菌株支持。
1 材料与分析
1.1 材料与试剂
5 个酸笋样品均于2019年8月采集自广西壮族自治区南宁市(E 108°22′,N 22°49′)北湖、淡村和五里亭菜市场,所有样品均为当地农户手工制作,编号依次为SS1~SS5。使用无菌自封袋包装样品并贴上标签置于保温采样箱中运送回实验室。
基因组提取试剂盒,德国QIAGEN公司;338F/806R和27F/1495R正反向引物,武汉天一辉远生物科技有限公司;DNA聚合酶、rTaq酶、10×Buffer和dNTP,宝生物工程(大连)有限公司;MRS和LB琼脂培养基,青岛海博生物技术有限公司。
1.2 主要仪器设备
Veriti FAST梯度PCR仪,美国ABI公司;UVPCDS8000凝胶成像分析系统,美国Protein Simple公司;Illumina MiSeq PE250高通量测序平台,美国Illumina公司;R930型机架式服务器,美国DELL公司;DG250型厌氧工作站,英国Don Whitley公司。
1.3 实验方法
1.3.1 宏基因组DNA的提取、PCR扩增和高通量测序
酸笋宏基因组DNA的提取:5份样品各称取2 g,参照提取试剂盒说明书提取酸笋宏基因组DNA。
PCR扩增和高通量测序:针对检测合格的基因16S rRNA V3~V4区域进行PCR扩增和清洁。为方便后续进行序列配对,在正反引物中添加1组barcode(核苷酸标签,含7个碱基),PCR扩增体系和程序均参照王强等[6]的方法进行操作,将纯化后的产物寄至上海美吉生物医药科技有限公司完成测序。
1.3.2 序列质控和生物信息学分析
参照崔梦君等[7]的方法对测序返回的fq序列文件进行质控,质控合格的序列上传至QIIME(v1.9.1)分析平台进行生物信息学分析。分别按照100%和97%相似度对有效序列进行序列归并和构建分类操作单元(operational taxonomic units,OTU)[8],同时将含有嵌合体序列的OTU进行删除[9],选择余下OTU代表性序列在RDP[10]、SILVA[11]和Greengenes[12]数据库中进行序列比对,并注释其分类学地位。在此基础上,进一步使用PICRUSt软件预测酸笋细菌的基因功能[13],参照蛋白质直系同源簇数据库(clusters of orthologous groups of proteins,COG)进行功能注释[14]。
1.3.3 乳酸菌的分离鉴定
称取10 g粉碎后的酸笋添加到90 mL生理盐水(高温高压蒸汽灭菌)中混合均匀,37 ℃,220 r/min的摇床中振荡30 min。参照倪慧等[15]的方法对酸笋中乳酸菌菌株进行初步分离、纯化与鉴定,并对过氧化氢酶阴性且革兰氏阳性的菌株进行保藏。菌株鉴定流程:(1)菌株DNA提取;(2)PCR扩增;(3)琼脂糖凝胶检测扩增产物;(4)PCR产物清洁;(5)连接转化;(6)挑取阳性克隆子。将扩增成功的阳性克隆子送至天一辉远(武汉)生物科技有限公司进行测序,返回的序列在NCBI网站进行比对[16],并完成系统发育树的构建。
1.3.4 数据处理
使用Origin 2017软件绘制曲线图与柱形图,使用R(v4.0.1)软件绘制菌群功能热图,使用在线作图网址(http://jvenn.toulouse.inra.fr/app/example.html)绘制Venn图,使用MEGA7.0软件构建系统发育树。
2 结果与分析
2.1 样本测序数据统计分析
纳入本研究的5个酸笋样品经测序共得到219 111条高质量序列,平均每个样品含有43 822条序列。本研究首先基于香农指数曲线和等级聚类曲线分析了样本测序深度及物种丰富度(图1)。

a-香农指数;b-等级聚类
图1 香农指数曲线和等级聚类曲线
Fig.1 The curves of Shannon index and rank abundance
样品SS1~SS5分别测得48 177、34 600、45 896、38 194和52 244条高质量序列,且当测序深度达到10 000条左右时,所有样品的香农指数曲线就均已到达平台期(图1-a),表明若测序深度继续增加,酸笋的细菌多样性不会再发生较大变化,说明本研究的测序深度合理,能够满足后续分析要求。样品SS5的曲线最平缓且跨度最大,样品SS2的曲线最陡峭且跨度最小(图1-b)。表明相较于其他样品,样品SS5的物种丰富度最高也最均匀,样品SS2的物种丰富度最低,不同样品在物种丰富度上呈现出一定的差别。
2.2 基于细菌门和属水平细菌多样性分析
本研究进一步基于门和属水平对酸笋的细菌类群进行分析,5个酸笋样品中的细菌类群共鉴定到了8个门和77个属,其中平均相对含量>0.5%的门和属见图2。酸笋中含有2个平均相对含量>0.5%的门,分别为硬壁菌门(Firmicutes,99.05%)和放线菌门(Actinobacteria,0.53%)(图2-a)。其中Actinobacteria在除SS1(0.65%)和SS5(1.74%)外的其他样品中占比均<0.25%。显然,Firmicutes为酸笋中的优势门。乳杆菌属(Lactobacillus)为酸笋中唯一的平均相对含量>0.5%的细菌属,其平均相对含量高达98.41%。除此之外,在样品SS4中相对含量>0.5%的属还包括魏斯代菌属(Weissella,0.84%),样品SS5中包括棒状杆菌属(Corynebacterium,0.65%)和异常球菌属(Deinococcus,0.58%)(图2-b)。由此可见,酸笋中的细菌类群主要为隶属于Firmicutes的Lactobacillus。陈晓东等[17]采用第2代测序技术解析广西壮族自治区柳州市和桂林市等6个地区的酸笋样品时得到了类似的结论。而鲍伟等[18]在使用同种方法解析浙江传统发酵蔬菜(如臭冬瓜、臭苋菜梗、酸笋和酸茭白等)的微生物类群过程中却发现,酸笋汁和酸笋固形物中的优势细菌属分别为克雷伯氏菌属(Klebsiella)和埃希菌属(Escherichia)。这表明不同地域,其环境等因素的不同可能导致当地酸笋中微生物类群亦有较大的差别,因而解析不同地区的酸笋细菌类群有助于全面认识酸笋中的微生物种类及特征。
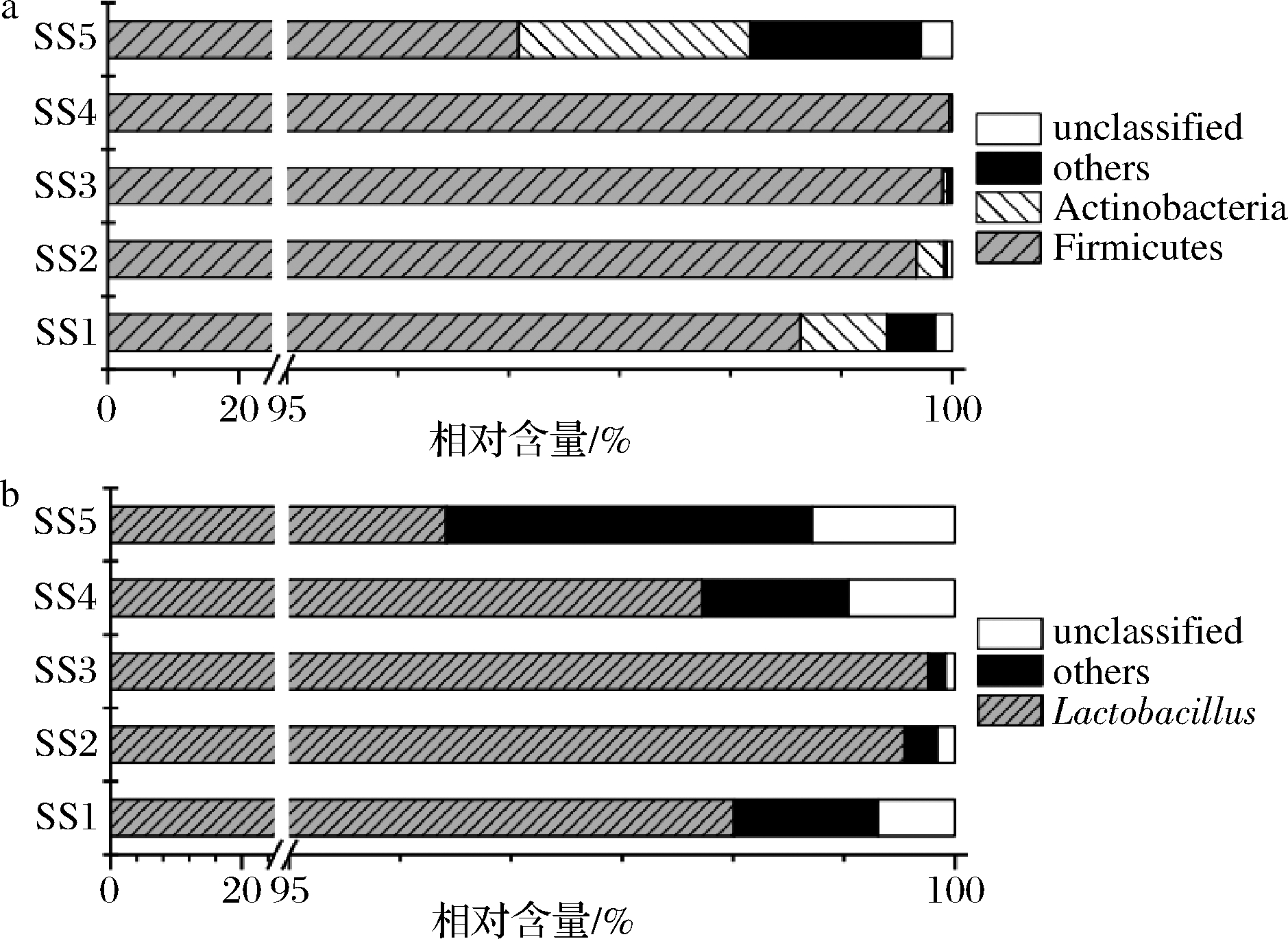
a-细菌门;b-细菌属
图2 酸笋中相对含量>0.5%的细菌门和细菌属
Fig.2 The phylum and genus with relative abundance more than 0.5% in fermented bamboo shoots
2.3 基于OTU水平细菌类群分析
本研究进一步从OTU水平来解析酸笋中的细菌类群,并利用韦恩图展示各样品中OTU的分布情况(图3)。检测出的全部有效序列经97%相似度划分得到1 333个OTU,每个样品平均包含533个OTU,样品SS1~SS5中分别含有547、237、421、425和1 037个OTU,特有OTU数分别为68、9、20、92和355个,包含的序列数占总序列数的比例依次为0.39%、0.05%、0.10%、1.14%和3.95%,而5个样品中共有的OTU数量为65个,所包含的序列数占总序列数的90.63%。由此可见,尽管不同样品间共有的物种种类并不多,但其在数量上却占到了极大的优势。

图3 各酸笋样品中的OTU分布韦恩图
Fig.3 OTU Venn diagram of fermented bamboo shoots
所有样品共同含有的平均相对含量>0.5%的核心OTU共有6个,分别为OTU4296、OTU5648、OTU1388、OTU220、OTU4815和OTU2404,平均相对含量分别为79.99%、1.96%、0.95%、0.78%、0.70%和0.68%(图4)。
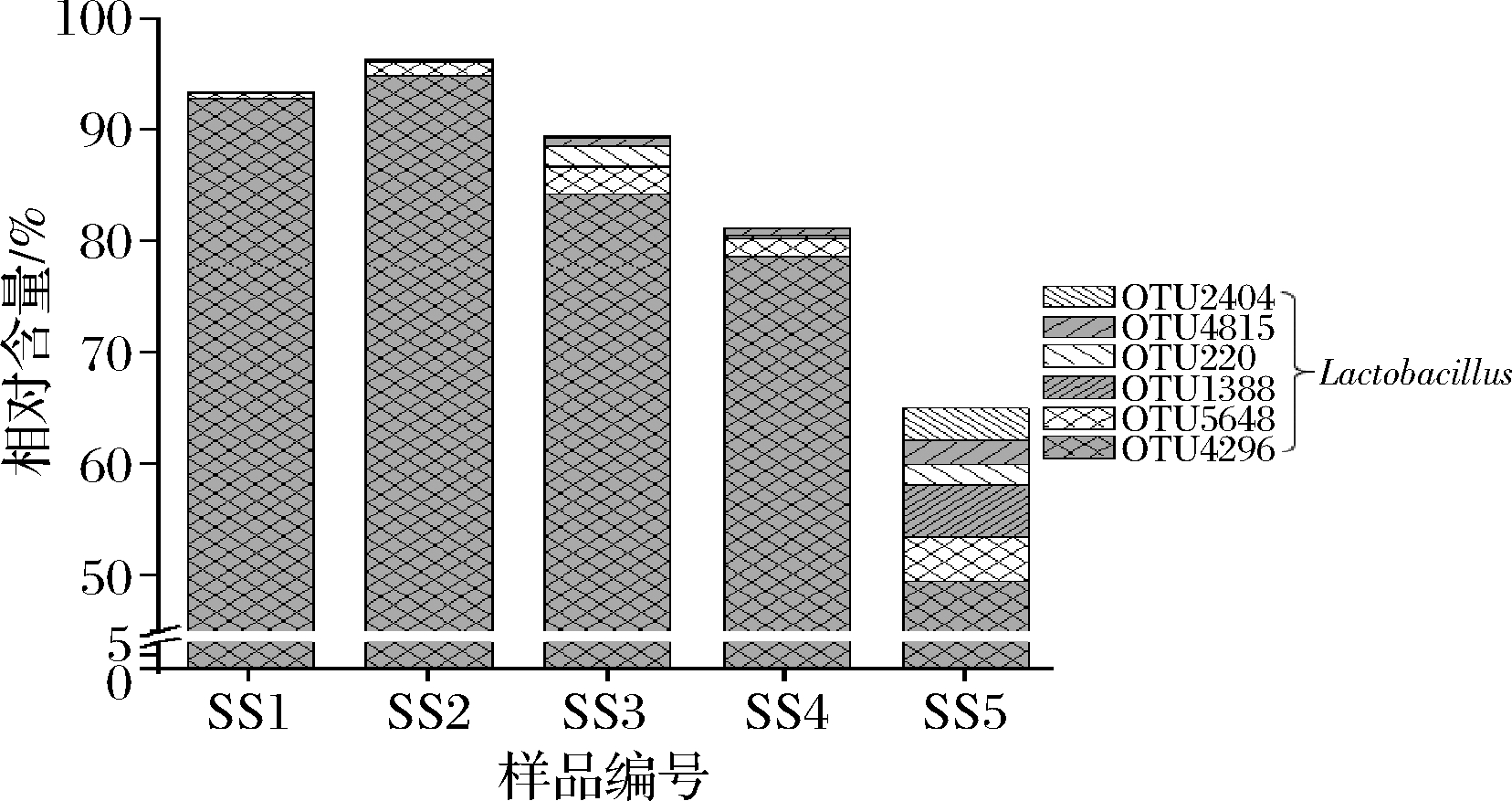
图4 平均相对含量>0.5%的核心OTU
Fig.4 Analysis of core OTU with average relative abundance more than 0.5%
值得注意的是,上述6个OTU均隶属于Lactobacillus,累计相对含量达85.07%,这一结果表明南宁地区酸笋中的核心细菌类群为Lactobacillus。有研究指出,隶属于Lactobacillus的部分种,例如L.plantarum所产生的乳酸和乙酸等能够破坏细菌的细胞膜从而产生抑菌效果,这一特性可能对酸笋的食用安全品质起到了一定的积极作用[19]。
2.4 酸笋细菌类群的功能预测分析
在对酸笋细菌类群结构进行解析的基础上,本研究还预测了其细菌类群所具有的潜在功能,全部有效序列通过PICRUSt预测共得到4 045个COG,分别注释到了22个功能大类(图5)。酸笋中的细菌类群除了在翻译、转录和细胞膜生物发生等维持细胞自身生长繁殖的功能上具有较高的表达以外,在氨基酸和碳水化合物的转运与代谢上也具有较高表达。这可能是由于蛋白质和糖类常被作为微生物发酵的营养物质来源,而微生物类群在消耗营养物质的过程中会引发一系列的产物转运和代谢反应。朱照华[20]比较了新鲜与发酵过的毛竹笋(后面统称为酸笋)的营养成分,发现酸笋中的蛋白质和总糖含量相对新鲜竹笋大幅减少,而膳食纤维含量有所上升,这表明酸笋中的微生物类群对其本身营养物质的含量变化有着较大的推动作用。
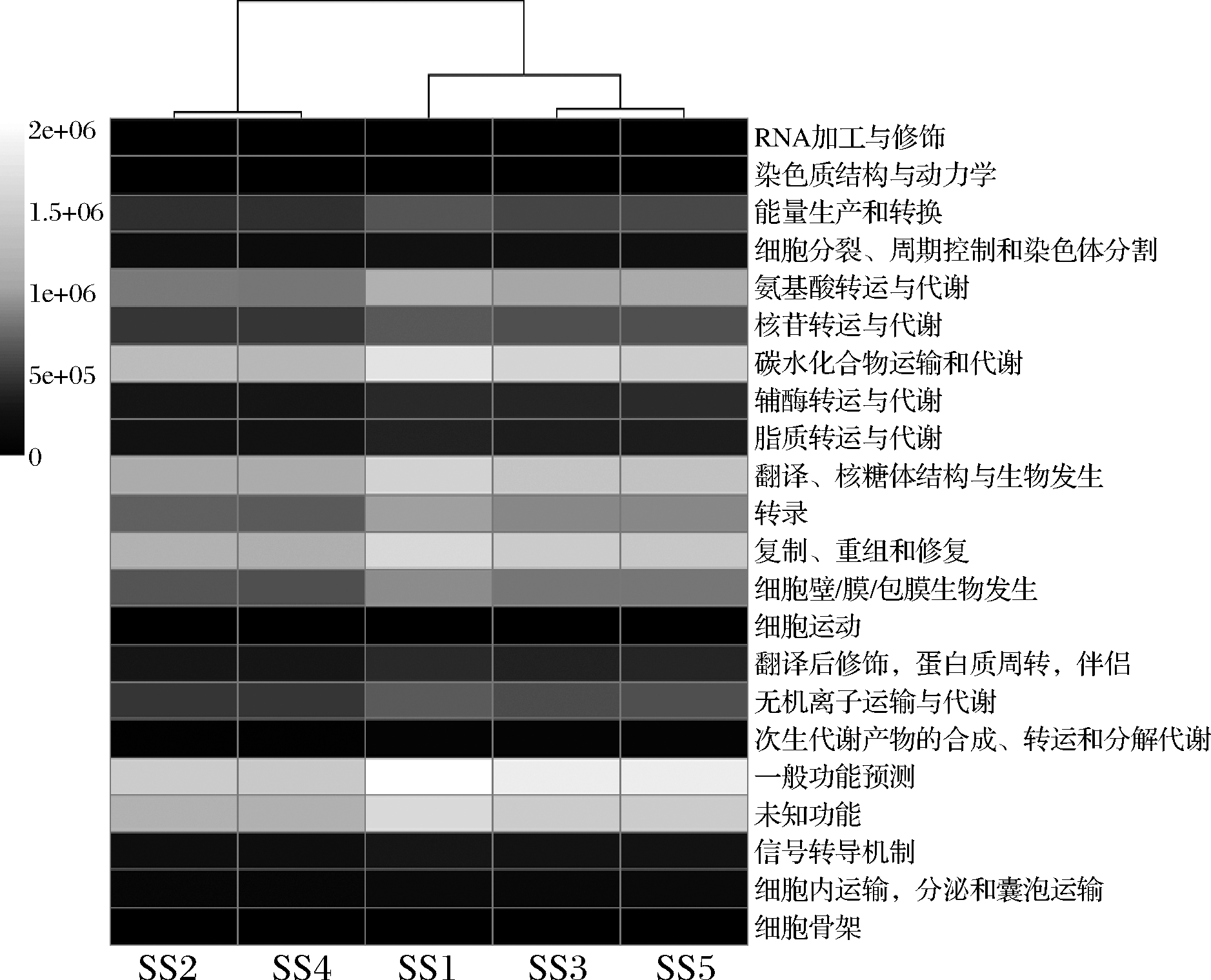
图5 不同酸笋中细菌类群基因功能分析热图
Fig.5 Heat map of gene function analysis of bacterial communities in different fermented bamboo shoots
2.5 乳酸菌多样性分析
测序结果显示酸笋中乳酸菌含量丰富,为进一步明确其中乳酸菌类群,本研究结合了传统微生物学方法对其乳酸菌进行了选择性培养。不同分离株的菌落形态如表1所示,分离株的菌落形态均为圆形,表面光滑;过氧化氢酶多为阴性,革兰氏染色均为阳性,镜检发现其细胞形状均为杆状。
从酸笋中分离的菌株SS-1、SS-2、SS-3和SS-4分别被鉴定为发酵乳杆菌(L.fermentum)、黑氏乳杆菌(L.hammesii)、沙克乳杆菌(L.sakei)和副蕈状芽胞杆菌(Bacillus paramycoides),隶属于Lactobacillus的分离株含量占到75%(图6)。
表1 不同分离株的形态学特征
Table 1 Morphological characteristics of different strains
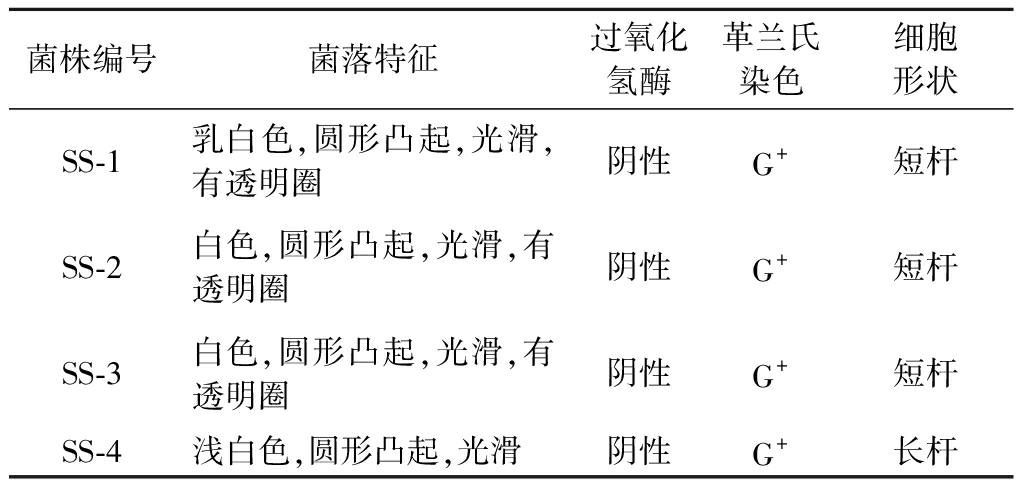
菌株编号菌落特征过氧化氢酶革兰氏染色细胞形状SS-1乳白色,圆形凸起,光滑,有透明圈阴性G+短杆SS-2白色,圆形凸起,光滑,有透明圈阴性G+短杆SS-3白色,圆形凸起,光滑,有透明圈阴性G+短杆SS-4浅白色,圆形凸起,光滑阴性G+长杆
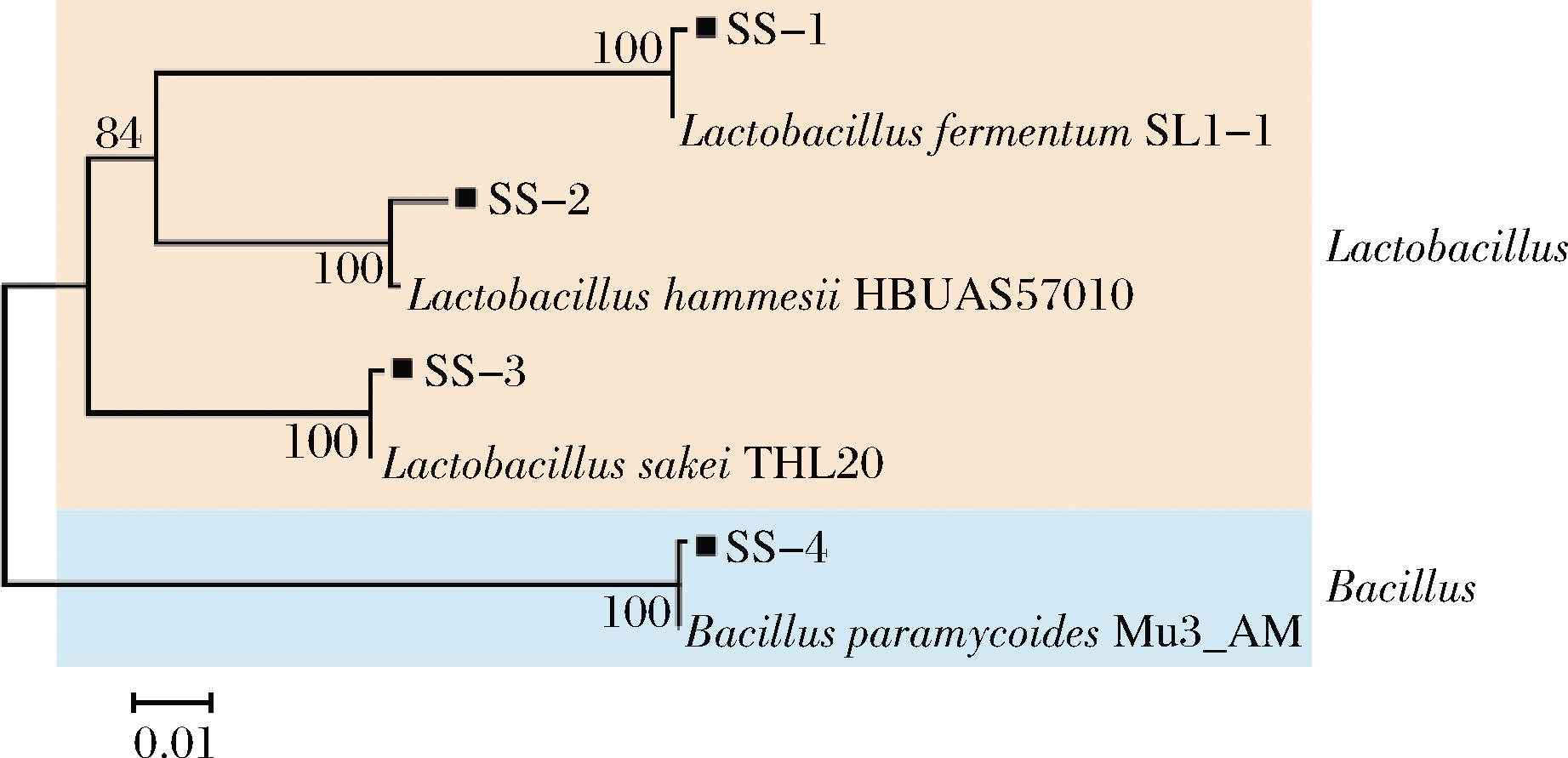
图6 酸笋中分离株系统发育树
Fig.6 Phylogenetic tree of strains isolated from fermented bamboo shoots
MiSeq高通量测序结果显示Lactobacillus在酸笋中的平均相对含量高达98.41%,但通过纯培养却只分离出3株乳杆菌。导致这种数量上的差异原因可能有2个:一是由于即使样品基质中的微生物已经失去活性或者死亡,但只要其DNA没有降解,MiSeq高通量测序技术就仍可以实现序列扩增[21];另一个可能是MRS培养基成分中缺少了酸笋中大部分乳酸菌所必需的营养元素,使其无法被培养出来。因而在后续研究中可以考虑采用培养组学的方法对酸笋中乳酸菌进行分离培养。
除此之外,测序结果并未显示出酸笋中含有芽胞杆菌属,但在纯培养过程中却分离出了1株芽胞杆菌。原因可能为芽胞杆菌属是空气中的常见菌属之一,样品在预处理过程中接触了外界空气,从而使其进入样品,继而被分离出来。
3 结论
MiSeq高通量测序结果显示南宁地区酸笋的细菌类群主要隶属于Lactobacillus,且在氨基酸和碳水化合物的转运与代谢功能上有着较高表达;同时乳酸菌分离鉴定结果显示分离株亦主要隶属于Lactobacillus。可见广西南宁地区的酸笋中细菌菌群主要为乳杆菌。
[1] THAKUR K, RAJANI C S, TOMAR S K, et al.Fermented bamboo shoots:A rich niche for bioprospecting lactic acid bacteria[J].Journal of Bacteriology Mycology, 2016, 3(4):1034.
[2] 孙宁, 雷敬玲, 吴晓青, 等.自然发酵酸笋中乳酸菌的筛选鉴定及益生特性研究[J].中国酿造, 2020, 39(6):63-68.
SUN N, LEI J L, WU X Q, et al.Screening, identification and probiotic characteristics of lactic acid bacteria from natural fermented sour bamboo shoots[J].China Brewing, 2020, 39(6):63-68.
[3] 陈正培, 蒋潮, 夏娴润, 等.柳州酸笋中降亚硝酸盐乳酸菌的筛选及鉴定[J].中国酿造, 2020, 39(3):26-31.
CHEN Z P, JIANG C, XIA X R, et al.Screening and identification of nitrite-degrading lactic acid bacteria from Liuzhou sour bamboo shoots[J].China Brewing, 2020, 39(3):26-31.
[4] 周金沙, 陈晓艺, 谭金萍, 等.基于Illumina MiSeq高通量测序技术分析广西无盐发酵酸笋中细菌多样性[J].中国酿造, 2019, 38(7):85-90.
ZHOU J S, CHEN X Y, TAN J P, et al.Analysis of bacterial diversity in Guangxi non-salt fermentation bamboo shoots based on Illumina MiSeq sequencing technology[J].China Brewing, 2019, 38(7):85-90.
[5] 郑文迪, 关倩倩, 刘长根, 等.基于GC-MS法对比广西地区酸菜和酸笋风味的差异[J].食品与发酵工业, 2020, 46(4):253-257.
ZHENG W D, GUAN Q Q, LIU Z G, et al.Comparison of flavors of Suancai and Suansun in Guangxi based on GC-MS[J].Food and Fermentation Industries, 2020, 46(4):253-257.
[6] 王强, 王玉荣, 陈江红, 等.龙山豆豉细菌多样性分析及其与当阳豆豉差异性比较[J].中国酿造, 2020, 39(1):26-30.
WANG Q, WANG Y R, CHEN J H, et al.Bacterial diversity in Longshan Douchi and differences comparison with Dangyang Douchi[J].China Brewing, 2020, 39(1):26-30.
[7] 崔梦君, 王玉荣, 葛东颖, 等.遵义地区莽椒细菌多样性及PICRUSt基因功能预测分析[J].食品与发酵工业, 2020, 46(4):106-112.
CUI M J, WANG Y R, GE D Y, et al.Study on bacterial diversity of Mangjiao in Zunyi region and PICRUSt-based predicted metagenomic analysis[J].Food and Fermentation Industries, 2020, 46(4):106-112.
[8] CALLAHAN B J, MCMURDIE P J, HOLMES S P.Exact sequence variants should replace operational taxonomic units in marker-gene data analysis[J].The ISME Journal, 2017, 11(12):2 639-2 643.
[9] HAAS B J, GEVERS D, EARL A M, et al.Chimeric 16S rRNA sequence formation and detection in sanger and 454-pyrosequenced PCR amplicons[J].Genome Research, 2011, 21(3):494-504.
[10] COLE J R, WANG Q, FISH J A, et al.Ribosomal database project:Data and tools for high throughput rRNA analysis[J].Nucleic Acids Research, 2014, 42(D1):D633-D642.
[11] YILMAZ P, PARFREY L W, YARZA P, et al.The SILVA and “all-species living tree project (LTP)” taxonomic frameworks[J].Nucleic Acids Research, 2014, 42(D1):D643-D648.
[12] ANUKAM K.Effects of ampicillin on the gut microbiome of an adult male as determined by 16S rRNA V4 metagenomics sequencing and greengenes bioinformatics suite[J].Journal of Advances in Microbiology, 2017, 7(4):1-18.
[13] AGRAWAL S, KINH C T, SCHWARTZ T, et al.Determining uncertainties in PICRUSt analysis-an easy approach for autotrophic nitrogen removal[J].Biochemical Engineering Journal, 2019, 152:107328.
[14] LANGILLE M G I, ZANEVELD J, CAPORASO J G, et al.Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J].Nature Biotechnology, 2013, 31(9):814-821.
[15] 倪慧, 王强, 魏冰倩, 等.恩施市泡萝卜中乳酸菌的分离鉴定及其对品质的影响[J].食品工业科技, 2019, 40(17):64-68;78.
NI H, WANG Q, WEI B Q, et al.Isolation and identification of lactic acid bacteria from pickled radish in Enshi and its effects on quality[J].Science and Technology of Food Industry, 2019, 40(17):64-68;78.
[16] PRUITT K D, TATUSOVA T, MAGLOTT D R.NCBI reference sequence (RefSeq):A curated non-redundant sequence database of genomes, transcripts and proteins[J].Nucleic Acids Research, 2007, 35(D1):D61-D65.
[17] 陈晓东, 朱志勇, 张韫, 等.基于宏基因组技术分析传统酸笋中微生物多样性[J].食品与生物技术学报, 2020, 39(8):89-98.
CHEN X D, ZHU Z Y, ZHANG Y, et al.Microbial community diversity analysis of traditional fermented bamboo shoots using metagenomic technology[J].Journal of Food Science and Biotechnology, 2020, 39(8):89-98.
[18] 鲍伟, 韩姣姣, 张旨轩, 等.基于高通量测序技术对浙江传统发酵蔬菜微生物多样性的解析[J].食品科学, 2021, 42(6):178-185.
BAO W, HAN J J, ZHANG Z X, et al.Analysis of microbial community diversity in traditional fermented vegetables in Zhejiang using high-throughput sequencing[J].Food Science, 2021, 42(6):178-185.
[19] MAO Y, ZHANG X J, XU Z H.Identification of antibacterial substances of Lactobacillus plantarum DY-6 for bacteriostatic action[J].Food Science & Nutrition, 2020, 8(6):2 854-2 863.
[20] 朱照华. 酸笋的营养成分检测及其主要风味物质的研究[D].南宁:广西大学, 2014.
ZHU Z H.Study on fermented bamboo shoot nutrition and flavor components[D].Nanning:Guangxi University, 2014.
[21] TIAN S N, TIAN Z, YANG H, et al.Detection of viable bacteria during sludge ozonation by the combination of ATP assay with PMA-Miseq sequencing[J].Water, 2017, 9(3):166.