酰胺类产品具有较高的工业价值,其中,最有代表性的产品是丙烯酰胺,其单体和聚合物广泛应用于石油、水处理、造纸、纺织等行业。酰胺类产品的传统工业生产方法为化学合成,但反应条件苛刻、转化率低以及副产物产率高。随着腈水合酶的发现和研究,条件温和、选择性强的生物催化法[1]正逐步替代化学合成法,已成功应用于工业生产丙烯酰胺、烟酰胺和5-氰基戊酰胺等酰胺类产品[2-4]。
腈水合酶(nitrile hydratase,NHase,EC 4.2.1.84)是一种金属酶,可以将腈类底物水合,从而转化为相应的酰胺类产物。玫瑰色红球菌J1(节杆菌属)来源的该酶是人们首次发现并报道的腈水合酶[5]。目前生产腈水合酶的微生物有红球菌属(Rhodococccus)、假单胞菌属(Pseudomonas)和短杆菌属(Brevibacterium)等[6-8],且其异源表达已在大肠杆菌(Escherichia coli)、谷氨酸棒状杆菌(Corynebacterium glutamicum)、毕赤酵母(Pichia pastoris)等模式菌株中实现[9-11]。自1985年起,第1代菌株N-774就已经被用于丙烯腈到丙烯酰胺的生物转化[12],这也是生物技术制造商业化学品的第1个成功案例[13]。目前,三菱公司已经可以达到超过20万t的丙烯酰胺年产量[14]。
腈水合酶虽然已被用于工业生产,但由于腈的水合是放热反应,且大多数的腈水合酶在超过50 ℃时会迅速失活[15],两者之间的矛盾一定程度上限制了腈水合酶更广泛的应用。因此,获取兼具较高酶活力和良好热稳定性的腈水合酶具有重要意义。
此前,本实验室通过基因挖掘,异源表达了温泉热碱芽孢杆菌(Caldalkalibacillus thermarum TA2.A1)来源的新型耐热腈水合酶,该酶在65 ℃的半衰期为3 h,具有优异的热稳定性[16],但野生型的催化酶活力较低。近年来,随着生物信息学工具的快速发展,越来越多的研究者运用分子动力学模拟进行理性设计改造天然酶[17],确定适合突变的最佳氨基酸残基,提高酶活力以适应工业需要。PAVLOVA等[18]应用随机加速分子动力学来模拟产物从酶活性位点的释放过程,并确定突变的关键残基,得到了酶活力提高32倍的突变体。
本研究以提升酶的催化活性为目标,采用分子动力学模拟的方法,定位对酶结构自由度有重要影响的氨基酸残基,通过定点突变以期打破氢键,提高底物通道入口的构象自由度。并对得到的有效突变体进行进一步的酶学性质和分子动力学模拟分析,探究突变体的工业生产适用性。
1 材料与方法
1.1 实验材料
克隆宿主大肠杆菌DH5α、异源表达宿主大肠杆菌BL21(DE3)均由本实验室保藏。质粒pET-24a(+)-Cal.t NHase携带温泉热碱芽孢杆菌来源的腈水合酶,为本实验室前期构建并保藏。
酵母提取物、胰蛋白胨,OXOID公司;感受态细胞制备试剂盒、异丙基-β-D-硫代吡喃半乳糖(isopropyl-β-D-thiogalactopyranoside,IPTG)、卡那霉素,上海生工生物工程有限公司;DNA Marker、Primer STAR Max DNA聚合酶、Dpn I限制性内切酶、Bradford蛋白定量检测试剂盒,TaKaRa公司(北京);丙烯腈,国药集团(中国上海);丙烯酰胺和脱硫生物素,Sigma公司;引物由苏州金唯智合成,本文所用引物和序列见表1。
表1 本文所用引物及其序列
Table 1 Primers used in this study

引物名称引物序列(5′-3′)N47A-FGGTCTGTACGCCTTAGATGAGTTCCGCCATGCCATTGAACN47A-RCTCATCTAAGGCGTACAGACCTTGACCGGCGGTACCAATTAATAAACN47E-FGGTCTGTACGAATTAGATGAGTTCCGCCATGCCATTGAACN47E-RCTCATCTAATTCGTACAGACCTTGACCGGCGGTACCAATTAATAAACN47F-FGGTCTGTACTTTTTAGATGAGTTCCGCCATGCCATTGAACN47F-RCTCATCTAAAAAGTACAGACCTTGACCGGCGGTACCAATTAATAAACN47H-FGGTCTGTACCATTTAGATGAGTTCCGCCATGCCATTGAACN47H-RCTCATCTAAATGGTACAGACCTTGACCGGCGGTACCAATTAATAAACN47K-FGGTCTGTACAAATTAGATGAGTTCCGCCATGCCATTGAACN47K-RCTCATCTAATTTGTACAGACCTTGACCGGCGGTACCAATTAATAAACN47L-FGGTCTGTACCTGTTAGATGAGTTCCGCCATGCCATTGAACN47L-RCTCATCTAACAGGTACAGACCTTGACCGGCGGTACCAATTAATAAACN47Q-FGGTCTGTACCAGTTAGATGAGTTCCGCCATGCCATTGAACN47Q-RCTCATCTAACTGGTACAGACCTTGACCGGCGGTACCAATTAATAAACN181A-FCCGGATGCCGCCGCCCATGGCAAAGGCGAAAGCCCGCN181A-RCCATGGGCGGCGGCATCCGGAAACACATGGGCACCGTGN181E-FCCGGATGCCGAAGCCCATGGCAAAGGCGAAAGCCCGCN181E-RCCATGGGCTTCGGCATCCGGAAACACATGGGCACCGTGN181F-FCCGGATGCCTTTGCCCATGGCAAAGGCGAAAGCCCGCN181F-RCCATGGGCAAAGGCATCCGGAAACACATGGGCACCGTGN181H-FCCGGATGCCCATGCCCATGGCAAAGGCGAAAGCCCGCN181H-RCCATGGGCATGGGCATCCGGAAACACATGGGCACCGTGN181K-FCCGGATGCCAAAGCCCATGGCAAAGGCGAAAGCCCGCN181K-RCCATGGGCTTTGGCATCCGGAAACACATGGGCACCGTGN181L-FCCGGATGCCCTGGCCCATGGCAAAGGCGAAAGCCCGCN181L-RCCATGGGCCAGGGCATCCGGAAACACATGGGCACCGTGN181Q-FCCGGATGCCCAGGCCCATGGCAAAGGCGAAAGCCCGCN181Q-RCCATGGGCCTGGGCATCCGGAAACACATGGGCACCGTG
注:引物序列中,下划线字母代表突变位点
100 mmol/L KPB缓冲液:K2HPO4 13.95 g、KH2PO4 10.92 g,用KH2PO4调节pH为7.4,定容至1 L。蛋白纯化用binding buffer:K2HPO4 2.84 g、NaCl 16.36 g、KCl 0.37 g,用盐酸调节pH至7.4后定容到1 L。washing buffer:在上述binding buffer中加入2.5 mmol/L脱硫生物素。
1.2 实验方法
1.2.1 计算机理性设计方法
分子动力学模拟使用NAMD软件。Cal.t NHase结构的PDB文件参考本实验室先前的同源建模结果[19]。模拟过程采用VPT系统,选择1 000步共轭梯度法使能量最小化后,再进行100 ns的全约束并放开平衡,步长为2 ps,计算出β亚基上各个氨基酸的涨落状况。对得到的轨迹文件使用VMD软件进行可视化分析,比较在不同温度下(300和360 K)Cal.t NHase的β亚基的均方根波动值(root mean square fluctuation,RMSF)。底物通道计算利用CAVER analyst 2.0软件,底物通道探针半径设置为0.9 Å,底物通道入口计算利用CAVER内嵌的Residue Graph和Contour模块。氢键的计算由NAMD软件中的hydrogen bond模块完成。蛋白结构绘图软件为PyMOL。RMSF曲线由Origin 2018绘制获得。
1.2.2 突变体构建及表达
以质粒pET-24a(+)-Cal.t NHase为模板,进行全质粒PCR(引物见表1),用Dpn I限制性内切酶消化以去除模板质粒,之后转入感受态细胞,运用Sanger测序法确认突变位点核苷酸序列。
将构建好的突变体转入BL21(DE3),在含有50 mg/L卡那霉素的LB平板上涂布,过夜培养,挑取单菌落接种到5 mL含有50 mg/L卡那霉素的LB培养基的试管中,摇床条件设定为37 ℃,200 r/min,培养8~10 h。试管种子液转接到含有50 mg/L卡那霉素的2YT培养基中,种子液接种量为1%,37 ℃、200 r/min,培养至菌体浓度OD600值为0.6~0.8时,加入终浓度为0.4 mmol/L的IPTG诱导蛋白表达,并将温度设置为30 ℃,培养16~18 h。
1.2.3 酶的分离纯化及浓度检测
诱导表达后的菌液用超高速离心机以10 000 r/min离心5 min,收集菌体,用binding buffer重悬菌体,按比例浓缩5倍,混匀后放置于冰盒中进行超声波破碎。随后,4 ℃,12 000 r/min 超高速离心30 min,取上清液弃沉淀,后用0.22 μm滤膜过滤,获得粗酶液,使用AKTA蛋白纯化仪通过strep亲和层析进行后续纯化。首先,先后用5倍体积的去离子水和binding buffer缓冲液以1 mL/min流速清洗并平衡柱子;参数稳定后上样,样品全部进入管道后换用binding buffer冲洗管道,直至紫外吸收值降低为0,再换用washing buffer洗脱,收集紫外吸收值>200 mAU的蛋白。用SDS-PAGE方法分析鉴定纯化后的蛋白表达情况,用Bradford法测定蛋白浓度,并将蛋白统一稀释到0.05 mg/mL以便后续测定酶活力。
1.2.4 酶活力检测
取10 μL纯化后的0.05 mg/mL的蛋白先于25 ℃金属浴下保温5 min,加入490 μL 200 mmol/L的丙烯腈底物,25 ℃下反应10 min,最后加入500 μL乙腈终止反应,反应终体积为1 mL。反应后样品经0.22 μm有机滤膜过滤后,用高效液相色谱测定产物生成量,检测器为紫外检测(检测波长为210 nm),色谱柱为C18柱(柱温为40 ℃),流动相为乙腈和水混合溶液(乙腈与水的体积比为1∶2),流速为0.6 mL/min,进样量为10 μL,进样时间为10 min。定义酶活力单位(U):25 ℃下每分钟催化丙烯腈生成1 μmol丙烯酰胺所需的酶量;定义比酶活(U/mg):1 mg纯酶具有的酶活力单位数。
1.2.5 热稳定性研究
设置金属浴温度为65 ℃,分别保温0、2、3、5 h后取样,测定剩余酶活力,以处理0 h的样品(未经热处理的酶)为对照,定义其酶活力为100%,依次计算其余样品相对酶活力,分析65 ℃下的纯酶热稳定性。
2 结果与分析
2.1 突变位点的选择
近年的研究发现,腈水合酶的底物通道主要由β亚基中的氨基酸残基构成,且底物通道对于该酶的催化性能具有决定性作用[20]。此外,研究理论指出,酶分子中的柔性loop区域可能对于酶的催化活性具有一定的影响[21]。根据上述报道,对位于腈水合酶底物通道入口处的loop结构进行研究(图1-a)。

a-底物通道结构及通道入口附近关键区域 (红色标出为R1区域,橙色标出为R2区域); b-底物通道入口关键氨基酸βAsn47和βAsn181之间的氢键
图1 Cal.t NHase结构及底物通道模拟分析
Fig.1 Structure modelling and key residues adjacent to substrate access tunnel of Cal.t NHase
RMSF是指一段时间内,原子相对于参考构象的结构变化,反映了原子的自由度[22]。当原子位于蛋白质上时,RMSF值可以反映蛋白质中各个氨基酸的柔性强度:RMSF值越大,柔性越大[23-24]。通过100 ns分子动力学模拟,对野生型Cal.t NHase上β亚基中各个氨基酸在温度300和360 K下的RMSF值进行计算。图2显示,其中,β亚基上的区域R1(βGly42-βLeu48)、R2(βAsn181-βSer188) 在360 K时较300 K时发生明显的柔性波动,为β亚基上的柔性区域。根据图1,Cal.t NHase底物通道入口loop区域由β亚基上的Gly42-Leu48七个氨基酸残基构成,与区域R1位置吻合。

图2 野生型β亚基在300和360 K下的RMSF
Fig.2 RMSF of wild type β subunit at 300 and 360 K
着重寻找柔性区域R1和R2上的特殊作用力,发现位于β亚基47位的天冬酰胺残基和181位的天冬酰胺残基在分子动力学模拟过程中会形成氢键(图1-b),且持续时间较长,推测这对氢键限制了区域R1的柔性。因此,本文拟将通过定点突变打断氢键,增加区域R1自由度,提高底物通道入口的构象可变性,从而提高腈水合酶酶活力。
2.2 Cal.t NHase的代表性氨基酸突变
为减小突变体库构建工作量,同时尽量提高小型突变体库中各氨基酸类型的全面性,选择7种具有代表性的氨基酸残基(极性、非极性、带电、不带电)进行突变,将β47位和β181位上的天冬酰胺分别突变为以下氨基酸(A、E、F、H、K、L、Q),构建共14个突变体。在OD600=0.5菌体浓度下,测定的各突变体相对酶活力如图3所示。
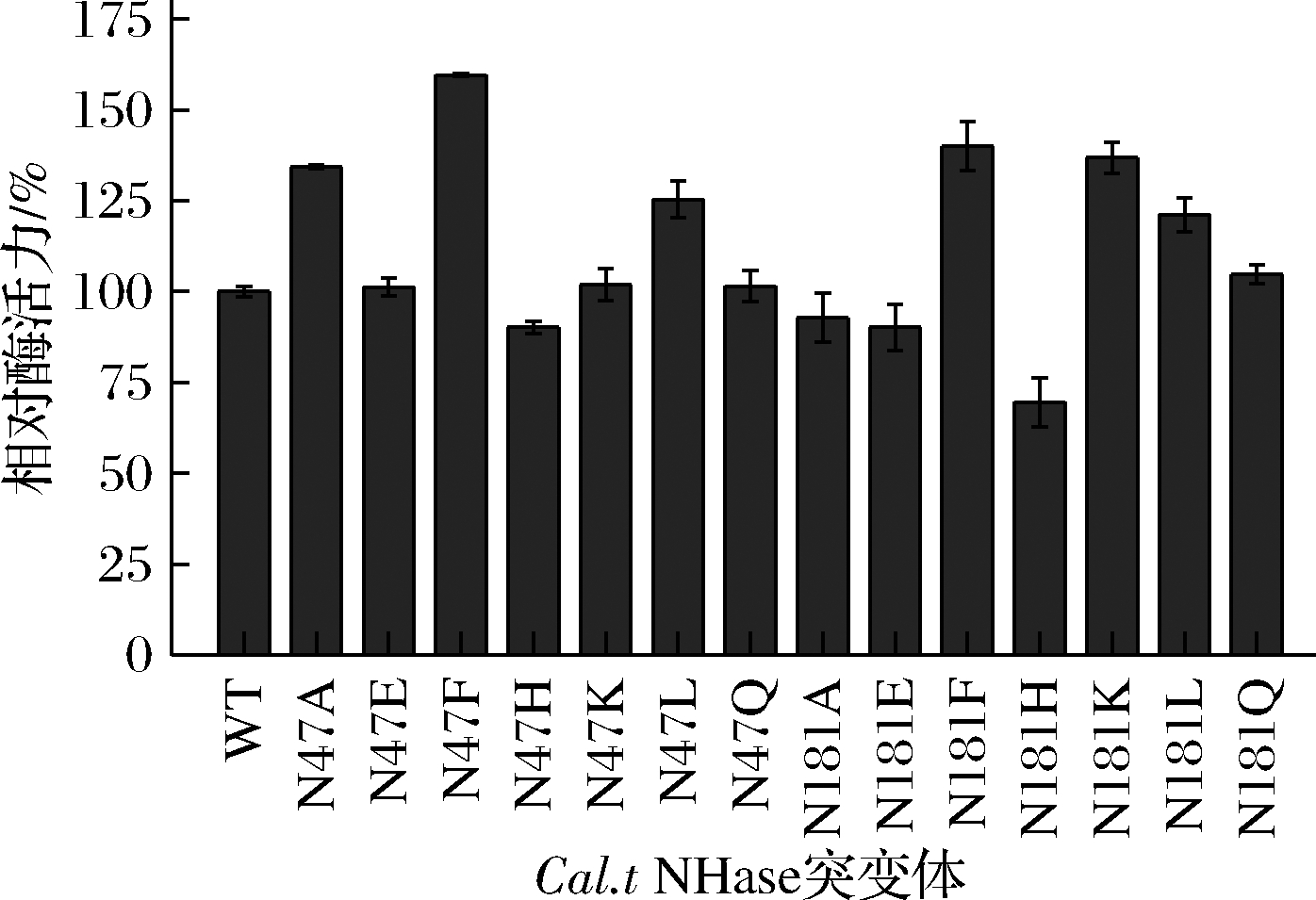
图3 Cal.t NHase突变体相对酶活力
Fig.3 Relative activity of Cal.t NHase mutants
选取其中突变为丙氨酸的2个突变体(βN47A、βN181A)和粗酶活力显著提高的4个突变体(βN47F、βN47L、βN181F、βN181K)进一步纯化(纯化后SDS-PAGE如图4所示)。
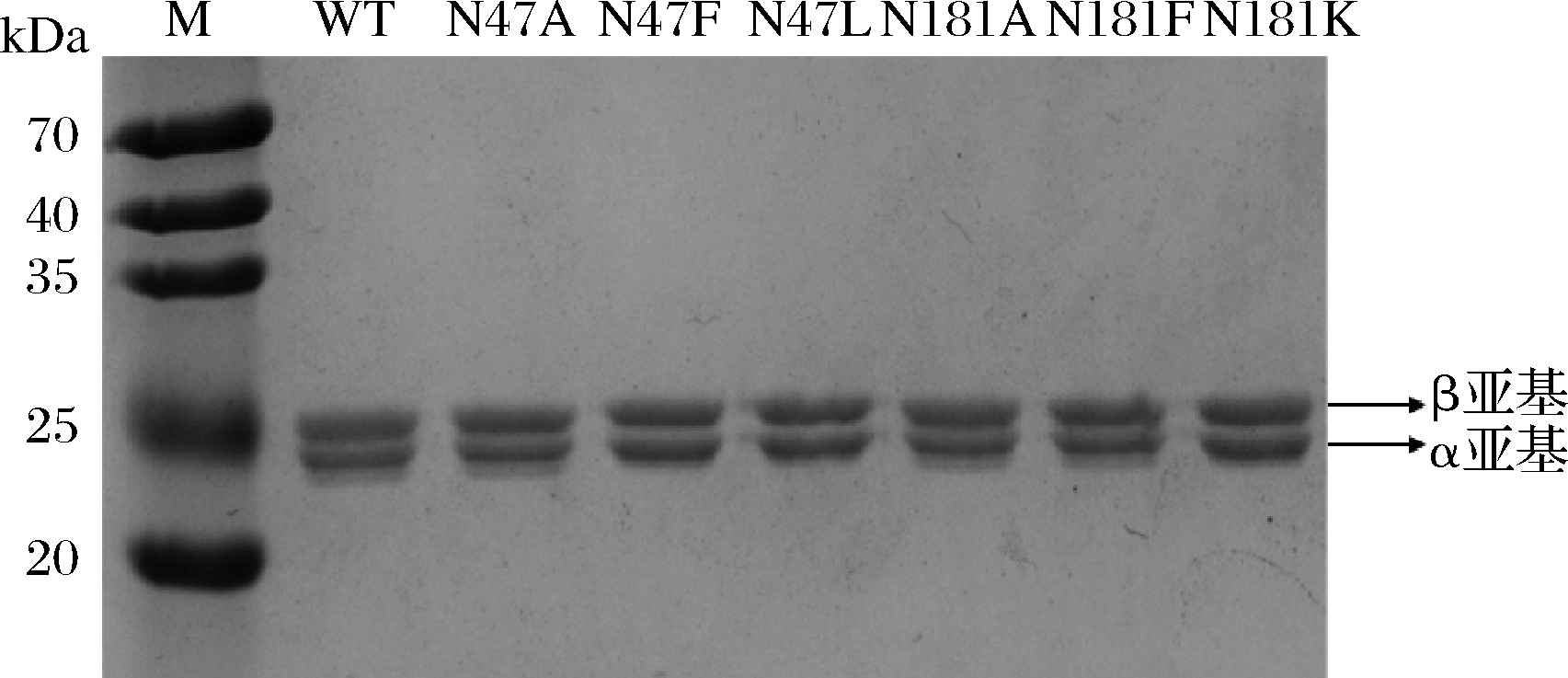
图4 Cal.t NHase SDS-PAGE图
Fig.4 SDS-PAGE of Cal.t NHase
测定的纯酶酶活力如图5所示。其中,得到的最优突变体为βN47F,比酶活力为592.92 U/mg,相对野生型的比酶活力为165.30 U/mg,提高了2.57倍。
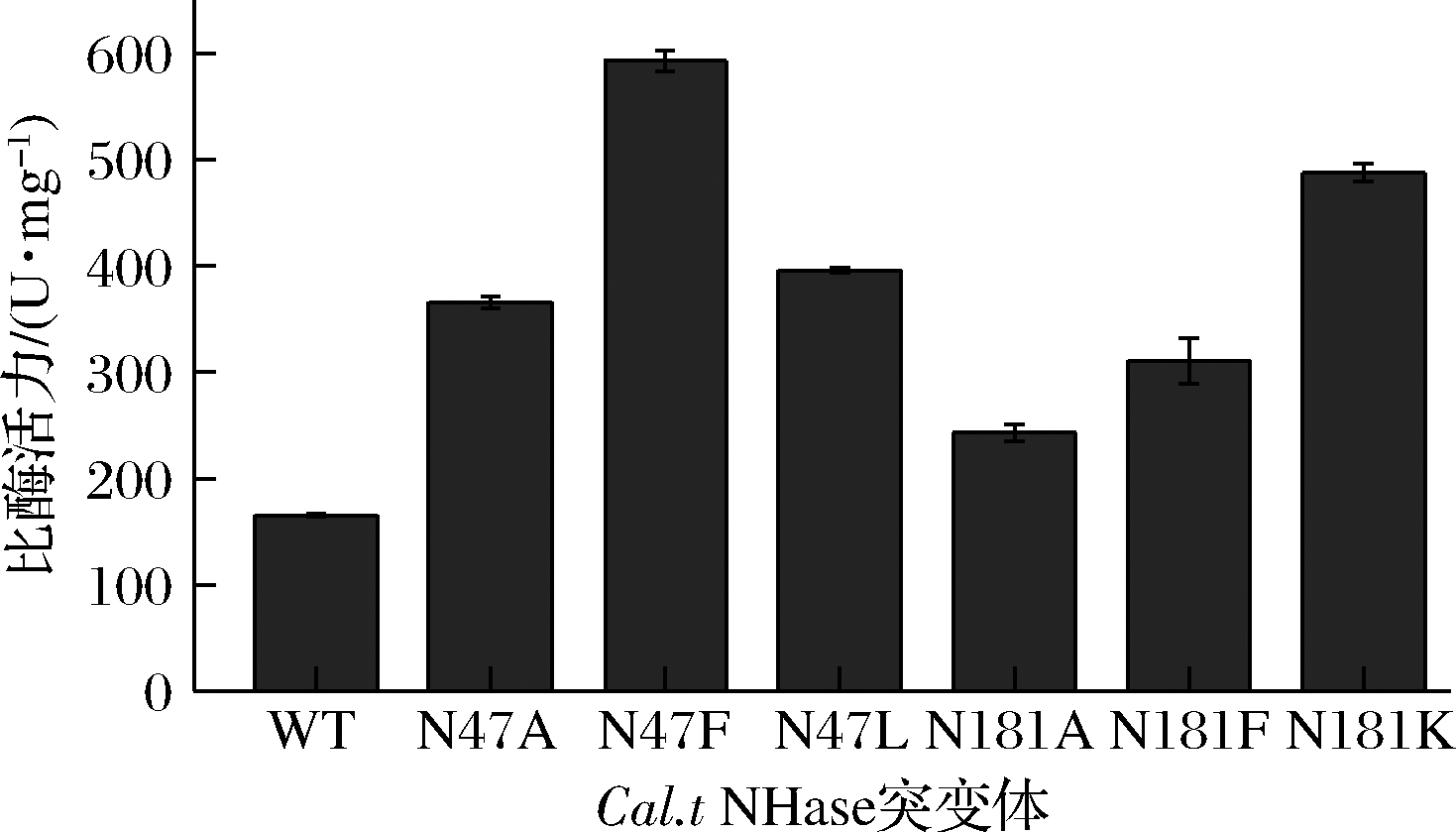
图5 Cal.t NHase突变体比酶活力
Fig.5 Specific activity of Cal.t NHase mutants
2.3 βN47F突变体的热稳定性
Cal.t NHase野生型及βN47F突变体的热稳定性如图6所示,野生型65 ℃下的半衰期为3 h,突变体βN47F 65 ℃处理3 h后仍残余56%酶活力。而嗜热假诺卡氏菌(Pseudonocardia thermophila JCM 3095)来源的腈水合酶(Pt NHase)65 ℃处理仅30 min,即残余酶活力不到10%[16]。突变体与野生型相比热稳定性有一定的提高,尤其与其他来源的腈水合酶相比,具有较明显的优势,证明βN47F在提高酶活力的同时保持了良好的热稳定性。

图6 Cal.t NHase βN47F热稳定性
Fig.6 Thermal stability of Cal.t NHase βN47F
2.4 突变体催化性能提高机制解析
通过蛋白质理性设计,获取了酶活力大幅提高的优势突变体βN47F。为了解析该突变体催化性能提高的机制,该研究首先通过trRosetta在线建模工具[25],以来源于Bacillus sp.RAPc8 (PDB ID:2DPP) 和Bacillus smithii SC-J05-1 (PDB ID:1V29) 的腈水合酶晶体结构为模板,构建了βN47F的三维模拟结构。由于β亚基上47位的天冬酰胺已突变为非极性残基苯丙氨酸,相应位点的残基侧链无法与β亚基上181位的天冬酰胺形成氢键,推测该突变破坏了原有的氢键相互作用,从而解除了Cal.t NHase底物通道入口loop区域的运动限制。在此基础上,通过对优势突变体βN47F进行100 ns的分子动力学模拟发现(图7),突变体底物通道入口loop区域的RMSF值较野生型Cal.t NHase有明显提高,即底物通道入口区域的柔性得到了提高。
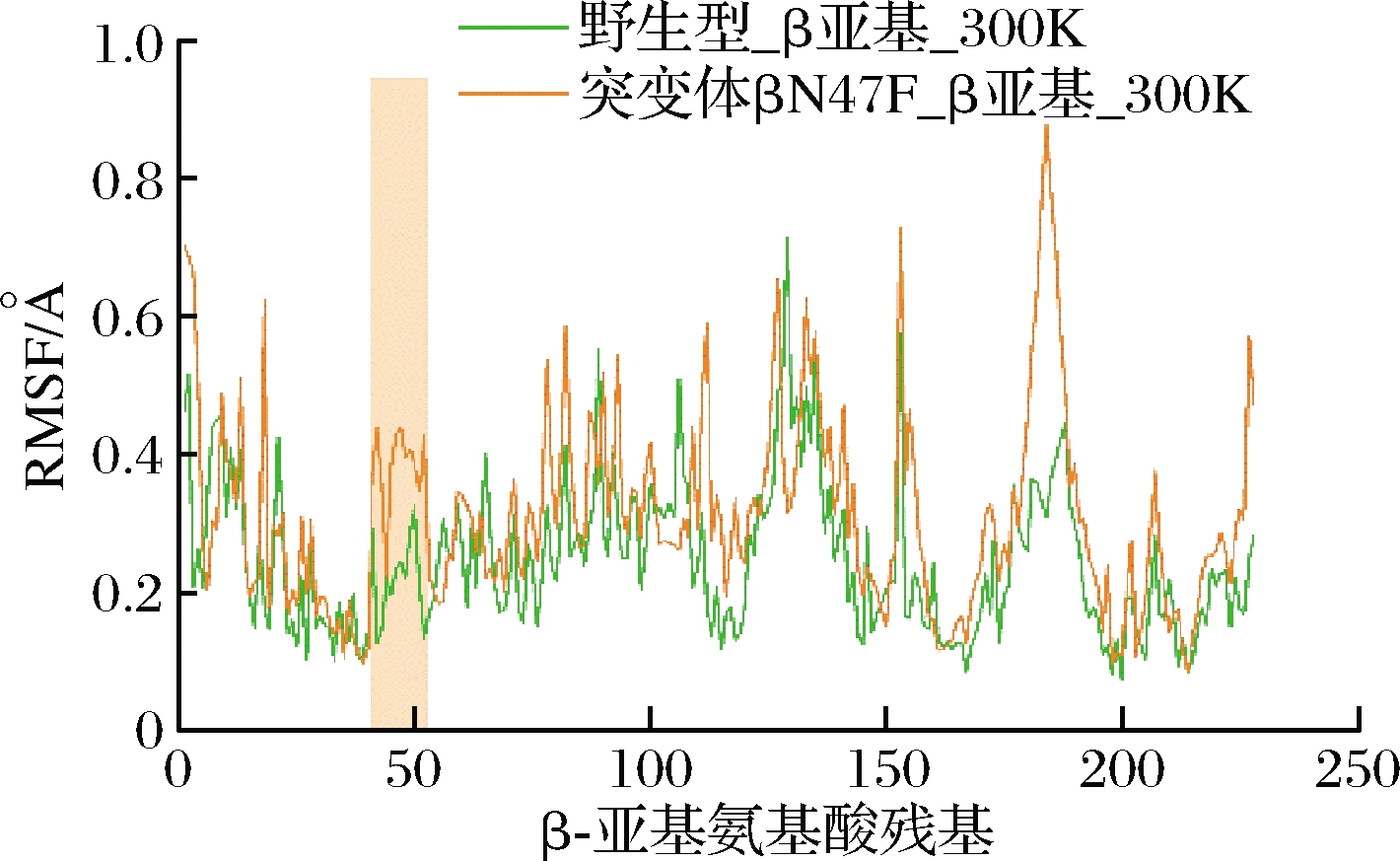
图7 300 K下野生型和突变体βN47F的RMSF值
Fig.7 RMSF values of wild-type and mutant βN47F at 300 K
利用PyMOL软件对所构建优势突变体βN47F进行分析(图8),发现突变体47位苯丙氨酸的侧链并未与其他任何氨基酸发生氢键等相互作用,表明野生型β亚基47位天冬酰胺侧链与181位天冬酰胺侧链的氢键已被成功打破。
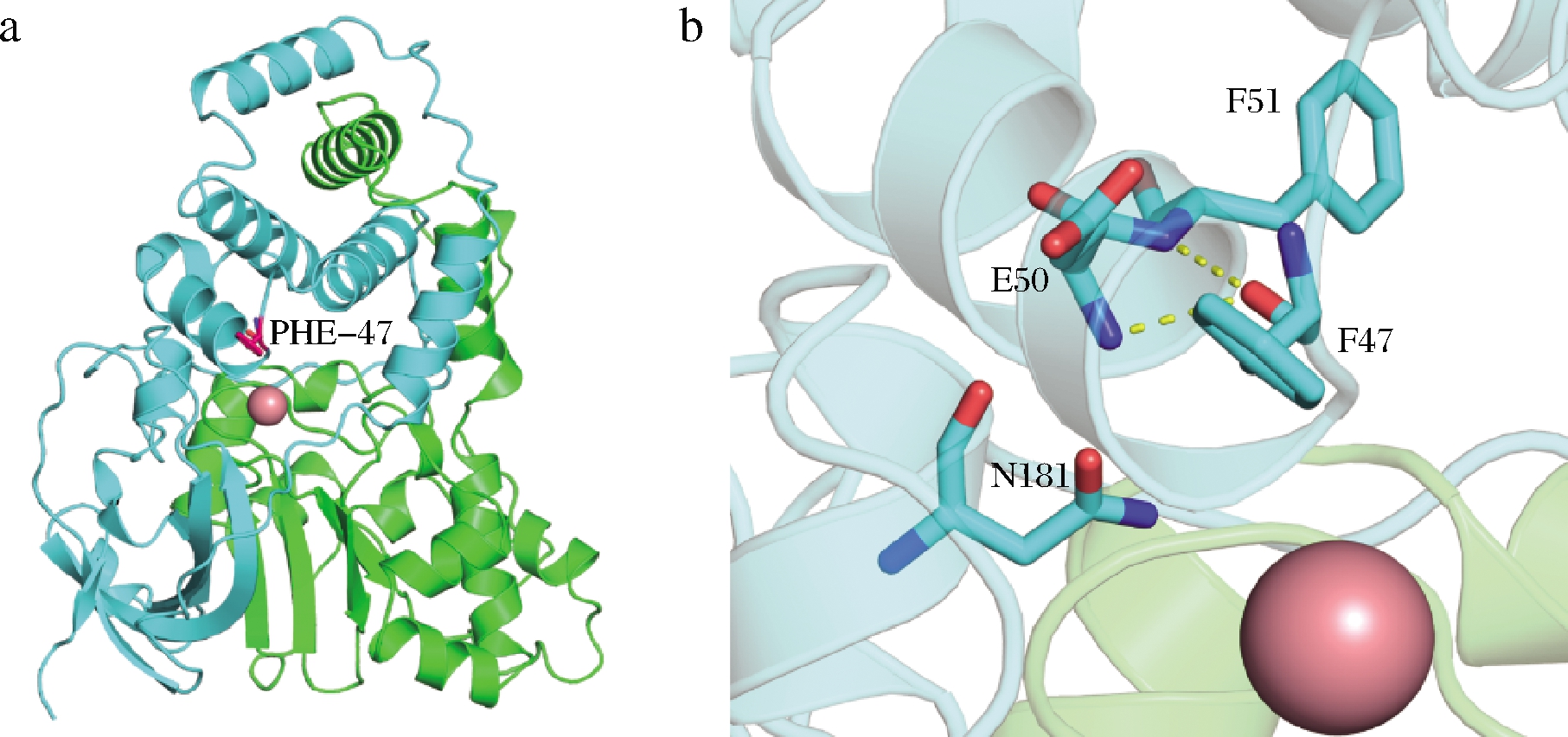
a-突变体βN47F的模拟结构; b-βPhe47与周围氨基酸残基的相互作用
图8 突变体βN47F模型图
Fig.8 Model diagram of mutant βN47F
利用CAVER软件对野生型Cal.t NHase及其突变体βN47F进行底物通道入口构象的分析(图9),发现突变后突变体βN47F的底物通道入口几何构象较野生型Cal.t NHase更宽大,表明突变后底物通道入口区域柔性的增强提升了该区域的构象可变性,拓宽了底物通道入口,更利于底物与腈水合酶的结合过程。
a-野生型底物通道入口构象;b-突变体βN47F底物通道入口构象
图9 野生型和突变体βN47F底物通道入口构象
Fig.9 Conformation of the substrate channel entrance of wild-type and mutant βN47F
3 结论
本文采用分子动力学模拟的方法,选取了位于腈水合酶底物通道入口附近的2个位点βN47、βN181进行突变,得到了酶活力显著提高的突变体βN47F。突变体比酶活力提高到592.92 U/mg,与野生型相比,提高了2.57倍。在提高酶活力的同时,突变体在65 ℃下的半衰期为3 h,保持了Cal.t NHase热稳定性良好的优势。对βN47F突变体催化性能提高机制进行解析,其三维结构显示β亚基47位上的天冬酰胺和β亚基181位的天冬酰胺之间形成的氢键相互作用力遭到破坏;分子动力学模拟和Caver软件分析显示突变体底物通道入口的柔性和构象可变性都得到了提高。兼具较高酶活和良好热稳定性的突变体的成功构建,增强了研究者对于理性设计方法改造蛋白提高酶活力的信心,进一步推动后续Cal.t NHase的工业化生产应用。
[1] WOHLGEMUTH R.Biocatalysis—Key to sustainable industrial chemistry[J].Current Opinion in Biotechnology, 2010, 21(6):713-724.
[2] JIAO S, LI F L, YU H M, et al.Advances in acrylamide bioproduction catalyzed with Rhodococcus cells harboring nitrile hydratase[J].Applied Microbiology and Biotechnology, 2020, 104(3):1 001-1 012.
[3] LI B F, SU J H, TAO J H.Enzyme and process development for production of nicotinamide[J].Organic Process Research & Development, 2010, 15(1):291-293.
[4] CHEN J, HUANG Y T, DENG S G, et al.Biotransformation of adiponitrile to 5-cyanovaleramide by Pseudomonas sp. SY031 resting cells[J].Advanced Materials Research, 2013, 791-793:204-207.
[5] ASANO Y, TANI Y, YAMADA H.A new enzyme “nitrile hydratase” which degrades acetonitrile in combination with amidase[J].Agricultural and Biological Chemistry, 1980, 44(9):2 251-2 252.
[6] PRASAD S, RAJ J, BHALLA T C.Optimisation of culture conditions for hyper production of nitrile hydratase of Rhodococcus rhodochrous PA-34[J].Indian Journal of Microbiology, 2004, 44(4):251-256.
[7] YAMADA H, RYUNO K, NAGASAWA T, et al.Optimum culture conditions for production by Pseudomonas chlororaphis B23 of nitrile hydratase[J].Agricultural and Biological Chemistry, 1986, 50(11):2 859-2 865.
[8] TAKASHIMA Y, KAWABE T, MITSUDA S.Factors affecting the production of nitrile hydratase by thermophilic Bacillus smithii SC-J05-1[J].Journal of Bioscience and Bioengineering, 2000, 89(3):282-284.
[9] WANG Z, LIU Z M, CUI W J, et al.Establishment of bioprocess for synthesis of nicotinamide by recombinant Escherichia coli expressing high-molecular-mass nitrile hydratase[J].Applied Biochemistry and Biotechnology, 2017, 182(4):1 458-1 466.
[10] KANG M S, HAN S S, KIM M Y, et al.High-level expression in Corynebacterium glutamicum of nitrile hydratase from Rhodococcus rhodochrous for acrylamide production[J].Applied Microbiology and Biotechnology, 2014, 98(10):4 379-4 387.
[11] SHI Y, YU H M, SUN X D, et al.Cloning of the nitrile hydratase gene from Nocardia sp. in Escherichia coli and Pichia pastoris and its functional expression using site-directed mutagenesis[J].Enzyme and Microbial Technology, 2004, 35(6-7):557-562.
[12] WATANABE I, SATOH Y, ENOMOTO K.Screening, isolation and taxonomical properties of microorganisms having acrylonitrile-hydrating activity[J].Agricultural and Biological Chemistry, 1987, 51(12):3 193-3 199.
[13] YAMADA H, KOBAYASHI M.Nitrile hydratase and its application to industrial production of acrylamide[J].Bioscience, Biotechnology, and Biochemistry, 1996, 60(9):1 391-1 400.
[14] SAKASHITA T, HASHIMOTO Y, OINUMA K I, et al.Transcriptional regulation of the nitrile hydratase gene cluster in Pseudomonas chlororaphis B23[J].Journal of Bacteriology, 2008, 190(12):4 210-4 217.
[15] COWAN D, CRAMP R, PEREIRA R, et al.Biochemistry and biotechnology of mesophilic and thermophilic nitrile metabolizing enzymes[J].Extremophiles, 1998, 2(3):207-216.
[16] 张赛兰, 李婷, 程中一, 等.新型耐热腈水合酶的异源表达及其催化工艺研究[J].食品与发酵工业, 2020, 46(14):108-113.
ZHANG S L, LI T, CHENG Z Y, et al.Heterologous expression of a novel thermostable nitrile hydratase and its catalytic process[J].Food and Fermentation Industries, 2020, 46(14):108-113.
[17] LUTZ S.Beyond directed evolution—Semi-rational protein engineering and design[J].Current Opinion in Biotechnology, 2010, 21(6):734-743.
[18] PAVLOVA M, KLVANA M, PROKOP Z, et al.Redesigning dehalogenase access tunnels as a strategy for degrading an anthropogenic substrate[J].Nature Chemical Biology, 2009, 5(10):727-733.
[19] 张赛兰. 新型耐热腈水合酶的异源表达及酶学性质研究[D].无锡:江南大学, 2020.
ZHANG S L.Heterologous expression and enzymatic property study of a novel thermostable nitrile hydratase[D].Wuxi:Jiangnan University, 2020.
[20] CHENG Z Y, CUI W J, LIU Z M, et al.A switch in a substrate tunnel for directing regioselectivity of nitrile hydratases towards a,w-dinitriles[J].Catalysis Science & Technology, 2016, 6(5):1 292-1 296.
[21] NESTL B M, HAUER B.Engineering of flexible loops in enzymes[J].ACS Catalysis, 2014, 4(9):3 201-3 211.
[22] YU H R, HUANG H.Engineering proteins for thermostability through rigidifying flexible sites[J].Biotechnology Advances, 2014, 32(2):308-315.
[23] TIAN J, WANG P, GAO S, et al.Enhanced thermostability of methyl parathion hydrolase from Ochrobactrum sp.M231 by rational engineering of a glycine to proline mutation[J].The FEBS Journal, 2010, 277(23):4 901-4 908.
[24] LIU J, YU H M, SHEN Z Y.Insights into thermal stability of thermophilic nitrile hydratases by molecular dynamics simulation[J].Journal of Molecular Graphics and Modelling, 2008, 27(4):529-535.
[25] YANG J Y, ANISHCHENKO I, PARK H, et al.Improved protein structure prediction using predicted interresidue orientations[J].Proceedings of the National Academy of Sciences, 2020, 117(3):1 496-1 503.